27 Análisis de supervivencia
27.1 Resumen
El análisis de supervivencia se centra en la descripción, para un individuo o grupo de individuos determinado, de un acontecimiento puntual denominado evento (aparición de una enfermedad, curación de una enfermedad, muerte, recaída tras la respuesta al tratamiento…) que se produce tras un periodo de tiempo denominado tiempo del evento (o tiempo de seguimiento (tiempo de seguimiento en los estudios basados en cohortes/poblaciones) durante el cual se observa a los individuos. Para determinar el tiempo de fracaso, es necesario definir un tiempo de origen (que puede ser la fecha de inclusión, la fecha de diagnóstico…).
El objetivo de la inferencia para el análisis de supervivencia es entonces el tiempo entre un origen y un evento. En la investigación médica actual, se utiliza ampliamente en los estudios clínicos para evaluar el efecto de un tratamiento, por ejemplo, o en la epidemiología del cáncer para evaluar una gran variedad de medidas de supervivencia del cáncer.
Suele expresarse mediante la probabilidad de supervivencia (survival probability), que es la probabilidad de que el suceso de interés no haya ocurrido en una duración t.
Censura: La censura se produce cuando al final del seguimiento, algunos de los individuos no han tenido el evento de interés, y por lo tanto su verdadero tiempo hasta el evento es desconocido. Aquí nos centraremos principalmente en la censura derecha, pero para más detalles sobre la censura y el análisis de supervivencia en general, puedes consultar las referencias.
27.2 Preparación
Cargar paquetes
Para realizar análisis de supervivencia en R, uno de los paquetes más utilizados es el de survival. Primero lo instalamos y luego lo cargamos, así como los demás paquetes que se utilizarán en esta sección:
En este manual destacamos p_load() de pacman, que instala el paquete si es necesario y lo carga para su uso. También puedes cargar los paquetes instalados con library() de R base. Consulta la página sobre fundamentos de R para obtener más información sobre los paquetes de R.
Esta página explora los análisis de supervivencia sobre el archivo linelist utilizado en la mayoría de las páginas anteriores y sobre el que aplicamos algunos cambios para tener unos datos de supervivencia adecuados.
Importar los datos
Importamos los datos de casos de una epidemia de ébola simulada. Si quieres seguir el proceso, clica para descargar linelist “limpio” (como archivo .rds). Importa los datos con la función import() del paquete rio (maneja muchos tipos de archivos como .xlsx, .csv, .rds - Mira la página de importación y exportación para más detalles).
# importar linelist
linelist_case_data <- rio::import("linelist_cleaned.rds")Gestión y transformación de datos
En resumen, los datos de supervivencia pueden describirse con las tres características siguientes:
- la variable dependiente o respuesta es el tiempo de espera hasta la ocurrencia de un evento bien definido,
- observaciones censuradas, en el sentido de que para algunas unidades el evento de interés no ha ocurrido en el momento en que se analizan los datos, y
- existen predictores o variables explicativas cuyo efecto sobre el tiempo de espera queremos evaluar o controlar.
Así, crearemos las diferentes variables necesarias para respetar esa estructura y ejecutaremos el análisis de supervivencia.
Definiremos:
- un nuevo dataframe
linelist_survpara este análisis - nuestro evento de interés como “death” (por lo tanto, nuestra probabilidad de supervivencia será la probabilidad de estar vivo después de un cierto tiempo después del momento de origen),
- el tiempo de seguimiento (
futime) como el tiempo transcurrido entre el momento del inicio y el momento del desenlace en días, - pacientes censurados son aquellos que se recuperaron o para los que no se conoce el resultado final, es decir, no se observó el evento “muerte” (
evento=0).
PRECAUCIÓN: Dado que en un estudio de cohortes real, la información sobre el momento de origen y el final del seguimiento se conoce dado que los individuos son observados, eliminaremos las observaciones en las que se desconozca la fecha de inicio o la fecha de desenlace. También se eliminarán los casos en los que la fecha de inicio sea posterior a la fecha de desenlace, ya que se consideran erróneos.
CONSEJO: Dado que el filtrado a mayor (>) o menor (<) de una fecha puede eliminar las filas con valores faltantes, la aplicación del filtro en las fechas incorrectas también eliminará las filas con fechas faltantes.
A continuación, utilizamos case_when() para crear una columna age_cat_small en la que sólo hay 3 categorías de edad.
#crea linelist_surv a partir de linelist_case_data
linelist_surv <- linelist_case_data %>%
dplyr::filter(
# eliminar observaciones con fechas de inicio o de resultado erróneas o ausentes
date_outcome > date_onset) %>%
dplyr::mutate(
# crear la var evento que es 1 si el paciente falleció y 0 si fue correctamente censurado
event = ifelse(is.na(outcome) | outcome == "Recover", 0, 1),
# crear la var de tiempo de seguimiento en días
futime = as.double(date_outcome - date_onset),
# crear una nueva variable de categoría de edad con sólo 3 estratos
age_cat_small = dplyr::case_when(
age_years < 5 ~ "0-4",
age_years >= 5 & age_years < 20 ~ "5-19",
age_years >= 20 ~ "20+"),
# el paso anterior creó la variable age_cat_small como carácter.
# ahora se convierte en factor y se especifican los niveles.
# Nótese que los valores NA siguen siendo NA y no se ponen en un nivel "desconocido" por ejemplo,
# ya que en los siguientes análisis tienen que ser eliminados.
age_cat_small = fct_relevel(age_cat_small, "0-4", "5-19", "20+")
)CONSEJO: Podemos verificar las nuevas columnas que hemos creado haciendo un resumen sobre futime y una tabulación cruzada entre event y outcome a partir del cual se ha creado. Además de esta verificación, es un buen hábito comunicar la mediana del tiempo de seguimiento al interpretar los resultados del análisis de supervivencia.
summary(linelist_surv$futime)## Min. 1st Qu. Median Mean 3rd Qu. Max.
## 1.00 6.00 10.00 11.98 16.00 64.00
# cruzar la nueva variable de evento y la de resultado a partir de la que se creó
# para asegurarse de que el código ha hecho lo que debía
linelist_surv %>%
tabyl(outcome, event)## outcome 0 1
## Death 0 1952
## Recover 1547 0
## <NA> 1040 0Ahora cruzamos la nueva var de age_cat_small y la antigua columna age_cat para asegurarnos de que las asignaciones son correctas
linelist_surv %>%
tabyl(age_cat_small, age_cat)## age_cat_small 0-4 5-9 10-14 15-19 20-29 30-49 50-69 70+ NA_
## 0-4 834 0 0 0 0 0 0 0 0
## 5-19 0 852 717 575 0 0 0 0 0
## 20+ 0 0 0 0 862 554 69 5 0
## <NA> 0 0 0 0 0 0 0 0 71Ahora revisamos las 10 primeras observaciones de los datos de linelist_surv mirando las variables específicas (incluyendo las de nueva creación).
linelist_surv %>%
select(case_id, age_cat_small, date_onset, date_outcome, outcome, event, futime) %>%
head(10)## case_id age_cat_small date_onset date_outcome outcome event futime
## 1 8689b7 0-4 2014-05-13 2014-05-18 Recover 0 5
## 2 11f8ea 20+ 2014-05-16 2014-05-30 Recover 0 14
## 3 893f25 0-4 2014-05-21 2014-05-29 Recover 0 8
## 4 be99c8 5-19 2014-05-22 2014-05-24 Recover 0 2
## 5 07e3e8 5-19 2014-05-27 2014-06-01 Recover 0 5
## 6 369449 0-4 2014-06-02 2014-06-07 Death 1 5
## 7 f393b4 20+ 2014-06-05 2014-06-18 Recover 0 13
## 8 1389ca 20+ 2014-06-05 2014-06-09 Death 1 4
## 9 2978ac 5-19 2014-06-06 2014-06-15 Death 1 9
## 10 fc15ef 5-19 2014-06-16 2014-07-09 Recover 0 23También podemos cruzar las columnas age_cat_small y gender para tener más detalles sobre la distribución de esta nueva columna por género. Utilizamos tabyl() y las funciones de adorno de janitor como se describe en la página de tablas descriptivas.
linelist_surv %>%
tabyl(gender, age_cat_small, show_na = F) %>%
adorn_totals(where = "both") %>%
adorn_percentages() %>%
adorn_pct_formatting() %>%
adorn_ns(position = "front")## gender 0-4 5-19 20+ Total
## f 482 (22.4%) 1184 (54.9%) 490 (22.7%) 2156 (100.0%)
## m 325 (15.0%) 880 (40.6%) 960 (44.3%) 2165 (100.0%)
## Total 807 (18.7%) 2064 (47.8%) 1450 (33.6%) 4321 (100.0%)27.3 Fundamentos del análisis de supervivencia
Construir un objeto de tipo surv-type
Primero utilizaremos Surv() de survival para construir un objeto de supervivencia a partir de las columnas de tiempo de seguimiento y evento.
El resultado de este paso es producir un objeto de tipo Surv que condensa la información de tiempo y si fue observado el evento de interés (muerte). Este objeto se utilizará en última instancia en el lado derecho de las fórmulas posteriores del modelo (véase la documentación).
# Utilizar la sintaxis Suv() para datos correctamente censurados
survobj <- Surv(time = linelist_surv$futime,
event = linelist_surv$event)Puedes revisar las primeras 10 filas de los datos de linelist_surv, viendo sólo algunas columnas importantes.
## case_id date_onset date_outcome futime outcome event
## 1 8689b7 2014-05-13 2014-05-18 5 Recover 0
## 2 11f8ea 2014-05-16 2014-05-30 14 Recover 0
## 3 893f25 2014-05-21 2014-05-29 8 Recover 0
## 4 be99c8 2014-05-22 2014-05-24 2 Recover 0
## 5 07e3e8 2014-05-27 2014-06-01 5 Recover 0
## 6 369449 2014-06-02 2014-06-07 5 Death 1
## 7 f393b4 2014-06-05 2014-06-18 13 Recover 0
## 8 1389ca 2014-06-05 2014-06-09 4 Death 1
## 9 2978ac 2014-06-06 2014-06-15 9 Death 1
## 10 fc15ef 2014-06-16 2014-07-09 23 Recover 0Y aquí están los primeros 10 elementos de survobj. Se imprime esencialmente como un vector de tiempo de seguimiento, con “+” a la derecha para representar si una observación fue censurada. Mira cómo los números se alinean arriba y abajo.
#imprimir los 50 primeros elementos del vector para ver cómo se presenta
head(survobj, 10)## [1] 5+ 14+ 8+ 2+ 5+ 5 13+ 4 9 23+Realización de los primeros análisis
A continuación, iniciamos nuestro análisis utilizando la función survfit() para producir un objeto survfit, que se ajusta a los cálculos por defecto para las estimaciones de Kaplan Meier (KM) de la curva de supervivencia global (marginal), que son de hecho una función escalonada con saltos en los tiempos de los eventos observados. El objeto survfit final contiene una o más curvas de supervivencia y se crea utilizando el objeto Surv como variable de respuesta en la fórmula del modelo.
NOTA: La estimación de Kaplan-Meier es una estimación no paramétrica de máxima verosimilitud (MLE) de la función de supervivencia. (ver recursos para más información).
El resumen de este objeto survfit dará lo que se llama una tabla de vida. Para cada paso de tiempo de seguimiento (time) en el que ocurrió un evento (en orden ascendente):
- el número de personas que estaban en riesgo de desarrollar el evento (personas que aún no tenían el evento ni estaban censuradas: (
n.risk) - los que sí desarrollaron el evento (
n.event) - y de lo anterior: la probabilidad de no desarrollar el evento (probabilidad de no morir, o de sobrevivir más allá de ese tiempo específico)
- por último, se obtienen y muestran el error estándar y el intervalo de confianza de esa probabilidad
Ajustamos las estimaciones de KM mediante la fórmula en la que el objeto Surv “survobj” anterior es la variable de respuesta. “~ 1” precisa que ejecutamos el modelo para la supervivencia global.
# ajustar las estimaciones de KM utilizando una fórmula donde el objeto Surv "survobj" es la variable de respuesta.
# "~ 1" significa que ejecutamos el modelo para la supervivencia global
linelistsurv_fit <- survival::survfit(survobj ~ 1)
#imprimir su resumen para más detalles
summary(linelistsurv_fit)## Call: survfit(formula = survobj ~ 1)
##
## time n.risk n.event survival std.err lower 95% CI upper 95% CI
## 1 4539 30 0.993 0.00120 0.991 0.996
## 2 4500 69 0.978 0.00217 0.974 0.982
## 3 4394 149 0.945 0.00340 0.938 0.952
## 4 4176 194 0.901 0.00447 0.892 0.910
## 5 3899 214 0.852 0.00535 0.841 0.862
## 6 3592 210 0.802 0.00604 0.790 0.814
## 7 3223 179 0.757 0.00656 0.745 0.770
## 8 2899 167 0.714 0.00700 0.700 0.728
## 9 2593 145 0.674 0.00735 0.660 0.688
## 10 2311 109 0.642 0.00761 0.627 0.657
## 11 2081 119 0.605 0.00788 0.590 0.621
## 12 1843 89 0.576 0.00809 0.560 0.592
## 13 1608 55 0.556 0.00823 0.540 0.573
## 14 1448 43 0.540 0.00837 0.524 0.556
## 15 1296 31 0.527 0.00848 0.511 0.544
## 16 1152 48 0.505 0.00870 0.488 0.522
## 17 1002 29 0.490 0.00886 0.473 0.508
## 18 898 21 0.479 0.00900 0.462 0.497
## 19 798 7 0.475 0.00906 0.457 0.493
## 20 705 4 0.472 0.00911 0.454 0.490
## 21 626 13 0.462 0.00932 0.444 0.481
## 22 546 8 0.455 0.00948 0.437 0.474
## 23 481 5 0.451 0.00962 0.432 0.470
## 24 436 4 0.447 0.00975 0.428 0.466
## 25 378 4 0.442 0.00993 0.423 0.462
## 26 336 3 0.438 0.01010 0.419 0.458
## 27 297 1 0.436 0.01017 0.417 0.457
## 29 235 1 0.435 0.01030 0.415 0.455
## 38 73 1 0.429 0.01175 0.406 0.452Al utilizar summary() podemos añadir la opción times y especificar ciertos tiempos en los que queremos ver la información de supervivencia
## Call: survfit(formula = survobj ~ 1)
##
## time n.risk n.event survival std.err lower 95% CI upper 95% CI
## 5 3899 656 0.852 0.00535 0.841 0.862
## 10 2311 810 0.642 0.00761 0.627 0.657
## 20 705 446 0.472 0.00911 0.454 0.490
## 30 210 39 0.435 0.01030 0.415 0.455
## 60 2 1 0.429 0.01175 0.406 0.452También podemos utilizar la función print(). El argumento print.rmean = TRUE se utiliza para obtener el tiempo medio de supervivencia y su error estándar (se).
NOTA: El tiempo medio de supervivencia restringido (RMST) es una medida de supervivencia específica cada vez más utilizada en el análisis de supervivencia del cáncer y que suele definirse como el área bajo la curva de supervivencia, dado que observamos a los pacientes hasta el tiempo restringido T (más detalles en la sección Recursos).
# imprimir el objeto linelistsurv_fit con el tiempo medio de supervivencia y su se.
print(linelistsurv_fit, print.rmean = TRUE)## Call: survfit(formula = survobj ~ 1)
##
## n events rmean* se(rmean) median 0.95LCL 0.95UCL
## [1,] 4539 1952 33.1 0.539 17 16 18
## * restricted mean with upper limit = 64CONSEJO: Podemos crear el objeto surv directamente en la función survfit() y ahorrarnos una línea de código. Esto se verá como: linelistsurv_quick <- survfit(Surv(futime, event) ~ 1, data=linelist_surv).
Riesgo acumulado
Además de la función summary(), también podemos utilizar la función str() que da más detalles sobre la estructura del objeto survfit(). Se trata de una lista de 16 elementos.
Entre estos elementos hay uno importante: el cumhaz, que es un vector numérico. Se puede trazar para mostrar el riesgo acumulado, siendo el riesgo la tasa instantánea de ocurrencia del evento (ver referencias).
str(linelistsurv_fit)## List of 16
## $ n : int 4539
## $ time : num [1:59] 1 2 3 4 5 6 7 8 9 10 ...
## $ n.risk : num [1:59] 4539 4500 4394 4176 3899 ...
## $ n.event : num [1:59] 30 69 149 194 214 210 179 167 145 109 ...
## $ n.censor : num [1:59] 9 37 69 83 93 159 145 139 137 121 ...
## $ surv : num [1:59] 0.993 0.978 0.945 0.901 0.852 ...
## $ std.err : num [1:59] 0.00121 0.00222 0.00359 0.00496 0.00628 ...
## $ cumhaz : num [1:59] 0.00661 0.02194 0.05585 0.10231 0.15719 ...
## $ std.chaz : num [1:59] 0.00121 0.00221 0.00355 0.00487 0.00615 ...
## $ type : chr "right"
## $ logse : logi TRUE
## $ conf.int : num 0.95
## $ conf.type: chr "log"
## $ lower : num [1:59] 0.991 0.974 0.938 0.892 0.841 ...
## $ upper : num [1:59] 0.996 0.982 0.952 0.91 0.862 ...
## $ call : language survfit(formula = survobj ~ 1)
## - attr(*, "class")= chr "survfit"Representar curvas de Kaplan-Meir
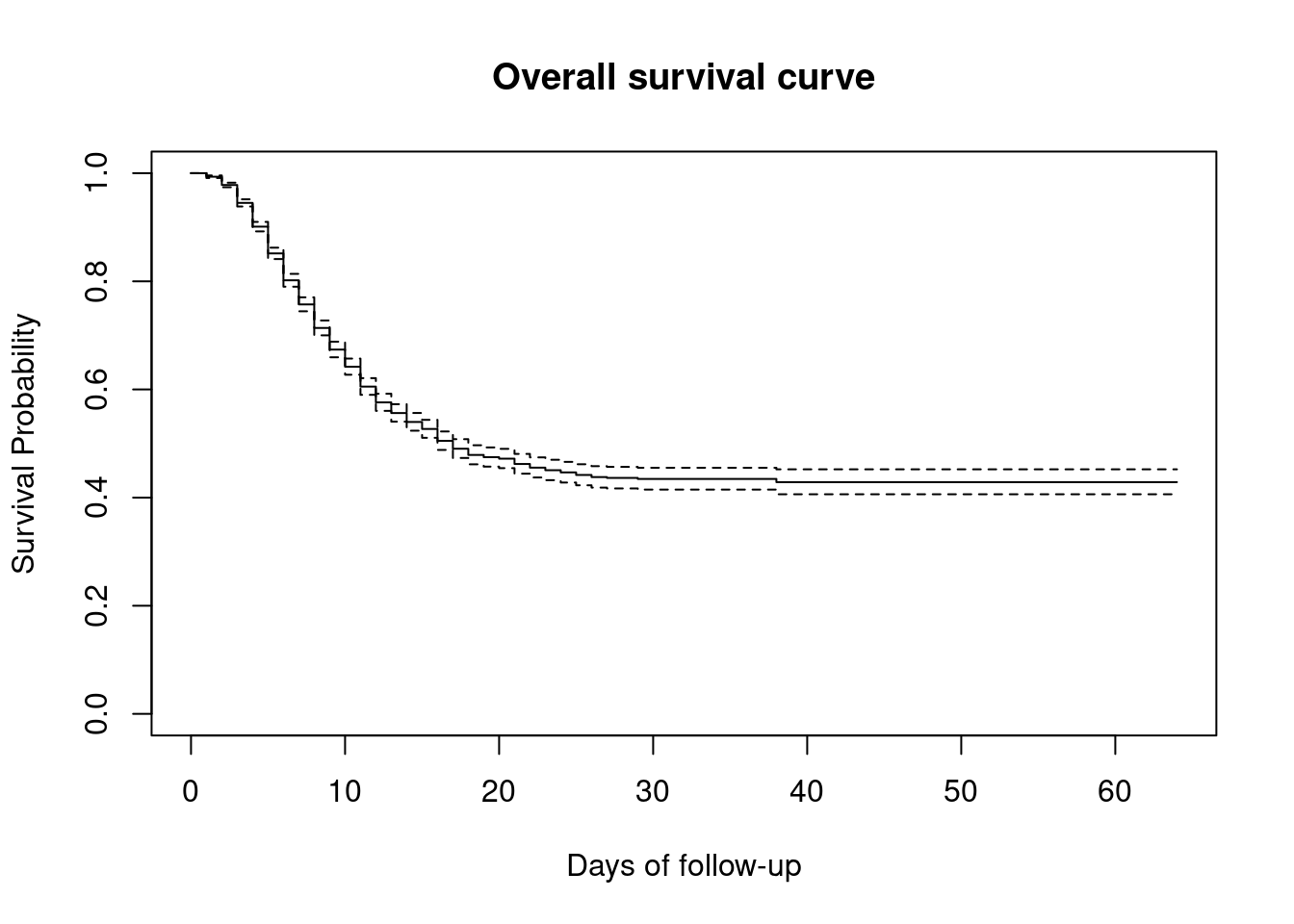
Una vez ajustadas las estimaciones de KM, podemos visualizar la probabilidad de estar vivo a lo largo de un tiempo determinado utilizando la función básica plot() que dibuja la “curva de Kaplan-Meier”. En otras palabras, la curva de abajo es una ilustración convencional de la experiencia de supervivencia en todo el grupo de pacientes.
Podemos verificar rápidamente el tiempo de seguimiento mínimo y máximo en la curva.
Una forma fácil de interpretarlo es decir que en el momento cero, todos los participantes están vivos y la probabilidad de supervivencia es entonces del 100%. Esta probabilidad disminuye con el tiempo a medida que los pacientes mueren. La proporción de participantes que sobreviven más allá de los 60 días de seguimiento se sitúa en torno al 40%.
plot(linelistsurv_fit,
xlab = "Days of follow-up", # etiqueta del eje-x
ylab="Survival Probability", # etiqueta del eje-y
main= "Overall survival curve" # título de la figura
)
El intervalo de confianza de las estimaciones de supervivencia de KM también se representa por defecto y puede descartarse añadiendo la opción conf.int = FALSE al comando plot().
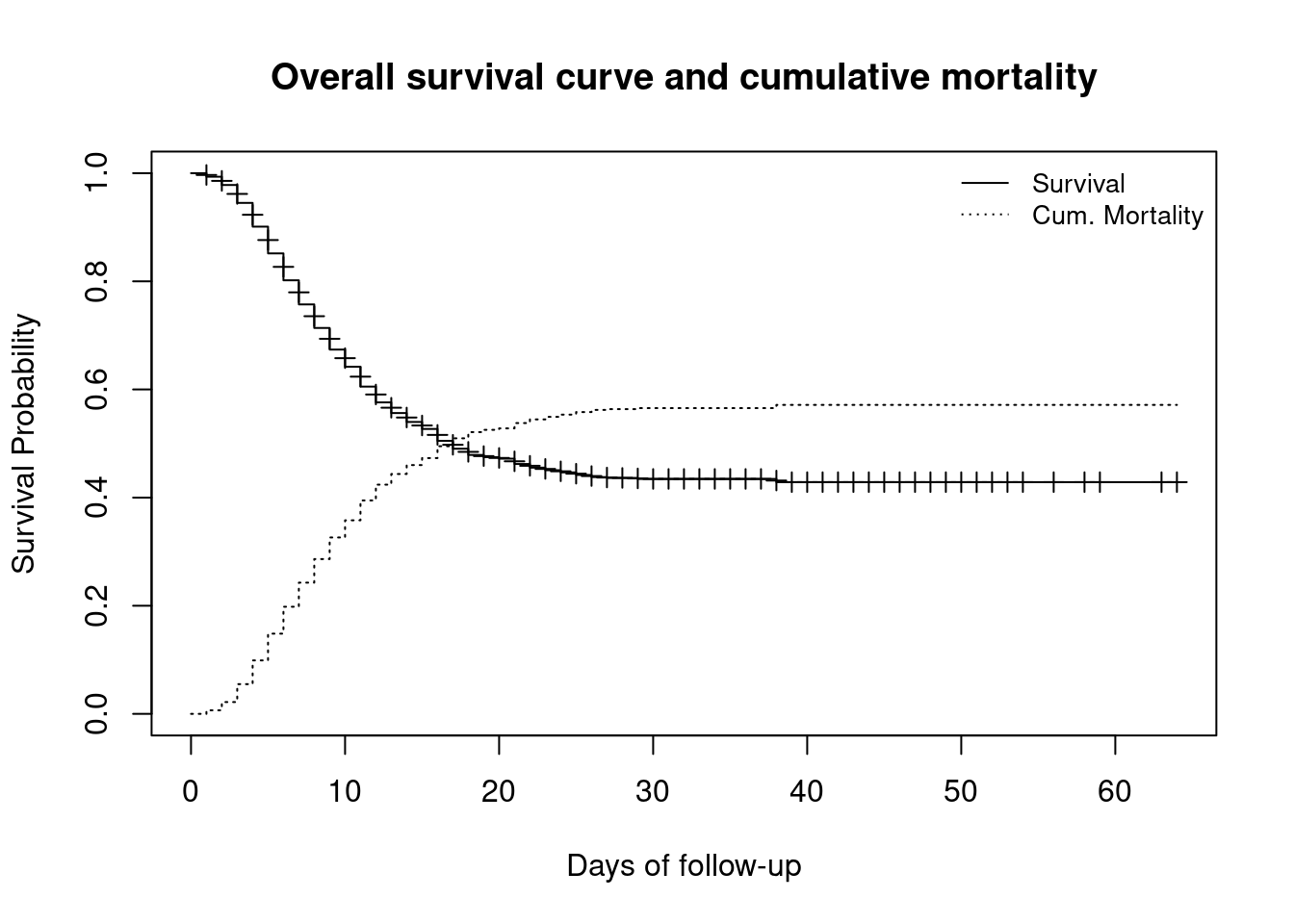
Dado que el evento de interés es “death”, dibujar una curva que describa los complementos de las proporciones de supervivencia llevará a dibujar las proporciones de mortalidad acumulada. Esto puede hacerse con lines(), que añade información a un gráfico existente.
# gráfico original
plot(
linelistsurv_fit,
xlab = "Days of follow-up",
ylab = "Survival Probability",
mark.time = TRUE, # marca los eventos en la curva: se imprime un "+" en cada evento
conf.int = FALSE, # no representa el intervalo de confianza
main = "Overall survival curve and cumulative mortality"
)
# dibujar una curva adicional a la anterior
lines(
linelistsurv_fit,
lty = 3, # use different line type for clarity
fun = "event", # dibuja los eventos acumulativos en lugar de la supervivencia
mark.time = FALSE,
conf.int = FALSE
)
# añade una leyenda al gráfico
legend(
"topright", # posición de la leyenda
legend = c("Survival", "Cum. Mortality"), # texto de la leyenda
lty = c(1, 3), # tipos de línea a utilizar en la leyenda
cex = .85, # parámetro que define el tamaño del texto de la leyenda
bty = "n" # no se dibujará ningún tipo de recuadro para la leyenda
)
27.4 Comparación de las curvas de supervivencia
Para comparar la supervivencia dentro de los diferentes grupos de nuestros participantes o pacientes observados, es posible que tengamos que observar primero sus respectivas curvas de supervivencia y luego realizar pruebas para evaluar la diferencia entre grupos independientes. Esta comparación puede referirse a grupos basados en el género, la edad, el tratamiento, la comorbilidad…
Test Log rank
El test Log rank (de rango logarítmico) es una prueba popular que compara toda la experiencia de supervivencia entre dos o más grupos independientes y puede considerarse como una prueba de si las curvas de supervivencia son idénticas (se superponen) o no (hipótesis nula de no diferencia de supervivencia entre los grupos). La función survdiff() del paquete survival permite ejecutar el test Log rank cuando especificamos rho = 0 (que es el valor predeterminado). Los resultados de la prueba dan un estadístico chi-cuadrado junto con un valor-p, ya que el estadístico log rank se distribuye aproximadamente como un test estadístico de chi-cuadrado.
En primer lugar, tratamos de comparar las curvas de supervivencia por grupos de género. Para ello, primero intentamos visualizarlo (comprobar si las dos curvas de supervivencia se superponen). Se creará un nuevo objeto survfit con una fórmula ligeramente diferente. Luego se creará el objeto survdiff.
Al suministrar ~ gender como lado derecho de la fórmula, ya no trazamos la supervivencia global sino por género.
# crear el nuevo objeto survfit basado en el género
linelistsurv_fit_sex <- survfit(Surv(futime, event) ~ gender, data = linelist_surv)Ahora podemos trazar las curvas de supervivencia por género. Observa el orden de los niveles de los estratos en la columna de género antes de definir los colores y la leyenda.
# establecer colores
col_sex <- c("lightgreen", "darkgreen")
# crear gráfico
plot(
linelistsurv_fit_sex,
col = col_sex,
xlab = "Days of follow-up",
ylab = "Survival Probability")
# añadir leyenda
legend(
"topright",
legend = c("Female","Male"),
col = col_sex,
lty = 1,
cex = .9,
bty = "n")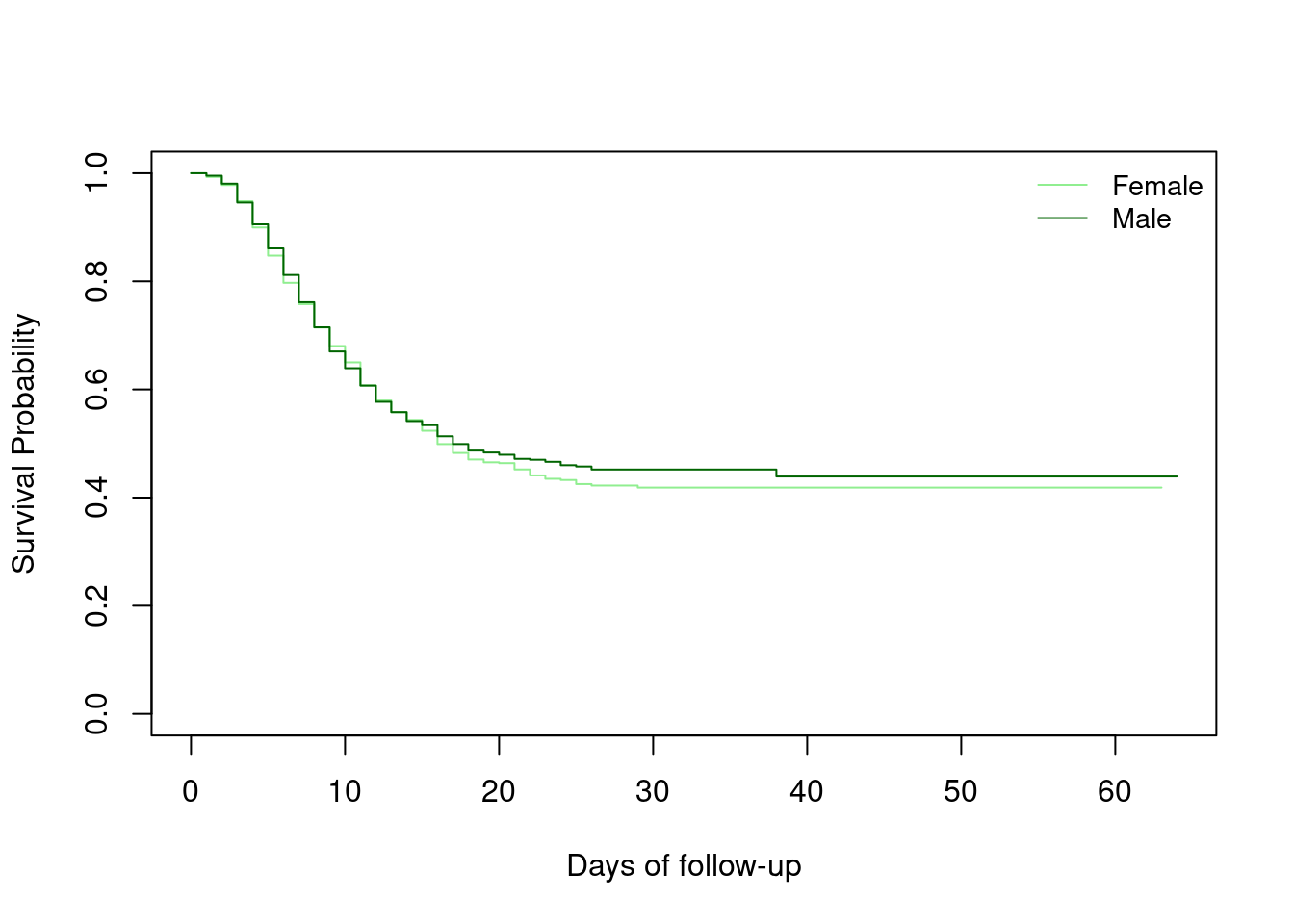
Y ahora podemos calcular la prueba de la diferencia entre las curvas de supervivencia utilizando survdiff()
#calcular el test de la diferencia entre las curvas de supervivencia
survival::survdiff(
Surv(futime, event) ~ gender,
data = linelist_surv
)## Call:
## survival::survdiff(formula = Surv(futime, event) ~ gender, data = linelist_surv)
##
## n=4321, 218 observations deleted due to missingness.
##
## N Observed Expected (O-E)^2/E (O-E)^2/V
## gender=f 2156 924 909 0.255 0.524
## gender=m 2165 929 944 0.245 0.524
##
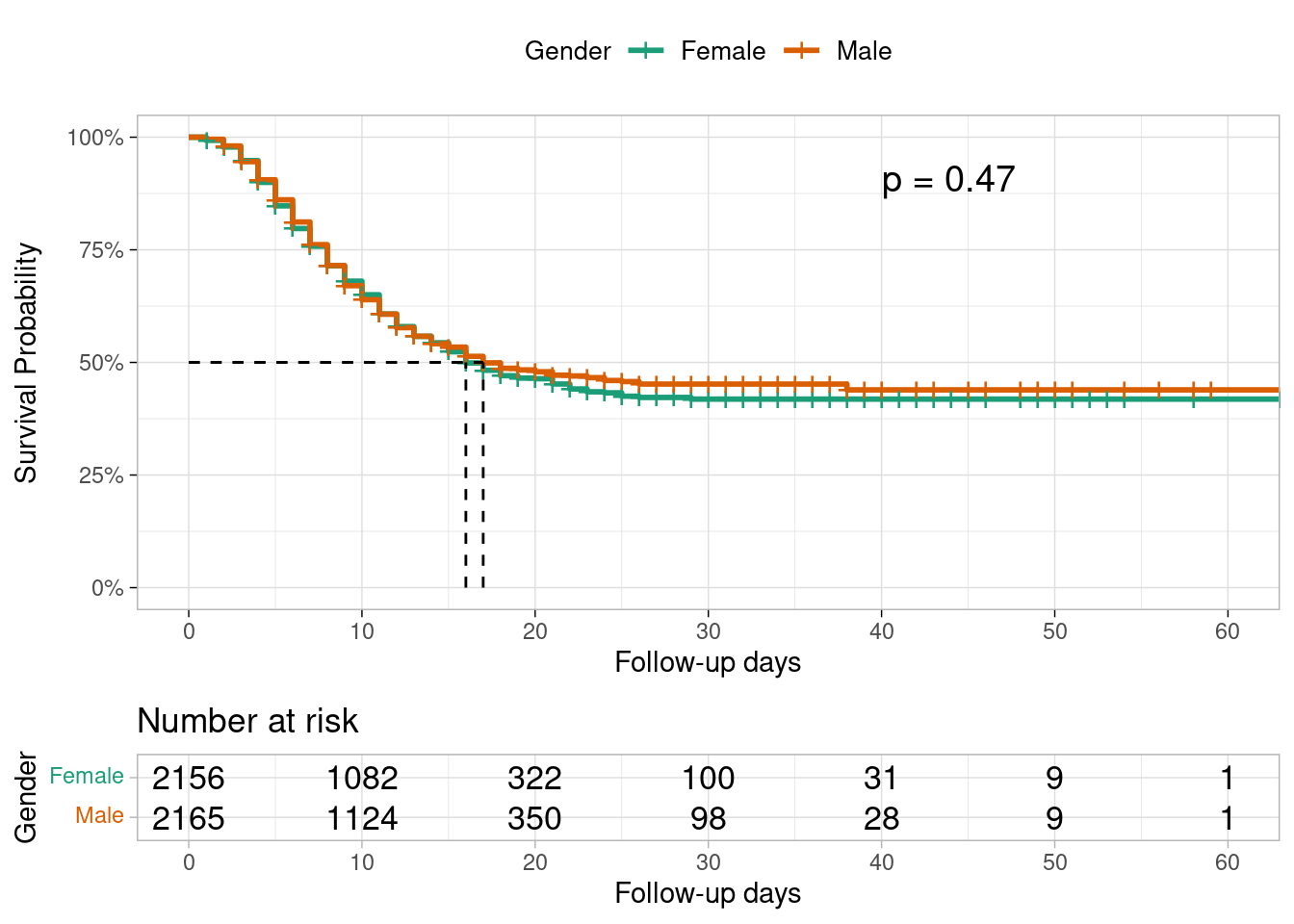
## Chisq= 0.5 on 1 degrees of freedom, p= 0.5Vemos que la curva de supervivencia de las mujeres y la de los hombres se superponen y la prueba de rango logarítmico no da pruebas de una diferencia de supervivencia entre mujeres y hombres.
Algunos otros paquetes de R permiten ilustrar curvas de supervivencia para diferentes grupos y probar la diferencia de una sola vez. Utilizando la función ggsurvplot() del paquete survminer, también podemos incluir en nuestra curva las tablas de riesgo impresas para cada grupo, así como el valor p del test log-rank.
PRECAUCIÓN: las funciones de survminer requieren que especifiques el objeto de supervivencia y que vuelvas a especificar los datos utilizados para ajustar el objeto de supervivencia. Recuerda hacer esto para evitar mensajes de error no específicos.
survminer::ggsurvplot(
linelistsurv_fit_sex,
data = linelist_surv, # especifica de nuevo los datos usados para ajustar linelistsurv_fit_sex
conf.int = FALSE, # no muestra el intervalo de confianza de las estimaciones de KM
surv.scale = "percent", # presenta las probabilidades en el eje y en %
break.time.by = 10, # presenta el eje temporal con un incremento de 10 días
xlab = "Follow-up days",
ylab = "Survival Probability",
pval = T, # imprime el valor p de la prueba Log-rank
pval.coord = c(40,.91), # imprime el valor p en estas coordenadas del gráfico
risk.table = T, # imprime la tabla de riesgos en la parte inferior
legend.title = "Gender", # características de la leyenda
legend.labs = c("Female","Male"),
font.legend = 10,
palette = "Dark2", # especifica la paleta de colores
surv.median.line = "hv", # dibuja líneas horizontales y verticales a las medianas de supervivencia
ggtheme = theme_light() # simplifica el fondo del gráfico
)
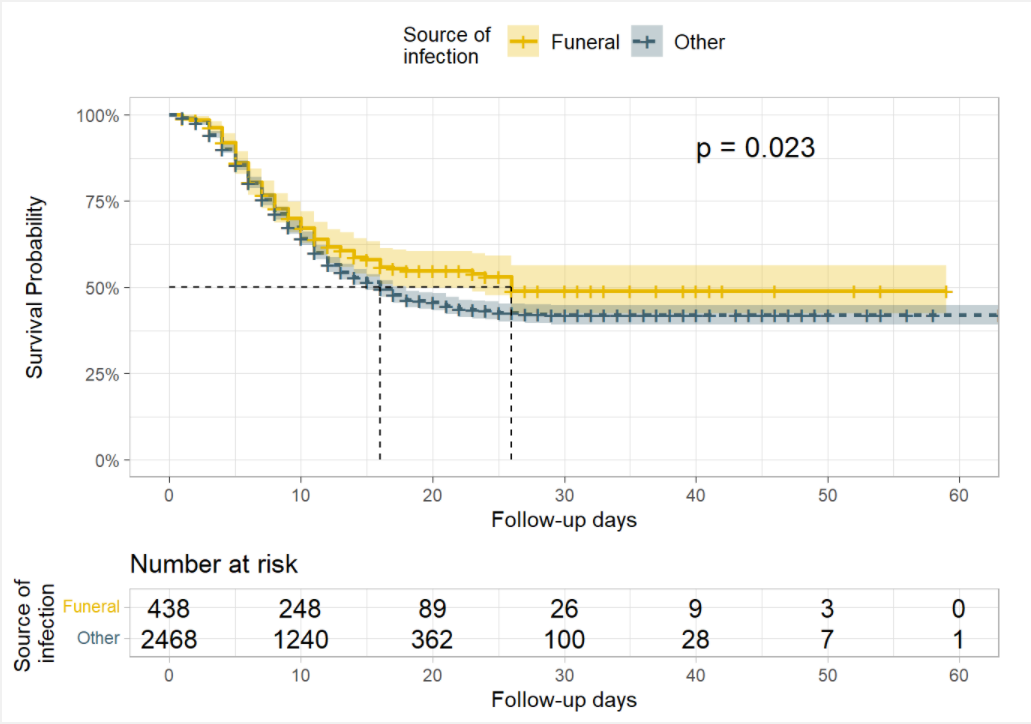
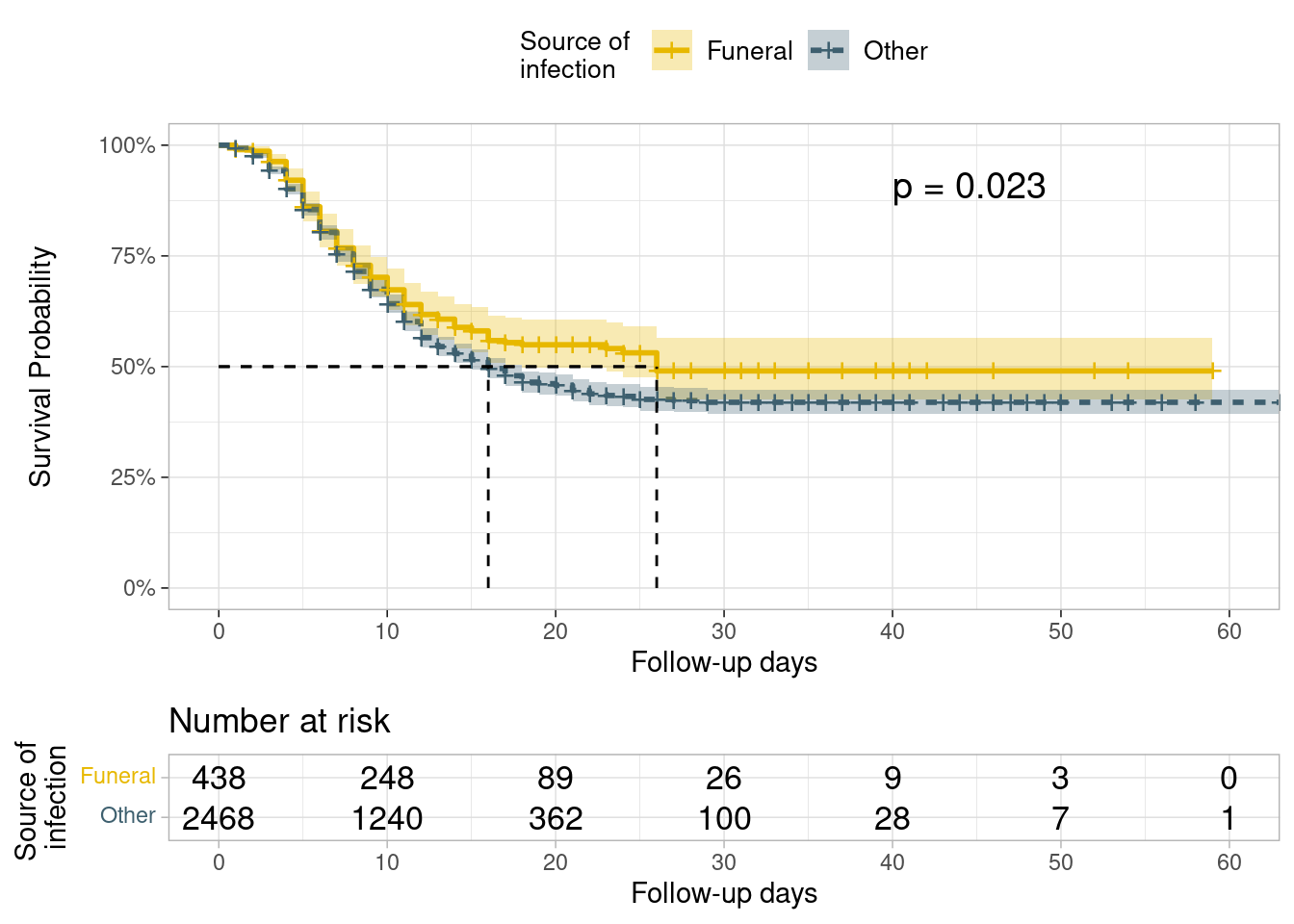
También podemos comprobar si hay diferencias en la supervivencia según la fuente de infección (fuente de contaminación).
En este caso, la prueba de rango logarítmico da pruebas suficientes de una diferencia en las probabilidades de supervivencia a alfa= 0,005. Las probabilidades de supervivencia de los pacientes que se infectaron en los funerales son mayores que las de los pacientes que se infectaron en otros lugares, lo que sugiere un beneficio para la supervivencia.
linelistsurv_fit_source <- survfit(
Surv(futime, event) ~ source,
data = linelist_surv
)
# gráfico
ggsurvplot(
linelistsurv_fit_source,
data = linelist_surv,
size = 1, linetype = "strata", # line types
conf.int = T,
surv.scale = "percent",
break.time.by = 10,
xlab = "Follow-up days",
ylab= "Survival Probability",
pval = T,
pval.coord = c(40,.91),
risk.table = T,
legend.title = "Source of \ninfection",
legend.labs = c("Funeral", "Other"),
font.legend = 10,
palette = c("#E7B800","#3E606F"),
surv.median.line = "hv",
ggtheme = theme_light()
)
27.5 Análisis de regresión de Cox
La regresión de riesgos proporcionales de Cox es una de las técnicas de regresión más populares para el análisis de supervivencia. También se pueden utilizar otros modelos, ya que el modelo de Cox requiere supuestos importantes que deben verificarse para un uso adecuado, como el supuesto de riesgos proporcionales: véanse las referencias.
En un modelo de regresión de riesgos proporcionales de Cox, la medida del efecto es la tasa de riesgo (HR), que es el riesgo de fracaso (o el riesgo de muerte en nuestro ejemplo), dado que el participante ha sobrevivido hasta un momento específico. Normalmente, nos interesa comparar grupos independientes con respecto a sus riesgos, y utilizamos una razón de riesgo, que es análoga a una razón de probabilidades en el entorno del análisis de regresión logística múltiple. La función cox.ph() del paquete de supervivencia se utiliza para ajustar el modelo. La función cox.zph() del paquete survival puede utilizarse para probar la suposición de riesgos proporcionales para un ajuste del modelo de regresión de Cox.
NOTA: Una probabilidad debe estar en el rango de 0 a 1. Sin embargo, el peligro representa el número esperado de eventos por una unidad de tiempo.
- Si la razón de riesgo (RR)de un predictor es cercana a 1, entonces ese predictor no afecta a la supervivencia,
- Si la RR es inferior a 1, entonces el predictor es protector (es decir, está asociado a una mejor supervivencia),
- y si la RR es mayor que 1, entonces el predictor se asocia a un mayor riesgo (o a una menor supervivencia).
Ajuste de un modelo de Cox
Primero podemos ajustar un modelo para evaluar el efecto de la edad y el sexo en la supervivencia. Con sólo imprimir el modelo, tenemos la información sobre:
- los coeficientes de regresión estimados
coefque cuantifican la asociación entre los predictores y el resultado, - su exponencial (para su interpretación,
exp(coef)) que produce la razón de riesgo, - su error estándar
se(coef), - la puntuación z: cuántos errores estándar se aleja el coeficiente estimado de 0,
- y el valor- p: la probabilidad de que el coeficiente estimado sea 0.
La función summary() aplicada al objeto del modelo de Cox ofrece más información, como el intervalo de confianza de la RR estimada y las diferentes puntuaciones de la prueba.
El efecto de la primera covariable, gender, se presenta en la primera fila. Se imprime genderm (masculino), lo que implica que el primer nivel de estrato (“f”), es decir, el grupo femenino, es el grupo de referencia para el género. Por lo tanto, la interpretación del parámetro de la prueba es la de los hombres en comparación con las mujeres. El valor p indica que no hay pruebas suficientes de un efecto del género sobre el peligro esperado o de una asociación entre el género y la mortalidad por todas las causas.
La misma falta de pruebas se observa en relación con el grupo de edad.
#ajustar el modelo cox
linelistsurv_cox_sexage <- survival::coxph(
Surv(futime, event) ~ gender + age_cat_small,
data = linelist_surv
)
#impresión del modelo ajustado
linelistsurv_cox_sexage## Call:
## survival::coxph(formula = Surv(futime, event) ~ gender + age_cat_small,
## data = linelist_surv)
##
## coef exp(coef) se(coef) z p
## genderm -0.03149 0.96900 0.04767 -0.661 0.509
## age_cat_small5-19 0.09400 1.09856 0.06454 1.456 0.145
## age_cat_small20+ 0.05032 1.05161 0.06953 0.724 0.469
##
## Likelihood ratio test=2.8 on 3 df, p=0.4243
## n= 4321, number of events= 1853
## (218 observations deleted due to missingness)
#resumen del modelo
summary(linelistsurv_cox_sexage)## Call:
## survival::coxph(formula = Surv(futime, event) ~ gender + age_cat_small,
## data = linelist_surv)
##
## n= 4321, number of events= 1853
## (218 observations deleted due to missingness)
##
## coef exp(coef) se(coef) z Pr(>|z|)
## genderm -0.03149 0.96900 0.04767 -0.661 0.509
## age_cat_small5-19 0.09400 1.09856 0.06454 1.456 0.145
## age_cat_small20+ 0.05032 1.05161 0.06953 0.724 0.469
##
## exp(coef) exp(-coef) lower .95 upper .95
## genderm 0.969 1.0320 0.8826 1.064
## age_cat_small5-19 1.099 0.9103 0.9680 1.247
## age_cat_small20+ 1.052 0.9509 0.9176 1.205
##
## Concordance= 0.514 (se = 0.007 )
## Likelihood ratio test= 2.8 on 3 df, p=0.4
## Wald test = 2.78 on 3 df, p=0.4
## Score (logrank) test = 2.78 on 3 df, p=0.4Fue interesante ejecutar el modelo y observar los resultados, pero un primer vistazo para verificar si se respetan los supuestos de riesgos proporcionales podría ayudar a ahorrar tiempo.
test_ph_sexage <- survival::cox.zph(linelistsurv_cox_sexage)
test_ph_sexage## chisq df p
## gender 0.454 1 0.50
## age_cat_small 0.838 2 0.66
## GLOBAL 1.399 3 0.71NOTA: Se puede especificar un segundo argumento llamado método cuando se calcula el modelo de Cox, que determina cómo se manejan los empates. El valor por defecto es “efron”, y las otras opciones son “breslow” y “exact”.
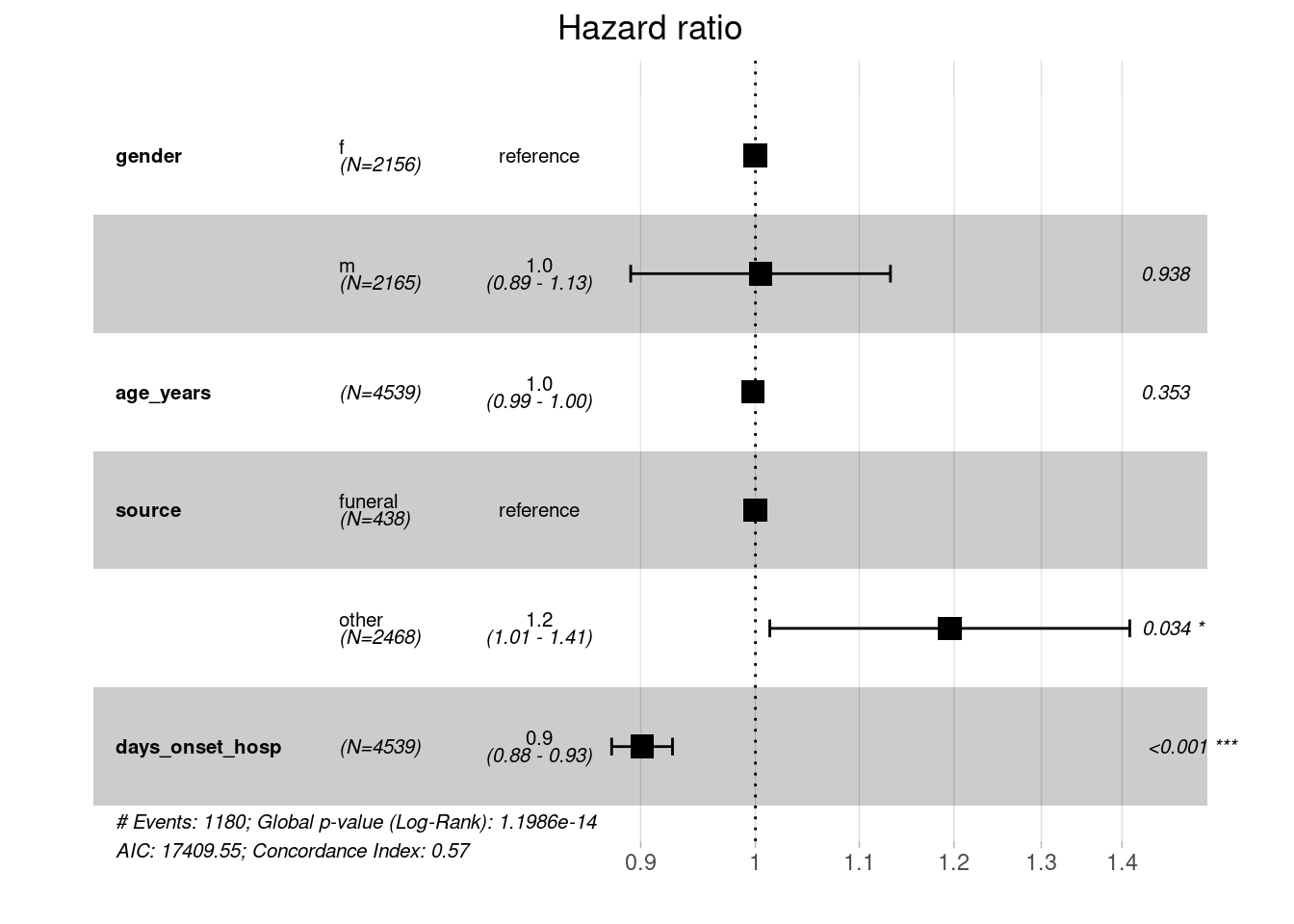
En otro modelo añadimos más factores de riesgo, como el origen de la infección y el número de días entre la fecha de inicio y el ingreso. Esta vez, primero verificamos la hipótesis de riesgos proporcionales antes de seguir adelante.
En este modelo, hemos incluido un predictor continuo (days_onset_hosp). En este caso, interpretamos las estimaciones de los parámetros como el aumento del logaritmo esperado del riesgo relativo por cada aumento de una unidad en el predictor, manteniendo los demás predictores constantes. Primero verificamos el supuesto de riesgos proporcionales.
#ajustar el modelo
linelistsurv_cox <- coxph(
Surv(futime, event) ~ gender + age_years+ source + days_onset_hosp,
data = linelist_surv
)
#comprobar el modelo de riesgo proporcional
linelistsurv_ph_test <- cox.zph(linelistsurv_cox)
linelistsurv_ph_test## chisq df p
## gender 0.45062 1 0.50
## age_years 0.00199 1 0.96
## source 1.79622 1 0.18
## days_onset_hosp 31.66167 1 1.8e-08
## GLOBAL 34.08502 4 7.2e-07La verificación gráfica de esta suposición puede realizarse con la función ggcoxzph() del paquete survminer.
survminer::ggcoxzph(linelistsurv_ph_test)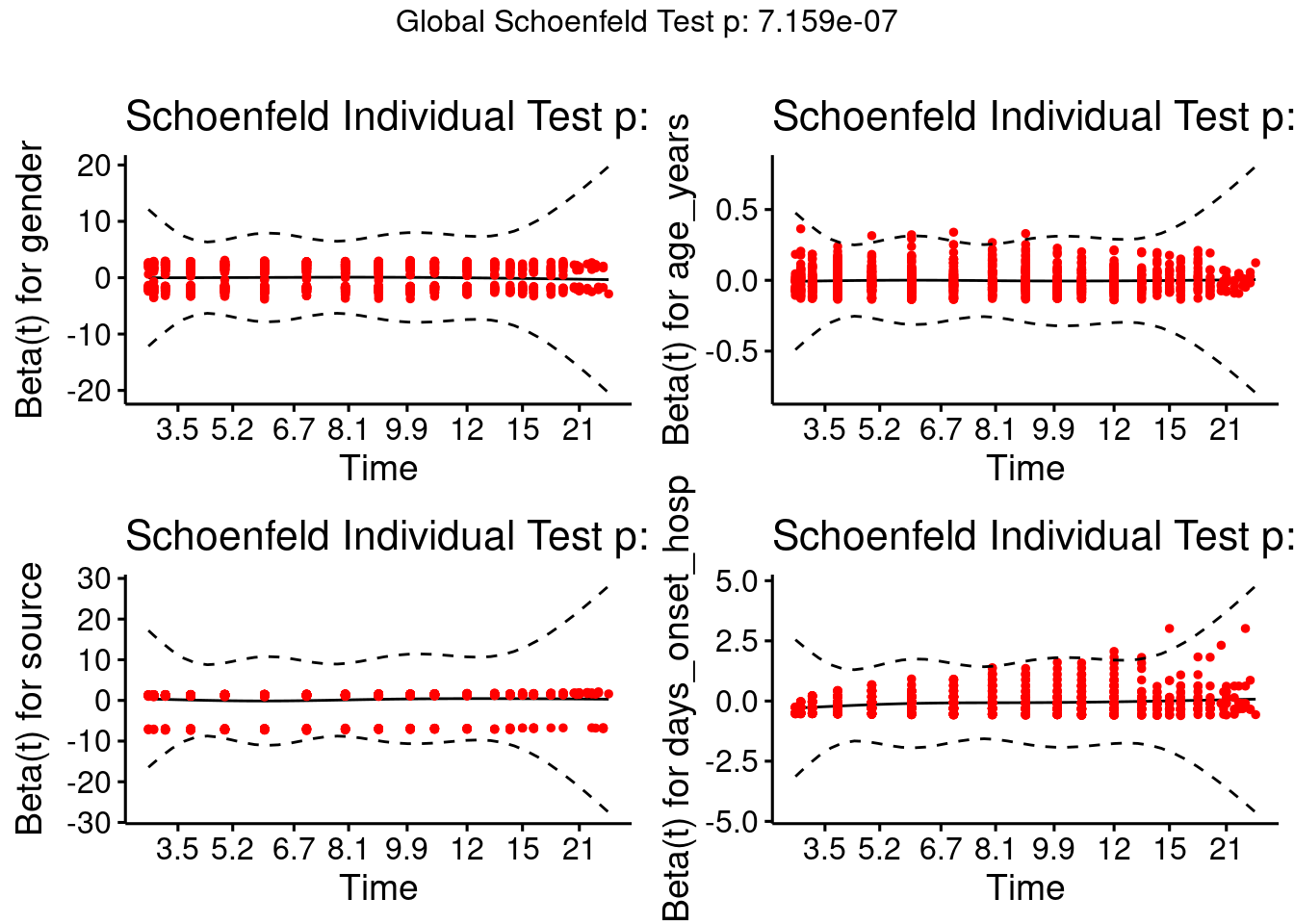
Los resultados del modelo indican que existe una asociación negativa entre la duración del inicio del ingreso y la mortalidad por todas las causas. El riesgo esperado es 0,9 veces menor en una persona que ingresa un día más tarde que otra, manteniendo el género constante. O, en una explicación más directa, un aumento de una unidad en la duración del inicio al ingreso se asocia con una disminución del 10,7% (coef *100) en el riesgo de muerte.
Los resultados muestran también una asociación positiva entre la fuente de infección y la mortalidad por todas las causas. Es decir, hay un mayor riesgo de muerte (1,21 veces) para los pacientes que tuvieron una fuente de infección distinta de los funerales.
#imprimir el resumen del modelo
summary(linelistsurv_cox)## Call:
## coxph(formula = Surv(futime, event) ~ gender + age_years + source +
## days_onset_hosp, data = linelist_surv)
##
## n= 2772, number of events= 1180
## (1767 observations deleted due to missingness)
##
## coef exp(coef) se(coef) z Pr(>|z|)
## genderm 0.004710 1.004721 0.060827 0.077 0.9383
## age_years -0.002249 0.997753 0.002421 -0.929 0.3528
## sourceother 0.178393 1.195295 0.084291 2.116 0.0343 *
## days_onset_hosp -0.104063 0.901169 0.014245 -7.305 2.77e-13 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## exp(coef) exp(-coef) lower .95 upper .95
## genderm 1.0047 0.9953 0.8918 1.1319
## age_years 0.9978 1.0023 0.9930 1.0025
## sourceother 1.1953 0.8366 1.0133 1.4100
## days_onset_hosp 0.9012 1.1097 0.8764 0.9267
##
## Concordance= 0.566 (se = 0.009 )
## Likelihood ratio test= 71.31 on 4 df, p=1e-14
## Wald test = 59.22 on 4 df, p=4e-12
## Score (logrank) test = 59.54 on 4 df, p=4e-12Podemos comprobar esta relación con una tabla:
linelist_case_data %>%
tabyl(days_onset_hosp, outcome) %>%
adorn_percentages() %>%
adorn_pct_formatting()## days_onset_hosp Death Recover NA_
## 0 44.3% 31.4% 24.3%
## 1 46.6% 32.2% 21.2%
## 2 43.0% 32.8% 24.2%
## 3 45.0% 32.3% 22.7%
## 4 41.5% 38.3% 20.2%
## 5 40.0% 36.2% 23.8%
## 6 32.2% 48.7% 19.1%
## 7 31.8% 38.6% 29.5%
## 8 29.8% 38.6% 31.6%
## 9 30.3% 51.5% 18.2%
## 10 16.7% 58.3% 25.0%
## 11 36.4% 45.5% 18.2%
## 12 18.8% 62.5% 18.8%
## 13 10.0% 60.0% 30.0%
## 14 10.0% 50.0% 40.0%
## 15 28.6% 42.9% 28.6%
## 16 20.0% 80.0% 0.0%
## 17 0.0% 100.0% 0.0%
## 18 0.0% 100.0% 0.0%
## 22 0.0% 100.0% 0.0%
## NA 52.7% 31.2% 16.0%Habría que considerar e investigar por qué existe esta asociación en los datos. Una posible explicación podría ser que los pacientes que viven lo suficiente como para ser ingresados más tarde tenían una enfermedad menos grave para empezar. Otra explicación, quizá más probable, es que, dado que utilizamos unos datos falsos simulados, este patrón no refleja la realidad.
27.6 Covariables tiempo-dependientes en modelos de supervivencia
Algunas de las siguientes secciones han sido adaptadas con permiso de la excelente introducción al análisis de supervivencia en R por la Dra. Emily Zabor
En la última sección hemos tratado el uso de la regresión de Cox para examinar las asociaciones entre las covariables de interés y los resultados de supervivencia, pero estos análisis dependen de que la covariable se mida en la línea de base, es decir, antes de que comience el tiempo de seguimiento del evento.
¿Qué ocurre si tienes interés en una covariable que se mide después del tiempo de seguimiento? O, ¿qué pasa si tienes una covariable que puede cambiar con el tiempo?
Por ejemplo, tal vez estés trabajando con datos clínicos en los que se repiten medidas de valores de laboratorio del hospital que pueden cambiar con el tiempo. Este es un ejemplo de una covariable dependiente del tiempo. Para abordar esto se necesita una configuración especial, pero afortunadamente el modelo de Cox es muy flexible y este tipo de datos también puede ser modelado con herramientas del paquete survival.
Configuración de covariables dependientes del tiempo
El análisis de covariables dependientes del tiempo en R requiere la configuración de unos datos especial. Si tienes interés, mira el documento del autor del paquete survival Using Time Dependent Covariates and Time Dependent Coefficients in the Cox Model.
Para ello, utilizaremos un nuevo conjunto de datos del paquete SemiCompRisks denominado BMT, que incluye datos de 137 pacientes de trasplante de médula ósea. Las variables en las que nos centraremos son:
-
T1- tiempo (en días) hasta la muerte o el último seguimiento -
delta1- indicador de muerte; 1-Muerto, 0-Vivo -
TA- tiempo (en días) hasta la enfermedad aguda de injerto contra huésped -
deltaA- indicador de la enfermedad aguda de injerto contra huésped;- 1 - Desarrolló la enfermedad aguda de injerto contra huésped
- 0 - Nunca desarrolló la enfermedad aguda de injerto contra huésped
Cargaremos este conjunto de datos del paquete survival utilizando el comando DE R base data(), que puede utilizarse para cargar datos que ya están incluidos en un paquete de R que se ha cargado. El dataframe BMT aparecerá en tu entorno de R.
data(BMT, package = "SemiCompRisks")Añadir un identificador único de paciente
No hay una columna de identificación única en los datos de BMT, que es necesaria para crear el tipo de conjunto de datos que queremos. Así que utilizamos la función rowid_to_column() del paquete tibble de tidyverse para crear una nueva columna de identificación llamada my_id (añade una columna al principio del dataframe con identificadores de fila secuenciales, empezando por el 1). Llamamos al dataframe bmt.
bmt <- rowid_to_column(BMT, "my_id")El conjunto de datos tiene ahora este aspecto:
Ampliar las filas de pacientes
A continuación, utilizaremos la función tmerge() con las funciones de ayuda event() y tdc() para crear el conjunto de datos reestructurado. Nuestro objetivo es reestructurar el conjunto de datos para crear una fila separada para cada paciente por cada intervalo de tiempo en el que tengan un valor diferente de deltaA. En este caso, cada paciente puede tener como máximo dos filas dependiendo de si desarrollaron la enfermedad aguda de injerto contra huésped durante el periodo de recogida de datos. Llamaremos a nuestro nuevo indicador para el desarrollo de la enfermedad aguda de injerto contra huésped agvhd.
-
tmerge()crea unos datos largos con múltiples intervalos de tiempo para los diferentes valores de las covariables de cada paciente -
event()crea el nuevo indicador de eventos para que vaya con los intervalos de tiempo recién creados -
tdc()crea la columna de covarianza dependiente del tiempo,agvhd, para que vaya con los intervalos de tiempo recién creados
td_dat <-
tmerge(
data1 = bmt %>% select(my_id, T1, delta1),
data2 = bmt %>% select(my_id, T1, delta1, TA, deltaA),
id = my_id,
death = event(T1, delta1),
agvhd = tdc(TA)
)Para ver qué hace esto, veamos los datos de los 5 primeros pacientes individuales.
Las variables de interés en los datos originales tenían este aspecto:
## my_id T1 delta1 TA deltaA
## 1 1 2081 0 67 1
## 2 2 1602 0 1602 0
## 3 3 1496 0 1496 0
## 4 4 1462 0 70 1
## 5 5 1433 0 1433 0El nuevo conjunto de datos para estos mismos pacientes tiene el siguiente aspecto:
## my_id T1 delta1 tstart tstop death agvhd
## 1 1 2081 0 0 67 0 0
## 2 1 2081 0 67 2081 0 1
## 3 2 1602 0 0 1602 0 0
## 4 3 1496 0 0 1496 0 0
## 5 4 1462 0 0 70 0 0
## 6 4 1462 0 70 1462 0 1
## 7 5 1433 0 0 1433 0 0Ahora algunos de nuestros pacientes tienen dos filas en el conjunto de datos correspondientes a intervalos en los que tienen un valor diferente de nuestra nueva variable, agvhd. Por ejemplo, el paciente 1 tiene ahora dos filas con un valor de agvhd de cero desde el tiempo 0 hasta el tiempo 67, y un valor de 1 desde el tiempo 67 hasta el tiempo 2081.
Regresión de Cox con covariables dependientes del tiempo
Ahora que hemos remodelado nuestros datos y añadido la nueva variable aghvd dependiente del tiempo, vamos a ajustar un modelo de regresión cox simple de una sola variable. Podemos utilizar la misma función coxph() que antes, sólo tenemos que cambiar nuestra función Surv() para especificar tanto el tiempo de inicio como el de finalización de cada intervalo utilizando los argumentos time1 = y time2 =.
bmt_td_model = coxph(
Surv(time = tstart, time2 = tstop, event = death) ~ agvhd,
data = td_dat
)
summary(bmt_td_model)## Call:
## coxph(formula = Surv(time = tstart, time2 = tstop, event = death) ~
## agvhd, data = td_dat)
##
## n= 163, number of events= 80
##
## coef exp(coef) se(coef) z Pr(>|z|)
## agvhd 0.3351 1.3980 0.2815 1.19 0.234
##
## exp(coef) exp(-coef) lower .95 upper .95
## agvhd 1.398 0.7153 0.8052 2.427
##
## Concordance= 0.535 (se = 0.024 )
## Likelihood ratio test= 1.33 on 1 df, p=0.2
## Wald test = 1.42 on 1 df, p=0.2
## Score (logrank) test = 1.43 on 1 df, p=0.2De nuevo, visualizaremos los resultados de nuestro modelo de Cox utilizando la función ggforest() del paquete urvminer.:
ggforest(bmt_td_model, data = td_dat)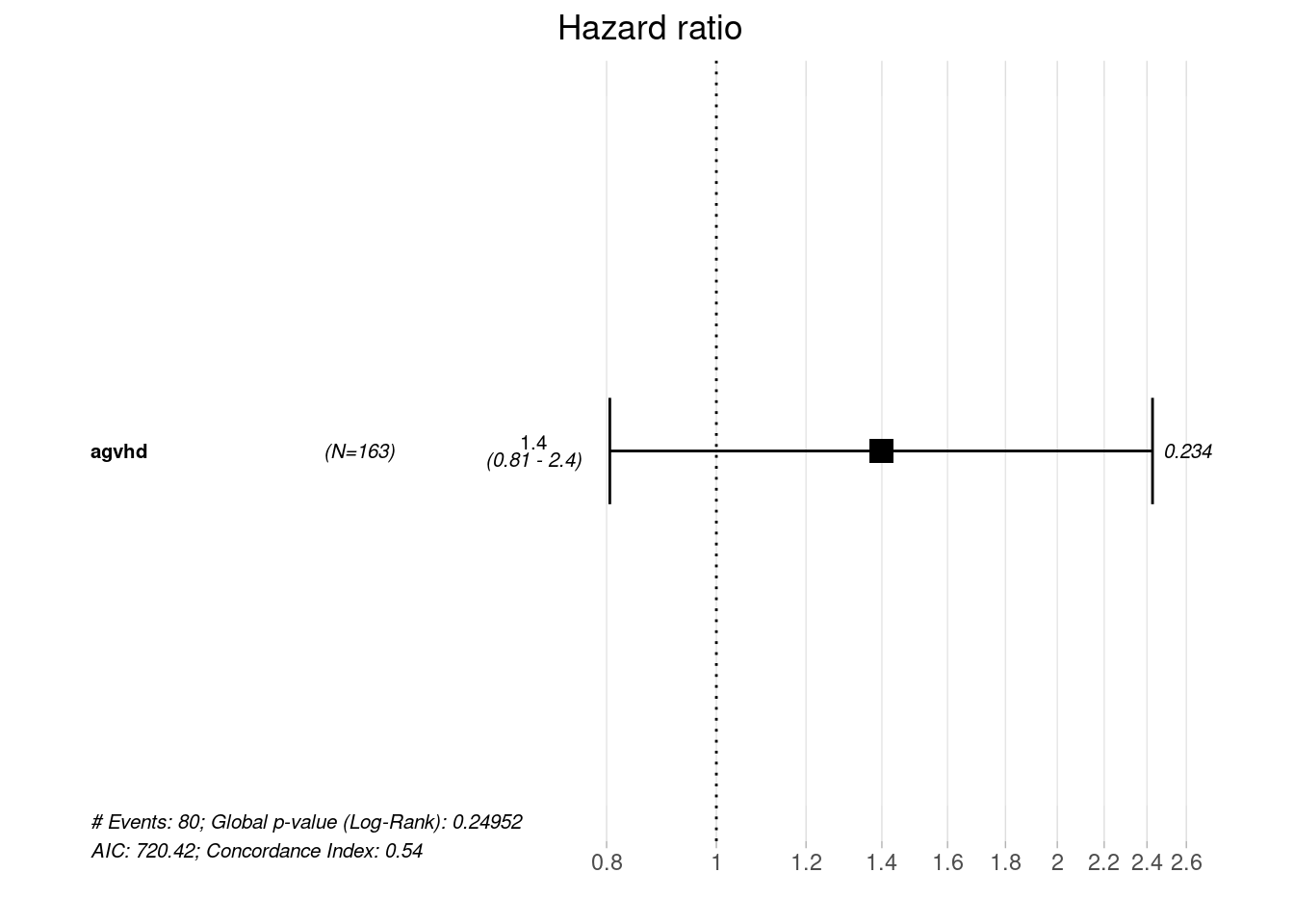
Como se puede ver en el gráfico de forest, el intervalo de confianza y el valor-p, no parece haber una fuerte asociación entre la muerte y la enfermedad aguda de injerto contra huésped en el contexto de nuestro modelo simple.
27.7 Recursos
Análisis de supervivencia Parte I: Conceptos básicos y primeros análisis
Análisis de supervivencia en R
Capítulo sobre modelos de supervivencia avanzados Princeton
Uso de covariables y coeficientes dependientes del tiempo en el modelo de Cox