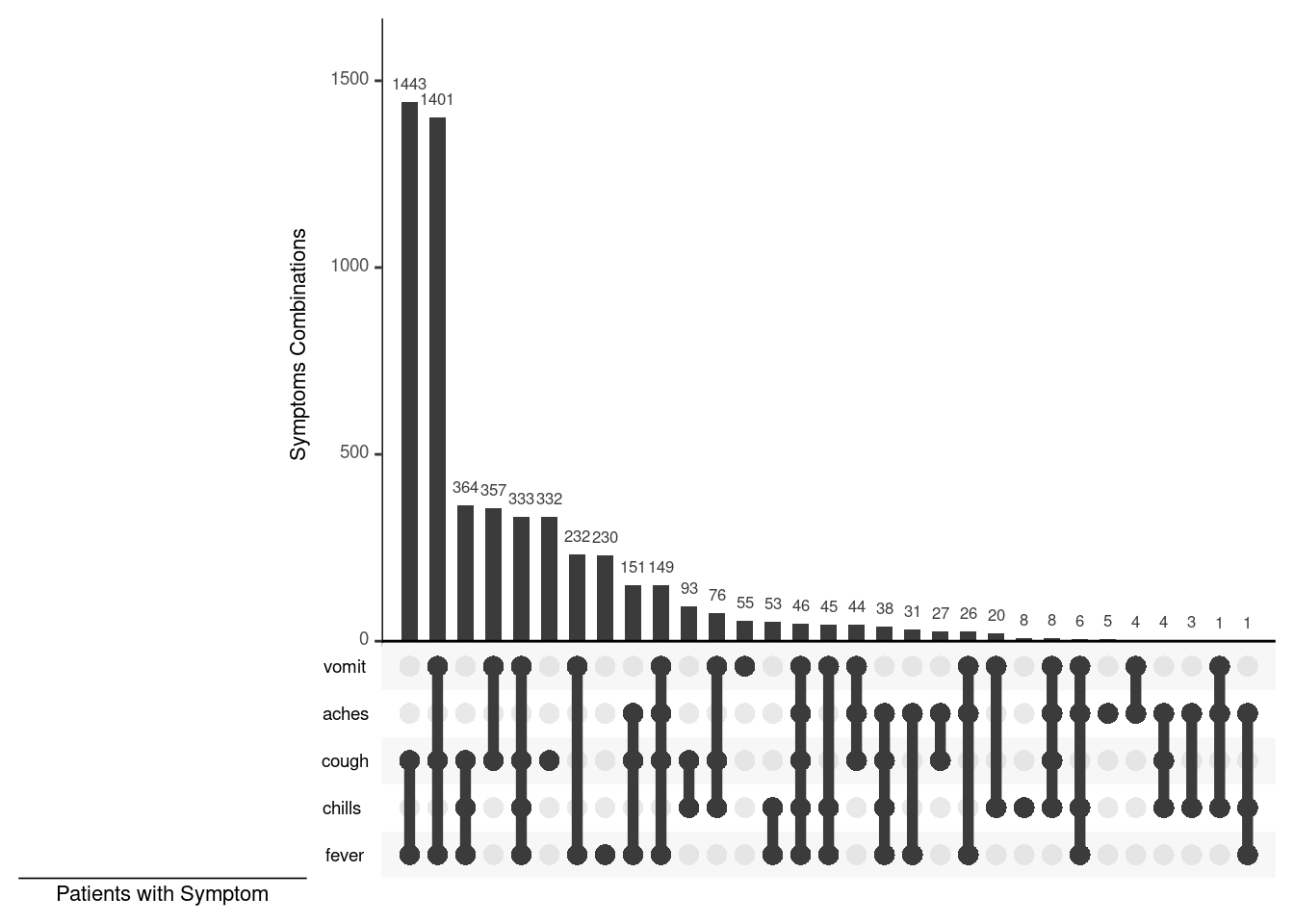
36 Análisis de combinaciones
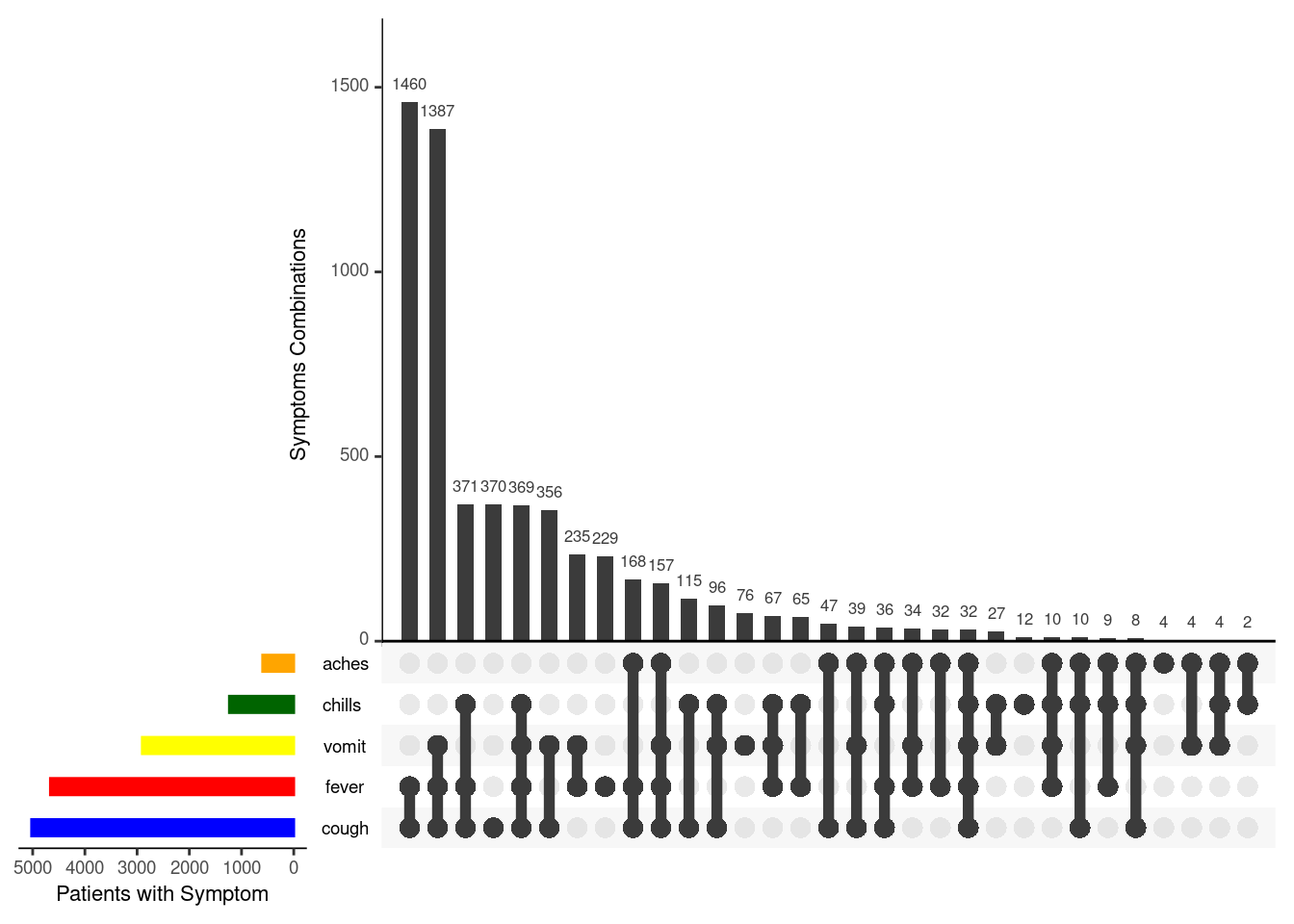
Este análisis representa la frecuencia de diferentes combinaciones de valores/respuestas. En este ejemplo, se representa la frecuencia con la que los casos mostraron varias combinaciones de síntomas.
Este análisis también se suele llamar:
- “Análisis de respuesta múltiple”
- “Análisis de conjuntos”
- “Análisis de combinaciones”
En el ejemplo del gráfico anterior, se muestran cinco síntomas. Debajo de cada barra vertical hay una línea y puntos que indican la combinación de síntomas que refleja la barra de arriba. A la derecha, las barras horizontales reflejan la frecuencia de cada síntoma individual.
El primer método que mostramos utiliza el paquete ggupset, y el segundo utiliza el paquete UpSetR.
36.1 Preparación
Cargar paquetes
Este trozo de código muestra la carga de los paquetes necesarios para los análisis. En este manual destacamos p_load() de pacman, que instala el paquete si es necesario y lo carga para su uso. También puedes cargar los paquetes instalados con library() de R base. Consulta la página sobre fundamentos de R para obtener más información sobre los paquetes de R.
pacman::p_load(
tidyverse, # gestión y visualización de datos
UpSetR, # paquete especial para gráficos combinados
ggupset) # paquete especial para gráficos combinadosImportar datos
Para empezar, importamos la lista de casos limpia de una epidemia de ébola simulada. Si quieres seguir el proceso, clica aquí para descargar linelist “limpio”, (como archivo .rds). Importa los datos con la función import() del paquete rio (maneja muchos tipos de archivos como .xlsx, .csv, .rds - ver la página de importación y exportación para más detalles).
# importar linelist de casos
linelist_sym <- import("linelist_cleaned.rds")Linelist incluye cinco variables “yes/no” sobre los síntomas declarados. Tendremos que transformar un poco estas variables para utilizar el paquete ggupset para hacer nuestro gráfico. Para ver los datos desplázate a la derecha para ver las variables de los síntomas).
Reformular los valores
Para alinearse con el formato esperado por ggupset, convertimos el “yes” y el “no” en el nombre real del síntoma, utilizando case_when() de dplyr. Si “no”, establecemos el valor en blanco, por lo que los valores son NA o el síntoma.
# crear columna con los síntomas nombrados, separados por punto y coma
linelist_sym_1 <- linelist_sym %>%
# convertir los valores "yes" y "no " en el propio nombre del síntoma
# si el valor anterior es "yes", el nuevo valor es "fever", de lo contrario se establece como faltante (NA)
mutate(fever = ifelse(fever == "yes", "fever", NA),
chills = ifelse(chills == "yes", "chills", NA),
cough = ifelse(cough == "yes", "cough", NA),
aches = ifelse(aches == "yes", "aches", NA),
vomit = ifelse(vomit == "yes", "vomit", NA))Ahora hacemos dos columnas finales:
- Concatenando (pegar) todos los síntomas del paciente (una columna de caracteres)
- Conviertiendo la columna anterior en una de tipo list, para que pueda ser aceptada por ggupset para hacer la trama
Consulta la página sobre Caracteres y cadenas para saber más sobre la función unite() de stringr
linelist_sym_1 <- linelist_sym_1 %>%
unite(col = "all_symptoms",
c(fever, chills, cough, aches, vomit),
sep = "; ",
remove = TRUE,
na.rm = TRUE) %>%
mutate(
# crea una copia de la columna all_symptoms, pero de clase "list" (necesaria para usar ggupset() en el siguiente paso)
all_symptoms_list = as.list(strsplit(all_symptoms, "; "))
)En los datos nuevos observa las dos columnas del extremo derecho: los valores combinados pegados y la lista
36.2 ggupset
Carga el paquete
pacman::p_load(ggupset)Crear el gráfico. Comenzamos con ggplot() y geom_bar(), pero luego añadimos la función especial scale_x_upset() de ggupset.
ggplot(
data = linelist_sym_1,
mapping = aes(x = all_symptoms_list)) +
geom_bar() +
scale_x_upset(
reverse = FALSE,
n_intersections = 10,
sets = c("fever", "chills", "cough", "aches", "vomit"))+
labs(
title = "Signs & symptoms",
subtitle = "10 most frequent combinations of signs and symptoms",
caption = "Caption here.",
x = "Symptom combination",
y = "Frequency in dataset")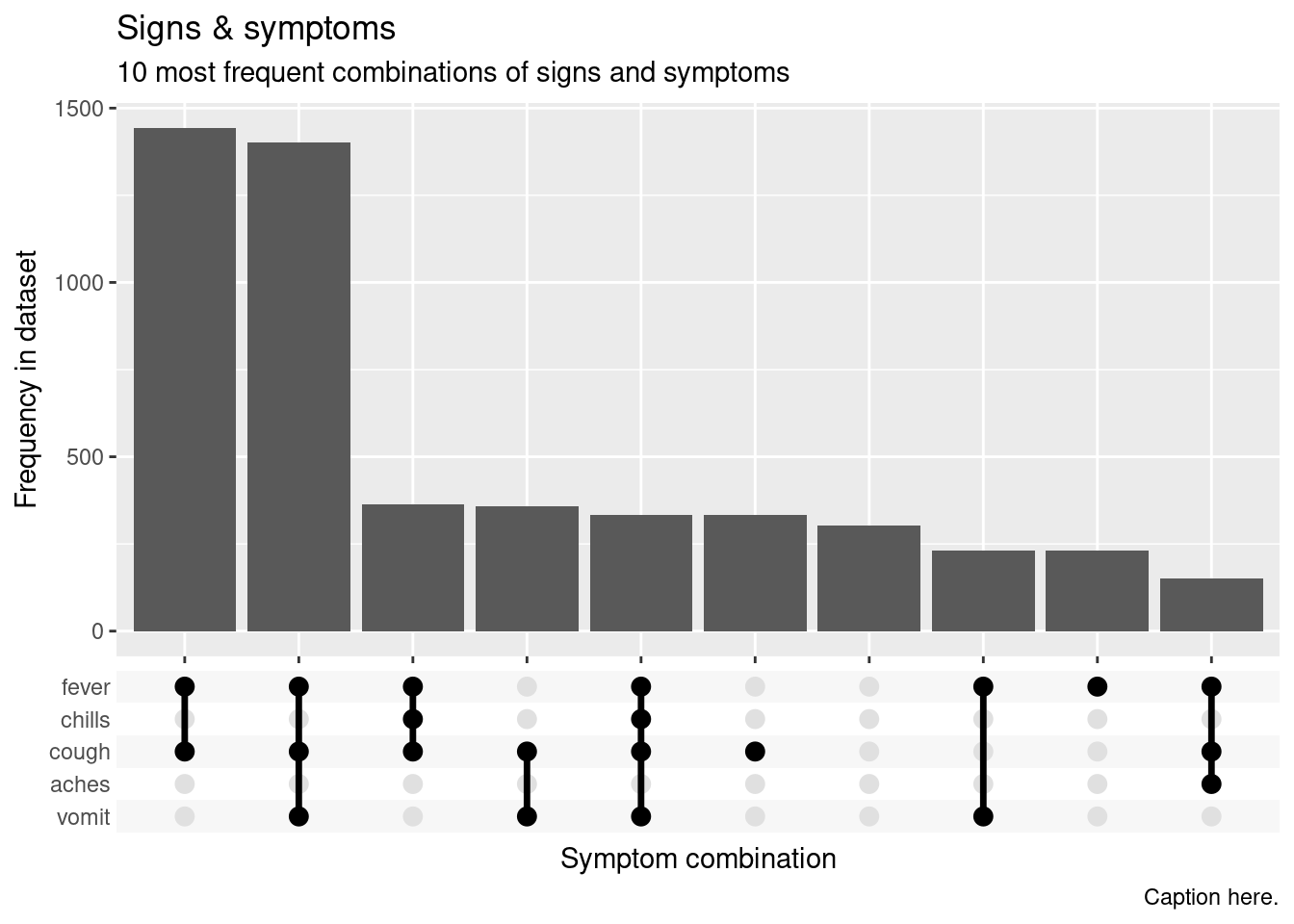
Puedes encontrar más información sobre ggupset en línea o fuera de línea en la documentación del paquete en su pestaña de Ayuda de RStudio ?ggupset.
36.3 UpSetR
El paquete UpSetR permite una mayor personalización del gráfico, pero puede ser más difícil de ejecutar:
Cargar paquete
pacman::p_load(UpSetR)Limpieza de datos
Debemos convertir los valores de los síntomas de linelist en 1 / 0.
linelist_sym_2 <- linelist_sym %>%
# convierte los valores "yes" y "no" en 1s y 0s
mutate(fever = ifelse(fever == "yes", 1, 0),
chills = ifelse(chills == "yes", 1, 0),
cough = ifelse(cough == "yes", 1, 0),
aches = ifelse(aches == "yes", 1, 0),
vomit = ifelse(vomit == "yes", 1, 0))Si está interesado en un comando más eficiente, puede aprovechar la función +(), que convierte en 1s y 0s basándose en una sentencia lógica. Este comando utiliza la función across() para cambiar varias columnas a la vez (lea más en Limpieza de datos y funciones básicas).
# Convierte eficazmente "yes" en 1 y 0
linelist_sym_2 <- linelist_sym %>%
# convierte los valores "yes" y "no" en 1s y 0s
mutate(across(c(fever, chills, cough, aches, vomit), .fns = ~+(.x == "yes")))Ahora haz el gráfico usando la función personalizada upset() - utilizando sólo las columnas de síntomas. Debes designar qué “conjuntos” comparar (los nombres de las columnas de síntomas). Alternativamente, utiliza nsets = y order.by = "freq" para mostrar sólo las X combinaciones principales.
# Crea el gráfico
linelist_sym_2 %>%
UpSetR::upset(
sets = c("fever", "chills", "cough", "aches", "vomit"),
order.by = "freq",
sets.bar.color = c("blue", "red", "yellow", "darkgreen", "orange"), # optional colors
empty.intersections = "on",
# nsets = 3,
number.angles = 0,
point.size = 3.5,
line.size = 2,
mainbar.y.label = "Symptoms Combinations",
sets.x.label = "Patients with Symptom")