28 Conceptos básicos de los SIG
28.1 Resumen
Los aspectos espaciales de tus datos pueden proporcionar mucha información sobre la situación del brote, y responder a preguntas como:
- ¿Dónde están los focos actuales de la enfermedad?
- ¿Cómo han cambiado los puntos conflictivos con el tiempo?
- ¿Cómo es el acceso a las instalaciones sanitarias? ¿Se necesitan mejoras?
El enfoque actual de esta página de los SIG (Sistemas de información geográfico; GIS, por sus siglas en inglés) es abordar las necesidades de la epidemiología aplicada en su respuesta a los brotes. Exploraremos los métodos básicos de visualización de datos espaciales utilizando los paquetes tmap y ggplot2. También recorreremos algunos de los métodos básicos de gestión y consulta de datos espaciales con el paquete sf. Por último, abordaremos brevemente conceptos de estadística espacial como las relaciones espaciales, la autocorrelación espacial y la regresión espacial utilizando el paquete spdep.
28.2 Términos clave
A continuación presentamos algunos términos clave. Para una introducción completa a los SIG y al análisis espacial, te sugerimos que revises uno de los tutoriales o cursos más largos que aparecen en la sección de Recursos.
Sistemas de Información Geográfica (SIG) - Un SIG es un marco o entorno de trabajo para recopilar, gestionar, analizar y visualizar datos espaciales.
Software SIG
Algunos de los programas de SIG más conocidos permiten la interacción “señalar y clicar” para el desarrollo de mapas y el análisis espacial. Estas herramientas tienen ventajas como no tener que aprender código y la facilidad de seleccionar y colocar manualmente los iconos y características en un mapa. He aquí dos de los más populares:
ArcGIS - Un software comercial de SIG desarrollado por la empresa ESRI, que es muy popular pero bastante caro.
QGIS - Un software de SIG gratuito de código abierto que puede hacer casi todo lo que ArcGIS puede hacer. Puedes descargar QGIS aquí
El uso de R como SIG puede parecer más intimidante al principio porque, en lugar de “señalar y clicar”, tiene una “interfaz de línea de comandos” (hay que codificar para adquirir el resultado deseado). Sin embargo, esto es una gran ventaja si se necesita producir mapas repetidamente o crear un análisis que sea reproducible.
Datos espaciales
Las dos formas principales de datos espaciales utilizadas en los SIG son los datos vectoriales y los ráster:
Datos vectoriales - Es el formato más común de datos espaciales utilizado en los SIG. Los datos vectoriales se componen de características geométricas de vértices y trayectorias. Los datos espaciales vectoriales pueden dividirse a su vez en tres tipos ampliamente utilizados:
Puntos - Un punto consiste en un par de coordenadas (x,y) que representan una ubicación específica en un sistema de coordenadas. Los puntos son la forma más básica de datos espaciales, y pueden utilizarse para denotar un caso (por ejemplo, el domicilio de un paciente) o una ubicación (por ejemplo, un hospital) en un mapa.
Líneas - Una línea está compuesta por dos puntos conectados. Las líneas tienen una longitud y pueden utilizarse para indicar cosas como carreteras o ríos.
Polígonos - Un polígono está compuesto por al menos tres líneas conectadas por puntos. Los polígonos tiene una longitud (es decir, el perímetro del área) así como una medida de área. Los polígonos pueden utilizarse para señalar una zona (por ejemplo, un pueblo) o una estructura (por ejemplo, la superficie real de un hospital).
Datos ráster - Es un formato alternativo para los datos espaciales. Los datos ráster son una matriz de celdas (por ejemplo, píxeles) en la que cada celda contiene información como la altura, la temperatura, la pendiente, la cubierta forestal, etc. Suelen ser fotografías aéreas, imágenes de satélite, etc. Las imágenes ráster también pueden utilizarse como “mapas base” debajo de los datos vectoriales.
Visualización de datos espaciales
Para representar visualmente los datos espaciales en un mapa, el software SIG requiere que se proporcione suficiente información sobre dónde deben estar las diferentes características y la relación de unas con otras. Si se utilizan datos vectoriales, como ocurre en la mayoría de los casos, esta información suele almacenarse en un archivo shapefile:
Shapefiles - Un shapefile es un formato de datos común para almacenar datos espaciales “vectoriales” consistentes en líneas, puntos o polígonos. Un shapefile es en realidad un grupo de al menos tres archivos - .shp, .shx y .dbf. Estos archivos deben estar en un determinado directorio (una misma carpeta) para que el shapefile se pueda leer. Estos archivos asociados pueden comprimirse en un archivo ZIP para enviarlos por correo electrónico o descargarlos de un sitio web.
El shapefile contendrá información sobre las características con las que estemos tratando, así como su ubicación en la superficie de la Tierra. Esto es importante porque, aunque la Tierra es un globo terráqueo, los mapas suelen ser bidimensionales; las decisiones sobre cómo “aplanar” los datos espaciales pueden tener un gran impacto en el aspecto y la interpretación del mapa resultante.
Sistemas de referencia de coordenadas (CRS-SRC) - Un SRC es un sistema basado en coordenadas que se utiliza para localizar accidentes geográficos en la superficie de la Tierra. Tiene unos cuantos componentes clave:
Sistema de coordenadas - Hay muchos sistemas de coordenadas diferentes, así que asegúrate de saber en qué sistema están tus coordenadas. Los grados de latitud/longitud son muy comunes, pero también puede que tu información esté guardada como coordenadas UTM.
Units - Asegúrate de saber cuáles son las unidades del sistema de coordenadas (por ejemplo, grados decimales, metros).
Datum - Es una versión particular modelada de la Tierra. Los datum ya estan establecidos y han sido revisados a lo largo de los años, así que asegúrate de que las capas de tu mapa utilizan el mismo datum.
Proyección - Es la referencia a la ecuación matemática que se utilizó para proyectar la tierra (realmente redonda) sobre una superficie plana (mapa).
Recuerda que puedes resumir los datos espaciales sin utilizar las herramientas cartográficas que se muestran a continuación. A veces basta con una simple tabla por zonas geográficas (por ejemplo, distrito, país, etc.).
28.3 Introducción a los SIG
Hay un par de elementos clave que deberás tener y en los que deberás pensar para hacer un mapa. Entre ellos están:
Datos: pueden estar con un formato de datos espaciales (como los shapefiles, como se ha indicado anteriormente) o pueden no estar en un formato espacial (por ejemplo, sólo como un csv).
Si tus datos no están en formato espacial, también necesitarás unos datos de referencia. Los datos de referencia consisten en la representación espacial de los datos y sus atributos relacionados, que incluirían el material que contiene la información de ubicación y dirección de características específicas.
Si estás trabajando con límites geográficos predefinidos (por ejemplo, regiones administrativas), probablemente puedas encontrar shapefiles de referencia que te puedes descargar de forma gratuita desde una agencia gubernamental u organización de intercambio de datos. En caso de duda, un buen punto de partida es buscar en Google “[regions] shapefile”
Si tus datos están guardados como direcciones, en vez de como latitud/longitud, puede que tengas que utilizar un motor de geocodificación que te permita hacer la “traducción” de una dirección específica a a datos en formato espacial.
Piensa cómo quieres presentar la información de los datos a tu audiencia. Hay muchos tipos diferentes de mapas, y es importante pensar qué tipo de mapa se ajusta mejor a tus necesidades.
Tipos de mapas para visualizar tus datos
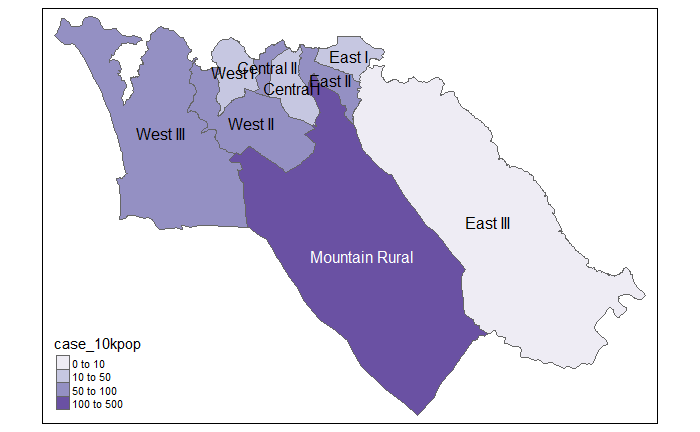
Mapa de coropletas: Es un tipo de mapa temático en el que se utiliza colores, sombreados o patrones para representar regiones geográficas en relación con el valor de un atributo. Por ejemplo, un valor mayor podría indicarse con un color más oscuro que un valor menor. Este tipo de mapa es particularmente útil cuando se visualiza una variable y cómo cambia a través de regiones o áreas geopolíticas definidas.
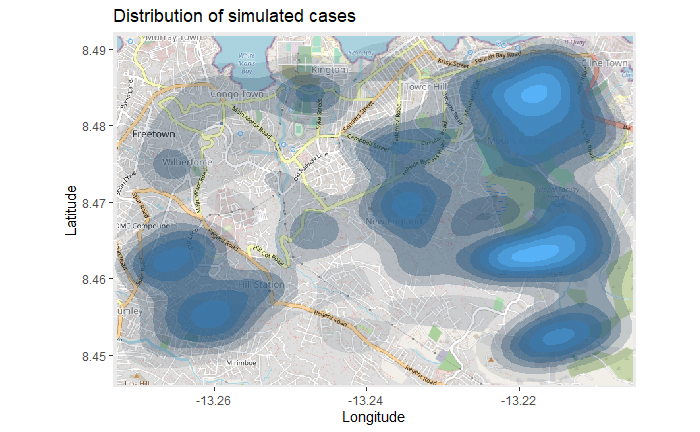
Mapa de densidad de calor de casos: Es un tipo de mapa temático en el que se utiliza colores para representar la intensidad de un valor, pero que no utiliza regiones definidas ni límites geopolíticos para agrupar los datos. Este tipo de mapa se suele utilizar para mostrar “puntos conflictivos” o zonas con una alta densidad o concentración de puntos.
Mapa de densidad de puntos: Es un tipo de mapa temático que utiliza puntos para representar los valores de los datos. Este tipo de mapa es el más adecuado para visualizar cómo están dispersos los datos en el mapa y buscar clústers visualmente.
Mapa de símbolos proporcionales (mapa de símbolos graduados): es un mapa temático similar a un mapa de coropletas, pero en lugar de utilizar el color para indicar el valor de un atributo, utiliza un símbolo (normalmente un círculo) cuyo tamaño esta en relación con el valor. Por ejemplo, un valor mayor podría indicarse con un símbolo de tamaño mayor que un valor menor. Este tipo de mapa se utiliza mejor cuando se quiere visualizar el tamaño o la cantidad de los datos en distintas regiones geográficas.
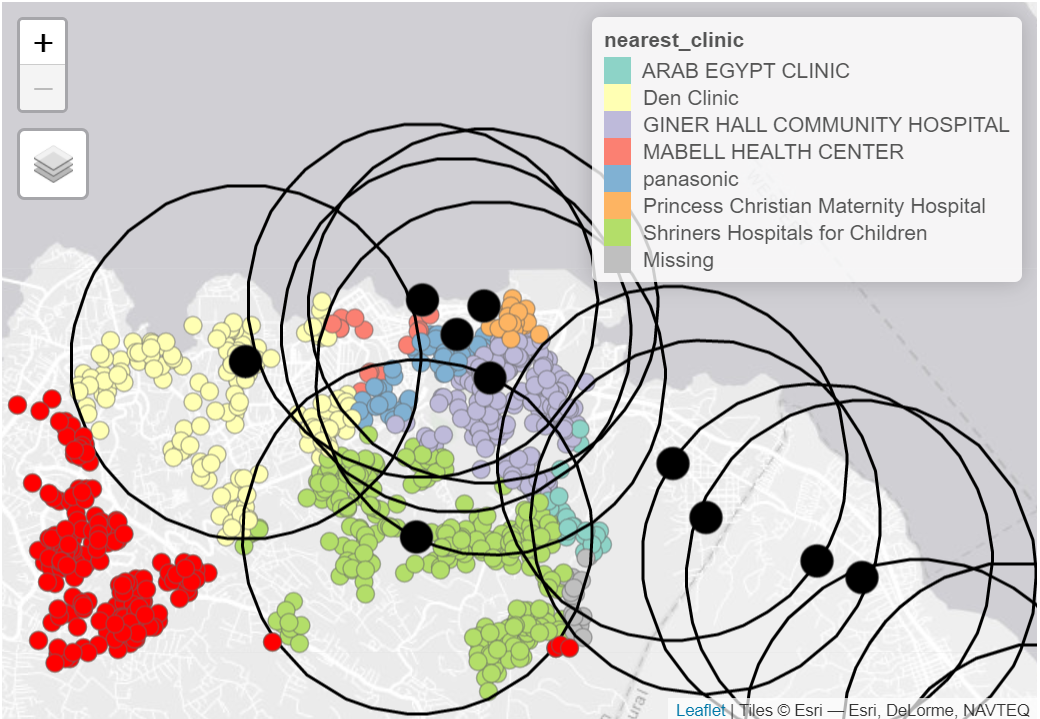
También puedes combinar varios tipos de visualizaciones diferentes para mostrar patrones geográficos complejos. Por ejemplo, los casos (puntos) del siguiente mapa están coloreados según su centro sanitario más cercano (véase la leyenda). Los círculos grandes de color negro muestran las zonas de captación de los centros sanitarios de un determinado radio, y los puntos/círculos rojos brillantes son los que no tienen ninguna zona de captación de centros sanitarios dentro de ese radio determinado:
Nota: El enfoque principal de esta página del SIG se centra en el contexto de respuesta a brotes en el terreno. Por lo tanto, el contenido de esta página cubrirá la manipulación, visualización y análisis básicos de datos espaciales.
28.4 Preparación
Cargar paquetes
Este trozo de código muestra cómo puedes cargar de los paquetes necesarios para el análisis espacial que vamos a realizar. En este manual destacamos la función p_load() del paquete pacman: esta instala el paquete (si aún no está instalado) y lo carga para su uso. También puedes cargar los paquetes uno por uno con la funcion library() de R base., si ya los tienes instalados. Consulta la página sobre los Fundamentos de R para obtener más información sobre los paquetes de R.
pacman::p_load(
rio, # para importar datos
here, # para localizar archivos
tidyverse, # para limpiar, manejar y graficar los datos (incluye el paquete ggplot2)
sf, # para manejar datos espaciales usando el formato Simple Feature
tmap, # para producir mapas simples, funciona tanto para mapas interactivos como estáticos
janitor, # para limpiar los nombres de las columnas
OpenStreetMap, # para añadir el mapa base OSM en el mapa ggplot
spdep # estadísticas espaciales
) En este enlace de CRAN “Spatial Task View” puedes ver un resumen de todos los paquetes de R que pueden trabajar con datos espaciales.
Ejemplos con una base de datos
A muestra de ejemplo, trabajaremos con una muestra aleatoria de 1000 casos del brote de ébola simulado en el dataframe linelist (computacionalmente, una base de datos pequeña hace que sea más fácil trabajar con este ejemplo). Si quieres seguir el proceso, clica para descargar linelist “limpio” (como archivo .rds).
Los resultados que tengas al correr el código de este ejercicio puede que sean un poco diferentes de los que se aquí mostramos. Esto es porque vamos a estar trabajando con una muestra aleatoria de los casos.
Importa los datos con la función import() del paquete rio (este paquete puede manejar muchos tipos de archivos, como .xlsx, .csv, .rds - véase la página de importación y exportación para más detalles).
# importar linelist limpio
linelist <- import("linelist_cleaned.rds") Para seleccionar la muestra aleatoria de 1000 filas utiliza sample() de R base.
# genera 1000 números de fila aleatorios, a partir del número de filas de linelist
sample_rows <- sample(nrow(linelist), 1000)
# subconjunto de linelist para mantener sólo las filas de muestra, y todas las columnas
linelist <- linelist[sample_rows,]Ahora queremos convertir este linelist, que es de tipo “dataframe”, en un objeto de tipo “sf” (spatial features, por sus siglas en inglés). Dado que linelist tiene dos columnas “lon” y “lat” que representan la longitud y latitud de la residencia de cada caso, esto será fácil.
Utilizamos la función st_as_sf() del paquete sf (spatial features) para crear el nuevo objeto que llamamos linelist_sf. Este nuevo objeto tiene el mismo aspecto que la linelist, pero las columnas lon y lat han sido designadas como columnas de coordenadas, y se ha asignado un sistema de referencia de coordenadas (CRS) para poder visualizar los puntos. Este CRS es el númermo 4326 que corresponde a coordenadas especificas basadas en el Sistema Geodésico Mundial 1984 (WGS84) - que es el estándar para las coordenadas GPS.
Este es el aspecto del dataframe original linelist. En esta demostración, sólo utilizaremos la columna date_onset y geometry (que se construyó a partir de los campos de longitud y latitud anteriores y es la última columna del dataframe).
Archivos shapefiles de límites administrativos
Sierra Leona: Archivos shapefiles de los límites administrativos
Primero, descárgate todos los límites administrativos de Sierra Leona del sitio web de Humanitarian Data Exchange (HDX). Como alternativa, también te puedes descargar estos archivos y todos los demás datos de ejemplo que usamos para este manual a través de nuestro paquete R, como se explica en la página descargando el manual y los datos.
Ahora vamos a hacer lo siguiente para guardar el shapefile del nivel administrativo 3 en R:
- Importar el shapefile
- Limpiar los nombres de las columnas
- Filtrar las filas para mantener sólo las áreas de interés
Para importar un shapefile utilizamos la función read_sf() del paquete sf, y usamos la función here() para que R encuentre el archivo. En nuestro caso, el archivo se encuentra dentro de nuestro proyecto R en las subcarpetas “data”, “gis” y “shp”, con nombre de archivo “sle_adm3.shp” (para más información ver las páginas sobre Importación y exportación y Proyectos en R). En tu caso, tendrás que indicar la localización específica donde tienes tus archivos en tu ordenador.
A continuación utilizamos clean_names() del paquete janitor para estandarizar los nombres de las columnas del shapefile. También utilizamos filter() para mantener sólo las filas con admin2name de “Western Area Urban” o “Western Area Rural”.
# Limpieza de nivel ADM3
sle_adm3 <- sle_adm3_raw %>%
janitor::clean_names() %>% # estandariza los nombres de las columnas
filter(admin2name %in% c("Western Area Urban", "Western Area Rural")) # filter to keep certain areasA continuación puedes ver el aspecto del shapefile después de la importación y limpieza. Desplázate a la derecha para ver cómo hay columnas con el nivel de administración 0 (país), el nivel de administración 1, el nivel de administración 2 y, finalmente, el nivel de administración 3. Cada nivel tiene un nombre y un identificador único “pcode”. El pcode se expande con cada nivel de administración creciente, por ejemplo, SL (Sierra Leona) -> SL04 (Occidental) -> SL0410 (Zona Occidental Rural) -> SL040101 (Koya Rural).
Datos de población
Sierra Leona: Población por ADM3
Al igual que hemos hecho antes, te puedes descargar estos datos
de HDX) o a través de nuestro paquete R epirhandbook como se explica en esta página. Utilizamos import() para cargar el archivo .csv. Al igual que antes, pasamos el archivo importado a clean_names() para estandarizar la sintaxis de los nombres de las columnas.
# Población del nivel ADM3
sle_adm3_pop <- import(here("data", "gis", "population", "sle_admpop_adm3_2020.csv")) %>%
clean_names() Este es el aspecto del archivo de población. Desplázate a la derecha para ver cómo cada jurisdicción tiene columnas con la población male, female, y la población total y el desglose de la población en columnas por grupos de edad.
Instalaciones sanitarias
Sierra Leone: Health facility data from OpenStreetMap
Una vez más, hemos descargado la localización de los centros sanitarios desde el HDX aquí o mediante las instrucciones de la página descarga de manuales y datos.
Importamos el shapefile de puntos de las instalaciones con read_sf(), limpiamos de nuevo los nombres de las columnas y filtramos para mantener sólo los puntos etiquetados como “hospital”, “clinic” o “doctors”.
# Archivo shapefile OSM de centros sanitarios
sle_hf <- sf::read_sf(here("data", "gis", "shp", "sle_hf.shp")) %>%
clean_names() %>%
filter(amenity %in% c("hospital", "clinic", "doctors"))Aquí está el dataframe resultante – desplázate a la derecha para ver el nombre de la instalación y las coordenadas geométricas (geometry).
28.5 Trazado de coordenadas
La forma más sencilla de representar las coordenadas X-Y (longitud/latitud, puntos) de los casos es dibujarlas como puntos directamente desde el objeto linelist_sf que ya creamos en la sección de preparación.
El paquete tmap nos permite visualizar este tipo de informacion tanto de modo estático (modo “plot”) como de modo interactivo (modo “view”) con sólo unas pocas líneas de código. La sintaxis de tmap es similar a la de ggplot2, de forma que los comandos se añaden unos a otros con +. Lee más detalles en esta viñeta.
- Lo primero es establecer qué modo tmap queremos. En este caso utilizaremos el modo “plot”, que produce salidas estáticas.
tmap_mode("plot") # choose either "view" or "plot"Abajo puedes ver que, por ahora, sólo estamos trazando los puntos. Utilizamos el tm_shape() con el objeto linelist_sf. A continuación, añadimos la informacion de los puntos mediante tm_dots(), especificando el tamaño y el color deseados. Recuerda que el linelist_sf es un objeto sf, con lo que ya tenemos designadas las dos columnas que contienen las coordenadas lat/long y el sistema de referencia de coordenadas (CRS):
# Sólo los casos (puntos)
tm_shape(linelist_sf) + tm_dots(size=0.08, col='blue')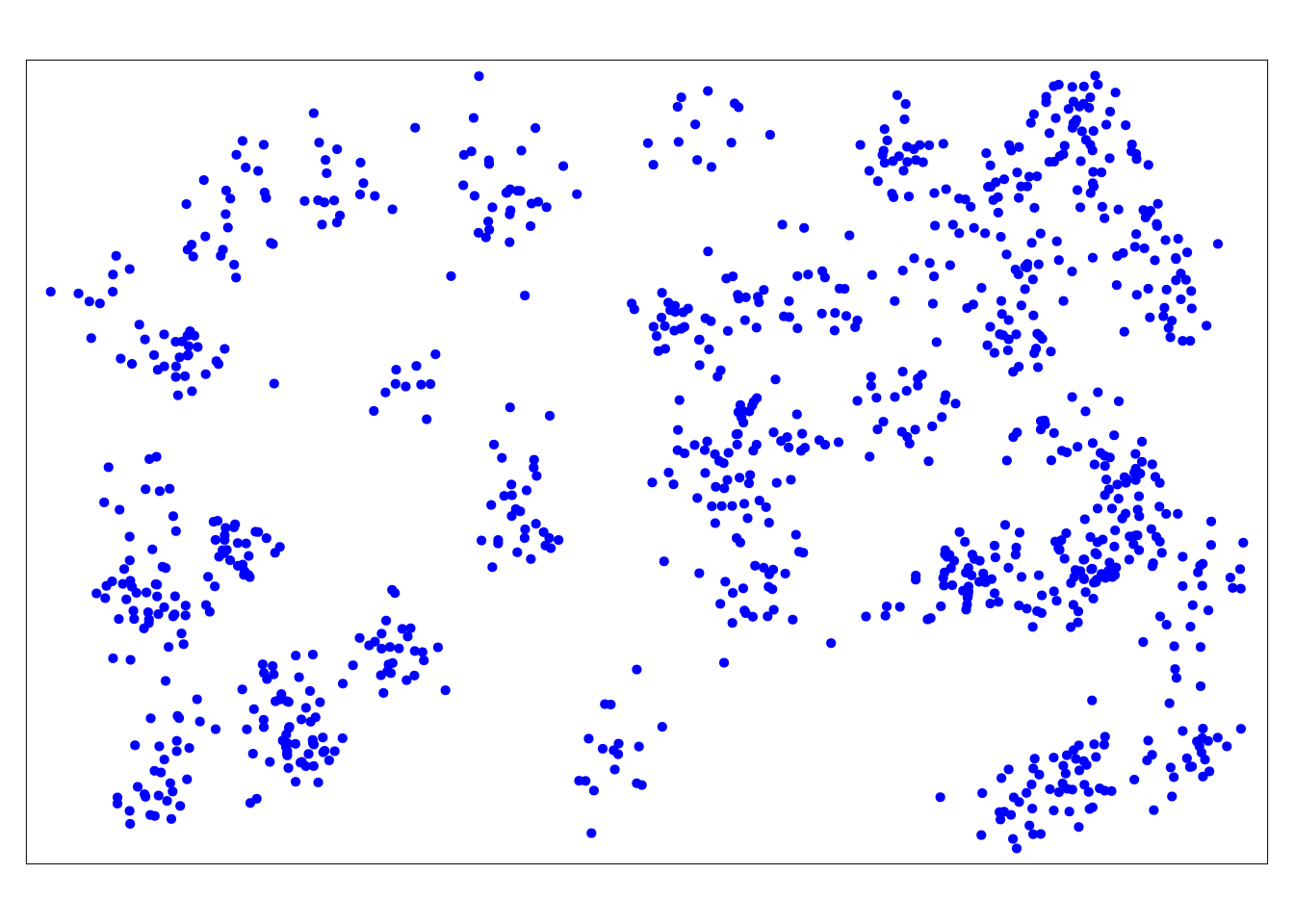
Por sí solos, los puntos no nos dicen mucho. Así que también tenemos que dibujar los límites administrativos:
Para esto, vamos a usar tm_shape() (documentación), pero en vez de proporcionar el shapefile de los puntos de los casos, proporcionamos el shapefile de los límites administrativos (polígonos).
Con el argumento bbox =(bbox significa “bounding box”) podemos especificar los límites de las coordenadas, lo cual nos ayuda a hacer zoom a una zona específica de interés. Primero mostramos la visualización del mapa sin bbox, y luego con él.
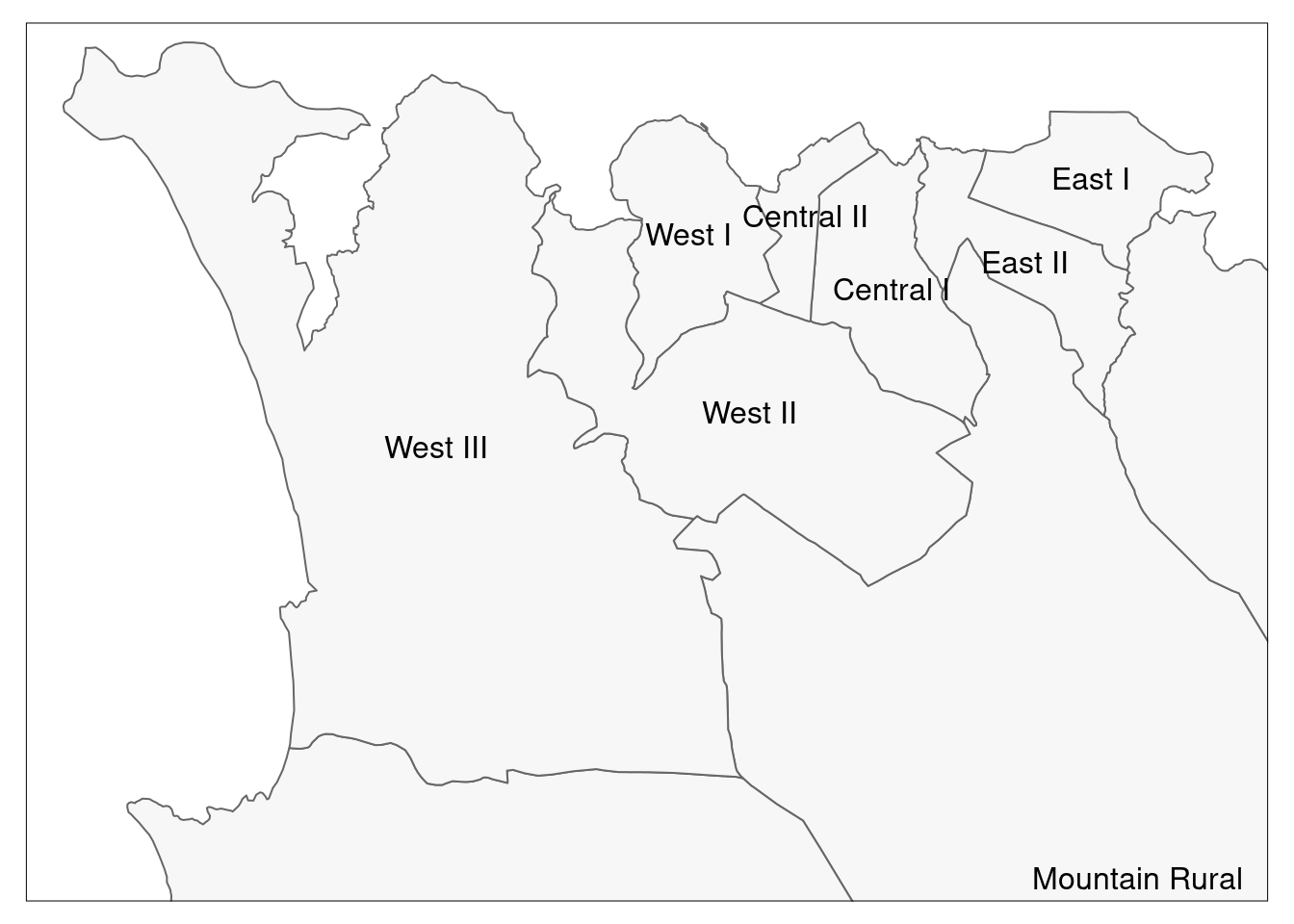
# Sólo los límites administrativos (polígonos)
tm_shape(sle_adm3) + # shapefile de límites administrativos
tm_polygons(col = "#F7F7F7")+ # muestra los polígonos en gris claro
tm_borders(col = "#000000", # muestra las fronteras con color y línea gruesa
lwd = 2) +
tm_text("admin3name") # texto de columna a mostrar para cada polígono
# Same as above, but with zoom from bounding box
tm_shape(sle_adm3,
bbox = c(-13.3, 8.43, # esquina
-13.2, 8.5)) + # esquina opuesta
tm_polygons(col = "#F7F7F7") +
tm_borders(col = "#000000", lwd = 2) +
tm_text("admin3name")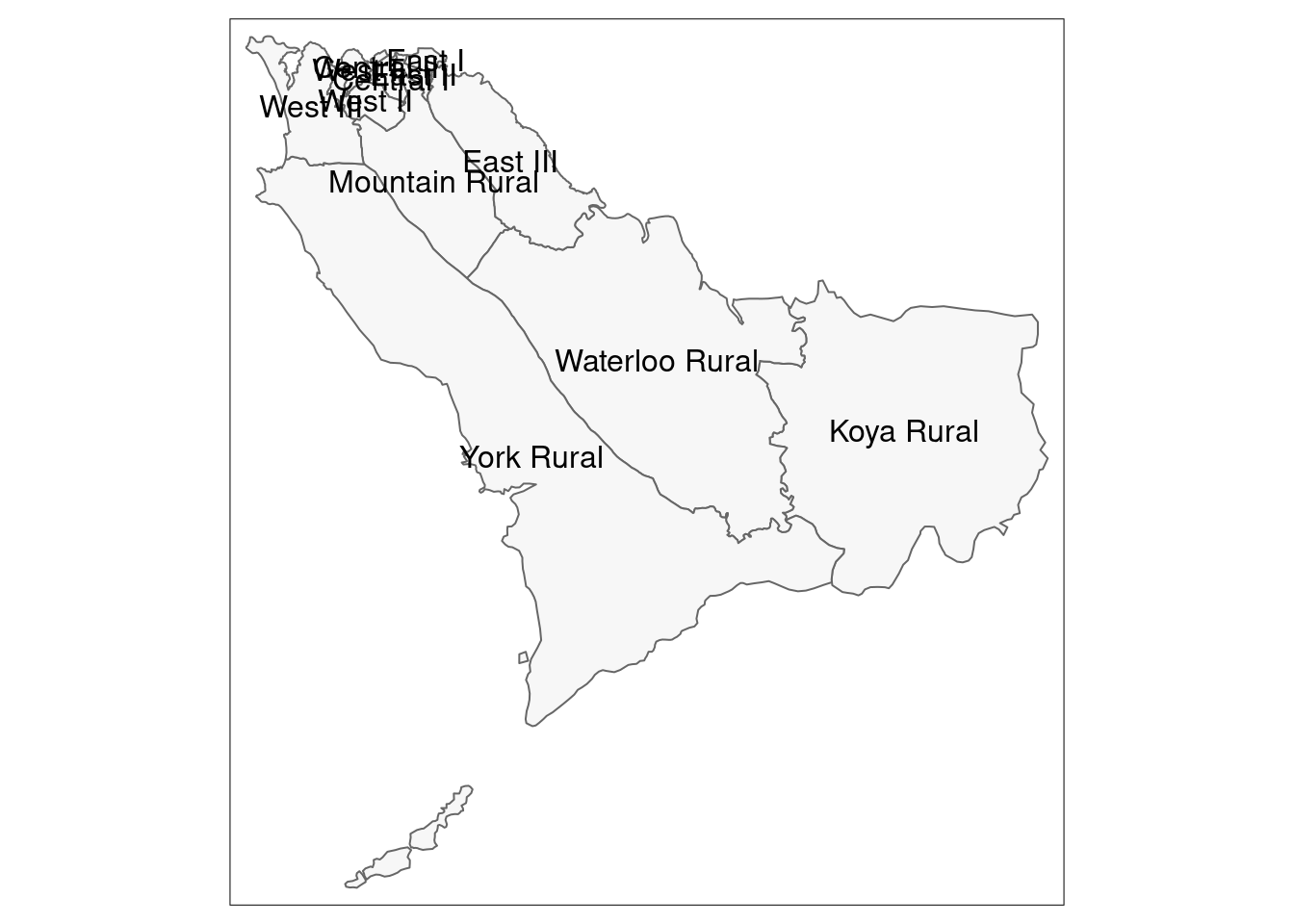
Con esto ya podemos trazar los puntos y los polígonos juntos:
# Todo junto
tm_shape(sle_adm3, bbox = c(-13.3, 8.43, -13.2, 8.5)) + #
tm_polygons(col = "#F7F7F7") +
tm_borders(col = "#000000", lwd = 2) +
tm_text("admin3name")+
tm_shape(linelist_sf) +
tm_dots(size=0.08, col='blue', alpha = 0.5) +
tm_layout(title = "Distribution of Ebola cases") # da título al mapa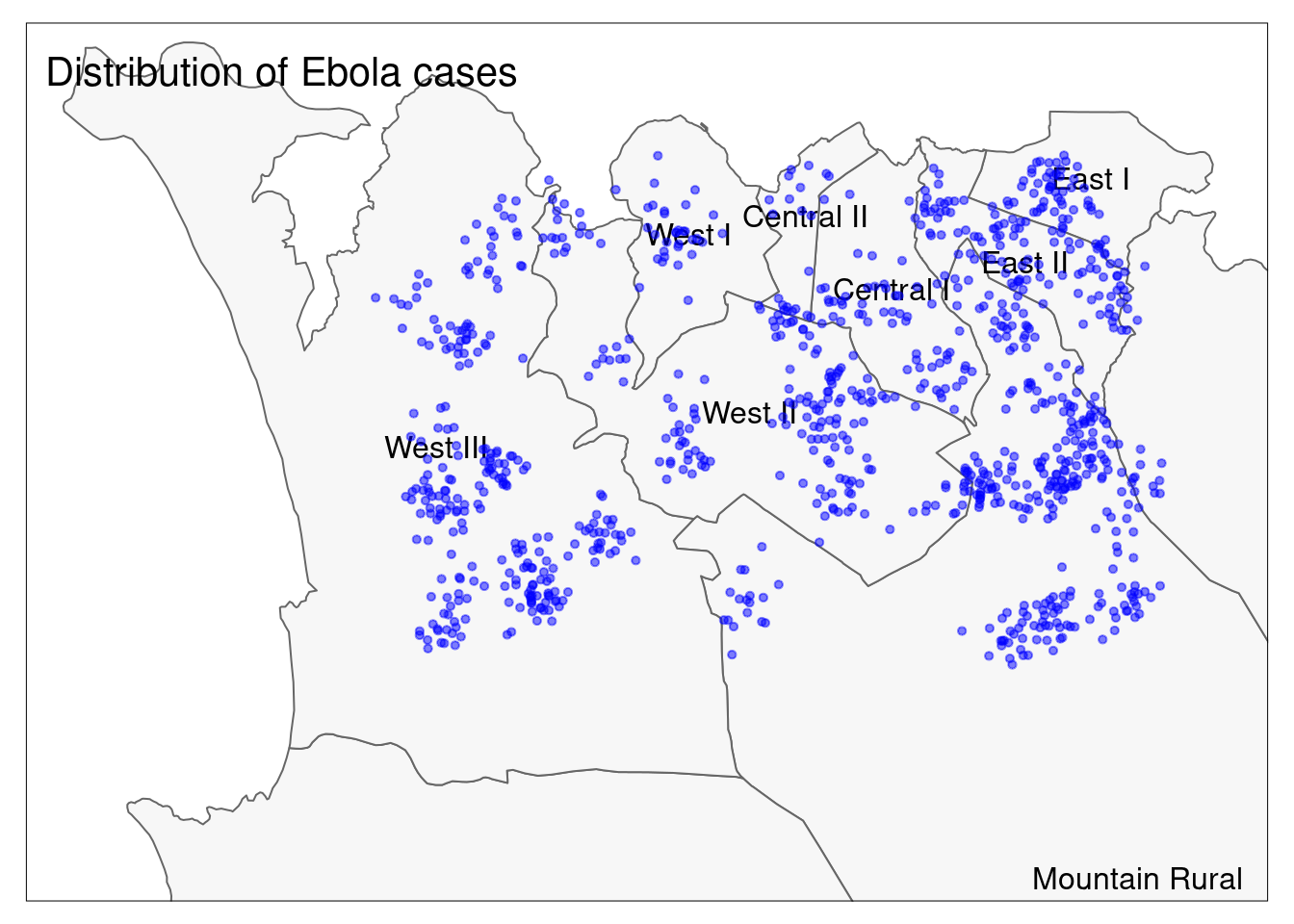
Para leer una buena comparación de las opciones de mapeo en R, consulta esta entrada del blog.
28.6 Uniones espaciales
Es posible que estés familiarizado/a con la unión de datos entre bases de datos. En la página unión de datos de este manual se trata varios métodos para unir bases de datos. Una unión espacial tiene un propósito similar, pero aprovecha las relaciones espaciales entre bases de datos. En lugar de confiar en los valores comunes de las columnas para hacer coincidir correctamente las observaciones, puede utilizar distintos tipos de relaciones espaciales, como por ejemplo que una característica esté contenida dentro de otra, que sea el “vecino más cercano” de otra característica, o que esté dentro de un buffer de determinado radio de otra.
El paquete sf ofrece varios métodos para las uniones espaciales. Puedes explorar la documentación del método st_join() y otros tipos de uniones espaciales en esta referencia.
Puntos en el polígono
Asignación espacial de unidades administrativas a los casos
Aquí se plantea un dilema interesante: la lista de casos no contiene ninguna información sobre las unidades administrativas de los mismos. Aunque lo ideal es recoger dicha información durante la fase inicial de recogida de datos, también podemos asignar unidades administrativas a los casos individuales basándonos en sus relaciones espaciales (es decir, el punto se cruza con un polígono).
A continuación, haremos una intersección espacial de las ubicaciones de nuestros casos (puntos) con los límites de la ADM3 (polígonos):
- Comenzamos con la linelist (casos/puntos)
- Continuamos con la unión espacial a los límites administrativos, estableciendo el tipo de unión en “st_intersects”
- Utilizamos
select()para mantener sólo algunas de las nuevas columnas de los límites administrativos (este paso lo realizamos un poco más abajo)
linelist_adm <- linelist_sf %>%
# une el fichero de límites administrativos a la lista de casos, basándose en la intersección espacial
sf::st_join(sle_adm3, join = st_intersects)¡Todas las columnas de sle_adms se han añadido a linelist! Cada caso tiene ahora columnas que detallan los niveles administrativos a los que pertenece. En este ejemplo, sólo queremos mantener dos de las nuevas columnas (las de nivel administrativo 3), así que haremos select() de los nombres de las columnas antiguas de la linelist, y sólo las dos adicionales de interés:
linelist_adm <- linelist_sf %>%
# une el fichero de límites administrativos a la lista de casos, basándose en la intersección espacial
sf::st_join(sle_adm3, join = st_intersects) %>%
# Mantiene los antiguos nombres de columna y dos nuevos admin de interés
select(names(linelist_sf), admin3name, admin3pcod)Aquí os mostramos los diez primeros casos para que veais que se ha añadido la información correspondiente de sus jurisdicciones a nivel de administración 3 (ADM3), basándose en la región del polígono donde se cruza el punto.
# Ahora se verán los nombres ADM3 adjuntos a cada caso
linelist_adm %>% select(case_id, admin3name, admin3pcod)## Simple feature collection with 1000 features and 3 fields
## Geometry type: POINT
## Dimension: XY
## Bounding box: xmin: -13.27276 ymin: 8.448185 xmax: -13.20684 ymax: 8.490442
## Geodetic CRS: WGS 84
## First 10 features:
## case_id admin3name admin3pcod geometry
## 2566 692ce9 West III SL040208 POINT (-13.26064 8.452916)
## 5703 06a734 West II SL040207 POINT (-13.23151 8.47192)
## 4993 e8bcf6 West III SL040208 POINT (-13.25903 8.457545)
## 2040 998070 West II SL040207 POINT (-13.23581 8.469372)
## 5198 e81e72 Mountain Rural SL040102 POINT (-13.22054 8.464097)
## 1006 50548e West III SL040208 POINT (-13.25568 8.459691)
## 2247 edf379 West III SL040208 POINT (-13.26688 8.462608)
## 3242 49d337 West I SL040206 POINT (-13.24739 8.482858)
## 205 b55ec2 West II SL040207 POINT (-13.24654 8.465635)
## 1968 5a65bb Mountain Rural SL040102 POINT (-13.21564 8.4629)Ahora podemos describir nuestros casos por unidad administrativa, algo que no podíamos hacer antes de la unión espacial.
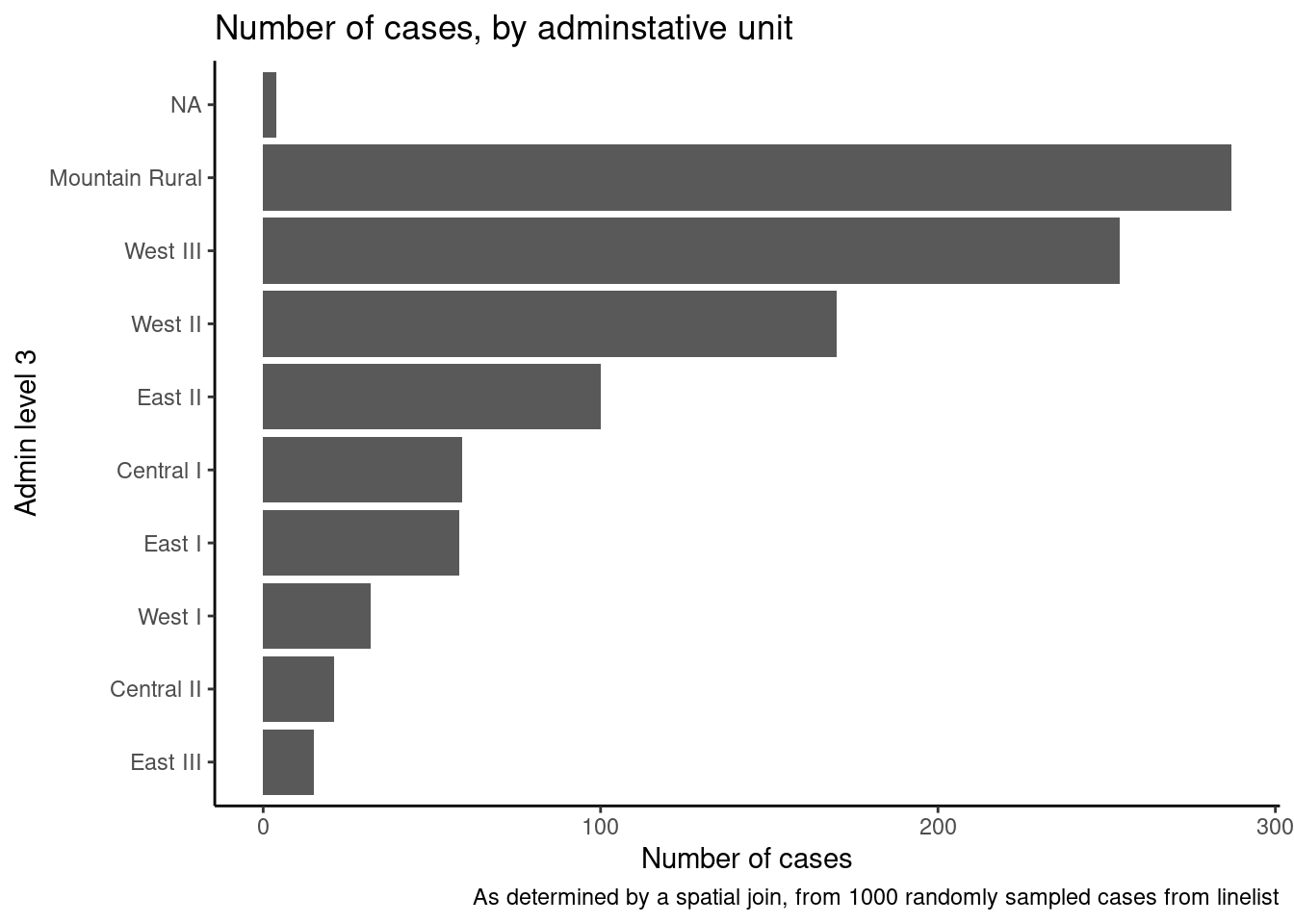
# Crear un nuevo dataframe que contiene los recuentos de casos por unidad administrativa
case_adm3 <- linelist_adm %>% # comienza con linelist con nuevas cols administrativas
as_tibble() %>% # convierte a tibble para una mejor visualización
group_by(admin3pcod, admin3name) %>% # agrupa por unidad administrativa, tanto por nombre como por pcode
summarise(cases = n()) %>% # resumen y recuento de filas
arrange(desc(cases)) # ordena en orden descendente
case_adm3## # A tibble: 10 × 3
## # Groups: admin3pcod [10]
## admin3pcod admin3name cases
## <chr> <chr> <int>
## 1 SL040102 Mountain Rural 287
## 2 SL040208 West III 254
## 3 SL040207 West II 170
## 4 SL040204 East II 100
## 5 SL040201 Central I 59
## 6 SL040203 East I 58
## 7 SL040206 West I 32
## 8 SL040202 Central II 21
## 9 SL040205 East III 15
## 10 <NA> <NA> 4También podemos crear un gráfico de barras con el número de casos por unidad administrativa.
En este ejemplo, utilizamos ggplot() con linelist_adm para poder aplicar funciones que manejan factores, como fct_infreq() que ordena las barras por frecuencia (véase la página sobre Factores para ver algunos consejos).
ggplot(
data = linelist_adm, # comienza con linelist que contiene información de la unidad administrativa
mapping = aes(
x = fct_rev(fct_infreq(admin3name))))+ # el eje-x son unidades admin, ordenadas por frecuencia (invertida)
geom_bar()+ # crea barras, la altura es el número de filas
coord_flip()+ # invierte los ejes X e Y para facilitar la lectura de las unidades adm
theme_classic()+ # simplifica el fondo
labs( # títulos y etiquetas
x = "Admin level 3",
y = "Number of cases",
title = "Number of cases, by adminstative unit",
caption = "As determined by a spatial join, from 1000 randomly sampled cases from linelist"
)
Vecino más cercano
Encontrar el centro sanitario más cercano
Podría ser útil saber dónde se encuentran los centros sanitarios (clínicas/hospitales) en relación con los focos de la enfermedad.
Podemos utilizar el método de unión st_nearest_feature de la función st_join() (paquete sf) para visualizar el centro sanitario más cercano a cada uno de los casos.
- Comenzamos con el shapefile linelist
linelist_sf - Unimos espacialmente con
sle_hf, que es la ubicación de los centros sanitarios y las clínicas (puntos), estableciendo el tipo de unión en “st_nearest_feature”
# Centro sanitario más cercano a cada caso
linelist_sf_hf <- linelist_sf %>% # comienza con el shapefile delinelist
st_join(sle_hf, join = st_nearest_feature) %>% # datos de la clínica más cercana unidos a los datos del caso
select(case_id, osm_id, name, amenity) %>% # mantiene las columnas de interés, incluyendo id, nombre, tipo y geometría del centro sanitario
rename("nearest_clinic" = "name") # renombra para mayor claridadPodemos ver a continuación (primeras 50 filas) que cada caso tiene ahora datos sobre la clínica/hospital más cercano
Podemos ver que “Den Clinic” es el centro sanitario más cercano para aproximadamente el 30% de los casos.
# Contar casos por centro sanitario
hf_catchment <- linelist_sf_hf %>% # comienza con linelist incluyendo datos de la clínica más cercana
as.data.frame() %>% # convierte de shapefile a dataframe
count(nearest_clinic, # cuenta filas por "nombre" (de la clínica)
name = "case_n") %>% # asigna nueva columna de recuento como "case_n"
arrange(desc(case_n)) # ordena en orden descendente
hf_catchment # imprime en la consola## nearest_clinic case_n
## 1 Den Clinic 370
## 2 Shriners Hospitals for Children 341
## 3 GINER HALL COMMUNITY HOSPITAL 154
## 4 panasonic 50
## 5 Princess Christian Maternity Hospital 35
## 6 ARAB EGYPT CLINIC 21
## 7 <NA> 15
## 8 MABELL HEALTH CENTER 14Para visualizar los resultados, podemos utilizar tmap - esta vez en modo interactivo para facilitar la visualización
tmap_mode("view") # establece el modo tmap en interactivo
# representar los casos y los puntos de las clínicas
tm_shape(linelist_sf_hf) + # representa los casos
tm_dots(size=0.08, # casos coloreados por la clínica más cercana
col='nearest_clinic') +
tm_shape(sle_hf) + # puntos negros grandes para las clínicas
tm_dots(size=0.3, col='black', alpha = 0.4) +
tm_text("name") + # se superpone el nombre de la clínica
tm_view(set.view = c(-13.2284, 8.4699, 13), # ajusta el zoom (coordenadas centrales, zoom)
set.zoom.limits = c(13,14))+
tm_layout(title = "Cases, colored by nearest clinic")Buffers
También podemos explorar cuántos casos se encuentran a menos de 2,5 km (~30 minutos) de distancia a pie del centro sanitario más cercano. Esto se conoce como buffer o zona o ámbito de influencia.
Nota: Para un cálculo más preciso de la distancia, es mejor reproyectar el objeto sf al respectivo sistema de proyección cartográfica local, como UTM (Tierra proyectada sobre una superficie plana). En este ejemplo, para simplificar, nos ceñiremos al sistema de coordenadas geográficas del Sistema Geodésico Mundial (WGS84) (la Tierra representada en una superficie esférica/redonda, por lo que las unidades están en grados decimales). Utilizaremos una conversión general de: 1 grado decimal = ~111km.
Puedes ver más información sobre proyecciones cartográficas y sistemas de coordenadas en este artículo de esri. Este blog habla de los diferentes tipos de proyecciones cartográficas y de cómo se puede elegir una proyección adecuada en función del área de interés y del contexto de su mapa/análisis.
En primer lugar, se crea un buffer circular con un radio de ~2,5km alrededor de cada centro sanitario. Esto se hace con la función st_buffer() de tmap. Como la unidad del mapa está en grados decimales lat/long, tenemos que proporcionar el valor de dist en grados decimales, es decir, tenemos que proporcionar un valor de “0,02” (2,5/111 = 0.02 grados decimales, correspondiente a ~2,5km). Si el sistema de coordenadas del mapa está en metros, el número debe proporcionarse en metros.
sle_hf_2k <- sle_hf %>%
st_buffer(dist=0.02) # grados decimales que se corresponden con aproximadamente 2.5km A continuación, trazamos las zonas de influencia propiamente dichas, con ese buffer:
tmap_mode("plot")
# Crear buffers circulares
tm_shape(sle_hf_2k) +
tm_borders(col = "black", lwd = 2)+
tm_shape(sle_hf) + # trazar las instalaciones clínicas en grandes puntos rojos
tm_dots(size=0.3, col='black') 
En segundo lugar, intersectamos estos buffers con los casos (puntos) utilizando st_join() y el tipo de unión st_intersects. Es decir, ponemos juntos los datos de los buffers y los de los puntos con los que se cruzan.
# Intersecta los casos con los buffers
linelist_sf_hf_2k <- linelist_sf_hf %>%
st_join(sle_hf_2k, join = st_intersects, left = TRUE) %>%
filter(osm_id.x==osm_id.y | is.na(osm_id.y)) %>%
select(case_id, osm_id.x, nearest_clinic, amenity.x, osm_id.y)Ahora podemos contabilizar cuántos de nuestros casos no estan dentro de ningun buffer (sus puntos no se cruzan con ningún buffer): nrow(linelist_sf_hf_2k[is.na(linelist_sf_hf_2k$osm_id.y),]). Con este código vemos que falta información para este valor (estamos buscando NA), y por tanto, inferimos que estos casos viven a más de 30 minutos a pie del centro sanitario más cercano.
# Casos que no se cruzaron con ninguno de los buffers de los centros sanitarios
linelist_sf_hf_2k %>%
filter(is.na(osm_id.y)) %>%
nrow()## [1] 1000Podemos visualizar los resultados de forma que los casos que no se cruzan con ningún buffer aparezcan en rojo.
tmap_mode("view")
# Primero muestra los casos en puntos
tm_shape(linelist_sf_hf) +
tm_dots(size=0.08, col='nearest_clinic') +
# representa las instalaciones sanitarias en puntos negros grandes
tm_shape(sle_hf) +
tm_dots(size=0.3, col='black')+
# A continuación, se superpone el buffer de los centros sanitarios en polilíneas
tm_shape(sle_hf_2k) +
tm_borders(col = "black", lwd = 2) +
# Se resaltan los casos que no forman parte de ningún centro sanitario
# en puntos rojos
tm_shape(linelist_sf_hf_2k %>% filter(is.na(osm_id.y))) +
tm_dots(size=0.1, col='red') +
tm_view(set.view = c(-13.2284,8.4699, 13), set.zoom.limits = c(13,14))+
# añade el título
tm_layout(title = "Cases by clinic catchment area")Otras uniones espaciales
Los siguientes son valores alternativos para el argunmento join (puedes encontrar más información en documentation)
- st_contains_properly
- st_contains
- st_covered_by
- st_covers
- st_crosses
- st_disjoint
- st_equals_exact
- st_equals
- st_is_within_distance
- st_nearest_feature
- st_overlaps
- st_touches
- st_within
28.7 Mapas de coropletas
Los mapas de coropletas pueden ser útiles para visualizar los datos por áreas predefinidas, como por ejemplo unidades administrativas o áreas de salud. Cuando se responde a un brote esto puede ser muy útil para dirigir los recursos a zonas específicas con altas tasas de incidencia, por ejemplo.
Ahora que tenemos los nombres de las unidades administrativas asignados a todos los casos (véase la sección sobre uniones espaciales, más arriba), podemos empezar a mapear el número de casos por zonas (mapa de coropletas).
Como también tenemos datos de población por ADM3, podemos añadir esta información a la tabla case_adm3 creada anteriormente.
Comenzamos con el dataframe creado en el paso anterior case_adm3, que es una tabla resumen de cada unidad administrativa y su número de casos.
- Los datos de la población
sle_adm3_popse unen utilizando unleft_join()de dplyr utilizando la columnaadmin3pcoddel dataframecase_adm3, y la columnaadm_pcodeen el dataframesle_adm3_pop, que tienen la misma información. Véase la página sobre la unión de datos. -
select()se aplica al nuevo dataframe, para mantener sólo las columnas que nos interesan -totales la población total. - Los casos por cada 10.000 habitantes se calculan como una nueva columna con
mutate().
# Añadir datos de población y calcular los casos por cada 10K habitantes
case_adm3 <- case_adm3 %>%
left_join(sle_adm3_pop, # añade columnas del conjunto de datos de población
by = c("admin3pcod" = "adm3_pcode")) %>% # join basado en valores comunes entre estas dos columnas
select(names(case_adm3), total) %>% # conserva sólo las columnas importantes, incluida la población total
mutate(case_10kpop = round(cases/total * 10000, 3)) # crea una nueva columna con la tasa de casos por 10000, redondeada a 3 decimales
case_adm3 # imprime en la consola para su visualización## # A tibble: 10 × 5
## # Groups: admin3pcod [10]
## admin3pcod admin3name cases total case_10kpop
## <chr> <chr> <int> <int> <dbl>
## 1 SL040102 Mountain Rural 287 33993 84.4
## 2 SL040208 West III 254 210252 12.1
## 3 SL040207 West II 170 145109 11.7
## 4 SL040204 East II 100 99821 10.0
## 5 SL040201 Central I 59 69683 8.47
## 6 SL040203 East I 58 68284 8.49
## 7 SL040206 West I 32 60186 5.32
## 8 SL040202 Central II 21 23874 8.80
## 9 SL040205 East III 15 500134 0.3
## 10 <NA> <NA> 4 NA NAPara poder mapear estos datos, tenemos que unir esta tabla con el shapefile de polígonos ADM3:
case_adm3_sf <- case_adm3 %>% # begin with cases & rate by admin unit
left_join(sle_adm3, by="admin3pcod") %>% # join a datos del shapefile por columna común
select(objectid, admin3pcod, # mantiene sólo ciertas columnas de interés
admin3name = admin3name.x, # limpia el nombre de una columna
admin2name, admin1name,
cases, total, case_10kpop,
geometry) %>% # mantiene la geometría para poder trazar los polígonos
drop_na(objectid)%>% # elimina las filas vacías
st_as_sf() # convierte a shapefilePara mapear los resultados en un mapa estático:
# tmap mode
tmap_mode("plot") # ver mapa estático
# plot polygons
tm_shape(case_adm3_sf) +
tm_polygons("cases") + # colorea según la columna de número de casos
tm_text("admin3name") # visualización del nombre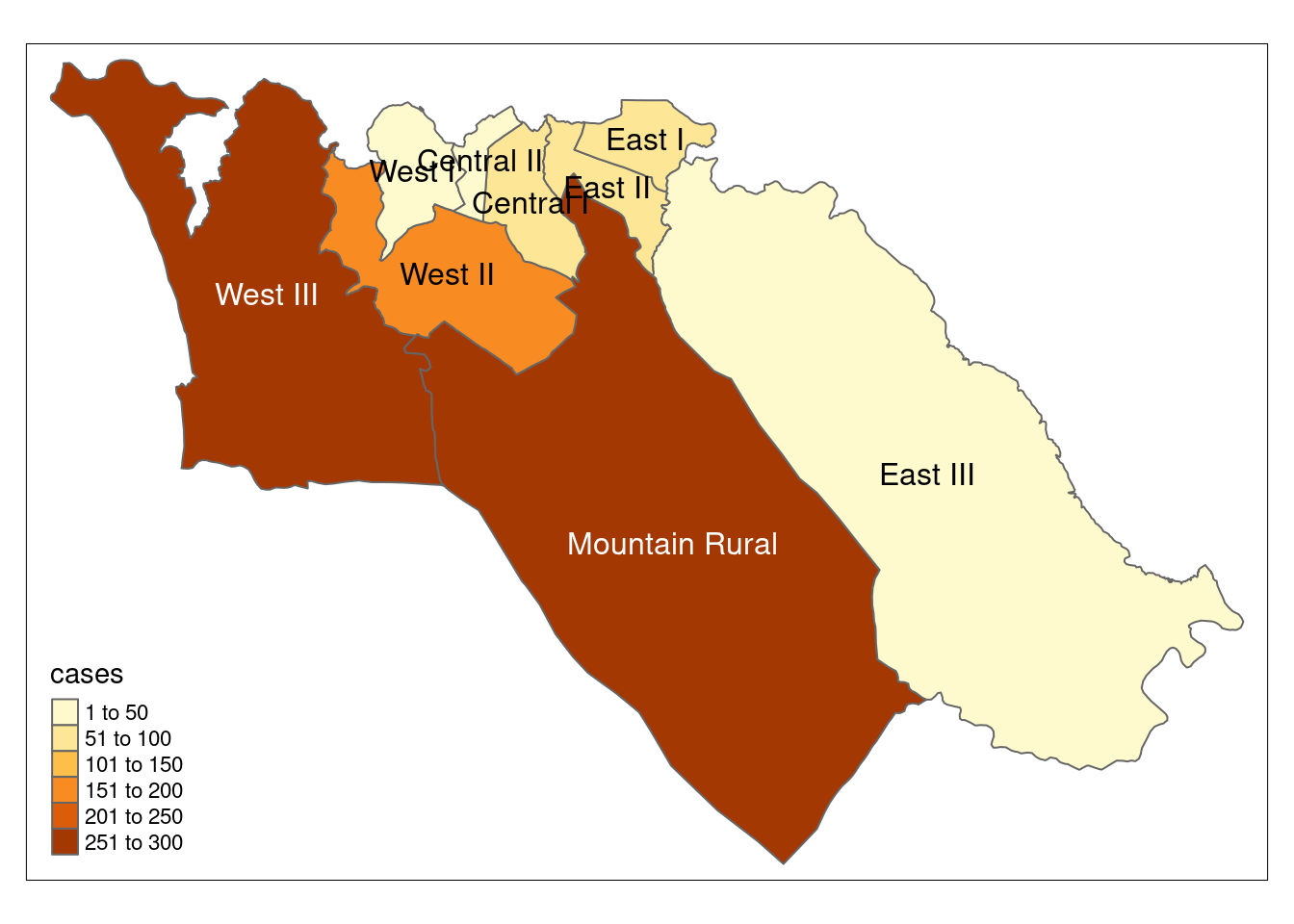
También podemos mapear las tasas de incidencia:
# Casos por 10.000 habitantes
tmap_mode("plot") # modo de visualización estático
# representación del mapa
tm_shape(case_adm3_sf) + # polígonos del gráfico
tm_polygons("case_10kpop", # colorea según la columna que contiene la tasa de casos
breaks=c(0, 10, 50, 100), # define los puntos de ruptura para los colores
palette = "Purples" # usa una paleta de colores púrpura
) +
tm_text("admin3name") # muestra el texto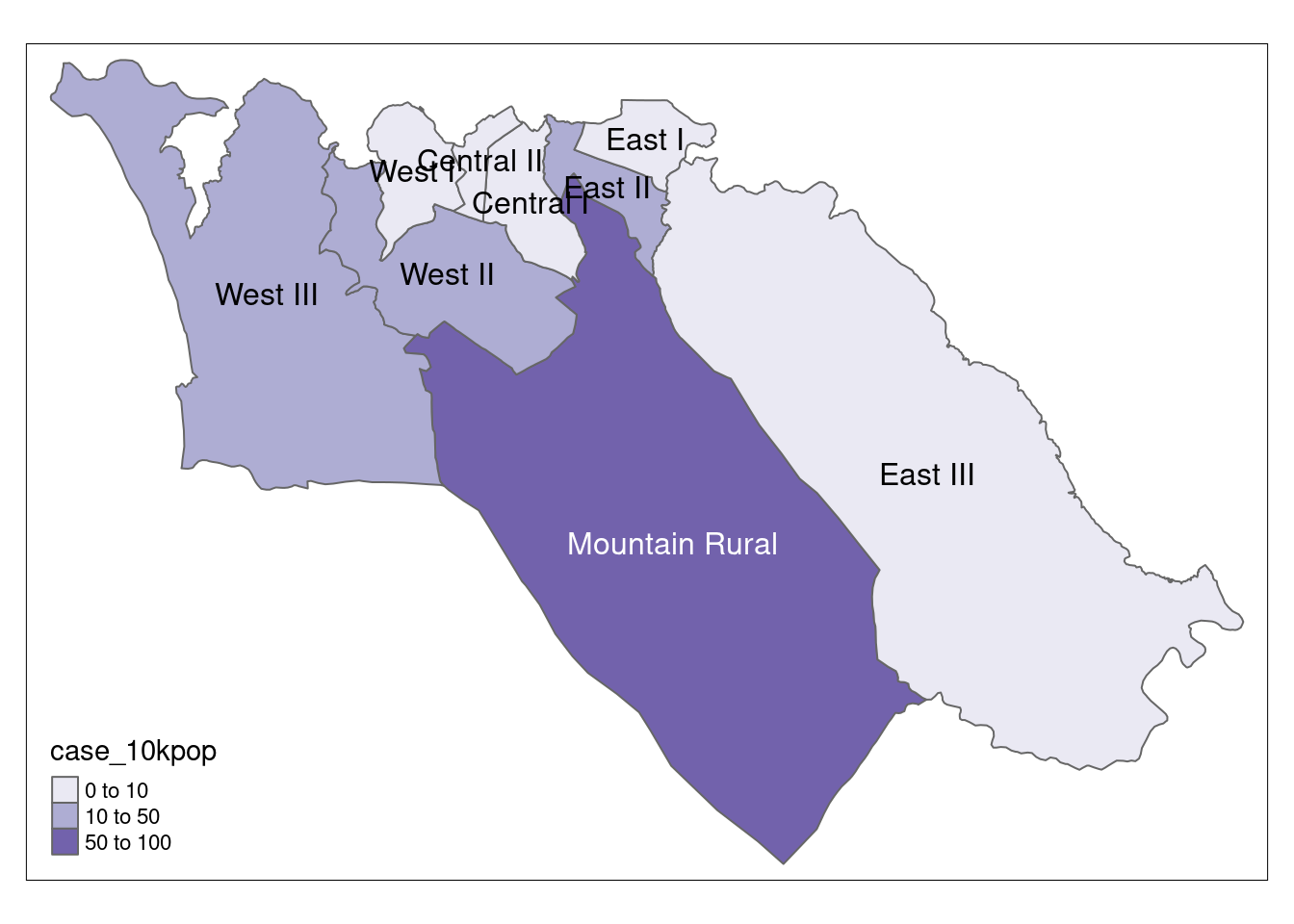
28.8 Mapeo con ggplot2
Si ya conoces el uso de ggplot2, puedes utilizar ese paquete para crear mapas estáticos de tus datos. La función geom_sf() dibujará diferentes objetos en función de las características de los datos. Por ejemplo, puedes utilizar geom_sf() en un ggplot() utilizando datos sf con geometría de polígonos para crear un mapa de coropletas.
Para ilustrar cómo funciona esto, podemos empezar con el archivo shape de polígonos ADM3 que hemos utilizado antes. Recordemos que se trata de regiones de nivel administrativo 3 en Sierra Leona:
sle_adm3## Simple feature collection with 12 features and 19 fields
## Geometry type: MULTIPOLYGON
## Dimension: XY
## Bounding box: xmin: -13.29894 ymin: 8.094272 xmax: -12.91333 ymax: 8.499809
## Geodetic CRS: WGS 84
## # A tibble: 12 × 20
## objectid admin3name admin3pcod admin3ref_n admin2name admin…¹ admin…² admin…³ admin…⁴ admin…⁵ date valid_on valid_to shape_l…⁶ shape…⁷ rowca…⁸ rowca…⁹ rowca…˟ rowca…˟ geometry
## * <dbl> <chr> <chr> <chr> <chr> <chr> <chr> <chr> <chr> <chr> <date> <date> <date> <dbl> <dbl> <chr> <chr> <chr> <chr> <MULTIPOLYGON [°]>
## 1 155 Koya Rural SL040101 Koya Rural Western Area Rur… SL0401 Western SL04 Sierra… SL 2016-08-01 2016-10-17 NA 0.638 1.37e-2 SLE SLE004 SLE004… SLE004… (((-13.02082 8.381989, -…
## 2 156 Mountain Rural SL040102 Mountain Rural Western Area Rur… SL0401 Western SL04 Sierra… SL 2016-08-01 2016-10-17 NA 0.293 3.18e-3 SLE SLE004 SLE004… SLE004… (((-13.21496 8.474341, -…
## 3 157 Waterloo Rural SL040103 Waterloo Rural Western Area Rur… SL0401 Western SL04 Sierra… SL 2016-08-01 2016-10-17 NA 0.723 1.36e-2 SLE SLE004 SLE004… SLE004… (((-13.12215 8.413156, -…
## 4 158 York Rural SL040104 York Rural Western Area Rur… SL0401 Western SL04 Sierra… SL 2016-08-01 2016-10-17 NA 1.24 1.98e-2 SLE SLE004 SLE004… SLE004… (((-13.24441 8.095223, -…
## 5 159 Central I SL040201 Central I Western Area Urb… SL0402 Western SL04 Sierra… SL 2016-08-01 2016-10-17 NA 0.0688 1.88e-4 SLE SLE004 SLE004… SLE004… (((-13.22646 8.489716, -…
## 6 160 East I SL040203 East I Western Area Urb… SL0402 Western SL04 Sierra… SL 2016-08-01 2016-10-17 NA 0.0575 1.43e-4 SLE SLE004 SLE004… SLE004… (((-13.2129 8.494033, -1…
## 7 161 East II SL040204 East II Western Area Urb… SL0402 Western SL04 Sierra… SL 2016-08-01 2016-10-17 NA 0.0840 1.49e-4 SLE SLE004 SLE004… SLE004… (((-13.22653 8.491883, -…
## 8 162 Central II SL040202 Central II Western Area Urb… SL0402 Western SL04 Sierra… SL 2016-08-01 2016-10-17 NA 0.0488 6.51e-5 SLE SLE004 SLE004… SLE004… (((-13.23154 8.491768, -…
## 9 163 West III SL040208 West III Western Area Urb… SL0402 Western SL04 Sierra… SL 2016-08-01 2016-10-17 NA 0.302 1.70e-3 SLE SLE004 SLE004… SLE004… (((-13.28529 8.497354, -…
## 10 164 West I SL040206 West I Western Area Urb… SL0402 Western SL04 Sierra… SL 2016-08-01 2016-10-17 NA 0.0695 1.82e-4 SLE SLE004 SLE004… SLE004… (((-13.24677 8.493453, -…
## 11 165 West II SL040207 West II Western Area Urb… SL0402 Western SL04 Sierra… SL 2016-08-01 2016-10-17 NA 0.149 5.47e-4 SLE SLE004 SLE004… SLE004… (((-13.25698 8.485518, -…
## 12 167 East III SL040205 East III Western Area Urb… SL0402 Western SL04 Sierra… SL 2016-08-01 2016-10-17 NA 0.327 3.10e-3 SLE SLE004 SLE004… SLE004… (((-13.20465 8.485758, -…
## # … with abbreviated variable names ¹admin2pcod, ²admin1name, ³admin1pcod, ⁴admin0name, ⁵admin0pcod, ⁶shape_leng, ⁷shape_area, ⁸rowcacode0, ⁹rowcacode1, ˟rowcacode2, ˟rowcacode3Podemos utilizar la función left_join() de dplyr para añadir al objeto shapefile los datos que queremos mapear. En este caso, vamos a utilizar el dataframe case_adm3 que creamos anteriormente para resumir los recuentos de casos por región administrativa. También podemos utilizar este mismo enfoque para mapear cualquier dato almacenado en un dataframe.
sle_adm3_dat <- sle_adm3 %>%
inner_join(case_adm3, by = "admin3pcod") # inner join = sólo se conservan los datos de ambos objetos
select(sle_adm3_dat, admin3name.x, cases) # imprime las variables seleccionadas en la consola## Simple feature collection with 9 features and 2 fields
## Geometry type: MULTIPOLYGON
## Dimension: XY
## Bounding box: xmin: -13.29894 ymin: 8.384533 xmax: -13.12612 ymax: 8.499809
## Geodetic CRS: WGS 84
## # A tibble: 9 × 3
## admin3name.x cases geometry
## <chr> <int> <MULTIPOLYGON [°]>
## 1 Mountain Rural 287 (((-13.21496 8.474341, -13.21479 8.474289, -13.21465 8.474296, -13.21455 8.474298, -...
## 2 Central I 59 (((-13.22646 8.489716, -13.22648 8.48955, -13.22644 8.489513, -13.22663 8.489229, -1...
## 3 East I 58 (((-13.2129 8.494033, -13.21076 8.494026, -13.21013 8.494041, -13.2096 8.494025, -13...
## 4 East II 100 (((-13.22653 8.491883, -13.22647 8.491853, -13.22642 8.49186, -13.22633 8.491814, -1...
## 5 Central II 21 (((-13.23154 8.491768, -13.23141 8.491566, -13.23144 8.49146, -13.23131 8.491294, -1...
## 6 West III 254 (((-13.28529 8.497354, -13.28456 8.496497, -13.28403 8.49621, -13.28338 8.496086, -1...
## 7 West I 32 (((-13.24677 8.493453, -13.24669 8.493285, -13.2464 8.493132, -13.24627 8.493131, -1...
## 8 West II 170 (((-13.25698 8.485518, -13.25685 8.485501, -13.25668 8.485505, -13.25657 8.485504, -...
## 9 East III 15 (((-13.20465 8.485758, -13.20461 8.485698, -13.20449 8.485757, -13.20431 8.485577, -...Para hacer un gráfico de columnas de los recuentos de casos por región utilizando ggplot2, podemos llamar a geom_col() de la siguiente manera:
ggplot(data=sle_adm3_dat) +
geom_col(aes(x=fct_reorder(admin3name.x, cases, .desc=T), # reordena el eje-x por 'casos' descendentes
y=cases)) + # el eje-y es el número de casos por región
theme_bw() +
labs( # establecer el texto de la figura
title="Number of cases, by administrative unit",
x="Admin level 3",
y="Number of cases"
) +
guides(x=guide_axis(angle=45)) # ajusta las etiquetas del eje-x a 45 grados para que encajen mejor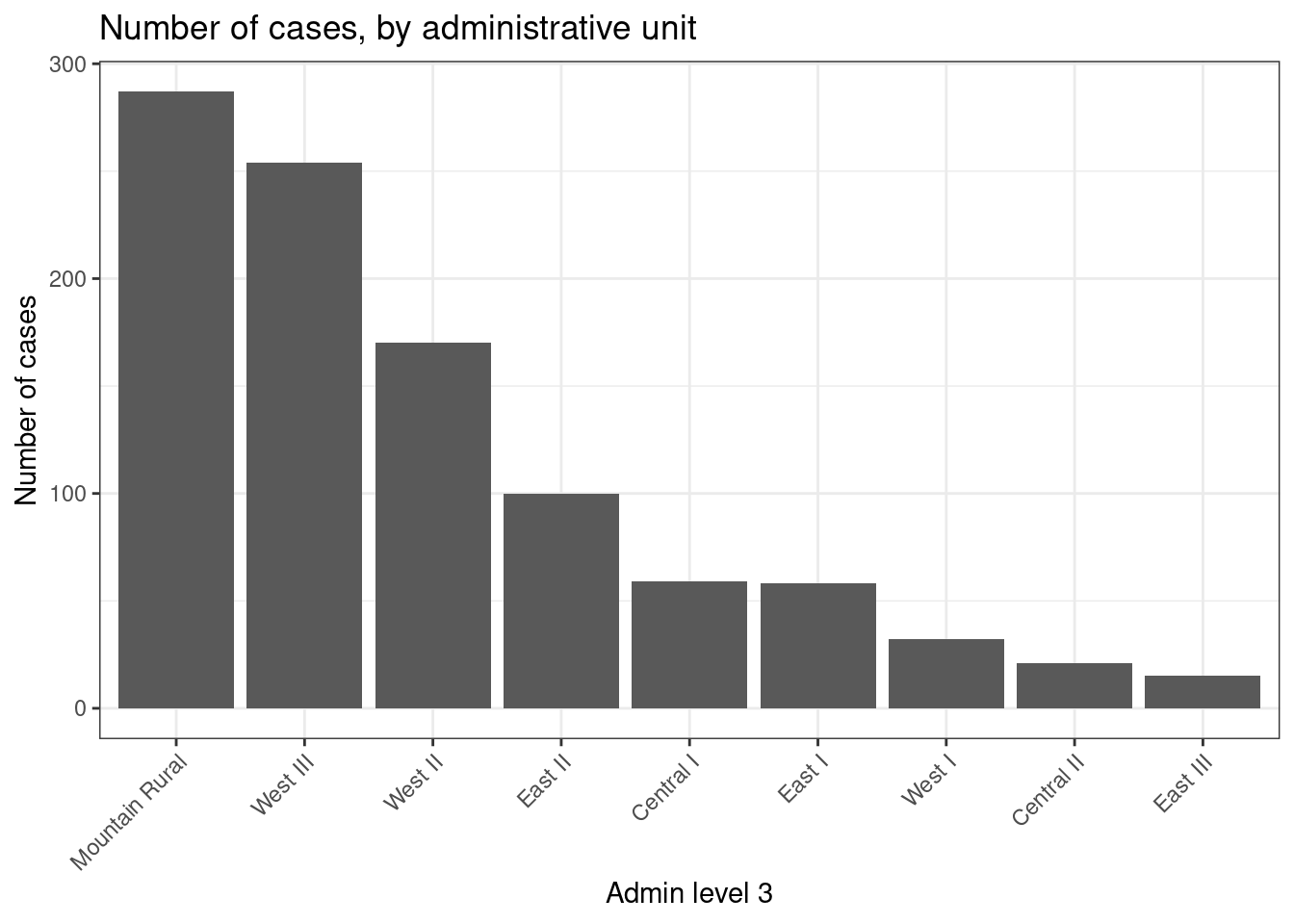
Si queremos utilizar ggplot2 para hacer un mapa de coropletas de los recuentos de casos, podemos utilizar una sintaxis similar para llamar a la función geom_sf():
ggplot(data=sle_adm3_dat) +
geom_sf(aes(fill=cases)) # configura el relleno para que varíe según la variable de recuento de casos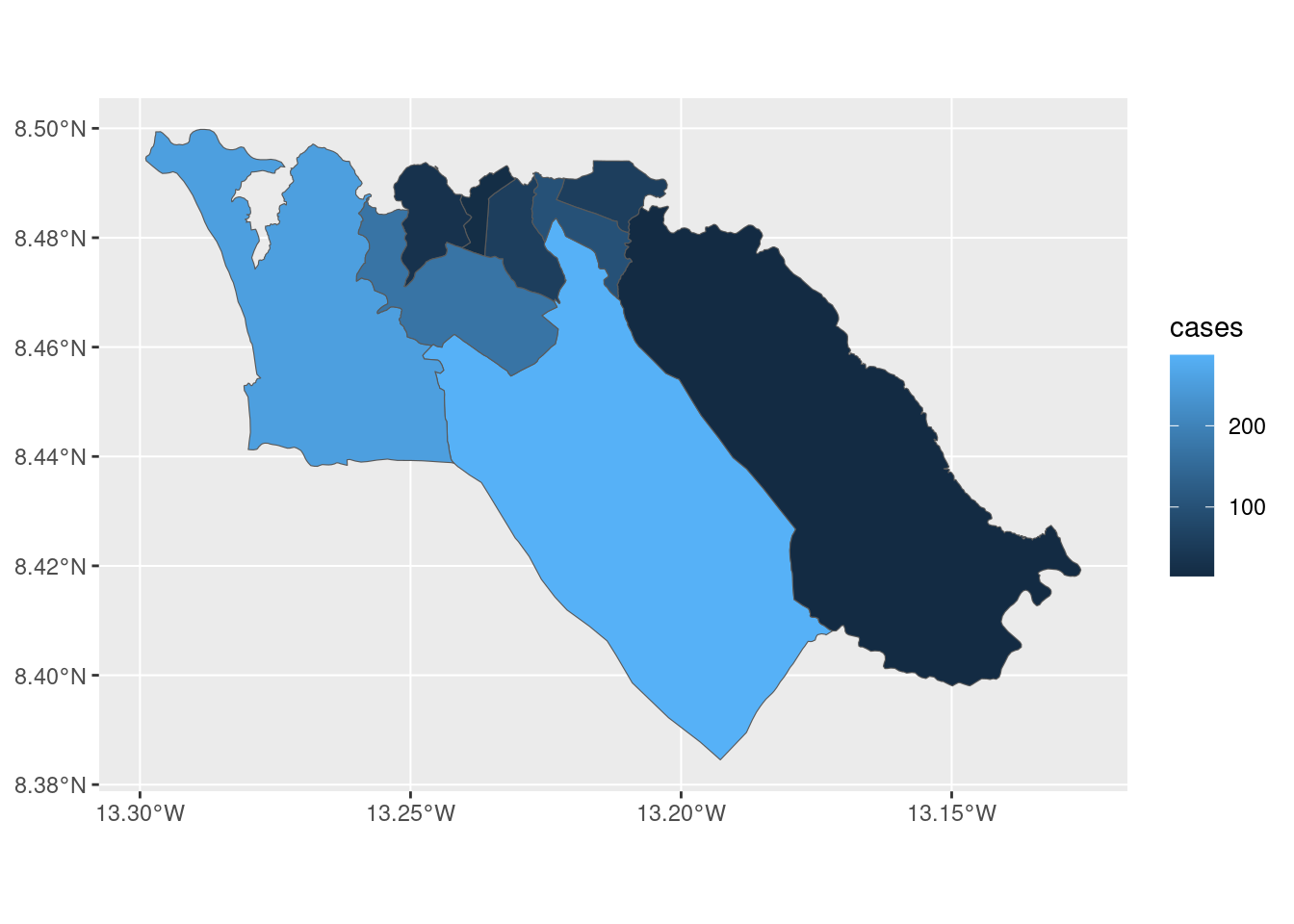
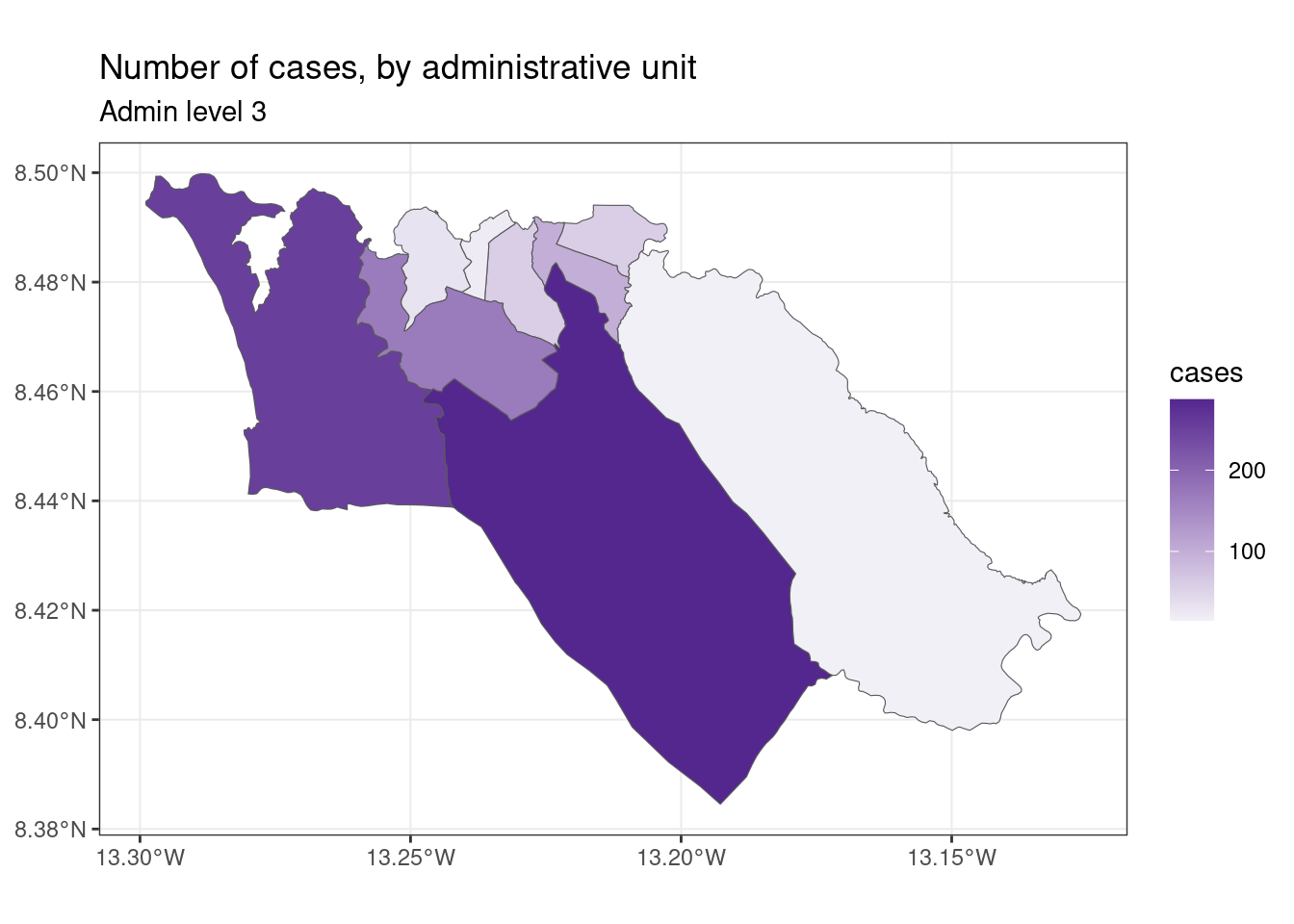
A continuación, podemos personalizar la apariencia de nuestro mapa utilizando una sintaxis que sea consistente en ggplot2, por ejemplo:
ggplot(data=sle_adm3_dat) +
geom_sf(aes(fill=cases)) +
scale_fill_continuous(high="#54278f", low="#f2f0f7") + # cambia el gradiente de color
theme_bw() +
labs(title = "Number of cases, by administrative unit", # establece el texto de la figura
subtitle = "Admin level 3"
)
Para las personas que se sientan cómodas trabajando con ggplot2, geom_sf() ofrece una implementación simple y directa que es adecuada para las visualizaciones básicas de mapas. Para saber más, mira la viñeta de geom_sf() o el libro de ggplot2.
28.9 Mapas base
OpenStreetMap
A continuación describimos cómo conseguir un mapa base para realizar un mapa con ggplot2 utilizando las características de OpenStreetMap. Existen métodos alternativos que incluyen el uso de ggmap que requiere el registro gratuito con Google (detalles).
OpenStreetMap es un proyecto de colaboración para crear un mapa editable y gratuito del mundo. Los datos de geolocalización subyacentes (por ejemplo, ubicaciones de ciudades, carreteras, características naturales, aeropuertos, escuelas, hospitales, caminos, etc.) se consideran el resultado principal del proyecto.
Primero cargamos el paquete OpenStreetMap, del que obtendremos nuestro mapa base.
A continuación, creamos el objeto map, que definimos mediante la función openmap() del paquete OpenStreetMap (documentación). Proporcionamos lo siguiente:
-
upperLeftylowerRight: Estas son dos pares de coordenadas que especifican los límites del marco del mapa base.- En este caso hemos puesto los máximos y mínimos de las filas del listado, para que el mapa responda dinámicamente a los datos
zoom =(si es nulo se determina automáticamente)type =qué tipo de mapa base - aquí hemos enumerado varias posibilidades y el código utiliza actualmente la primera ([1]) “osm”mergeTiles= elegimos TRUE para que las capas se fusionen en uno solo
# cargar el paquete
pacman::p_load(OpenStreetMap)
# Ajustar mapa base por rango de coordenadas lat/long. Se elige el tipo de mosaico
map <- openmap(
upperLeft = c(max(linelist$lat, na.rm=T), max(linelist$lon, na.rm=T)), # límites del mosaico del mapa base
lowerRight = c(min(linelist$lat, na.rm=T), min(linelist$lon, na.rm=T)),
zoom = NULL,
type = c("osm", "stamen-toner", "stamen-terrain", "stamen-watercolor", "esri","esri-topo")[1])Si trazamos este mapa ahora mismo, usando autoplot.OpenStreetMap() del paquete OpenStreetMap, verás que las unidades en los ejes no son coordenadas de latitud/longitud. Se está utilizando un sistema de coordenadas diferente. Para mostrar correctamente las residencias de los casos (que se almacenan en lat/long), se debe cambiar esto.
autoplot.OpenStreetMap(map)
Vamos a convertir el mapa a latitud/longitud con la función openproj() del paquete OpenStreetMap. Proporcionamos el mapa base map y también el Sistema de Referencia de Coordenadas (CRS) que queremos. Lo hacemos proporcionando la cadena de caracteres “proj.4” para la proyección WGS 1984, pero también se puede proporcionar el CRS de otras maneras. (ver esta página para entender mejor qué es una cadena proj.4)
# Projección WGS84
map_latlon <- openproj(map, projection = "+proj=longlat +ellps=WGS84 +datum=WGS84 +no_defs")Ahora cuando creamos el gráfico vemos que a lo largo de los ejes están las coordenadas de latitud y longitud. El sistema de coordenadas ha sido convertido. Ahora nuestros casos se trazarán correctamente si se superponen:
# Dibujar el mapa. Se debe usar "autoplot" para trabajar con ggplot
autoplot.OpenStreetMap(map_latlon)
Consulta estos dos tutoriales aquí y aquí para obtener más información sobre este tema.
28.10 Mapas de calor de densidad contorneada
A continuación describimos cómo conseguir un mapa de calor de densidad contorneada de casos, sobre un mapa base, comenzando con un listado (una fila por caso).
- Crear un mapa base a partir de OpenStreetMap, como se ha descrito anteriormente.
- Trazar los casos de
linelistutilizando las columnas de latitud y longitud. - Convertir los puntos en un mapa de calor de densidad con
stat_density_2d()de ggplot2,
Cuando tenemos un mapa base con coordenadas de latitud y longitud, podemos trazar nuestros casos encima utilizando las coordenadas de latitud y longitud de su residencia.
Partiendo de la función autoplot.OpenStreetMap() para crear el mapa base, se pueden añadir las funciones de ggplot2, como se muestra con geom_point() a continuación:
# Dibujar el mapa. Se debe usar "autoplot" para trabajar con ggplot
autoplot.OpenStreetMap(map_latlon)+ # comienza con el mapa base
geom_point( # añade puntos xy de las columnas lon y lat de linelist
data = linelist,
aes(x = lon, y = lat),
size = 1,
alpha = 0.5,
show.legend = FALSE) + # elimina la leyenda por completo
labs(x = "Longitude", # títulos y etiquetas
y = "Latitude",
title = "Cumulative cases")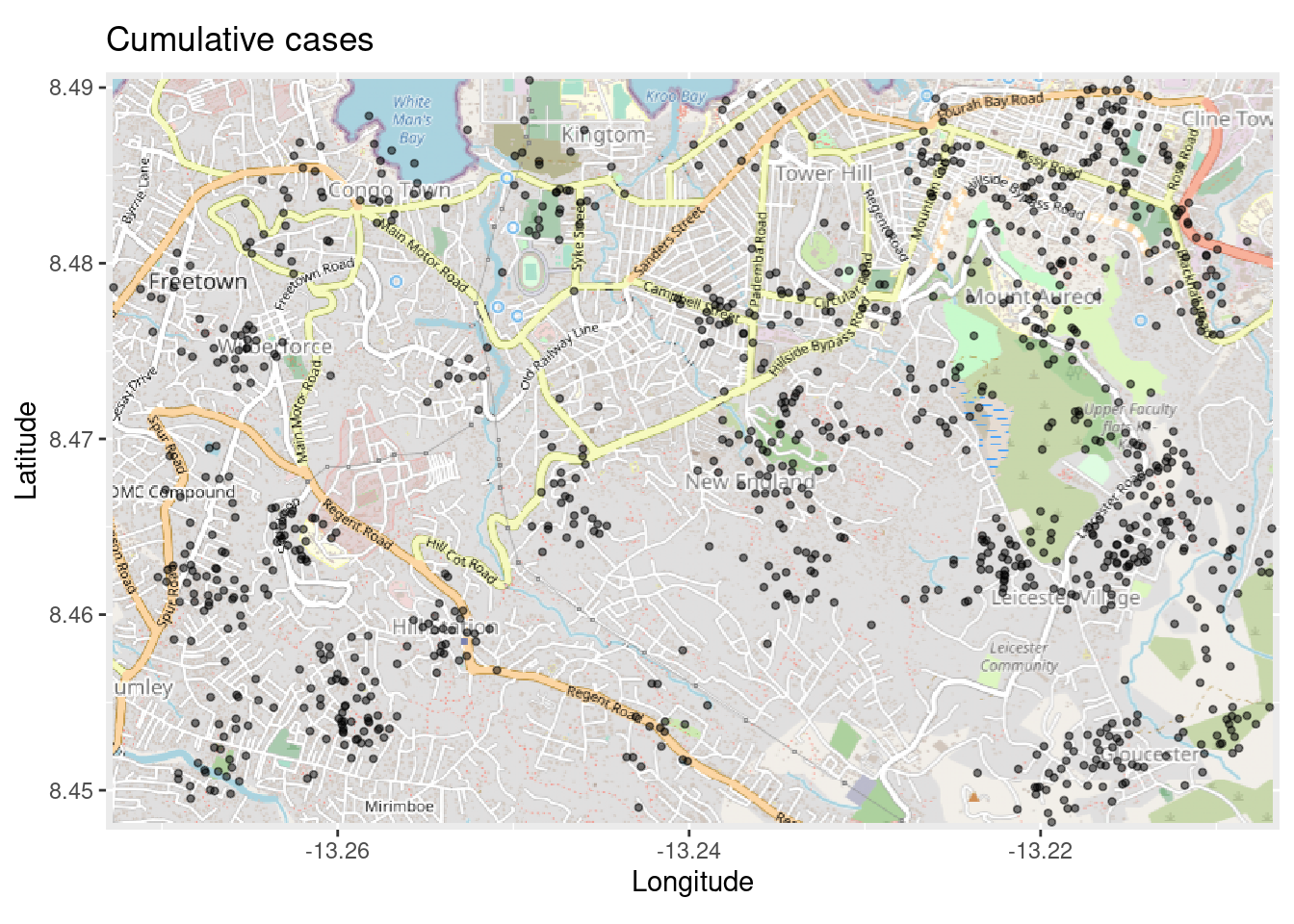
El mapa anterior puede ser difícil de interpretar, especialmente con los puntos superpuestos. Para mejorar esto, vamos a trazar un mapa de densidad en 2d utilizando la función ggplot2 stat_density_2d(). Sguemos utilizando las coordenadas lat/lon del listado, pero ahora estamos realizando una estimación de la densidad del núcleo en 2D y los resultados se muestran con líneas de contorno - como un mapa topográfico. Puedes leer esta documentación completa para saber más.
# comienza con el mapa base
autoplot.OpenStreetMap(map_latlon)+
# añadir el mapa de densidad
ggplot2::stat_density_2d(
data = linelist,
aes(
x = lon,
y = lat,
fill = ..level..,
alpha = ..level..),
bins = 10,
geom = "polygon",
contour_var = "count",
show.legend = F) +
# especificar escala de color
scale_fill_gradient(low = "black", high = "red")+
# etiquetas
labs(x = "Longitude",
y = "Latitude",
title = "Distribution of cumulative cases")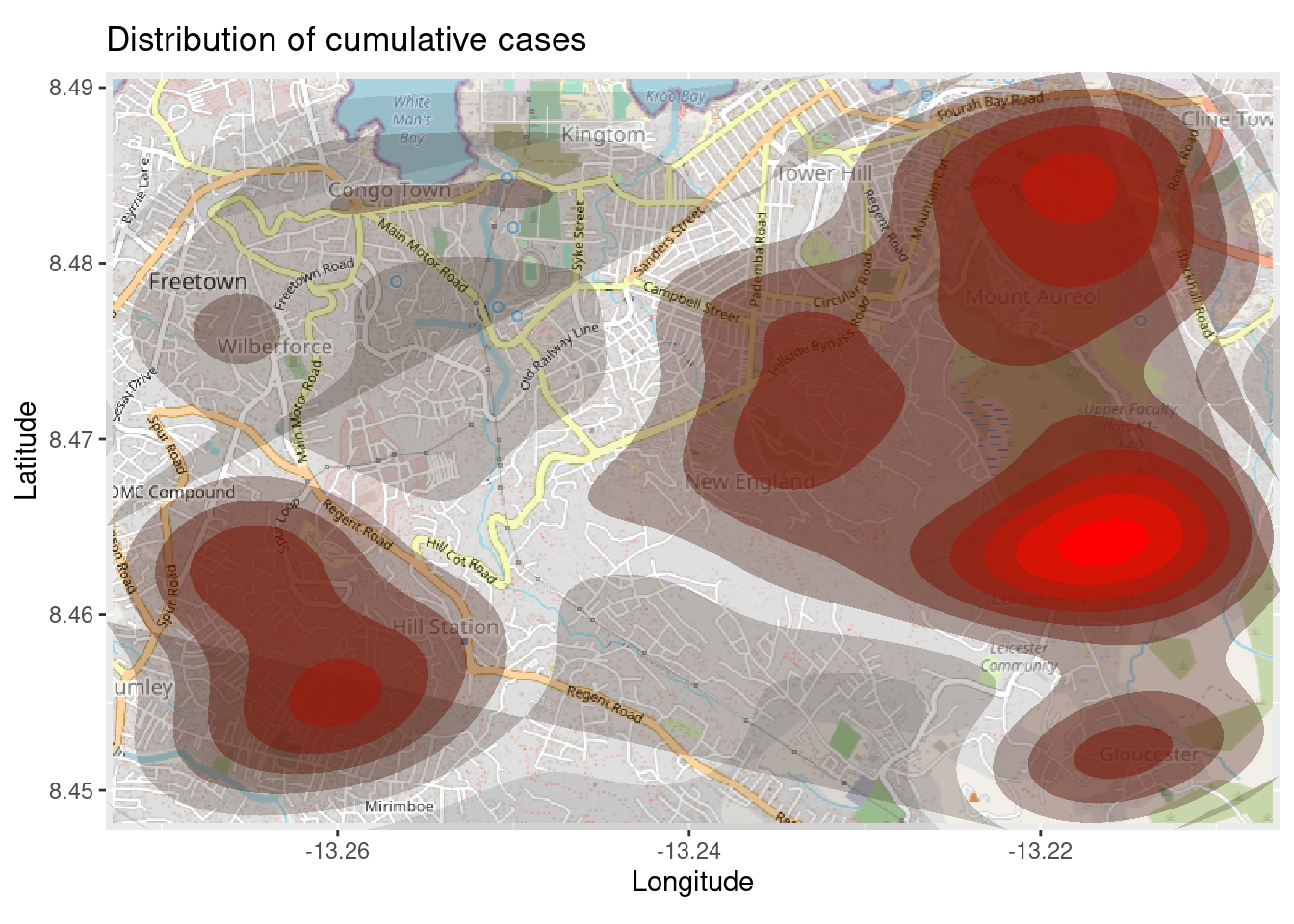
Mapa de calor de series temporales
El mapa de calor de densidad anterior muestra los casos acumulados. Podemos examinar el brote a lo largo del tiempo y del espacio haciendo un facetado del mapa de calor basado en el mes de inicio de los síntomas, si tenemos esta información en el linelist.
Comenzamos con linelist, creando una nueva columna con el Año y el Mes de inicio. La función format() de R base cambia la forma en que se muestra una fecha. En este caso queremos “AAAA-MM”.
# Extraer mes de inicio
linelist <- linelist %>%
mutate(date_onset_ym = format(date_onset, "%Y-%m"))
# Examinar los valores
table(linelist$date_onset_ym, useNA = "always")##
## 2014-04 2014-05 2014-06 2014-07 2014-08 2014-09 2014-10 2014-11 2014-12 2015-01 2015-02 2015-03 2015-04 <NA>
## 1 9 16 36 104 190 182 123 101 85 57 30 27 39Ahora, simplemente introducimos el facetado a través de ggplot2 en el mapa de calor de densidad. Se aplica facet_wrap(), utilizando la nueva columna como filas. Fijamos el número de columnas de facetas en 4 paraque se vea mejor.
# paquetes
pacman::p_load(OpenStreetMap, tidyverse)
# comienza con el mapa base
autoplot.OpenStreetMap(map_latlon)+
# añadir el mapa de densidad
ggplot2::stat_density_2d(
data = linelist,
aes(
x = lon,
y = lat,
fill = ..level..,
alpha = ..level..),
bins = 10,
geom = "polygon",
contour_var = "count",
show.legend = F) +
# especificar escala de color
scale_fill_gradient(low = "black", high = "red")+
# etiquetas
labs(x = "Longitude",
y = "Latitude",
title = "Distribution of cumulative cases over time")+
# facetar el gráfico por mes-año de inicio
facet_wrap(~ date_onset_ym, ncol = 4) 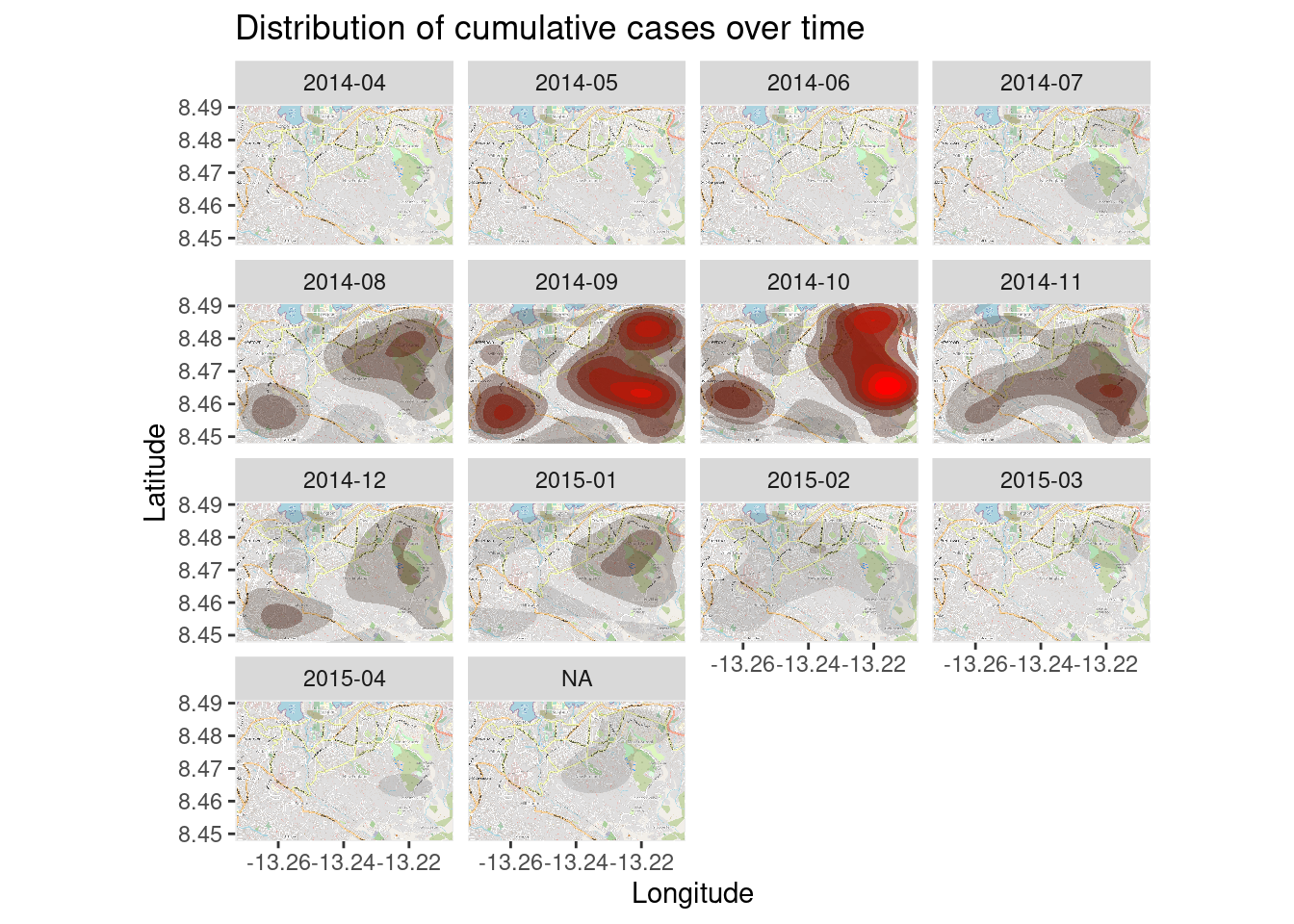
28.11 Estadísticas espaciales
La mayor parte de nuestra discusión hasta ahora se ha centrado en la visualización de datos espaciales. En algunos casos, también puede interesarte utilizar estadísticas espaciales para cuantificar las relaciones espaciales de los atributos de tus datos. En esta sección se ofrece una breve visión general de algunos conceptos claves de la estadística espacial y se sugiere algunos recursos que te resultarán útiles si deseas realizar análisis espaciales más exhaustivos.
Relaciones espaciales
Antes de poder calcular cualquier estadística espacial, tenemos que especificar las relaciones entre las características de nuestros datos. Hay muchas formas de conceptualizar las relaciones espaciales, pero un modelo sencillo y comúnmente aplicable es el de la adyacencia, es decir, que esperamos una relación geográfica entre las zonas que comparten una frontera o son “vecinas” unas de otras.
Podemos cuantificar las relaciones de adyacencia entre los polígonos de las regiones administrativas en los datos sle_adm3 que hemos estado utilizando con el paquete spdep. Especificaremos la contigüidad queen, que significa que las regiones serán vecinas si comparten al menos un punto a lo largo de sus fronteras. La alternativa sería la contigüidad rook, que requiere que las regiones compartan un borde - en nuestro caso, con polígonos irregulares, la distinción es trivial, pero en algunos casos la elección entre queen y rook puede ser influyente.
sle_nb <- spdep::poly2nb(sle_adm3_dat, queen=T) # crear vecinos
sle_adjmat <- spdep::nb2mat(sle_nb) # crear matriz que resuma las relaciones de vecindad
sle_listw <- spdep::nb2listw(sle_nb) # crear el objeto listw (lista de pesos) -- lo necesitaremos más adelante
sle_nb## Neighbour list object:
## Number of regions: 9
## Number of nonzero links: 30
## Percentage nonzero weights: 37.03704
## Average number of links: 3.333333
round(sle_adjmat, digits = 2)## [,1] [,2] [,3] [,4] [,5] [,6] [,7] [,8] [,9]
## 1 0.00 0.20 0.00 0.20 0.00 0.2 0.00 0.20 0.20
## 2 0.25 0.00 0.00 0.25 0.25 0.0 0.00 0.25 0.00
## 3 0.00 0.00 0.00 0.50 0.00 0.0 0.00 0.00 0.50
## 4 0.25 0.25 0.25 0.00 0.00 0.0 0.00 0.00 0.25
## 5 0.00 0.33 0.00 0.00 0.00 0.0 0.33 0.33 0.00
## 6 0.50 0.00 0.00 0.00 0.00 0.0 0.00 0.50 0.00
## 7 0.00 0.00 0.00 0.00 0.50 0.0 0.00 0.50 0.00
## 8 0.20 0.20 0.00 0.00 0.20 0.2 0.20 0.00 0.00
## 9 0.33 0.00 0.33 0.33 0.00 0.0 0.00 0.00 0.00
## attr(,"call")
## spdep::nb2mat(neighbours = sle_nb)La matriz de arriba muestra las relaciones entre las 9 regiones de nuestros datos sle_adm3. Una puntuación de 0 indica que dos regiones no son vecinas, mientras que cualquier valor distinto de 0 indica una relación de vecindad. Los valores de la matriz se han escalado para que cada región tenga un peso total de 1 en la fila.
La mejor manera de visualizar estas relaciones de vecindad es dibujarlas:
plot(sle_adm3_dat$geometry) + # representar las fronteras de las regiones
spdep::plot.nb(sle_nb,as(sle_adm3_dat, 'Spatial'), col='grey', add=T) # añadir relaciones de vecindad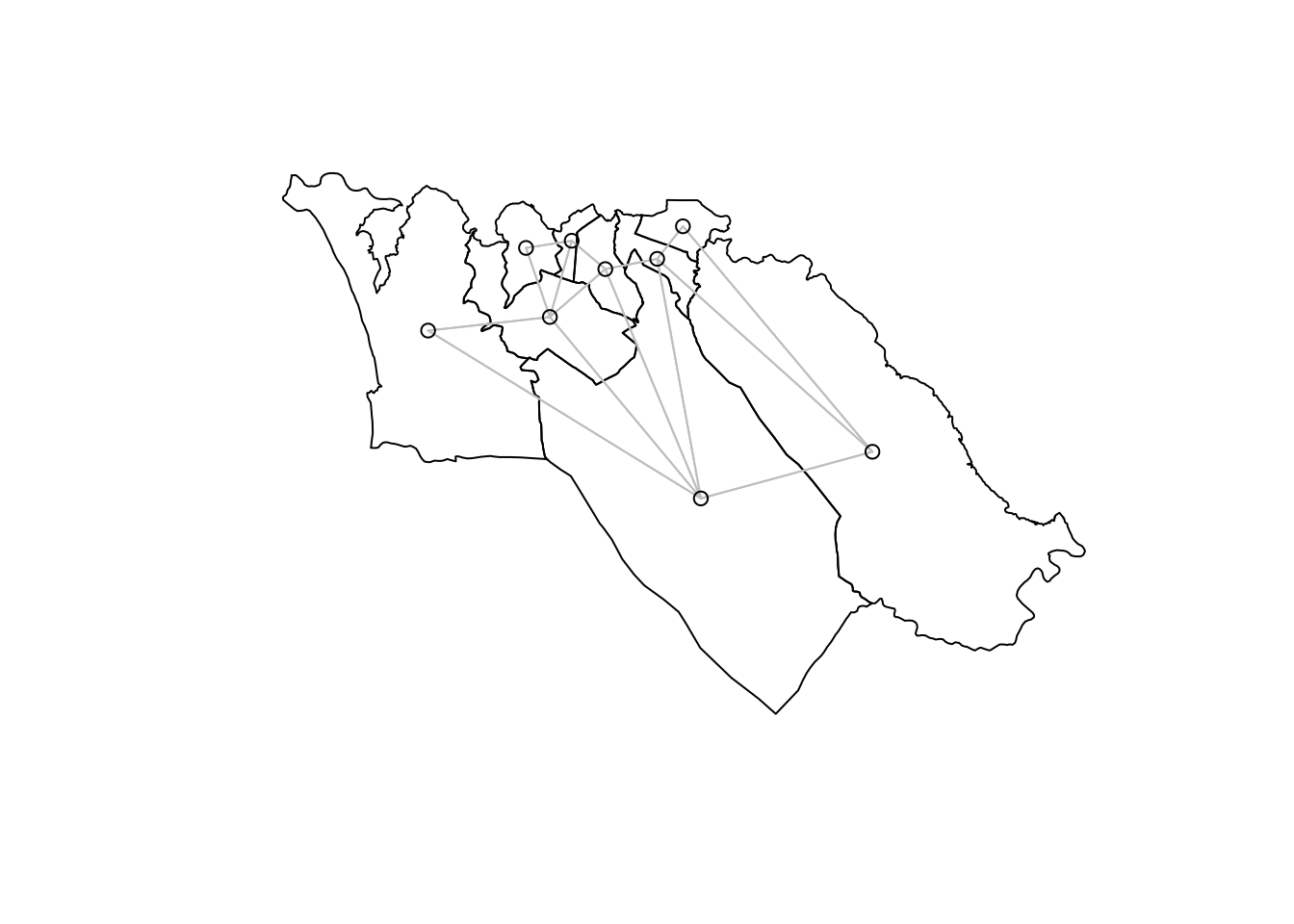
Hemos utilizado un enfoque de adyacencia para identificar los polígonos vecinos; los vecinos que identificamos también se denominan a veces vecinos por contigüidad. Pero ésta es sólo una forma de elegir qué regiones se espera que tengan una relación geográfica. Los enfoques alternativos más comunes para identificar las relaciones geográficas generan vecinos basados en la distancia; brevemente, estos son:
K-vecinos más cercanos - Basándose en la distancia entre los centroides (el centro ponderado geográficamente de cada región poligonal), selecciona las n regiones más cercanas como vecinas. También se puede especificar un umbral de proximidad de distancia máxima. En spdep, puedes utilizar
knearneigh()(documentación).Vecinos de umbral de distancia - Selecciona todos los vecinos dentro de un umbral de distancia. En spdep, estas relaciones de vecindad pueden ser identificadas usando
dnearneigh()(documentación).
Autocorrelación espacial
La tan citada primera ley de la geografía de Tobler afirma que “todo está relacionado con todo lo demás, pero las cosas cercanas están más relacionadas que las lejanas”. En epidemiología, esto suele significar que el riesgo de un determinado resultado sanitario en una región determinada es más similar al de sus regiones vecinas que al de las lejanas. Este concepto se ha formalizado como autocorrelación espacial: la propiedad estadística de que las características geográficas con valores similares se agrupan en el espacio. Las medidas estadísticas de autocorrelación espacial pueden utilizarse para cuantificar el alcance de la agrupación espacial de tus datos, localizar dónde se produce la agrupación e identificar patrones compartidos de autocorrelación espacial entre distintas variables de los datos. Esta sección ofrece una visión general de algunas medidas comunes de autocorrelación espacial y cómo calcularlas en R.
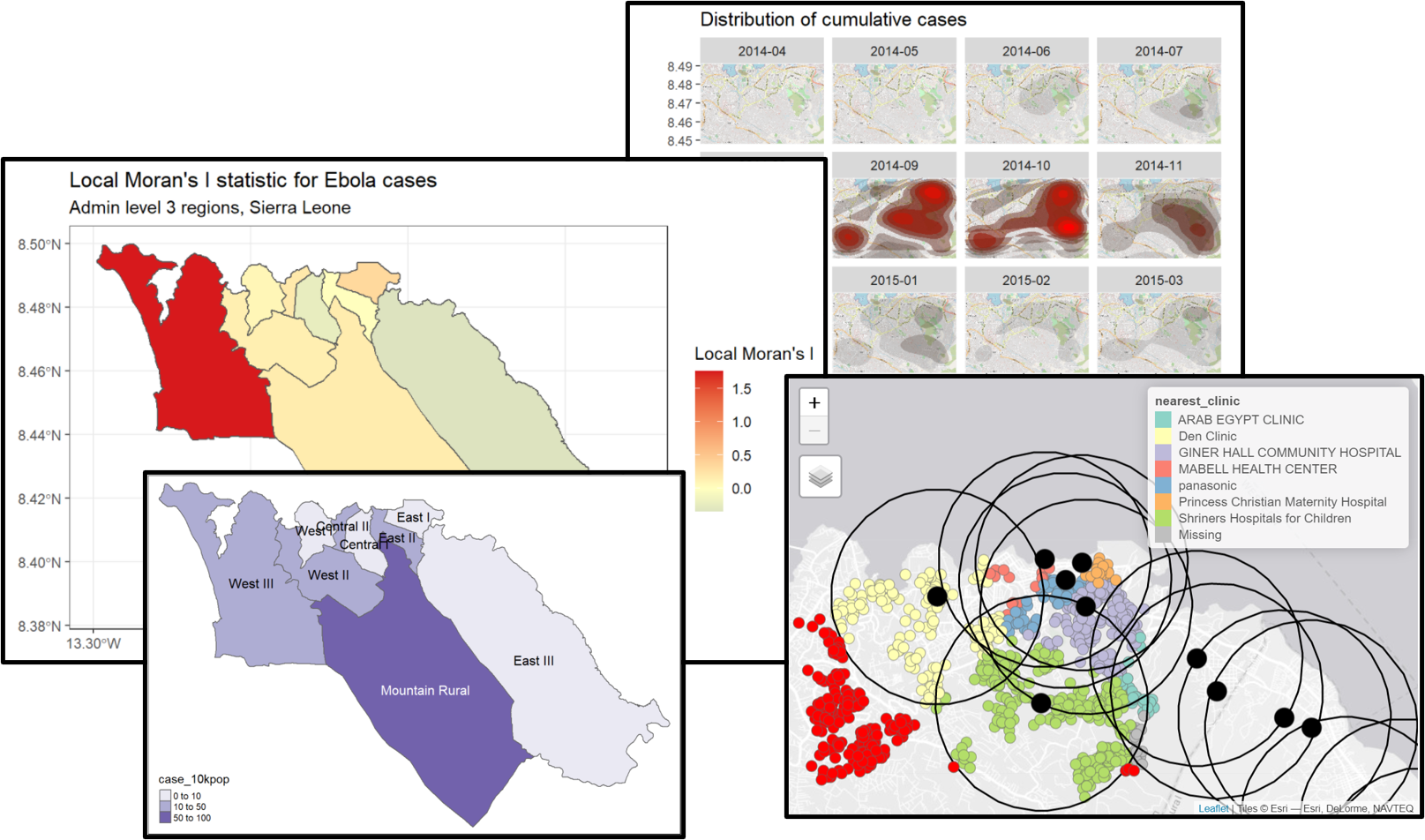
I de Moran - Se trata de una estadística de resumen global de la correlación entre el valor de una variable en una región y los valores de la misma variable en las regiones vecinas. La estadística I de Moran suele oscilar entre -1 y 1. Un valor de 0 indica que no hay ningún patrón de correlación espacial, mientras que los valores más cercanos a 1 o -1 indican una mayor autocorrelación espacial (valores similares cercanos) o dispersión espacial (valores disímiles cercanos), respectivamente.
Como ejemplo, calcularemos la estadística I de Moran para cuantificar la autocorrelación espacial en los casos de Ébola que hemos mapeado antes (recordemos que se trata de un subconjunto de casos de la epidemia simulada del dataframe linelist). El paquete spdep tiene una función, moran.test, que puede hacer este cálculo por nosotros:
moran_i <-spdep::moran.test(sle_adm3_dat$cases, # vector numérico con variable de interés.
listw=sle_listw) # objeto listw que resume las relaciones entre vecinos
moran_i # imprimir resultados del test I de Moran##
## Moran I test under randomisation
##
## data: sle_adm3_dat$cases
## weights: sle_listw
##
## Moran I statistic standard deviate = 1.768, p-value = 0.03853
## alternative hypothesis: greater
## sample estimates:
## Moran I statistic Expectation Variance
## 0.24346395 -0.12500000 0.04343311El resultado de la función moran.test() nos muestra una estadística I de Moran de round(moran_i$estimate[1],2). Esto indica la presencia de autocorrelación espacial en nuestros datos; en concreto, sugiere que es probable que las regiones con un número similar de casos de Ébola estén próximas entre sí. El valor p proporcionado por moran.test() se genera mediante la comparación con la expectativa bajo la hipótesis nula de ausencia de autocorrelación espacial, y puede utilizarse si se necesita informar de los resultados de una prueba de hipótesis formal.
I de Moran local - Podemos descomponer la estadística I de Moran (global) calculada anteriormente para identificar la autocorrelación espacial localizada; es decir, para identificar grupos específicos en nuestros datos. Esta estadística, que a veces se denomina indicador local de asociación espacial (LISA), resume el grado de autocorrelación espacial alrededor de cada región individual. Puede ser útil para encontrar puntos “calientes” y “fríos” en el mapa.
Para mostrar un ejemplo, podemos calcular y mapear la I de Moran local para los recuentos de casos de Ébola utilizados anteriormente, con la función local_moran() de spdep:
# calcular I de Moran local
local_moran <- spdep::localmoran(
sle_adm3_dat$cases, # variable de interés
listw=sle_listw # objeto listw con pesos de vecindad
)
# unir los resultados a los datos de sf
sle_adm3_dat<- cbind(sle_adm3_dat, local_moran)
# representar el mapa
ggplot(data=sle_adm3_dat) +
geom_sf(aes(fill=Ii)) +
theme_bw() +
scale_fill_gradient2(low="#2c7bb6", mid="#ffffbf", high="#d7191c",
name="Local Moran's I") +
labs(title="Local Moran's I statistic for Ebola cases",
subtitle="Admin level 3 regions, Sierra Leone")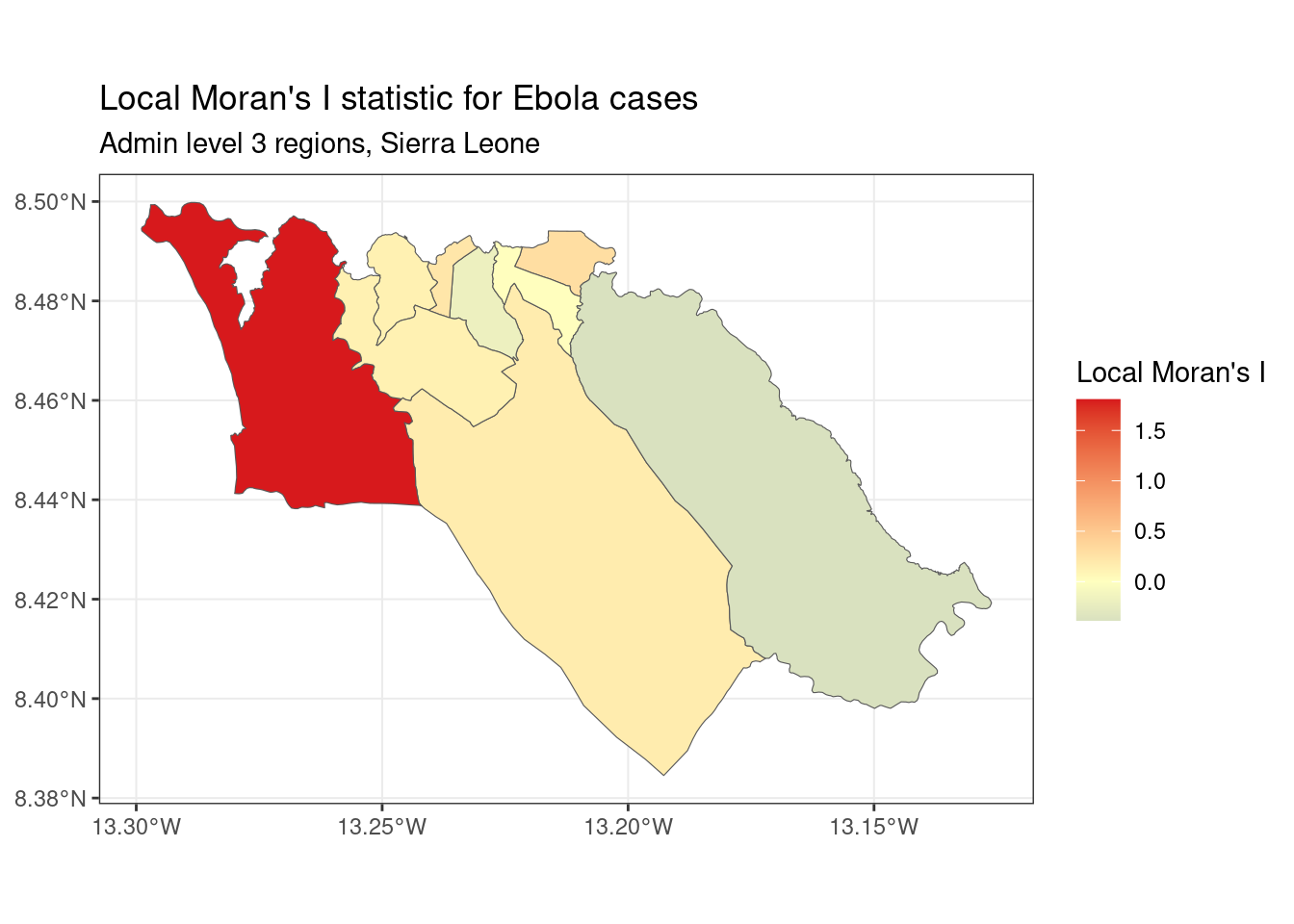
Getis-Ord Gi * - Esta es otra estadística que se utiliza comúnmente para el análisis de puntos calientes; en gran parte, la popularidad de esta estadística se relaciona con su uso en la herramienta de análisis de puntos calientes en ArcGIS. Se basa en la suposición de que, normalmente, la diferencia del valor de una variable entre regiones vecinas debería seguir una distribución normal. Utiliza un enfoque de puntuación z para identificar las regiones que tienen valores significativamente más altos (punto caliente) o significativamente más bajos (punto frío) de una variable específica, en comparación con sus vecinos.
Podemos calcular y asignar la estadística Gi* utilizando la función localG() de spdep:
# Realizar análisis G local
getis_ord <- spdep::localG(
sle_adm3_dat$cases,
sle_listw
)
# join results to sf data
sle_adm3_dat$getis_ord <- getis_ord
# representar el mapa
ggplot(data=sle_adm3_dat) +
geom_sf(aes(fill=getis_ord)) +
theme_bw() +
scale_fill_gradient2(low="#2c7bb6", mid="#ffffbf", high="#d7191c",
name="Gi*") +
labs(title="Getis-Ord Gi* statistic for Ebola cases",
subtitle="Admin level 3 regions, Sierra Leone")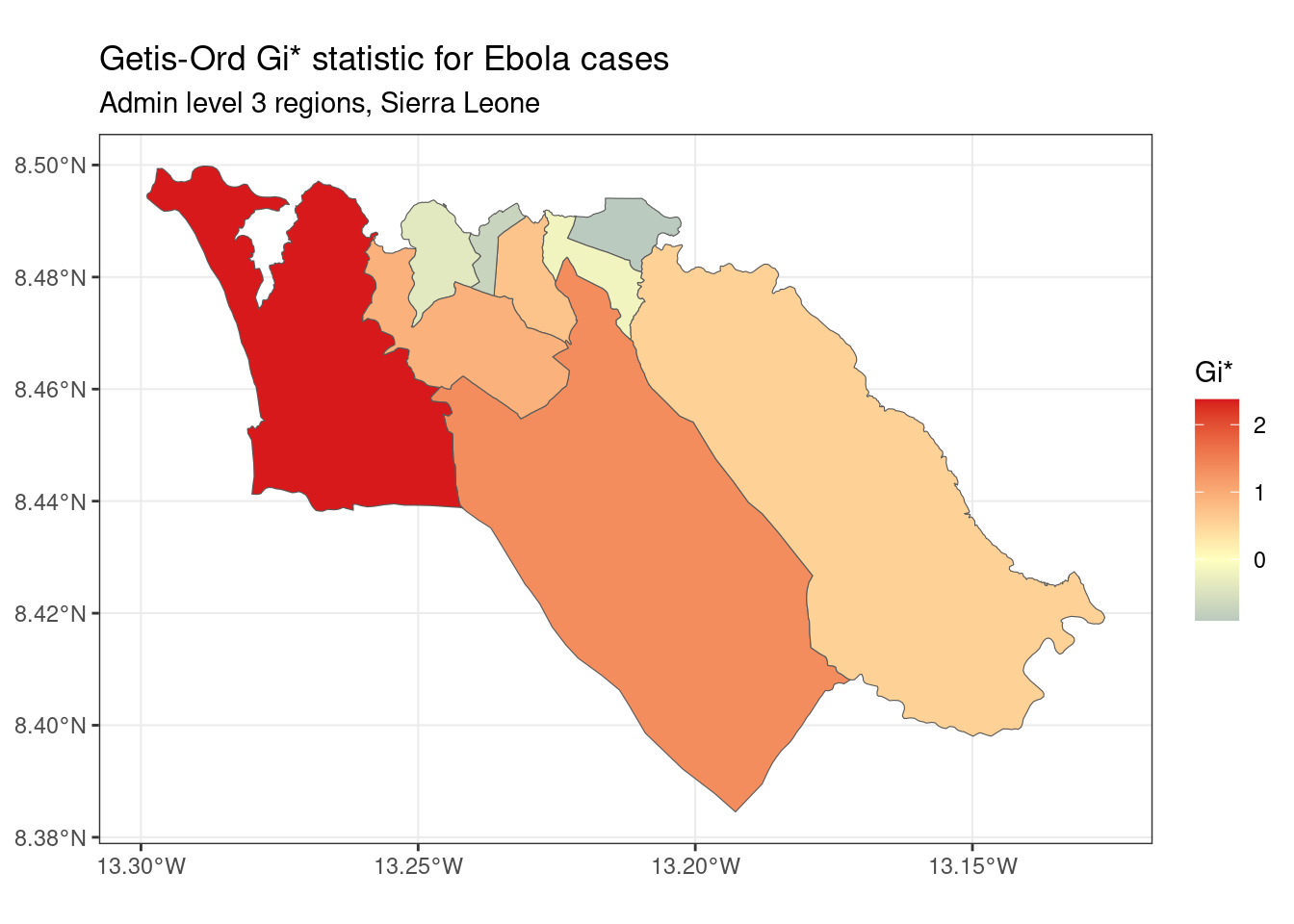
Como puedes ver, el mapa de Getis-Ord Gi* tiene un aspecto ligeramente diferente del mapa de Moran local elaborado anteriormente. Esto refleja que el método utilizado para calcular estas dos estadísticas es ligeramente diferente. Cuál de ellas debes utilizar depende de tu caso de uso específico y de la pregunta de investigación de interés.
Prueba L de Lee - Es una prueba estadística de correlación espacial bivariada. Permite comprobar si el patrón espacial de una determinada variable x es similar al patrón espacial de otra variable, y, que se supone que está relacionada espacialmente con x.
Para dar un ejemplo, vamos a probar si el patrón espacial de los casos de Ébola de la epidemia simulada está correlacionado con el patrón espacial de la población. Para empezar, necesitamos tener una variable population en nuestros datos sle_adm3. Podemos utilizar la variable total del dataframe sle_adm3_pop que hemos cargado anteriormente.
Podemos visualizar rápidamente los patrones espaciales de las dos variables una al lado de la otra, para ver si se parecen:
tmap_mode("plot")
cases_map <- tm_shape(sle_adm3_dat) + tm_polygons("cases") + tm_layout(main.title="Cases")
pop_map <- tm_shape(sle_adm3_dat) + tm_polygons("population") + tm_layout(main.title="Population")
tmap_arrange(cases_map, pop_map, ncol=2) # organizar en 2 x 1 facetas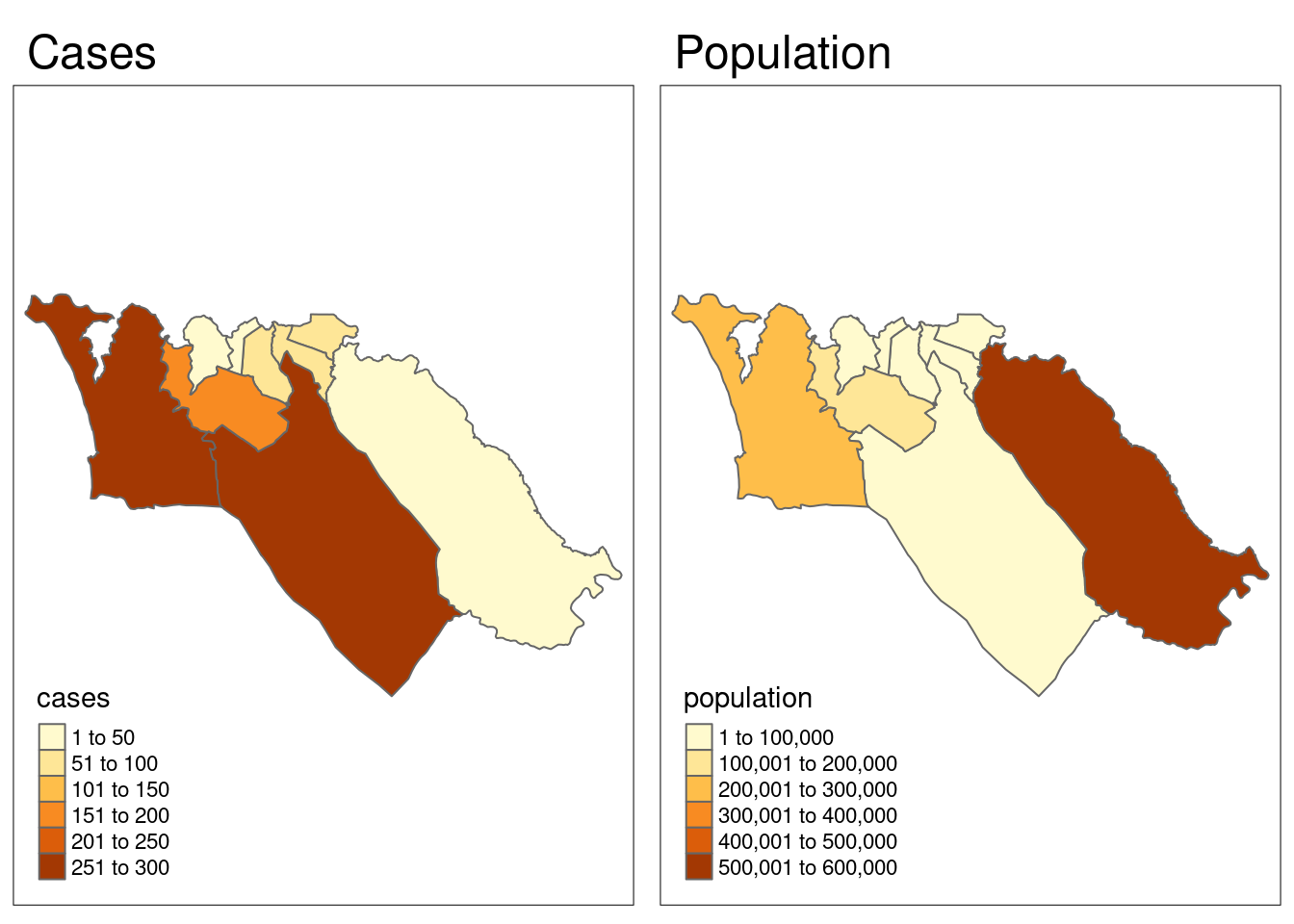
Visualmente, los patrones parecen no parecen muy similares. Podemos utilizar la función lee.test() de spdep para comprobar estadísticamente si el patrón de autocorrelación espacial de las dos variables está relacionado. La estadística L será cercana a 0 si no hay correlación entre los patrones, cercana a 1 si hay una fuerte correlación positiva (es decir, los patrones son similares), y cercana a -1 si hay una fuerte correlación negativa (es decir, los patrones son inversos).
lee_test <- spdep::lee.test(
x=sle_adm3_dat$cases, # variable 1 a comparar
y=sle_adm3_dat$population, # variable 2 a comparar
listw=sle_listw # objeto listw con pesos de vecindad
)
lee_test##
## Lee's L statistic randomisation
##
## data: sle_adm3_dat$cases , sle_adm3_dat$population
## weights: sle_listw
##
## Lee's L statistic standard deviate = -0.94386, p-value = 0.8274
## alternative hypothesis: greater
## sample estimates:
## Lee's L statistic Expectation Variance
## -0.14347788 -0.03860690 0.01234515El resultado anterior muestra que la estadística L de Lee para nuestras dos variables fue round(lee_test$estimate[1],2), lo que indica una débil correlación negativa. Esto confirma nuestra evaluación visual de que el patrón de los casos y la población no están relacionados entre sí, y proporciona pruebas de que el patrón espacial de los casos no es estrictamente un resultado de la densidad de población en las zonas de alto riesgo.
La estadística L de Lee puede ser útil para hacer este tipo de inferencias sobre la relación entre variables distribuidas espacialmente; sin embargo, para describir la naturaleza de la relación entre dos variables con más detalle, o ajustar por confusión, tenemos que aplicar técnicas de regresión espacial. Describeremos brevemente algunas de estas en la siguiente sección.
Regresión espacial
Es posible que quieras hacer inferencias estadísticas sobre las relaciones entre las variables de tus datos espaciales. En estos casos, es útil considerar las técnicas de regresión espacial, es decir, los enfoques de regresión que consideran explícitamente la organización espacial de las unidades en los datos. Algunas de las razones por las que puedes necesitar considerar modelos de regresión espacial, en lugar de modelos de regresión estándar como los GLM, incluyen:
Los modelos de regresión estándar asumen que los residuos son independientes entre sí. En presencia de una autocorrelación espacial fuerte, es probable que los residuos de un modelo de regresión estándar también estén autocorrelacionados espacialmente, violando así este supuesto. Esto puede dar lugar a problemas de interpretación de los resultados del modelo, en cuyo caso sería preferible un modelo espacial.
Los modelos de regresión también suelen suponer que el efecto de una variable x es constante en todas las observaciones. En el caso de la heterogeneidad espacial, los efectos que deseamos estimar pueden variar a lo largo del espacio, y podemos estar interesados en cuantificar esas diferencias. En este caso, los modelos de regresión espacial ofrecen más flexibilidad para estimar e interpretar los efectos.
Los detalles de los enfoques de regresión espacial están fuera del alcance de este manual. En su lugar, esta sección ofrece una visión general de los modelos de regresión espacial más comunes y sus usos, y te remite a referencias que pueden ser útiles por si se deseas profundizar en este ámbito.
Modelos de error espacial - Estos modelos suponen que los términos de error entre unidades espaciales están correlacionados, en cuyo caso los datos violarían los supuestos de un modelo OLS estándar. Los modelos de error espacial también se denominan a veces modelos autorregresivos simultáneos (SAR). Pueden ajustarse utilizando la función errorsarlm() del paquete spatialreg (funciones de regresión espacial que solían formar parte de spdep).
Modelos de desfase espacial - Estos modelos suponen que la variable dependiente de una región i está influida no sólo por el valor de las variables independientes en i, sino también por los valores de esas variables en las regiones vecinas a i. Al igual que los modelos de error espacial, los modelos de desfase espacial también se describen a veces como modelos autorregresivos simultáneos (SAR). Pueden ajustarse utilizando la función lagsarlm() del paquete spatialreg.
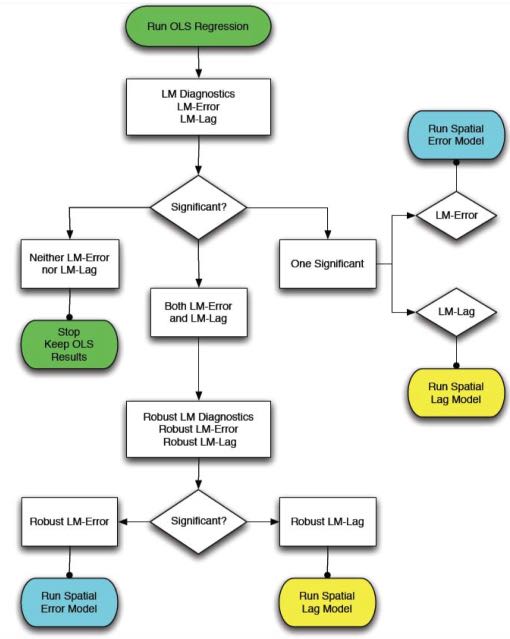
El paquete spdep contiene varias pruebas de diagnóstico útiles para decidir entre los modelos OLS estándar, de desfase espacial y de error espacial. Estas pruebas, denominadas diagnósticos del multiplicador de Lagrange, pueden utilizarse para identificar el tipo de dependencia espacial en sus datos y elegir el modelo más apropiado. La función lm.LMtests() puede utilizarse para calcular todos los diagnósticos del multiplicador de Lagrange. Anselin (1988) también proporciona una útil herramienta de diagrama de flujo para decidir qué modelo de regresión espacial utilizar basándose en los resultados de las pruebas del multiplicador de Lagrange:
Modelos jerárquicos bayesianos: Los enfoques bayesianos se utilizan habitualmente para algunas aplicaciones del análisis espacial, sobre todo para el mapeo de enfermedades. Se prefieren en los casos en los que los datos de los casos están escasamente distribuidos (por ejemplo, en el caso de un resultado raro) o son estadísticamente “ruidosos”, ya que pueden utilizarse para generar estimaciones “suavizadas” del riesgo de enfermedad al tener en cuenta el proceso espacial latente subyacente. Esto puede mejorar la calidad de las estimaciones. También permiten que el investigador especifique previamente (mediante la elección de “priors” (valores pre-establecidos)) los patrones complejos de correlación espacial que pueden existir en los datos, los cuales pueden tomar en cuenta la variación espacialmente dependiente e independiente en las variables independientes y dependientes. En R, los modelos jerárquicos bayesianos pueden ajustarse utilizando el paquete CARbayes (véase la viñeta) o R-INLA (véase este sitio web y el libro de texto). A través de R también puedes usar software externo que realice estimaciones bayesianas, como JAGS o WinBUGS.
28.12 Recursos
Funciones simples de R y viñeta del paquete sf
Introducción a la elaboración de mapas con R, visión general de los diferentes paquetes
Datos espaciales en R (curso EarthLab)
Libro de texto Applied Spatial Data Analysis in R
SpatialEpiApp - una aplicación Shiny que se puede descargar como un paquete de R, lo que le permite proporcionar sus propios datos y llevar a cabo la cartografía, el análisis de conglomerados y las estadísticas espaciales.
Taller de introducción a la econometría espacial en R