32 Curvas epidémicas
Una curva epidémica (también conocida como “curva epi”) es un gráfico epidemiológico básico que se suele utilizar para visualizar el patrón temporal de aparición de la enfermedad entre un grupo o epidemia de casos.
El análisis de la curva epidémica puede revelar tendencias temporales, valores atípicos, la magnitud del brote, el periodo de exposición más probable, los intervalos de tiempo entre las generaciones de casos, e incluso puede ayudar a identificar el modo de transmisión de una enfermedad no identificada (por ejemplo, fuente puntual, fuente común continua, propagación de persona a persona). En el sitio web de los CDC de EE.UU. se puede encontrar una lección en línea sobre la interpretación de las curvas epidémicas.
En esta página mostramos dos enfoques para producir curva epidémicas en R:
- El paquete incidence2, que puede producir una curva epi con simples comandos
- El paquete ggplot2, que permite una personalización avanzada mediante comandos más complejos
También se abordan casos de uso específicos como:
- Grágicos de datos de recuento agregados
- Facetado o producción de múltiplos gráficos pequeños
- Aplicación de medias móviles
- Mostrar qué datos son “provisionales” o están sujetos a retrasos en la presentación de informes
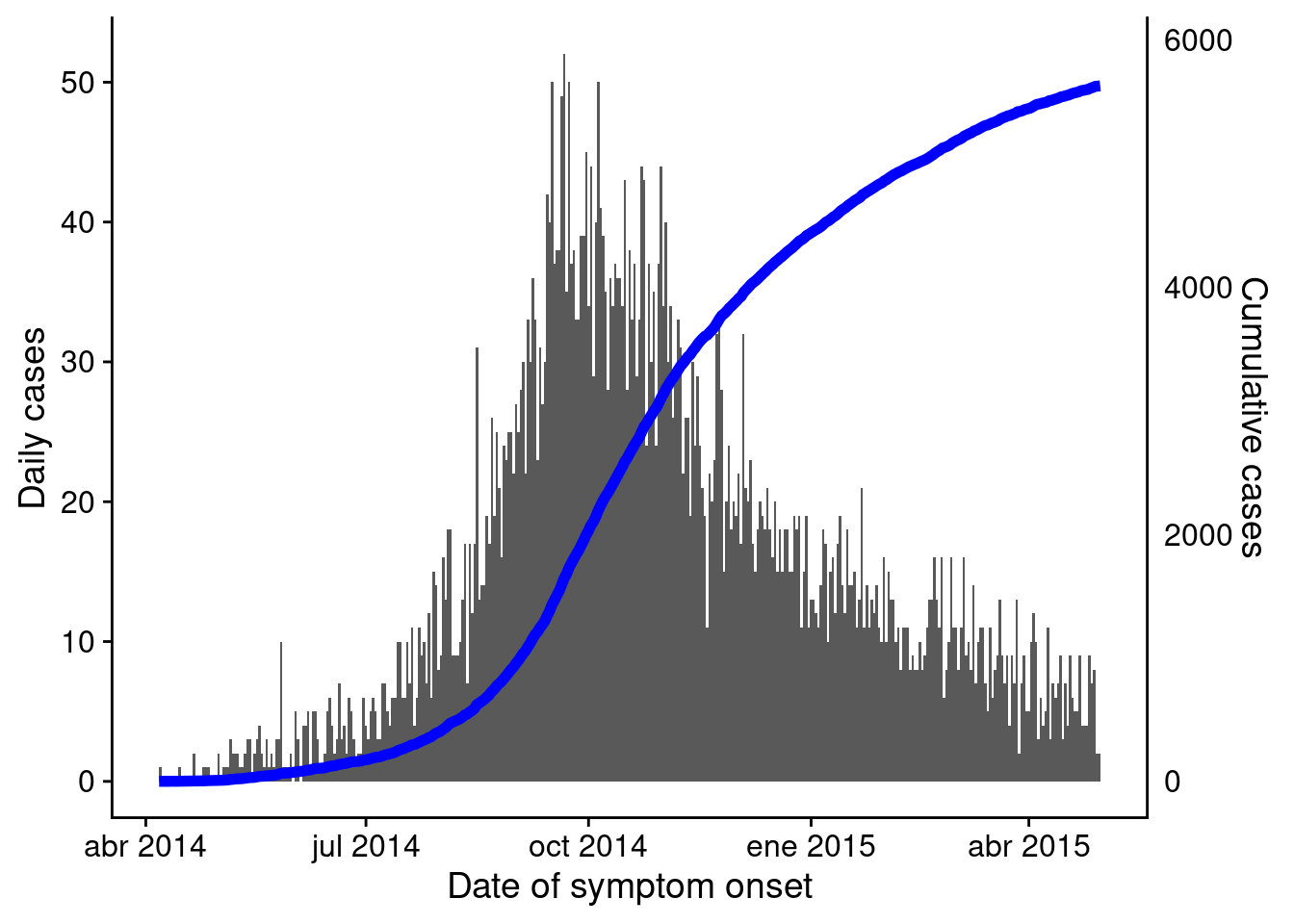
- Superposición de la incidencia de casos acumulados mediante un segundo eje
32.1 Preparación
Paquetes
Este trozo de código muestra la carga de los paquetes necesarios para los análisis. En este manual destacamos p_load() de pacman, que instala el paquete si es necesario y lo carga para su uso. También puede cargar los paquetes instalados con library() de R base. Consulta la página sobre fundamentos de R para obtener más información sobre los paquetes de R.
pacman::p_load(
rio, # importación/exportación de archivos
here, # rutas relativas
lubridate, # trabajar con fechas/semanas epid.
aweek, # paquete alternativo para trabajar con fechas/semanas epid.
incidence2, # epicurvas de datos linelist
i2extras, # suplemento a incidence2
stringr, # buscar y manipular cadenas de caracteres
forcats, # trabajar con factores
RColorBrewer, # paletas de colores de colorbrewer2.org
tidyverse # gestión de datos + gráficos ggplot2
) Importar datos
En esta sección se utilizan dos conjuntos de datos de ejemplo:
-
Linelistcon casos individuales de una epidemia simulada - Recuentos agregados por hospital de la misma epidemia simulada
Los datos se importan mediante la función import() del paquete rio. Consulta la página sobre importación y exportación para conocer las distintas formas de importar datos.
Linelist con casos
Importamos los datos de casos de una epidemia de ébola simulada. Si deseas descargar los datos para seguirlos paso a paso, consulta las instrucciones en la página Descargando el manual y los datos. Asumimos que el archivo está en el directorio de trabajo, por lo que no se especifican subcarpetas en esta ruta de archivo.
linelist <- import("linelist_cleaned.rds")A continuación se muestran las primeras 50 filas.
Recuentos de casos agregados por hospital
A efectos del manual, los datos de recuentos semanales agregados por hospital se crean a partir de linelist con el siguiente código.
# importar los datos de los recuentos a R
count_data <- linelist %>%
group_by(hospital, date_hospitalisation) %>%
summarize(n_cases = dplyr::n()) %>%
filter(date_hospitalisation > as.Date("2013-06-01")) %>%
ungroup()A continuación se muestran las primeras 50 filas:
Establecer parámetros
Para la producción de un informe, es posible que desees establecer parámetros editables como la fecha para la que los datos sean actuales (la “data_date”). A continuación, puedes hacer referencia al objeto data_date en tu código cuando apliques filtros o en subtítulos dinámicos.
## establece la fecha del informe
## nota: se puede establecer en Sys.Date() para la fecha actual
data_date <- as.Date("2015-05-15")Verificar las fechas
Verifica que cada columna de fecha relevante es de tipo Date y tiene un rango de valores apropiado. Puedes hacerlo simplemente utilizando hist() para histogramas, o range() con na.rm=TRUE, o con ggplot() como se indica a continuación.
# comprobar el intervalo de fechas de inicio
ggplot(data = linelist)+
geom_histogram(aes(x = date_onset))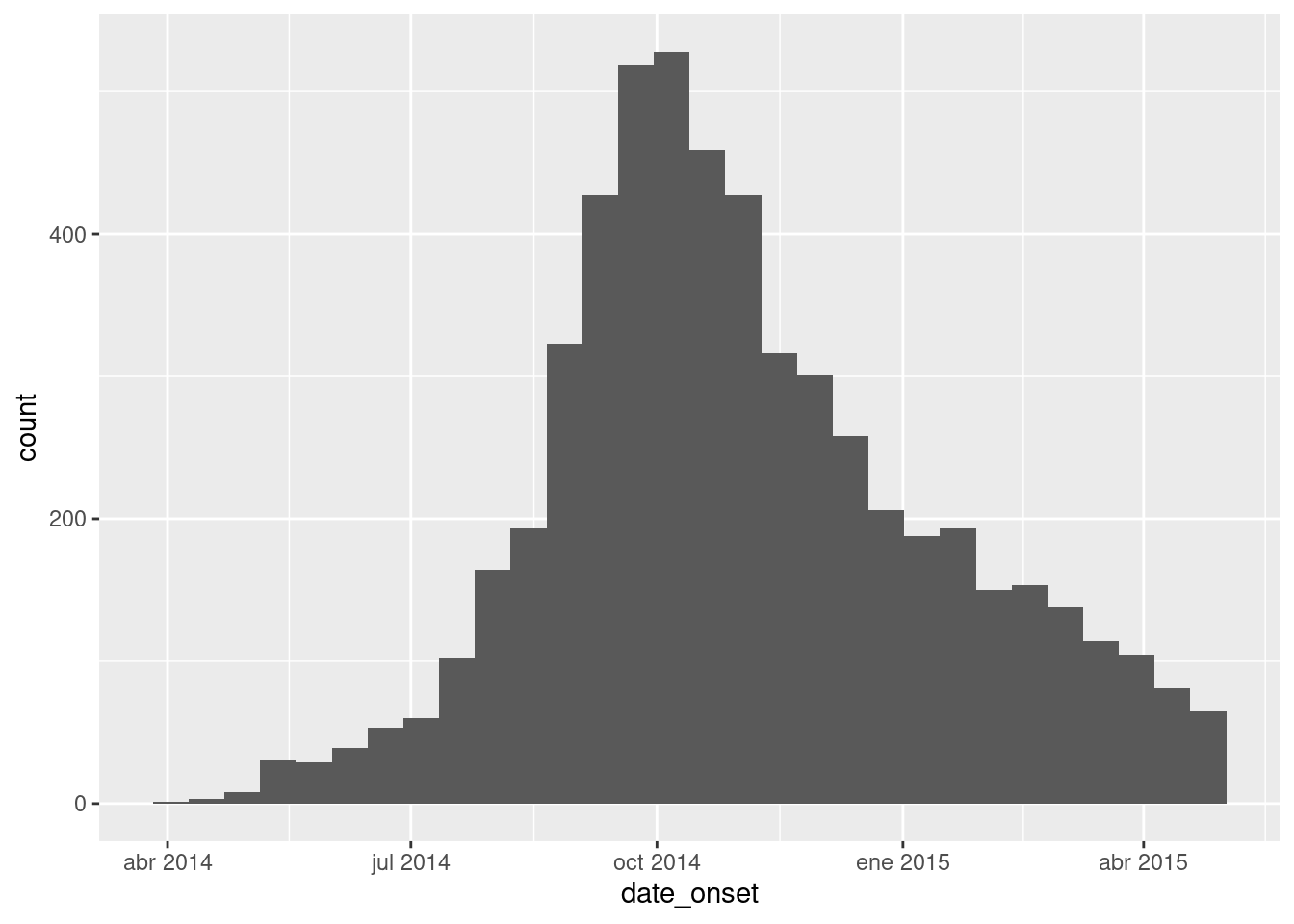
32.2 Epicurves con el paquete incidence2
A continuación mostramos cómo hacer curvas epidémicas utilizando el paquete incidence2. Los autores de este paquete han intentado que el usuario pueda crear y modificar curvas epidémicas sin necesidad de conocer la sintaxis de ggplot2. Gran parte de esta página está adaptada de las viñetas del paquete, que se pueden encontrar en la página de github de incidence2.
Ejemplo sencillo
Se requieren 2 pasos para trazar una curva epidémica con el paquete incidence2:
-
Crear un objeto de incidencia (utilizando la función
incidence())- Proporcionar los datos
- Especificar la columna de fecha a
date_index = - Especificar el
interval =en el que deben agregarse los casos (diario, semanal, mensual..) - Especificar cualquier columna de agrupación (por ejemplo, sexo, hospital, resultado)
-
Representarr el objeto de incidencia
- Especificar etiquetas, colores, títulos, etc.
A continuación, cargamos el paquete incidence2, creamos el objeto de incidencia a partir de linelist en la columna date_onset y agregamos los casos por día. A continuación, imprimimos un resumen del objeto de incidencia.
# carga el paquete incidence2
pacman::p_load(incidence2)
# crea el objeto de incidencia, agregando los casos por día
epi_day <- incidence( # crea un objeto de incidencia
x = linelist, # conjunto de datos
date_index = date_onset, # columna de fecha
interval = "day" # intervalo de agrupación de fechas
)El objeto epi_day2 tiene el aspecto de un tibble (como una trama de datos) y puede imprimirse o manipularse como un dataframe.
class(epi_day)## [1] "incidence2" "incidence_df" "tbl_df" "tbl" "data.frame"Este es el aspecto que tiene cuando se imprime. Tiene una columna date_index y una columna count.
epi_day## An incidence object: 367 x 2
## date range: [2014-04-07] to [2015-04-30]
## cases: 5632
## interval: 1 day
## cumulative: FALSE
##
## date_index count
## <date> <int>
## 1 2014-04-07 1
## 2 2014-04-15 1
## 3 2014-04-21 2
## 4 2014-04-25 1
## 5 2014-04-26 1
## 6 2014-04-27 1
## 7 2014-05-01 2
## 8 2014-05-03 1
## 9 2014-05-04 1
## 10 2014-05-05 1
## # … with 357 more rowsTambién puedes imprimir un resumen del objeto:
# imprime el resumen del objeto de incidencia
summary(epi_day)## date range: [2014-04-07] to [2015-04-30]
## cases: 5632
## interval: 1 day
## cumulative: FALSE
## timespan: 389 daysPara trazar el objeto de incidencia, utiliza plot() en el nombre del objeto de incidencia. En segundo plano, se llama a la función plot.incidence2(), por lo que para leer la documentación específica de incidence2** se ejecutaría ?plot.incidence2.
# representa el objeto de incidencia
plot(epi_day)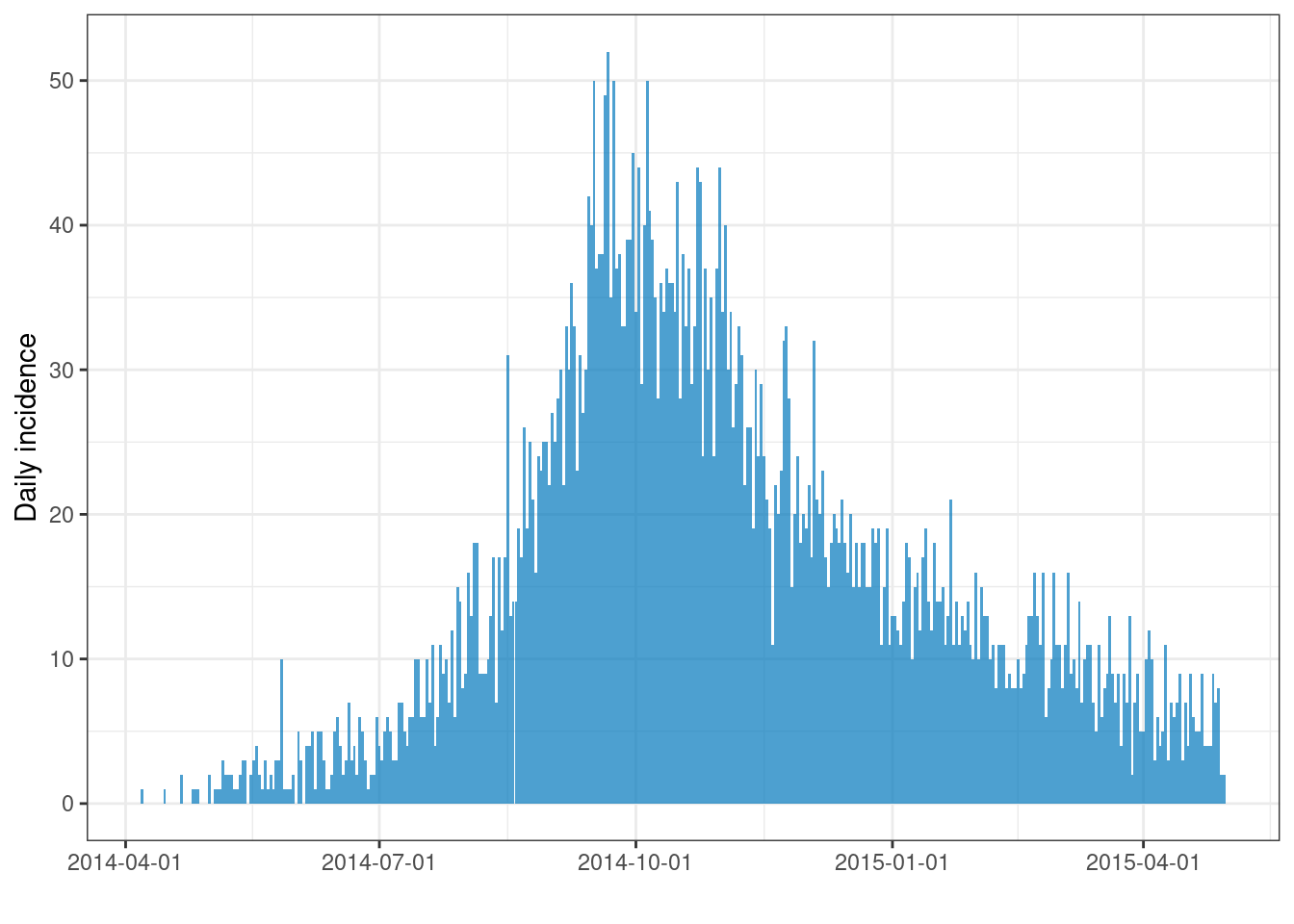
Si notas muchas líneas blancas verticales diminutas, intenta ajustar el tamaño de la imagen. Por ejemplo, si exportas el gráfico con ggsave(), puede proporcionar números a width = y height =. Si amplías el gráfico esas líneas pueden desaparecer.
Cambiar el intervalo de tiempo de la agregación de casos
El argumento interval = de incidence() define cómo se agrupan las observaciones en barras verticales.
Especificar el intervalo
incidence2 proporciona flexibilidad y una sintaxis comprensible para especificar cómo quieres agregar tus casos en una curva epidémica de barras Proporcione un valor como los siguientes al argumento interval =. Puedes escribir cualquiera de los siguientes en plural (por ejemplo, “weeks”), y puedes añadir números antes (por ejemplo, “3 months”).
| Opción de argumento | Más explicaciones |
|---|---|
| Número (1, 7, 13, 14, etc.) | Número de días por intervalo |
| “week” | nota: el lunes es el día de inicio predeterminad |
| “2 weeks” | o 3, 4, 5… |
| “Sunday week” | semanas que comienzan en domingo (también se puede utilizar el Thursday, etc.) |
| “2 Sunday weeks” | o 3, 4, 5… |
| “MMWRweek” | la semana comienza en domingo - ver US CDC |
| “month” | El 1 de mes |
| “quarter” | 1er mes del trimestre |
| “2 months” | o 3, 4, 5… |
| “year” | Primer día del año natural |
A continuación se muestran ejemplos de cómo se ven los diferentes intervalos cuando se aplican a linelist. Observa cómo el formato por defecto y la frecuencia de las etiquetas de fecha en el eje-x cambian a medida que cambia el intervalo de fecha.
# Crear los objetos de incidencia (con diferentes intervalos)
#############################################################
# Semanal ( semana desde el lunes, por defecto)
epi_wk <- incidence(linelist, date_onset, interval = "Monday week")
# Semana desde el domingo
epi_Sun_wk <- incidence(linelist, date_onset, interval = "Sunday week")
# Tres semanas (semana del lunes por defecto)
epi_2wk <- incidence(linelist, date_onset, interval = "2 weeks")
# Mensual
epi_month <- incidence(linelist, date_onset, interval = "month")
# Trimestral
epi_quarter <- incidence(linelist, date_onset, interval = "quarter")
# Años
epi_year <- incidence(linelist, date_onset, interval = "year")
# Graficar los objetos de incidencia (+ títulos para mayor claridad)
####################################################################
plot(epi_wk)+ labs(title = "Monday weeks")
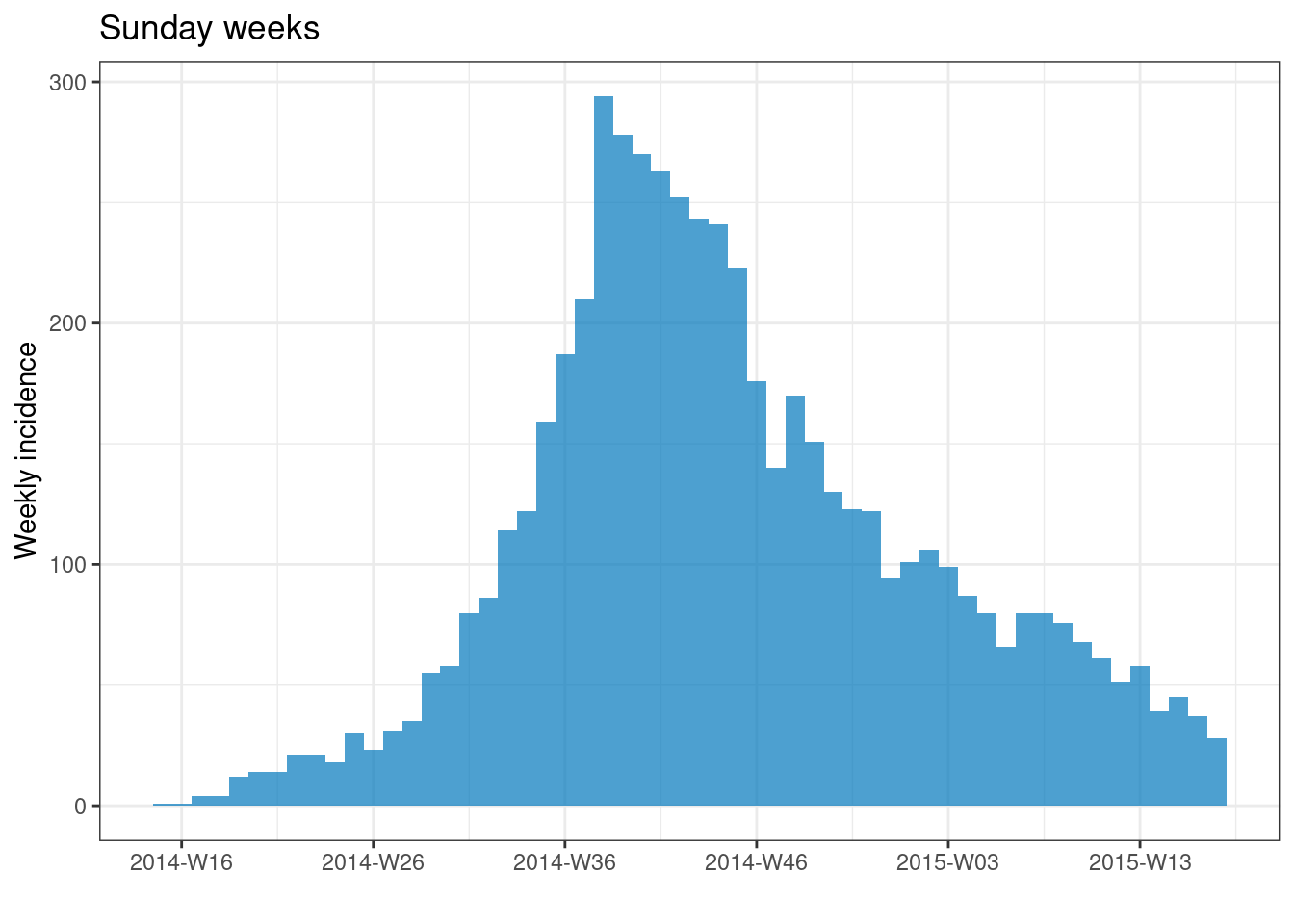
plot(epi_Sun_wk)+ labs(title = "Sunday weeks")
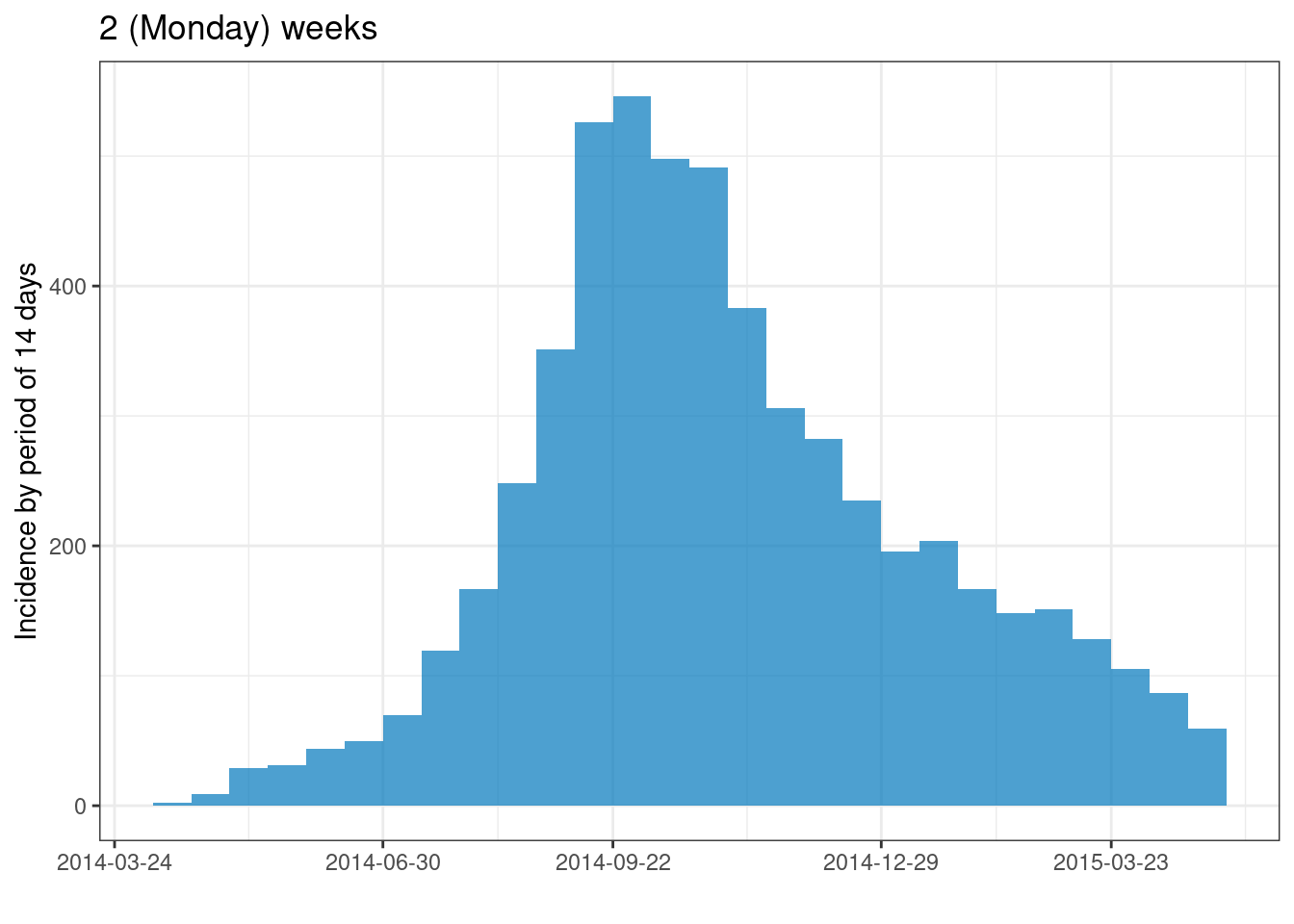
plot(epi_2wk)+ labs(title = "2 (Monday) weeks")
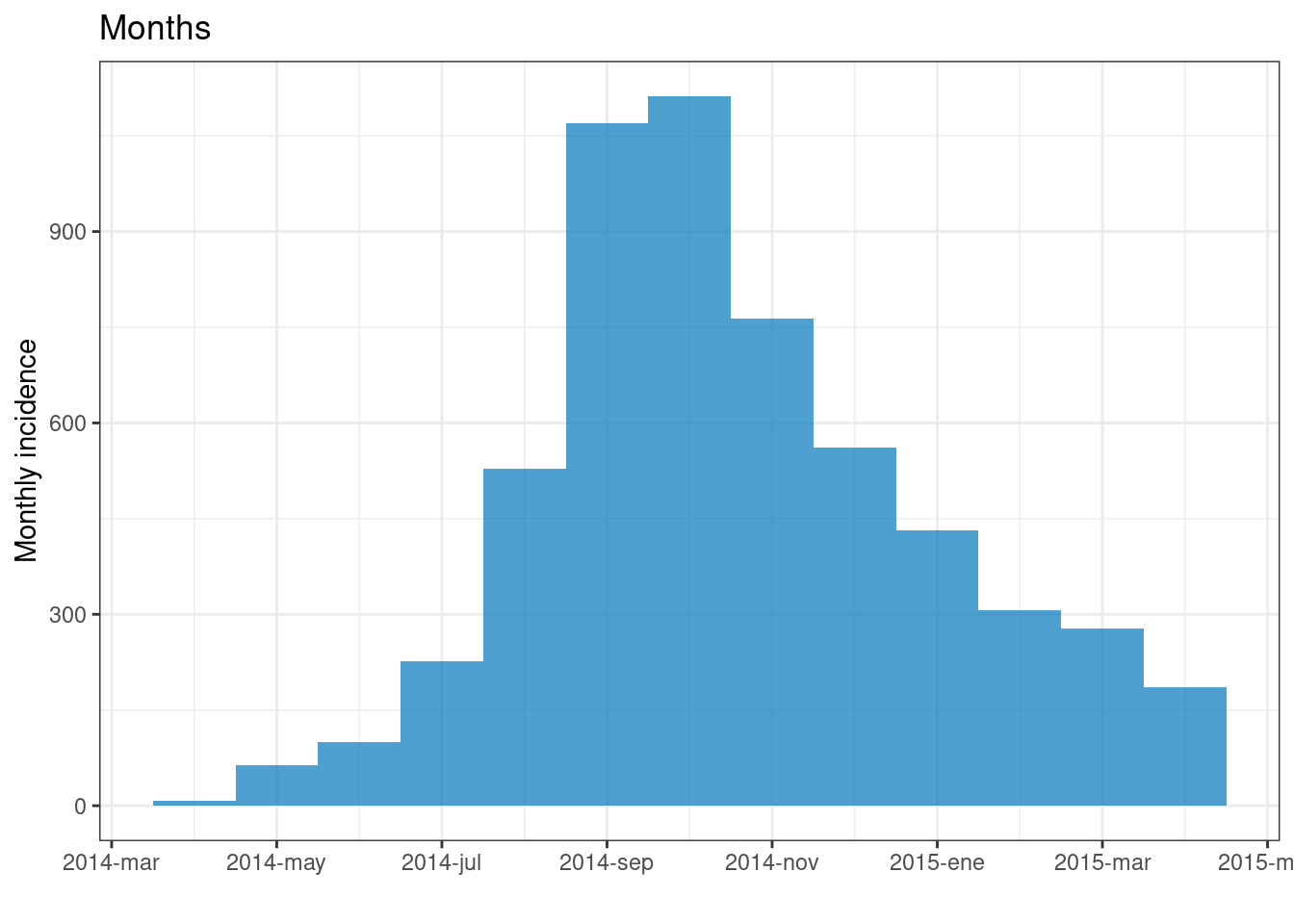
plot(epi_month)+ labs(title = "Months")
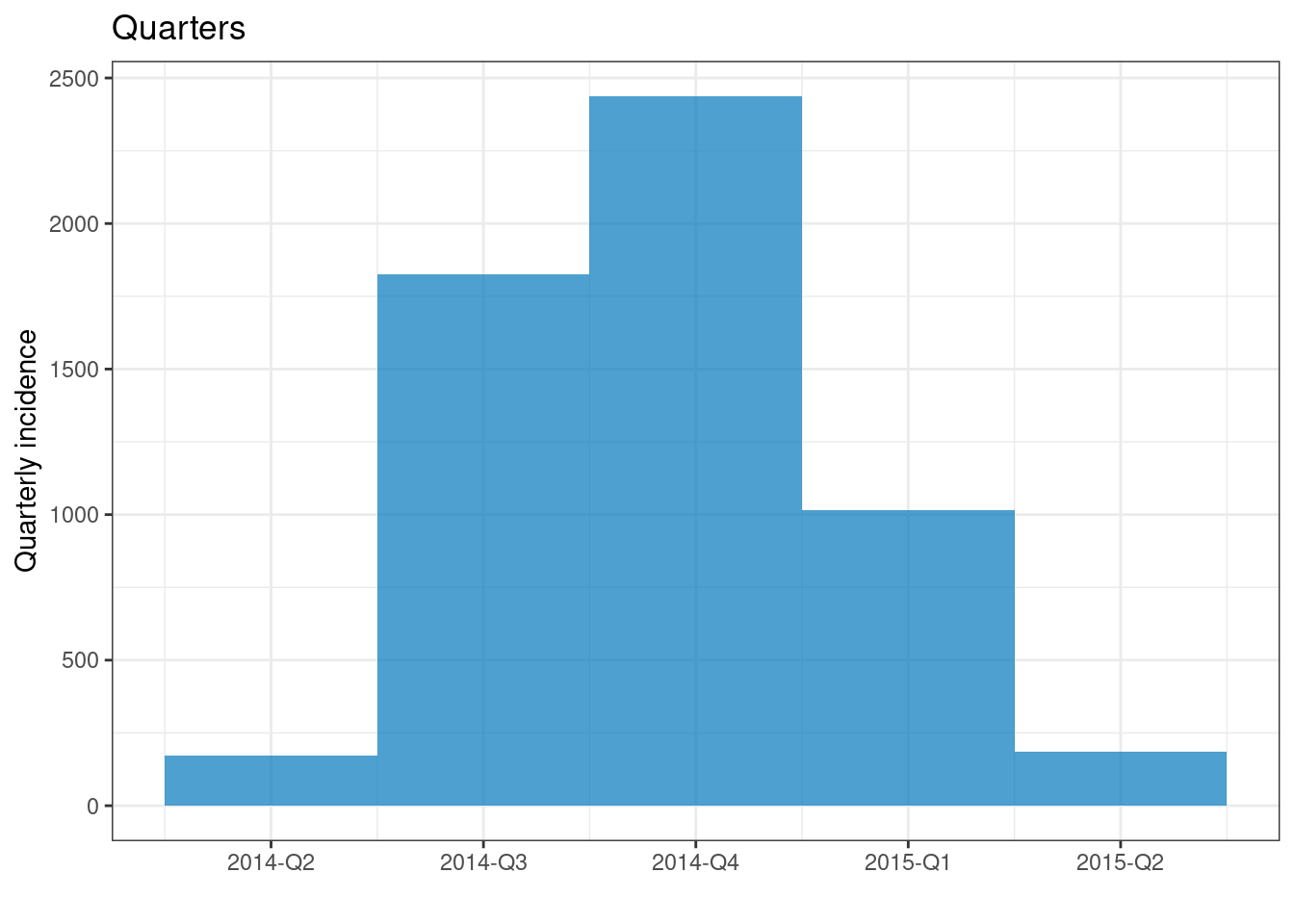
plot(epi_quarter)+ labs(title = "Quarters")
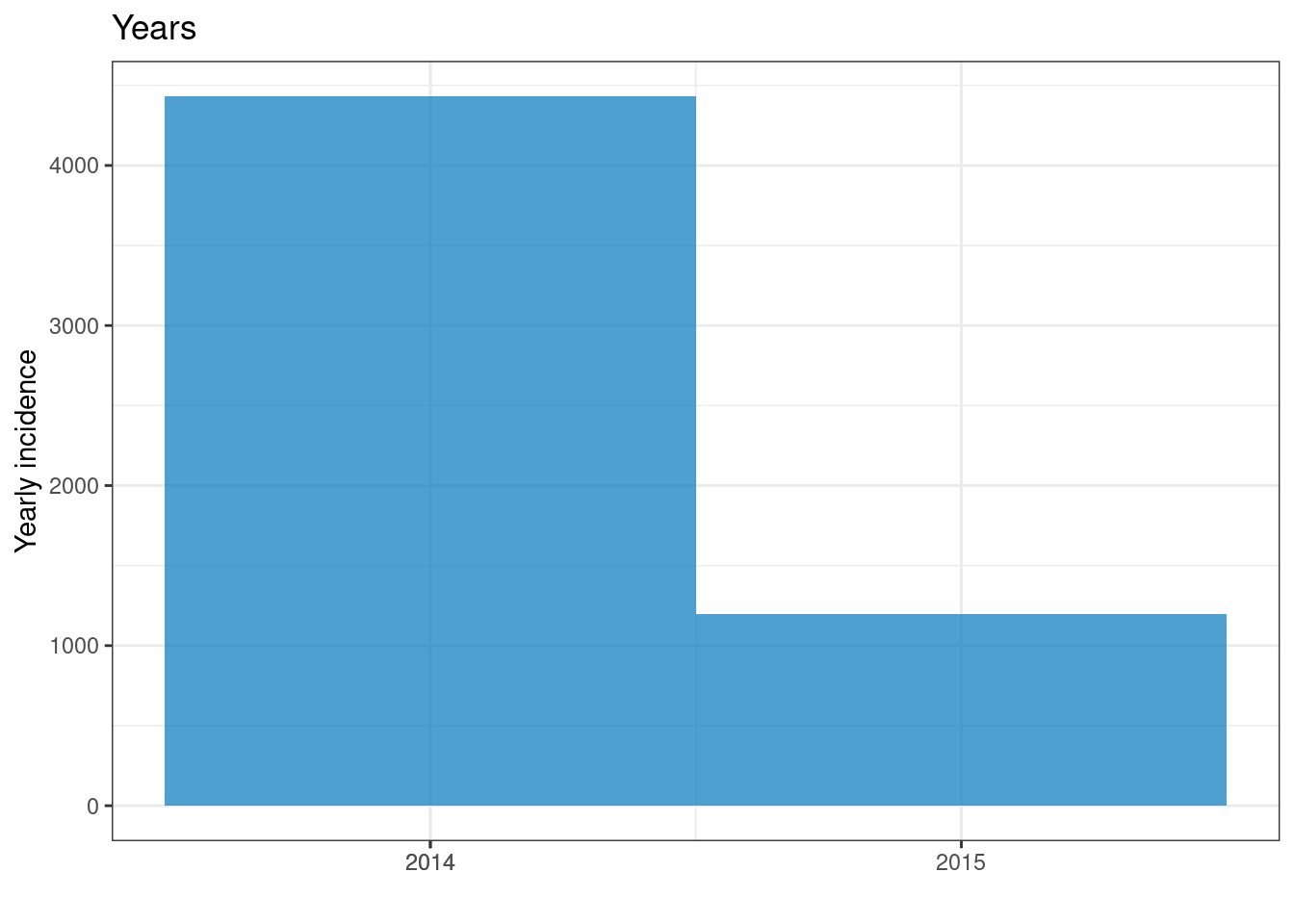
plot(epi_year)+ labs(title = "Years")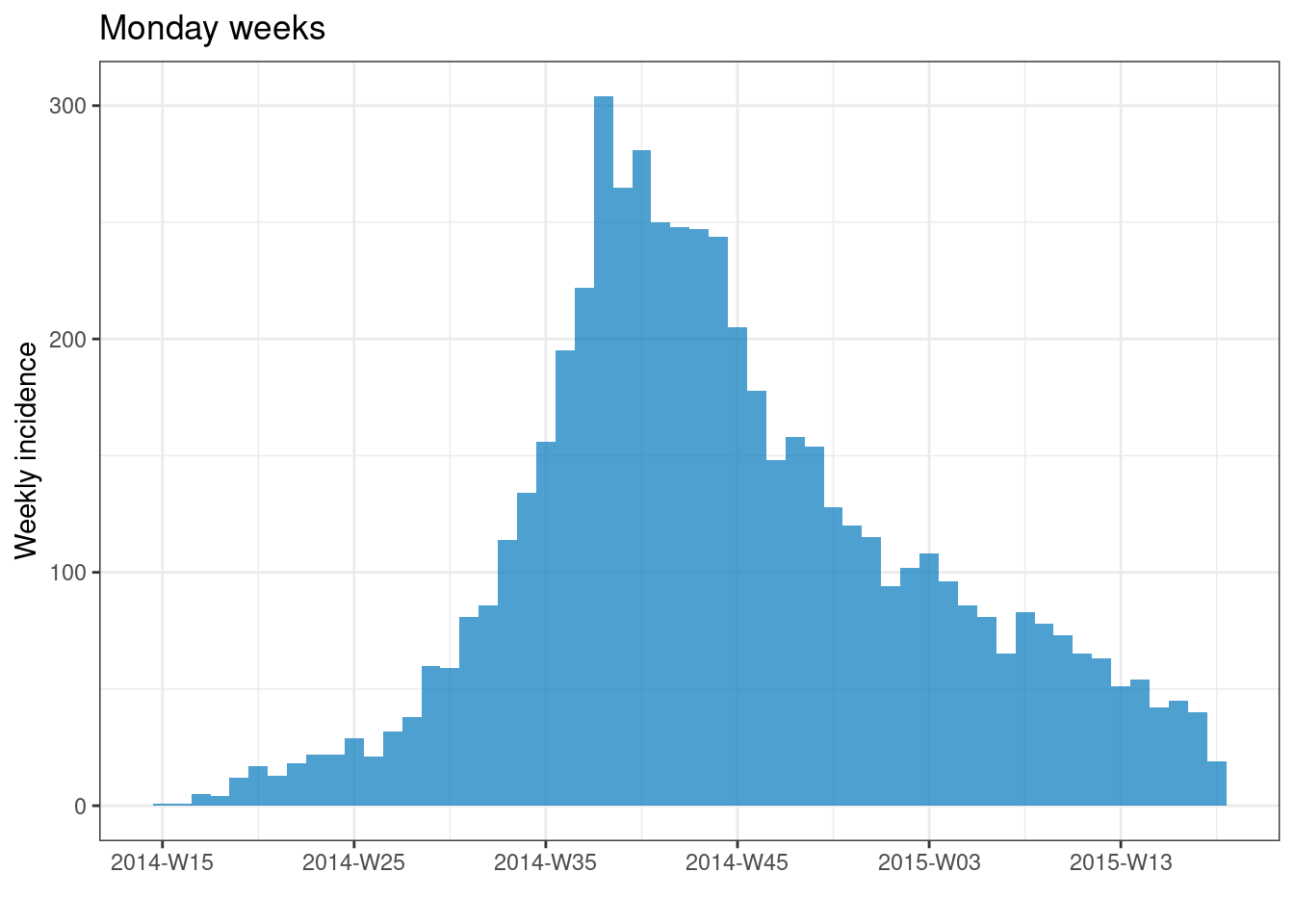
Primera fecha
Puedes especificar opcionalmente un valor de tipo Date (por ejemplo, as.Date("2016-05-01")) a firstdate = en el comando incidence(). Si se da, los datos se recortarán a este rango y los intervalos comenzarán en esta fecha.
Grupos
Los grupos se especifican en el comando incidence(), y pueden utilizarse para colorear las barras o para facetar los datos. Para especificar los grupos en tus datos, escribe el nombre de la(s) columna(s) en el argumento groups = del comando incidence() (sin comillas alrededor del nombre de la columna). Si especificas varias columnas, pon sus nombres dentro de c().
Puedes especificar que los casos con valores faltantes en las columnas de agrupación sean listados como un grupo NA distinto estableciendo na_as_group = TRUE. De lo contrario, se excluirán del gráfico.
Para colorear las barras por una columna de agrupación, debes proporcionar de nuevo el nombre de la columna en
fill =del comandoplot().Para crear una faceta basada en una columna de agrupación, consulta la sección siguiente sobre facetas con incidence2.
En el ejemplo siguiente, los casos de todo el brote se agrupan por su categoría de edad. Los valores faltantes se incluyen como grupo. El intervalo de la curva epidémica es de semanas.
# Crear objeto de incidencia, con datos agrupados por categoría de edad
age_outbreak <- incidence(
linelist, # conjunto de datos
date_index = date_onset, # columna de fecha
interval = "week", # agregación de casos semanal desde el lunes
groups = age_cat, # se establece age_cat como grupo
na_as_group = TRUE) # valores faltantes asignados a su propio grupo
# dibujar el objeto de incidencia agrupado
plot(
age_outbreak, # objeto de incidencia con age_cat como grupo
fill = age_cat)+ # age_cat se utiliza para el color de relleno de la barra (debe haberse establecido anteriormente como columna de grupo)
labs(fill = "Age Category") # cambia el título de la leyenda de "age_cat" por defecto (esta es una modificación de ggplot2)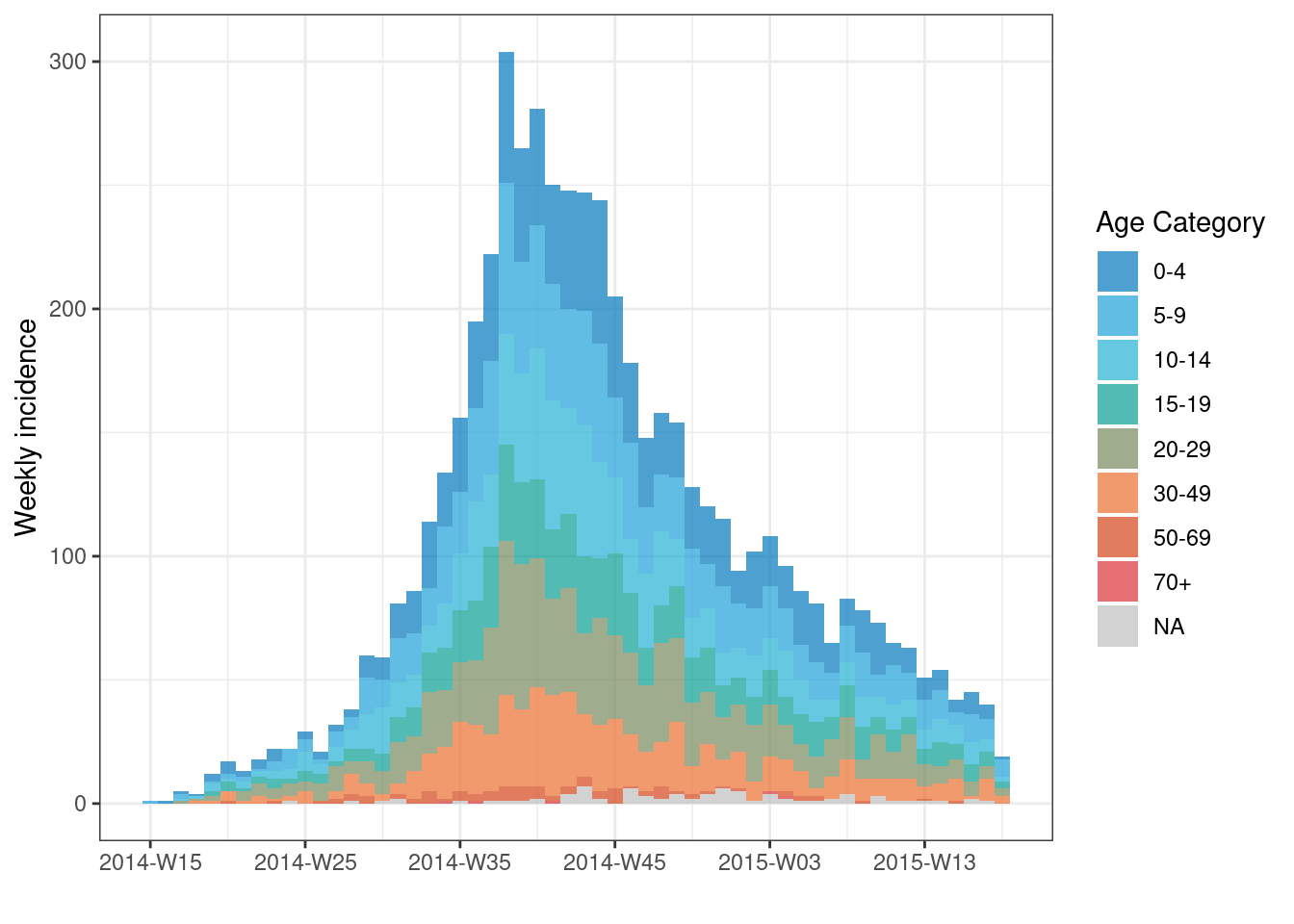 CONSEJO: Cambia el título de la leyenda añadiendo
CONSEJO: Cambia el título de la leyenda añadiendo + el comando de ggplot2 labs(fill = "su título")
También puedes hacer que las barras agrupadas se muestren una al lado de la otra estableciendo stack = FALSE en plot(), como se muestra a continuación:
# Crea el objeto de incidencia de recuentos mensuales.
monthly_gender <- incidence(
linelist,
date_index = date_onset,
interval = "month",
groups = gender # Establece gender como columna de agrupación
)
plot(
monthly_gender, # objeto de incidencia
fill = gender, # muestra barras coloreadas por género
stack = FALSE) # lado a lado (no apiladas)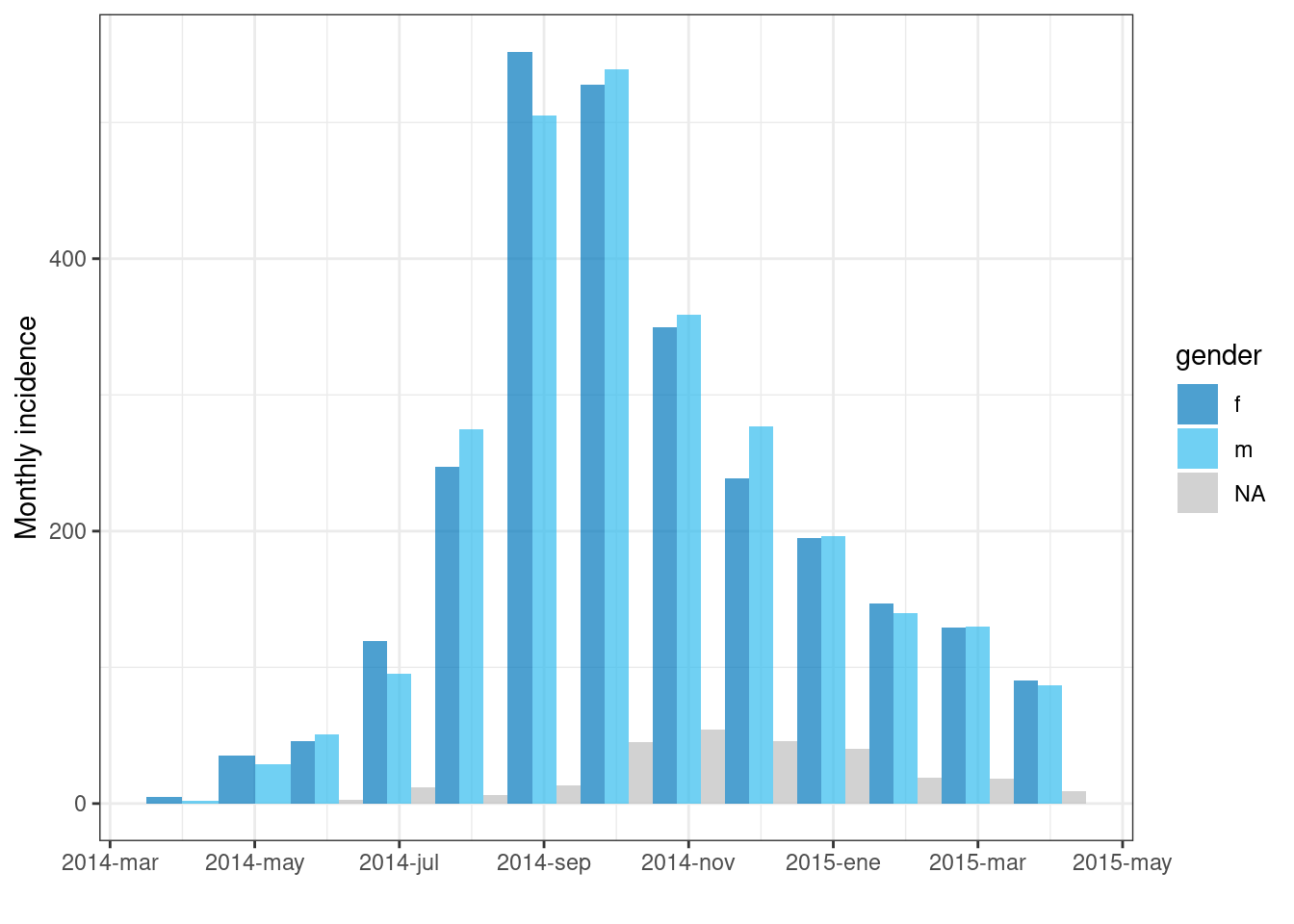
Puedes establecer el argumento na_as_group = a FALSE en el comando incidence() para eliminar las filas con valores faltantes del gráfico.
Datos filtrados
Para trazar la curva epidémica de un subconjunto de datos:
- Filtrar los datos del listado
- Proporcionar los datos filtrados al comando
incidence() - Trazar el objeto de incidencia
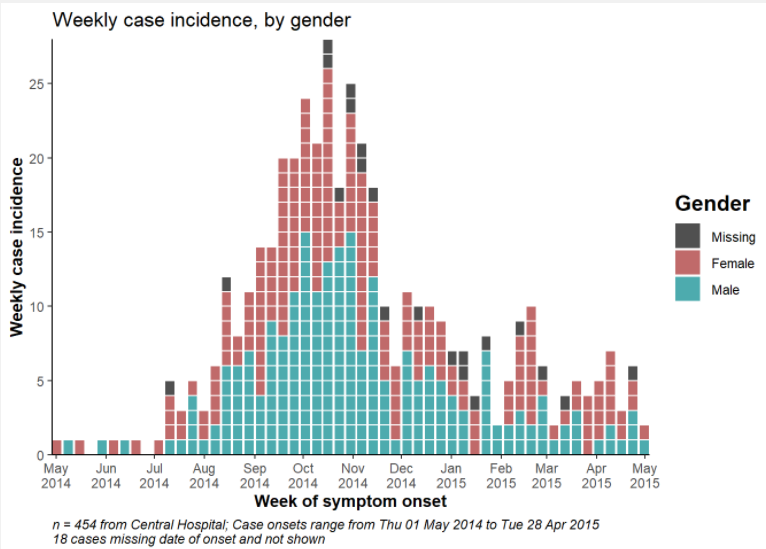
El ejemplo siguiente utiliza datos filtrados para mostrar sólo los casos del Central Hospital.
# filtrar linelist
central_data <- linelist %>%
filter(hospital == "Central Hospital")
# crear objeto de incidencia usando los datos filtrados
central_outbreak <- incidence(central_data, date_index = date_onset, interval = "week")
# representar el objeto de incidencia
plot(central_outbreak, title = "Weekly case incidence at Central Hospital")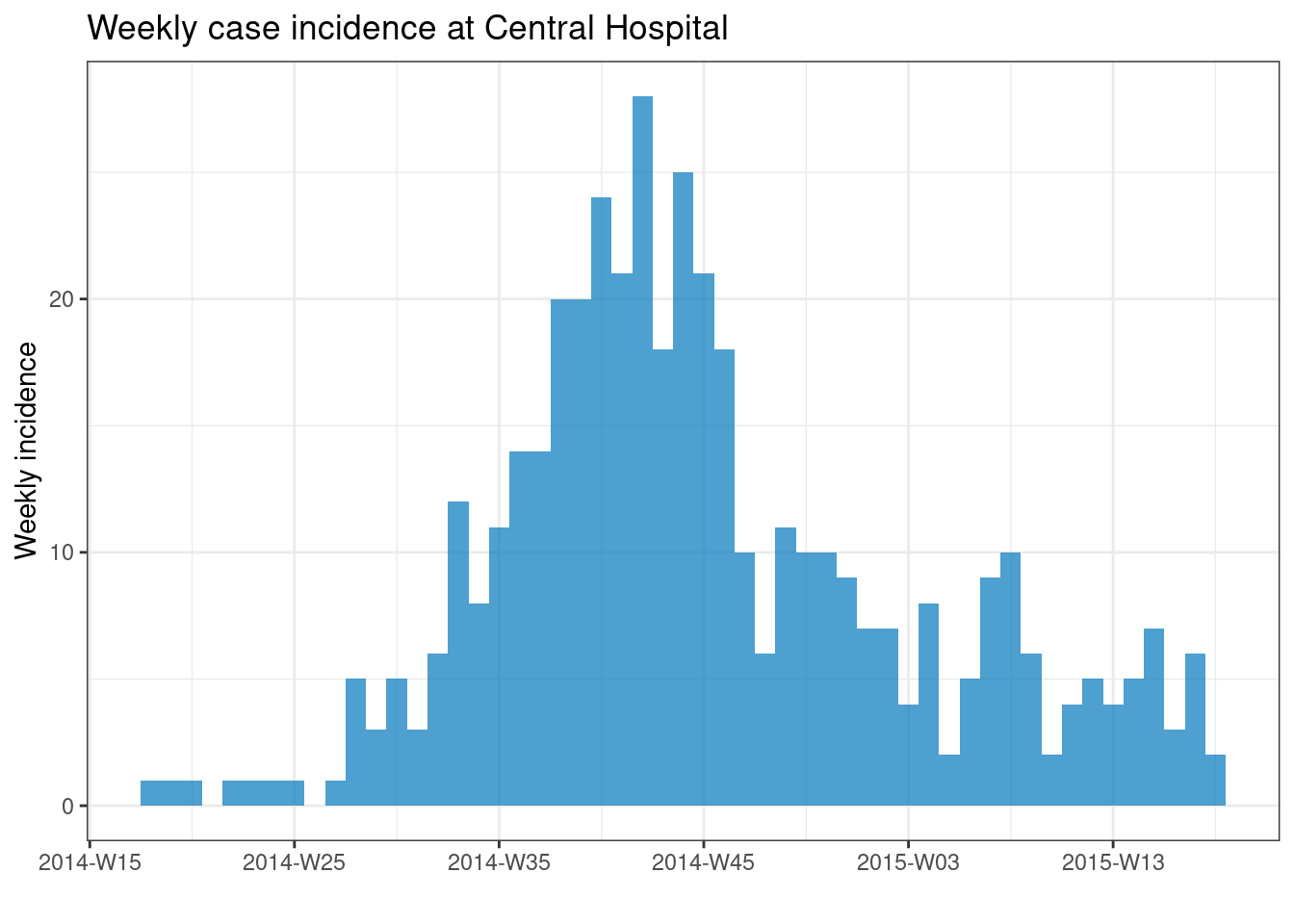
Recuentos agregados
Si los datos originales son agregados (recuentos), cuando crees el objeto de incidencia con incidence() proporciona el nombre de la columna que contiene los recuentos de casos al argumento count =.
Por ejemplo, este dataframe count_data son casos de linelist agregados en recuentos diarios por hospital. Las primeras 50 filas tienen este aspecto:
Si comienzas tu análisis con datos de recuento diario como el conjunto de datos anterior, tu comando incidence() para convertirlo en una curva epidémica semanal por hospital tendría el siguiente aspecto:
epi_counts <- incidence( # crea un objeto de incidencia semanal
count_data, # conjunto de datos con recuentos agregados por día
date_index = date_hospitalisation, # columna con fechas
count = n_cases, # columna con recuentos
interval = "week", # agrega recuentos diarios por semanas
groups = hospital # agrupa por hospital
)
# dibuja la curva epidemiológica semanal de incidencia, con barras apiladas por hospital
plot(epi_counts, # objeto incidencia
fill = hospital) # colorear las barras por hospital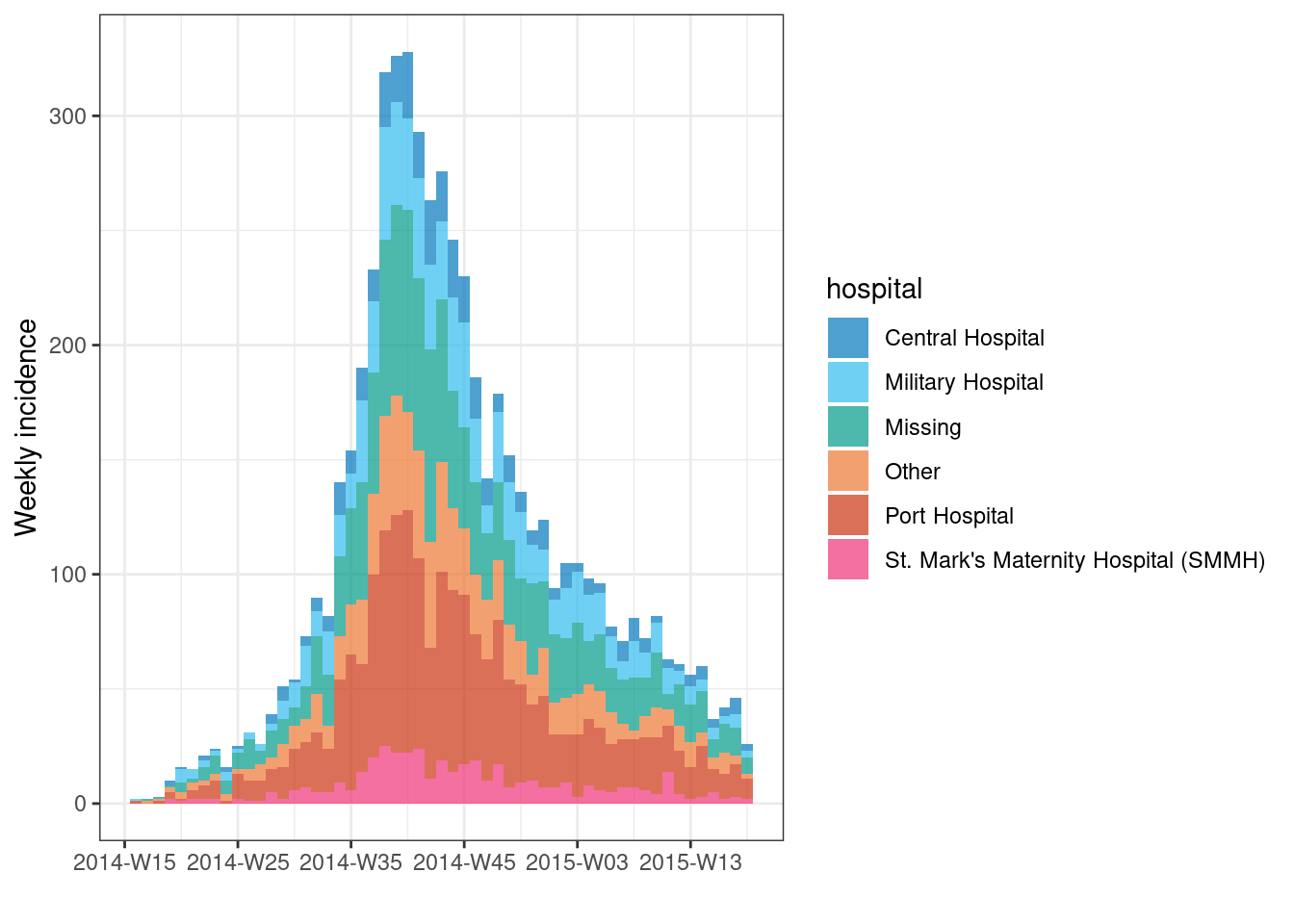
Facetas/pequeños múltiplos
Facetar los datos por grupos (es decir, producir “pequeños múltiples”):
Especificar la columna a facetar en
groups =Utilizar el comando facet_plot() en lugar de
plot()Especificar qué columnas de agrupación utilizar como
fill =y cuáles utilizar comofacets =
A continuación, establecemos las columnas hospital y outcome como columnas de agrupación en el comando incidence(). A continuación, en facet_plot() trazamos la curva epidémica, especificando que queremos una curva epidémica diferente para cada hospital y que dentro de cada curva epidémica las barras deben estar apiladas y coloreadas por outcome.
epi_wks_hosp_out <- incidence(
linelist, # conjunto de datos
date_index = date_onset, # columna con fechas
interval = "month", # barras mensuales
groups = c(outcome, hospital) # agrupa por resultado (outcome) y hospital
)
# gráfica
incidence2::facet_plot(
epi_wks_hosp_out, # objeto de incidencia
facets = hospital, # columna a facetar
fill = outcome) # columna a rellenar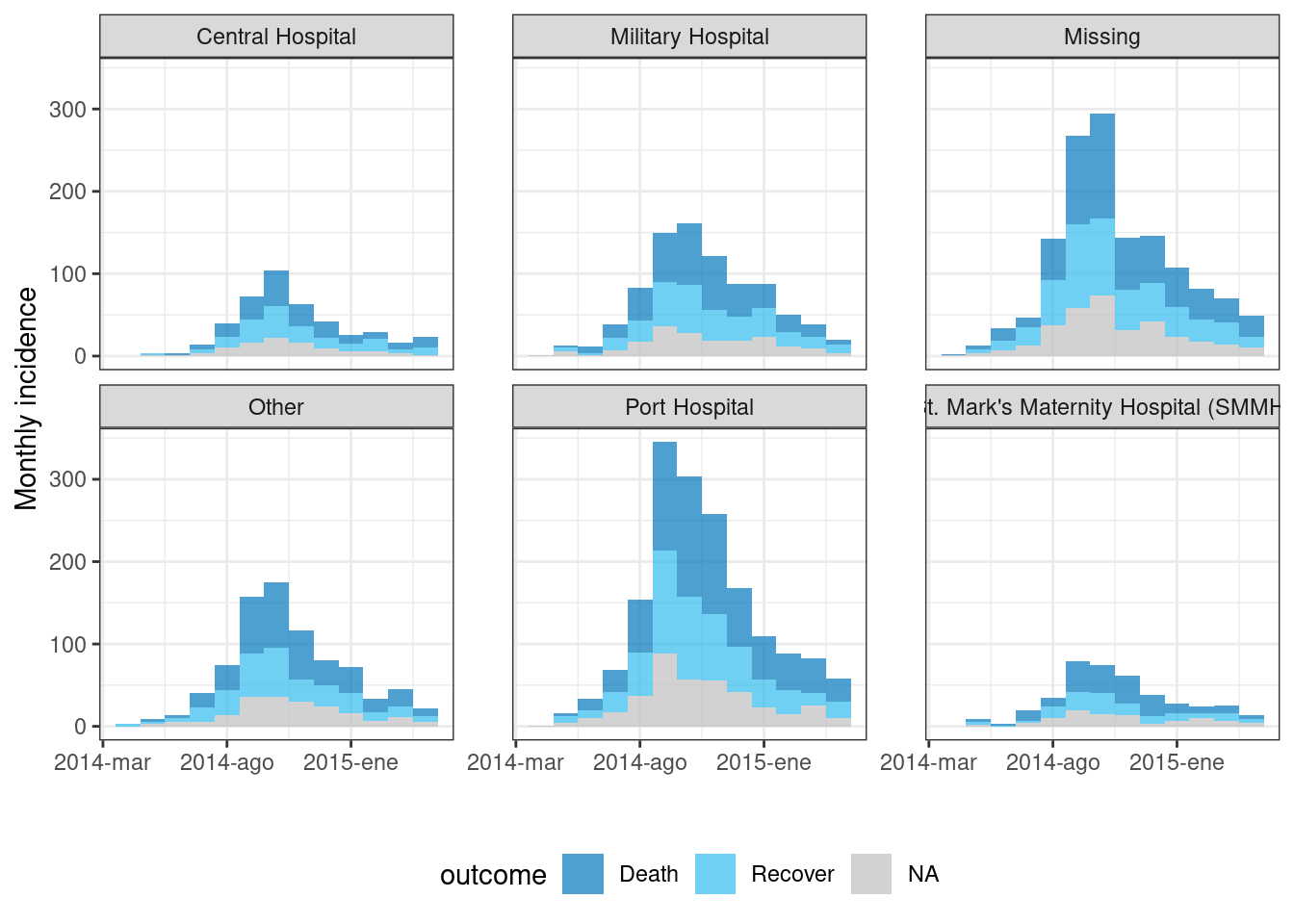
Ten en cuenta que el paquete ggtree (utilizado para mostrar árboles filogenéticos) también tiene una función facet_plot() - por eso especificamos incidence2::facet_plot() arriba.
Modificaciones con plot()
Una curva epidémica producida por incidence2 puede ser modificada a través de estos argumentos dentro de la función plot().
Aquí están los argumentos de plot() que modifican la apariencia de las barras:
| Argumento | Descripción | Ejemplos |
|---|---|---|
fill = |
Color de la barra. Nombre de color o Nombre de la columna previamente especificada en groups = en el comando incidence()
|
fill = "red", o fill = gender
|
color = |
Colorea alrededor de cada barra, o de cada agrupación dentro de una barra | border = "white" |
legend = |
Ubicación de la leyenda | Puede ser “bottom”, “top”, “left”, “right”, o “none” |
alpha = |
Transparencia de las barras/cajas | 1 es totalmente opaco, 0 es totalmente transparente |
width = |
Valor entre 0 y 1 que indica el tamaño de las barras relativo a su intervalo de tiempo | width = .7 |
show_cases = |
Lógico; si es TRUE, cada caso se muestra como una caja. Muestra mejor en brotes pequeños. | show_cases = TRUE |
Aquí están los argumentos de plot() que modifican el eje de la fecha:
| Argumento(s) | Descripción |
|---|---|
centre_dates = |
TRUE/FALSE en cuanto a si la fecha aparece bajo el centro de las barras, o al principio de las mismas |
date_format = |
Ajusta el formato de visualización de la fecha con la sintaxis strptime (“%”). Sólo funciona si centre_dates = FALSE (detalles más abajo). |
n.breaks = |
Número aproximado de interrupciones de la etiqueta del eje-x |
angle = |
Ángulo de las etiquetas de fecha del eje-x (número de grados) |
size = |
Tamaño del texto en puntos |
Ten en cuenta que el argumento date_breaks = sólo funciona si centre_dates = FALSE. Proporciona un valor de carácter entre comillas utilizando la sintaxis strptime que se indica a continuación, como se detalla en la página Trabajar con fechas. Puedes utilizar \n para una “nueva línea”.
%d = Número de día del mes (5, 17, 28, etc.)
%j = Número de día del año (día juliano 001-366)
%a = Día de la semana abreviado (Mon, Tue, Wed, etc.)
%A = Día de la semana completo (Monday, Tuesday, etc.))
%w = Número del día de la semana (0-6, el domingo es 0)
%u = Número del día de la semana (1-7, el lunes es 1)
%W = Número de la semana (00-53, el lunes es el comienzo de la semana)
%U = Número de la semana (01-53, el domingo es el comienzo de la semana)
%m = Número del mes (ej. 01, 02, 03, 04)
%b = Mes abreviado (Jan, Feb, etc. )
%B = Mes completo (January, February, etc.)
%y = Año de 2 dígitos (por ejemplo, 89)
%Y = Año de 4 dígitos (por ejemplo, 1989)
%h = horas (reloj de 24 horas)
%m = minutos
%s = segundos
%z = desviación de GMT
%Z = Zona horaria (carácter)
Estos son los argumentos de plot() que modifican las etiquetas de los gráficos:
| Argumento(s) | Descripción |
|---|---|
title = |
Título del gráfico |
xlab = |
Título del eje-x |
ylab = |
Título del eje-y |
size = |
Tamaño del texto del eje-x en pts (utiliza ggplot’s theme() para ajustar otros tamaños) |
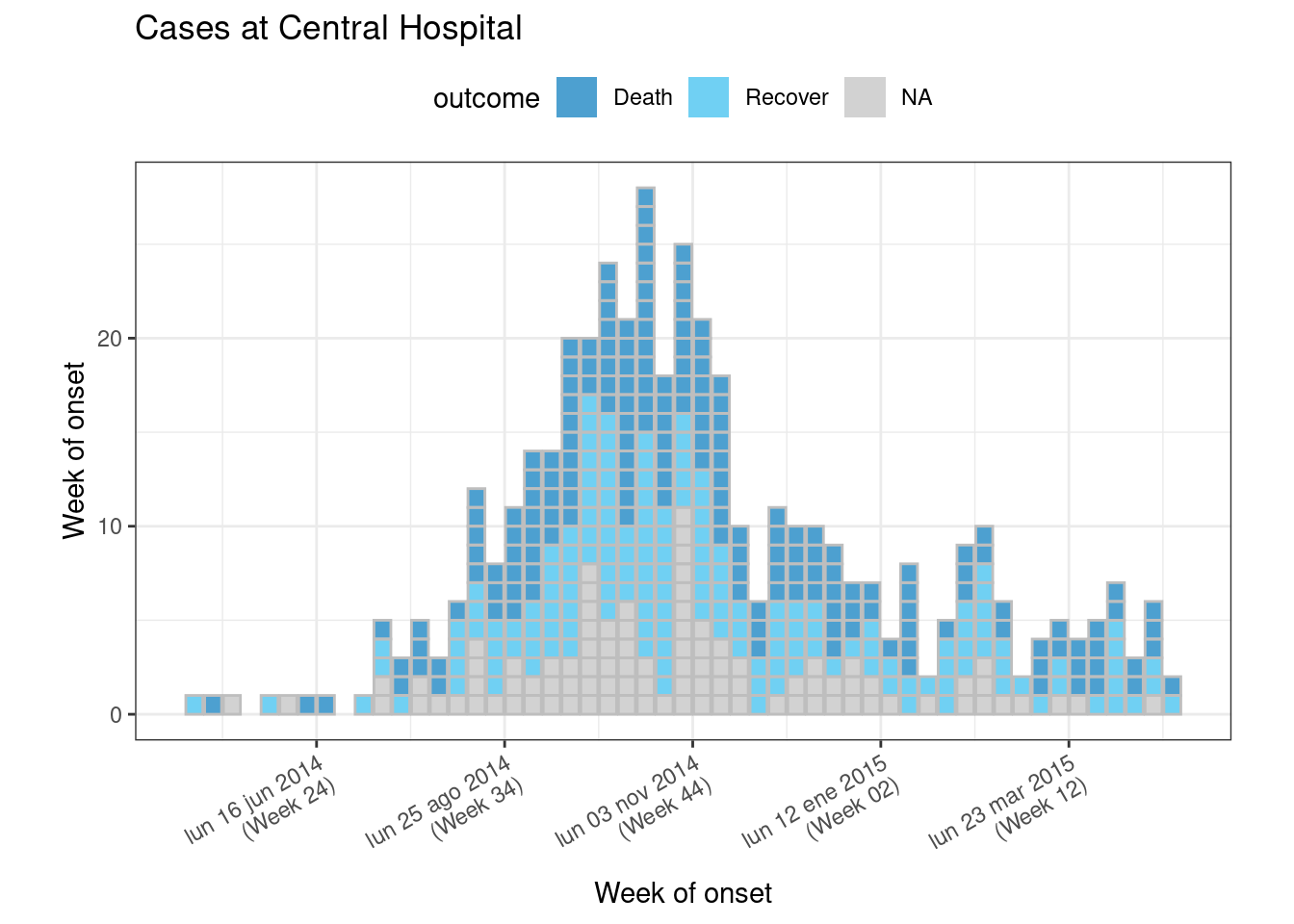
Un ejemplo que utiliza muchos de los argumentos anteriores:
# filtrar linelist
central_data <- linelist %>%
filter(hospital == "Central Hospital")
# crear objeto de incidencia usando los datos filtrados
central_outbreak <- incidence(
central_data,
date_index = date_onset,
interval = "week",
groups = outcome)
# representar el objeto de incidencia
plot(
central_outbreak,
fill = outcome, # color de la barra/caja
legend = "top", # leyenda en la parte superior
title = "Cases at Central Hospital", # título
xlab = "Week of onset", # etiqueta del eje-x
ylab = "Week of onset", # etiqueta del eje-y
show_cases = TRUE, # muestra cada caso como un cuadrado individual
alpha = 0.7, # transparencia
border = "grey", # borde de la caja
angle = 30, # ángulo de las etiquetas de fecha
centre_dates = FALSE, # etiquetas de fecha en el borde de la barra
date_format = "%a %d %b %Y\n(Week %W)" # ajusta cómo se muestran las fechas
)
Para ajustar aún más la apariencia del gráfico, consulta la sección siguiente sobre modificaciones con ggplot().
Modificaciones con ggplot2
Puedes modificar aún más un gráfico de incidence2 añadiendo modificaciones de ggplot2 con un + después del cierre de la función plot() de incidence, como se demuestra a continuación.
A continuación, el gráfico de incidence2 termina y luego se utilizan los comandos de ggplot2 para modificar los ejes, añadir una leyenda y ajustar la fuente en negrita y el tamaño del texto.
Ten en cuenta que si añades scale_x_date(), la mayor parte del formato de fecha de plot() se sobrescribirá. Consulta la sección de curvas epidémicas de ggplot() y la página del Manual Consejos de ggplot para más opciones.
# filtrar linelist
central_data <- linelist %>%
filter(hospital == "Central Hospital")
# crear objeto de incidencia usando los datos filtrados
central_outbreak <- incidence(
central_data,
date_index = date_onset,
interval = "week",
groups = c(outcome))
# representar el objeto de incidencia
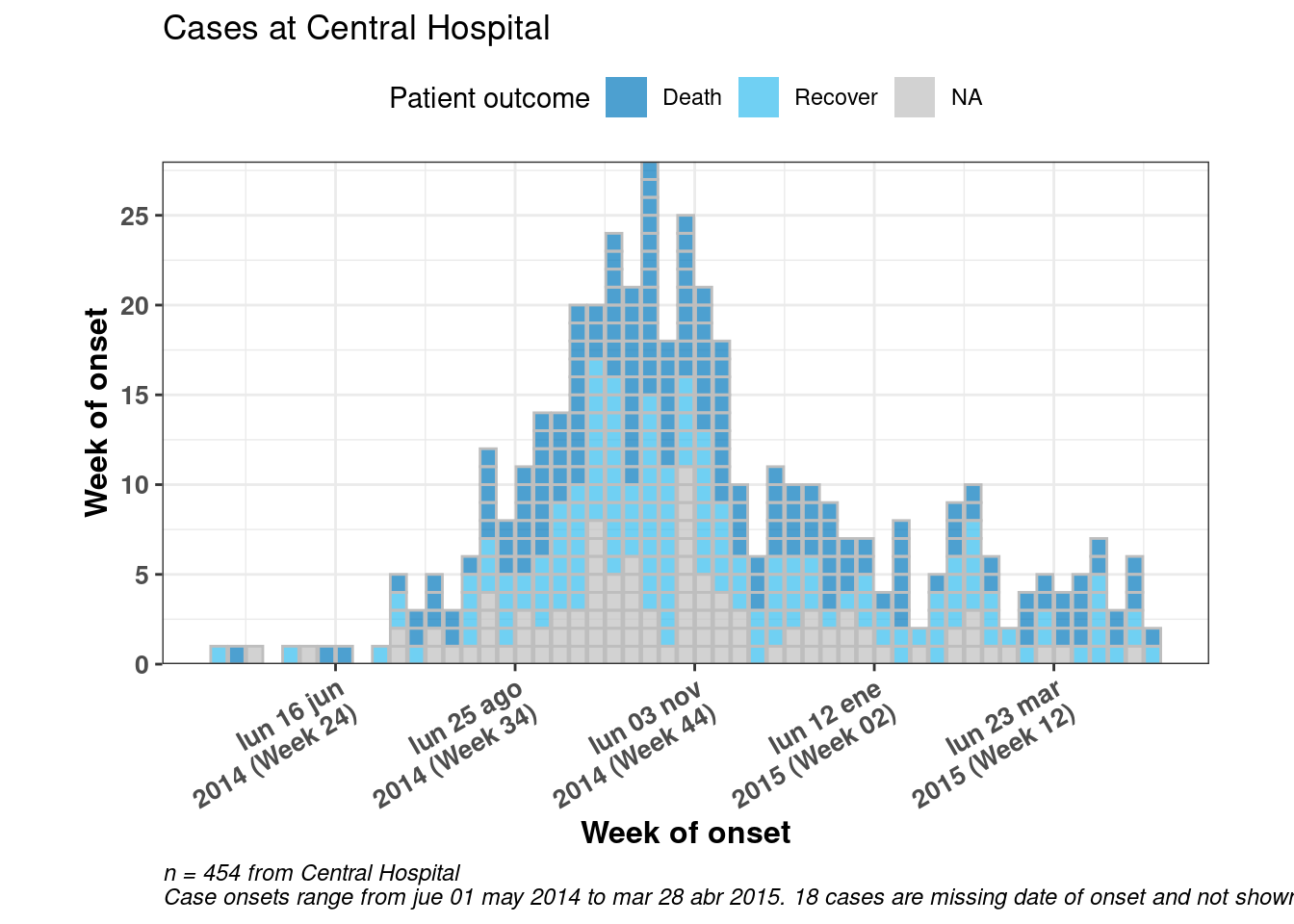
plot(
central_outbreak,
fill = outcome, # color de la barra/caja
legend = "top", # leyenda en la parte superior
title = "Cases at Central Hospital", # título
xlab = "Week of onset", # etiqueta del eje-x
ylab = "Week of onset", # etiqueta del eje-y
show_cases = TRUE, # muestra cada caso como un cuadrado individual
alpha = 0.7, # transparencia
border = "grey", # borde de la caja
centre_dates = FALSE,
date_format = "%a %d %b\n%Y (Week %W)",
angle = 30, # ángulo de las etiquetas de fecha
)+
scale_y_continuous(
breaks = seq(from = 0, to = 30, by = 5), # especifica incrementos del eje-y por 5
expand = c(0,0))+ # elimina el espacio sobrante por debajo de 0 en el eje-y
# añadeir leyenda dinámica
labs(
fill = "Patient outcome", # Título de la leyenda
caption = stringr::str_glue( # leyenda dinámica - ver página sobre caracteres y cadenas
"n = {central_cases} from Central Hospital
Case onsets range from {earliest_date} to {latest_date}. {missing_onset} cases are missing date of onset and not shown",
central_cases = nrow(central_data),
earliest_date = format(min(central_data$date_onset, na.rm=T), format = '%a %d %b %Y'),
latest_date = format(max(central_data$date_onset, na.rm=T), format = '%a %d %b %Y'),
missing_onset = nrow(central_data %>% filter(is.na(date_onset)))))+
# ajustar la negrita y la posición de los encabezados
theme(
axis.title = element_text(size = 12, face = "bold"), # títulos de los ejes más grandes y en negrita
axis.text = element_text(size = 10, face = "bold"), # tamaño del texto del eje y negrita
plot.caption = element_text(hjust = 0, face = "italic") # mover el título a la izquierda
)
Cambiar los colores
Especificar una paleta
Proporciona el nombre de una paleta predefinida al argumento col_pal = en plot(). El paquete incidence2 viene con 2 paletas predefinidas: “vibrant” y “muted”. En “vibrant” los primeros 6 colores son distintos y en “muted” los primeros 9 colores son distintos. Después de estos números, los colores son interpolaciones/intermediarios de otros colores. Estas paletas predefinidas se pueden encontrar en este sitio web. Las paletas excluyen el gris, que está reservado para los datos que faltan (utiliza na_color = para cambiar este valor por defecto).
# Crear objeto de incidencia, con datos agrupados por categoría de edad.
age_outbreak <- incidence(
linelist,
date_index = date_onset, # fecha de inicio para el eje-x
interval = "week", # agregación semanal de casos
groups = age_cat)
# dibujar la curvaepi con la paleta por defecto
plot(age_outbreak, fill = age_cat, title = "'vibrant' default incidence2 palette")
# dibujar con una paleta de colores diferente
#plot(age_outbreak, fill = age_cat, col_pal = muted, title = "'muted' incidence2 palette")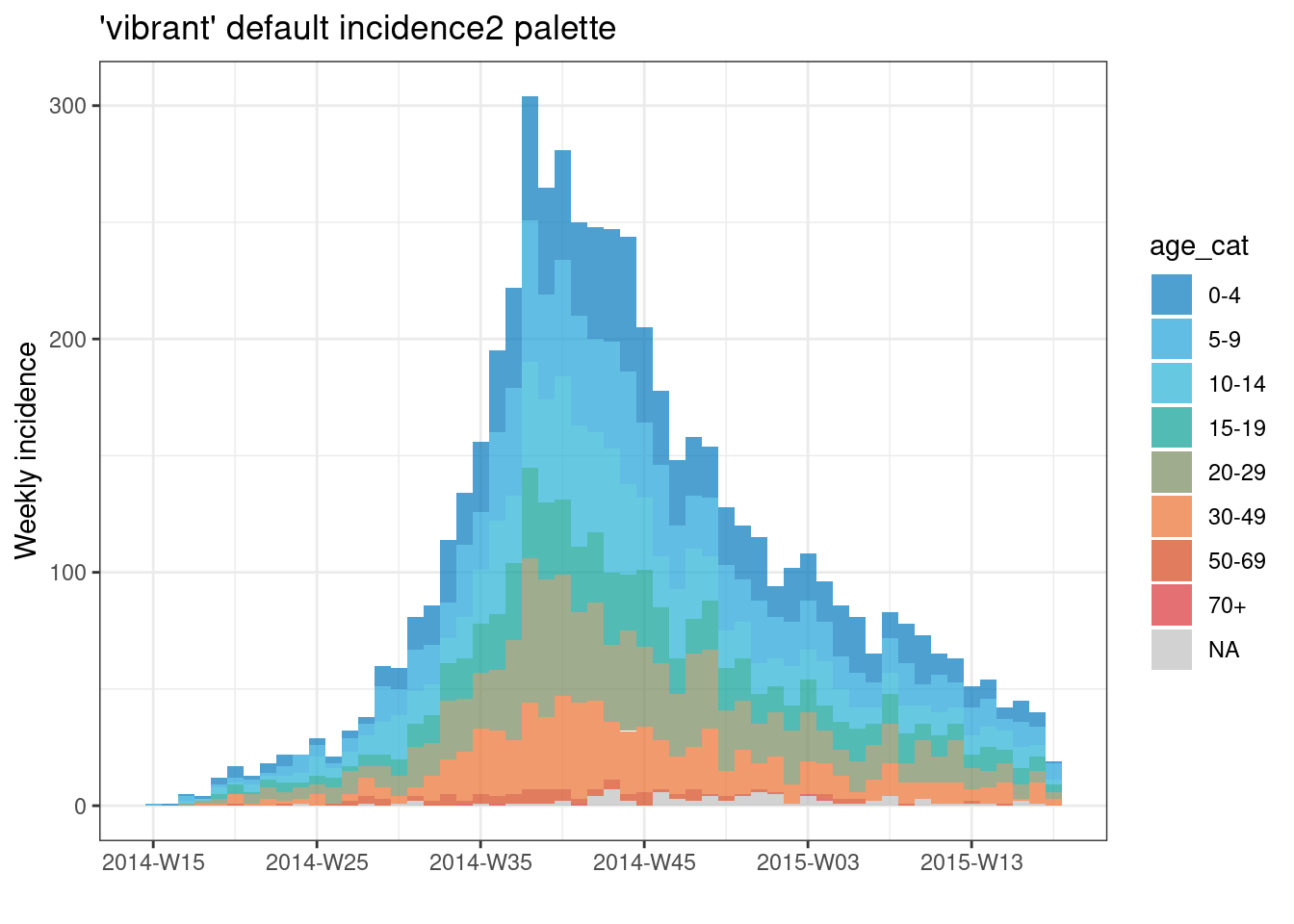
También puedes utilizar una de las paletas de R base (pon el nombre de la paleta sin comillas).
# dibujar con una paleta de R base
plot(age_outbreak, fill = age_cat, col_pal = heat.colors, title = "base R heat.colors palette")
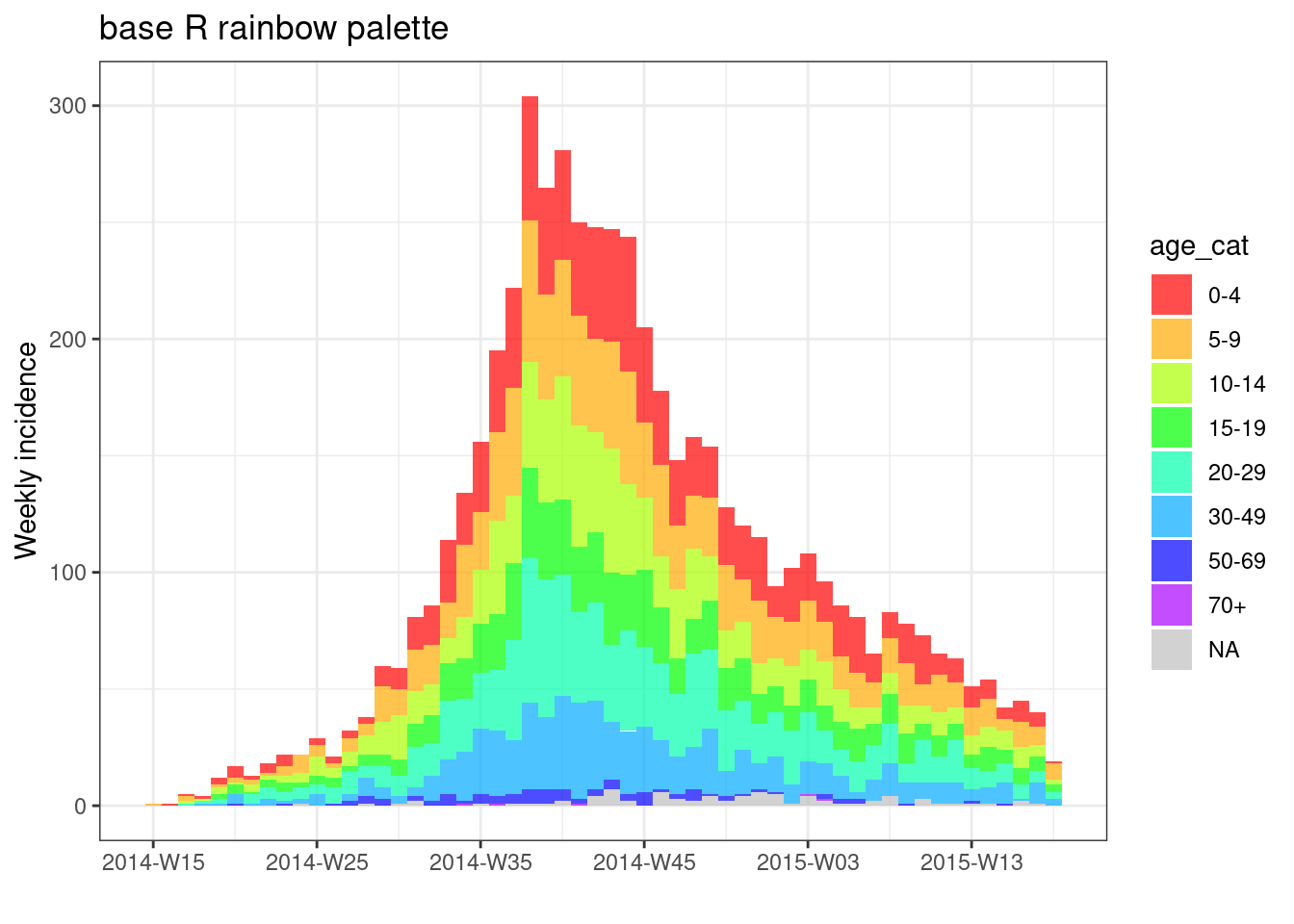
# dibujar con una paleta de R base
plot(age_outbreak, fill = age_cat, col_pal = rainbow, title = "base R rainbow palette")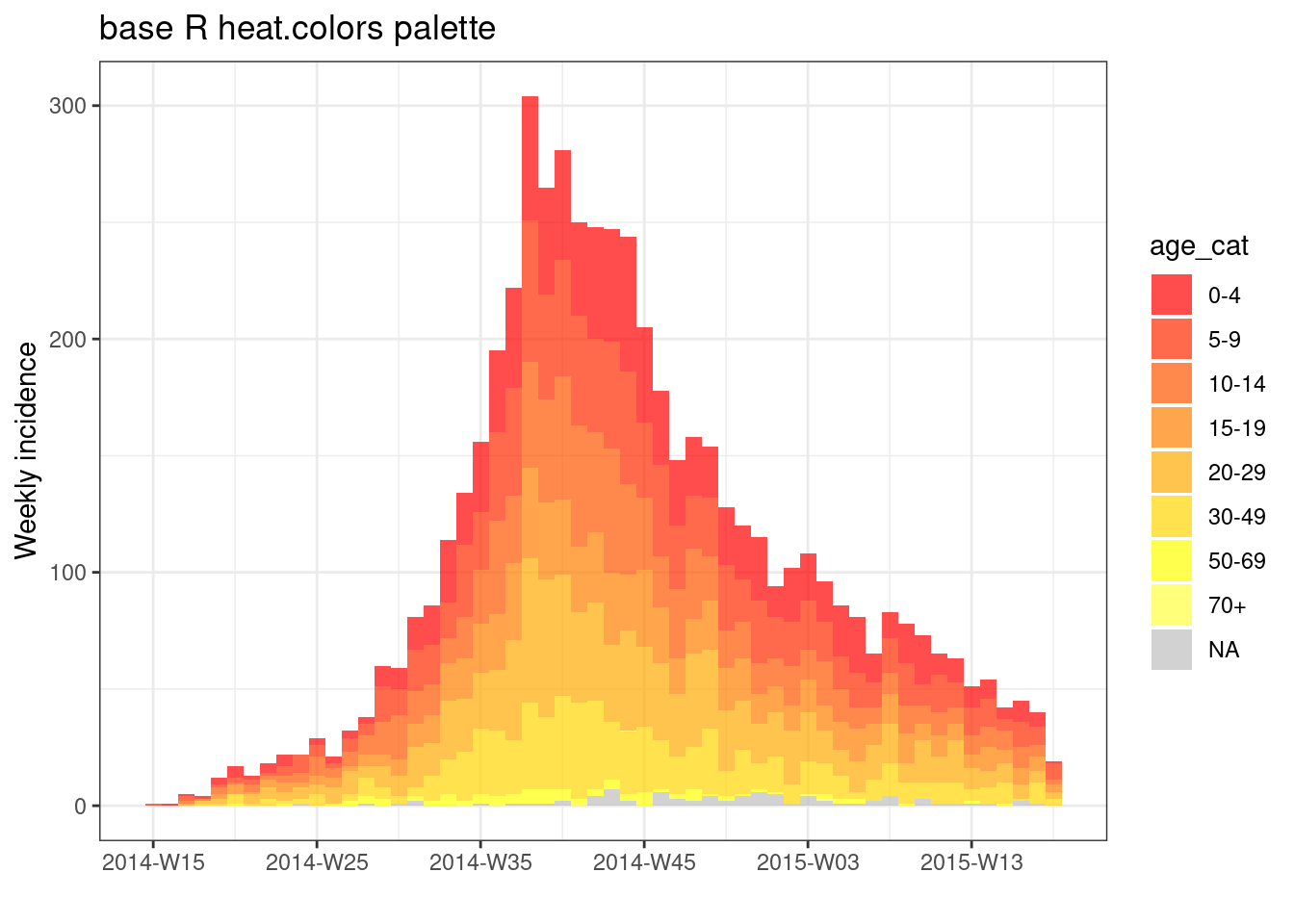
También puedes añadir una paleta de colores del paquete viridis o del paquete RColorBrewer. Primero hay que cargar esos paquetes, y luego añadir sus respectivas funciones scale_fill_*() con un +, como se muestra a continuación.
pacman::p_load(RColorBrewer, viridis)
# representar con una paleta de colores
plot(age_outbreak, fill = age_cat, title = "Viridis palette")+
scale_fill_viridis_d(
option = "inferno", # esquema de color, prueba también "plasma" o el predeterminado
name = "Age Category", # nombre de la leyenda
na.value = "grey") # para valores faltantes
# representar con una paleta de colores
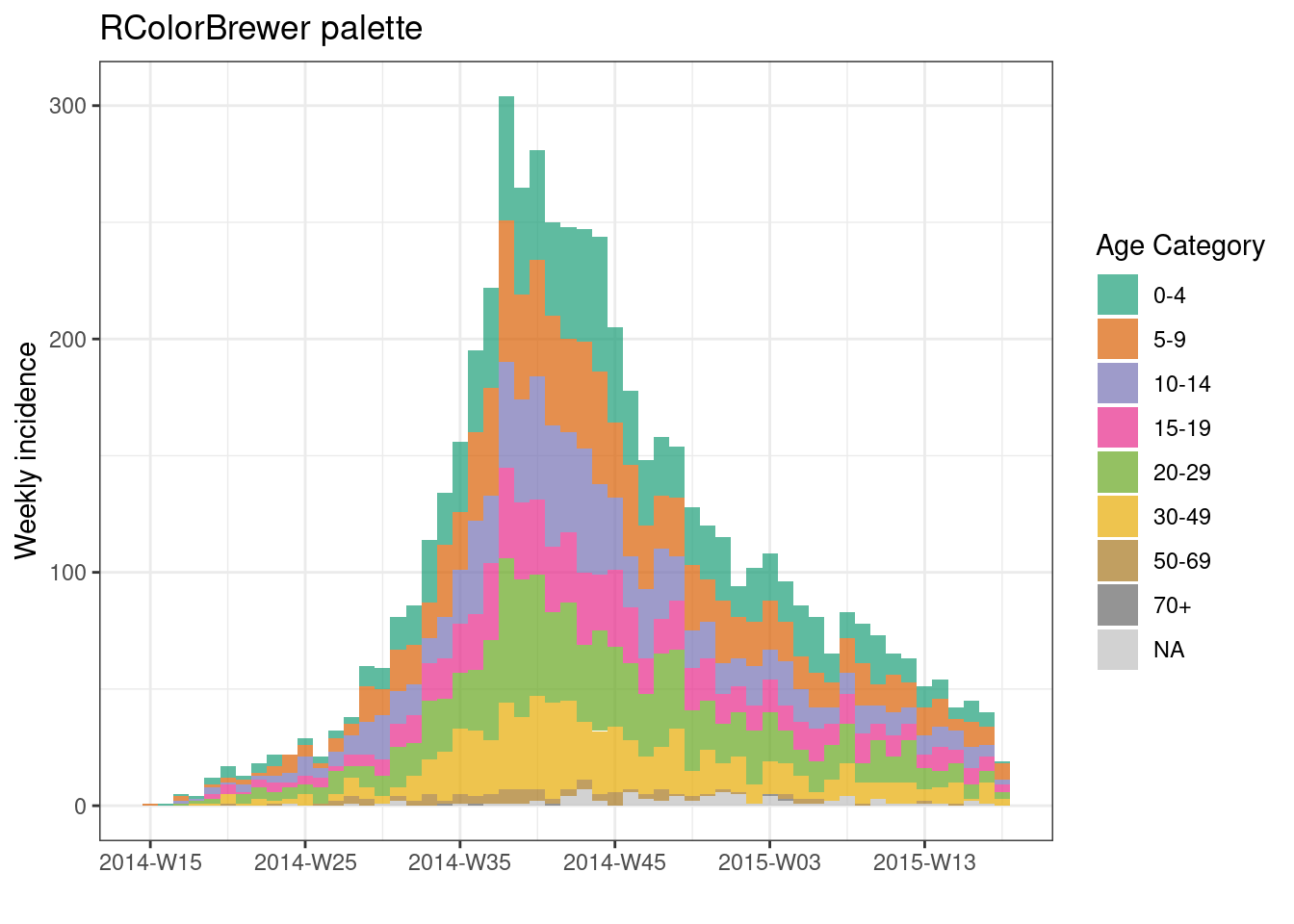
plot(age_outbreak, fill = age_cat, title = "RColorBrewer palette")+
scale_fill_brewer(
palette = "Dark2", # paleta de colores, prueba también Accent, Dark2, Paired, Pastel1, Pastel2, Set1, Set2, Set3
name = "Age Category", # nombre de la leyenda
na.value = "grey") # for missing values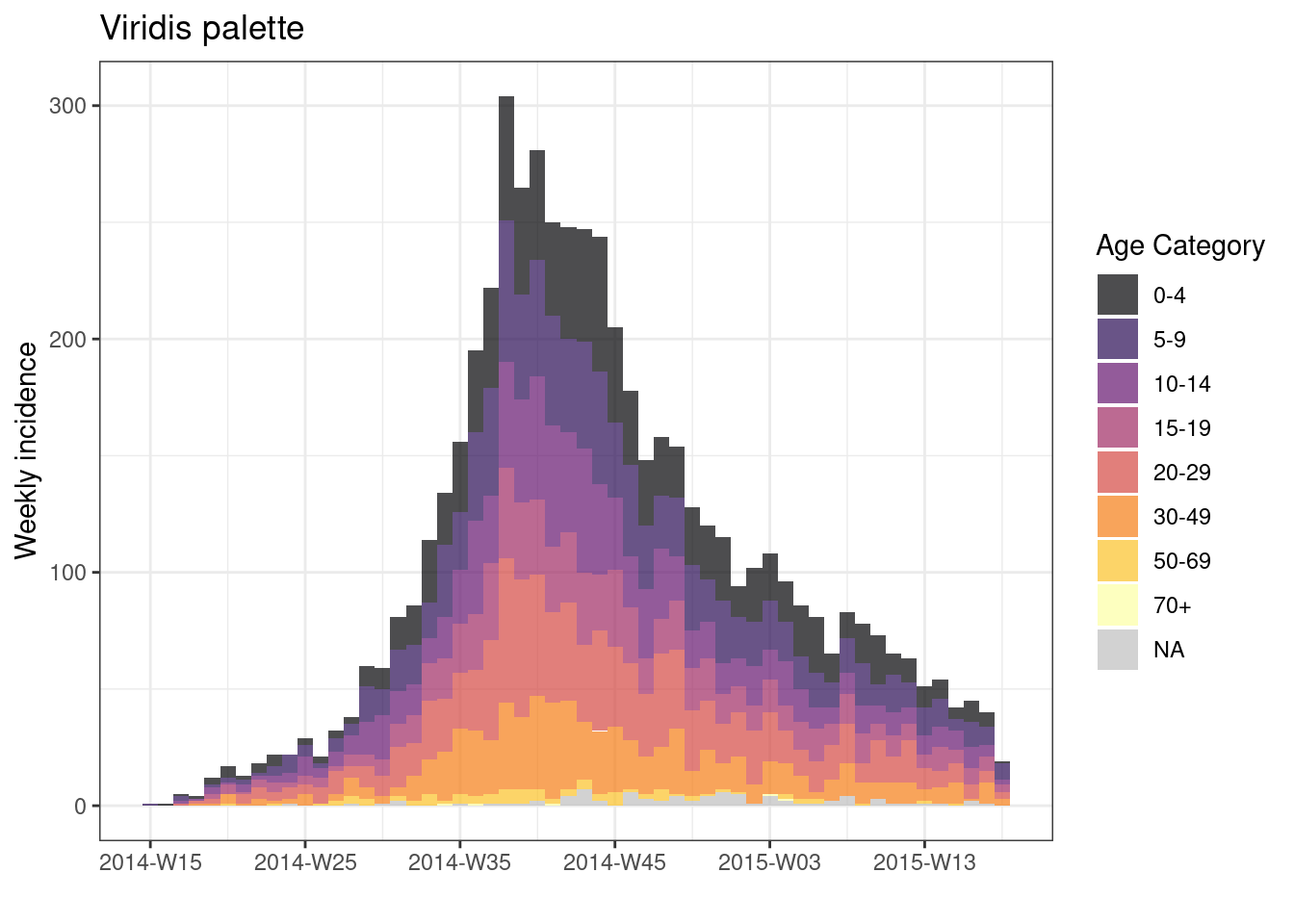
Especificar manualmente
Para especificar los colores manualmente, añade la función scale_fill_manual() de ggplot2 a la función plot() con un + y proporciona el vector de nombres de colores o códigos HEX al argumento values =. El número de colores listados debe ser igual al número de grupos. Ten en cuenta que si los valores faltantes son un grupo - pueden ser convertidos a un valor de carácter como “Missing” durante la preparación de los datos con la función fct_explicit_na() como se explica en la página sobre Factores.
# colores especificados manualmente
plot(age_outbreak, fill = age_cat, title = "Manually-specified colors")+
scale_fill_manual(
values = c("darkgreen", "darkblue", "purple", "grey", "yellow", "orange", "red", "lightblue"), # colores
name = "Age Category") # Nombre para la leyenda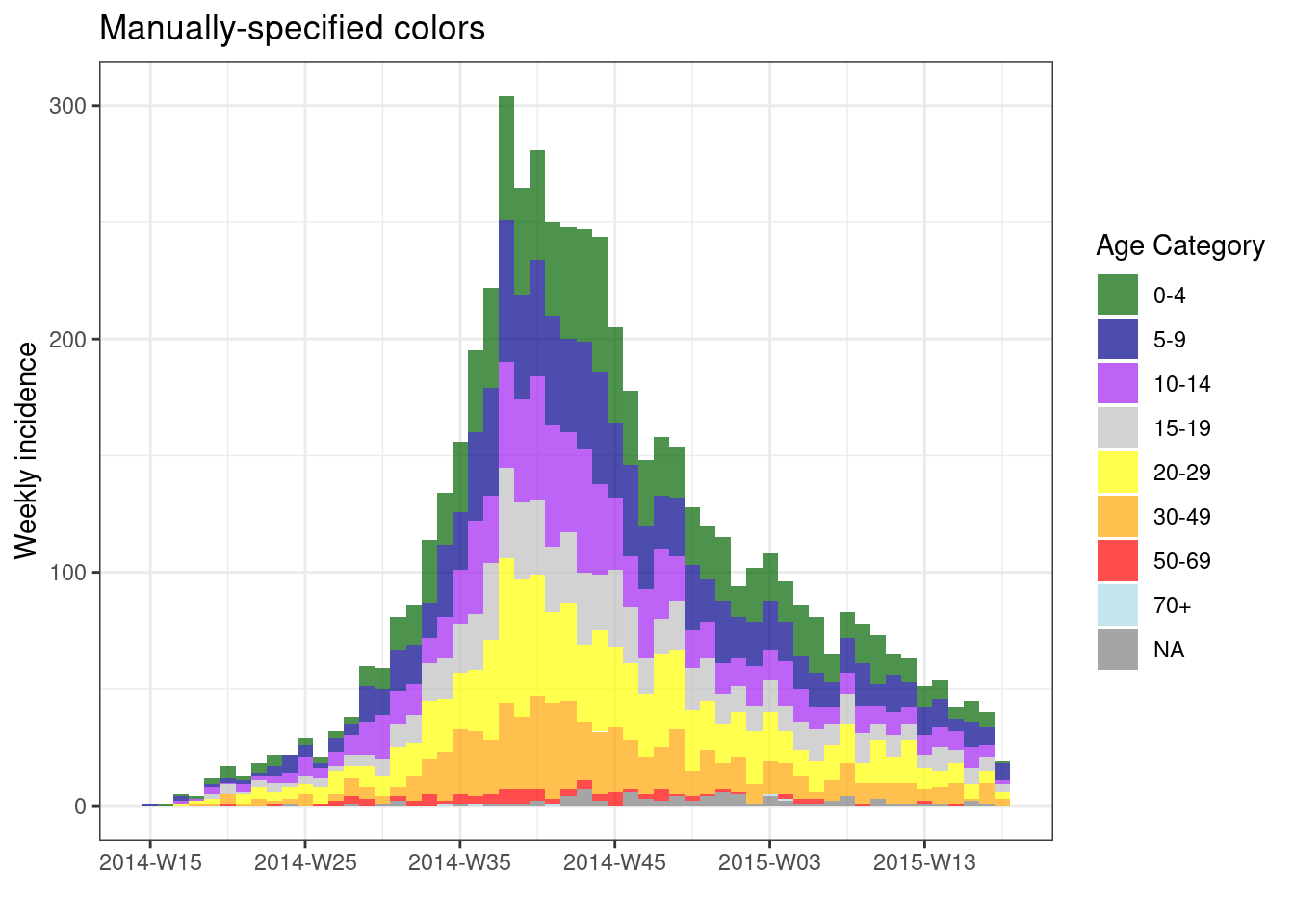
Como se menciona en la página Consejos de ggplot, puedes crear tus propias paletas utilizando colorRampPalette() sobre un vector de colores y especificando el número de colores que deseas. Esta es una buena manera de obtener muchos colores en una rampa especificando unos pocos.
my_cols <- c("darkgreen", "darkblue", "purple", "grey", "yellow", "orange")
my_palette <- colorRampPalette(my_cols)(12) # expand the 6 colors above to 12 colors
my_palette## [1] "#006400" "#00363F" "#00097E" "#3A0BAF" "#821ADD" "#A84BE2" "#B592CB" "#C9C99B" "#E7E745" "#FFF600" "#FFCD00" "#FFA500"Ajustar el orden de los niveles
Para ajustar el orden de aparición de los grupos (en el gráfico y en la leyenda), la columna de agrupación debe ser de tipo Factor. Consulta la página sobre Factores para obtener más información.
En primer lugar, veamos una curva epidémica semanal por hospital con la ordenación por defecto:
# ORIGINAL - hospital NO como factor
####################################
# crea objeto de incidencia semanal, filas agrupadas por hospital y semana
hospital_outbreak <- incidence(
linelist,
date_index = date_onset,
interval = "week",
groups = hospital)
# representar el objeto de incidencia
plot(hospital_outbreak, fill = hospital, title = "ORIGINAL - hospital not a factor")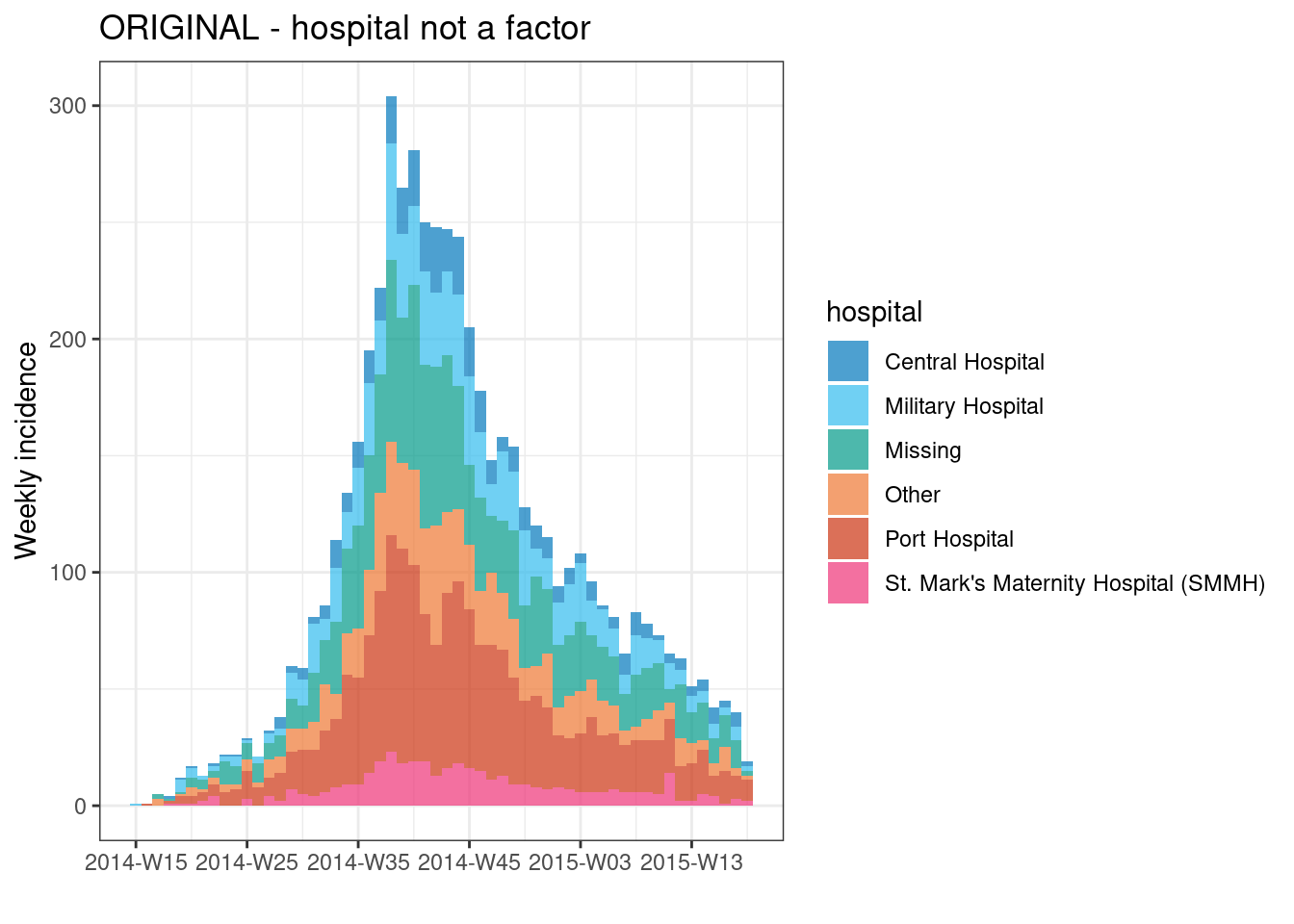
Ahora, para ajustar el orden de manera que los “Missing” y “Otros” estén en la parte superior de la curva epidémica podemos hacer lo siguiente:
- Cargar el paquete forcats, para trabajar con factores
- Ajustar los datos - en este caso vamos a definir un nuevo dataset (
plot_data) en el que:- la columna
genderse define como un factor el orden de los niveles se establecen confct_relevel()de manera que “Other” y “Missing” son los primeros, por lo que aparecen en la parte superior de las barras
- la columna
- El objeto de incidencia se crea y se traza como antes
- Añadimos modificaciones de ggplot2
-
scale_fill_manual()para asignar manualmente los colores para que “Missing” sea gris y “Other” sea beige
-
# MODIFICADO - hospital como factor
###################################
# carga paquete forcats para trabajar con factores
pacman::p_load(forcats)
# Convierte la columna hospital en factor y ajusta los niveles
plot_data <- linelist %>%
mutate(hospital = fct_relevel(hospital, c("Missing", "Other"))) # Establece " Missing " y " Other " como niveles superiores
# crea objeto de incidencia semanal, agrupado por hospital y semana
hospital_outbreak_mod <- incidence(
plot_data,
date_index = date_onset,
interval = "week",
groups = hospital)
# representar el objeto de incidencia
plot(hospital_outbreak_mod, fill = hospital)+
# colores especificados manualmente
scale_fill_manual(values = c("grey", "beige", "darkgreen", "green2", "orange", "red", "pink"))+
# etiquetas añadidas mediante ggplot
labs(
title = "MODIFIED - hospital as factor", # título del gráfico
subtitle = "Other & Missing at top of epicurve",
y = "Weekly case incidence", # título del eje-y
x = "Week of symptom onset", # título del eje-x
fill = "Hospital") # título de la leyenda 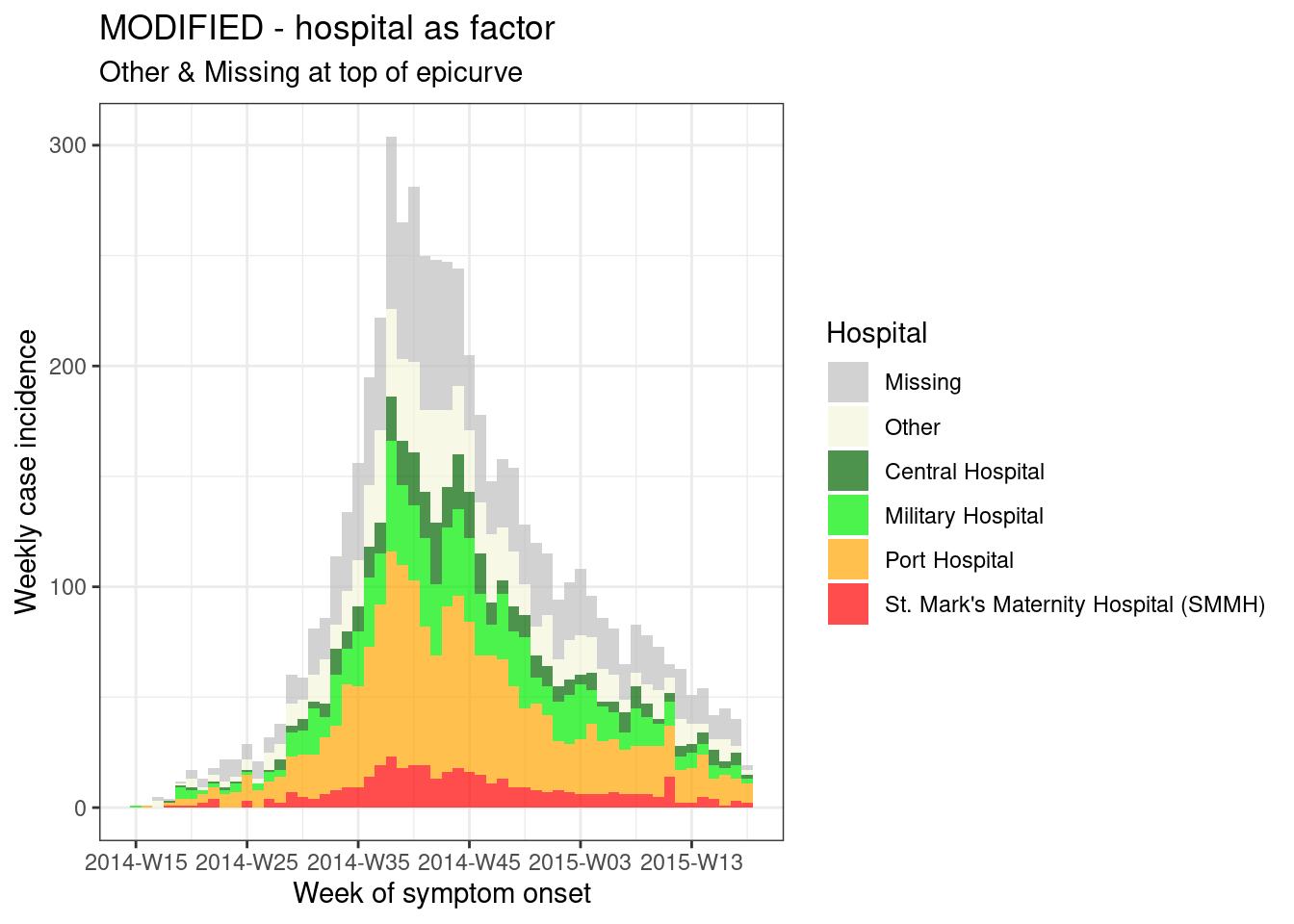
CONSEJO: Si deseas invertir el orden de la leyenda solamente, añade este comando guides(fill = guide_legend(reverse = TRUE))de ggplot2.
Líneas de cuadrícula verticales
Si utilizas la configuración predeterminada de incidence2, puedes observar que las líneas de cuadrícula verticales aparecen en cada etiqueta de fecha y una vez entre cada etiqueta de semana. Esto puede dar lugar a que las líneas de cuadrícula se crucen con la parte superior de algunas barras.
Puedes eliminar todas las líneas de la cuadrícula añadiendo el comando theme_classic() de ggplot2.
# Crea objeto de incidencia
a <- incidence(
central_data,
date_index = date_onset,
interval = "Monday weeks"
)
# Cuadrículas por defecto
plot(a, title = "Default lines")
# Intervalos de cuadrícula especificados
# NO FUNCIONA CON INCIDENCE2 1.0.0
# plot(a, title = "Weekly lines")+
# scale_x_date(
# date_breaks = "4 weeks", # las líneas verticales mayores se alinean en semanas
# date_minor_breaks = "weeks", # líneas verticales menores semanales
# date_labels = "%a\n%d\n%b") # formato de las etiquetas de fecha
# Sin cuadrículas
plot(a, title = "No lines")+
theme_classic() # elimina todas las cuadrículas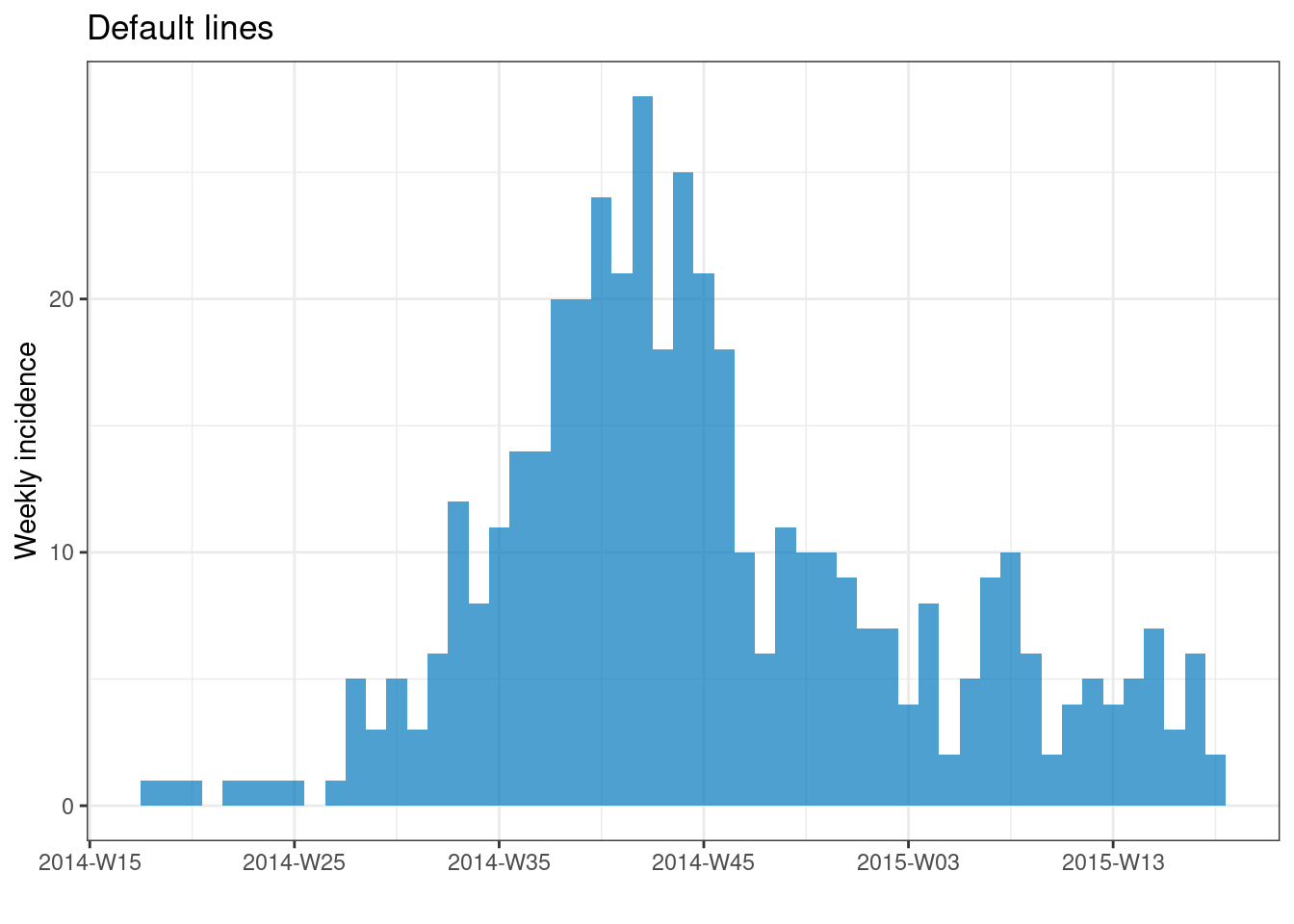
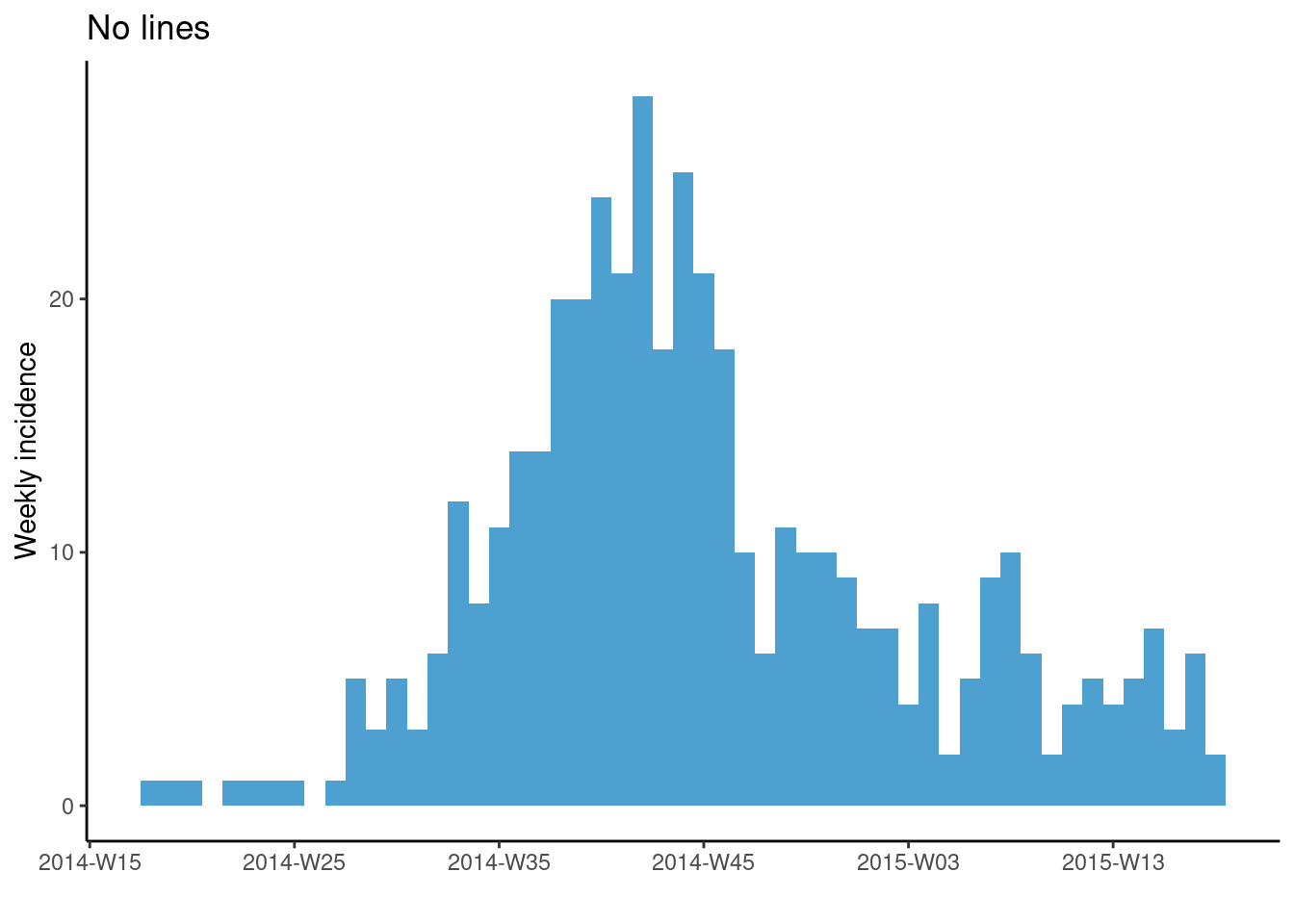
Ten en cuenta, sin embargo, que si utiliza semanas, los argumentos date_breaks y date_minor_breaks sólo funcionan para las semanas del lunes. Si tus semanas emepiezan por otro día de la semana tendrás que proporcionar manualmente un vector de fechas a los argumentos breaks = y minor_breaks =. Consulta la sección de ggplot2 para ver ejemplos de esto utilizando seq.Date().
Incidencia acumulada
En versiones anteriores de incidence2 se podía usar la función cumulate(). Esto se ha eliminado en la versión más reciente del paquete.
Ver la sección más abajo en esta página para el método alternativo para trazar la incidencia acumulativa con ggplot2 - por ejemplo para superponer una línea de incidencia acumulativa sobre una curva epidémica.
Media móvil
Puedes añadir una media móvil a un gráfico de incidence2 fácilmente con add_rolling_average() del paquete i2extras. Pasa tu objeto incidence2 a esta función, y luego a plot(). Establece en before = el número de días anteriores que deseas incluir en la media móvil (por defecto es 2). Si tus datos están agrupados, la media móvil se calculará por grupo.
rolling_avg <- incidence( # Crea objeto de incidencia
linelist,
date_index = date_onset,
interval = "week",
groups = gender) %>%
i2extras::add_rolling_average(before = 6) # añade medias móviles (en este caso, por sexo)
# dibuja la gráfica
plot(rolling_avg) # faceted automatically because rolling average on groups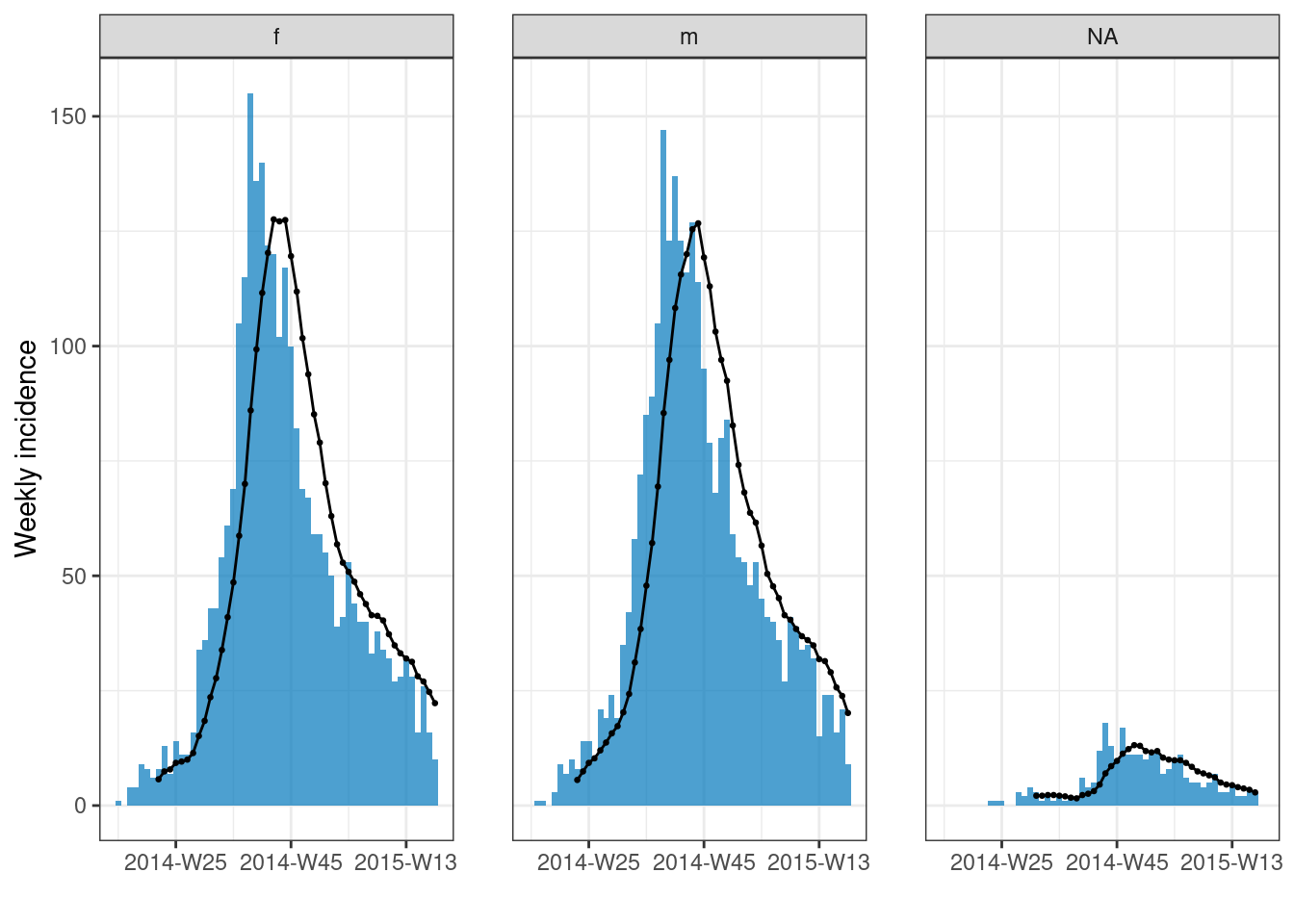
Para aprender a aplicar las medias móviles de forma más general sobre los datos, consulta la página del Manual sobre medias móviles.
32.3 Curvas epidémicas con ggplot2
El uso de ggplot() para construir tu curva epidémica permite más flexibilidad y personalización, pero requiere más esfuerzo y comprensión de cómo funciona ggplot().
A diferencia de lo que ocurre con el paquete incidence2, hay que controlar manualmente la agregación de los casos por tiempo (en semanas, meses, etc.) y los intervalos de las etiquetas en el eje de fechas. Esto debe gestionarse cuidadosamente.
Estos ejemplos utilizan un subconjunto de los datos de linelist: sólo los casos del Hospital Central.
Para producir una curva epidémica con ggplot() hay tres elementos principales:
- Un histograma, con los casos de la lista de líneas agregados en “bins” distinguidos por puntos específicos de “ruptura”.
- Escalas para los ejes y sus etiquetas
- Temas para la apariencia del gráfico, incluyendo títulos, etiquetas, subtítulos, etc.
Especificaciones de las barras
Aquí mostramos cómo especificar cómo se agregarán los casos en los intervalos del histograma (“barras”). Es importante reconocer que la agregación de los casos en los intervalos del histograma no son necesariamente los mismos intervalos que las fechas que aparecerán en el eje-x.
A continuación se muestra el código más sencillo para producir curvas epidémicas diarias y semanales.
En el comando general ggplot() se proporciona el conjunto de datos en data =. Sobre esta base, se añade la geometría de un histograma con un +. Dentro de geom_histogram(), mapeamos la estética de tal manera que la columna date_onset se mapea al eje-x. También dentro de geom_histogram() pero no dentro de aes() establecemos la anchura de las barras del histograma con binwidth =, en días. Si esta sintaxis de ggplot2 es confusa, revisa la página sobre Conceptos básicos de ggplot.
PRECAUCIÓN: El trazado de casos semanales mediante el uso de binwidth = 7 inicia la primera barra de 7 días en el primer caso, ¡que podría ser cualquier día de la semana! Para crear semanas específicas, véase la sección siguiente.
# diariamente
ggplot(data = central_data) + # establecer datos
geom_histogram( # añadir histograma
mapping = aes(x = date_onset), # asignar la columna de fecha al eje-x
binwidth = 1)+ # casos agrupados por 1 día
labs(title = "Central Hospital - Daily") # título
# semanalmente
ggplot(data = central_data) + # establecer datos
geom_histogram( # añadir histograma
mapping = aes(x = date_onset), # asignar la columna de fecha al eje-x
binwidth = 7)+ # casos agrupados cada 7 días, empezando por el primer caso (!)
labs(title = "Central Hospital - 7-day bins, starting at first case") # título
Observamos que el primer caso de este conjunto de datos del Hospital Central tuvo un inicio de síntomas el:
## [1] "jueves 01 may, 2014"Para especificar manualmente las pausas del histograma, no utilices el argumento binwidth =, y en su lugar suministra un vector de fechas a breaks =.
Crea el vector de fechas con la función seq.Date() de R base. Esta función espera argumentos to =, from =, y by =. Por ejemplo, el comando siguiente devuelve fechas mensuales que comienzan en el 15 de enero y terminan en el 28 de junio.
monthly_breaks <- seq.Date(from = as.Date("2014-02-01"),
to = as.Date("2015-07-15"),
by = "months")
monthly_breaks # imprime## [1] "2014-02-01" "2014-03-01" "2014-04-01" "2014-05-01" "2014-06-01" "2014-07-01" "2014-08-01" "2014-09-01" "2014-10-01" "2014-11-01" "2014-12-01" "2015-01-01" "2015-02-01" "2015-03-01" "2015-04-01" "2015-05-01"
## [17] "2015-06-01" "2015-07-01"Este vector puede proporcionarse a geom_histogram() como breaks =:
# mensualmente
ggplot(data = central_data) +
geom_histogram(
mapping = aes(x = date_onset),
breaks = monthly_breaks)+ # proporciona el vector predefinido de rupturas
labs(title = "Monthly case bins") # título
Una secuencia simple de fechas semanales puede ser devuelta estableciendo by = "week". Por ejemplo:
Una alternativa a la provisión de fechas específicas de inicio y fin es escribir un código dinámico para que los intervalos semanales comiencen el lunes anterior al primer caso. Utilizaremos estos vectores de fechas a lo largo de los ejemplos siguientes.
# Secuencia de fechas semanales del lunes para el HOSPITAL CENTRAL
weekly_breaks_central <- seq.Date(
from = floor_date(min(central_data$date_onset, na.rm=T), "week", week_start = 1), # lunes anterior al primer caso
to = ceiling_date(max(central_data$date_onset, na.rm=T), "week", week_start = 1), # lunes posterior al último caso
by = "week")Descompongamos el código anterior, que es bastante desalentador:
- El valor “from” (fecha más temprana) se crea de la siguiente manera: el valor mínimo de fecha (
min()conna.rm=TRUE) en la columnadate_onsetse introduce enfloor_date()del paquete lubridate.floor_date()ajustado a “week” devuelve la fecha de inicio de la “semana” de esos casos, dado que el día de inicio de cada semana es un lunes (week_start = 1). - Asimismo, el valor “to” (fecha final) se crea utilizando la función inversa
ceiling_date()para devolver el lunes posterior al último caso. - El argumento “by” de
seq.Date()puede establecerse en cualquier número de días, semanas o meses. - Utiliza
week_start = 7para las semanas de domingo
Como vamos a utilizar estos vectores de fechas a lo largo de esta página, también definimos uno para todo el brote (el anterior es sólo para el Hospital Central).
# Secuencia para todo el brote
weekly_breaks_all <- seq.Date(
from = floor_date(min(linelist$date_onset, na.rm=T), "week", week_start = 1), # lunes anterior al primer caso
to = ceiling_date(max(linelist$date_onset, na.rm=T), "week", week_start = 1), # lunes posterior al último caso
by = "week")Estas salidas de seq.Date() pueden utilizarse para crear los saltos de las casillas del histograma, pero también los saltos de las etiquetas de fecha, que pueden ser independientes de las casillas. Lea más sobre las etiquetas de fecha en secciones posteriores.
CONSEJO: Para un comando ggplot() más sencillo, guarda los saltos de cubo y los saltos de etiqueta de fecha como vectores con nombre por adelantado, y simplemente proporciona sus nombres a breaks =..
Ejemplo de curva epidémica semanal
A continuación se muestra un código de ejemplo detallado para producir curvas epidémicas semanales para las semanas del lunes, con barras alineadas, etiquetas de fecha y líneas de cuadrícula verticales. Esta sección es para el usuario que necesita el código rápidamente. Para entender cada aspecto (temas, etiquetas de fecha, etc.) en profundidad, continúa con las secciones siguientes. Es importante tener en cuenta:
- Las pausas del histograma se definen con
seq.Date(), como se ha explicado anteriormente, para comenzar el lunes anterior al caso más antiguo y terminar el lunes posterior al último caso - El intervalo de las etiquetas de fecha se especifica mediante
date_breaks =dentro descale_x_date() - El intervalo de líneas verticales menores entre etiquetas de fecha se especifica en
date_minor_breaks = -
expand = c(0,0)en los ejes x e y elimina el exceso de espacio a cada lado de los ejes, lo que también asegura que las etiquetas de fecha comiencen desde la primera barra.
# ALINEACIÓN TOTAL DE LA SEMANA DEL LUNES
#########################################
# Definir secuencia de rupturas semanales
weekly_breaks_central <- seq.Date(
from = floor_date(min(central_data$date_onset, na.rm=T), "week", week_start = 1), # lunes anterior al primer caso
to = ceiling_date(max(central_data$date_onset, na.rm=T), "week", week_start = 1), # lunes posterior al último caso
by = "week") # bins are 7-days
ggplot(data = central_data) +
# crear histograma: especificar puntos de ruptura: comienza el lunes anterior al primer caso, finaliza el lunes posterior al último caso
geom_histogram(
# estética del mapeo
mapping = aes(x = date_onset), # columna de fecha asignada al eje-x
# histogram bin breaks
breaks = weekly_breaks_central, # rupturas del histograma definidas previamente
# Barras
color = "darkblue", # color de las líneas alrededor de las barras
fill = "lightblue" # color del relleno de las barras
)+
# Etiquetas del eje-x
scale_x_date(
expand = c(0,0), # elimina el exceso de espacio del eje-x antes y después de las barras de casos
date_breaks = "4 weeks", # las etiquetas de fecha y las cuadrículas verticales mayores aparecen cada 3 semanas de lunes
date_minor_breaks = "week", # las líneas verticales menores aparecen cada semana de lunes
date_labels = "%a\n%d %b\n%Y")+ # formato de las etiquetas de fecha
# eje-y
scale_y_continuous(
expand = c(0,0))+ # elimina el exceso de espacio del eje-y por debajo de 0 (alinea el histograma con el eje-x)
# temas estéticos
theme_minimal()+ # simplifica el fondo del gráfico
theme(
plot.caption = element_text(hjust = 0, # texto a la izquierda
face = "italic"), # y en cursiva
axis.title = element_text(face = "bold"))+ # títulos de los ejes en negrita
# etiquetas con texto dinámico
labs(
title = "Weekly incidence of cases (Monday weeks)",
subtitle = "Note alignment of bars, vertical gridlines, and axis labels on Monday weeks",
x = "Week of symptom onset",
y = "Weekly incident cases reported",
caption = stringr::str_glue("n = {nrow(central_data)} from Central Hospital; Case onsets range from {format(min(central_data$date_onset, na.rm=T), format = '%a %d %b %Y')} to {format(max(central_data$date_onset, na.rm=T), format = '%a %d %b %Y')}\n{nrow(central_data %>% filter(is.na(date_onset)))} cases missing date of onset and not shown"))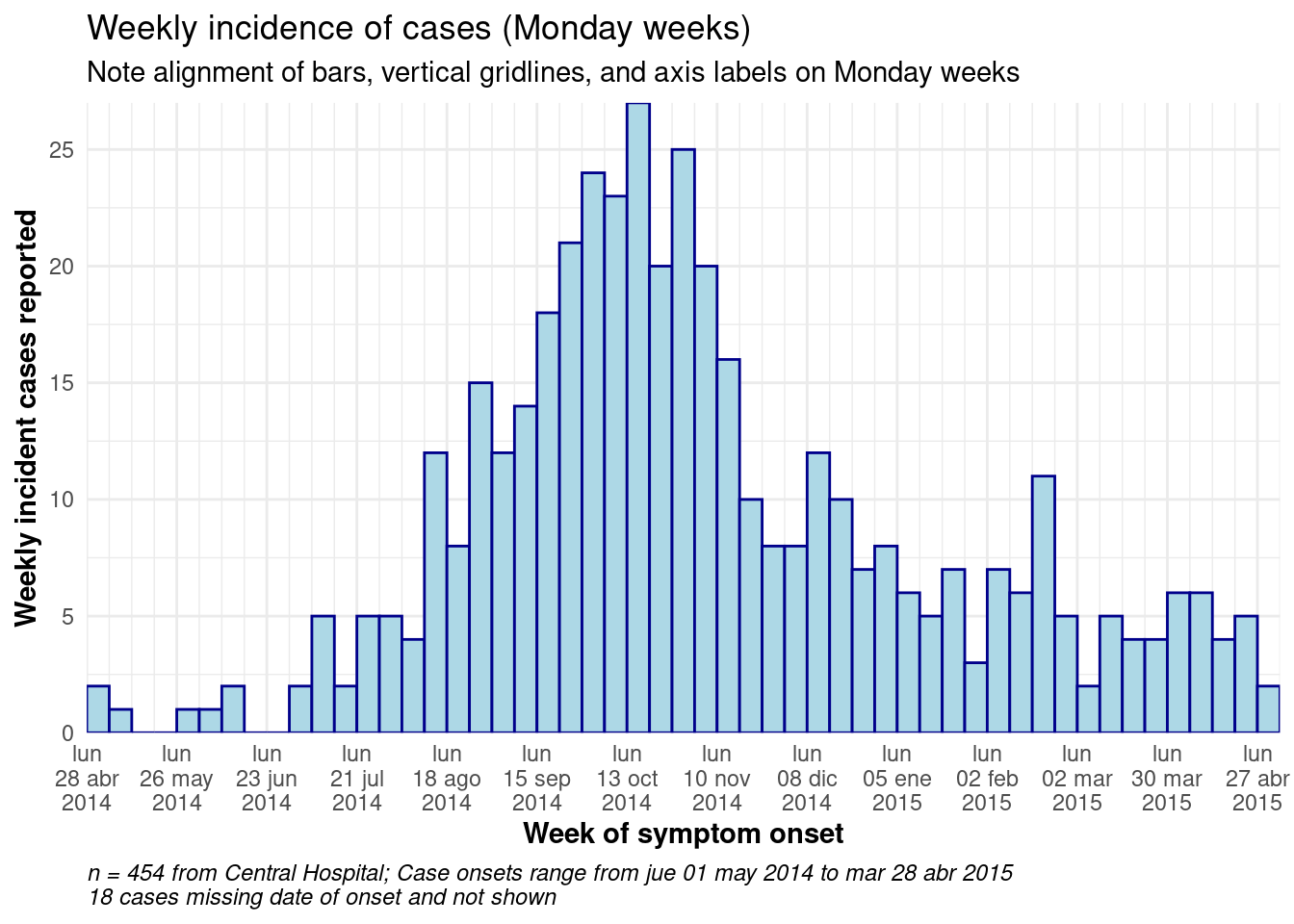
Semanas dominicales
Para conseguir el gráfico anterior para las semanas desde los domingos son necesarias algunas modificaciones, ya que los date_breaks = "weeks" sólo funcionan para las semanas de los lunes.
- Los puntos de ruptura de las franjas del histograma deben fijarse en los domingos (
week_start = 7) - Dentro de
scale_x_date(), los saltos de fecha similares deben proporcionarse abreaks =yminor_breaks =para asegurar que las etiquetas de fecha y las líneas verticales de la cuadrícula se alineen los domingos.
Por ejemplo, el comando scale_x_date() para las semanas del domingo podría tener este aspecto:
scale_x_date(
expand = c(0,0),
# especificar el intervalo de las etiquetas de fecha y las principales Cuadrícula
breaks = seq.Date(
from = floor_date(min(central_data$date_onset, na.rm=T), "week", week_start = 7), # domingo anterior al primer caso
to = ceiling_date(max(central_data$date_onset, na.rm=T), "week", week_start = 7), # domingo posterior al último caso
by = "4 weeks"),
# especificar el intervalo de la Cuadrícula vertical menor
minor_breaks = seq.Date(
from = floor_date(min(central_data$date_onset, na.rm=T), "week", week_start = 7), # domingo anterior al primer caso
to = ceiling_date(max(central_data$date_onset, na.rm=T), "week", week_start = 7), # domingo posterior al último caso
by = "week"),
# formato de etiqueta de fecha
date_labels = "%a\n%d %b\n%Y")+ # día, sobre mes abreviado, año de 2 dígitosAgrupar/colorear por valor
Las barras del histograma pueden colorearse por grupos y “apilarse”. Para designar la columna de agrupación, haz los siguientes cambios. Consulta la página de Conceptos básicos de ggplot para más detalles.
- Dentro del mapeo estético del histograma
aes(), asigna el nombre de la columna a los argumentosgroup =yfill = - Elimina cualquier argumento
fill =fuera deaes(), ya que anulará el de dentro - Los argumentos dentro de
aes()se aplicarán por grupo, mientras que los de fuera se aplicarán a todas las barras (por ejemplo, es posible que quierascolor =fuera, para que cada barra tenga el mismo borde)
Este es el aspecto que tendría el comando aes() para agrupar y colorear las barras por gender:
aes(x = date_onset, group = gender, fill = gender)Aquí se aplica:
ggplot(data = linelist) + # empieza con linelist (muchos hospitales)
# realizar el histograma: especifica los puntos de corte: empieza el lunes anterior al primer caso, termina el lunes posterior al último caso
geom_histogram(
mapping = aes(
x = date_onset,
group = hospital, # agrupa los datos por hospital
fill = hospital), # relleno de las barras (color interior) por hospital
# las saltos son las semanas de los lunes
breaks = weekly_breaks_all, # secuencia de saltos de semanas de lunes para todo el brote, definidos en el código anterior
# Color alrededor de las barras
color = "black")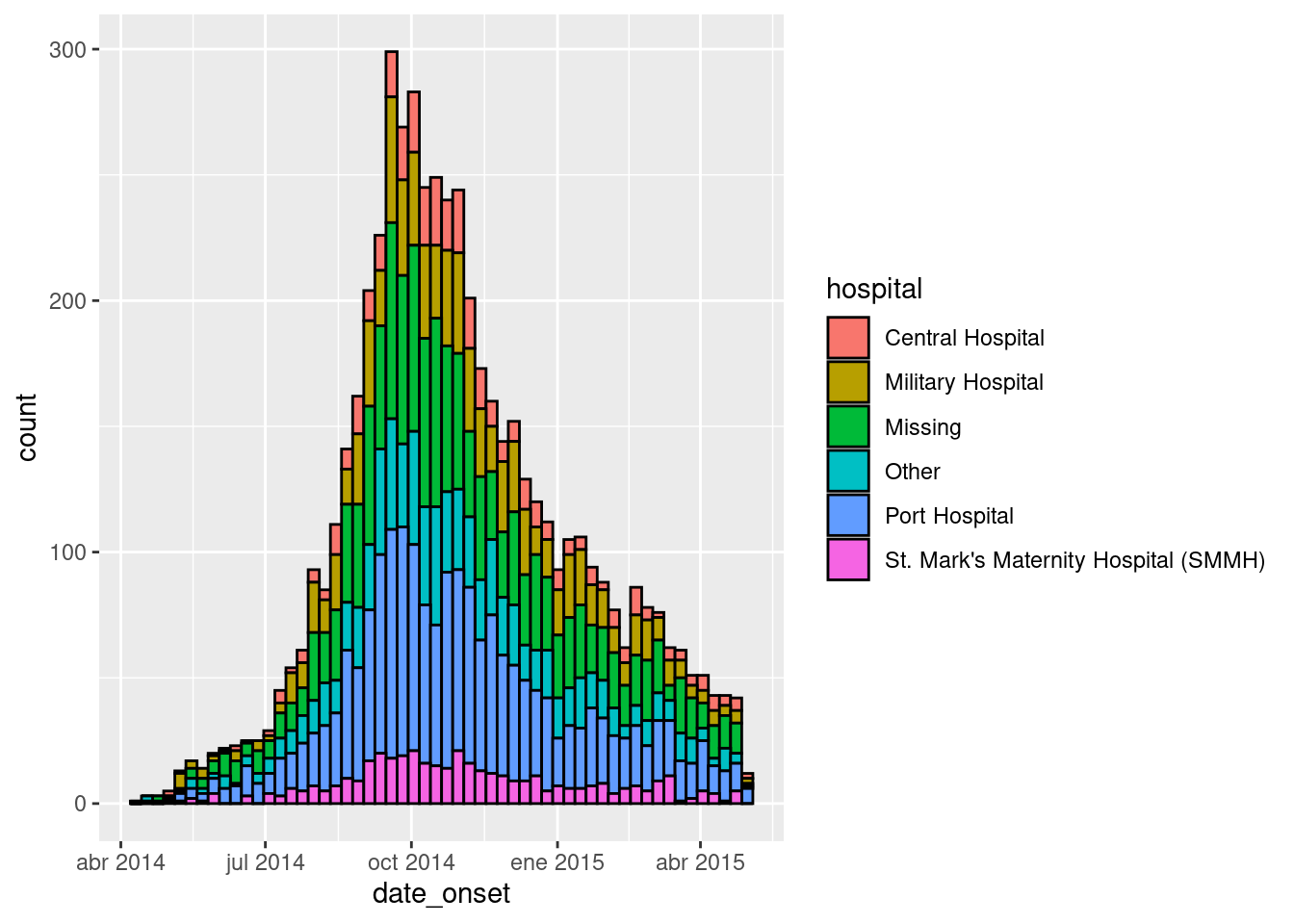
Ajustar los colores
- Para establecer manualmente el relleno de cada grupo, utiliza
scale_fill_manual()(nota:scale_color_manual()es diferente).- Utiliza el argumento
values =para aplicar un vector de colores. - Utiliza
na.value =para especificar un color para los valoresNA. - Utiliza el argumento
labels =para cambiar el texto de los elementos de la leyenda. Para estar seguro, proporciónalo como un vector, comoc("old" = "new", "old" = "new")o ajusta los valores en los propios datos. - Utiliza
name =para dar un título adecuado a la leyenda
- Utiliza el argumento
- Consulta la página sobre Conceptos básicos de ggplot para obtener más información sobre escalas y paletas de colores.
ggplot(data = linelist)+ # empieza con linelist (muchos hospitales)
# realizar el histograma
geom_histogram(
mapping = aes(x = date_onset,
group = hospital, # casos agrupados por hospital
fill = hospital), # relleno de las barras por hospital
# saltos
breaks = weekly_breaks_all, # secuencia de saltos de semanas de lunes, definidos en el código anterior
# Color alrededor de las barras
color = "black")+ # color del borde de cada barra
# especificación manual de colores
scale_fill_manual(
values = c("black", "orange", "grey", "beige", "blue", "brown"),
labels = c("St. Mark's Maternity Hospital (SMMH)" = "St. Mark's"),
name = "Hospital") # especifica los colores de relleno (valores) - ¡atención al orden!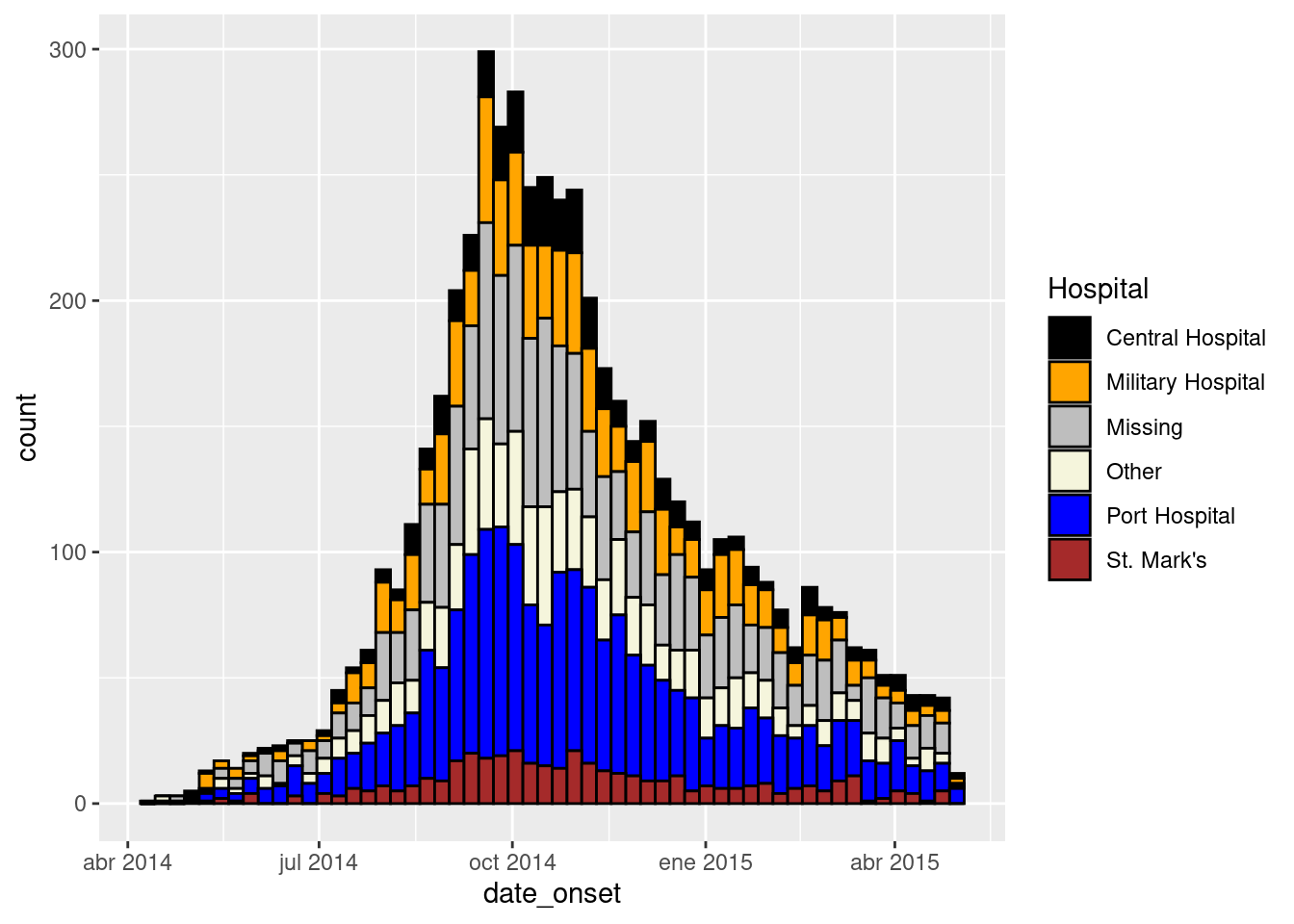
Ajustar el orden de los niveles
El orden en que se apilan las barras agrupadas se ajusta mejor clasificando la columna de agrupación como tipo Factor. A continuación, puedes designar el orden de los niveles de los factores (y sus etiquetas de visualización). Consulta la página sobre Factores o consejos de ggplot para obtener más detalles.
Antes de realizar el gráfico, utiliza la función fct_relevel() del paquete forcats para convertir la columna de agrupación en de tipo factor y ajustar manualmente el orden de los niveles, como se detalla en la página sobre Factores.
# carga el paquete forcats para trabajar con factores
pacman::p_load(forcats)
# Definir nuevo conjunto de datos con hospital como factor
plot_data <- linelist %>%
mutate(hospital = fct_relevel(hospital, c("Missing", "Other"))) # Convierte a factor y establece "Missing" y "Other" como niveles superiores para aparecer en la curvaepi
levels(plot_data$hospital) # imprime los niveles en orden## [1] "Missing" "Other" "Central Hospital" "Military Hospital" "Port Hospital"
## [6] "St. Mark's Maternity Hospital (SMMH)"En el siguiente gráfico, las únicas diferencias con respecto al anterior es que la columna hospital se ha consolidado como en el caso anterior, y utilizamos guides() para invertir el orden de la leyenda, de modo que “Missing” se encuentra en la parte inferior de la leyenda.
ggplot(plot_data) + # Utiliza el NUEVO conjunto de datos con el hospital como factor reordenado
# realizar el histograma
geom_histogram(
mapping = aes(x = date_onset,
group = hospital, # casos agrupados por hospital
fill = hospital), # relleno de las barras por hospital
breaks = weekly_breaks_all, # secuencia de pausas de semanas de lunes para todo el brote, definidas en la parte superior de la sección ggplot
color = "black")+ # color del borde de cada barra
# Etiquetas del eje-x
scale_x_date(
expand = c(0,0), # elimina el exceso de espacio del eje-x antes y después de las barras de casos
date_breaks = "3 weeks", # las etiquetas aparecen cada 3 semanas de lunes
date_minor_breaks = "week", # las líneas verticales aparecen cada semana de lunes
date_labels = "%d\n%b\n'%y")+ # formato de las etiquetas de fecha
# eje-y
scale_y_continuous(
expand = c(0,0))+ # elimina el exceso de espacio del eje-y por debajo de 0
# especificación manual de colores, ¡atención al orden!
scale_fill_manual(
values = c("grey", "beige", "black", "orange", "blue", "brown"),
labels = c("St. Mark's Maternity Hospital (SMMH)" = "St. Mark's"),
name = "Hospital")+
# temas de estética
theme_minimal()+ # simplificar el fondo del gráfico
theme(
plot.caption = element_text(face = "italic", # texto en cursiva a la izquierda
hjust = 0),
axis.title = element_text(face = "bold"))+ # títulos de los ejes en negrita
# etiquetas
labs(
title = "Weekly incidence of cases by hospital",
subtitle = "Hospital as re-ordered factor",
x = "Week of symptom onset",
y = "Weekly cases")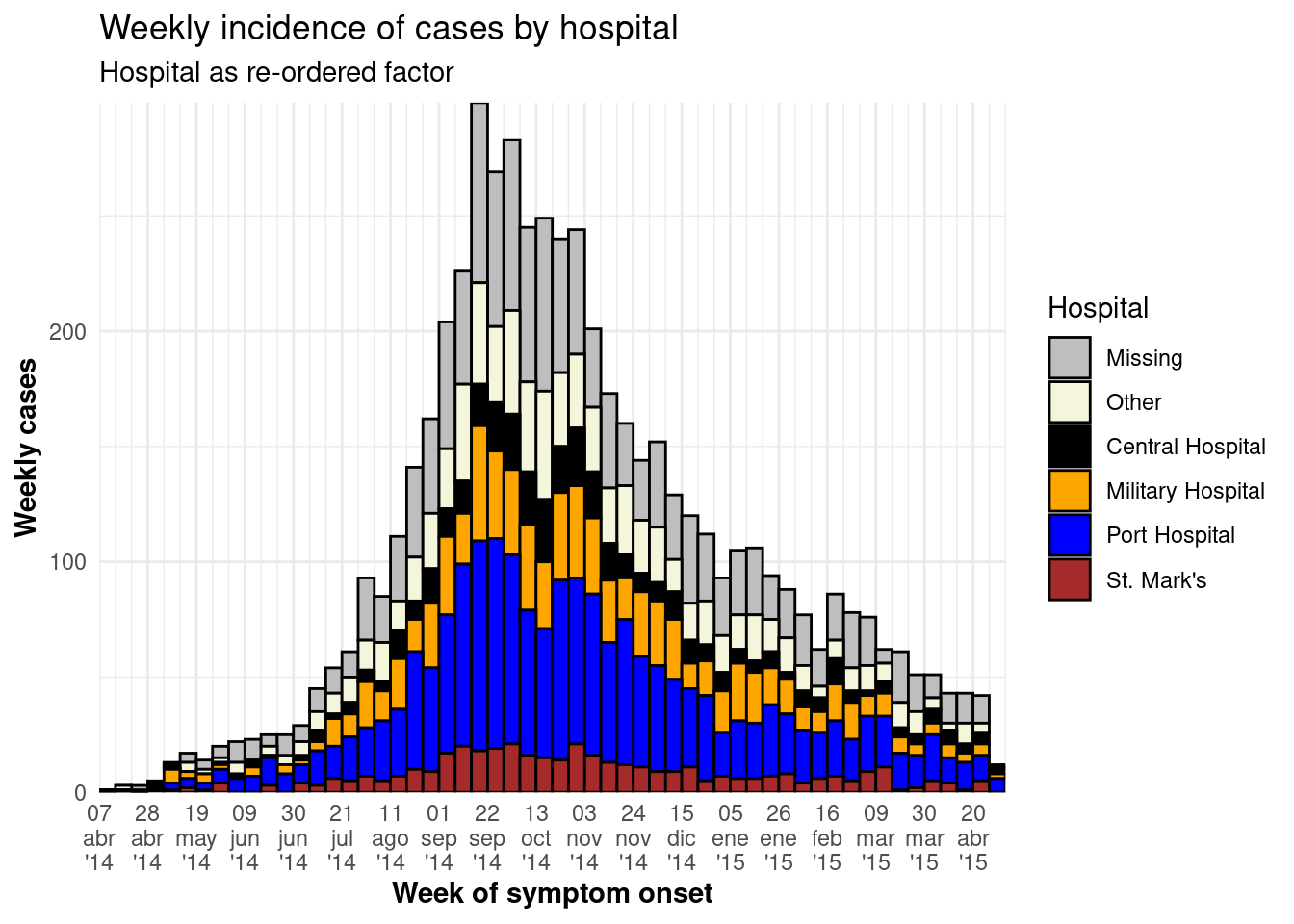
CONSEJO: Para invertir solamente el orden de la leyenda, añade este comando ggplot2: guides(fill = guide_legend(reverse = TRUE)).
Ajustar la leyenda
Lee más sobre las leyendas y las escalas en la página Consejos de ggplot. Aquí hay algunos puntos destacados:
- Edita el título de la leyenda, ya sea en la función de escala o con
labs(fill = "Título de la leyenda")(si estás usandocolor =estético, entonces usalabs(color = "")) -
theme(legend.title = element_blank())para no tener título de leyenda -
theme(legend.position = "top")(“bottom”, “left”, “right”, o “none” para eliminar la leyenda) -
theme(legend.direction = "horizontal")leyenda horizontal -
guides(fill = guide_legend(reverse = TRUE))para invertir el orden de la leyenda
Barras de lado a lado
La visualización lado a lado de las barras de grupo (en lugar de apiladas) se especifica dentro de geom_histogram() con position = "dodge" colocado fuera de aes().
Si hay más de dos grupos de valores, éstos pueden resultar difíciles de leer. Considera la posibilidad de utilizar un gráfico facetado (múltiples pequeños). Para mejorar la legibilidad en este ejemplo, se han eliminado los valores de género que faltan.
ggplot(central_data %>% drop_na(gender))+ # comienza con los casos del Hospital Central eliminando el género faltante
geom_histogram(
mapping = aes(
x = date_onset,
group = gender, # casos agrupados por género
fill = gender), # barras rellenas por género
# cortes del histograma
breaks = weekly_breaks_central, # secuencia de fechas semanales para el brote del Central - definida en la parte superior de la sección ggplot
color = "black", # color del borde de la barra
position = "dodge")+ # barras LADO A LADO
# Las etiquetas en el eje-x
scale_x_date(expand = c(0,0), # elimina el exceso de espacio del eje-x antes y después de las barras de casos
date_breaks = "3 weeks", # las etiquetas aparecen cada 3 semanas de lunes
date_minor_breaks = "week", # las líneas verticales aparecen cada semana de lunes
date_labels = "%d\n%b\n'%y")+ # formato de las etiquetas de fecha
# eje-y
scale_y_continuous(expand = c(0,0))+ # elimina el exceso de espacio del eje-y por debajo de 0
#scale of colors and legend labels
scale_fill_manual(values = c("brown", "orange"), # especificación manual de colores, ¡atención al orden!
na.value = "grey" )+
# temas de estética
theme_minimal()+ # un conjunto de temas para simplificar el gráfico
theme(plot.caption = element_text(face = "italic", hjust = 0), # texto a la izquierda en cursiva
axis.title = element_text(face = "bold"))+ # títulos de los ejes en negrita
# etiquetas
labs(title = "Weekly incidence of cases, by gender",
subtitle = "Subtitle",
fill = "Gender", # proporciona un nuevo título para la leyenda
x = "Week of symptom onset",
y = "Weekly incident cases reported")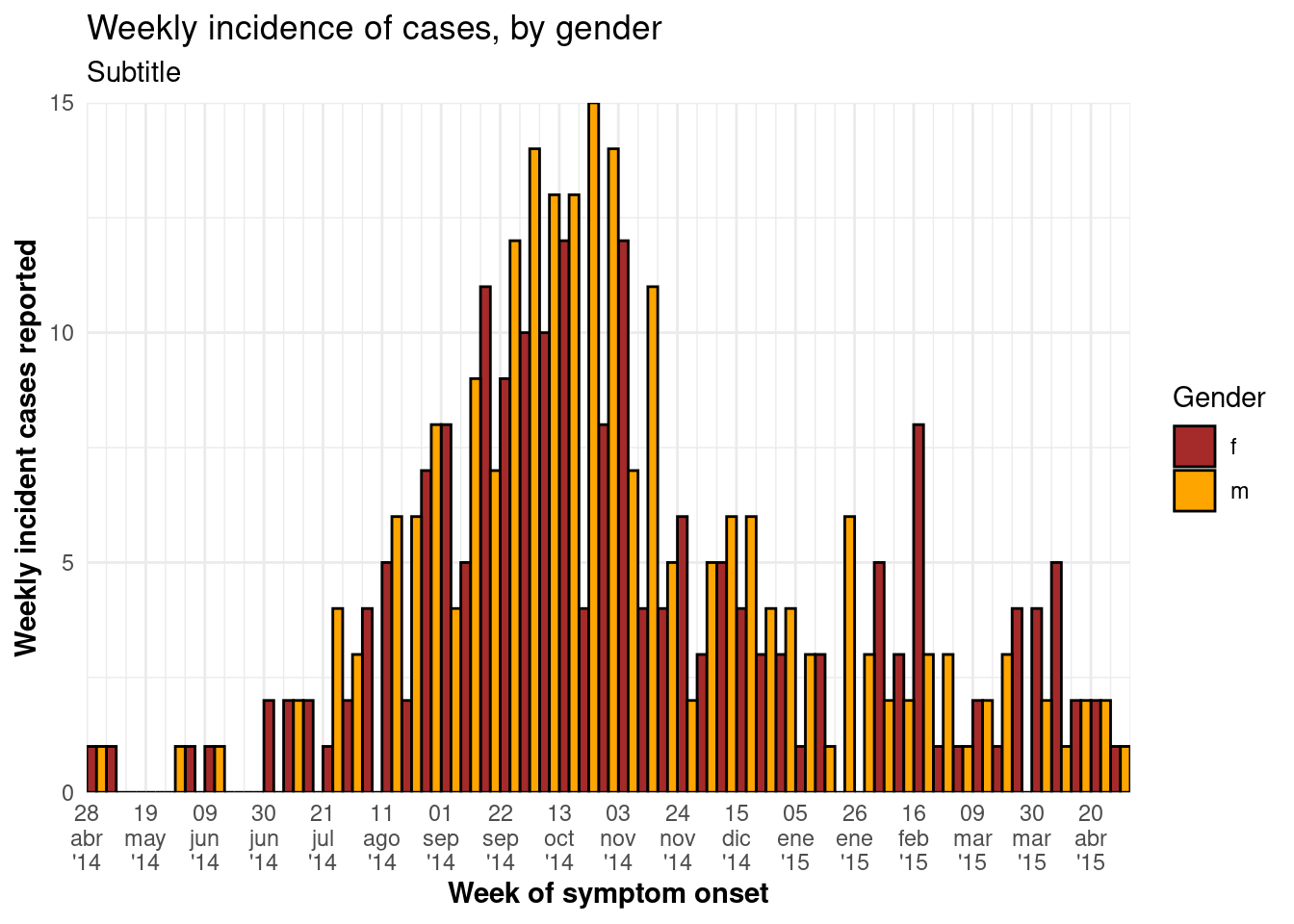
Límites del eje
Hay dos maneras de limitar la extensión de los valores del eje.
Por lo general, la forma preferida es utilizar el comando coord_cartesian(), que acepta xlim = c(min, max) y ylim = c(min, max) (donde proporcionas los valores mínimos y máximos). Esto actúa como un “zoom” sin eliminar realmente ningún dato, lo que es importante para las estadísticas y las medidas de resumen.
Alternativamente, puedes establecer valores de fecha máximos y mínimos utilizando limits = c() dentro de scale_x_date(). Por ejemplo:
scale_x_date(limits = c(as.Date("2014-04-01"), NA)) # fija una fecha mínima pero deja abierta la máxima. Asimismo, si deseas que el eje-x se extienda hasta una fecha concreta (por ejemplo, la fecha actual), aunque no se hayan notificado nuevos casos, puedes utilizar
scale_x_date(limits = c(NA, Sys.Date()) # Asegura que el eje de fecha se extenderá hasta la fecha actual PELIGRO: Ten cuidado al establecer los cortes o límites de la escala del eje-y (por ejemplo, de 0 a 30 por 5: seq(0, 30, 5)). Tales números estáticos pueden cortar tu gráfica demasiado si los datos cambian para superar el límite!
Ejes de fecha etiquetas/cuadrículas
CONSEJO: Recuerda que las etiquetas de los ejes de fecha son independientes de la agregación de los datos en barras, pero visualmente puede ser importante alinear las franjas, las etiquetas de fecha y las líneas verticales de la cuadrícula.
Para modificar las etiquetas de fecha y las líneas de la cuadrícula, utiliza scale_x_date() de una de estas maneras:
-
Si los intervalos de tu histograma son días, semanas de lunes, meses o años:
- Utiliza
date_breaks =para especificar el intervalo de las etiquetas y las líneas principales de la cuadrícula (por ejemplo, “day”, “week”, “3 weeks”, “month”, o “year”) - Utiliza
date_minor_breaks =para especificar el intervalo de las líneas verticales menores (entre las etiquetas de fecha) - Añade
expand = c(0,0)para comenzar las etiquetas en la primera barra - Usa
date_labels =para especificar el formato de las etiquetas de fecha - mira la página de trabajar con fechas para consejos (usa\npara una nueva línea)
- Utiliza
-
Si las franjas de tu histograma son semanas de domingo:
- Usa
breaks =yminor_breaks =proporcionando una secuencia de saltos de fecha para cada una - Puedes seguir utilizando
date_labels =yexpand =para formatear, como se ha descrito anteriormente
- Usa
Algunas notas:
- Consulta la sección de apertura de ggplot para obtener instrucciones sobre cómo crear una secuencia de fechas utilizando
seq.Date(). - Consulta esta página o la página Trabajar con fechas para obtener consejos sobre la creación de etiquetas con fechas.
Demostraciones
A continuación se hace una demostración de gráficos en los que los intervalos y las etiquetas de los gráficos/líneas de la cuadrícula están alineados y no alineados:
# Intervalos de 7 días + etiquetas de lunes
###########################################
ggplot(central_data) +
geom_histogram(
mapping = aes(x = date_onset),
binwidth = 7, # intervalos de 7 días con inicio en el primer caso
color = "darkblue",
fill = "lightblue") +
scale_x_date(
expand = c(0,0), # elimina el exceso de espacio bajo el eje-x y después de las barras de casos
date_breaks = "3 weeks", # lunes cada 3 semanas
date_minor_breaks = "week", # semanas de lunes
date_labels = "%a\n%d\n%b\n'%y")+ # formato de etiqueta
scale_y_continuous(
expand = c(0,0))+ # elimina el espacio sobrante bajo el eje-x y alinea
labs(
title = "MISALIGNED",
subtitle = "! CAUTION: 7-day bars start Thursdays at first case\nDate labels and gridlines on Mondays\nNote how ticks don't align with bars")
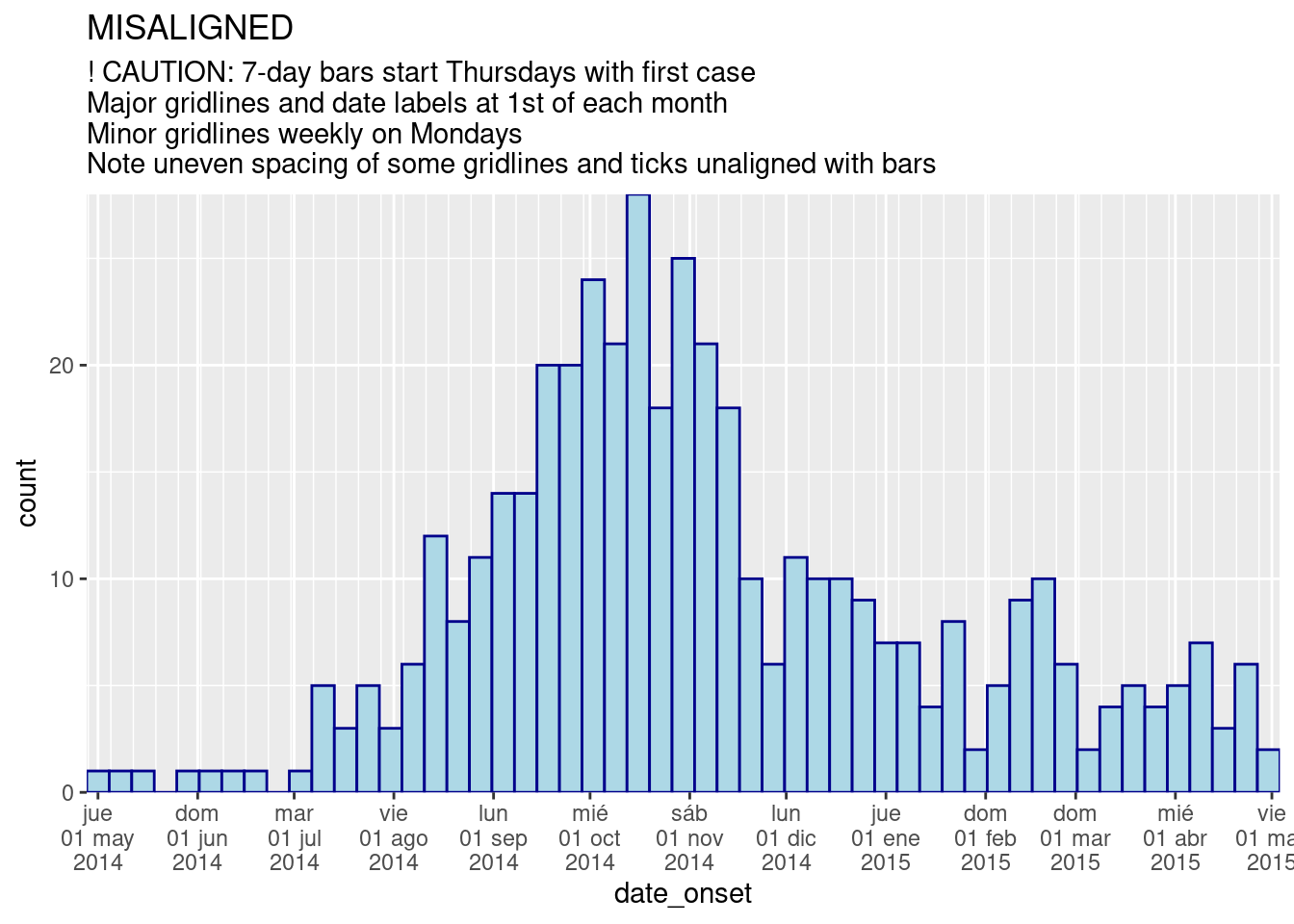
# intervalos de 7 días + Meses
##############################
ggplot(central_data) +
geom_histogram(
mapping = aes(x = date_onset),
binwidth = 7,
color = "darkblue",
fill = "lightblue") +
scale_x_date(
expand = c(0,0), # elimina el exceso de espacio bajo el eje-x y después de las barras de casos
date_breaks = "months", # 1º de mes
date_minor_breaks = "week", # semanas de lunes
date_labels = "%a\n%d %b\n%Y")+ # formato de etiqueta
scale_y_continuous(
expand = c(0,0))+ # elimina el espacio sobrante bajo el eje-x y alinea
labs(
title = "MISALIGNED",
subtitle = "! CAUTION: 7-day bars start Thursdays with first case\nMajor gridlines and date labels at 1st of each month\nMinor gridlines weekly on Mondays\nNote uneven spacing of some gridlines and ticks unaligned with bars")
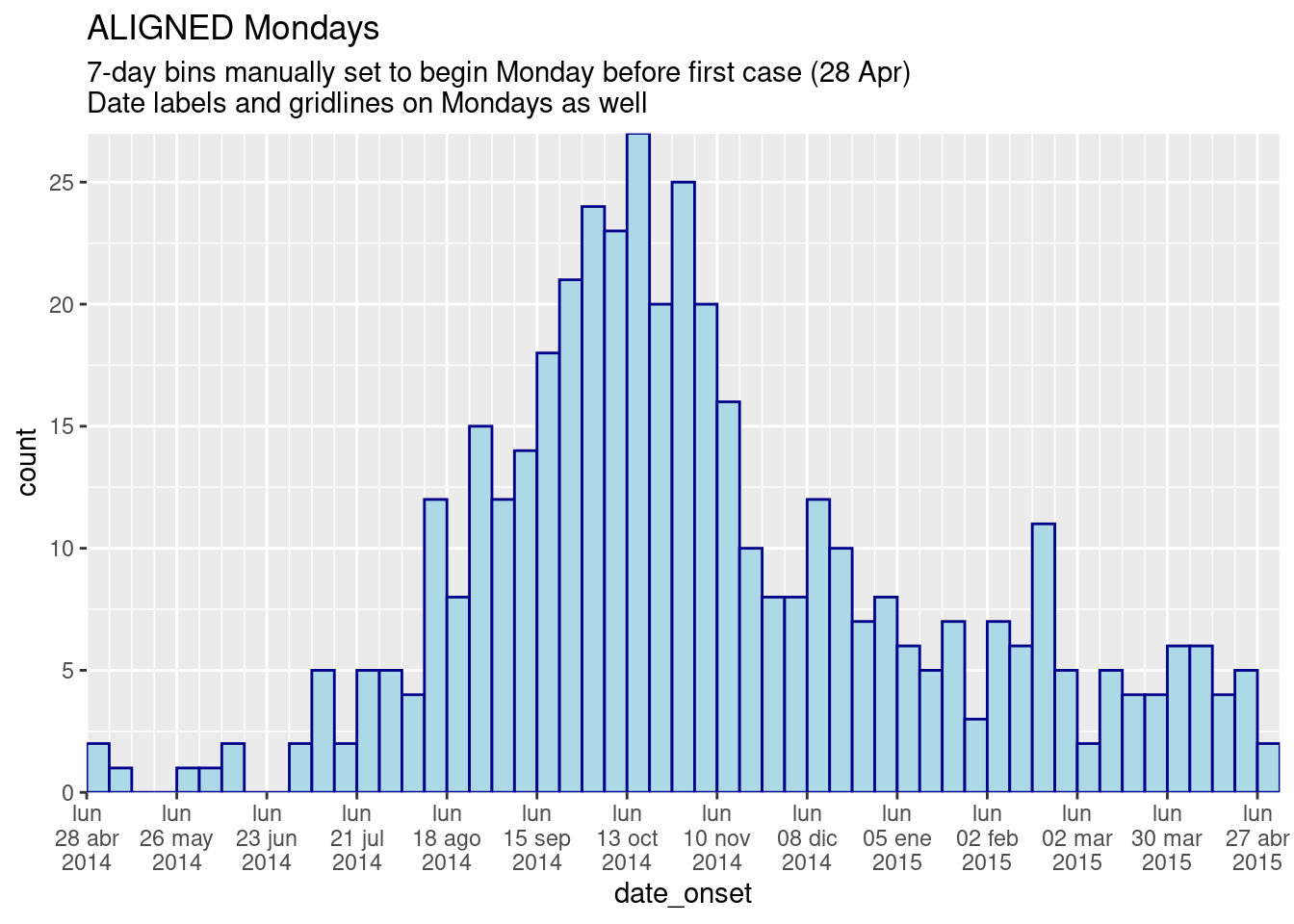
# ALINEACIÓN TOTAL DEL LUNES: especificar que los saltos manuales de las cajas sean los lunes
#############################################################################################
ggplot(central_data) +
geom_histogram(
mapping = aes(x = date_onset),
# los cortes del histograma se establecen en 7 días a partir del lunes anterior al primer caso
breaks = weekly_breaks_central, # definido anteriormente en esta página
color = "darkblue",
fill = "lightblue") +
scale_x_date(
expand = c(0,0), # elimina el espacio sobrante antes y después de las barras de casos
date_breaks = "4 weeks", # Lunes cada 4 semanas
date_minor_breaks = "week", # semanas de lunes
date_labels = "%a\n%d %b\n%Y")+ # formato de etiqueta
scale_y_continuous(
expand = c(0,0))+ # elimina el espacio sobrante bajo el eje-x y alinea
labs(
title = "ALIGNED Mondays",
subtitle = "7-day bins manually set to begin Monday before first case (28 Apr)\nDate labels and gridlines on Mondays as well")
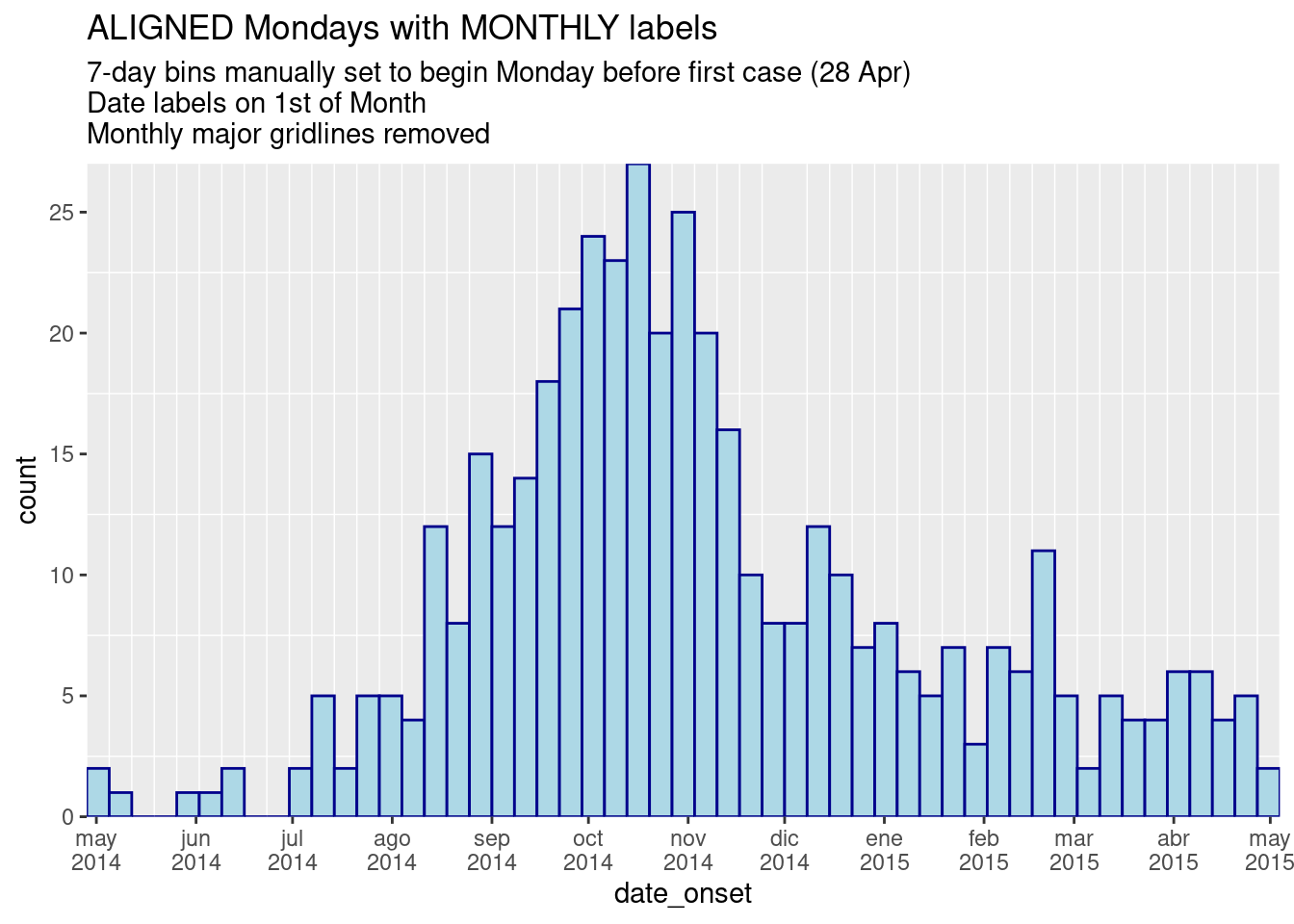
# ALINEACIÓN TOTAL DE LUNES CON ETIQUETAS DE MESES:
###################################################
ggplot(central_data) +
geom_histogram(
mapping = aes(x = date_onset),
# los cortes del histograma se establecen en 7 días a partir del lunes anterior al primer caso
breaks = weekly_breaks_central, # definido anteriormente en esta página
color = "darkblue",
fill = "lightblue") +
scale_x_date(
expand = c(0,0), # elimina el exceso de espacio en el eje-x antes y después de las barras de casos
date_breaks = "months", # Lunes cada 4 semanas
date_minor_breaks = "week", # semanas de lunes
date_labels = "%b\n%Y")+ # formato de etiqueta
scale_y_continuous(
expand = c(0,0))+ # elimina el espacio sobrante bajo el eje-x y alinea
theme(panel.grid.major = element_blank())+ # Elimina las cuadrículas principales ( coinciden con el día 1 de cada mes)
labs(
title = "ALIGNED Mondays with MONTHLY labels",
subtitle = "7-day bins manually set to begin Monday before first case (28 Apr)\nDate labels on 1st of Month\nMonthly major gridlines removed")
# ALINEACIÓN TOTAL DEL DOMINGO: especificar los puntos de corte manualmente Y las etiquetas que serán domingos
##############################################################################################################
ggplot(central_data) +
geom_histogram(
mapping = aes(x = date_onset),
# cortes del histograma fijados en 7 días a partir del domingo anterior al primer caso
breaks = seq.Date(from = floor_date(min(central_data$date_onset, na.rm=T), "week", week_start = 7),
to = ceiling_date(max(central_data$date_onset, na.rm=T), "week", week_start = 7),
by = "7 days"),
color = "darkblue",
fill = "lightblue") +
scale_x_date(
expand = c(0,0),
# rupturas de etiquetas de fecha y cuadrículas principales establecidas cada 3 semanas a partir del domingo anterior al primer caso
breaks = seq.Date(from = floor_date(min(central_data$date_onset, na.rm=T), "week", week_start = 7),
to = ceiling_date(max(central_data$date_onset, na.rm=T), "week", week_start = 7),
by = "3 weeks"),
# Cuadrículas menores fijadas en semanal a partir del domingo anterior al primer caso
minor_breaks = seq.Date(from = floor_date(min(central_data$date_onset, na.rm=T), "week", week_start = 7),
to = ceiling_date(max(central_data$date_onset, na.rm=T), "week", week_start = 7),
by = "7 days"),
date_labels = "%a\n%d\n%b\n'%y")+ # formato de etiqueta
scale_y_continuous(
expand = c(0,0))+ # elimina el espacio sobrante bajo el eje-x y ajusta
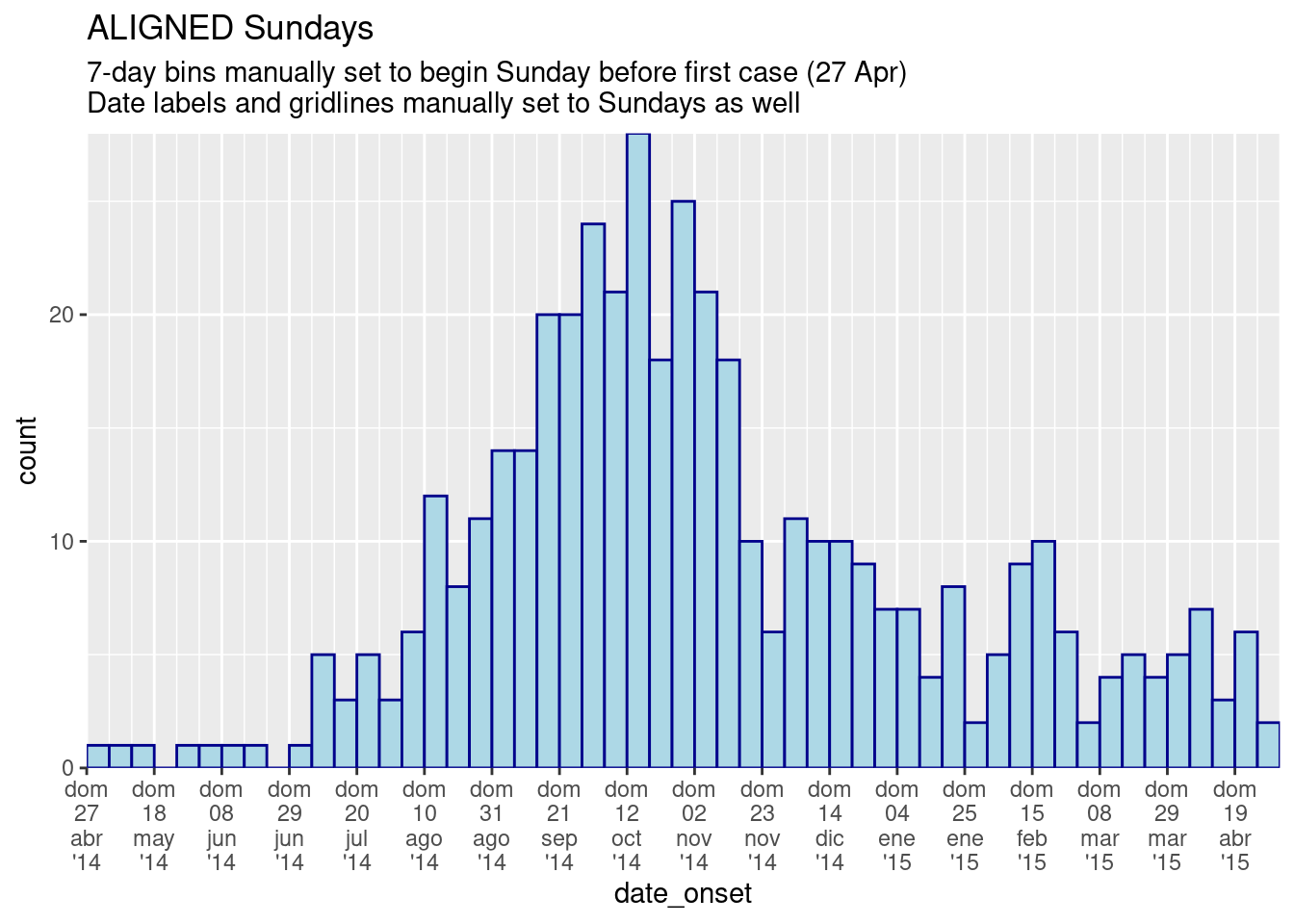
labs(title = "ALIGNED Sundays",
subtitle = "7-day bins manually set to begin Sunday before first case (27 Apr)\nDate labels and gridlines manually set to Sundays as well")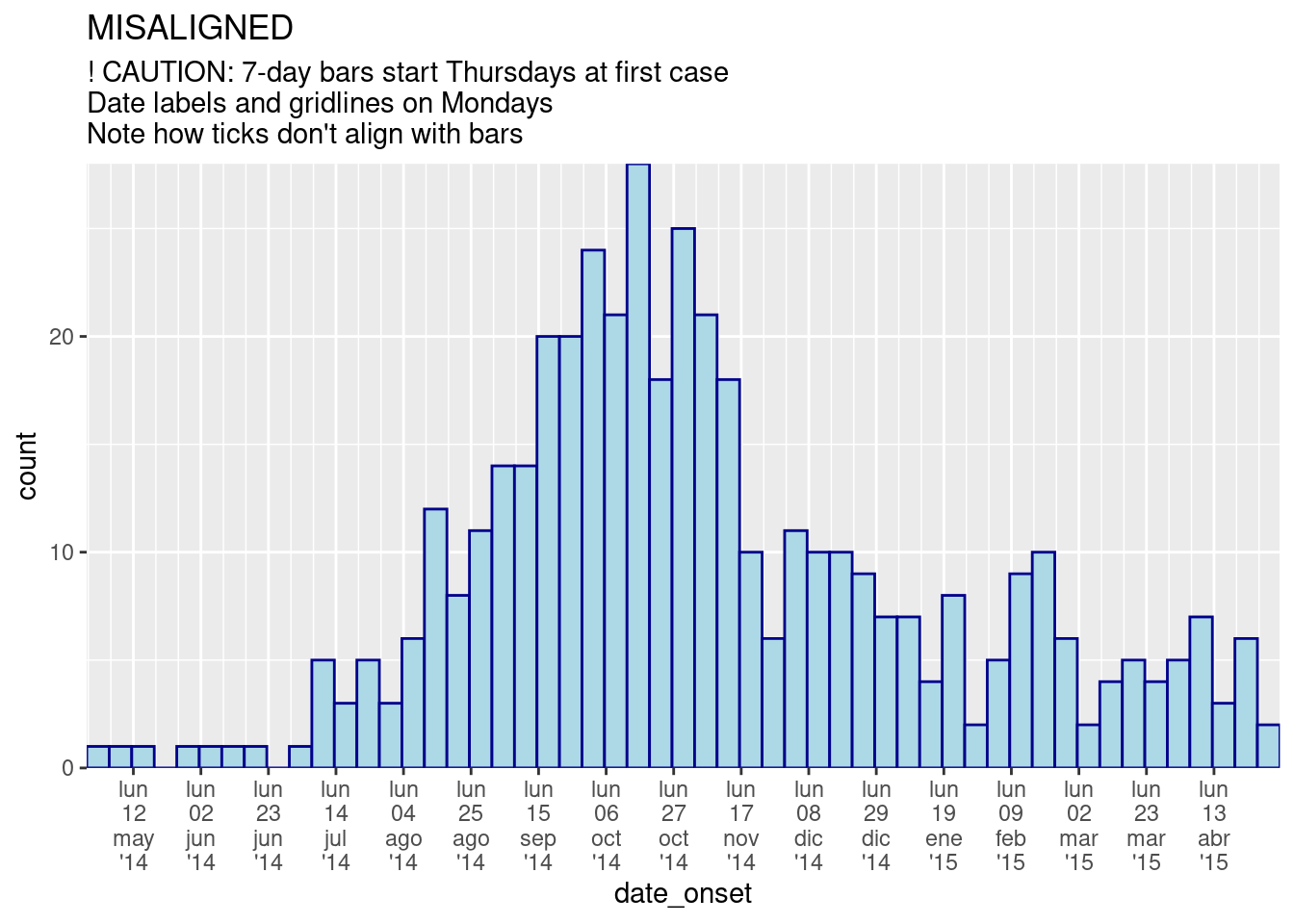
Datos agregados
A menudo, en lugar de un listado, se comienza con recuentos agregados de instalaciones, distritos, etc. Se puede hacer una curva epidémica con ggplot() pero el código será ligeramente diferente. Esta sección utilizará los datos de count_data que fue importado anteriormente, en la sección de preparación de datos. Este conjunto de datos es linelist agregado a los recuentos de día-hospital. A continuación se muestran las primeras 50 filas.
Representar recuentos diarios
Podemos trazar una curva epidémica diaria a partir de estos recuentos diarios. Aquí están las diferencias con el código:
- Dentro del mapeo estético
aes(), especificay =como columna de recuento (en este caso, el nombre de la columna esn_cases) - Añadir el argumento
stat = "identity"dentro degeom_histogram(), que especifica que la altura de la barra debe ser el valory =y no el número de filas, como es el valor por defecto - Añade el argumento
width =para evitar las líneas blancas verticales entre las barras. Para los datos diarios, establece el valor 1. Para los datos semanales, escribe 7. Para los datos de recuento mensual, las líneas blancas son un problema (cada mes tiene un número diferente de días) - considera la posibilidad de transformar el eje x en un factor categórico ordenado (meses) y utilizargeom_col().
ggplot(data = count_data)+
geom_histogram(
mapping = aes(x = date_hospitalisation, y = n_cases),
stat = "identity",
width = 1)+ # para recuentos diarios, establece anchura = 1 para evitar espacios en blanco entre barras
labs(
x = "Date of report",
y = "Number of cases",
title = "Daily case incidence, from daily count data")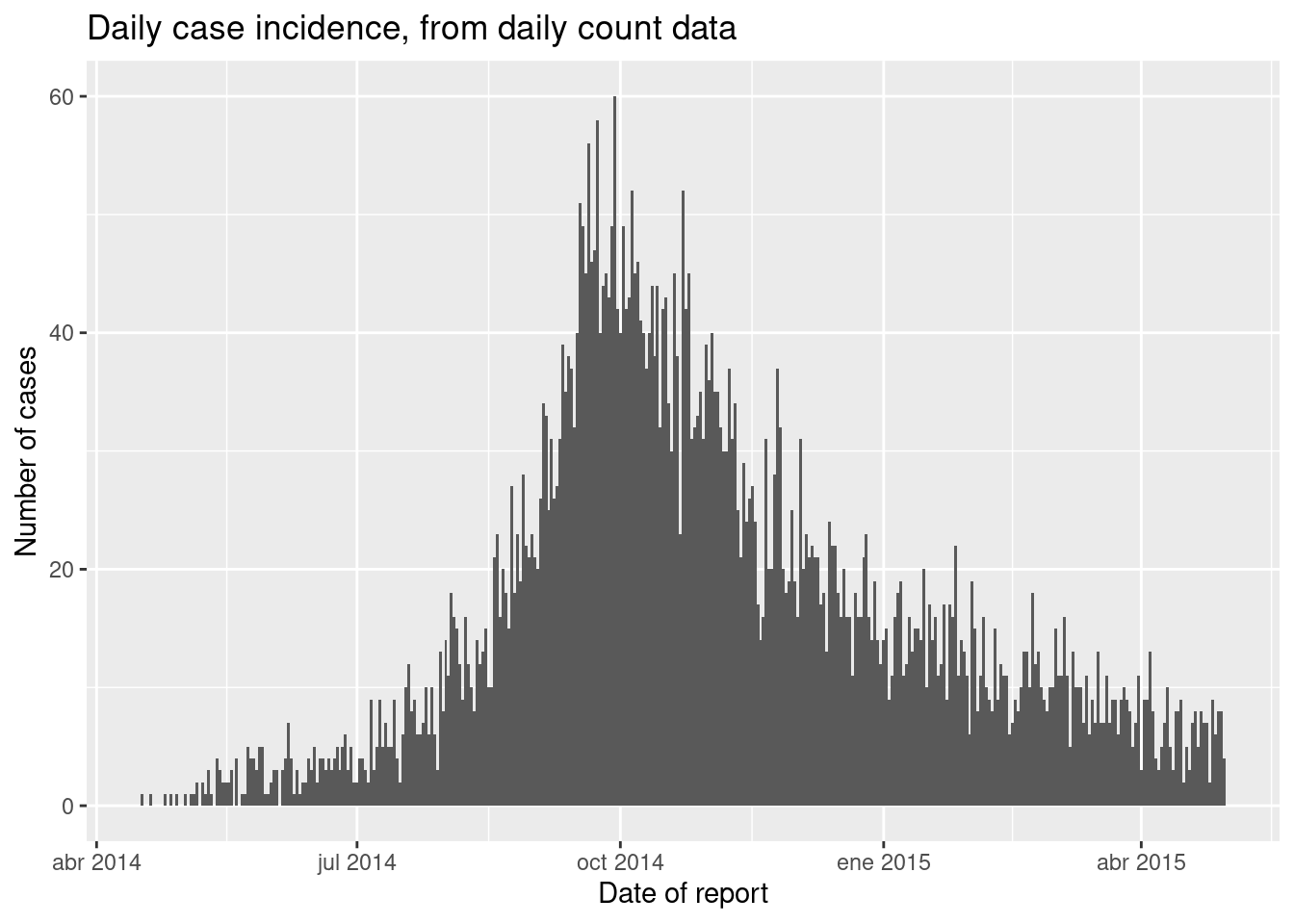
representar recuentos semanales
Si tus datos ya son recuentos de casos por semana, podrían parecerse a este conjunto de datos (llamado count_data_weekly):
A continuación se muestran las primeras 50 filas de count_data_weekly. Puedes ver que los recuentos se han agregado en semanas. Cada semana se muestra por el primer día de la semana (lunes por defecto).
Ahora trace de manera que x =la columna epiweek. Recuerda añadir y = la columna de recuentos al mapeo estético, y añadir stat = "identity" como se ha explicado anteriormente.
ggplot(data = count_data_weekly)+
geom_histogram(
mapping = aes(
x = epiweek, # el eje-x es epiweek (como tipo Fecha)
y = n_cases_weekly, # altura del eje-y en los recuentos semanales de casos
group = hospital, # agrupa las barras y colorea por hospital
fill = hospital),
stat = "identity")+ # esto también es necesario al dibujar datos de recuento
# etiquetas para el eje-x
scale_x_date(
date_breaks = "2 months", # etiquetas cada 2 meses
date_minor_breaks = "1 month", # cuadrículas cada mes
date_labels = '%b\n%Y')+ # etiquetado por mes con año debajo
# Elige la paleta de colores (utiliza el paquete RColorBrewer)
scale_fill_brewer(palette = "Pastel2")+
theme_minimal()+
labs(
x = "Week of onset",
y = "Weekly case incidence",
fill = "Hospital",
title = "Weekly case incidence, from aggregated count data by hospital")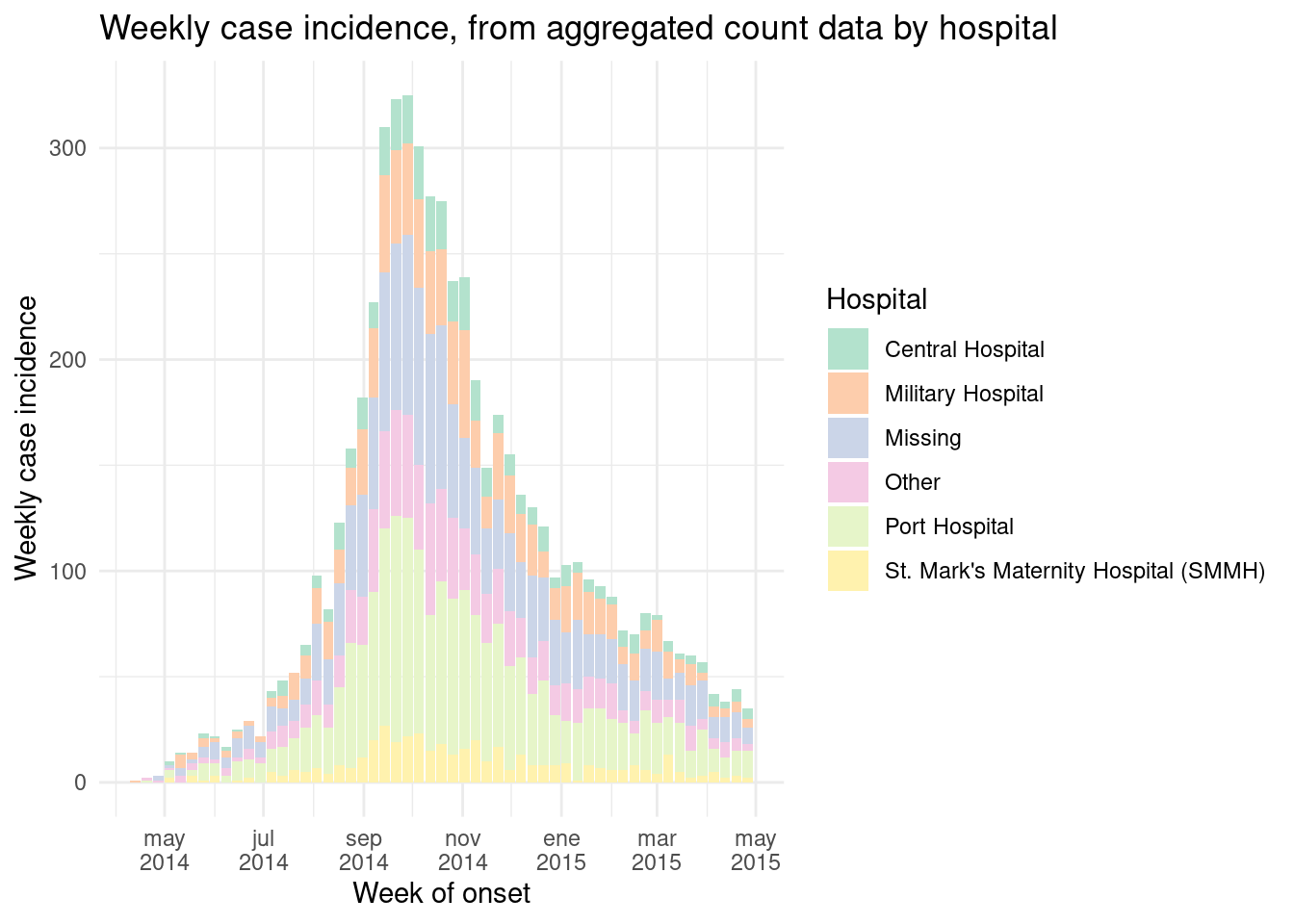
Medias móviles
Consulta la página sobre medias móviles para obtener una descripción detallada y varias opciones. A continuación se muestra una opción para calcular medias móviles con el paquete slider. En este enfoque, la media móvil se calcula antes de representarla:
- Agrega los datos en recuentos según sea necesario (diario, semanal, etc.) (véase la página de Agrupar datos)
- Crea una nueva columna para contener la media móvil, creada con
slide_index()del paquete slider - Dibuja la media móvil como una
geom_line()encima (después) del histograma de la curva epidémica
Es muy útil la viñeta en línea del paquete slider
# cargar paquete
pacman::p_load(slider) # slider used to calculate rolling averages
# crea un conjunto de datos con los recuentos diarios y la media móvil de 7 días
################################################################################
ll_counts_7day <- linelist %>% # comienza con linelist
## count cases by date
count(date_onset, name = "new_cases") %>% # nombra la nueva columna con los recuentos como "new_cases"
drop_na(date_onset) %>% # elimina los casos en los que falta date_onset
## calcular el número medio de casos en una ventana de 7 días
mutate(
avg_7day = slider::slide_index( # crea una nueva columna
new_cases, # calcula basándose en el valor de la columna new_cases
.i = date_onset, # el índice es date_onset col, por lo que las fechas no presentes se incluyen en la ventana
.f = ~mean(.x, na.rm = TRUE), # la función es mean() con los valores faltantes eliminados
.before = 6, # la ventana es el día y los 6 días anteriores
.complete = FALSE), # debe ser FALSE para que unlist() funcione en el siguiente paso
avg_7day = unlist(avg_7day)) # convierte el tipo lista en tipo numérico
# realizar el gráfico
#####################
ggplot(data = ll_counts_7day) + # comienza con el nuevo conjunto de datos definido anteriormente
geom_histogram( # crea el histograma de la curva epidemiológica
mapping = aes(
x = date_onset, # columna de fecha como eje-x
y = new_cases), # la altura es el número de casos nuevos diarios
stat = "identity", # la altura es el valor-y
fill="#92a8d1", # color frío para las barras
colour = "#92a8d1", # mismo color para el borde de las barras
)+
geom_line( # crea la linea de la media móvil
mapping = aes(
x = date_onset, # columna de fecha para el eje-x
y = avg_7day, # valor-y establecido con la columna de media móvil
lty = "7-day \nrolling avg"), # nombre de la línea en la leyenda
color="red", # color de la línea
size = 1) + # anchura de la línea
scale_x_date( # escala de fechas
date_breaks = "1 month",
date_labels = '%d/%m',
expand = c(0,0)) +
scale_y_continuous( # escala del eje-y
expand = c(0,0),
limits = c(0, NA)) +
labs(
x="",
y ="Number of confirmed cases",
fill = "Legend")+
theme_minimal()+
theme(legend.title = element_blank()) # elimina el título de la leyenda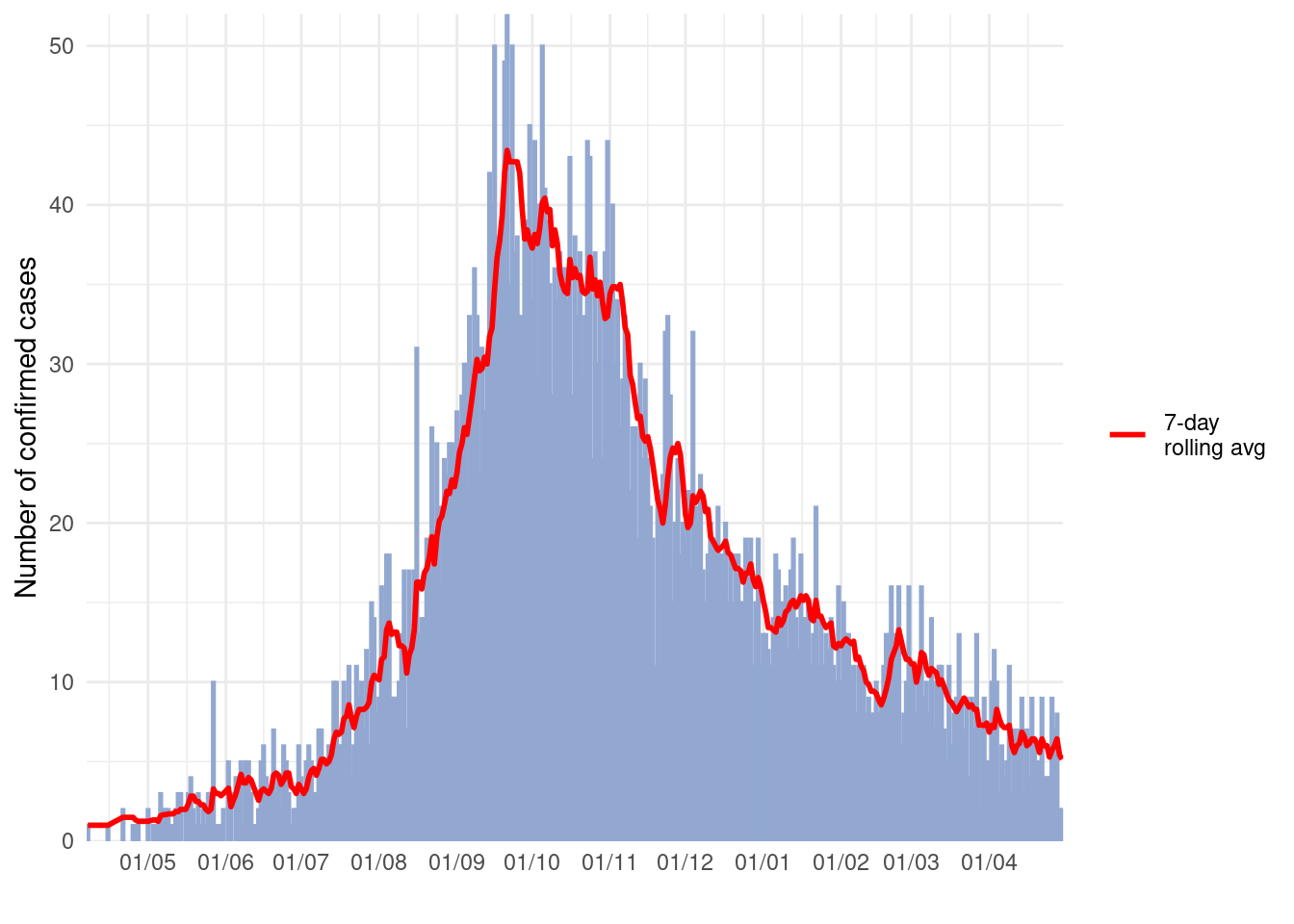
Facetas/pequeñas múltiples
Al igual que con otros ggplots, puedes crear gráficos facetados (“pequeños múltiples”). Como se explica en la página Consejos de ggplot de este manual, puedes utilizar facet_wrap() o facet_grid(). Aquí lo mostramos con facet_wrap(). Para las curvas epidémicas, facet_wrap() es típicamente más fácil, ya que es probable que sólo necesites facetar una columna.
La sintaxis general es facet_wrap(rows ~ cols), donde a la izquierda de la tilde (~) está el nombre de una columna que se extiende a través de las “filas” del gráfico facetado, y a la derecha de la tilde está el nombre de una columna que se extiende a través de las “columnas” del gráfico facetado. Lo más sencillo es utilizar un nombre de columna, a la derecha de la tilde: facet_wrap(~age_cat).
Ejes libres
Tendrás que decidir si las escalas de los ejes para cada faceta son “fijas” (por defecto), o “libres” (lo que significa que cambiarán en función de los datos dentro de la faceta). Haz esto con el argumento scales = dentro de facet_wrap() especificando “free_x” o “free_y”, o “free”.
Número de columnas y filas de las facetas
Se puede especificar con ncol = y nrow = dentro de facet_wrap().
Orden de los paneles Para cambiar el orden de aparición, cambia el orden de los niveles de la columna de factores utilizada para crear las facetas.
Estética
El tamaño y tipo de la fuente, el color de la franja, etc, se pueden modificar mediante theme() con argumentos como:
-
strip.text = element_text()(size, colour, face, angle..(tamaño, color, cara, ángulo) -
strip.background = element_rect()(e.g. element_rect(fill=“grey”))
-
strip.position =(posición “bottom”, “top”, “left”, o “right” (Abajo, arriba, izquierda o derecha))
Etiquetas de banda Las etiquetas de los gráficos de facetas pueden modificarse a través de las “etiquetas” de la columna como factor, o mediante el uso de un “etiquetador”.
Haz un etiquetador como este, usando la función as_labeller() de ggplot2. A continuación, proporciona el argumento labeller = en facet_wrap() como se muestra a continuación.
my_labels <- as_labeller(c(
"0-4" = "Ages 0-4",
"5-9" = "Ages 5-9",
"10-14" = "Ages 10-14",
"15-19" = "Ages 15-19",
"20-29" = "Ages 20-29",
"30-49" = "Ages 30-49",
"50-69" = "Ages 50-69",
"70+" = "Over age 70"))Un ejemplo de gráfico facetado - facetado por la columna age_cat.
# crear el gráfico
##################
ggplot(central_data) +
geom_histogram(
mapping = aes(
x = date_onset,
group = age_cat,
fill = age_cat), # los argumentos dentro de aes() se aplican por grupo
color = "black", # los argumentos fuera de aes() se aplican a todos los datos
# cortes del histograma
breaks = weekly_breaks_central)+ # vector de fechas predefinido (véase más arriba en esta página)
# Las etiquetas del eje-x
scale_x_date(
expand = c(0,0), # elimina el exceso de espacio del eje-x debajo y después de las barras de casos
date_breaks = "2 months", # elimina el exceso de espacio del eje-x debajo y después de las barras de casos
date_minor_breaks = "1 month", # las líneas verticales aparecen cada 1 mes
date_labels = "%b\n'%y")+ # formato de las etiquetas de fecha
# eje-y
scale_y_continuous(expand = c(0,0))+ # elimina el exceso de espacio en el eje y entre la parte inferior de las barras y las etiquetas
# temas estéticos
theme_minimal()+ # un conjunto de temas para simplificar el gráfico
theme(
plot.caption = element_text(face = "italic", hjust = 0), # leyenda a la izquierda en cursiva
axis.title = element_text(face = "bold"),
legend.position = "bottom",
strip.text = element_text(face = "bold", size = 10),
strip.background = element_rect(fill = "grey"))+ # títulos de los ejes en negrita
# crear facetas
facet_wrap(
~age_cat,
ncol = 4,
strip.position = "top",
labeller = my_labels)+
# Etiquetas
labs(
title = "Weekly incidence of cases, by age category",
subtitle = "Subtitle",
fill = "Age category", # proporciona un nuevo título para la leyenda
x = "Week of symptom onset",
y = "Weekly incident cases reported",
caption = stringr::str_glue("n = {nrow(central_data)} from Central Hospital; Case onsets range from {format(min(central_data$date_onset, na.rm=T), format = '%a %d %b %Y')} to {format(max(central_data$date_onset, na.rm=T), format = '%a %d %b %Y')}\n{nrow(central_data %>% filter(is.na(date_onset)))} cases missing date of onset and not shown"))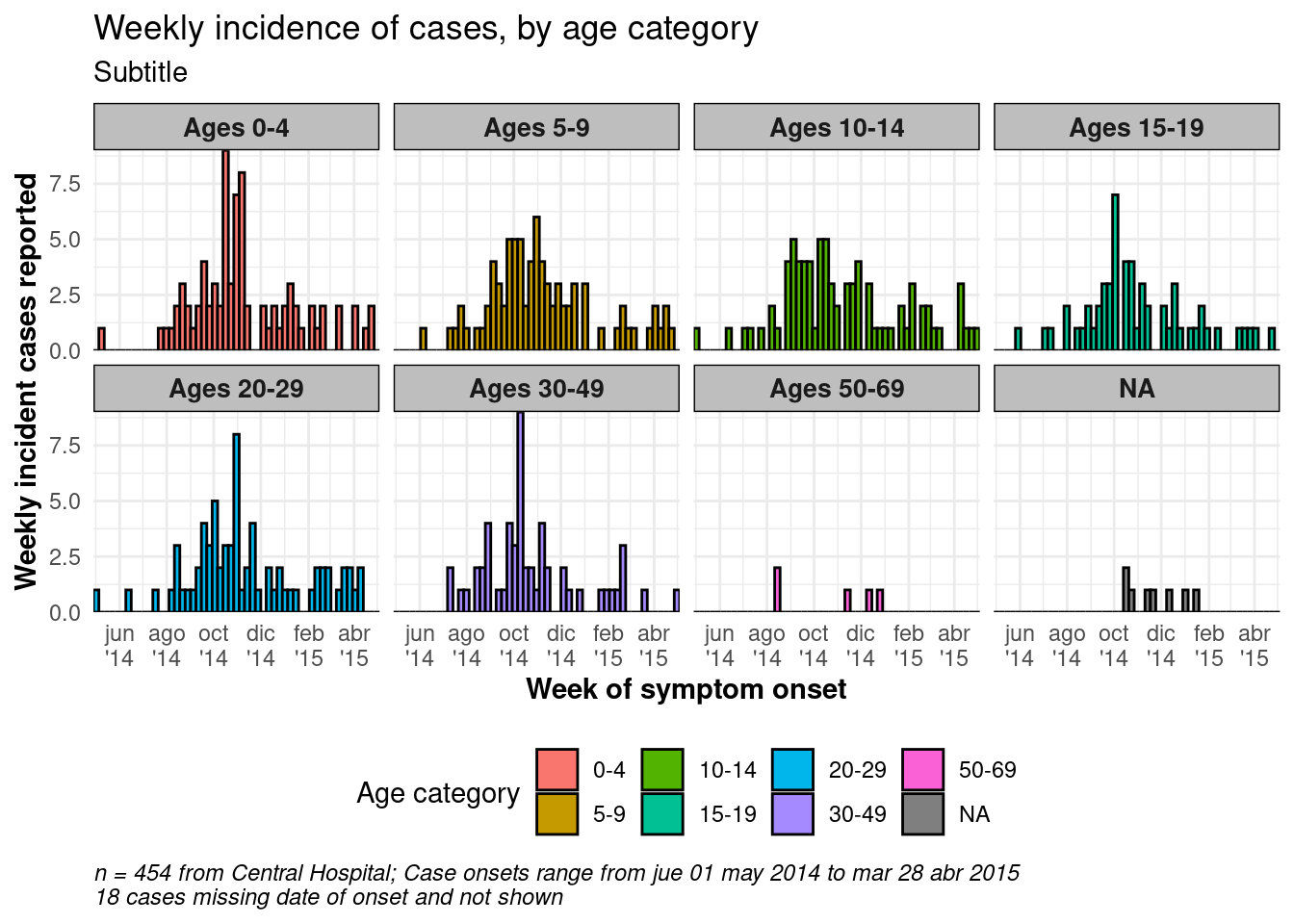
Consulta este enlace para obtener más información sobre las etiquetadoras.
Conjunto de la Epidemia como fondo de la faceta
Para mostrar el conjunto de la epidemia como fondo de cada faceta, añade la función gghighlight() con paréntesis vacíos al ggplot. Esto es del paquete gghighlight. Observa que el máximo del eje Y en todas las facetas se basa ahora en el pico de toda la epidemia. Hay más ejemplos de este paquete en la página Consejos de ggplot.
ggplot(central_data) +
# curvaepi por grupo
geom_histogram(
mapping = aes(
x = date_onset,
group = age_cat,
fill = age_cat), # los argumentos dentro de aes() se aplican por grupo
color = "black", # los argumentos fuera de aes() se aplican a todos los datos
# cortes del histograma
breaks = weekly_breaks_central)+ # vector de fechas predefinido (véase la parte superior de la sección ggplot)
# añade gris epidémico de fondo a cada faceta
gghighlight::gghighlight()+
# etiquetas en el eje-x
scale_x_date(
expand = c(0,0), # elimina el exceso de espacio en el eje-x debajo y después de las barras de casos
date_breaks = "2 months", # las etiquetas aparecen cada 2 meses
date_minor_breaks = "1 month", # las líneas verticales aparecen cada 1 mes
date_labels = "%b\n'%y")+ # formato de las etiquetas de fecha
# eje-y
scale_y_continuous(expand = c(0,0))+ # elimina el exceso de espacio del eje-y por debajo de 0
# aesthetic themes
theme_minimal()+ # una serie de temas para simplificar el gráfico
theme(
plot.caption = element_text(face = "italic", hjust = 0), # ajustado a la izquierda en cursiva
axis.title = element_text(face = "bold"),
legend.position = "bottom",
strip.text = element_text(face = "bold", size = 10),
strip.background = element_rect(fill = "white"))+ # títulos de los ejes en negrita
# crear facetas
facet_wrap(
~age_cat, # cada gráfico es un valor de age_cat
ncol = 4, # número de columnas
strip.position = "top", # posición del título/tira de la faceta
labeller = my_labels)+ # etiquetado definido arriba
# Etiquetas
labs(
title = "Weekly incidence of cases, by age category",
subtitle = "Subtitle",
fill = "Age category", # proporciona un nuevo título para la leyenda
x = "Week of symptom onset",
y = "Weekly incident cases reported",
caption = stringr::str_glue("n = {nrow(central_data)} from Central Hospital; Case onsets range from {format(min(central_data$date_onset, na.rm=T), format = '%a %d %b %Y')} to {format(max(central_data$date_onset, na.rm=T), format = '%a %d %b %Y')}\n{nrow(central_data %>% filter(is.na(date_onset)))} cases missing date of onset and not shown"))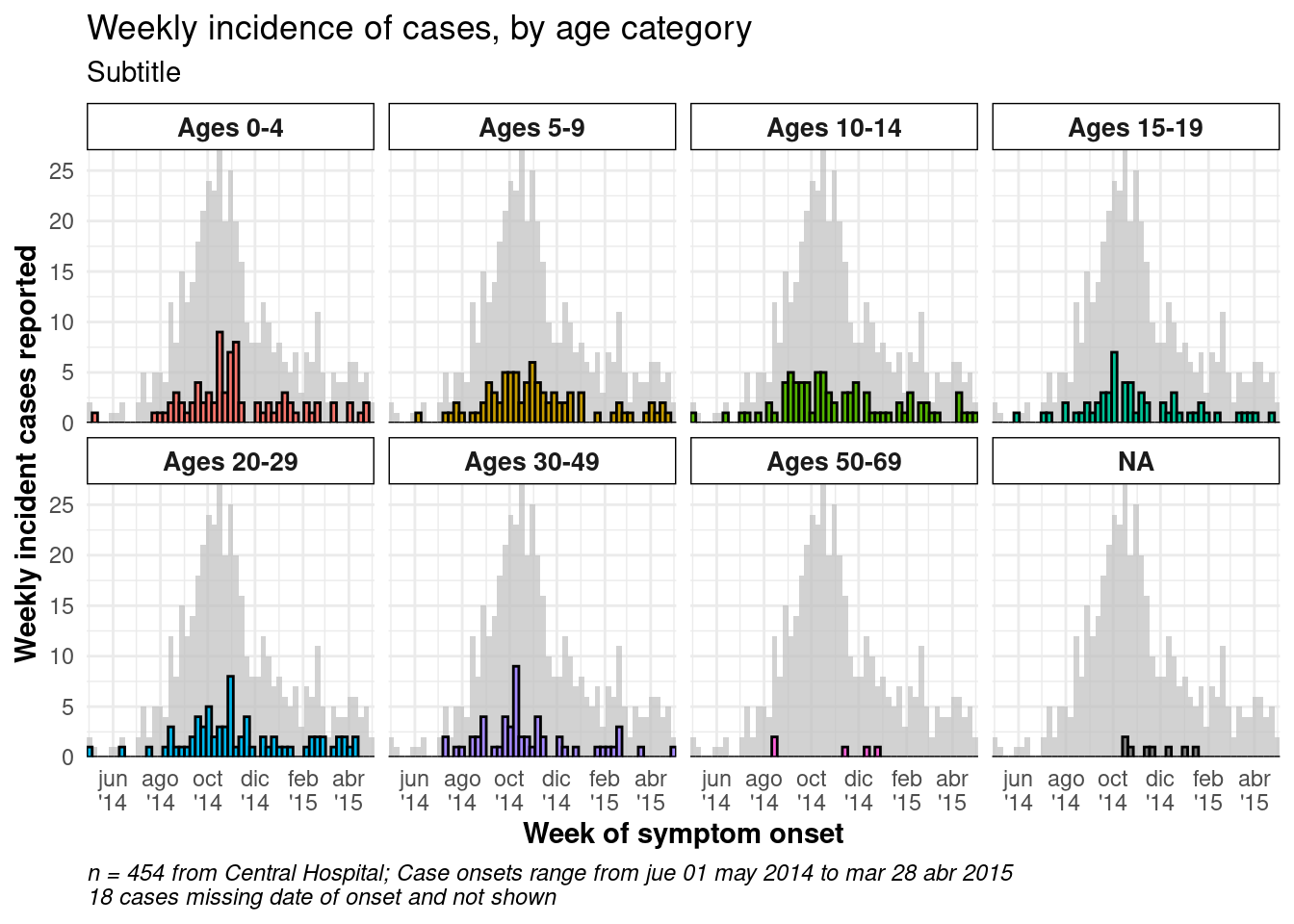
Una faceta con datos
Si quieres tener una caja de facetas que contenga todos los datos, duplica todo el conjunto de datos y trata los duplicados como un solo valor de facetas. Una función de “ayuda” CreateAllFacet() a continuación puede ayudar con esto (gracias a esta entrada del blog). Cuando se ejecuta, el número de filas se duplica y habrá una nueva columna llamada facet en la que las filas duplicadas tendrán el valor “all”, y las filas originales tienen el valor original de la columna facet. Ahora sólo tienes que hacer la faceta con la columna facet.
Aquí está la función de ayuda. Ejecútala para que esté disponible para ti.
# Definir la función de ayuda
CreateAllFacet <- function(df, col){
df$facet <- df[[col]]
temp <- df
temp$facet <- "all"
merged <-rbind(temp, df)
# asegura que el valor de la faceta es un factor
merged[[col]] <- as.factor(merged[[col]])
return(merged)
}Ahora aplica la función de ayuda a los datos, en la columna age_cat:
# Crea un conjunto de datos duplicado y con una nueva columna "facet" para mostrar "todas" las categorías de edad como otro nivel de faceta
central_data2 <- CreateAllFacet(central_data, col = "age_cat") %>%
# establecer niveles del factor
mutate(facet = fct_relevel(facet, "all", "0-4", "5-9",
"10-14", "15-19", "20-29",
"30-49", "50-69", "70+"))## Warning: 1 unknown level in `f`: 70+
# comprobar niveles
table(central_data2$facet, useNA = "always")##
## all 0-4 5-9 10-14 15-19 20-29 30-49 50-69 <NA>
## 454 84 84 82 58 73 57 7 9Los cambios más importantes en el comando ggplot() son:
- Los datos utilizados son ahora
central_data2(el doble de filas, con la nueva columna “facet”) - La etiquetadora tendrá que ser actualizada, si se utiliza
- Opcional: para conseguir facetas apiladas verticalmente: la columna de la faceta se mueve al lado de las filas de la ecuación y a la derecha se sustituye por “.” (
facet_wrap(facet\~.)), yncol = 1. También puede ser necesario ajustar la anchura y la altura de la imagen png guardada (verggsave()en Conceptos básicos de ggplot).
ggplot(central_data2) +
# curvas epidemiológicas actuales por grupo
geom_histogram(
mapping = aes(
x = date_onset,
group = age_cat,
fill = age_cat), # los argumentos dentro de aes() se aplican por grupo
color = "black", # los argumentos fuera de aes() se aplican a todos los datos
# histogram breaks
breaks = weekly_breaks_central)+ # vector de fechas predefinido (véase la parte superior de la sección ggplot)
# Etiquetas del eje-x
scale_x_date(
expand = c(0,0), # elimina el exceso de espacio en el eje-x debajo y después de las barras de casos
date_breaks = "2 months", # las etiquetas aparecen cada 2 meses
date_minor_breaks = "1 month", # las líneas verticales aparecen cada 1 mes
date_labels = "%b\n'%y")+ # formato de las etiquetas de fecha
# eje-y
scale_y_continuous(expand = c(0,0))+ # elimina el exceso de espacio en el eje-y entre la parte inferior de las barras y las etiquetas
# temas estéticos
theme_minimal()+ # un conjunto de temas para simplificar el gráfico
theme(
plot.caption = element_text(face = "italic", hjust = 0), # ajusta a la izquierda en cursiva
axis.title = element_text(face = "bold"),
legend.position = "bottom")+
# crear facetas
facet_wrap(facet~. , # cada gráfico es un valor de faceta
ncol = 1)+
# Etiquetas
labs(title = "Weekly incidence of cases, by age category",
subtitle = "Subtitle",
fill = "Age category", # proporcionar un nuevo título para la leyenda
x = "Week of symptom onset",
y = "Weekly incident cases reported",
caption = stringr::str_glue("n = {nrow(central_data)} from Central Hospital; Case onsets range from {format(min(central_data$date_onset, na.rm=T), format = '%a %d %b %Y')} to {format(max(central_data$date_onset, na.rm=T), format = '%a %d %b %Y')}\n{nrow(central_data %>% filter(is.na(date_onset)))} cases missing date of onset and not shown"))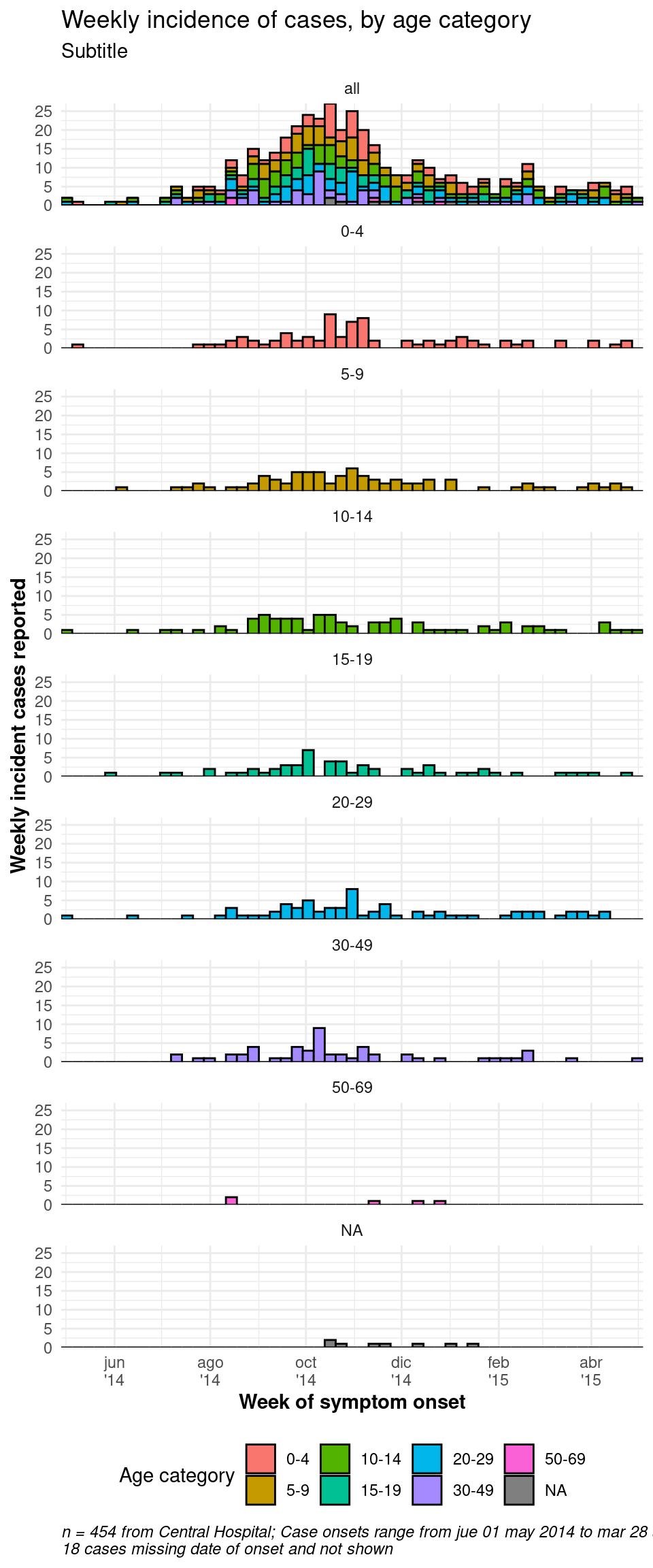
32.4 Datos provisionales
Los datos más recientes que se muestran en las curvas epidémicas deben marcarse a menudo como provisionales, o sujetos a retrasos en los informes. Esto puede hacerse añadiendo una línea vertical y/o un rectángulo sobre un número determinado de días. Aquí hay dos opciones:
- Utiliza
annotate():- Para una línea utiliza
annotate(geom = "segment"). Proporcionax,xend,y, eyend. Ajusta el tamaño, el tipo de línea (lty)y el color. - Para un rectángulo utiliza
annotate(geom = "rect"). Proporcionaxmin/xmax/ymin/ymax. Ajusta el color y el alpha.
- Para una línea utiliza
- Agrupar los datos por estado tentativo y colorear esas barras de forma diferente
PRECAUCIÓN: Puedes intentar geom_rect() para dibujar un rectángulo, pero el ajuste de la transparencia no funciona en un contexto de listado. Esta función superpone un rectángulo para cada observación/fila!. Utiliza un alfa muy bajo (por ejemplo, 0,01), u otro enfoque.
Uso de annotate()
- Dentro de
annotate(geom = "rect"), los argumentosxminyxmaxdeben tener entradas del tipo Date. - Ten en cuenta que, como estos datos se agregan en barras semanales, y la última barra se extiende hasta el lunes siguiente al último punto de datos, la región sombreada puede parecer que abarca 4 semanas
- Este es un ejemplo de
annotate()en línea
ggplot(central_data) +
# histograma
geom_histogram(
mapping = aes(x = date_onset),
breaks = weekly_breaks_central, # vector de fechas predefinido - véase la parte superior de la sección ggplot
color = "darkblue",
fill = "lightblue") +
# escalas
scale_y_continuous(expand = c(0,0))+
scale_x_date(
expand = c(0,0), # elimina el exceso de espacio del eje-x debajo y después de las barras de casos
date_breaks = "1 month", # 1º de mes
date_minor_breaks = "1 month", # 1º de mes
date_labels = "%b\n'%y")+ # formato de etiqueta
# etiquetas y tema
labs(
title = "Using annotate()\nRectangle and line showing that data from last 21-days are tentative",
x = "Week of symptom onset",
y = "Weekly case indicence")+
theme_minimal()+
# añadir un rectángulo rojo semitransparente a los datos provisionales
annotate(
"rect",
xmin = as.Date(max(central_data$date_onset, na.rm = T) - 21), # la nota debe estar incluida en as.Date()
xmax = as.Date(Inf), # la nota debe estar incluida en in as.Date()
ymin = 0,
ymax = Inf,
alpha = 0.2, # alpha fácil e intuitivo de ajustar usando annotate()
fill = "red")+
# añadir línea vertical negra sobre otras capas
annotate(
"segment",
x = max(central_data$date_onset, na.rm = T) - 21, # 21 días antes del último dato
xend = max(central_data$date_onset, na.rm = T) - 21,
y = 0, # la línea comienza en y = 0
yend = Inf, # línea hasta la parte superior del gráfico
size = 2, # tamaño de la línea
color = "black",
lty = "solid")+ # linetype e.g. "solid", "dashed" (sólida, rayada)
# añadir texto en el rectángulo
annotate(
"text",
x = max(central_data$date_onset, na.rm = T) - 15,
y = 15,
label = "Subject to reporting delays",
angle = 90)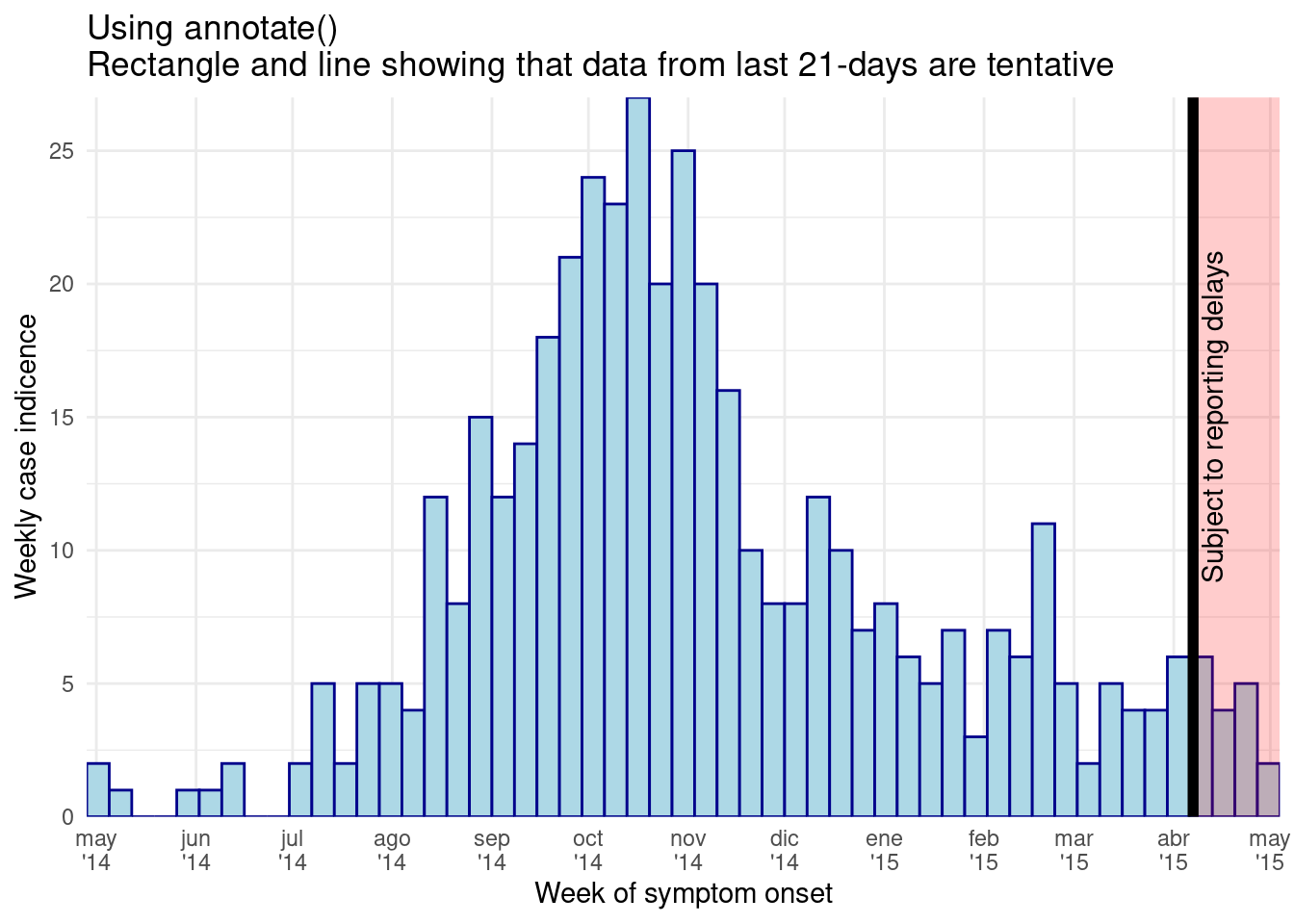
La misma línea vertical negra se puede conseguir con el código de abajo, pero usando geom_vline() se pierde la capacidad de controlar la altura:
geom_vline(xintercept = max(central_data$date_onset, na.rm = T) - 21,
size = 2,
color = "black")Color de las barras
Un enfoque alternativo podría ser ajustar el color o la visualización de las propias barras de datos tentativos. Podrías crear una nueva columna en la etapa de preparación de los datos y utilizarla para agrupar los datos, de manera que el aes(fill = ) de los datos tentativos pueda tener un color o un alfa diferente al de las otras barras.
# añadir columna
############
plot_data <- central_data %>%
mutate(tentative = case_when(
date_onset >= max(date_onset, na.rm=T) - 7 ~ "Tentative", # provisional si es en los últimos 7 días
TRUE ~ "Reliable")) # todos los demás se considerarán definitivos
# Gráfico
######
ggplot(plot_data, aes(x = date_onset, fill = tentative)) +
# histograma
geom_histogram(
breaks = weekly_breaks_central, # vector de datos predefinido, véase la parte superior de la página ggplot
color = "black") +
# escalas
scale_y_continuous(expand = c(0,0))+
scale_fill_manual(values = c("lightblue", "grey"))+
scale_x_date(
expand = c(0,0), # elimina el exceso de espacio en el eje-x debajo y después de las barras de casose bars
date_breaks = "3 weeks", # lunes cada 3 semanas
date_minor_breaks = "week", # semanas de lunes
date_labels = "%d\n%b\n'%y")+ # formato de etiqueta
# etiquetas y tema
labs(title = "Show days that are tentative reporting",
subtitle = "")+
theme_minimal()+
theme(legend.title = element_blank()) # elimina el título de la leyenda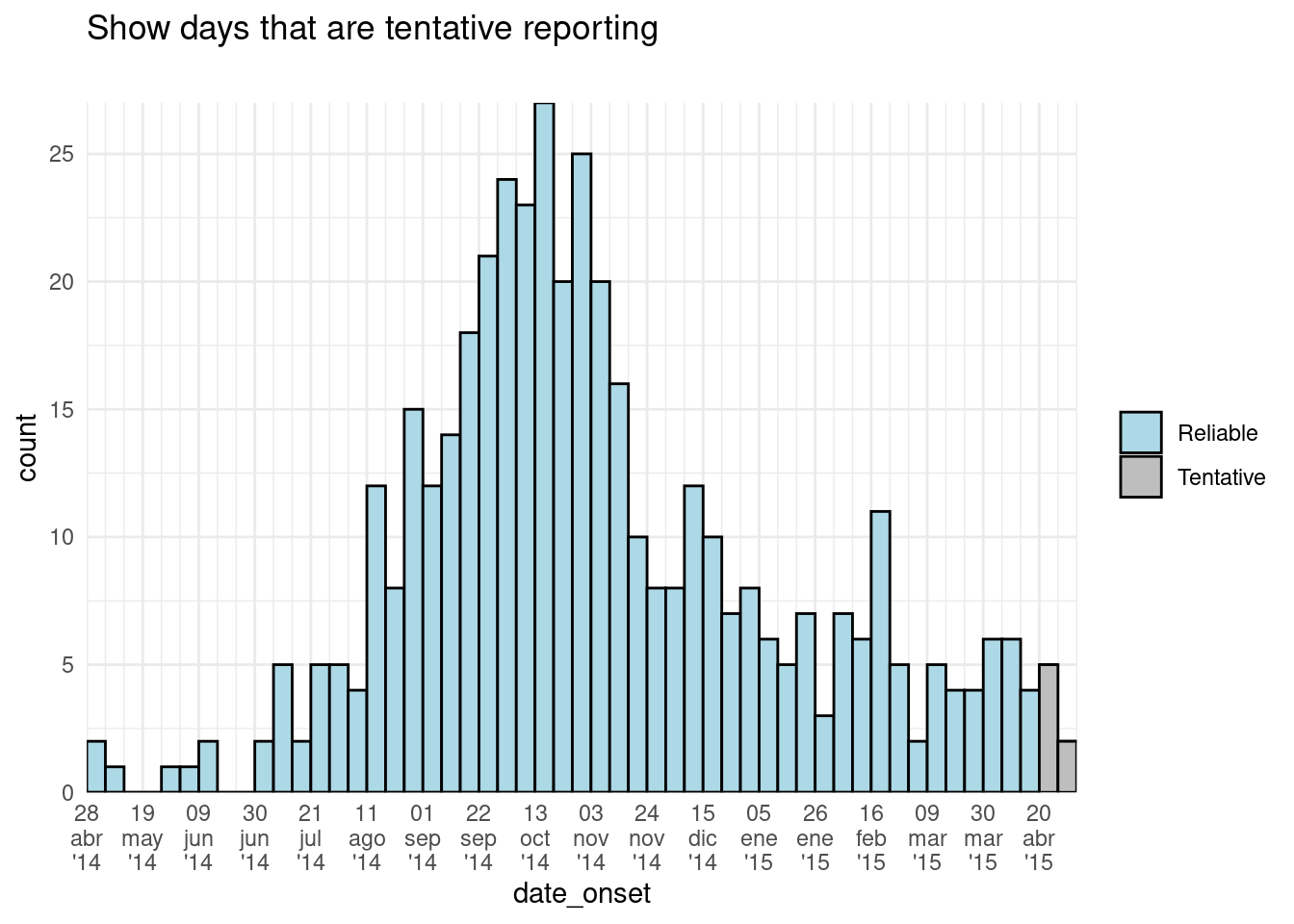
32.5 Etiquetas de fecha de varios niveles
Si deseas etiquetas de fecha de varios niveles (por ejemplo, mes y año) sin duplicar los niveles de etiquetas inferiores, considera uno de los enfoques siguientes:
Recuerda - puedes utilizar herramientas como \n dentro de los argumentos date_labels o labels para poner partes de cada etiqueta en una nueva línea inferior. Sin embargo, el código de abajo le ayuda a usar años o meses (por ejemplo) en una línea inferior y sólo una vez. Algunas notas sobre el código de abajo:
- Los recuentos de casos se agregan en semanas por motivos estéticos. Véase la página de Epicurves (sección de datos agregados) para más detalles.
- Se utiliza una línea
geom_area()en lugar de un histograma, ya que el enfoque de facetas que se presenta a continuación no funciona bien con los histogramas.
Agregar a los recuentos semanales
# Crear conjunto de datos de recuentos de casos por semana
##########################################################
central_weekly <- linelist %>%
filter(hospital == "Central Hospital") %>% # filtra linelist
mutate(week = lubridate::floor_date(date_onset, unit = "weeks")) %>%
count(week) %>% # resume el recuento semanal de casos
drop_na(week) %>% # elimina los casos en los que falta onset_date
complete( # completa todas las semanas sin casos notificados
week = seq.Date(
from = min(week),
to = max(week),
by = "week"),
fill = list(n = 0)) # convertir los nuevos valores NA en 0 casosHacer gráficos
# gráfico con borde en la caja del año
######################################
ggplot(central_weekly) +
geom_area(aes(x = week, y = n), # crea una línea, especifica x e y
stat = "identity") + # porque la altura de la línea es el número de casos
scale_x_date(date_labels="%b", # formato de etiqueta de fecha muestra el mes
date_breaks="month", # etiquetas de fecha el día 1 de cada mes
expand=c(0,0)) + # elimina el espacio sobrante en cada extremoach end
scale_y_continuous(
expand = c(0,0))+ # elimina el espacio sobrante debajo del eje-x
facet_grid(~lubridate::year(week), # faceta sobre el año (de la columna de clase Date)
space="free_x",
scales="free_x", # los ejes-x se adaptan al rango de datos (no son "fijos")
switch="x") + # etiquetas de facetas (año) en la parte inferior
theme_bw() +
theme(strip.placement = "outside", # colocación de las etiquetas de las facetas
strip.background = element_rect(fill = NA, # las etiquetas de facetas sin relleno con borde gris
colour = "grey50"),
panel.spacing = unit(0, "cm"))+ # sin espacio entre paneles de facetas
labs(title = "Nested year labels, grey label border")
# gráfico sin borde en la caja del año
#######################################
ggplot(central_weekly,
aes(x = week, y = n)) + # establecer x e y para todo el gráfico
geom_line(stat = "identity", # crear línea, la altura de la línea es el número de casos
color = "#69b3a2") + # color de la línea
geom_point(size=1, color="#69b3a2") + # crear puntos en los datos semanales
geom_area(fill = "#69b3a2", # relleno del área bajo la línea
alpha = 0.4)+ # relleno transparente
scale_x_date(date_labels="%b", # formato de etiqueta de fecha mostrar mes
date_breaks="month", # etiquetas de fecha el día 1 de cada mes
expand=c(0,0)) + # elimina el espacio sobrante
scale_y_continuous(
expand = c(0,0))+ # elimina el espacio sobrante bajo el eje-x
facet_grid(~lubridate::year(week), # faceta sobre el año (de la columna de clase Date)
space="free_x",
scales="free_x", # los ejes-x se adaptan al rango de datos (no son "fijos")
switch="x") + # etiquetas de facetas (año) en la parte inferior
theme_bw() +
theme(strip.placement = "outside", # colocación de etiqueta de faceta
strip.background = element_blank(), # sin fondo de etiqueta de faceta
panel.grid.minor.x = element_blank(),
panel.border = element_rect(colour="grey40"), # borde gris de la faceta PANEL
panel.spacing=unit(0,"cm"))+ # sin espacio entre paneles de facetas
labs(title = "Nested year labels - points, shaded, no label border")
Las técnicas anteriores fueron adaptadas de este y este post en stackoverflow.com.
32.6 Doble eje
Aunque hay fuertes discusiones sobre la validez de los ejes duales dentro de la comunidad de visualización de datos, muchos supervisores de epi todavía quieren ver una curva epidémica o un gráfico similar con un porcentaje superpuesto con un segundo eje. Esto se discute más ampliamente en la página Consejos de ggplot, pero a continuación se muestra un ejemplo utilizando el método cowplot:
- Se hacen dos gráficos distintos y luego se combinan con el paquete cowplot.
- Los gráficos deben tener exactamente el mismo eje x (límites establecidos) o de lo contrario los datos y las etiquetas no se alinearán
- Cada uno de ellos utiliza
theme_cowplot()y uno de ellos tiene el eje-y desplazado a la derecha del gráfico
# cargar paquete
pacman::p_load(cowplot)
# Crea el primer gráfico, el histograma de la curvaepi
######################################################
plot_cases <- linelist %>%
# representa casos por semana
ggplot()+
# crea el histograma
geom_histogram(
mapping = aes(x = date_onset),
# intervalos semanales desde el lunes anterior al primer caso hasta el lunes posterior al último caso
breaks = weekly_breaks_all)+ # vector predefinido de fechas semanales (véase la parte superior de la sección ggplot)
# especifica el principio y el final del eje de fechas para alinearlo con otros gráficos
scale_x_date(
limits = c(min(weekly_breaks_all), max(weekly_breaks_all)))+ # min/max de las rupturas predefinidas semanales del histograma
# etiquetas
labs(
y = "Daily cases",
x = "Date of symptom onset"
)+
theme_cowplot()
# crea un segundo gráfico con el porcentaje de fallecidos por semana
####################################################################
plot_deaths <- linelist %>% # comienza con linelist
group_by(week = floor_date(date_onset, "week")) %>% # crea la columna semana
# resume para obtener el porcentaje semanal de casos que fallecieron
summarise(n_cases = n(),
died = sum(outcome == "Death", na.rm=T),
pct_died = 100*died/n_cases) %>%
# comienza el gráfico
ggplot()+
# línea de porcentaje semanal de fallecidos
geom_line( # crea la línea del porcentaje de fallecidos
mapping = aes(x = week, y = pct_died), # especifica la altura-y como columna pct_died
stat = "identity", # establece la altura de la línea al valor de la columna pct_dead, no al número de filas (que es por defecto)
size = 2,
color = "black")+
# Mismos límites del eje-fecha que el otro gráfico - alineación perfecta
scale_x_date(
limits = c(min(weekly_breaks_all), max(weekly_breaks_all)))+ # min/max de los cortes semanales predefinidos del histograma
# ajustes del eje-y
scale_y_continuous( # ajusta el eje-y
breaks = seq(0,100, 10), # establece los intervalos de ruptura del eje porcentual
limits = c(0, 100), # establece la extensión del eje porcentual
position = "right")+ # mueve el eje porcentual a la derecha
# etiqueta del eje-Y, sin etiqueta del eje-x
labs(x = "",
y = "Percent deceased")+ # etiqueta del eje de porcentajes
theme_cowplot() # añade esto para que los dos gráficos se fusionen bienAhora utiliza cowplot para superponer los dos gráficos. Se ha prestado atención a la alineación del eje-x, al lado del eje-y y al uso de theme_cowplot().
aligned_plots <- cowplot::align_plots(plot_cases, plot_deaths, align="hv", axis="tblr")
ggdraw(aligned_plots[[1]]) + draw_plot(aligned_plots[[2]])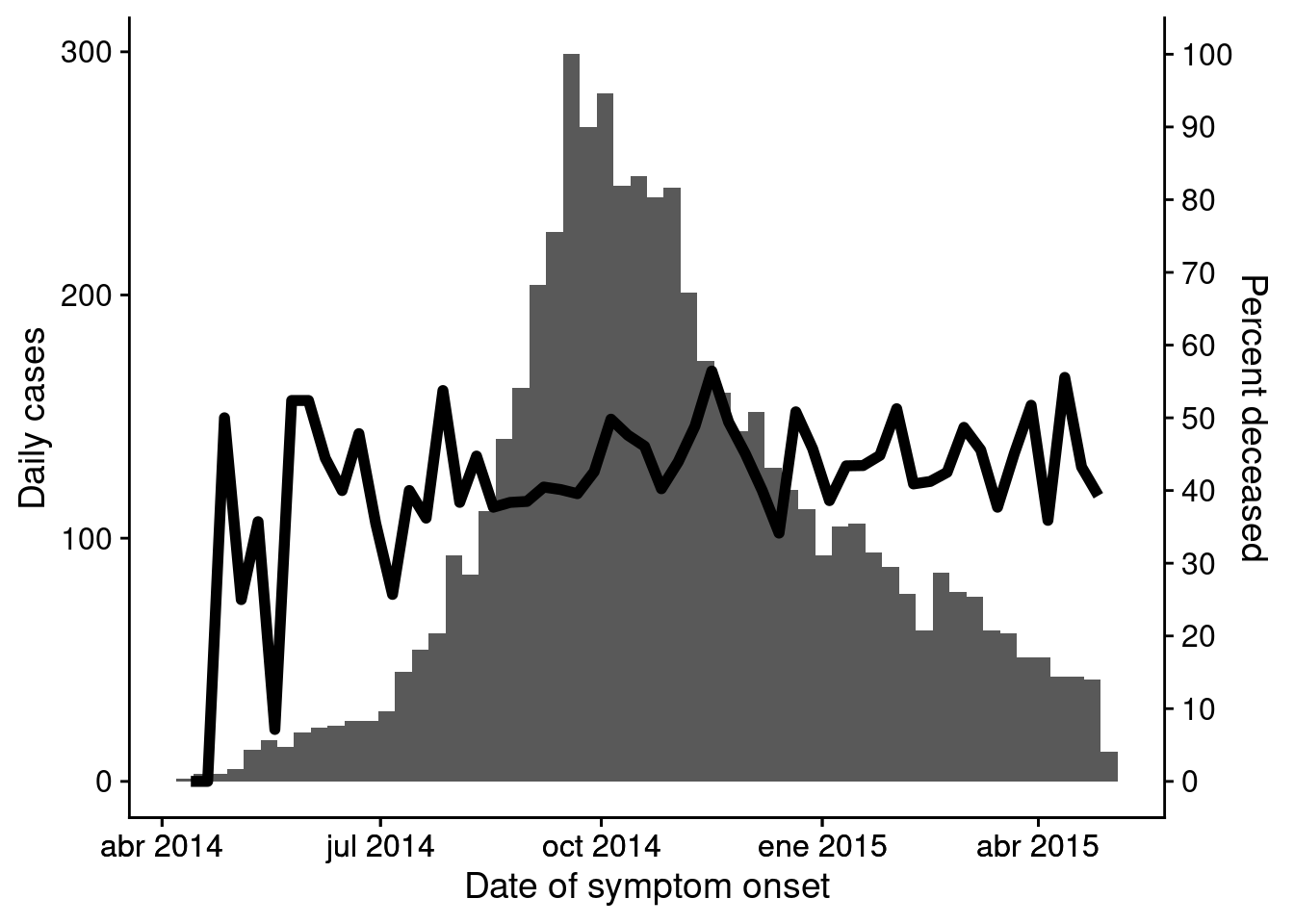
32.7 Incidencia acumulada
Nota: Si utilizas incidence2, consulta la sección sobre cómo puede producirse la incidencia acumulada con una función simple. Esta página abordará cómo calcular la incidencia acumulada y dibujarla con ggplot().
Si se empieza con una lista de casos, crea una nueva columna que contenga el número acumulado de casos por día en un brote utilizando cumsum() de R base:
cumulative_case_counts <- linelist %>%
count(date_onset) %>% # recuento de filas por día (devuelto en la columna "n")
mutate(
cumulative_cases = cumsum(n) # nueva columna del número acumulado de filas en cada fecha
)A continuación se muestran las 10 primeras filas:
Esta columna acumulativa puede entonces ser dibujada contra date_onset, usando geom_line():
plot_cumulative <- ggplot()+
geom_line(
data = cumulative_case_counts,
aes(x = date_onset, y = cumulative_cases),
size = 2,
color = "blue")
plot_cumulative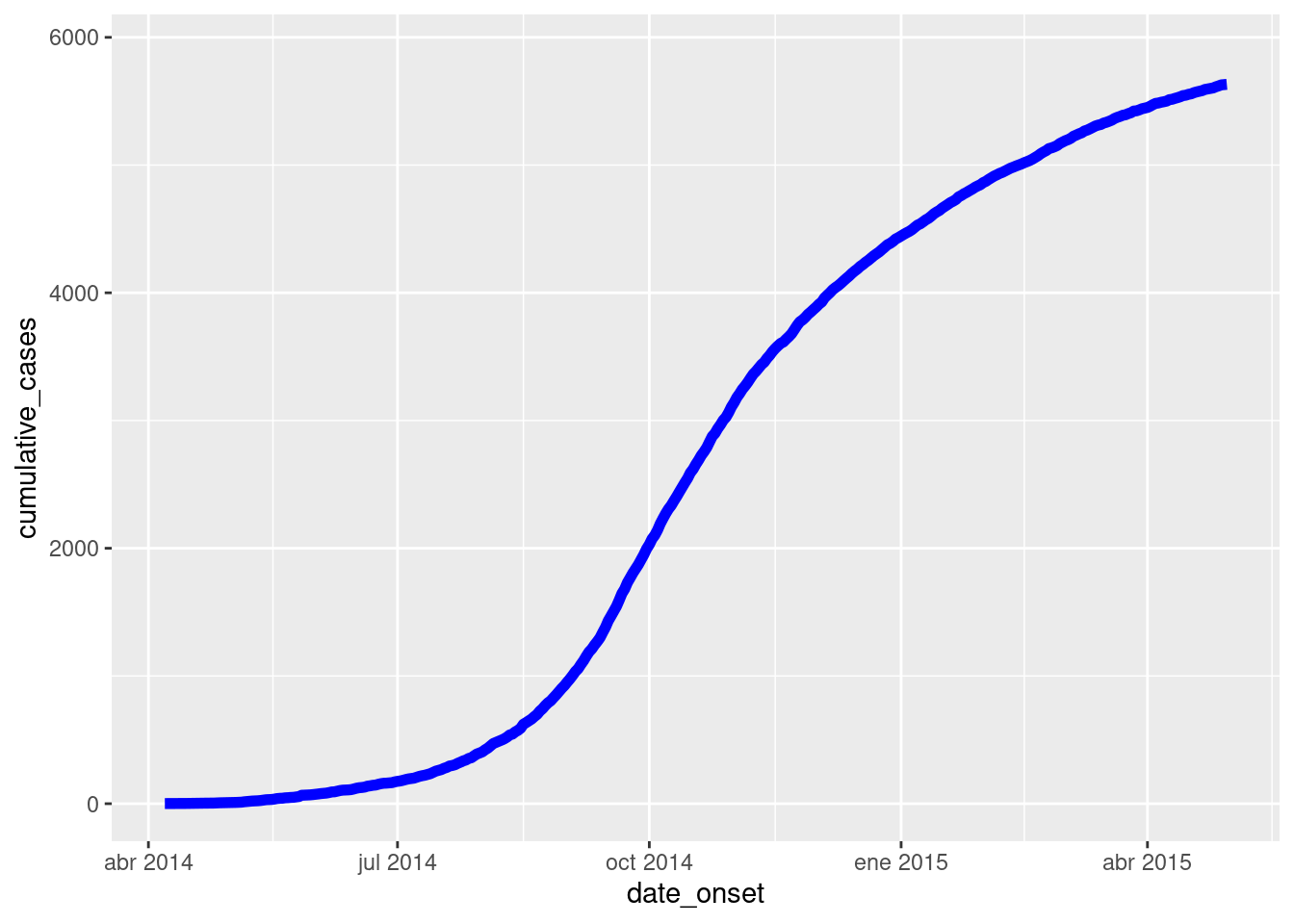
También se puede superponer a la curva epidémica, con doble eje utilizando el método cowplot descrito anteriormente y en la página Consejos de ggplot:
# cargar paquete
pacman::p_load(cowplot)
# Crea el primer gráfico, el histograma de la curvaepi
plot_cases <- ggplot()+
geom_histogram(
data = linelist,
aes(x = date_onset),
binwidth = 1)+
labs(
y = "Daily cases",
x = "Date of symptom onset"
)+
theme_cowplot()
# crea un segundo gráfico de la línea de casos acumulados
plot_cumulative <- ggplot()+
geom_line(
data = cumulative_case_counts,
aes(x = date_onset, y = cumulative_cases),
size = 2,
color = "blue")+
scale_y_continuous(
position = "right")+
labs(x = "",
y = "Cumulative cases")+
theme_cowplot()+
theme(
axis.line.x = element_blank(),
axis.text.x = element_blank(),
axis.title.x = element_blank(),
axis.ticks = element_blank())Ahora utiliza cowplot para superponer los dos gráficos. Se ha prestado atención a la alineación del eje-x, al lado del eje-y y al uso de theme_cowplot().
aligned_plots <- cowplot::align_plots(plot_cases, plot_cumulative, align="hv", axis="tblr")
ggdraw(aligned_plots[[1]]) + draw_plot(aligned_plots[[2]])