37 Cadenas de transmisión
37.1 Resumen
La principal herramienta para manejar, analizar y visualizar las cadenas de transmisión y los datos de rastreo de contactos es el paquete epicontacts, desarrollado por la gente de RECON. Prueba con el gráfico interactivo que se muestra a continuación pasando el cursor por encima de los nodos para obtener más información, arrastrándolos para moverlos y clicando sobre ellos para resaltar los casos posteriores.
37.2 Preparación
Cargar paquetes
Primero carga los paquetes estándar necesarios para la importación y manipulación de datos. En este manual destacamos p_load() de pacman, que instala el paquete si es necesario y lo carga para su uso. También se pueden cargar paquetes con library() desde R base. Consulta la página Fundamentos de R para obtener más información sobre los paquetes de R.
pacman::p_load(
rio, # Importación de ficheros
here, # Localizador de ficheros
tidyverse, # Gestión de datos + gráficos ggplot2
remotes # Instalación de paquetes desde github
)Necesitarás la versión de desarrollo de epicontacts, que puede instalarse desde github utilizando la función p_install_github() de pacman. Sólo necesitas ejecutar este comando una vez, no cada vez que utilizas el paquete (a partir de entonces, puedes utilizar sólo p_load()).
pacman::p_install_gh("reconhub/epicontacts@timeline")Importar datos
Importamos el conjunto de datos de casos de una epidemia de ébola simulada. Si deseas descargar los datos para seguirlos paso a paso, consulta las instrucciones en la página de descargando el manual y los datos. El conjunto de datos se importa utilizando la función import() del paquete rio. Consulta la página sobre importación y exportación para conocer las distintas formas de importar datos.
# importar linelist
linelist <- import("linelist_cleaned.xlsx")A continuación se muestran las primeras 50 filas del listado. Son especialmente interesantes las columnas case_id, generation, infector, y source.
Creación de un objeto epicontacts
A continuación, tenemos que crear un objeto epicontacts, que requiere dos tipos de datos:
- un listado de casos en los que las columnas son variables y las filas corresponden a casos únicos
- una lista de bordes que definen los vínculos entre los casos sobre la base de sus identificadores únicos (pueden ser contactos, eventos de transmisión, etc.)
Como ya tenemos un listado, sólo tenemos que crear una lista de aristas entre los casos, más concretamente entre sus ID. Podemos extraer los enlaces de transmisión del listado vinculando la columna infector con la columna case_id. En este punto también podemos añadir “propiedades de borde”, con lo que nos referimos a cualquier variable que describa el vínculo entre los dos casos, no los casos en sí. Por ejemplo, añadiremos una variable location que describa la ubicación del evento de transmisión, y una variable de duración que describa la duración del contacto en días.
En el código siguiente, la función transmute() de dplyr es similar a mutate, excepto que sólo mantiene las columnas que hemos especificado dentro de la función. La función drop_na filtrará cualquier fila en la que las columnas especificadas tengan un valor NA; en este caso, sólo queremos mantener las filas en las que se conoce el infector.
## generar contactos
contacts <- linelist %>%
transmute(
infector = infector,
case_id = case_id,
location = sample(c("Community", "Nosocomial"), n(), TRUE),
duration = sample.int(10, n(), TRUE)
) %>%
drop_na(infector)Ahora podemos crear el objeto epicontacts utilizando la función make_epicontacts. Necesitamos especificar qué columna del listado apunta al identificador único del caso, así como qué columnas de los contactos apuntan a los identificadores únicos de los casos involucrados en cada enlace. Estos enlaces son direccionales en el sentido de que la infección va del infector al caso, por lo que necesitamos especificar los argumentos from y to. Por lo tanto, también establecemos el argumento directed a TRUE, que afectará a las operaciones futuras.
## generar el objeto epicontactos
epic <- make_epicontacts(
linelist = linelist,
contacts = contacts,
id = "case_id",
from = "infector",
to = "case_id",
directed = TRUE
)Al examinar los objetos epicontacts, podemos ver que la columna case_id del listado ha sido renombrada a id y las columnas case_id e infector de los contactos han sido renombradas a from y to. Esto garantiza la coherencia en las operaciones posteriores de manipulación, visualización y análisis.
## ver el objeto epicontactos
epic##
## /// Epidemiological Contacts //
##
## // class: epicontacts
## // 5,888 cases in linelist; 3,800 contacts; directed
##
## // linelist
##
## # A tibble: 5,888 × 30
## id generation date_inf…¹ date_onset date_hos…² date_out…³ outcome gender age age_u…⁴ age_y…⁵ age_cat age_c…⁶ hospi…⁷ lon lat infec…⁸ source wt_kg ht_cm ct_bl…⁹ fever chills cough aches vomit temp time_…˟
## <chr> <dbl> <date> <date> <date> <date> <chr> <chr> <dbl> <chr> <dbl> <fct> <fct> <chr> <dbl> <dbl> <chr> <chr> <dbl> <dbl> <dbl> <chr> <chr> <chr> <chr> <chr> <dbl> <chr>
## 1 5fe599 4 2014-05-08 2014-05-13 2014-05-15 NA <NA> m 2 years 2 0-4 0-4 Other -13.2 8.47 f547d6 other 27 48 22 no no yes no yes 36.8 <NA>
## 2 8689b7 4 NA 2014-05-13 2014-05-14 2014-05-18 Recover f 3 years 3 0-4 0-4 Missing -13.2 8.45 <NA> <NA> 25 59 22 <NA> <NA> <NA> <NA> <NA> 36.9 09:36
## 3 11f8ea 2 NA 2014-05-16 2014-05-18 2014-05-30 Recover m 56 years 56 50-69 55-59 St. Ma… -13.2 8.46 <NA> <NA> 91 238 21 <NA> <NA> <NA> <NA> <NA> 36.9 16:48
## 4 b8812a 3 2014-05-04 2014-05-18 2014-05-20 NA <NA> f 18 years 18 15-19 15-19 Port H… -13.2 8.48 f90f5f other 41 135 23 no no no no no 36.8 11:22
## 5 893f25 3 2014-05-18 2014-05-21 2014-05-22 2014-05-29 Recover m 3 years 3 0-4 0-4 Milita… -13.2 8.46 11f8ea other 36 71 23 no no yes no yes 36.9 12:60
## 6 be99c8 3 2014-05-03 2014-05-22 2014-05-23 2014-05-24 Recover f 16 years 16 15-19 15-19 Port H… -13.2 8.46 aec8ec other 56 116 21 no no yes no yes 37.6 14:13
## 7 07e3e8 4 2014-05-22 2014-05-27 2014-05-29 2014-06-01 Recover f 16 years 16 15-19 15-19 Missing -13.2 8.46 893f25 other 47 87 21 <NA> <NA> <NA> <NA> <NA> 37.3 14:33
## 8 369449 4 2014-05-28 2014-06-02 2014-06-03 2014-06-07 Death f 0 years 0 0-4 0-4 Missing -13.2 8.46 133ee7 other 0 11 22 no no yes no yes 37 09:25
## 9 f393b4 4 NA 2014-06-05 2014-06-06 2014-06-18 Recover m 61 years 61 50-69 60-64 Missing -13.2 8.46 <NA> <NA> 86 226 22 no no yes no yes 36.4 11:16
## 10 1389ca 4 NA 2014-06-05 2014-06-07 2014-06-09 Death f 27 years 27 20-29 25-29 Missing -13.3 8.47 <NA> <NA> 69 174 22 no no yes no no 35.9 10:55
## # … with 5,878 more rows, 2 more variables: bmi <dbl>, days_onset_hosp <dbl>, and abbreviated variable names ¹date_infection, ²date_hospitalisation, ³date_outcome, ⁴age_unit, ⁵age_years, ⁶age_cat5, ⁷hospital,
## # ⁸infector, ⁹ct_blood, ˟time_admission
##
## // contacts
##
## # A tibble: 3,800 × 4
## from to location duration
## <chr> <chr> <chr> <int>
## 1 f547d6 5fe599 Nosocomial 10
## 2 f90f5f b8812a Community 10
## 3 11f8ea 893f25 Nosocomial 10
## 4 aec8ec be99c8 Nosocomial 3
## 5 893f25 07e3e8 Nosocomial 2
## 6 133ee7 369449 Community 5
## 7 996f3a 2978ac Nosocomial 1
## 8 133ee7 57a565 Nosocomial 1
## 9 37a6f6 fc15ef Community 1
## 10 9f6884 2eaa9a Community 2
## # … with 3,790 more rows37.3 Manipulación
Subconjunto
El método subset() para los objetos epicontacts permite, entre otras cosas, filtrar las redes en función de las propiedades del listado (“atributos de nodos”) y de la base de datos de contactos (“atributos de aristas”). Estos valores deben pasarse como listas con nombre al argumento respectivo. Por ejemplo, en el código que sigue mantenemos en linelist sólo los casos masculinos que tienen una fecha de infección entre abril y julio de 2014 (las fechas se especifican como rangos), y los enlaces de transmisión que se produjeron en el hospital.
sub_attributes <- subset(
epic,
node_attribute = list(
gender = "m",
date_infection = as.Date(c("2014-04-01", "2014-07-01"))
),
edge_attribute = list(location = "Nosocomial")
)
sub_attributes##
## /// Epidemiological Contacts //
##
## // class: epicontacts
## // 69 cases in linelist; 1,898 contacts; directed
##
## // linelist
##
## # A tibble: 69 × 30
## id generation date_inf…¹ date_onset date_hos…² date_out…³ outcome gender age age_u…⁴ age_y…⁵ age_cat age_c…⁶ hospi…⁷ lon lat infec…⁸ source wt_kg ht_cm ct_bl…⁹ fever chills cough aches vomit temp time_…˟
## <chr> <dbl> <date> <date> <date> <date> <chr> <chr> <dbl> <chr> <dbl> <fct> <fct> <chr> <dbl> <dbl> <chr> <chr> <dbl> <dbl> <dbl> <chr> <chr> <chr> <chr> <chr> <dbl> <chr>
## 1 5fe599 4 2014-05-08 2014-05-13 2014-05-15 NA <NA> m 2 years 2 0-4 0-4 Other -13.2 8.47 f547d6 other 27 48 22 no no yes no yes 36.8 <NA>
## 2 893f25 3 2014-05-18 2014-05-21 2014-05-22 2014-05-29 Recover m 3 years 3 0-4 0-4 Milita… -13.2 8.46 11f8ea other 36 71 23 no no yes no yes 36.9 12:60
## 3 2978ac 4 2014-05-30 2014-06-06 2014-06-08 2014-06-15 Death m 12 years 12 10-14 10-14 Port H… -13.2 8.48 996f3a other 67 112 22 no no yes no yes 36.5 16:03
## 4 57a565 4 2014-05-28 2014-06-13 2014-06-15 NA Death m 42 years 42 30-49 40-44 Milita… -13.3 8.46 133ee7 other 84 186 22 no no yes no no 36.9 11:14
## 5 fc15ef 6 2014-06-14 2014-06-16 2014-06-17 2014-07-09 Recover m 19 years 19 15-19 15-19 Missing -13.2 8.48 37a6f6 other 68 174 22 no no yes no no 36.5 12:42
## 6 99e8fa 7 2014-06-24 2014-06-28 2014-06-29 2014-07-09 Recover m 19 years 19 15-19 15-19 Port H… -13.2 8.47 ab634e other 71 150 21 no no yes no yes 37 12:43
## 7 f327be 6 2014-06-14 2014-07-12 2014-07-13 2014-07-14 Death m 31 years 31 30-49 30-34 St. Ma… -13.2 8.46 a15e13 other 68 188 24 no no yes no no 37.6 16:17
## 8 90e5fe 5 2014-06-18 2014-07-13 2014-07-14 2014-07-16 <NA> m 67 years 67 50-69 65-69 Port H… -13.3 8.46 ea3740 other 100 233 20 <NA> <NA> <NA> <NA> <NA> 36.6 17:45
## 9 a47529 5 2014-06-13 2014-07-17 2014-07-18 2014-07-26 Death m 45 years 45 30-49 45-49 Milita… -13.2 8.48 a2086d other 72 214 21 no no yes no yes 37.5 11:36
## 10 da8ecb 5 2014-06-20 2014-07-18 2014-07-20 2014-08-01 <NA> m 12 years 12 10-14 10-14 Missing -13.2 8.48 eb2277 funer… 69 157 21 <NA> <NA> <NA> <NA> <NA> 36.9 16:27
## # … with 59 more rows, 2 more variables: bmi <dbl>, days_onset_hosp <dbl>, and abbreviated variable names ¹date_infection, ²date_hospitalisation, ³date_outcome, ⁴age_unit, ⁵age_years, ⁶age_cat5, ⁷hospital, ⁸infector,
## # ⁹ct_blood, ˟time_admission
##
## // contacts
##
## # A tibble: 1,898 × 4
## from to location duration
## <chr> <chr> <chr> <int>
## 1 f547d6 5fe599 Nosocomial 10
## 2 11f8ea 893f25 Nosocomial 10
## 3 aec8ec be99c8 Nosocomial 3
## 4 893f25 07e3e8 Nosocomial 2
## 5 996f3a 2978ac Nosocomial 1
## 6 133ee7 57a565 Nosocomial 1
## 7 4802b1 bbfa93 Nosocomial 8
## 8 a75c7f 7f5a01 Nosocomial 3
## 9 b799eb bc2adf Nosocomial 7
## 10 a15e13 f327be Nosocomial 5
## # … with 1,888 more rowsPodemos utilizar la función thin para filtrar linelist para incluir los casos que se encuentran en los contactos estableciendo el argumento what = "linelist", o filtrar los contactos para incluir los casos que se encuentran en linelist estableciendo el argumento what = "contacts". En el código siguiente, filtramos aún más el objeto epicontactos para mantener sólo los enlaces de transmisión que implican los casos masculinos infectados entre abril y julio que habíamos filtrado anteriormente. Podemos ver que sólo dos enlaces de transmisión conocidos se ajustan a esa especificación.
sub_attributes <- thin(sub_attributes, what = "contacts")
nrow(sub_attributes$contacts)## [1] 2Además de la subdivisión por atributos de nodos y aristas, las redes pueden podarse para incluir sólo los componentes que están conectados a ciertos nodos. El argumento cluster_id toma un vector de IDs de casos y devuelve linelist de individuos que están vinculados, directa o indirectamente, a esos IDs. En el código siguiente, podemos ver que un total de 13 casos del listado están involucrados en los clusters que contienen 2ae019 y 71577a.
## [1] 13El método subset() para los objetos epicontacts también permite filtrar por tamaño de cluster usando los argumentos cs, cs_min y cs_max. En el código siguiente, estamos manteniendo sólo los casos vinculados a clusters de 10 casos o más, y podemos ver que 271 casos del listado están involucrados en tales clusters.
## [1] 271Acceso a los IDs
La función get_id() recupera información sobre los ID de los casos en el conjunto de datos, y puede parametrizarse como sigue:
- linelist: IDs en los datos del listado
- contacts: IDs en el conjunto de datos de los contactos (“desde” y “hasta” combinados)
- from: IDs en la columna “from” de los datos del contacto
- to los identificadores de la columna “a” de los datos de los contactos
- all: Las identificaciones que aparecen en cualquier parte de cualquiera de los conjuntos de datos
- common: identificaciones que aparecen tanto en el conjunto de datos de contactos como en linelist
Por ejemplo, ¿cuáles son las diez primeras identificaciones de los datos de contactos?
contacts_ids <- get_id(epic, "contacts")
head(contacts_ids, n = 10)## [1] "f547d6" "f90f5f" "11f8ea" "aec8ec" "893f25" "133ee7" "996f3a" "37a6f6" "9f6884" "4802b1"¿Cuántas identificaciones se encuentran tanto en linelist como en los contactos?
length(get_id(epic, "common"))## [1] 435237.4 Visualización
Representación básica
Todas las visualizaciones de los objetos epicontacts son manejadas por la función plot. En primer lugar, filtraremos el objeto epicontacts para incluir solo los casos con fechas de inicio en junio de 2014 utilizando la función de subconjunto, y solo incluiremos los contactos vinculados a esos casos utilizando la función thin.
## subconjunto objeto epicontactos
sub <- epic %>%
subset(
node_attribute = list(date_onset = c(as.Date(c("2014-06-30", "2014-06-01"))))
) %>%
thin("contacts")A continuación, podemos crear el gráfico básico e interactivo de la siguiente manera:
## dibuja el objeto epicontactos
plot(
sub,
width = 700,
height = 700
)Puedes mover los nodos arrastrándolos, pasar por encima de ellos para obtener más información y clicar sobre ellos para resaltar los casos conectados.
Hay un gran número de argumentos para modificar este gráfico. Aquí cubriremos los principales, pero consulta la documentación a través de ?vis_epicontacts (la función a la que se llama cuando se utiliza plot en un objeto epicontacts) para obtener una descripción completa de los argumentos de la función.
Visualizar los atributos de los nodos
El color, la forma y el tamaño del nodo se pueden asignar a una columna determinada en linelist utilizando los argumentos node_color, node_shape y node_size. Esto es similar a la sintaxis aes que puede reconocer ggplot2.
Los colores, formas y tamaños específicos de los nodos pueden especificarse de la siguiente manera:
Colores a través del argumento
col_pal, ya sea proporcionando una lista de nombres para la especificación manual de cada color como se hace a continuación, o proporcionando una función de paleta de colores comocolorRampPalette(c("black", "red", "orange")), que proporcionaría un gradiente de colores entre los especificados.Formas pasando una lista con nombre al argumento
shapes, especificando una forma para cada elemento único en la columna del listado especificada por el argumentonode_shape. Ver encodeawesomelas formas disponibles.Tamaño pasando un rango de tamaño de los nodos al argumento
size_range.
Aquí un ejemplo, donde el color representa el resultado, la forma el género y el tamaño la edad:
Visualizar los atributos de los bordes
El color, tamaño y el tipo de línea de los bordes pueden asignarse a una columna determinada del dataframe de los contactos utilizando los argumentos edge_color, edge_width y edge_linetype. Los colores y tamaño específicos de los bordes se pueden especificar como sigue:
Colores a través del argumento
edge_col_pal, de la misma manera que se utiliza paracol_pal.Tamaño pasando un rango de tamaño de los nodos al argumento
width_range.
Aquí un ejemplo:
plot(
sub,
node_color = "outcome",
node_shape = "gender",
node_size = 'age',
col_pal = c(Death = "firebrick", Recover = "green"),
shapes = c(f = "female", m = "male"),
size_range = c(40, 60),
edge_color = 'location',
edge_linetype = 'location',
edge_width = 'duration',
edge_col_pal = c(Community = "orange", Nosocomial = "purple"),
width_range = c(1, 3),
height = 700,
width = 700
)Eje temporal
También podemos visualizar la red a lo largo de un eje temporal asignando el argumento x_axis a una columna del listado. En el ejemplo siguiente, el eje-x representa la fecha de inicio de los síntomas. También hemos especificado el argumento arrow_size para asegurarnos que las flechas no son demasiado grandes, y hemos establecido label = FALSE para que la figura esté menos recargada.
plot(
sub,
x_axis = "date_onset",
node_color = "outcome",
col_pal = c(Death = "firebrick", Recover = "green"),
arrow_size = 0.5,
node_size = 13,
label = FALSE,
height = 700,
width = 700
)Hay un gran número de argumentos adicionales para especificar aún más cómo se visualiza esta red a lo largo de un eje temporal, que puede comprobar a través de ?vis_temporal_interactive (la función que se llama cuando se utiliza plot en un objeto epicontacts con el x_axis especificado). A continuación veremos algunos.
Especificar la forma del árbol de transmisión
Hay dos formas principales que puede adoptar el árbol de transmisión, especificadas mediante el argumento network_shape. La primera es una forma ramificada, como se muestra arriba, en la que un borde recto conecta dos nodos cualesquiera. Esta es la representación más intuitiva, pero puede dar lugar a la superposición de aristas en una red densamente conectada. La segunda forma es rectangle, que produce un árbol parecido a una filogenia. Por ejemplo:
plot(
sub,
x_axis = "date_onset",
network_shape = "rectangle",
node_color = "outcome",
col_pal = c(Death = "firebrick", Recover = "green"),
arrow_size = 0.5,
node_size = 13,
label = FALSE,
height = 700,
width = 700
)A cada nodo del caso se le puede asignar una posición vertical única mediante el argumento position_dodge. La posición de los casos no conectados (es decir, sin contactos reportados) se especifica utilizando el argumento unlinked_pos.
plot(
sub,
x_axis = "date_onset",
network_shape = "rectangle",
node_color = "outcome",
col_pal = c(Death = "firebrick", Recover = "green"),
position_dodge = TRUE,
unlinked_pos = "bottom",
arrow_size = 0.5,
node_size = 13,
label = FALSE,
height = 700,
width = 700
)La posición del nodo padre respecto a los nodos hijos puede especificarse mediante el argumento parent_pos. La opción por defecto es colocar el nodo padre en el centro, sin embargo puede colocarse en la parte inferior (parent_pos = 'bottom') o en la parte superior (parent_pos = 'top').
Cómo guardar gráficos y valores
Puedes guardar un gráfico como un archivo html interactivo y autónomo con la función visSave del paquete VisNetwork:
plot(
sub,
x_axis = "date_onset",
network_shape = "rectangle",
node_color = "outcome",
col_pal = c(Death = "firebrick", Recover = "green"),
parent_pos = "top",
arrow_size = 0.5,
node_size = 13,
label = FALSE,
height = 700,
width = 700
) %>%
visNetwork::visSave("network.html")Guardar estas salidas de red como una imagen es desafortunadamente menos fácil y requiere que guardes el archivo como un html y luego tomes una captura de pantalla de este archivo usando el paquete webshot. En el código siguiente, estamos convirtiendo el archivo html guardado anteriormente en un PNG:
webshot(url = "network.html", file = "network.png")Líneas de tiempo
También se pueden incluir líneas de tiempo en la red, que se representan en el eje de abscisas de cada caso. Esto puede servir para visualizar la ubicación de los casos, por ejemplo, o el tiempo hasta el resultado. Para generar una línea de tiempo, tenemos que crear un data.frame de al menos tres columnas que indiquen el ID del caso, la fecha de inicio del “evento” y la fecha de finalización del “evento”. También se puede añadir cualquier número de otras columnas que luego se pueden asignar a las propiedades de los nodos y aristas de la línea de tiempo. En el código siguiente, generamos una línea de tiempo que va desde la fecha de inicio de los síntomas hasta la fecha del desenlace, y mantenemos las variables de desenlace y hospital que utilizamos para definir la forma y el color de los nodos. Ten en cuenta que puede tener más de una fila/evento de la línea de tiempo por caso, por ejemplo si un caso es transferido entre varios hospitales.
## generar la línea temporal
timeline <- linelist %>%
transmute(
id = case_id,
start = date_onset,
end = date_outcome,
outcome = outcome,
hospital = hospital
)A continuación, pasamos el elemento de la línea de tiempo al argumento timeline. Podemos mapear los atributos de la línea de tiempo a los colores, formas y tamaños de los nodos de la línea de tiempo de la misma manera definida en las secciones anteriores, excepto que tenemos dos nodos: el nodo de inicio y el nodo final de cada línea de tiempo, que tienen argumentos separados. Por ejemplo, tl_start_node_color define qué columna de la línea de tiempo se asigna al color del nodo inicial, mientras que tl_end_node_shape define qué columna de la línea de tiempo se asigna a la forma del nodo final. También podemos asignar el color, la anchura, el tipo de línea y las etiquetas al borde de la línea de tiempo mediante los argumentos tl_edge_.
Consulta ?vis_temporal_interactive (la función a la que se llama cuando se traza un objeto epicontacto) para obtener documentación detallada sobre los argumentos. Cada argumento está anotado también en el código de abajo:
## define las formas
shapes <- c(
f = "female",
m = "male",
Death = "user-times",
Recover = "heartbeat",
"NA" = "question-circle"
)
## define los colores
colours <- c(
Death = "firebrick",
Recover = "green",
"NA" = "grey"
)
## realizar el gráfico
plot(
sub,
## coordenada x máxima a la fecha de inicio
x_axis = "date_onset",
## utiliza una forma de red rectangular
network_shape = "rectangle",
## asigna las formas de los nodos a la columna de género
node_shape = "gender",
## no queremos asignar el color del nodo a ninguna columna - esto es importante ya que el
## valor por defecto es asignar al id del nodo, lo que desordenará el esquema de color
node_color = NULL,
## establecer el tamaño del nodo caso a 30 (como no es un caracter, node_size no es
## asignado a una columna sino que se interpreta como el tamaño real del nodo)
node_size = 30,
## establece la anchura del enlace de transmisión en 4 (como no es un carácter, edge_width
## no se asigna a una columna, sino que se interpreta como la anchura real del borde).
edge_width = 4,
## proporciona el objeto timeline
timeline = timeline,
## asigna la forma del nodo final a la columna del resultado en el objeto timeline
tl_end_node_shape = "outcome",
## establece el tamaño del nodo final en 15 (como no es un carácter, este
## argumento no se asigna a una columna sino que se interpreta como el
## tamaño real del nodo)
tl_end_node_size = 15,
## asigna el color del borde de timeline a la columna hospital
tl_edge_color = "hospital",
## establece el ancho del borde de la línea de tiempo en 2 (como no es un carácter, este argumento
## argumento no se asigna a una columna, sino que se interpreta como la anchura real
## del borde).
tl_edge_width = 2,
## asigna las etiquetas de los bordes a la variable hospital
tl_edge_label = "hospital",
## especifica la forma para cada atributo de nodo (definido anteriormente)
shapes = shapes,
## especifica la paleta de colores (definida anteriormente)
col_pal = colours,
## establece el tamaño de la flecha en 0.5
arrow_size = 0.5,
## usa dos columnas en la leyenda
legend_ncol = 2,
## establece el tamaño de la fuente
font_size = 15,
## define el formato de las fechas
date_labels = c("%d %b %Y"),
## no muestra las etiquetas ID debajo de los nodos
label = FALSE,
## especifica la altura
height = 1000,
## especifica la anchura
width = 1200,
## asegura que cada nodo case tiene una única coordenada y - esto es muy importante
## cuando se utilizan líneas de tiempo, de lo contrario tendrá líneas de tiempo superpuestas de
## diferentes casos
position_dodge = TRUE
)## Warning in assert_timeline(timeline, x, x_axis): 5865 timeline row(s) removed as ID not found in linelist or start/end date is NA37.5 Análisis
Resumiendo
Podemos obtener una visión general de algunas de las propiedades de la red utilizando la función summary.
## resume el objeto epicontactos
summary(epic)##
## /// Overview //
## // number of unique IDs in linelist: 5888
## // number of unique IDs in contacts: 5511
## // number of unique IDs in both: 4352
## // number of contacts: 3800
## // contacts with both cases in linelist: 56.868 %
##
## /// Degrees of the network //
## // in-degree summary:
## Min. 1st Qu. Median Mean 3rd Qu. Max.
## 0.0000 0.0000 1.0000 0.5392 1.0000 1.0000
##
## // out-degree summary:
## Min. 1st Qu. Median Mean 3rd Qu. Max.
## 0.0000 0.0000 0.0000 0.5392 1.0000 6.0000
##
## // in and out degree summary:
## Min. 1st Qu. Median Mean 3rd Qu. Max.
## 0.000 1.000 1.000 1.078 1.000 7.000
##
## /// Attributes //
## // attributes in linelist:
## generation date_infection date_onset date_hospitalisation date_outcome outcome gender age age_unit age_years age_cat age_cat5 hospital lon lat infector source wt_kg ht_cm ct_blood fever chills cough aches vomit temp time_admission bmi days_onset_hosp
##
## // attributes in contacts:
## location durationPor ejemplo, podemos ver que sólo el 57% de los contactos tienen ambos casos en linelist; esto significa que no tenemos datos del listado sobre un número significativo de casos involucrados en estas cadenas de transmisión.
Características de los pares
La función get_pairwise() permite procesar la(s) variable(s) del listado según cada par de los datos de contactos. En el siguiente ejemplo, la fecha de inicio de la enfermedad se extrae del listado para calcular la diferencia entre la fecha de inicio de la enfermedad para cada par. El valor que se obtiene de esta comparación representa el intervalo de serie (si).
si <- get_pairwise(epic, "date_onset")
summary(si)## Min. 1st Qu. Median Mean 3rd Qu. Max. NA's
## 0.00 5.00 9.00 11.01 15.00 99.00 1820
tibble(si = si) %>%
ggplot(aes(si)) +
geom_histogram() +
labs(
x = "Serial interval",
y = "Frequency"
)## `stat_bin()` using `bins = 30`. Pick better value with `binwidth`.## Warning: Removed 1820 rows containing non-finite values (`stat_bin()`).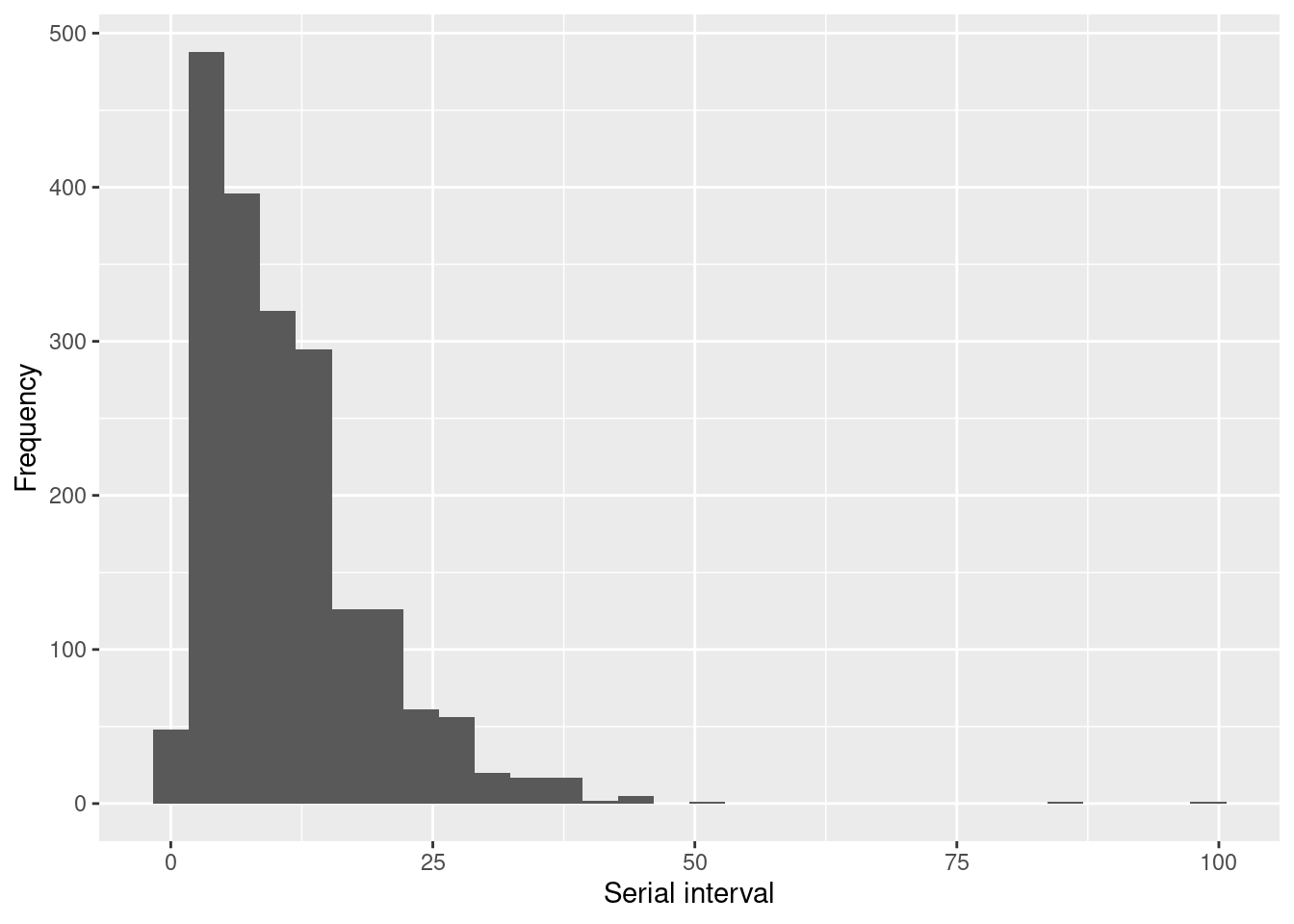
La función get_pairwise() interpretará el tipo de la columna que se utiliza para la comparación, y ajustará su método de comparación de los valores en consecuencia. Para los números y las fechas (como en el ejemplo de si), la función restará los valores. Cuando se aplica a columnas que son caracteres o categóricas, get_pairwise() pegará los valores. Dado que la función también permite un procesamiento arbitrario (véase el argumento “f”), estas combinaciones discretas pueden ser fácilmente tabuladas y analizadas.
head(get_pairwise(epic, "gender"), n = 10)## [1] "f -> m" NA "m -> m" NA "m -> f" "f -> f" NA "f -> m" NA "m -> f"
get_pairwise(epic, "gender", f = table)## values.to
## values.from f m
## f 464 516
## m 510 468
fisher.test(get_pairwise(epic, "gender", f = table))##
## Fisher's Exact Test for Count Data
##
## data: get_pairwise(epic, "gender", f = table)
## p-value = 0.03758
## alternative hypothesis: true odds ratio is not equal to 1
## 95 percent confidence interval:
## 0.6882761 0.9892811
## sample estimates:
## odds ratio
## 0.8252575En este caso, se observa una asociación significativa entre los vínculos de transmisión y el género.
Identificación de clusters
La función get_clusters() puede utilizarse para identificar componentes conectados en un objeto epicontacts. En primer lugar, la utilizamos para recuperar un data.frame que contenga la información de los clusters:
clust <- get_clusters(epic, output = "data.frame")
table(clust$cluster_size)##
## 1 2 3 4 5 6 7 8 9 10 11 12 13 14
## 1536 1680 1182 784 545 342 308 208 171 100 99 24 26 42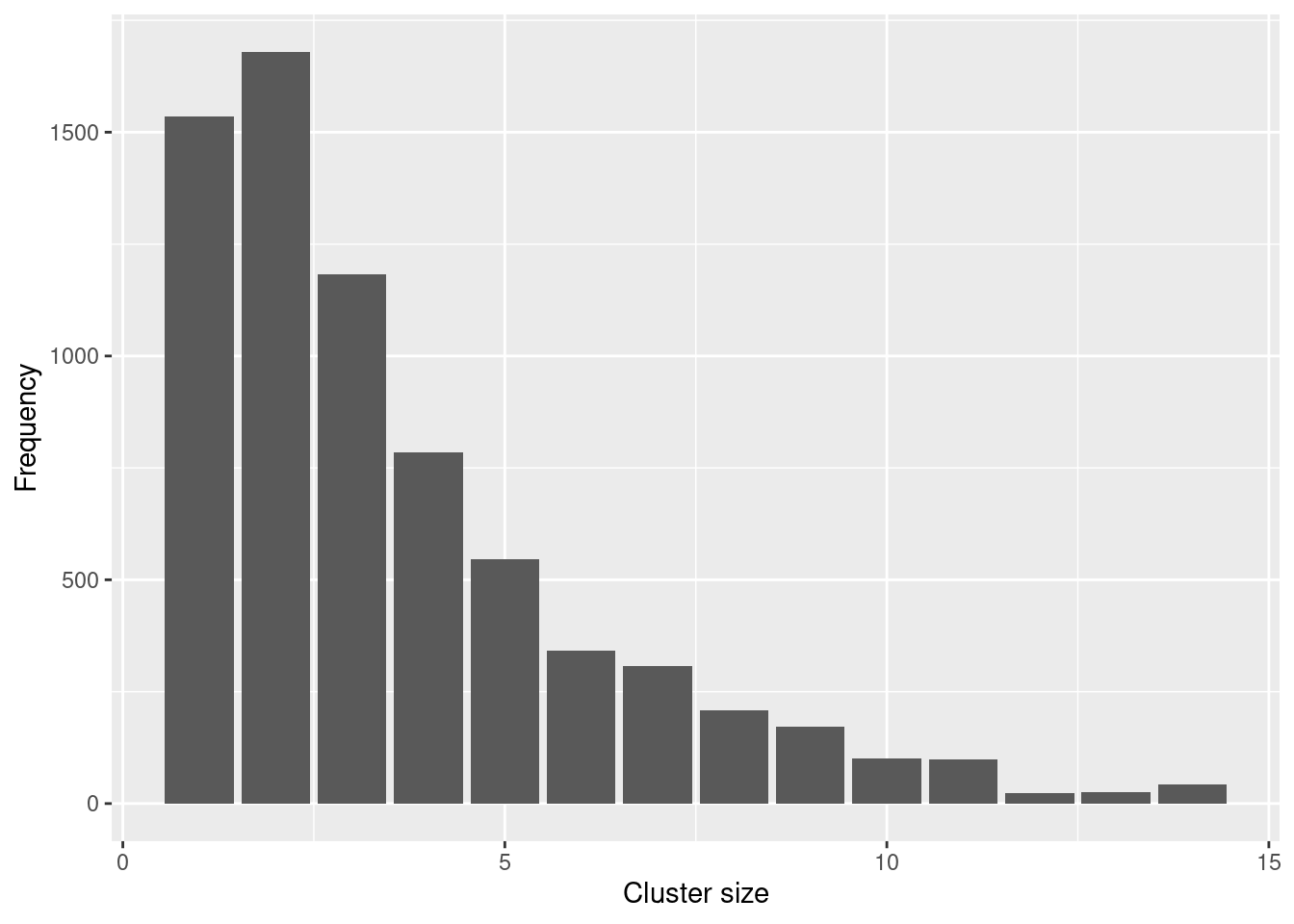
Veamos los clusters más grandes. Para ello, añadimos la información de los clústers al objeto epicontacts y luego lo subconjuntamos para mantener sólo los clústers más grandes:
Cálculo de grados
El grado de un nodo corresponde a su número de aristas o conexiones con otros nodos. get_degree() proporciona un método sencillo para calcular este valor para las redes de epicontacts. Un grado alto en este contexto indica un individuo que estuvo en contacto con muchos otros. El argumento type indica que queremos contar tanto el grado de entrada como el de salida, el argumento only_linelist indica que sólo queremos calcular el grado para los casos del listado.
deg_both <- get_degree(epic, type = "both", only_linelist = TRUE)¿Qué personas son las que tienen más de 10 contactos?
## 916d0a 858426 6833d7 f093ea 11f8ea 3a4372 38fc71 c8c4d5 a127a7 02d8fd
## 7 6 6 6 5 5 5 5 5 5¿Cuál es el número medio de contactos?
mean(deg_both)## [1] 1.07847337.6 Recursos
La página de epicontacts ofrece una visión general de las funciones del paquete e incluye algunas viñetas más detalladas.
La página de github de epicontacts puede utilizarse para plantear problemas y solicitar funciones.