31 Consejos de ggplot
En esta página cubriremos consejos y trucos para hacer que tus ggplots sean nítidos y elegantes. Consulta la página sobre Conceptos básicos de ggplot para conocer los fundamentos.
Hay varios tutoriales extensos de ggplot2 enlazados en la sección de Recursos. También puedes descargar esta hoja de trucos de visualización de datos con ggplot desde el sitio web de RStudio. Recomendamos encarecidamente que busques inspiración en la galería de gráficos de R y en Data-to-viz.
31.1 Preparación
Cargar paquetes
Este trozo de código muestra la carga de los paquetes necesarios para los análisis. En este manual destacamos p_load() de pacman, que instala el paquete si es necesario y lo carga para su uso. También puedes cargar los paquetes instalados con library() de R base. Consulta la página sobre fundamentos de R para obtener más información sobre los paquetes de R.
pacman::p_load(
tidyverse, # incluye ggplot2 y otros
rio, # importar/exportar
here, # localizador de ficheros
stringr, # trabajar con caracteres
scales, # transformar números
ggrepel, # etiquetas colocadas inteligentemente
gghighlight, # resaltar una parte del gráfico
RColorBrewer # escalas de color
)Importar datos
Para esta página, importamos el conjunto de datos de casos de una epidemia de ébola simulada. Si quieres seguir el proceso, clica para descargar linelist “limpio” (como archivo .rds). Importa los datos con la función import() del paquete rio (maneja muchos tipos de archivos como .xlsx, .csv, .rds - ver la página de importación y exportación para más detalles).
linelist <- rio::import("linelist_cleaned.rds")A continuación se muestran las primeras 50 filas de linelist.
31.2 Escalas para el color, relleno, ejes, etc.
En ggplot2, cuando la estética de los datos trazados (por ejemplo, el tamaño, el color, la forma, el relleno, el eje de trazado) se asigna a las columnas de los datos, la visualización exacta se puede ajustar con el correspondiente comando “scale”. En esta sección explicamos algunos ajustes de escala comunes.
Esquemas de color
Una cosa que puede ser inicialmente difícil de entender con ggplot2 es el control de los esquemas de color. Ten en cuenta que esta sección discute el color de los objetos a representar (geoms/shapes) como puntos, barras, líneas, mosaicos, etc. Para ajustar el color del texto accesorio, los títulos o el color de fondo, consulta la sección Temas de la página Conceptos básicos de ggplot.
Para controlar el “color” de los objetos de la gráfica se ajustará la estética del color = (el color exterior) o la estética del relleno, fill = (el color interior). Una excepción a este patrón es geom_point(), donde realmente sólo se llega a controlar color =, que controla el color del punto (interior y exterior).
Al establecer el color o el relleno puedes utilizar nombres de colores reconocidos por R como "red" (ver lista completa o introducir ?colors), o un color hexadecimal específico como "#ff0505".
# histograma -
ggplot(data = linelist, mapping = aes(x = age))+ # establece datos y ejes
geom_histogram( # muestra el histograma
binwidth = 7, # anchura de los cuadrados
color = "red", # color de la línea de los cuadrados
fill = "lightblue") # color del interior del cuadrado (fill)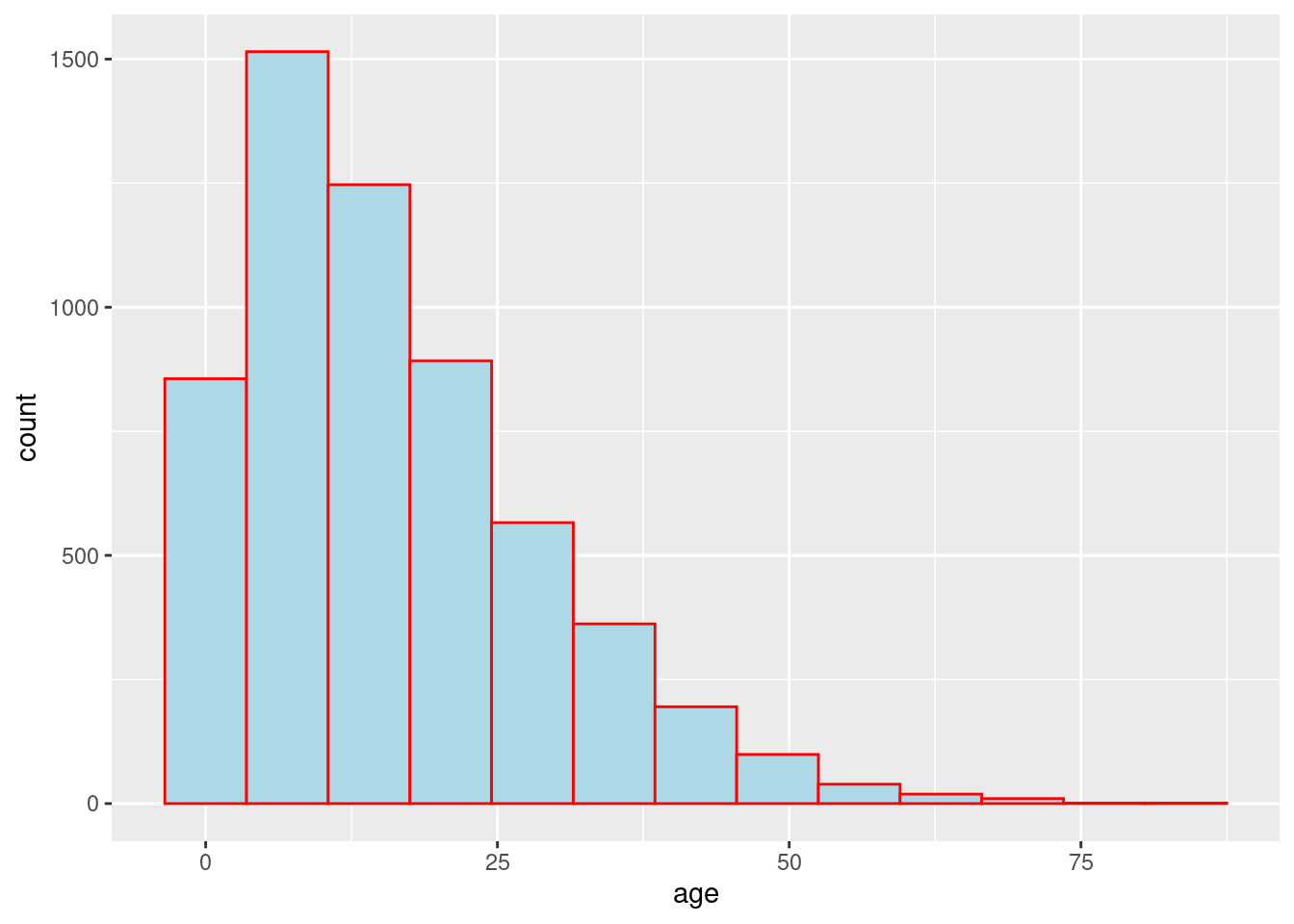
Como se explica en la sección asignación de datos al gráfico de Conceptos básicos de ggplot sobre la estética como fill = y color = puede definirse fuera de una sentencia mapping = aes() o dentro de ella. Si está fuera de aes(), el valor asignado debe ser estático (por ejemplo, color = "blue") y se aplicará a todos los datos trazados por geom. Si está dentro, la estética debe asignarse a una columna, como color = hospital, y la expresión variará según el valor de esa fila en los datos. Algunos ejemplos:
# Color estático para los puntos y para la línea
ggplot(data = linelist, mapping = aes(x = age, y = wt_kg))+
geom_point(color = "purple")+
geom_vline(xintercept = 50, color = "orange")+
labs(title = "Static color for points and line")
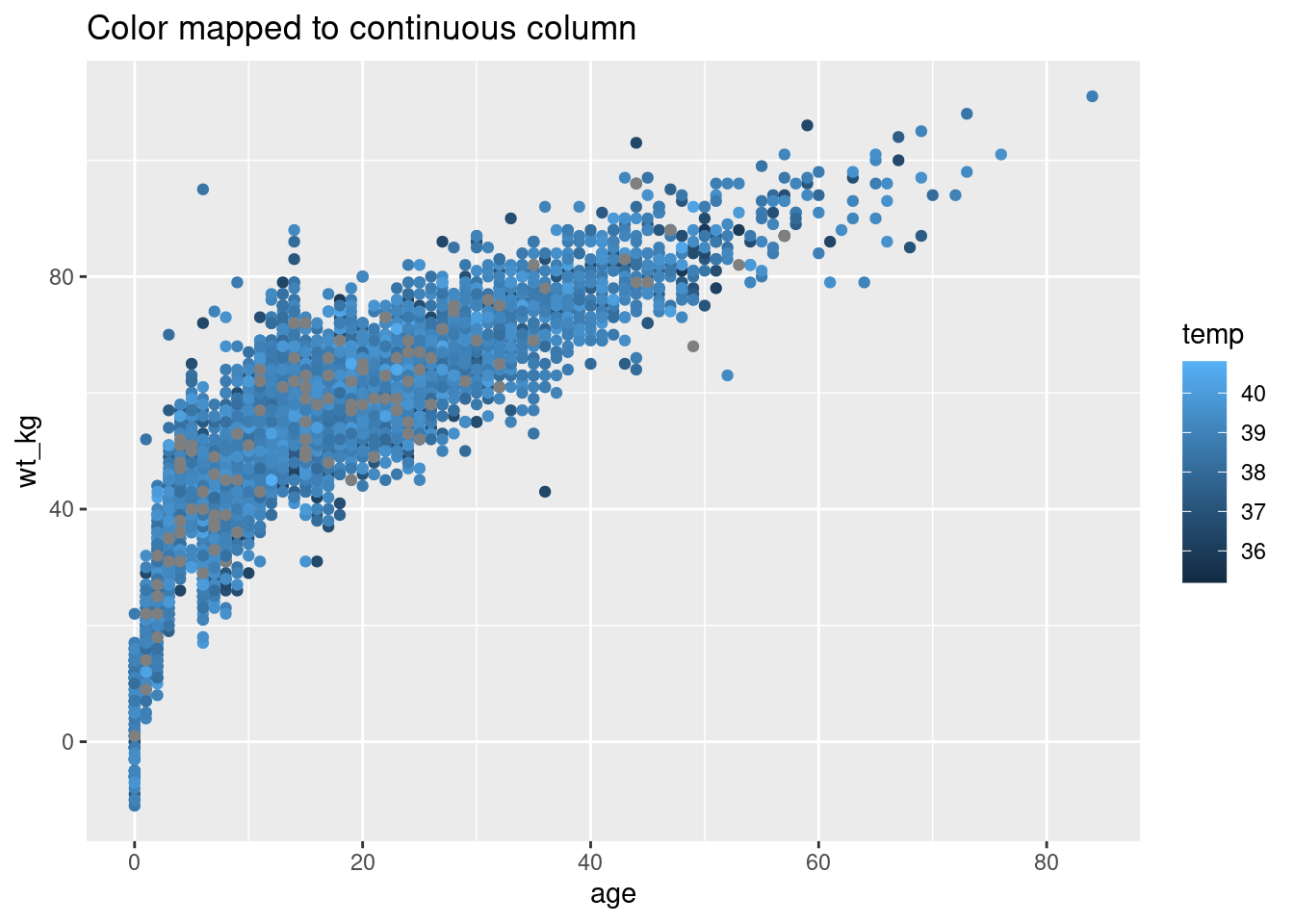
# Color asignado a la columna continua
ggplot(data = linelist, mapping = aes(x = age, y = wt_kg))+
geom_point(mapping = aes(color = temp))+
labs(title = "Color mapped to continuous column")
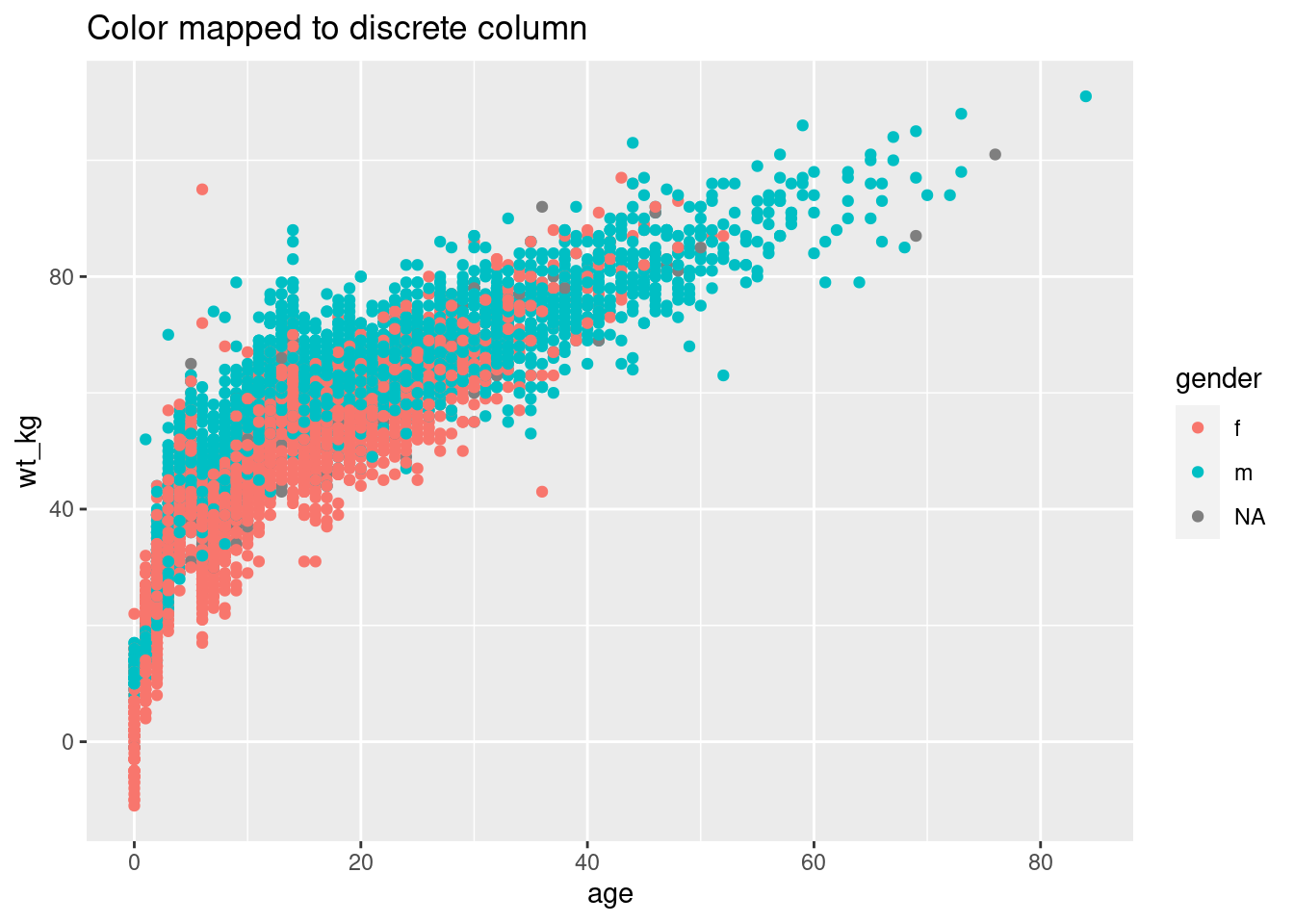
# Color asignado a una columna discreta
ggplot(data = linelist, mapping = aes(x = age, y = wt_kg))+
geom_point(mapping = aes(color = gender))+
labs(title = "Color mapped to discrete column")
# gráfico de barras, relleno a columna discreta, color a valor estático
ggplot(data = linelist, mapping = aes(x = hospital))+
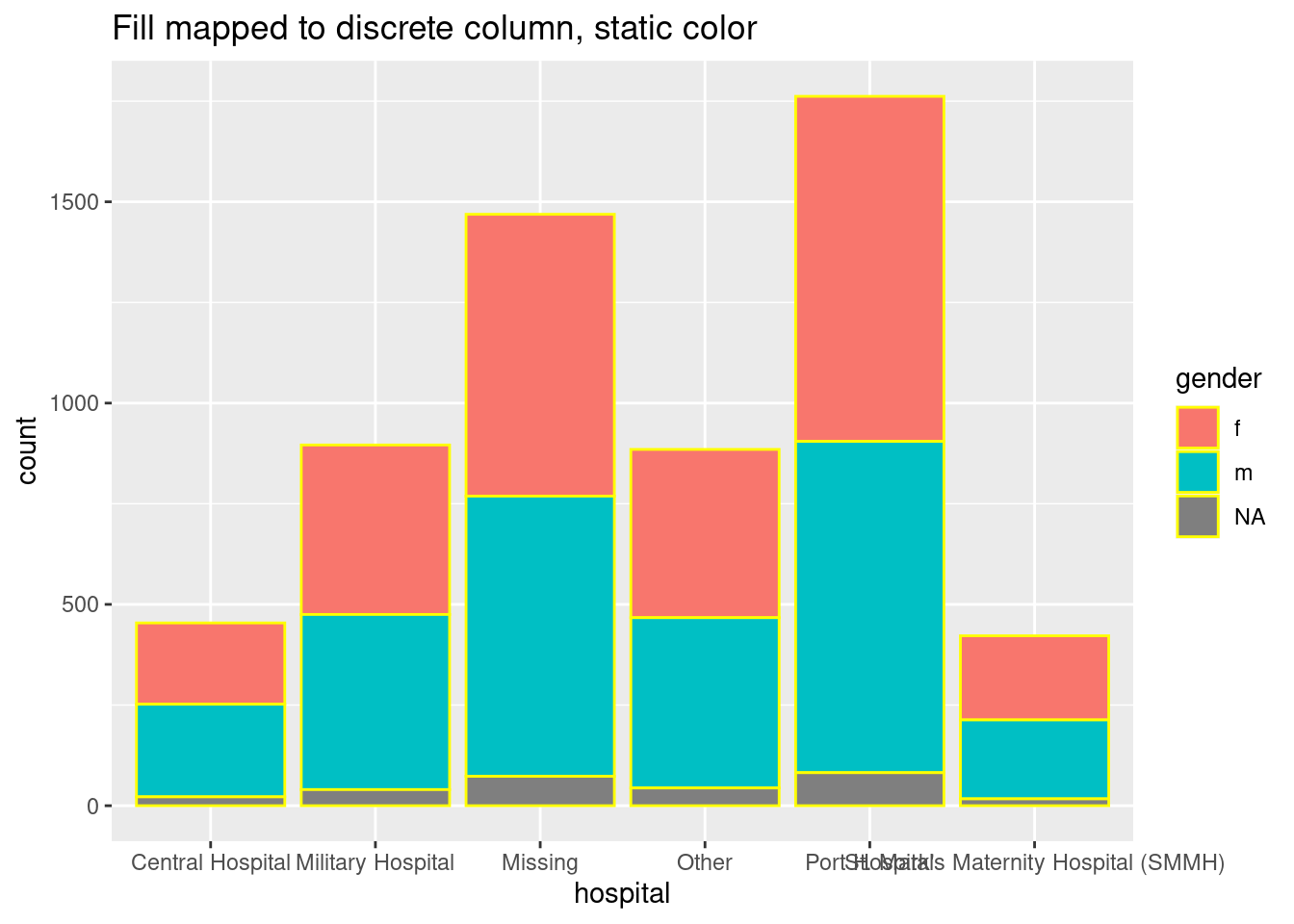
geom_bar(mapping = aes(fill = gender), color = "yellow")+
labs(title = "Fill mapped to discrete column, static color")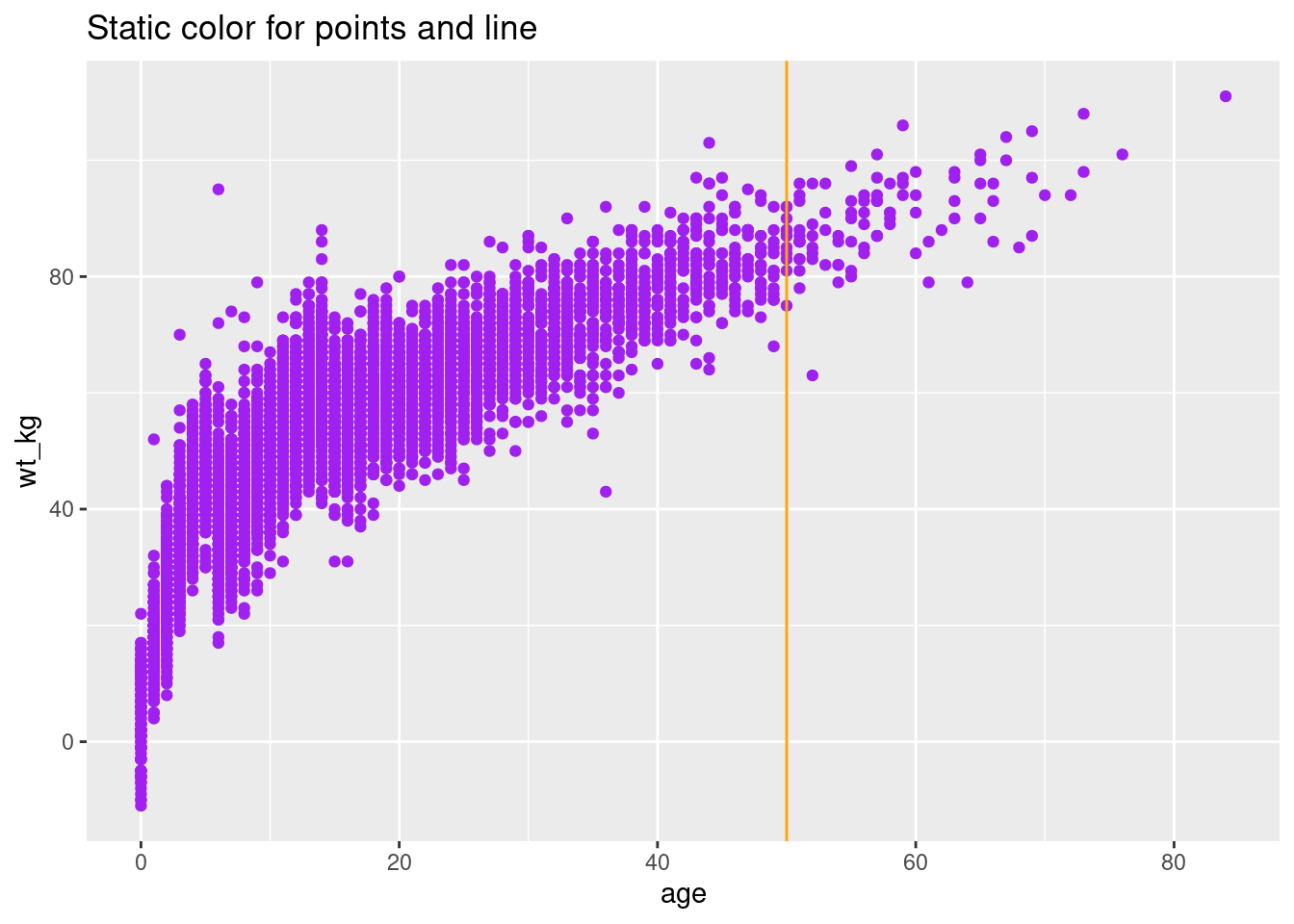
Escalas
Una vez que se asigna una columna a una estética de la gráfica (por ejemplo, x =, y =, fill =, color =…), tu gráfica ganará una escala / leyenda. Mira arriba cómo la escala puede ser continua, discreta, de fecha, etc. dependiendo del tipo de la columna asignada. Si tienes múltiples estéticas asignadas a las columnas, tu gráfico tendrá múltiples escalas.
Puedes controlar las escalas con la función scales_() apropiada. Las funciones de escala de ggplot() tienen 3 partes que se escriben así: scale_AESTHETIC_METHOD().
- La primera parte,
scale_(), es fija. - La segunda parte, la ESTÉTICA, debe ser la estética para la que deseas ajustar la escala (
_fill_,_shape_,_color_,_size_,_alpha_…) - las opciones aquí también incluyen_x_e_y_. - La tercera parte, el MÉTODO, será
_discrete(),continuous(),_date(),_gradient(), o_manual()dependiendo del tipo de la columna y de cómo se quiera controlar. Hay otros, pero estos son los más utilizados.
Asegúrate de utilizar la función correcta para la escala. De lo contrario, tu comando de escala no parecerá cambiar nada. Si tienes varias escalas, puedes utilizar varias funciones de escala para ajustarlas. Por ejemplo:
Argumentos de la escala
Cada tipo de escala tiene sus propios argumentos, aunque hay cierto solapamiento. Consulta la función escribiendo ?scale_color_discrete en la consola de R para ver la documentación de los argumentos de la función.
Para escalas continuas, utiliza breaks = para proporcionar una secuencia de valores con seq() (to =, from =, y by = como se muestra en el ejemplo siguiente. Fija expand = c(0,0) para eliminar el espacio de relleno alrededor de los ejes (esto se puede utilizar en cualquier escala _x_ o _y_.
En el caso de las escalas discretas, puedes ajustar el orden de aparición de los niveles con los breaks =, y cómo se muestran los valores con el argumento labels =. Proporciona un vector de caracteres a cada uno de ellos (véase el ejemplo siguiente). También puedes dejar de lado NA fácilmente estableciendo na.translate = FALSE.
Los matices de las escalas de fechas se tratan más ampliamente en la página de curvas epidémicas.
Ajustes manuales
Uno de los trucos más útiles es utilizar las funciones de escalado “manual” para asignar explícitamente los colores que se deseen. Son funciones con la sintaxis scale_xxx_manual() (por ejemplo, scale_colour_manual() o scale_fill_manual()). Cada uno de los argumentos siguientes se demuestra en el ejemplo de código que sigue.
- Asignar colores a los valores de los datos con el argumento
values = - Especificar un color para
NAconna.value = - Cambiar cómo se escriben los valores en la leyenda con el argumento
labels = - Cambiar el título de la leyenda con
name =
A continuación, creamos un gráfico de barras y mostramos cómo aparece por defecto, y luego con tres escalas ajustadas - la escala continua del eje-y, la escala discreta del eje-x, y el ajuste manual del relleno (color interior de la barra).
# LÍNEA BASE - sin ajuste de escala
ggplot(data = linelist)+
geom_bar(mapping = aes(x = outcome, fill = gender))+
labs(title = "Baseline - no scale adjustments")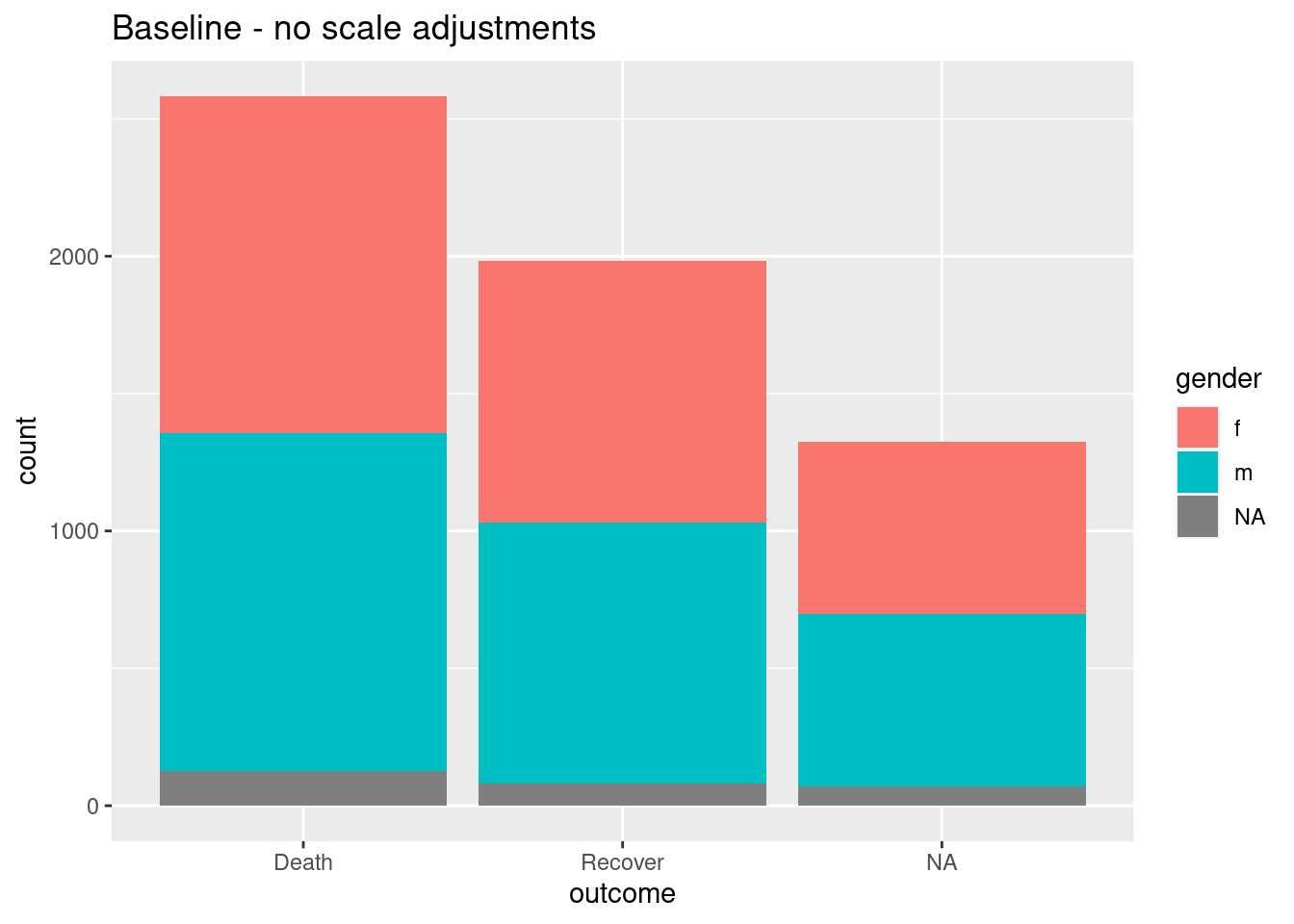
# ESCALAS AJUSTADAS
ggplot(data = linelist)+
geom_bar(mapping = aes(x = outcome, fill = gender), color = "black")+
theme_minimal()+ # fondo simplificado
scale_y_continuous( # escala continua para el eje-y (recuentos)
expand = c(0,0), # sin relleno
breaks = seq(from = 0,
to = 3000,
by = 500))+
scale_x_discrete( # escala discreta para el eje x (género)
expand = c(0,0), # sin relleno
drop = FALSE, # muestra todos los niveles del factor (incluso si no están en los datos)
na.translate = FALSE, # elimina los resultados NA del gráfico
labels = c("Died", "Recovered"))+ # Cambia la visualización de los valores
scale_fill_manual( # Especifica manualmente el relleno (color interior de la barra)
values = c("m" = "violetred", # valores de referencia en los datos para asignar colores
"f" = "aquamarine"),
labels = c("m" = "Male", # reetiqueta la leyenda (utiliza la asignación "=" para evitar errores)
"f" = "Female",
"Missing"),
name = "Gender", # título de la leyenda
na.value = "grey" # asigna un color para los valores que faltan
)+
labs(title = "Adjustment of scales") # ajusta el título de la leyenda de relleno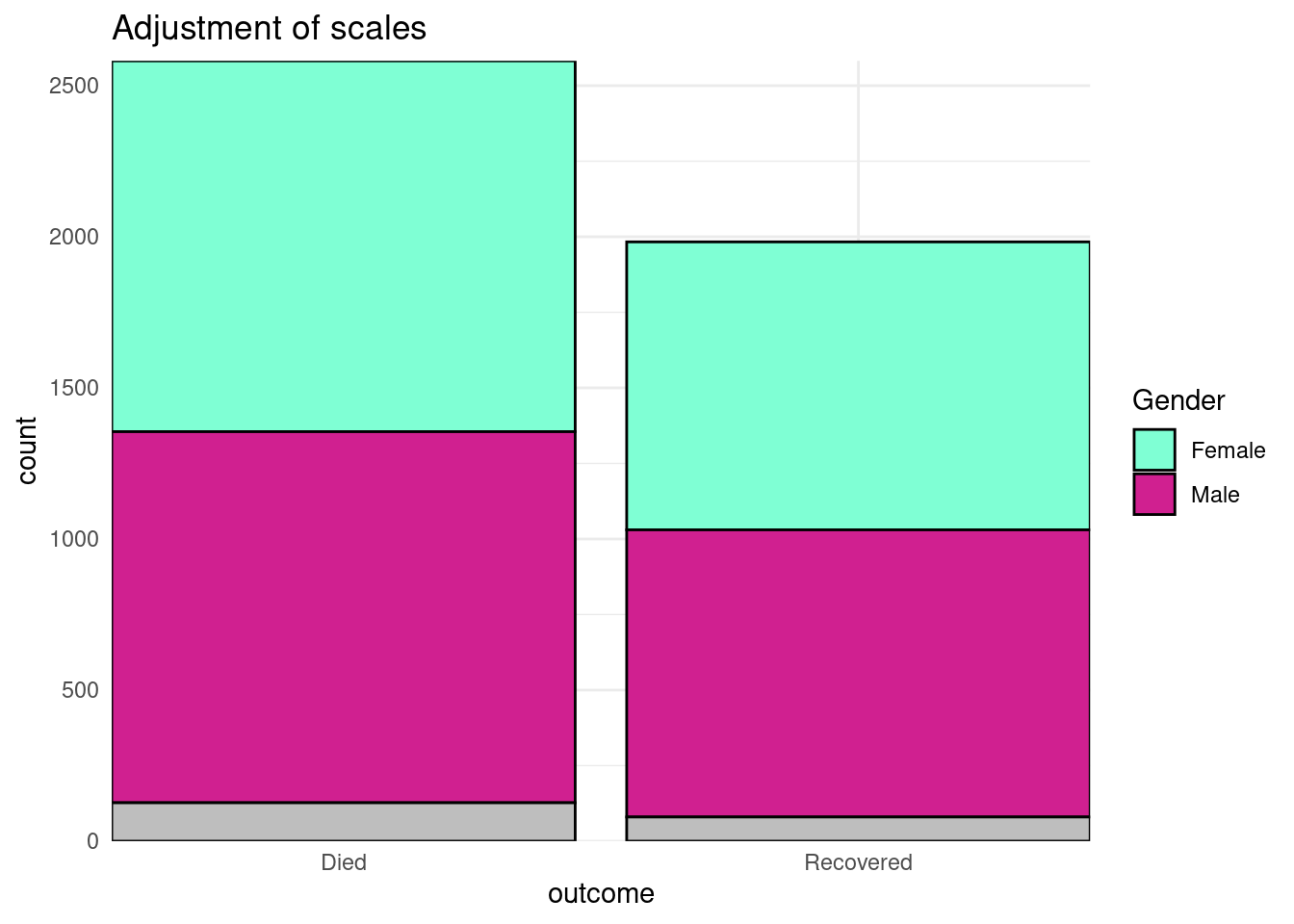
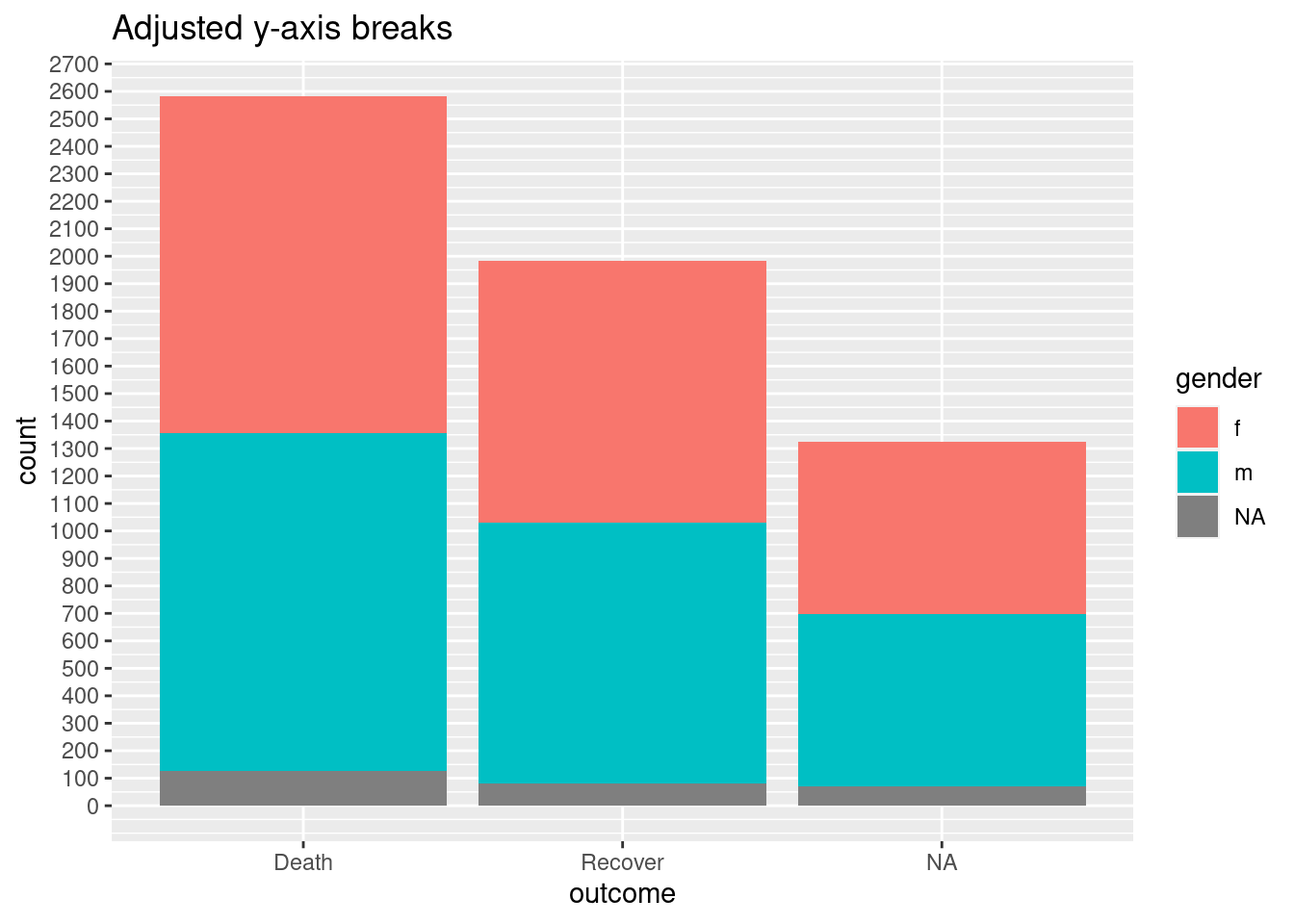
Escalas de ejes continuos
Cuando los datos se mapean a los ejes del gráfico, éstos también pueden ajustarse con comandos de escalas. Un ejemplo común es el ajuste de la visualización de un eje (por ejemplo, el eje-y) que se asigna a una columna con datos continuos.
Es posible que queramos ajustar los descansos o la visualización de los valores en ggplot utilizando scale_y_continuous(). Como se indicó anteriormente, utiliza el argumento breaks = para proporcionar una secuencia de valores que servirán como “saltos” a lo largo de la escala. Estos son los valores en los que se mostrarán los números. A este argumento, puedes proporcionar un vector c() que contenga los valores de ruptura deseados, o puedes proporcionar una secuencia regular de números utilizando la función seq() de R base. Esta función seq() acepta to =, from =, y by =.
# LÍNEA BASE - sin ajuste de escala
ggplot(data = linelist)+
geom_bar(mapping = aes(x = outcome, fill = gender))+
labs(title = "Baseline - no scale adjustments")
#
ggplot(data = linelist)+
geom_bar(mapping = aes(x = outcome, fill = gender))+
scale_y_continuous(
breaks = seq(
from = 0,
to = 3000,
by = 100)
)+
labs(title = "Adjusted y-axis breaks")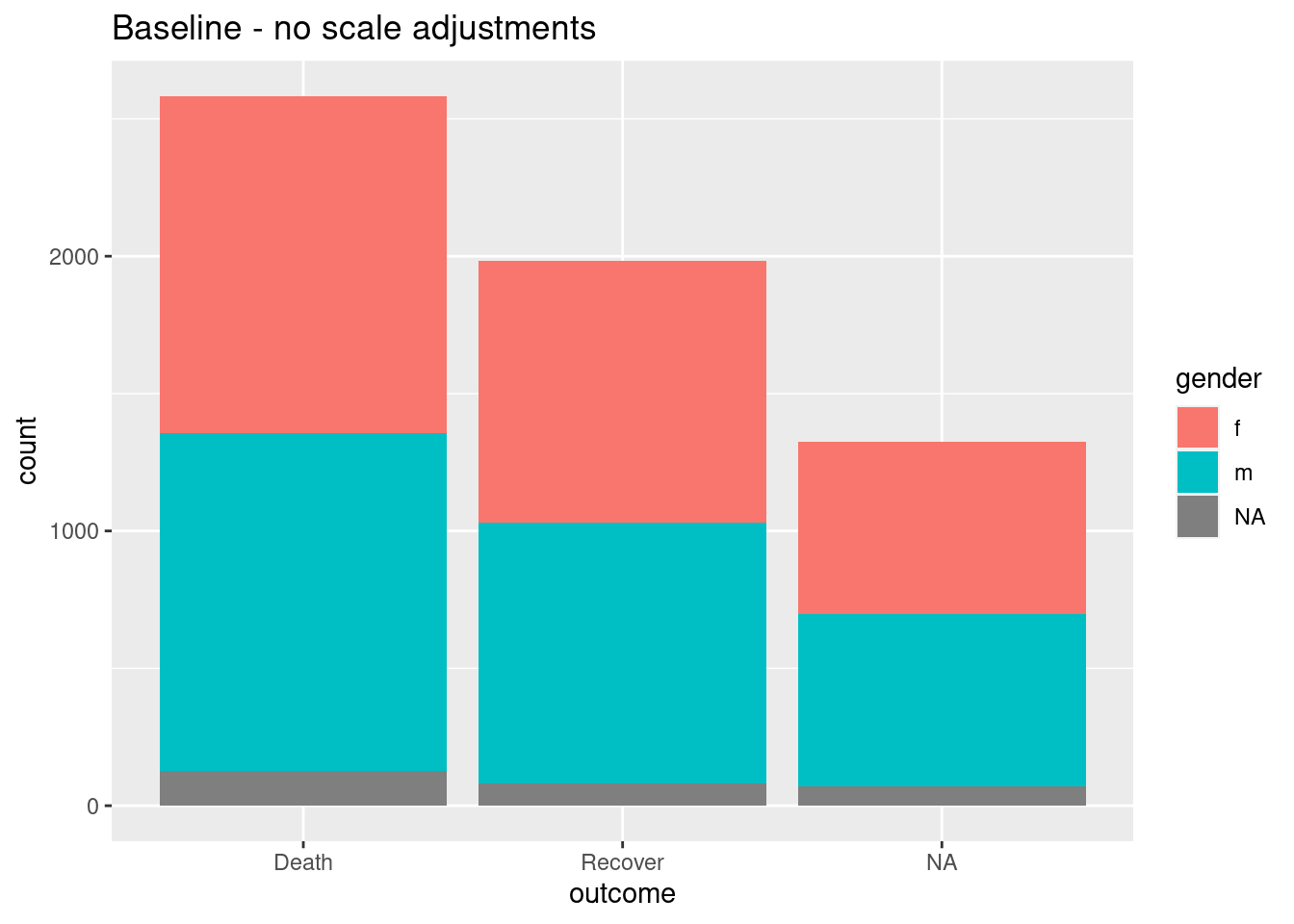
Mostrar porcentajes
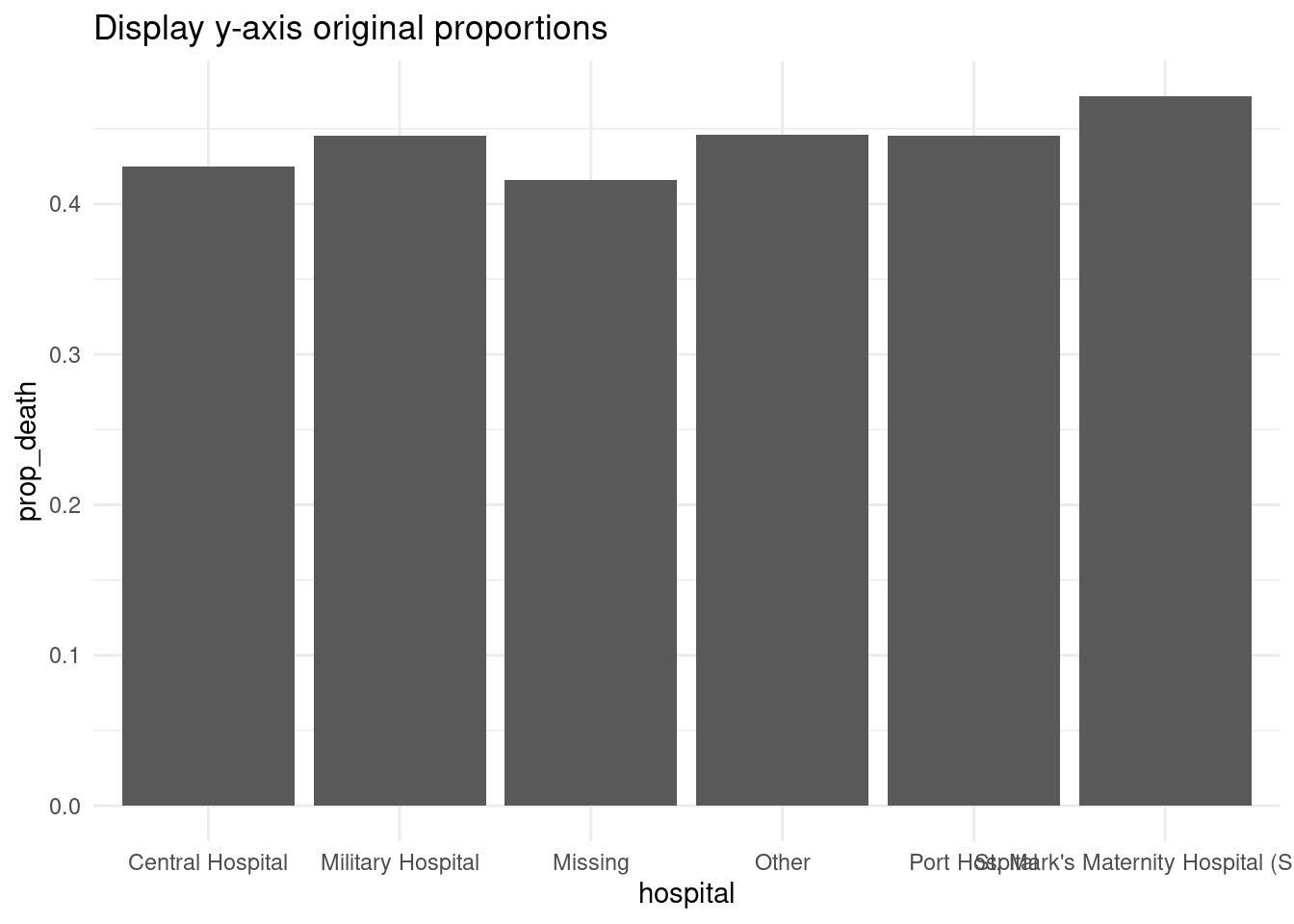
Si los valores de datos originales son proporciones, puedes mostrarlos fácilmente como porcentajes con “%” proporcionando labels = scales::percent en el comando de escalas, como se muestra a continuación.
Aunque una alternativa sería convertir los valores en caracteres y añadir un carácter “%” al final, este enfoque causará complicaciones porque tus datos ya no serán valores numéricos continuos.
# Proporciones originales del eje-y
###################################
linelist %>% # empieza con linelist
group_by(hospital) %>% # agrupa los datos por hospital
summarise( # crea columnas de resumen
n = n(), # número total de filas en el grupo
deaths = sum(outcome == "Death", na.rm=T), # número de defunciones en el grupo
prop_death = deaths/n) %>% # proporción de defunciones en el grupo
ggplot( # comienza a trazar
mapping = aes(
x = hospital,
y = prop_death))+
geom_col()+
theme_minimal()+
labs(title = "Display y-axis original proportions")
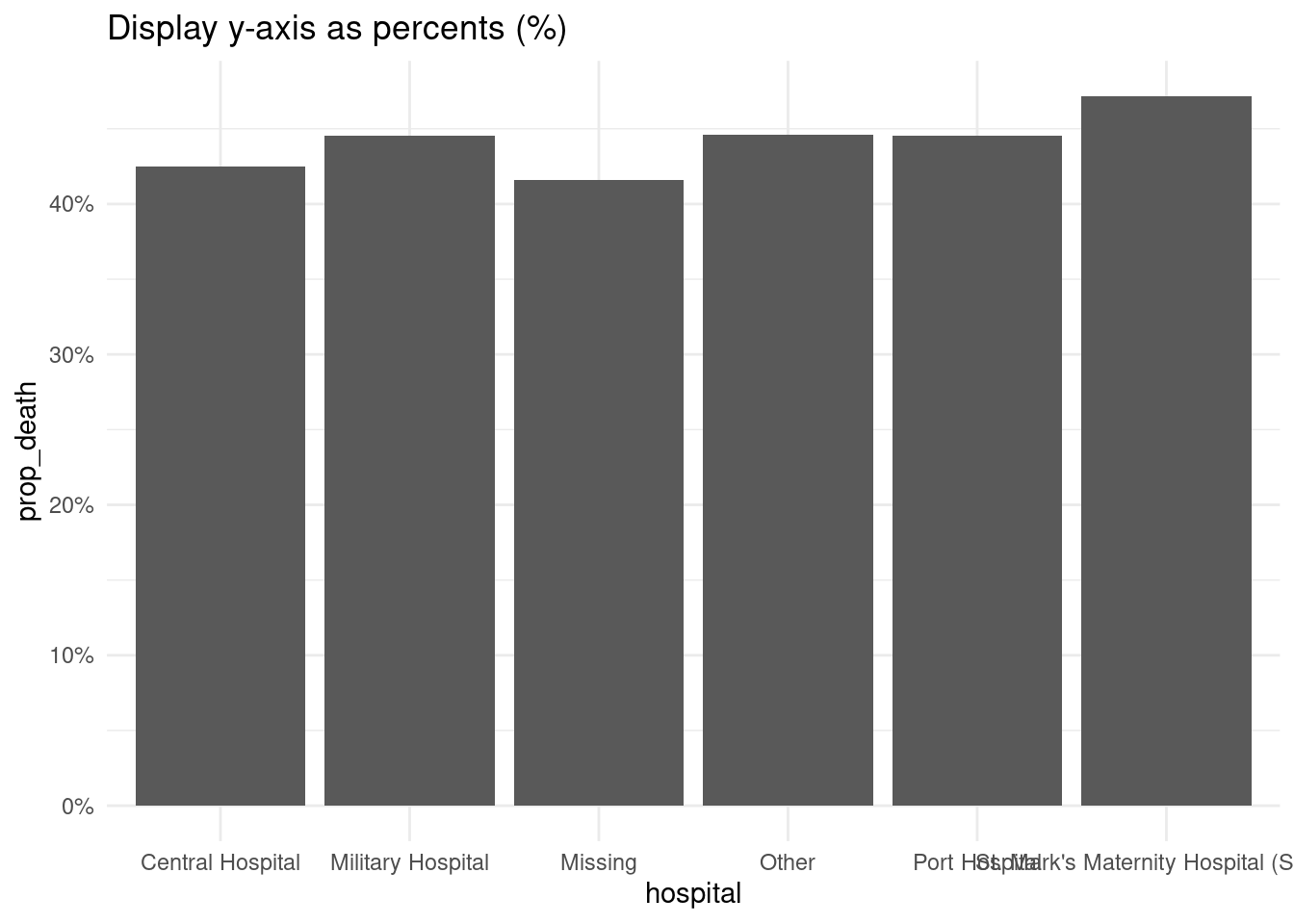
# Mostrar las proporciones del eje-y como porcentajes
#####################################################
linelist %>%
group_by(hospital) %>%
summarise(
n = n(),
deaths = sum(outcome == "Death", na.rm=T),
prop_death = deaths/n) %>%
ggplot(
mapping = aes(
x = hospital,
y = prop_death))+
geom_col()+
theme_minimal()+
labs(title = "Display y-axis as percents (%)")+
scale_y_continuous(
labels = scales::percent # muestra las proporciones como porcentajes
)
Escala logarítmica
Para transformar un eje continuo a escala logarítmica, añade trans = "log2" al comando de escala. A modo de ejemplo, creamos un dataframe de regiones con sus respectivos valores de preparedness_index y casos acumulados.
plot_data <- data.frame(
region = c("A", "B", "C", "D", "E", "F", "G", "H", "I"),
preparedness_index = c(8.8, 7.5, 3.4, 3.6, 2.1, 7.9, 7.0, 5.6, 1.0),
cases_cumulative = c(15, 45, 80, 20, 21, 7, 51, 30, 1442)
)
plot_data## region preparedness_index cases_cumulative
## 1 A 8.8 15
## 2 B 7.5 45
## 3 C 3.4 80
## 4 D 3.6 20
## 5 E 2.1 21
## 6 F 7.9 7
## 7 G 7.0 51
## 8 H 5.6 30
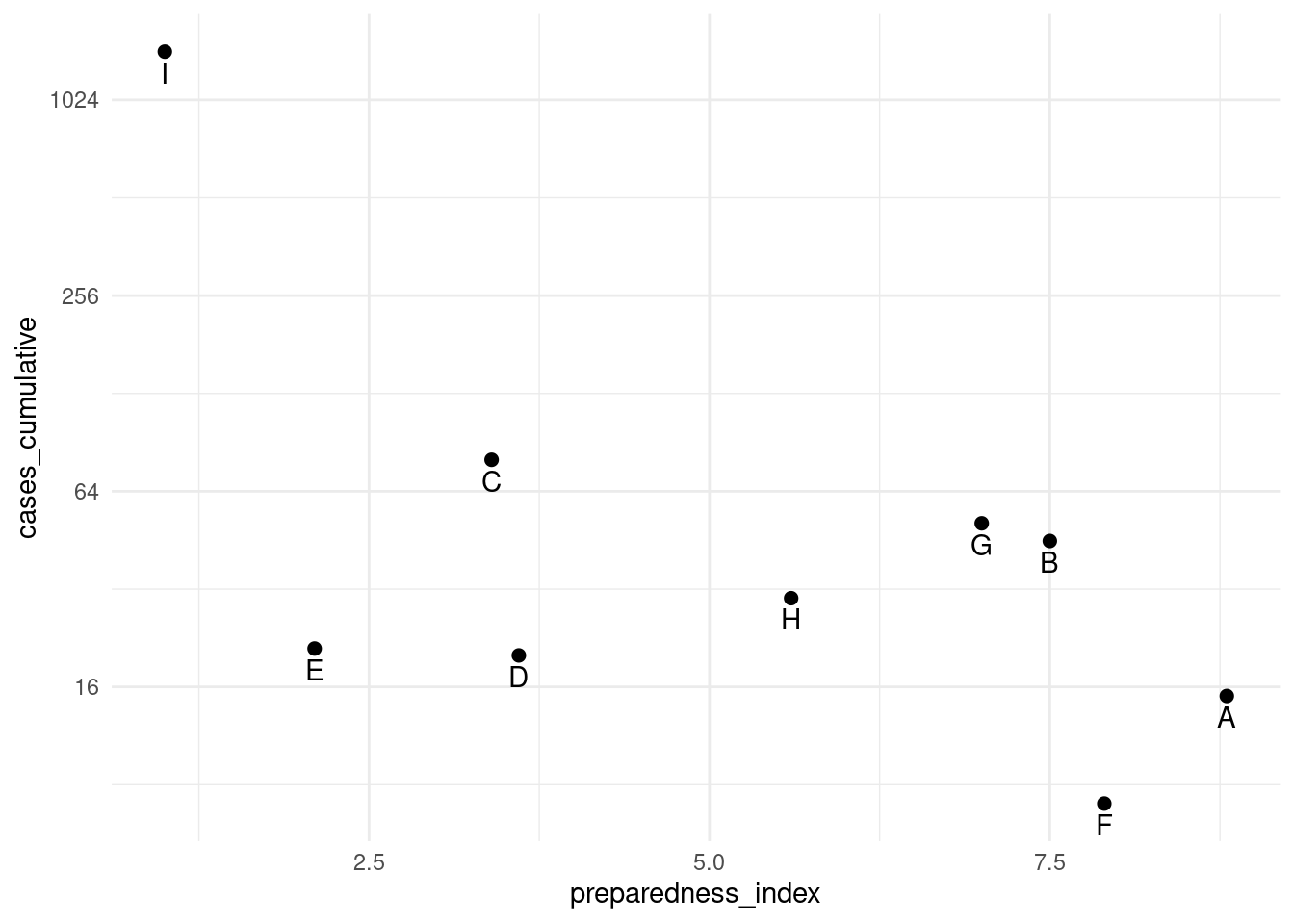
## 9 I 1.0 1442Los casos acumulados de la región “I” son mucho mayores que los de las demás regiones. En circunstancias como ésta, puedes optar por mostrar el eje-y utilizando una escala logarítmica para que el lector pueda ver las diferencias entre las regiones con menos casos acumulados.
# Eje-y original
preparedness_plot <- ggplot(data = plot_data,
mapping = aes(
x = preparedness_index,
y = cases_cumulative))+
geom_point(size = 2)+ # puntos para cada región
geom_text(
mapping = aes(label = region),
vjust = 1.5)+ # añade etiquetas de texto
theme_minimal()
preparedness_plot # imprime el gráfico original
# imprime con el eje-y transformado
preparedness_plot+ # comienza con el gráfico anterior
scale_y_continuous(trans = "log2") # añade transformación al eje-y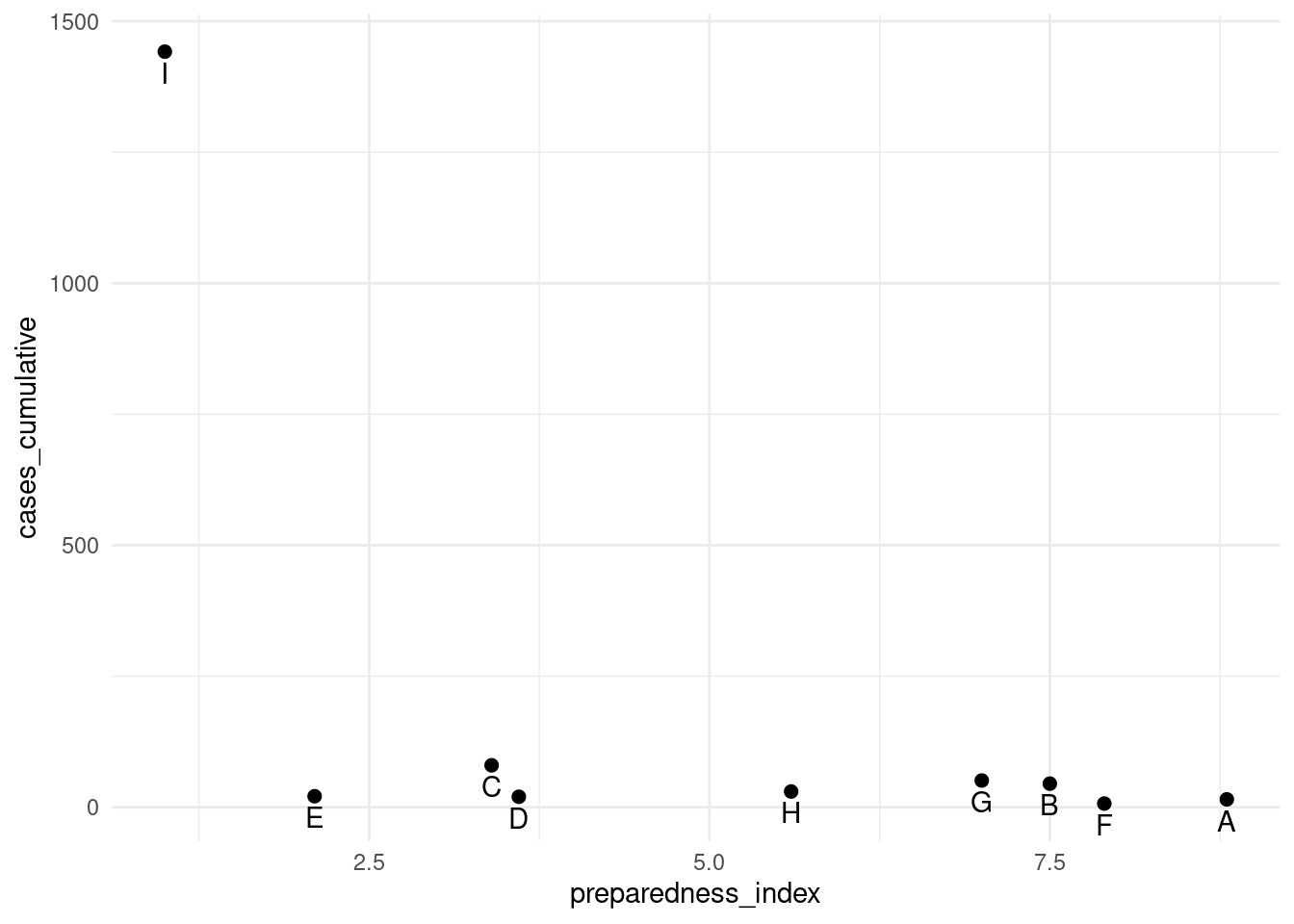
Escalas de gradiente
Las escalas de degradado de relleno pueden implicar matices adicionales. Los valores predeterminados suelen ser bastante agradables, pero es posible que desees ajustar los valores, los cortes, etc.
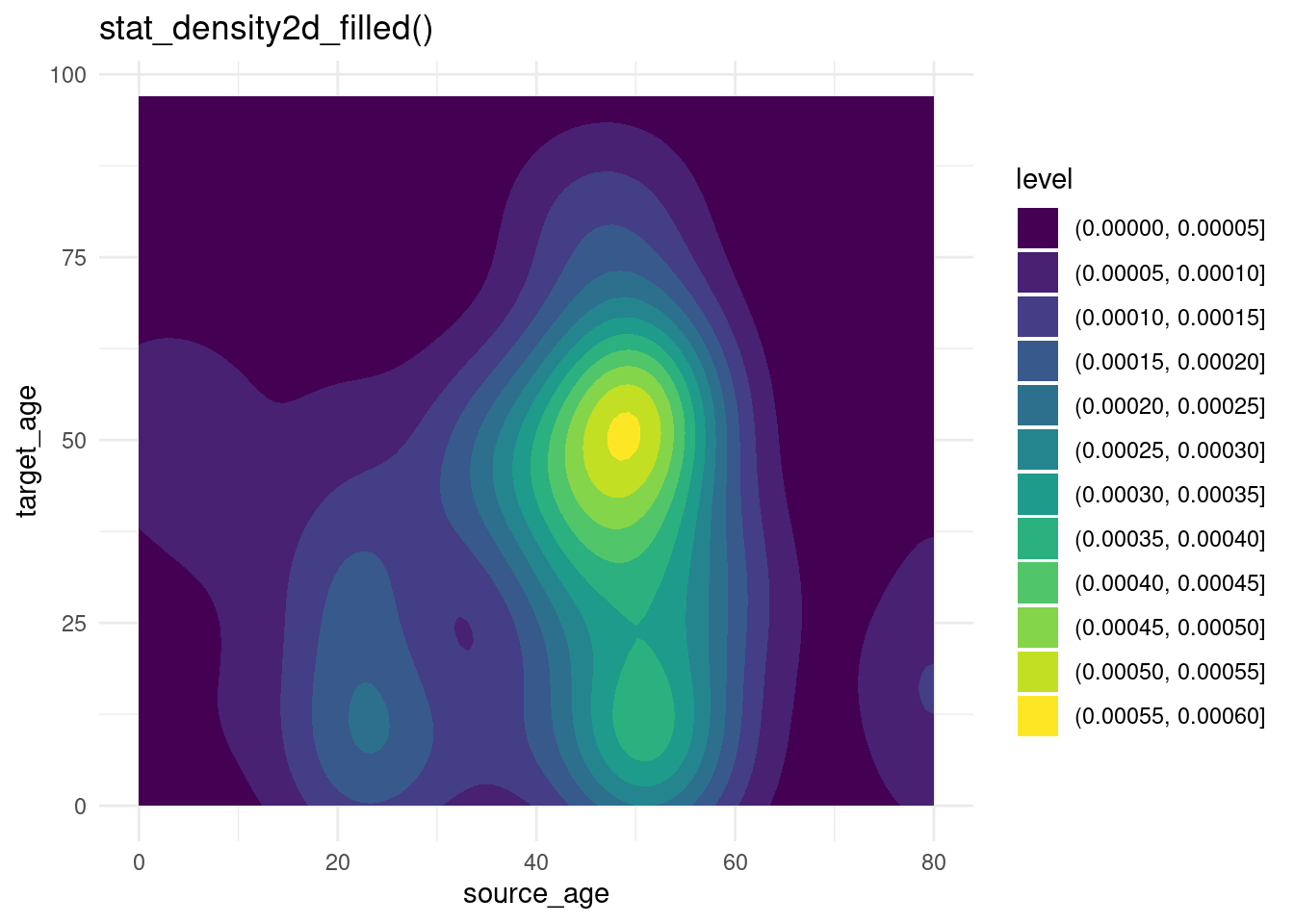
Para demostrar cómo ajustar una escala de colores continua, utilizaremos unos datos de la página de Rastreo de contactos que contiene las edades de los casos y de sus casos de origen.
case_source_relationships <- rio::import(here::here("data", "godata", "relationships_clean.rds")) %>%
select(source_age, target_age) A continuación, producimos un gráfico de densidad de mapa de calor “rasterizado”. No vamos a desarrollar cómo (ver el enlace en el párrafo anterior), pero nos centraremos en cómo podemos ajustar la escala de colores. Lee más sobre la función stat_density2d() de ggplot2 aquí. Observa cómo la escala de relleno es continua.
trans_matrix <- ggplot(
data = case_source_relationships,
mapping = aes(x = source_age, y = target_age))+
stat_density2d(
geom = "raster",
mapping = aes(fill = after_stat(density)),
contour = FALSE)+
theme_minimal()Ahora mostramos algunas variaciones en la escala de relleno:
trans_matrix
trans_matrix + scale_fill_viridis_c(option = "plasma")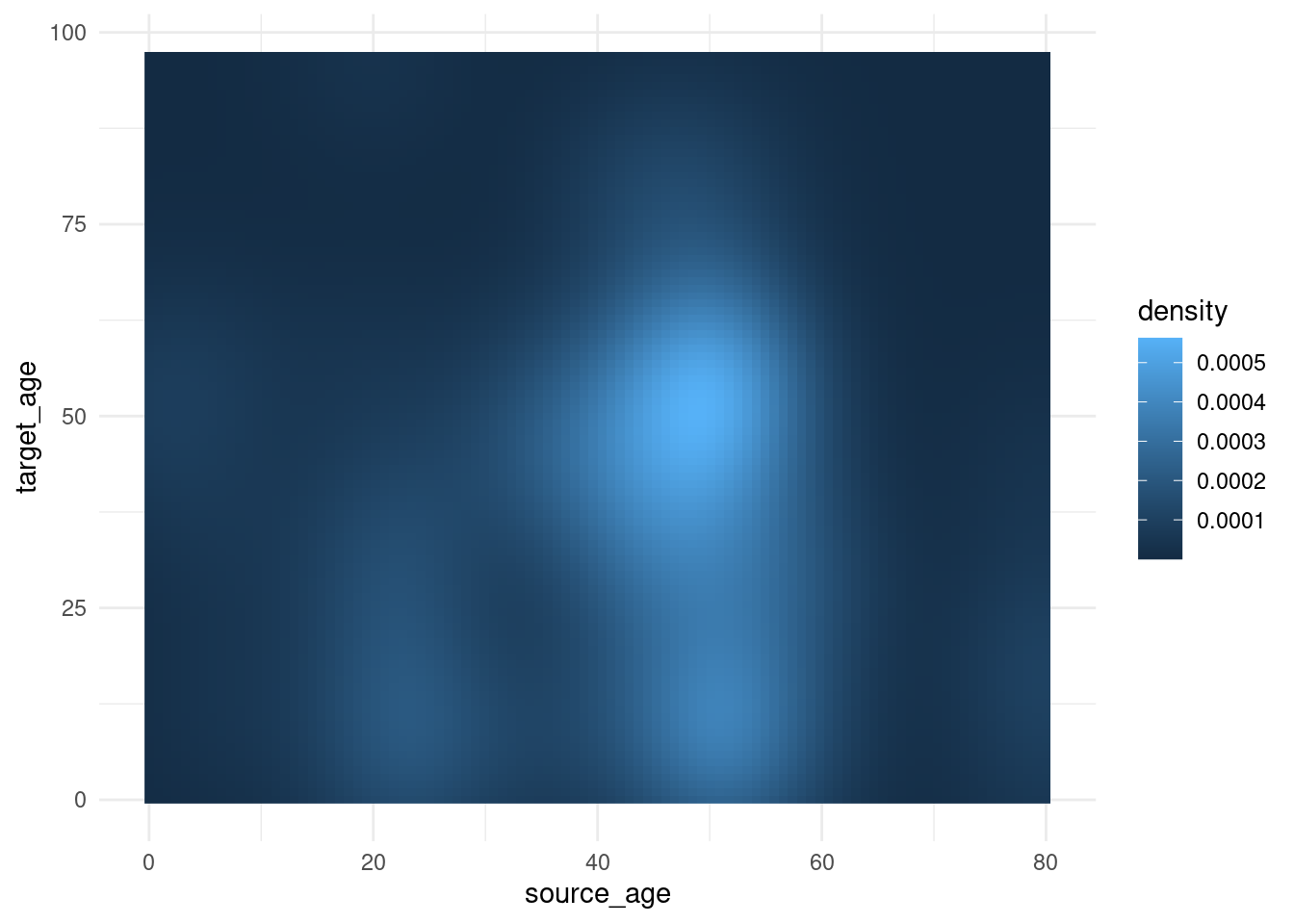
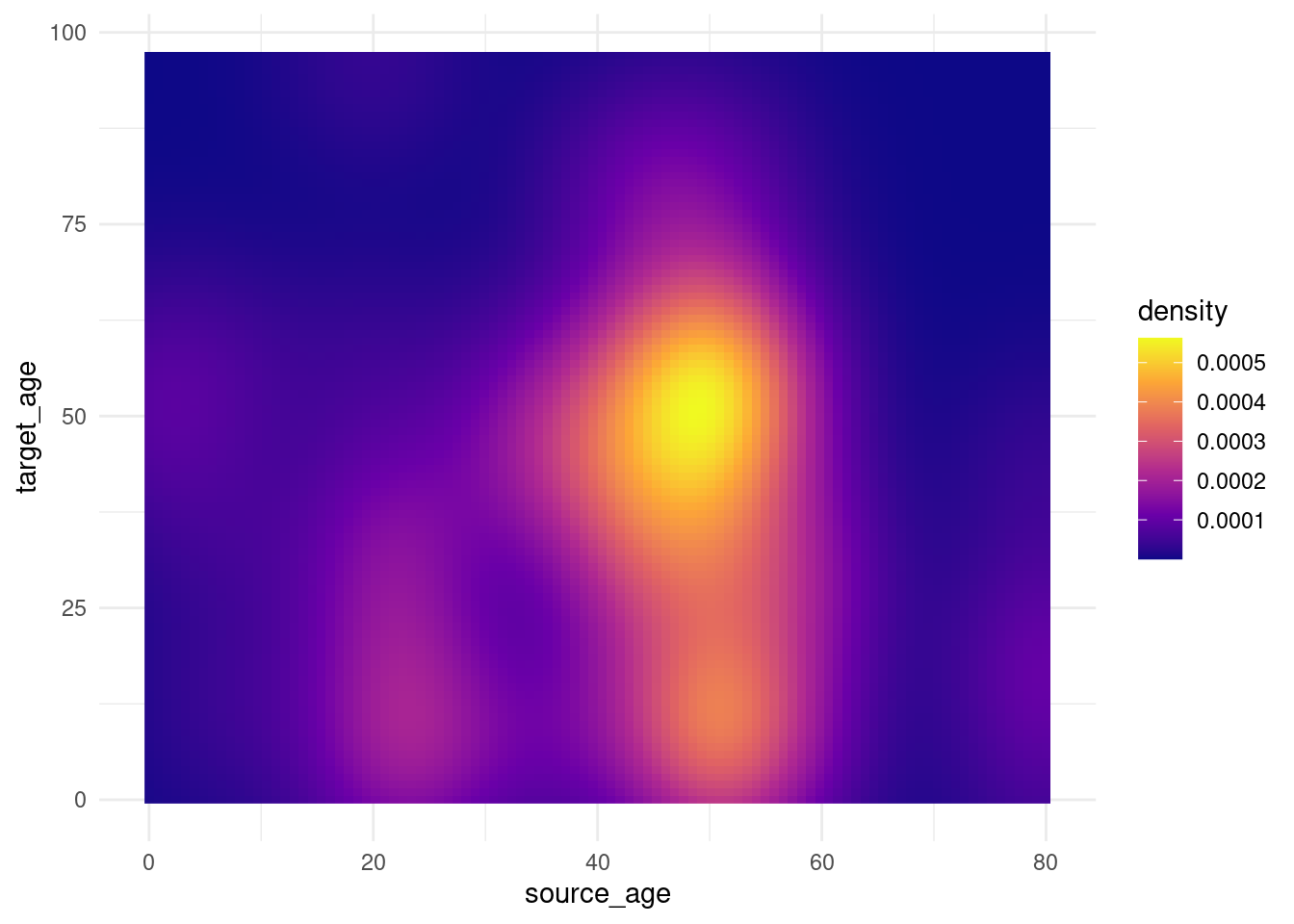
Ahora mostramos algunos ejemplos de cómo ajustar realmente los puntos de ruptura de la escala:
-
scale_fill_gradient()acepta dos colores (high/low) -
scale_fill_gradientn()acepta un vector de cualquier longitud de colores avalues =(los valores intermedios serán interpolados) - Usa scales::rescale() para ajustar la posición de los colores a lo largo del gradiente; reescala tu vector de posiciones para que esté entre 0 y 1.
trans_matrix +
scale_fill_gradient( # escala de gradiente de 2 lados
low = "aquamarine", # valor bajo
high = "purple", # valor alto
na.value = "grey", # valor para NA
name = "Density")+ # Título de la leyenda
labs(title = "Manually specify high/low colors")
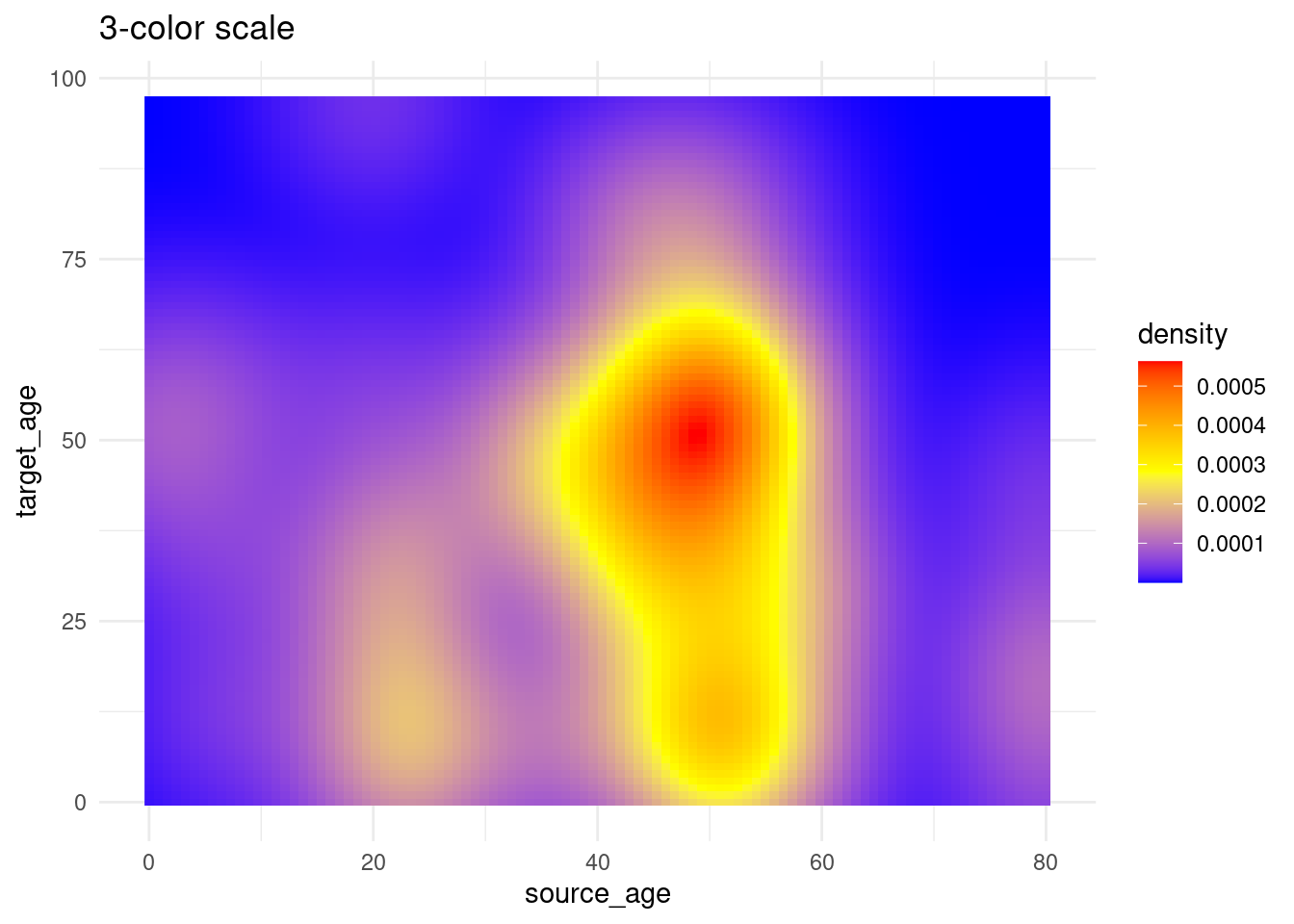
# 3+ colores en la escala
trans_matrix +
scale_fill_gradientn( # escala de 3 colores (bajo/medio/alto)
colors = c("blue", "yellow","red") # proporciona los colores en el vector
)+
labs(title = "3-color scale")
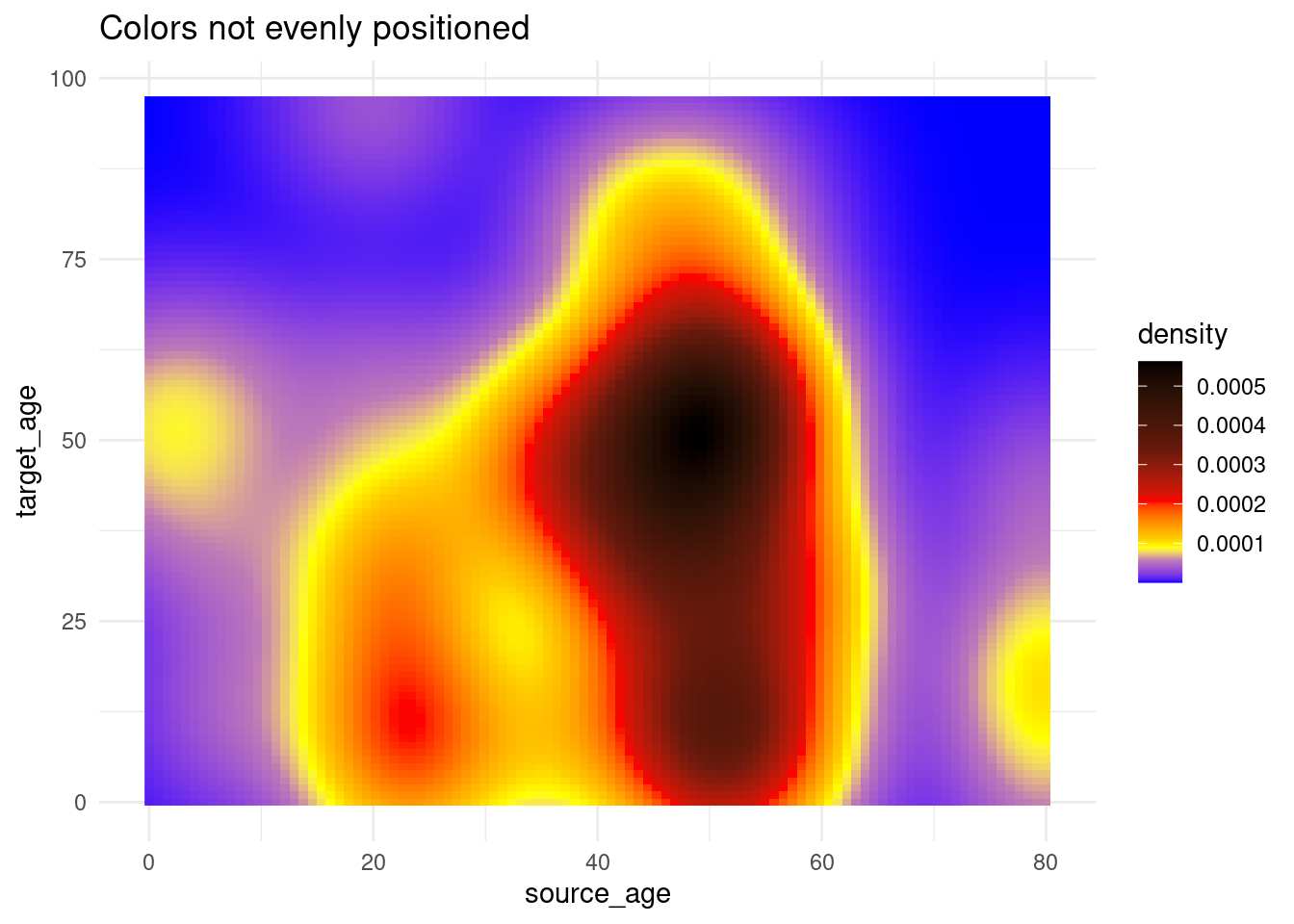
# Uso de rescale() para ajustar la colocación de los colores a lo largo de la escala
trans_matrix +
scale_fill_gradientn( # proporciona cualquier número de colores
colors = c("blue", "yellow","red", "black"),
values = scales::rescale(c(0, 0.05, 0.07, 0.10, 0.15, 0.20, 0.3, 0.5)) # las posiciones de los colores se reescalan entre 0 y 1
)+
labs(title = "Colors not evenly positioned")
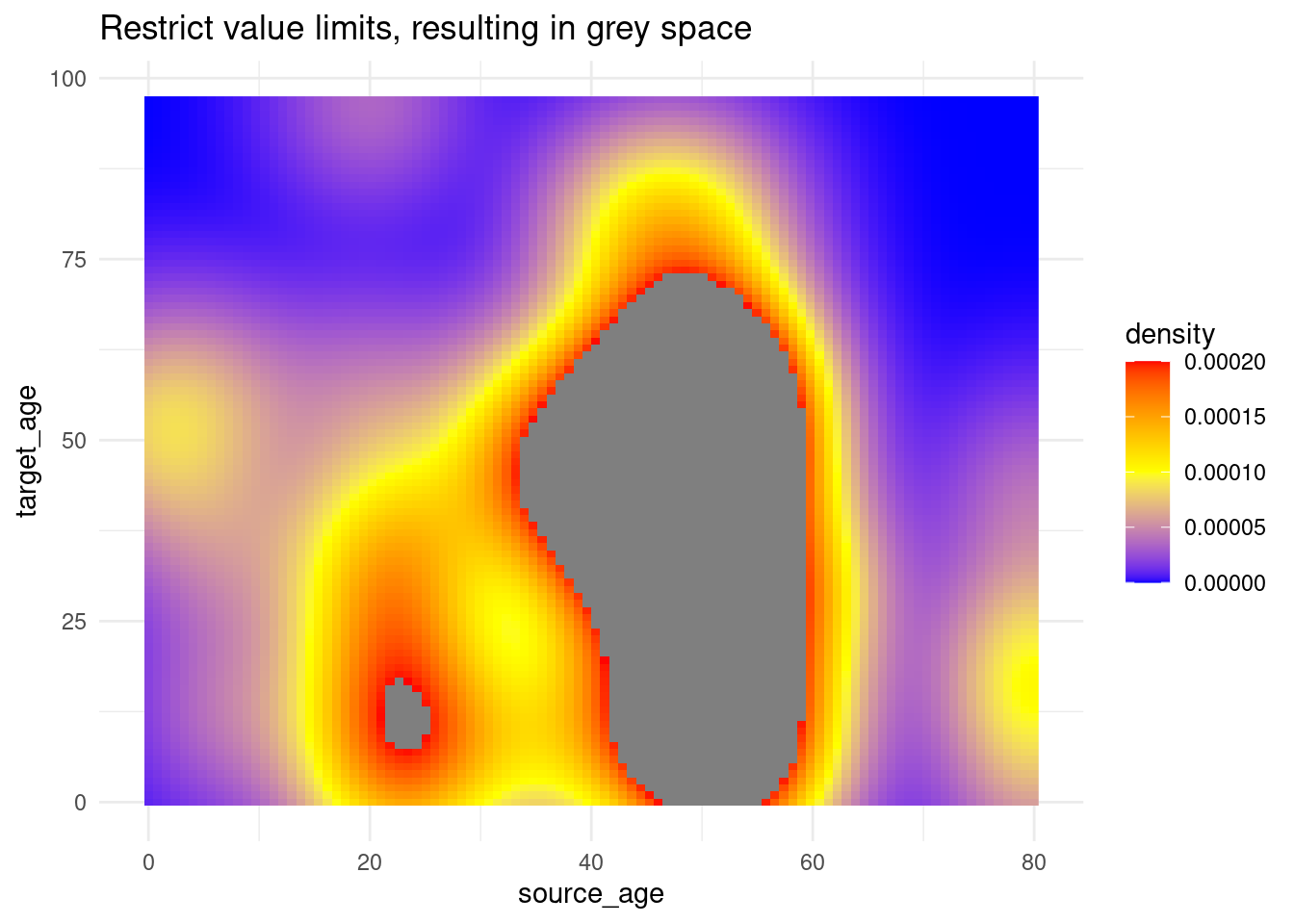
# uso de valores de corte que obtienen color de relleno
trans_matrix +
scale_fill_gradientn(
colors = c("blue", "yellow","red"),
limits = c(0, 0.0002))+
labs(title = "Restrict value limits, resulting in grey space")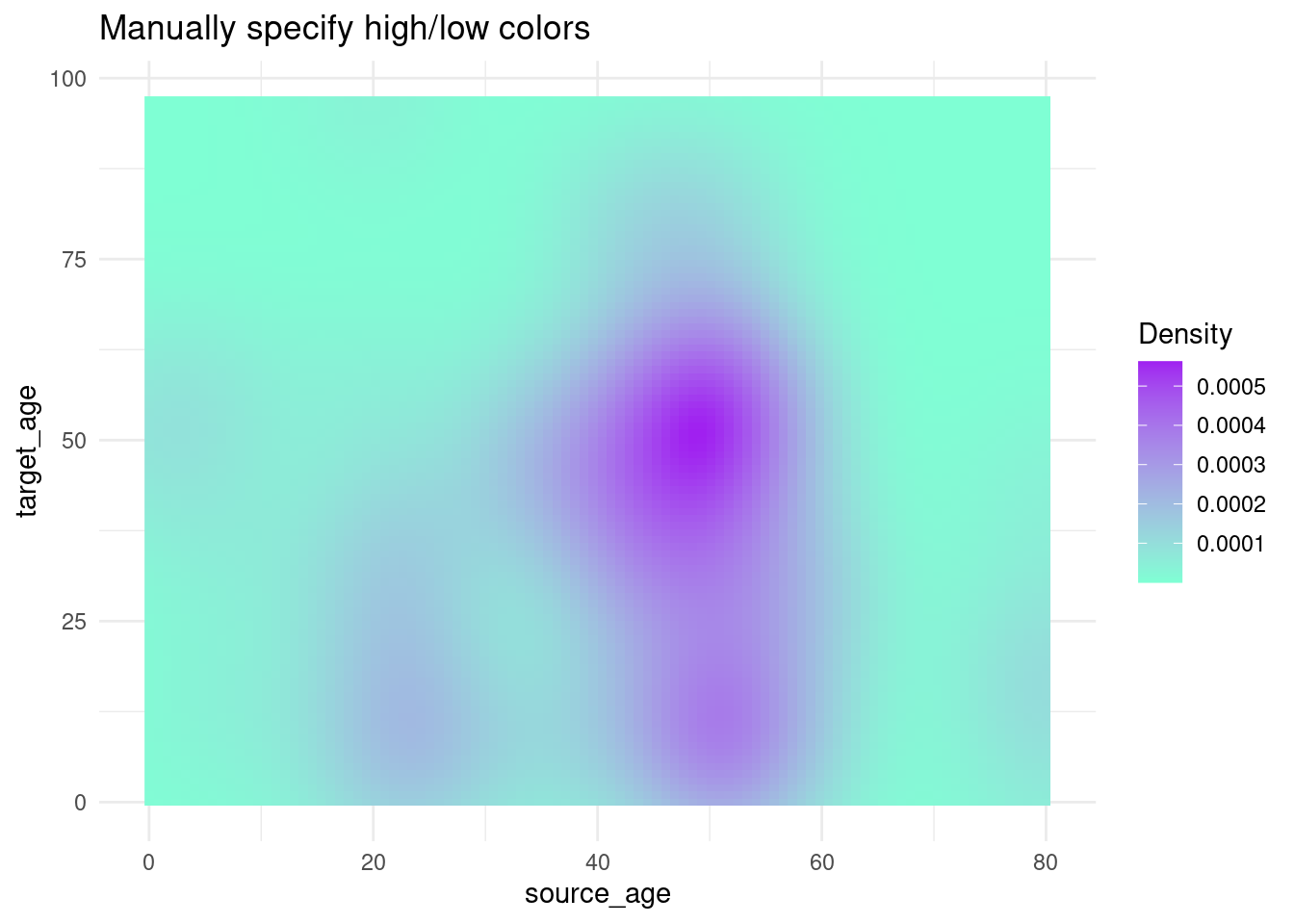
Paletas
Colorbrewer y Viridis
En general, si deseas paletas predefinidas, puedes utilizar las funciones scale_xxx_brewer o scale_xxx_viridis_y.
Las funciones ‘brewer’ pueden dibujar desde las paletas de colorbrewer.org.
Las funciones ‘viridis’ se basan en las paletas viridis (¡difíciles para los daltónicos!), que “proporcionan mapas de color que son perceptivamente uniformes tanto en color como en blanco y negro. También están diseñadas para ser percibidas por espectadores con formas comunes de daltonismo”. (lee más aquí y aquí). Define si la paleta es discreta, continua o con intervalos especificando esto al final de la función (por ejemplo, discreta es scale_xxx_viridis_d).
Se aconseja que pruebes tu esquema en este simulador de daltonismo. Si tienes un esquema de color rojo/verde, prueba en su lugar un esquema “caliente-frío” (rojo-azul) como se describe aquí
Aquí hay un ejemplo de la página de Conceptos básicos de ggplot, utilizando varios esquemas de color.
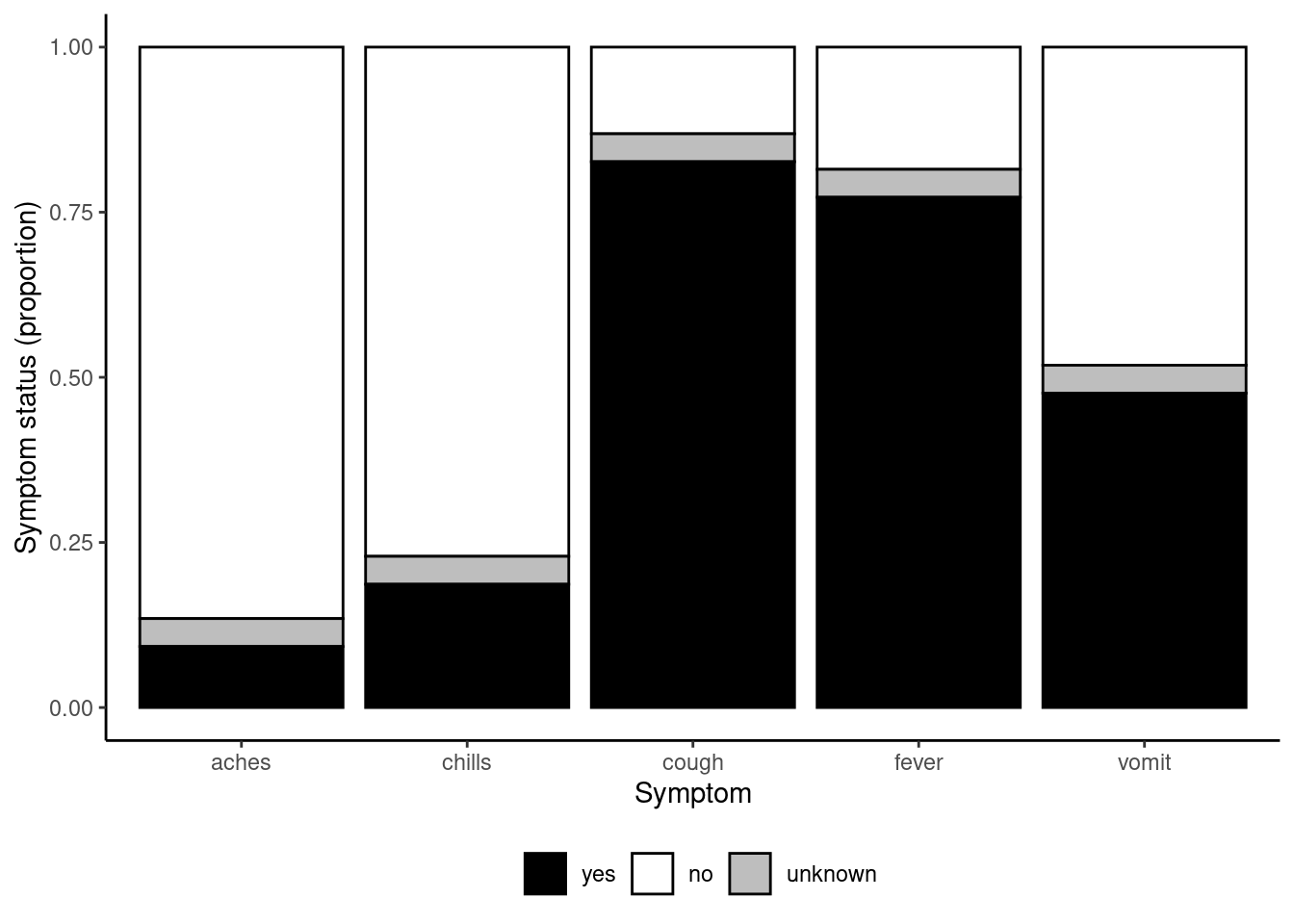
symp_plot <- linelist %>% # comienza con linelist
select(c(case_id, fever, chills, cough, aches, vomit)) %>% # selecciona columnas
pivot_longer( # pivotea largo
cols = -case_id,
names_to = "symptom_name",
values_to = "symptom_is_present") %>%
mutate( # reemplaza los valores faltantes
symptom_is_present = replace_na(symptom_is_present, "unknown")) %>%
ggplot( # ¡comienza ggplot!
mapping = aes(x = symptom_name, fill = symptom_is_present))+
geom_bar(position = "fill", col = "black") +
theme_classic() +
theme(legend.position = "bottom")+
labs(
x = "Symptom",
y = "Symptom status (proportion)"
)
symp_plot # imprime con colores por defecto
#################################
# imprimir con colores especificados manualmente
symp_plot +
scale_fill_manual(
values = c("yes" = "black", # define explícitamente los colores
"no" = "white",
"unknown" = "grey"),
breaks = c("yes", "no", "unknown"), # ordena los factores correctamente
name = "" # establece la leyenda sin título
)
#################################
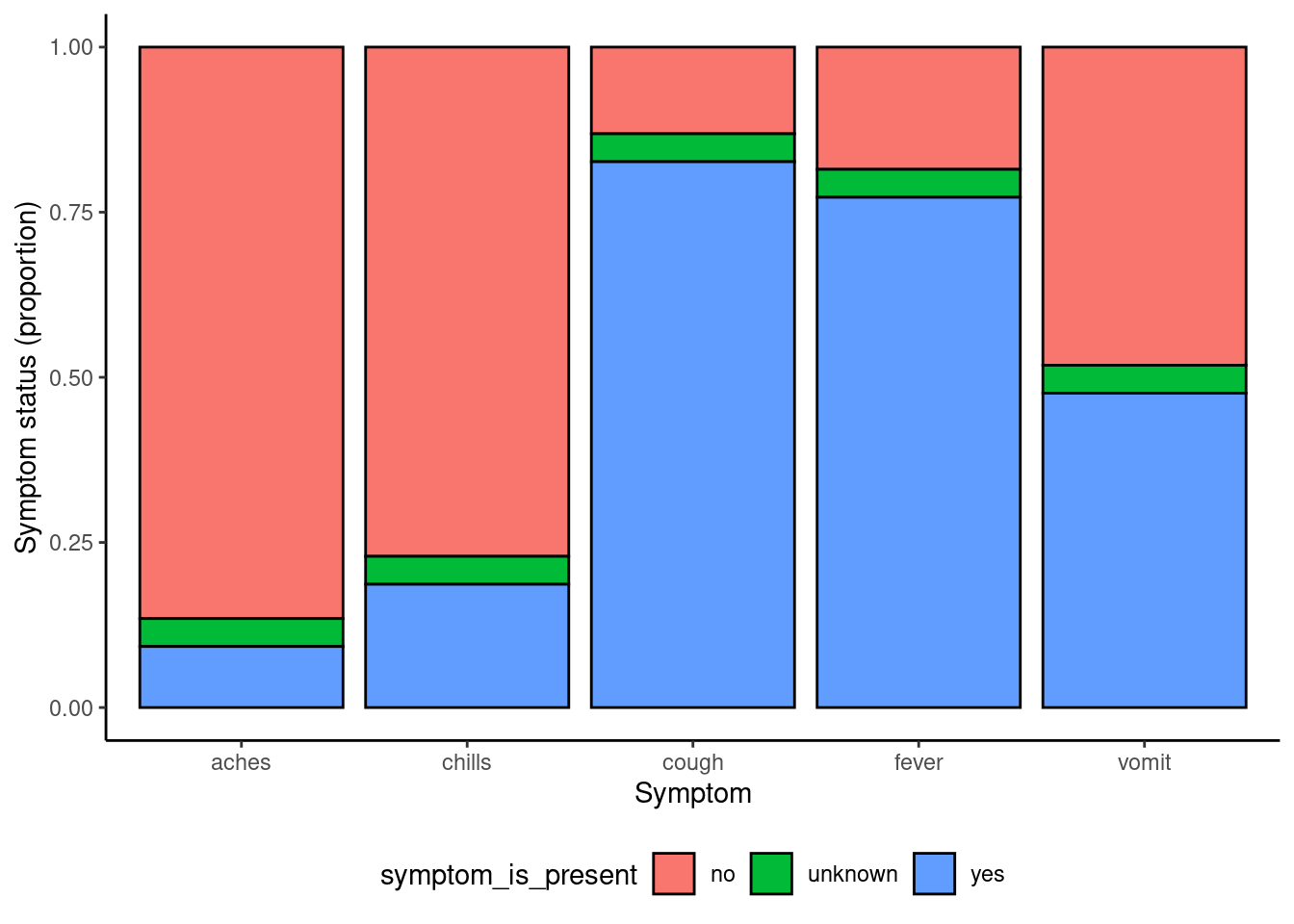
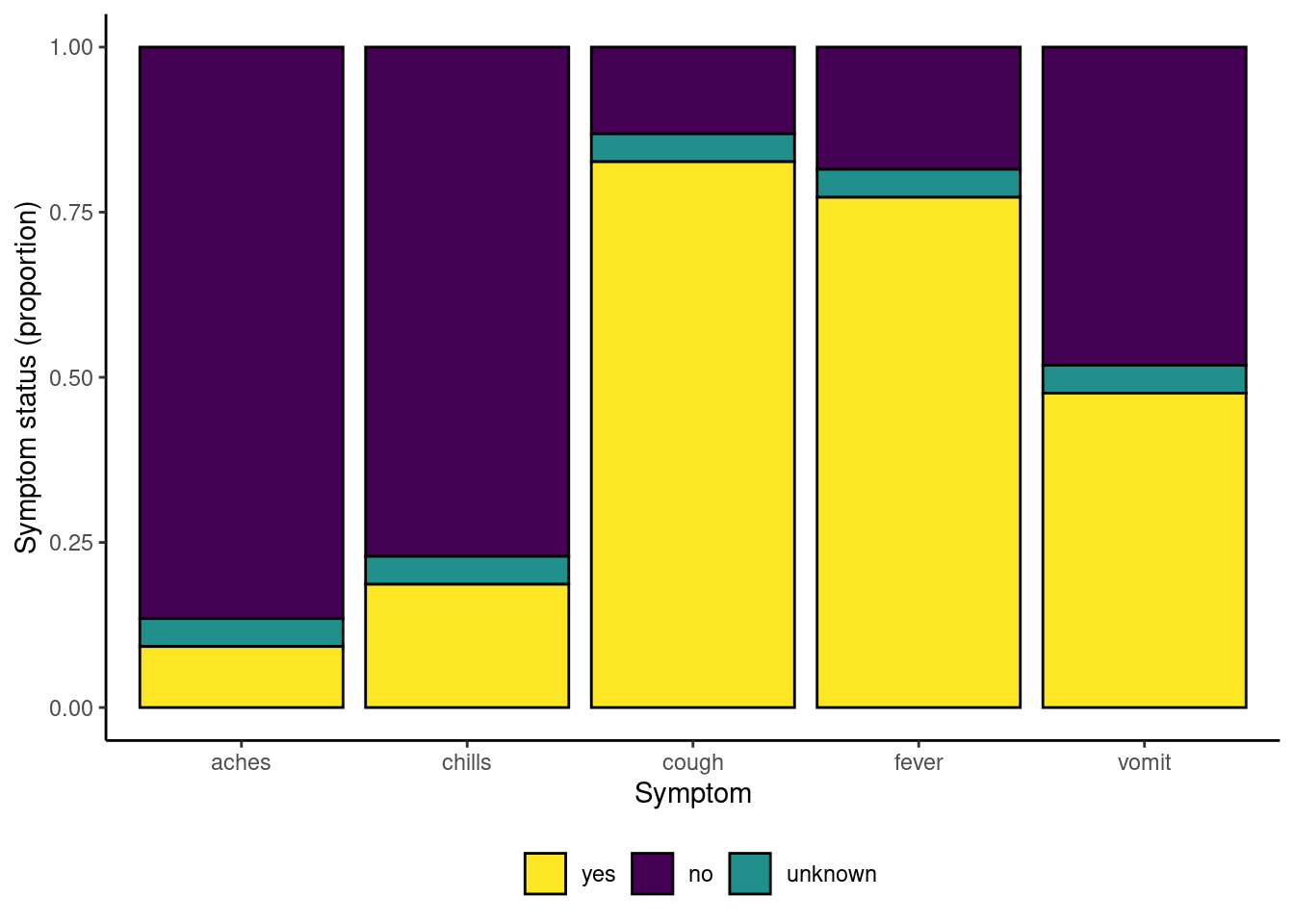
# imprimir con colores discretos viridis
symp_plot +
scale_fill_viridis_d(
breaks = c("yes", "no", "unknown"),
name = ""
)
31.3 Cambiar el orden de las variables discretas
Cambiar el orden en que aparecen las variables discretas es a menudo difícil de entender para las personas que son nuevas en los gráficos de ggplot2. Sin embargo, es fácil de entender cómo hacer esto una vez que se entiende cómo ggplot2 maneja las variables discretas por debajo. En general, si se utiliza una variable discreta, se convierte automáticamente en un tipo de factor - que ordena los factores por orden alfabético por defecto. Para manejar esto, simplemente tienes que reordenar los niveles de los factores para reflejar el orden en que te gustaría que aparecieran en el gráfico. Para obtener información más detallada sobre cómo reordenar los objetos de factor, consulta la página Factores.
Podemos ver un ejemplo común utilizando los grupos de edad - por defecto el grupo de 5 a 9 años se colocará en medio de los grupos de edad (dado el orden alfanumérico), pero podemos moverlo detrás del grupo de 0 a 4 años del gráfico renivelando los factores.
ggplot(
data = linelist %>% drop_na(age_cat5), # elimina las filas en las que falta age_cat5
mapping = aes(x = fct_relevel(age_cat5, "5-9", after = 1))) + # factor de renivelación
geom_bar() +
labs(x = "Age group", y = "Number of hospitalisations",
title = "Total hospitalisations by age group") +
theme_minimal()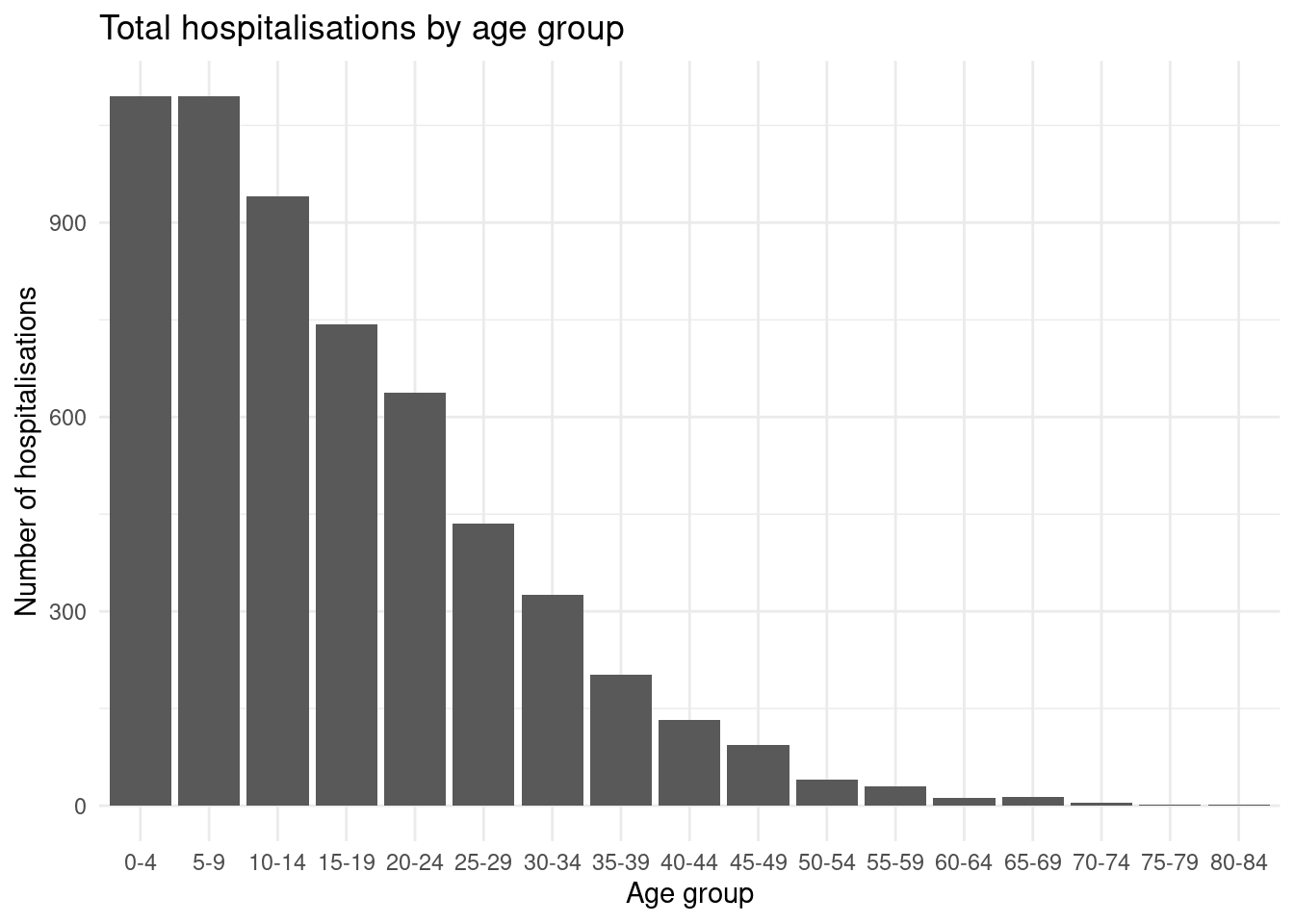
31.3.0.1 ggthemr
También considera utilizar el paquete ggthemr. Puedes descargar este paquete desde Github usando estas instrucciones. Ofrece paletas que son muy agradables estéticamente, pero ten en cuenta que estas suelen tener un número máximo de valores que puede ser limitante si quieres más de 7 u 8 colores.
31.4 Líneas de contorno
Los gráficos de contorno son útiles cuando se tienen muchos puntos que pueden cubrirse unos a otros (“sobretrazado”). Los datos de la fuente de casos utilizados anteriormente se trazan de nuevo, pero de forma más sencilla utilizando stat_density2d() y stat_density2d_filled() para producir niveles de contorno discretos - como un mapa topográfico. Lee más sobre las estadísticas aquí.
case_source_relationships %>%
ggplot(aes(x = source_age, y = target_age))+
stat_density2d()+
geom_point()+
theme_minimal()+
labs(title = "stat_density2d() + geom_point()")
case_source_relationships %>%
ggplot(aes(x = source_age, y = target_age))+
stat_density2d_filled()+
theme_minimal()+
labs(title = "stat_density2d_filled()")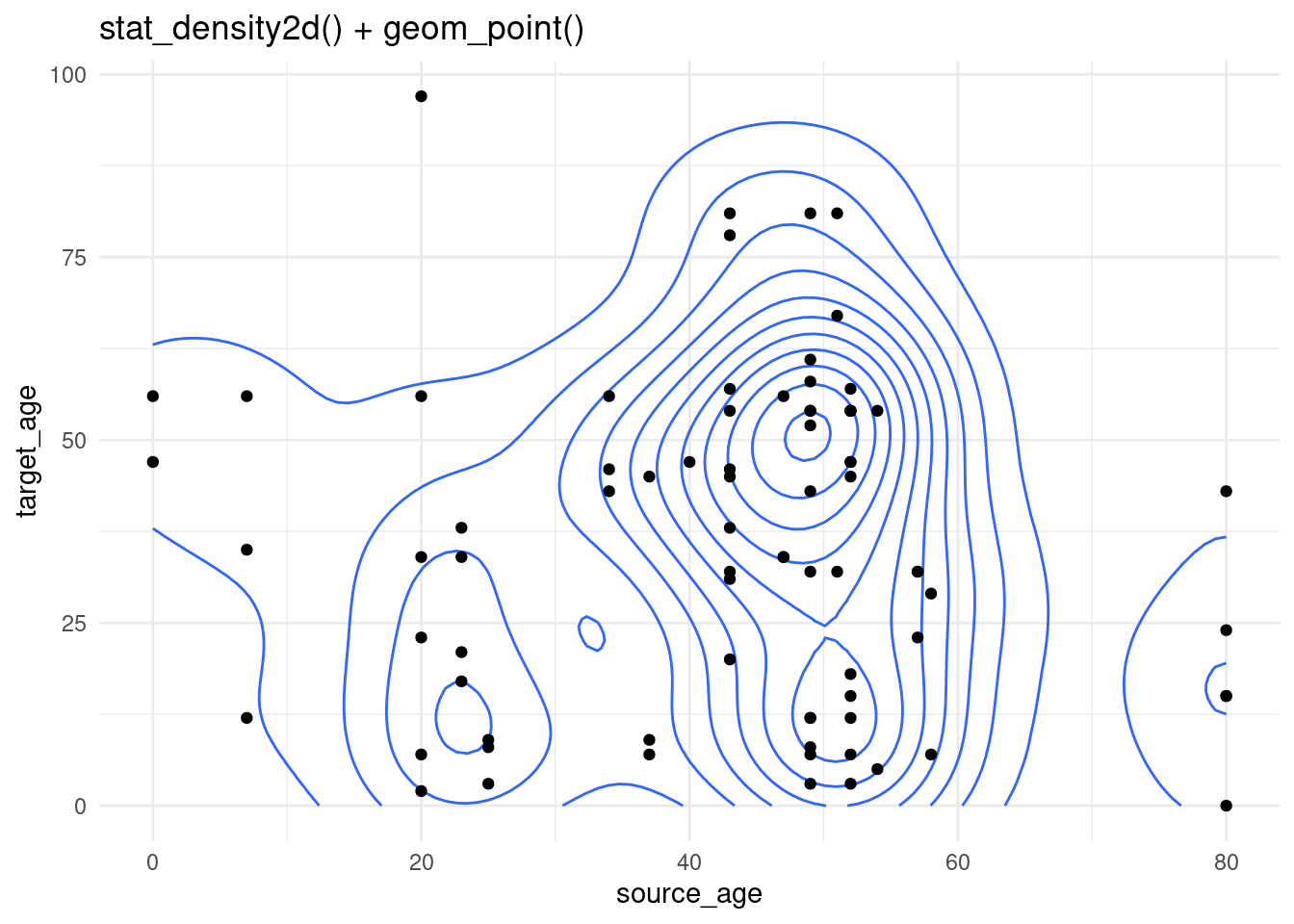
31.5 Distribuciones marginales
Para mostrar las distribuciones en los bordes de un gráfico de dispersión geom_point(), puedes utilizar el paquete ggExtra y su función ggMarginal(). Guarda tu ggplot original como un objeto, y pásalo a ggMarginal() como se muestra a continuación. Estos son los argumentos clave:
- Debe especificar el
type =como “histogram”, “density” “boxplot”, “violin”, o “densigram” - Por defecto, los gráficos marginales aparecerán para ambos ejes. Puedes establecer
margins =a “x” o “y” si sólo quieres uno. - Otros argumentos opcionales son
fill =(color de la barra),color =(color de la línea),size =(tamaño del gráfico en relación con el tamaño del margen, por lo que un número mayor hace que el gráfico marginal sea más pequeño). - Puedes proporcionar otros argumentos específicos del eje a
xparams =eyparams =. Por ejemplo, para tener diferentes tamaños de cubos de histograma, como se muestra a continuación.
Puedes hacer que los gráficos marginales reflejen grupos (columnas a las que se les ha asignado un color = en su estética mapeada de ggplot()). Si este es el caso, establece el argumento ggMarginal() groupColour = o groupFill = a TRUE, como se muestra a continuación.
Lee más en esta viñeta, en la galería de gráficos de R o en la documentación de la función R ?ggMarginal.
# Instalar/cargar ggExtra
pacman::p_load(ggExtra)
# Gráfico básico de dispersión de peso y edad
scatter_plot <- ggplot(data = linelist)+
geom_point(mapping = aes(y = wt_kg, x = age)) +
labs(title = "Scatter plot of weight and age")Para añadir histogramas marginales utiliza type = "histogram". Opcionalmente puedes establecer groupFill = TRUE para obtener histogramas apilados.
# con histogramas
ggMarginal(
scatter_plot, # añade histogramas marginales
type = "histogram", # especifica histogramas
fill = "lightblue", # relleno de barras
xparams = list(binwidth = 10), # otros parámetros para el eje-x marginal
yparams = list(binwidth = 5)) # otros parámetros para el eje-y marginal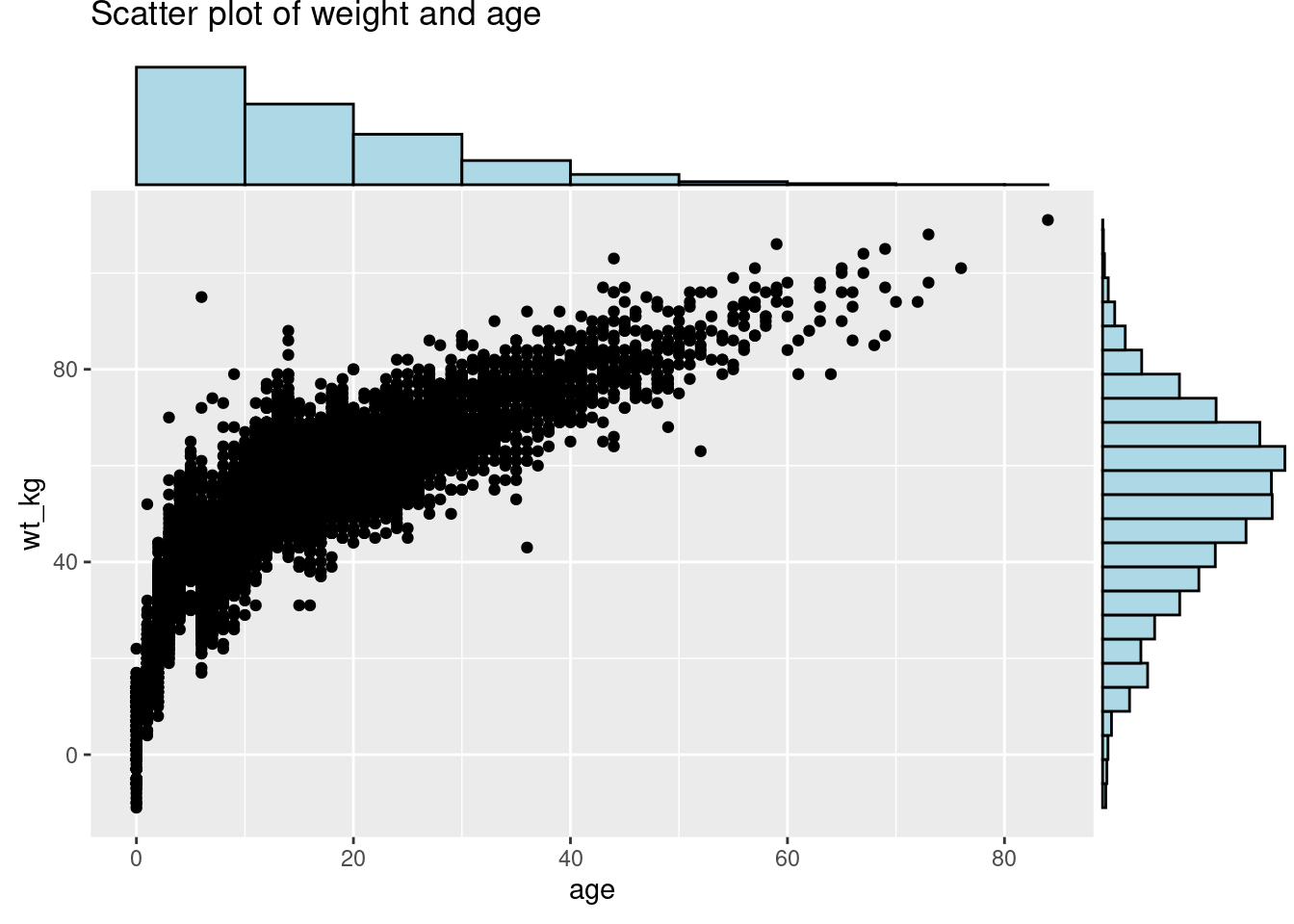
Gráfico de densidad marginal con valores agrupados/coloreados:
# Diagrama de dispersión, coloreado por resultado
# La columna Outcome se asigna como color en ggplot. groupFill en ggMarginal se establece en TRUE
scatter_plot_color <- ggplot(data = linelist %>% drop_na(gender))+
geom_point(mapping = aes(y = wt_kg, x = age, color = gender)) +
labs(title = "Scatter plot of weight and age")+
theme(legend.position = "bottom")
ggMarginal(scatter_plot_color, type = "density", groupFill = TRUE)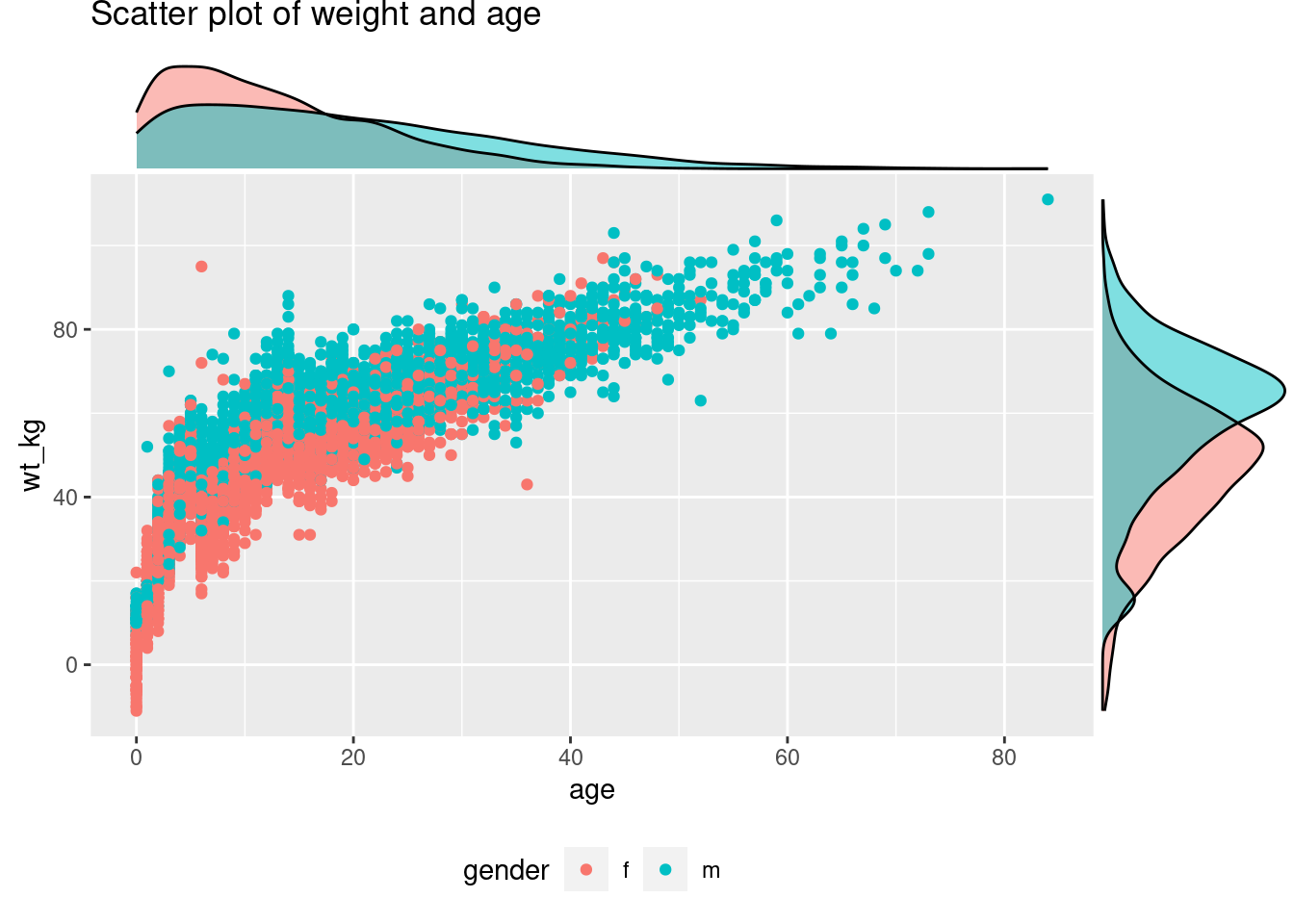
Establece el argumento size = para ajustar el tamaño relativo del gráfico marginal. Un número más pequeño hace un gráfico marginal más grande. También se establece el color =. A continuación se muestra un boxplot marginal, con la demostración del argumento margins = por lo que aparece en un solo eje:
# con boxplot
ggMarginal(
scatter_plot,
margins = "x", # sólo muestra el gráfico marginal del eje-x
type = "boxplot") 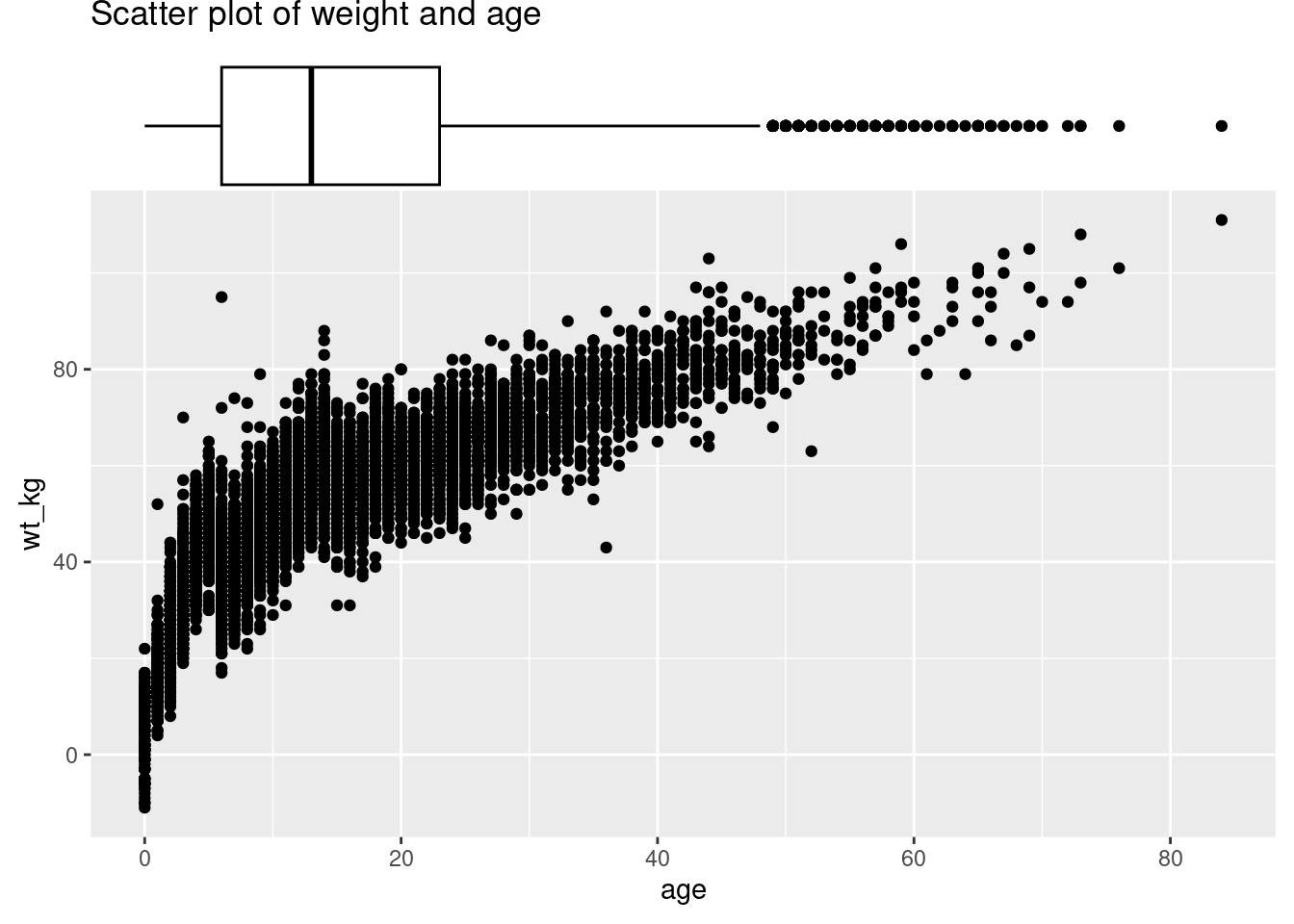
31.6 Etiquetado inteligente
En ggplot2, también es posible añadir texto a los gráficos. Sin embargo, esto viene con la notable limitación de que las etiquetas de texto a menudo chocan con los puntos de datos en un gráfico, haciendo que se vean desordenados o difíciles de leer. No hay una manera ideal de lidiar con esto en el paquete base, pero hay un complemento de ggplot2, conocido como ggrepel que hace que esto sea muy simple.
El paquete ggrepel proporciona dos nuevas funciones, geom_label_repel() y geom_text_repel(), que sustituyen a geom_label() y geom_text(). Basta con utilizar estas funciones en lugar de las funciones base para producir etiquetas ordenadas. Dentro de la función, mapea la estética aes() como siempre, pero incluye el argumento label = al que le proporciona un nombre de columna que contenga los valores que deseas mostrar (por ejemplo, id de paciente, o nombre, etc.). Puedes hacer etiquetas más complejas combinando columnas y nuevas líneas (\n) dentro de str_glue() como se muestra a continuación.
Algunos consejos:
- Utiliza
min.segment.length = 0para dibujar siempre segmentos de línea, omin.segment.length = Infpara no dibujarlos nunca - Utiliza
size =fuera deaes()para establecer el tamaño del texto - Utiliza
force =para cambiar el grado de repulsión entre las etiquetas y sus respectivos puntos (por defecto es 1) - Incluye
fill =dentro deaes()para tener la etiqueta coloreada por el valor- Puede aparecer una letra “a” en la leyenda - añade
guides(fill = guide_legend(override.aes = aes(color = NA)))+para eliminarla
- Puede aparecer una letra “a” en la leyenda - añade
Para verlo con mayor profundidad, consulta este tutorial
pacman::p_load(ggrepel)
linelist %>% # empieza con linelist
group_by(hospital) %>% # agrupa por hospital
summarise( # crea un nuevo conjunto de datos con valores de resumen por hospital
n_cases = n(), # número de casos por hospital
delay_mean = round(mean(days_onset_hosp, na.rm=T),1), # retraso medio por hospital
) %>%
ggplot(mapping = aes(x = n_cases, y = delay_mean))+ # envía los datos a ggplot
geom_point(size = 2)+ # añade puntos
geom_label_repel( # añade etiquetas de puntos
mapping = aes(
label = stringr::str_glue(
"{hospital}\n{n_cases} cases, {delay_mean} days") # cómo se muestra la etiqueta
),
size = 3, # tamaño del texto en las etiquetas
min.segment.length = 0)+ # muestra todos los segmentos de línea
labs( # añade etiquetas a los ejes
title = "Mean delay to admission, by hospital",
x = "Number of cases",
y = "Mean delay (days)")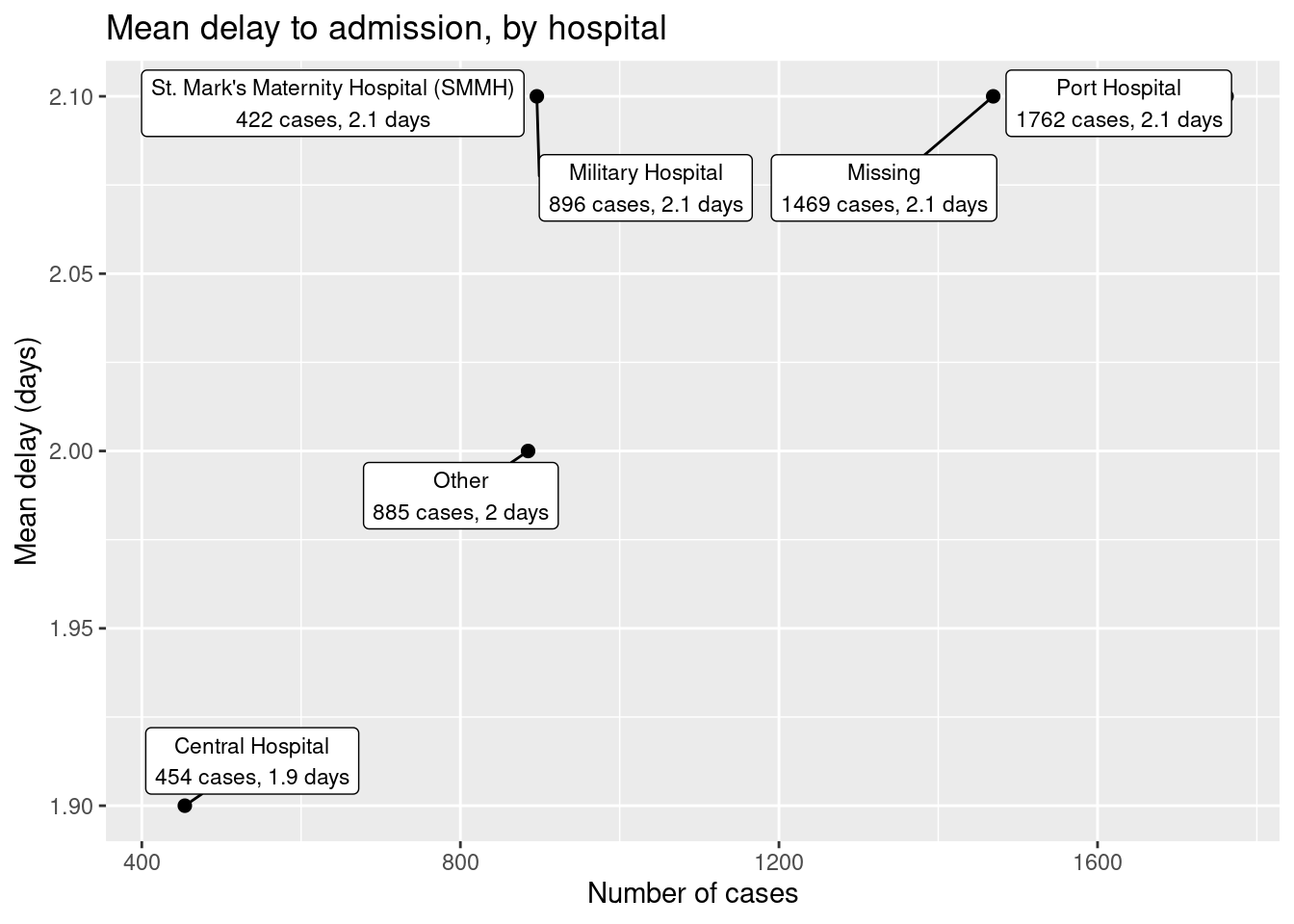
Puedes etiquetar sólo un subconjunto de los puntos de datos - utilizando la sintaxis estándar de ggplot() para proporcionar diferentes data = para cada geom del gráfico. A continuación, se trazan todos los casos, pero sólo se etiquetan algunos.
ggplot()+
# Todos los puntos en gris
geom_point(
data = linelist, # todos los datos proporcionados a esta capa
mapping = aes(x = ht_cm, y = wt_kg),
color = "grey",
alpha = 0.5)+ # gris y semitransparente
# Pocos puntos en negro
geom_point(
data = linelist %>% filter(days_onset_hosp > 15), # datos filtrados proporcionados a esta capa
mapping = aes(x = ht_cm, y = wt_kg),
alpha = 1)+ # por defecto negro y no transparente
# etiquetas para algunos puntos
geom_label_repel(
data = linelist %>% filter(days_onset_hosp > 15), # filtra los datos para las etiquetas
mapping = aes(
x = ht_cm,
y = wt_kg,
fill = outcome, # color de la etiqueta por resultado
label = stringr::str_glue("Delay: {days_onset_hosp}d")), # etiqueta creada con str_glue()
min.segment.length = 0) + # muestra segmentos de línea para todos
# elimina la letra "a" del interior de los cuadros de leyenda
guides(fill = guide_legend(override.aes = aes(color = NA)))+
# etiquetas de los ejes
labs(
title = "Cases with long delay to admission",
y = "weight (kg)",
x = "height(cm)")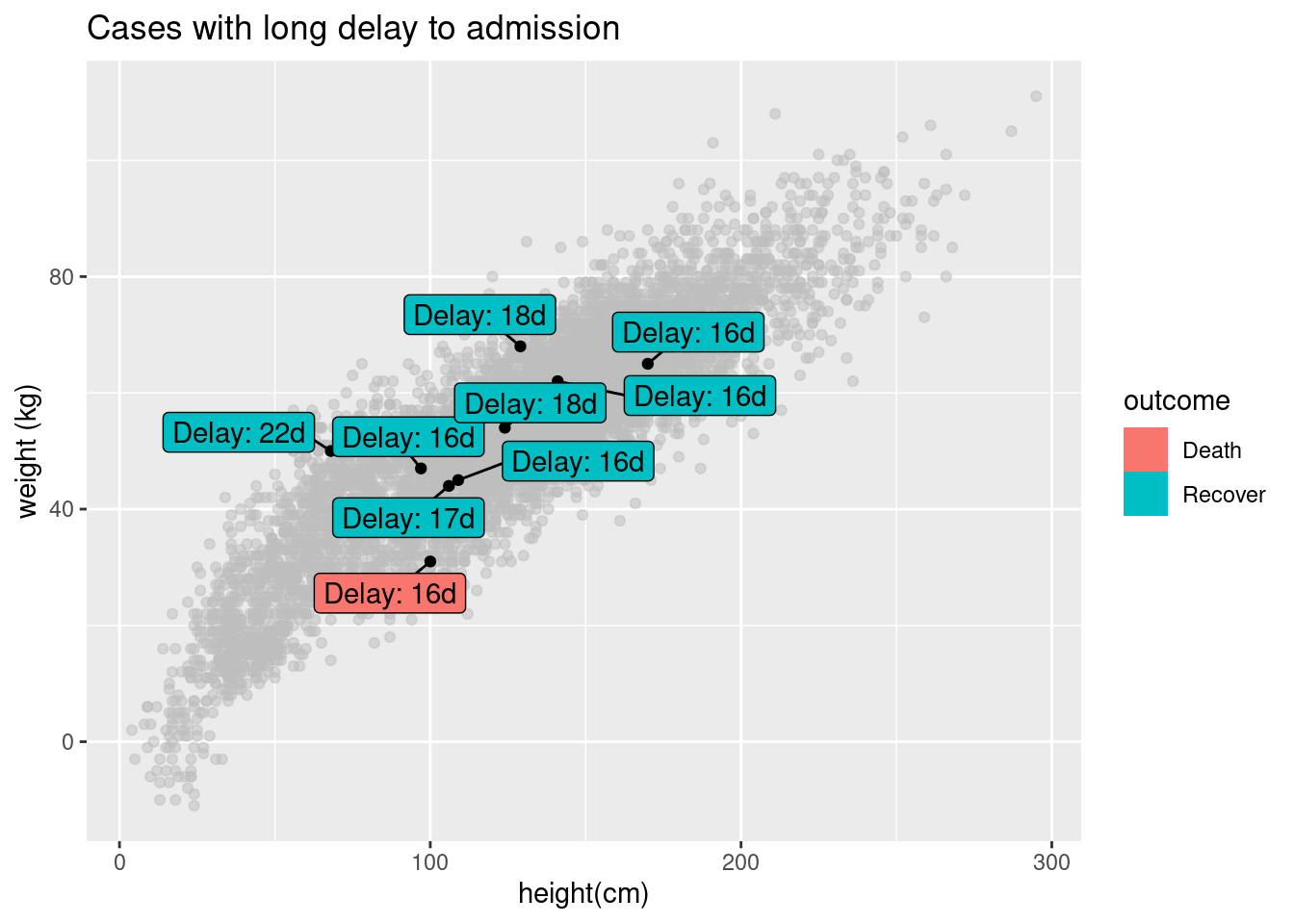
31.7 Ejes temporales
Trabajar con ejes de tiempo en ggplot puede parecer desalentador, pero se hace muy fácil con unas pocas funciones clave. Recuerda que cuando trabajes con el tiempo o la fecha debes asegurarte que las variables correctas están formateadas como tipo date o datetime - mira la página Trabajar con fechas para más información sobre esto, o la página Curvas epidémicas (sección ggplot) para ver ejemplos.
El conjunto de funciones más útil para trabajar con fechas en ggplot2 son las funciones de escala (scale_x_date(), scale_x_datetime(), y sus funciones afines del eje-y). Estas funciones permiten definir la frecuencia de las etiquetas de los ejes, y cómo formatear las etiquetas de los ejes. Para saber cómo dar formato a las fechas, vuelve a ver la sección de trabajar con fechas. Puedes utilizar los argumentos date_breaks y date_labels para especificar el aspecto de las fechas:
date_breakspermite especificar la frecuencia con la que se producen las rupturas de los ejes - se puede pasar aquí una cadena (por ejemplo,"3 months", or “2 days")date_labelspermite definir el formato en el que se muestran las fechas. Puedes pasar una cadena de formato de fecha a estos argumentos (por ejemplo,"%b-%d-%Y"):
# crea la curva epidemiológica por fecha de inicio cuando esté disponible
ggplot(linelist, aes(x = date_onset)) +
geom_histogram(binwidth = 7) +
scale_x_date(
# 1 salto cada 1 mes
date_breaks = "1 months",
# las etiquetas deben mostrar el mes y luego la fecha
date_labels = "%b %d"
) +
theme_classic()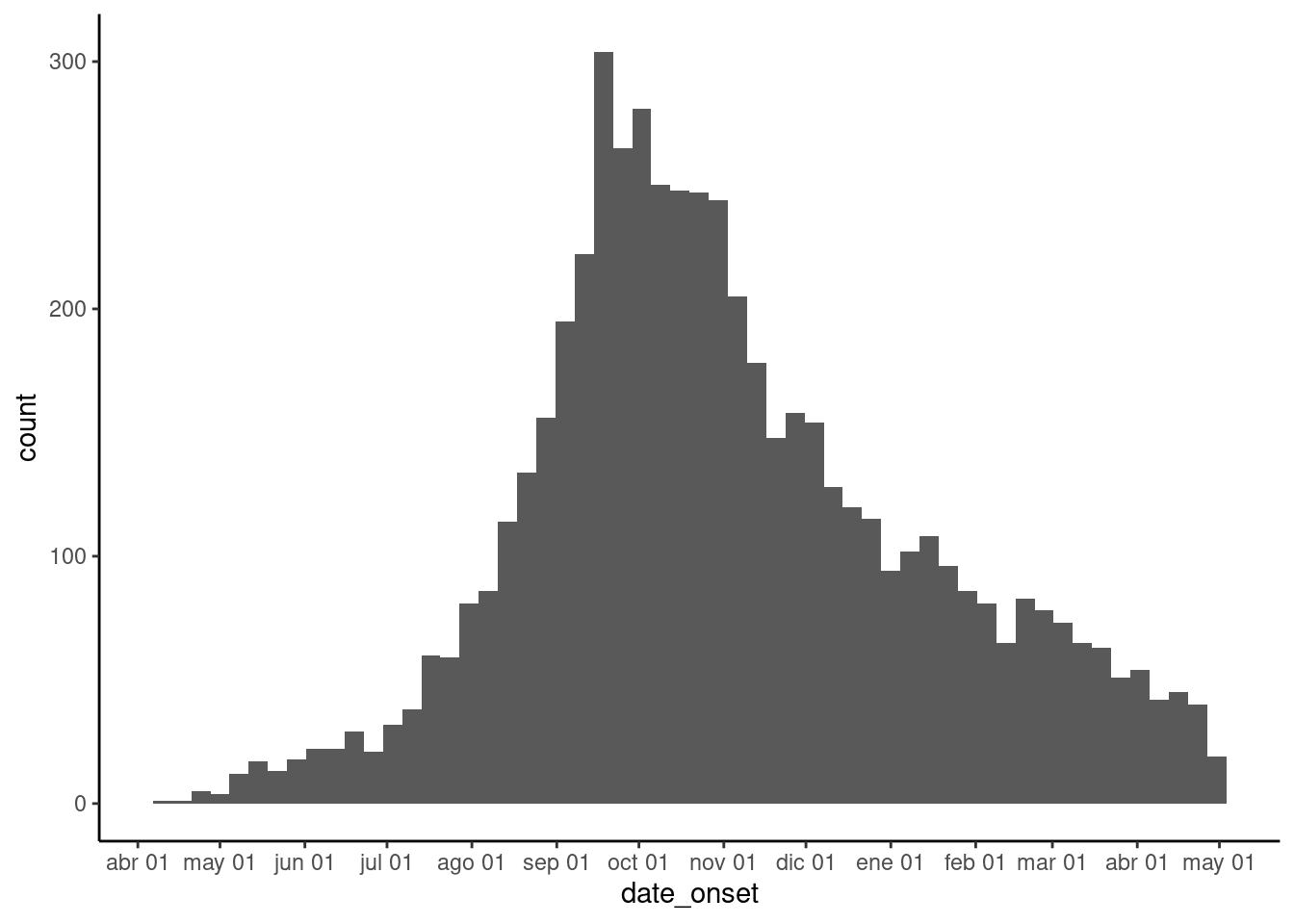
31.8 Resaltando
Resaltar elementos específicos en un gráfico es una forma útil de llamar la atención sobre una instancia específica de una variable, a la vez que se proporciona información sobre la dispersión de los datos. Aunque esto no es fácil de hacer en ggplot2 base, hay un paquete externo que puede ayudar a hacer esto conocido como gghighlight. Es fácil de usar dentro de la sintaxis de ggplot.
El paquete gghighlight utiliza la función gghighlight() para lograr este efecto. Para usar esta función, suministra una declaración lógica a la función - esto puede tener resultados bastante flexibles, pero aquí mostraremos un ejemplo de la distribución de edad de los casos en nuestro listado, resaltándolos por resultado.
# carga gghighlight
library(gghighlight)
# sustituye los valores NA por unknown en la variable outcome
linelist <- linelist %>%
mutate(outcome = replace_na(outcome, "Unknown"))
# produce a histogram of all cases by age
ggplot(
data = linelist,
mapping = aes(x = age_years, fill = outcome)) +
geom_histogram() +
gghighlight::gghighlight(outcome == "Death") # highlight instances where the patient has died.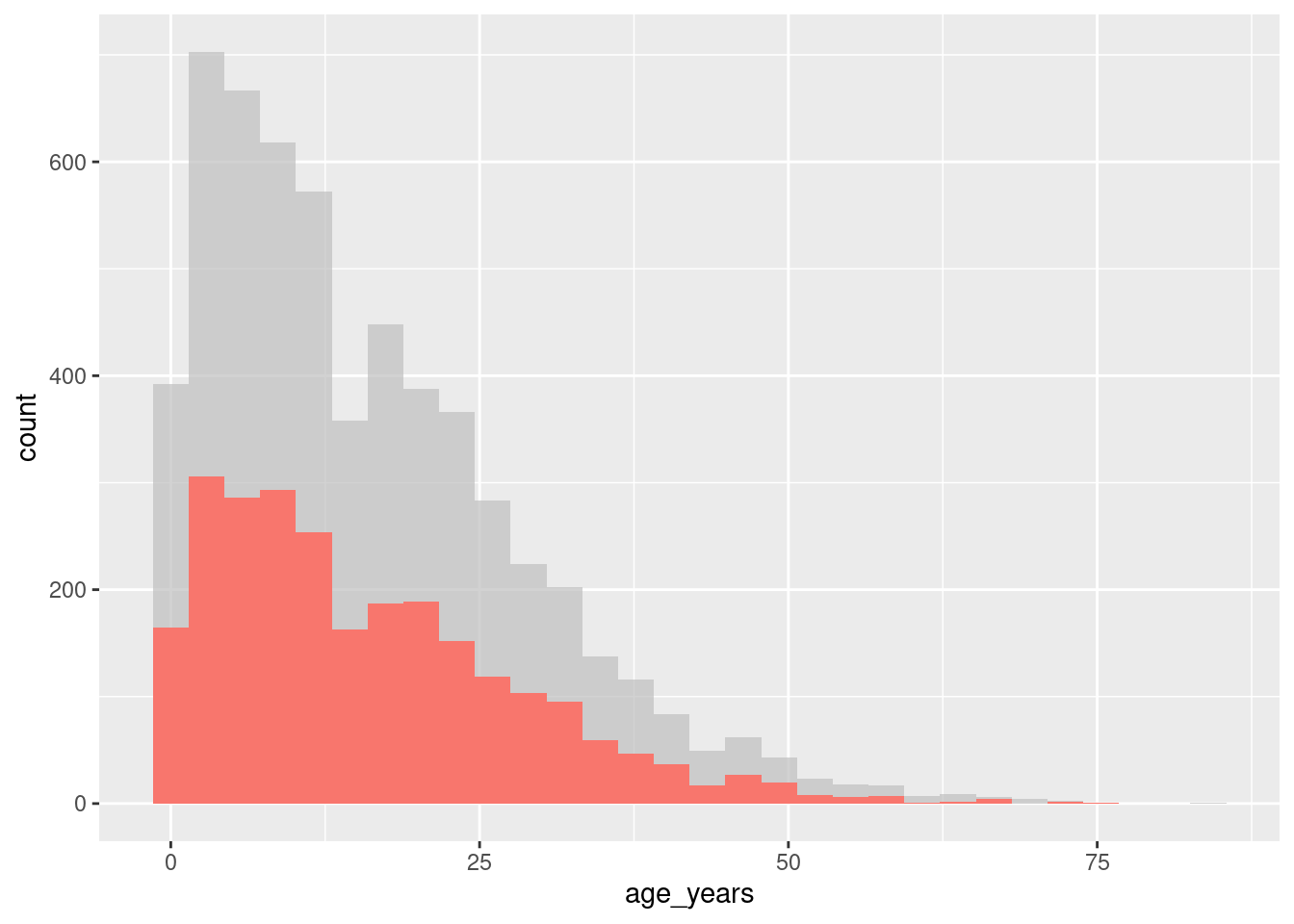
Esto también funciona bien con las funciones de facetas - ¡permite al usuario producir gráficos de facetas con los datos de fondo resaltados que no se aplican a la faceta! A continuación, contamos los casos por semana y trazamos las curvas de epidemia por hospital (color = y facet_wrap() ajustado a la columna hospital).
# produce un histograma de todos los casos por edad
linelist %>%
count(week = lubridate::floor_date(date_hospitalisation, "week"),
hospital) %>%
ggplot()+
geom_line(aes(x = week, y = n, color = hospital))+
theme_minimal()+
gghighlight::gghighlight() + # resalta los casos en los que el paciente ha fallecido.
facet_wrap(~hospital) # crea facetas por resultado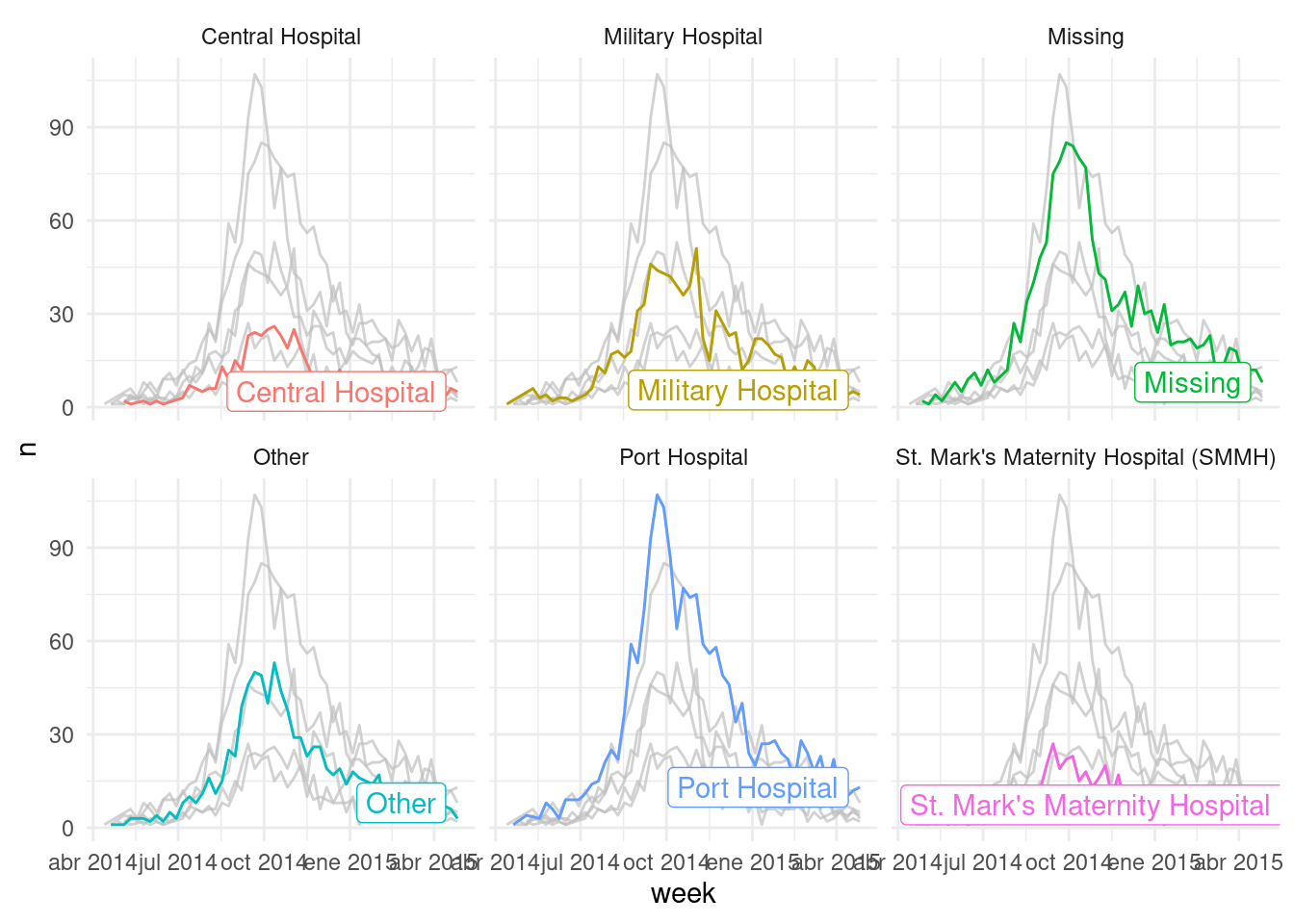
31.9 Representar múltiples conjuntos de datos
Ten en cuenta que alinear correctamente los ejes para trazar múltiples conjuntos de datos en el mismo gráfico puede ser difícil. Considera una de las siguientes estrategias:
- Fusionar los datos antes de trazarlos y convertirlos al formato “long” con una columna que refleje el conjunto de datos
- Utilizar Cowplot o un paquete similar para combinar dos gráficos (véase más abajo)
31.10 Combinar gráficos
Dos paquetes que son muy útiles para combinar gráficos son cowplot y patchwork. En esta página nos centraremos principalmente en cowplot, con el uso ocasional de patchwork.
Aquí está la introducción en línea a cowplot. Puedes leer la documentación más extensa de cada función en línea aquí. A continuación cubriremos algunos de los casos de uso y funciones más comunes.
El paquete cowplot funciona en tándem con ggplot2 - esencialmente, se utiliza para organizar y combinar ggplots y sus leyendas en figuras compuestas. También puede aceptar gráficos de R base.
pacman::p_load(
tidyverse, # manipulación y visualización de datos
cowplot, # combinar gráficos
patchwork # combinar gráficos
)Mientras que las facetas (descritas en la página de Conceptos básicos de ggplot) son un enfoque conveniente para el trazado, a veces no es posible obtener los resultados que deseas de su enfoque relativamente restrictivo. En este caso, se puede optar por combinar los gráficos pegándolos en un gráfico más grande. Hay tres paquetes bien conocidos que son excelentes para esto - cowplot, gridExtra, y patchwork. Sin embargo, estos paquetes hacen en gran medida las mismas cosas, por lo que nos centraremos en cowplot para esta sección.
plot_grid()
El paquete cowplot tiene una gama bastante amplia de funciones, pero el uso más fácil de él se puede lograr mediante el uso de plot_grid(). Se trata de una forma de organizar los gráficos predefinidos en una cuadrícula. Podemos trabajar a través de otro ejemplo con el conjunto de datos de la malaria - aquí podemos trazar el total de casos por distrito, y también mostrar la curva epidémica en el tiempo.
malaria_data <- rio::import(here::here("data", "malaria_facility_count_data.rds"))
# gráfico de barras del total de casos por distrito
p1 <- ggplot(malaria_data, aes(x = District, y = malaria_tot)) +
geom_bar(stat = "identity") +
labs(
x = "District",
y = "Total number of cases",
title = "Total malaria cases by district"
) +
theme_minimal()
# curva epidémica a lo largo del tiempo
p2 <- ggplot(malaria_data, aes(x = data_date, y = malaria_tot)) +
geom_col(width = 1) +
labs(
x = "Date of data submission",
y = "number of cases"
) +
theme_minimal()
cowplot::plot_grid(p1, p2,
# 1 columna y dos filas - apiladas una sobre otra
ncol = 1,
nrow = 2,
# el gráfico superior es 2/3 más alto que el segundo
rel_heights = c(2, 3))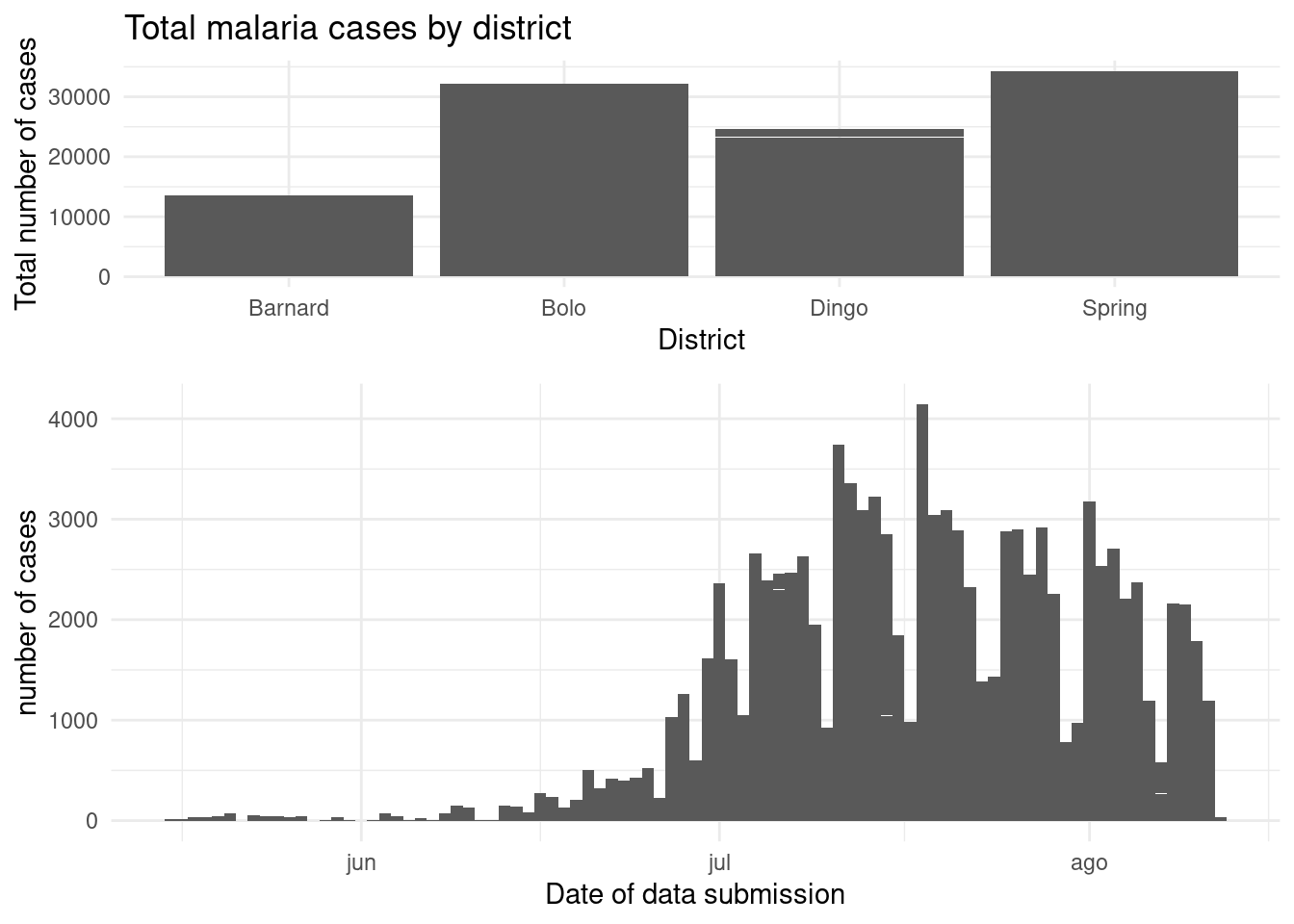
Combinar leyendas
Si tus gráficos tienen la misma leyenda, combinarlos es relativamente sencillo. Simplemente utiliza el método de cowplot anterior para combinar los gráficos, pero elimina la leyenda de uno de ellos (de-duplica).
Si tus gráficos tienen leyendas diferentes, debes utilizar un enfoque alternativo:
- Crea y guarda tus gráficos sin leyendas utilizando
theme(legend.position = "none") - Extrae las leyendas de cada gráfica utilizando get_legend() como se muestra a continuación - pero extrae las leyendas de los gráficos modificados para mostrar realmente la leyenda
- Combina las leyendas en un panel de leyendas
- Combina el panel de gráficos y leyendas
Para comprobarlo, mostramos las dos gráficos por separado, y luego dispuestas en una cuadrícula con sus propias leyendas mostrando (uso feo e ineficiente del espacio):
p1 <- linelist %>%
mutate(hospital = recode(hospital, "St. Mark's Maternity Hospital (SMMH)" = "St. Marks")) %>%
count(hospital, outcome) %>%
ggplot()+
geom_col(mapping = aes(x = hospital, y = n, fill = outcome))+
scale_fill_brewer(type = "qual", palette = 4, na.value = "grey")+
coord_flip()+
theme_minimal()+
labs(title = "Cases by outcome")
p2 <- linelist %>%
mutate(hospital = recode(hospital, "St. Mark's Maternity Hospital (SMMH)" = "St. Marks")) %>%
count(hospital, age_cat) %>%
ggplot()+
geom_col(mapping = aes(x = hospital, y = n, fill = age_cat))+
scale_fill_brewer(type = "qual", palette = 1, na.value = "grey")+
coord_flip()+
theme_minimal()+
theme(axis.text.y = element_blank())+
labs(title = "Cases by age")Así es como se ven los dos gráficos cuando se combinan usando plot_grid() sin combinar sus leyendas:
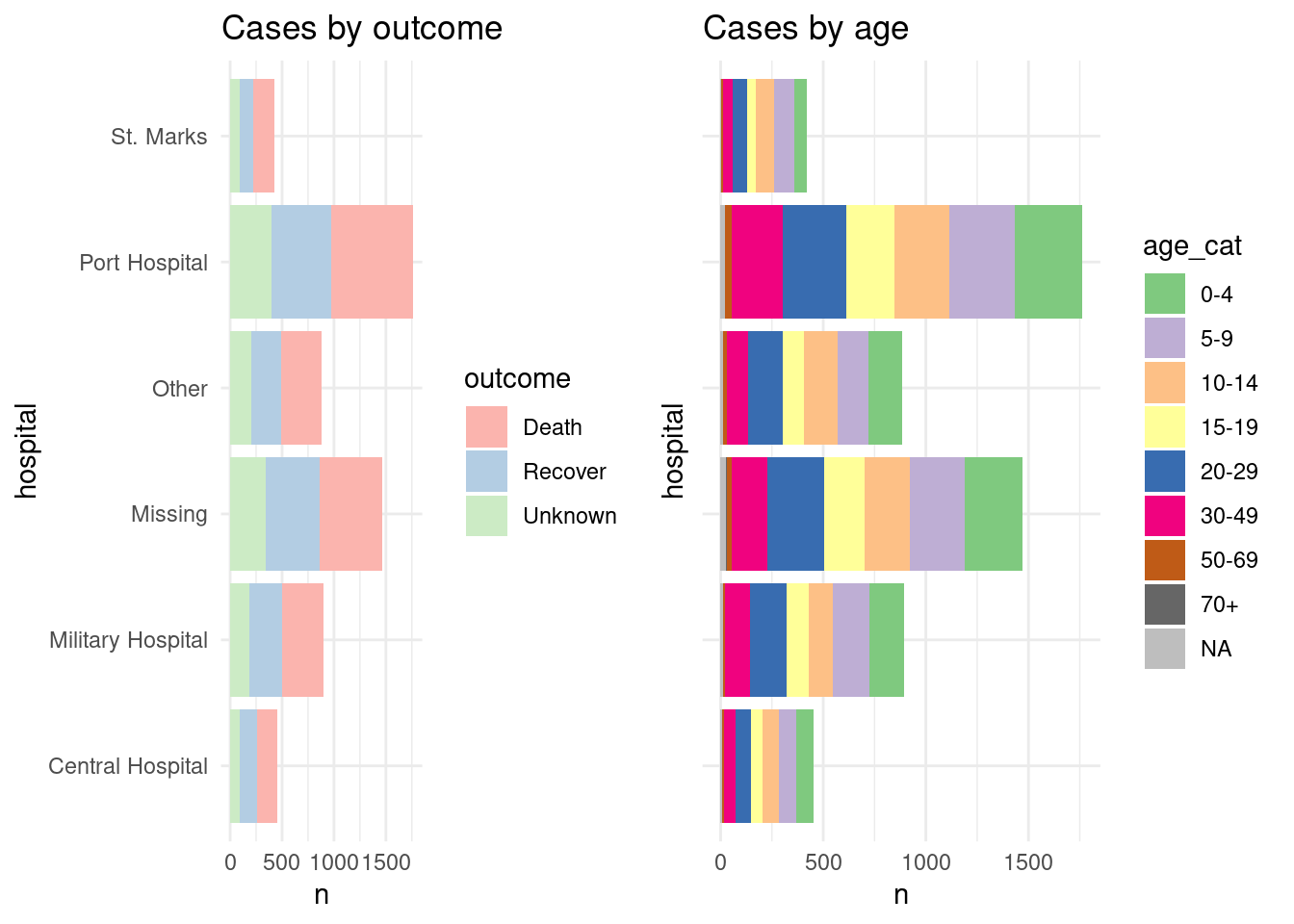
Y ahora mostramos cómo combinar las leyendas. Esencialmente lo que hacemos es definir cada gráfica sin su leyenda (theme(legend.position = "none"), y luego definimos la leyenda de cada gráfica por separado, utilizando la función get_legend() de cowplot. Cuando extraemos la leyenda del gráfico guardado, tenemos que añadir + la leyenda de nuevo, incluyendo la especificación de la colocación (“derecha”) y pequeños ajustes para la alineación de las leyendas y sus títulos. A continuación, combinamos las leyendas verticalmente, y luego combinamos los dos gráficos con las leyendas recién combinadas. Voila!
# Define plot 1 without legend
p1 <- linelist %>%
mutate(hospital = recode(hospital, "St. Mark's Maternity Hospital (SMMH)" = "St. Marks")) %>%
count(hospital, outcome) %>%
ggplot()+
geom_col(mapping = aes(x = hospital, y = n, fill = outcome))+
scale_fill_brewer(type = "qual", palette = 4, na.value = "grey")+
coord_flip()+
theme_minimal()+
theme(legend.position = "none")+
labs(title = "Cases by outcome")
# Define plot 2 without legend
p2 <- linelist %>%
mutate(hospital = recode(hospital, "St. Mark's Maternity Hospital (SMMH)" = "St. Marks")) %>%
count(hospital, age_cat) %>%
ggplot()+
geom_col(mapping = aes(x = hospital, y = n, fill = age_cat))+
scale_fill_brewer(type = "qual", palette = 1, na.value = "grey")+
coord_flip()+
theme_minimal()+
theme(
legend.position = "none",
axis.text.y = element_blank(),
axis.title.y = element_blank()
)+
labs(title = "Cases by age")
# extract legend from p1 (from p1 + legend)
leg_p1 <- cowplot::get_legend(p1 +
theme(legend.position = "right", # extract vertical legend
legend.justification = c(0,0.5))+ # so legends align
labs(fill = "Outcome")) # title of legend
# extract legend from p2 (from p2 + legend)
leg_p2 <- cowplot::get_legend(p2 +
theme(legend.position = "right", # extract vertical legend
legend.justification = c(0,0.5))+ # so legends align
labs(fill = "Age Category")) # title of legend
# create a blank plot for legend alignment
#blank_p <- patchwork::plot_spacer() + theme_void()
# create legends panel, can be one on top of the other (or use spacer commented above)
legends <- cowplot::plot_grid(leg_p1, leg_p2, nrow = 2, rel_heights = c(.3, .7))
# combine two plots and the combined legends panel
combined <- cowplot::plot_grid(p1, p2, legends, ncol = 3, rel_widths = c(.4, .4, .2))
combined # print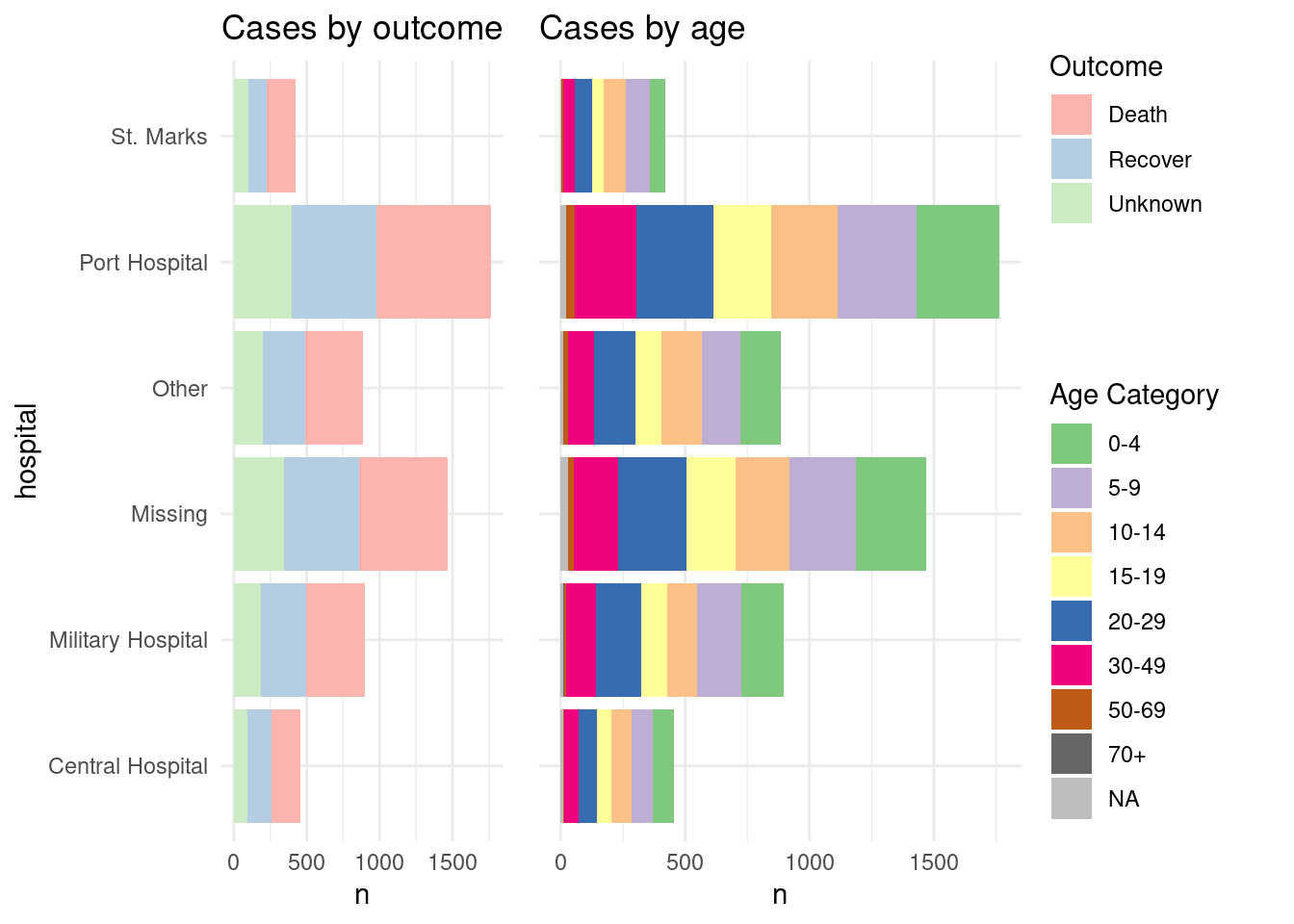
Esta solución fue aprendida de este post con un arreglo menor para alinear las leyendas de este post.
CONSEJO: Nota divertida: la “vaca” en cowplot viene del nombre del creador: Claus O. Wilke.
Gráficos insertados
Puedes insertar una gráfica en otra utilizando cowplot. Aquí hay cosas que hay que tener en cuenta:
- Define el gráfico principal con
theme_half_open()de cowplot; puede ser mejor tener la leyenda arriba o abajo - Define el gráfico de inserción. Lo mejor es tener un gráfico en el que no se necesite una leyenda. Puedes eliminar los elementos del tema del gráfico con
element_blank()como se muestra a continuación. - Combínalos aplicando
ggdraw()al gráfico principal, y luego añadiendodraw_plot()en el gráfico de inserción y especificando las coordenadas (x e y de la esquina inferior izquierda), la altura y la anchura como proporción de todo el gráfico principal.
# Define main plot
main_plot <- ggplot(data = linelist)+
geom_histogram(aes(x = date_onset, fill = hospital))+
scale_fill_brewer(type = "qual", palette = 1, na.value = "grey")+
theme_half_open()+
theme(legend.position = "bottom")+
labs(title = "Epidemic curve and outcomes by hospital")
# Define inset plot
inset_plot <- linelist %>%
mutate(hospital = recode(hospital, "St. Mark's Maternity Hospital (SMMH)" = "St. Marks")) %>%
count(hospital, outcome) %>%
ggplot()+
geom_col(mapping = aes(x = hospital, y = n, fill = outcome))+
scale_fill_brewer(type = "qual", palette = 4, na.value = "grey")+
coord_flip()+
theme_minimal()+
theme(legend.position = "none",
axis.title.y = element_blank())+
labs(title = "Cases by outcome")
# Combine main with inset
cowplot::ggdraw(main_plot)+
draw_plot(inset_plot,
x = .6, y = .55, #x = .07, y = .65,
width = .4, height = .4)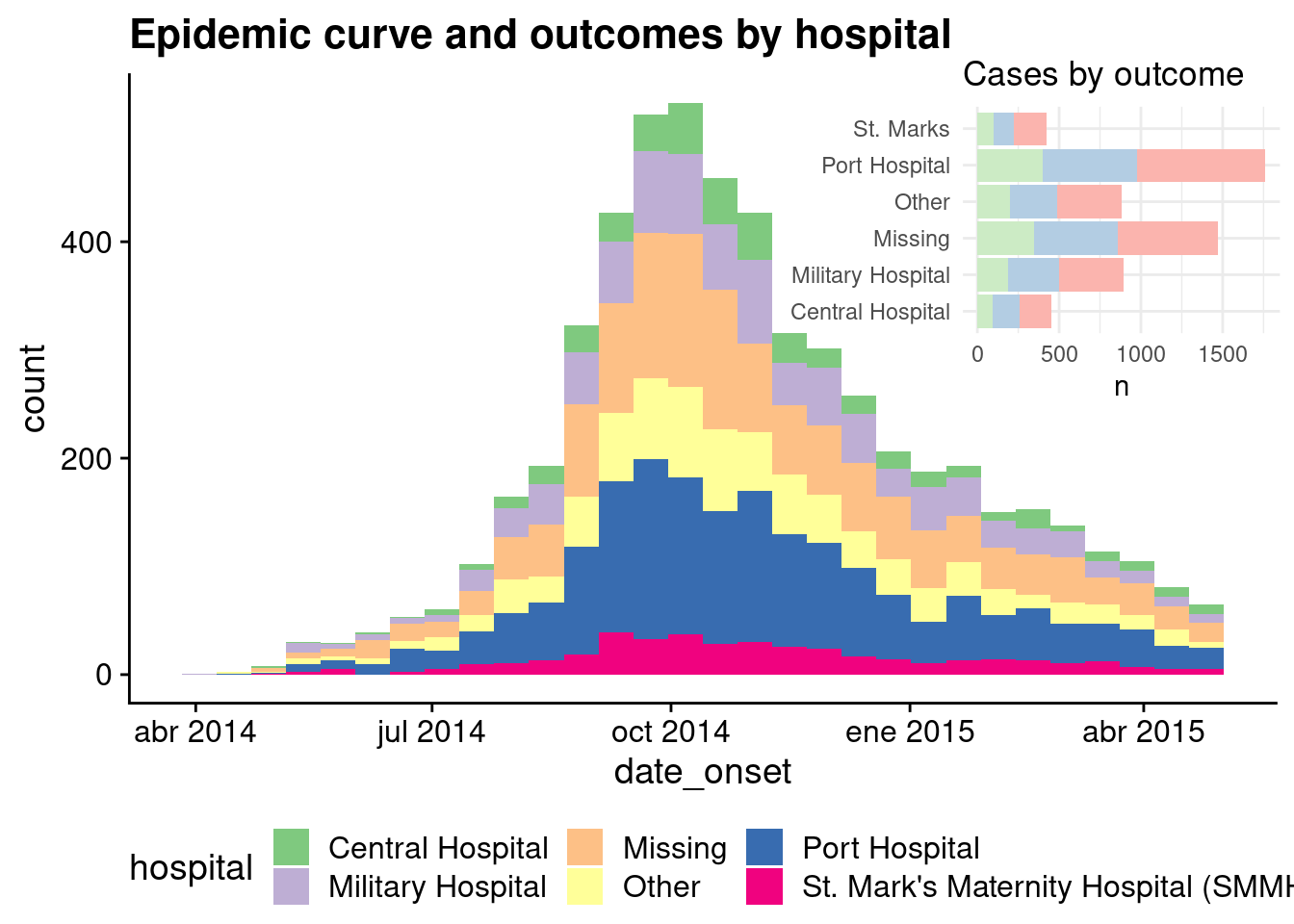
Esta técnica se explica mejor en estas dos viñetas:
31.11 Ejes dobles
Un eje-y secundario es a menudo una adición solicitada a un gráfico ggplot2. Aunque existe un fuerte debate sobre la validez de estos gráficos en la comunidad de visualización de datos, y a menudo no se recomiendan, es posible que tu jefe los quiera. A continuación, presentamos un método para conseguirlos: usa el paquete cowplot para combinar dos gráficos separados.
Este enfoque implica la creación de dos gráficos separados - uno con un eje-y a la izquierda, y el otro con un eje-y a la derecha. Ambos utilizarán un tema específico de theme_cowplot() y deben tener el mismo eje-x. Luego, en un tercer comando, los dos gráficos se alinean y se superponen. Las funcionalidades de cowplot, de las que ésta es sólo una, se describen en profundidad en este sitio.
Para demostrar esta técnica, superpondremos la curva epidémica con una línea del porcentaje semanal de pacientes fallecidos. Utilizamos este ejemplo porque la alineación de las fechas en el eje-x es más compleja que, por ejemplo, alinear un gráfico de barras con otro gráfico. Hay que tener en cuenta algunas cosas:
- Para la curva epidémica y la línea se agrupan en semanas antes de trazarlas y los
date_breaksydate_labelsson idénticos - lo hacemos para que los ejes-x de los dos gráficos sean los mismos cuando se superponen. - El eje-y se mueve a la derecha para el gráfico 2 con el argumento
position =descale_y_continuous(). - Ambos gráficos hacen uso de
theme_cowplot()
Observa que hay otro ejemplo de esta técnica en la página de curvas epidémicas: la superposición de la incidencia acumulada sobre la curva epidemica.
Hacer el gráfico 1
Esto es esencialmente la curva epidémica. Usamos geom_area() sólo para mostrar su uso (área bajo una línea, por defecto)
pacman::p_load(cowplot) # load/install cowplot
p1 <- linelist %>% # save plot as object
count(
epiweek = lubridate::floor_date(date_onset, "week")) %>%
ggplot()+
geom_area(aes(x = epiweek, y = n), fill = "grey")+
scale_x_date(
date_breaks = "month",
date_labels = "%b")+
theme_cowplot()+
labs(
y = "Weekly cases"
)
p1 # view plot 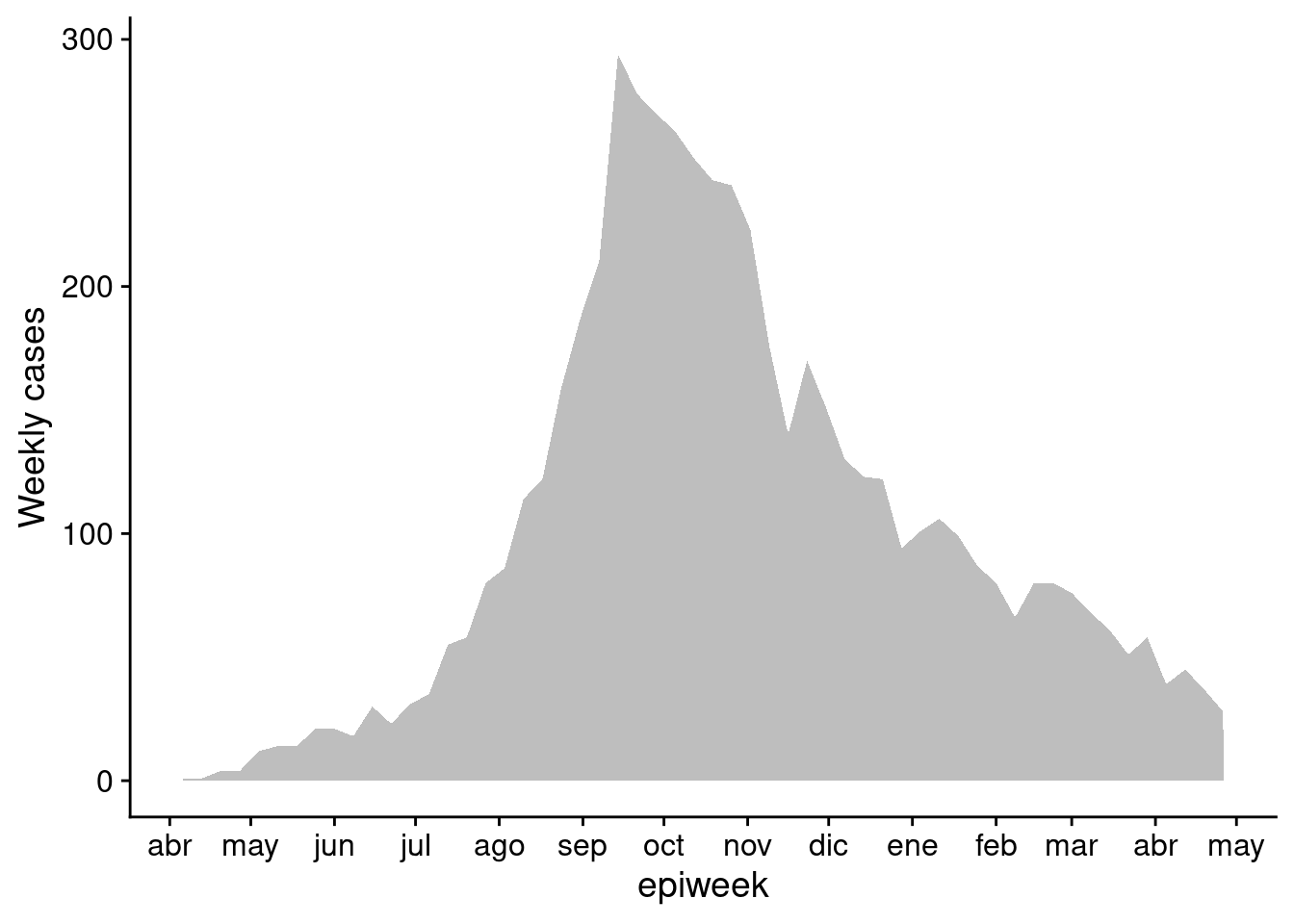
Hacer el gráfico 2 Crea el segundo gráfico mostrando una línea del porcentaje semanal de casos que murieron.
p2 <- linelist %>% # save plot as object
group_by(
epiweek = lubridate::floor_date(date_onset, "week")) %>%
summarise(
n = n(),
pct_death = 100*sum(outcome == "Death", na.rm=T) / n) %>%
ggplot(aes(x = epiweek, y = pct_death))+
geom_line()+
scale_x_date(
date_breaks = "month",
date_labels = "%b")+
scale_y_continuous(
position = "right")+
theme_cowplot()+
labs(
x = "Epiweek of symptom onset",
y = "Weekly percent of deaths",
title = "Weekly case incidence and percent deaths"
)
p2 # view plot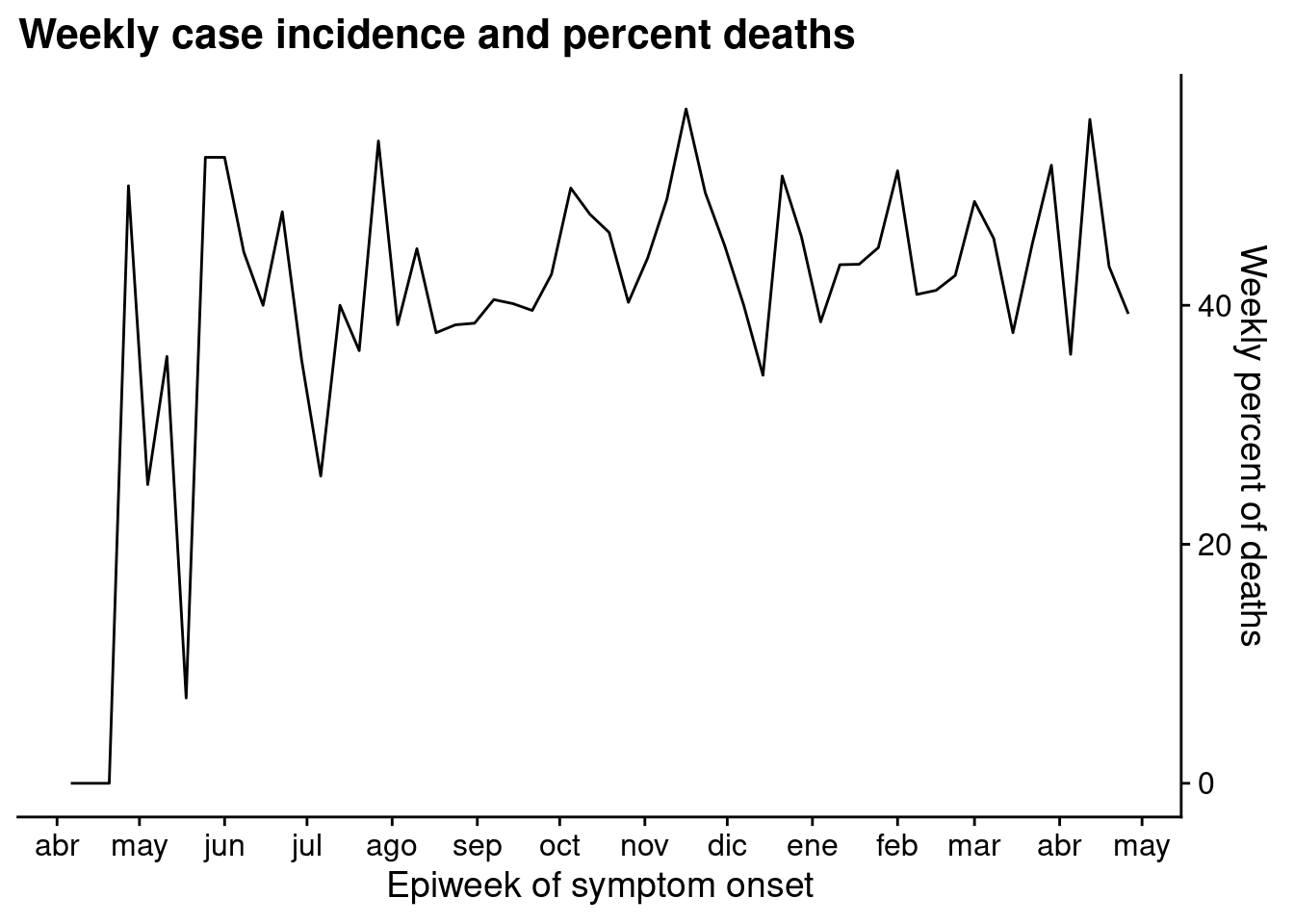
Ahora alineamos el gráfico utilizando la función align_plots(), especificando la alineación horizontal y vertical (“hv”, también podría ser “h”, “v”, “none”). También especificamos la alineación de todos los ejes (top, bottom, left, y right) con “tblr”. La salida es de tipo list (2 elementos).
Luego dibujamos los dos gráficos juntos usando ggdraw() (de cowplot) y referenciando las dos partes del objeto aligned_plots.
aligned_plots <- cowplot::align_plots(p1, p2, align="hv", axis="tblr") # align the two plots and save them as list
aligned_plotted <- ggdraw(aligned_plots[[1]]) + draw_plot(aligned_plots[[2]]) # overlay them and save the visual plot
aligned_plotted # print the overlayed plots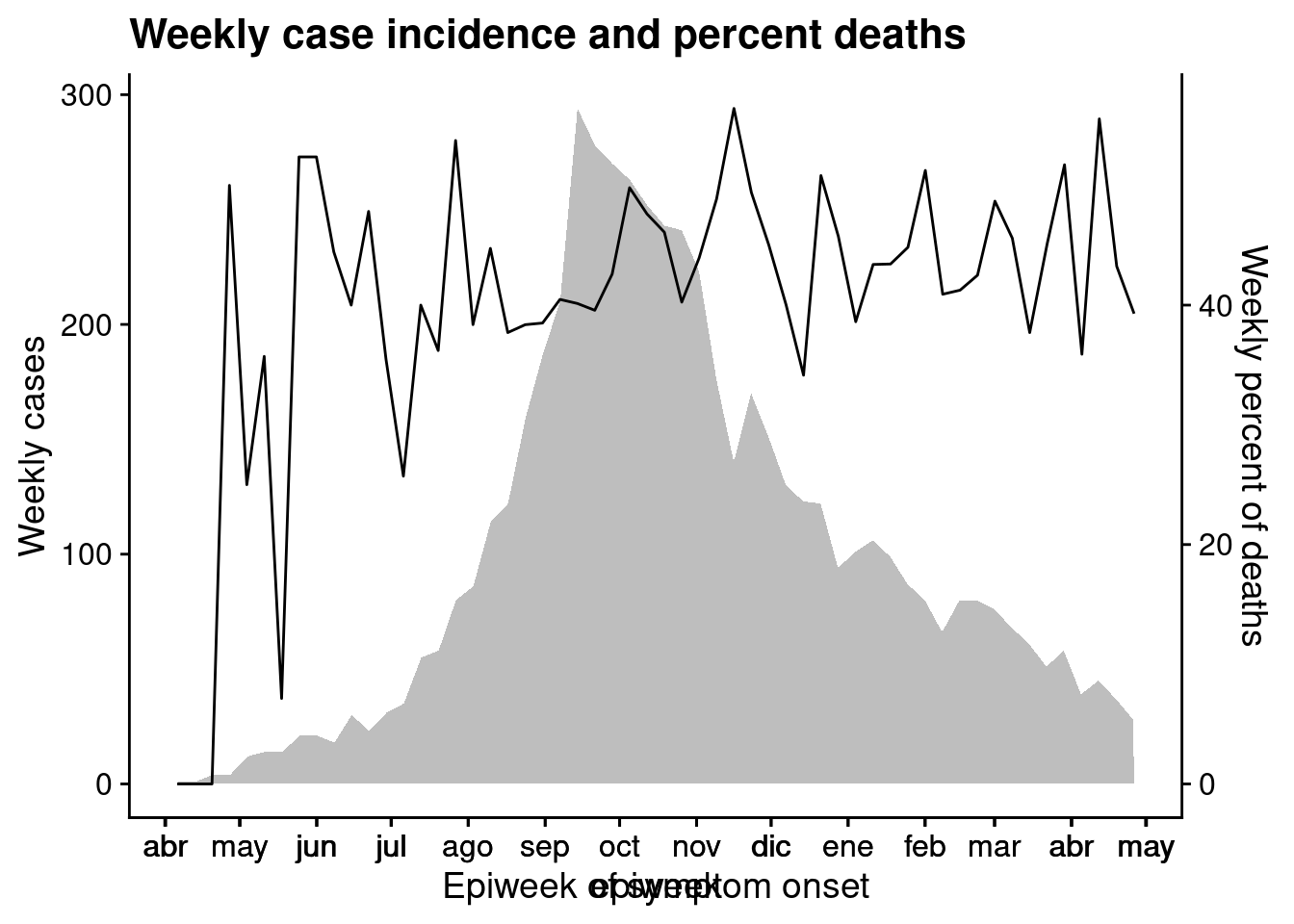
31.12 Paquetes para ayudarte
Hay algunos paquetes de R muy buenos diseñados específicamente para ayudarte a navegar por ggplot2:
Apuntar y clicar en ggplot2 con equisse
“Este complemento te permite explorar interactivamente tus datos visualizándolos con el paquete ggplot2. Te permite dibujar gráficos de barras, curvas, gráficos de dispersión, histogramas, boxplot y objetos sf, y luego exportar el gráfico o recuperar el código para reproducir el gráfico.”
Instala y luego lanza el complemento con el menú de RStudio o con esquisse::esquisser().
Ver la página de Github de esquisse
31.13 Miscelánea
Pantalla numérica
Puedes desactivar la notación científica ejecutando este comando antes de representar gráficamente.
options(scipen=999)O aplicar number_format() del paquete scales a un valor o columna específicos, como se muestra a continuación.
Utiliza las funciones del paquete scales para ajustar fácilmente la forma en que se muestran los números. Pueden aplicarse a las columnas del dataframe, pero se muestran en los números individuales a modo de ejemplo.
scales::number(6.2e5)## [1] "620 000"
scales::number(1506800.62, accuracy = 0.1,)## [1] "1 506 800.6"
scales::comma(1506800.62, accuracy = 0.01)## [1] "1,506,800.62"
scales::comma(1506800.62, accuracy = 0.01, big.mark = "." , decimal.mark = ",")## [1] "1.506.800,62"
scales::percent(0.1)## [1] "10%"
scales::dollar(56)## [1] "$56"
scales::scientific(100000)## [1] "1e+05"31.14 Recursos
Inspiración galería de gráficos de ggplot
Directrices para la presentación de los datos de vigilancia del Centro Europeo para la Prevención y el Control de las Enfermedades (ecdc)
Utilización de etiquetadoras para tiras de facetas y Etiquetadoras]
Ajuste del orden con factores
Reordenar una variable en ggplot2
R for Data Science en español - Factores
Leyendas
Ajustar el orden de las leyendas
Pies de foto
Alineación de las leyendas
Etiquetas
ggrepel
Cheetsheets
Trazado bonito con ggplot2