12 Pivotar datos

En la gestión de datos, se puede entender que el pivoteo se refiere a uno de los dos procesos:
La creación de tablas dinámicas, que son tablas de estadísticas que resumen los datos de una tabla más extensa
La conversión de una tabla de formato largo a formato ancho, o viceversa.
En esta página, nos centraremos en la última definición. La primera es un paso crucial en el análisis de datos, y se trata en las páginas Agrupar datos y Tablas descriptivas.
En esta página se tratan los formatos de los datos. Es útil conocer la idea de “datos ordenados”, en la que cada variable tiene su propia columna, cada observación tiene su propia fila y cada valor tiene su propia celda. Se puede encontrar más información sobre este tema en este capítulo en línea de R for Data Science.
12.1 Preparación
Cargar paquetes
Este trozo de código muestra la carga de los paquetes necesarios para los análisis. En este manual destacamos p_load() de pacman, que instala el paquete si es necesario y lo carga para su uso. También puede cargar los paquetes instalados con library() de R base. Consulta la página sobre Fundamentos de R para obtener más información sobre los paquetes de R.
pacman::p_load(
rio, # Importación de ficheros
here, # Localizador de archivos
tidyverse) # gestión de datos + gráficos ggplot2Recuento de casos de malaria
En esta página, utilizaremos unos datos ficticios de casos diarios de malaria, por centro y grupo de edad. Si quieres seguirlo, clica aquí para descargarlo (como archivo .rds). Importa los datos con la función import() del paquete rio (maneja muchos tipos de archivos como .xlsx, .csv, .rds - mira la página de importación y exportación para más detalles).
# Import data
count_data <- import("malaria_facility_count_data.rds")A continuación se muestran las primeras 50 filas.
Listado de casos de Linelist
En la parte posterior de esta página, también utilizaremos los datos de casos de una epidemia de ébola simulada. Si quieres seguir el proceso, clica aqui para descargar linelist “limpio” (como archivo .rds). Importa tus datos con la función import() del paquete rio (acepta muchos tipos de archivos como .xlsx, .rds, .csv - mira la página de importación y exportación para más detalles).
# importar tus datos
linelist <- import("linelist_cleaned.xlsx")12.2 De ancho a largo
“Formato ancho”
Los datos suelen introducirse y almacenarse en un formato “amplio”, en el que las características o respuestas de un sujeto se almacenan en una sola fila. Aunque esto puede ser útil para la presentación, no es ideal para algunos tipos de análisis.
Tomemos como ejemplo el set de datos count_data importado en la sección “Preparación”. Puedes ver que cada fila representa un “centro-día”. Los recuentos de casos reales (las columnas más a la derecha) se almacenan en un formato “ancho”, de modo que la información de cada grupo de edad en un día determinado del centro se almacena en una sola fila.
Cada observación de este conjunto de datos se refiere a los recuentos de paludismo en una de las 65 instalaciones en una fecha determinada, que va desde count_data$data_date %\>% min() hasta count_data$data_date %\>% max(). Estas instalaciones están situadas en una Province (Norte) y cuatro District (Spring, Bolo, Dingo y Barnard). Los datos proporcionan los recuentos globales de malaria, así como los recuentos específicos por edad en cada uno de los tres grupos de edad: <4 años, 5-14 años y 15 años o más.
Los datos “anchos” como éste no se ajustan a las normas de “datos ordenados”, porque los encabezados de las columnas no representan realmente “variables”, sino que representan valores de una hipotética variable “grupo de edad”.
Este formato puede ser útil para presentar la información en una tabla, o para introducir datos (por ejemplo, en Excel) a partir de formularios de informes de casos. Sin embargo, en la etapa de análisis, estos datos normalmente deben ser transformados a un formato “largo” más alineado con los estándares de “datos ordenados”. El paquete ggplot2, en particular, funciona mejor cuando los datos están en un formato “largo”.
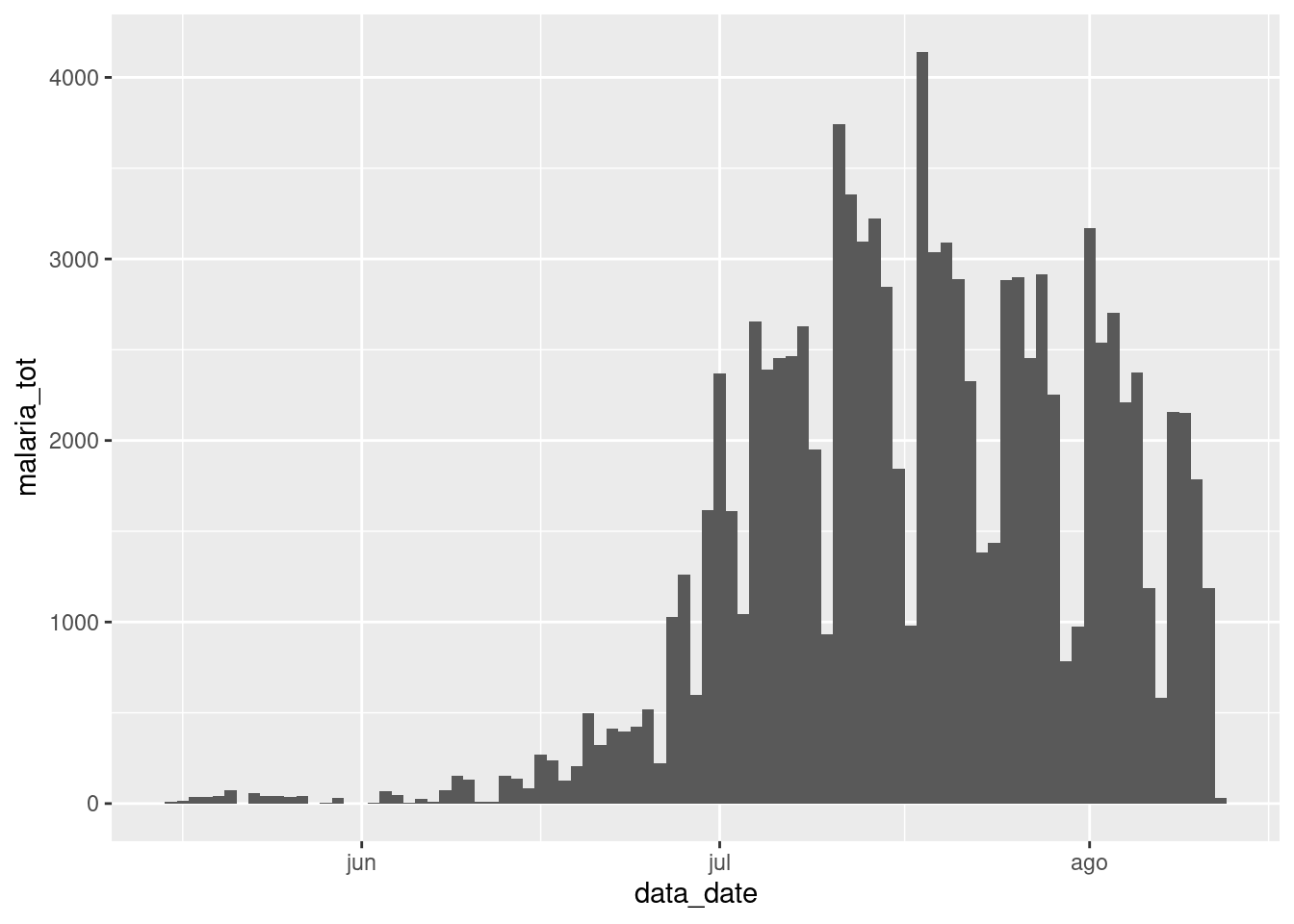
La visualización de los recuentos totales de malaria a lo largo del tiempo no plantea ninguna dificultad con los datos en su formato actual:
Sin embargo, ¿qué pasaría si quisiéramos mostrar las contribuciones relativas de cada grupo de edad a este recuento total? En este caso, necesitamos asegurarnos de que la variable de interés (grupo de edad), aparezca en el conjunto de datos en una sola columna que pueda pasarse a {ggplot2} el argumento aes() de “mapping aesthetics”.
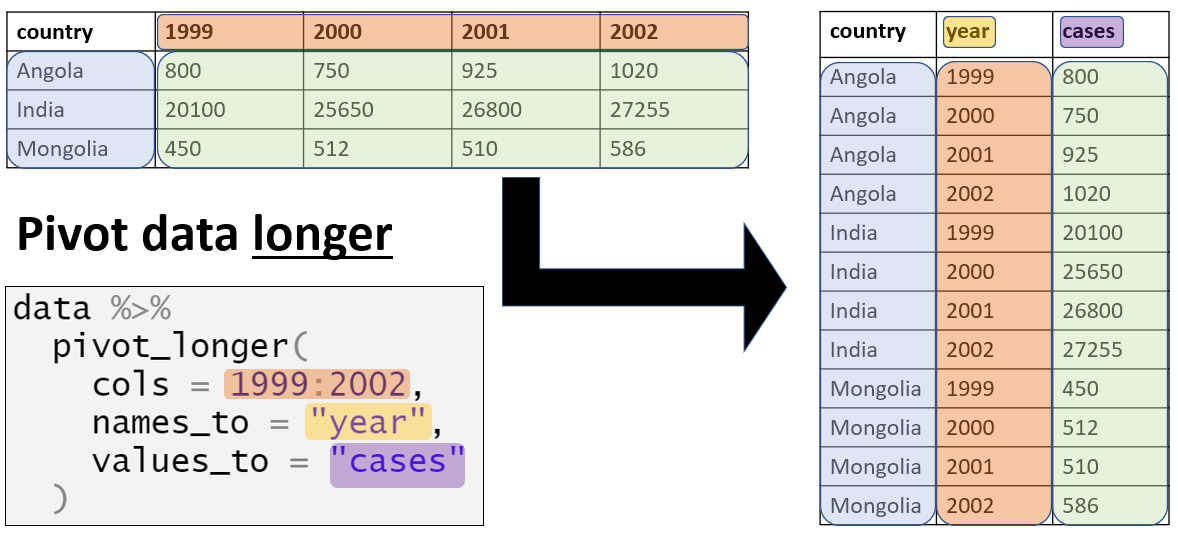
pivot_longer()
La función pivot_longer() de tidyr hace que los datos sean “largos”. tidyr forma parte de los paquetes tidyverse .
Acepta un rango de columnas para transformar (especificado a cols =). Por lo tanto, puede operar sólo en una parte de unos datos. Esto es útil para los datos de la malaria, ya que sólo queremos pivotar las columnas de recuento de casos.
En este proceso, terminará con dos “nuevas” columnas - una con las categorías (los antiguos nombres de las columnas), y otra con los valores correspondientes (por ejemplo, recuento de casos). Puedes aceptar los nombres por defecto para estas nuevas columnas, o puede especificar otros con names_to = y values_to = respectivamente.
Veamos pivot_longer() en acción…
Pivoteo estándar
Queremos utilizar la función pivot_longer() de tidyr para convertir los datos “anchos” en un formato “largo”. Concretamente, para convertir las cuatro columnas numéricas con datos sobre los recuentos de malaria en dos nuevas columnas: una que contenga los grupos de edad y otra que contenga los valores correspondientes.
df_long <- count_data %>%
pivot_longer(
cols = c(`malaria_rdt_0-4`, `malaria_rdt_5-14`, `malaria_rdt_15`, `malaria_tot`)
)
df_longObserva que el dataframe recién creado (df_long) tiene más filas (12.152 frente a 3.038); se ha hecho más largo. De hecho, es precisamente cuatro veces más largo, porque cada fila de los datos originales representa ahora cuatro filas en df_long, una para cada una de las observaciones de recuento de malaria (<4 años, 5-14 años, 15 años+ y total).
Además de ser más largo, el nuevo conjunto de datos tiene menos columnas (8 frente a 10), ya que los datos que antes se almacenaban en cuatro columnas (las que empiezan por el prefijo malaria_) se almacenan ahora en dos.
Dado que los nombres de estas cuatro columnas comienzan con el prefijo malaria_, podríamos haber hecho uso de la práctica función “tidyselect” starts_with() para conseguir el mismo resultado (véase la página Limpieza de datos y funciones básicas para conocer más sobre estas funciones de ayuda).
# proporcionar a la columna una función de ayuda tidyselect
count_data %>%
pivot_longer(
cols = starts_with("malaria_")
)## # A tibble: 12,152 × 8
## location_name data_date submitted_date Province District newid name value
## <chr> <date> <date> <chr> <chr> <int> <chr> <int>
## 1 Facility 1 2020-08-11 2020-08-12 North Spring 1 malaria_rdt_0-4 11
## 2 Facility 1 2020-08-11 2020-08-12 North Spring 1 malaria_rdt_5-14 12
## 3 Facility 1 2020-08-11 2020-08-12 North Spring 1 malaria_rdt_15 23
## 4 Facility 1 2020-08-11 2020-08-12 North Spring 1 malaria_tot 46
## 5 Facility 2 2020-08-11 2020-08-12 North Bolo 2 malaria_rdt_0-4 11
## 6 Facility 2 2020-08-11 2020-08-12 North Bolo 2 malaria_rdt_5-14 10
## 7 Facility 2 2020-08-11 2020-08-12 North Bolo 2 malaria_rdt_15 5
## 8 Facility 2 2020-08-11 2020-08-12 North Bolo 2 malaria_tot 26
## 9 Facility 3 2020-08-11 2020-08-12 North Dingo 3 malaria_rdt_0-4 8
## 10 Facility 3 2020-08-11 2020-08-12 North Dingo 3 malaria_rdt_5-14 5
## # … with 12,142 more rowso por posición:
# proporcionar columnas por posición
count_data %>%
pivot_longer(
cols = 6:9
)o por rango de nombres:
# proporcionar rango de columnas consecutivas
count_data %>%
pivot_longer(
cols = `malaria_rdt_0-4`:malaria_tot
)Estas dos nuevas columnas reciben los nombres por defecto de name y value, pero podemos cambiar estos valores por defecto para proporcionar nombres más significativos, que pueden ayudar a recordar lo que se almacena dentro, utilizando los argumentos names_to y values_to. Utilicemos los nombres age_group y counts:
df_long <-
count_data %>%
pivot_longer(
cols = starts_with("malaria_"),
names_to = "age_group",
values_to = "counts"
)
df_long## # A tibble: 12,152 × 8
## location_name data_date submitted_date Province District newid age_group counts
## <chr> <date> <date> <chr> <chr> <int> <chr> <int>
## 1 Facility 1 2020-08-11 2020-08-12 North Spring 1 malaria_rdt_0-4 11
## 2 Facility 1 2020-08-11 2020-08-12 North Spring 1 malaria_rdt_5-14 12
## 3 Facility 1 2020-08-11 2020-08-12 North Spring 1 malaria_rdt_15 23
## 4 Facility 1 2020-08-11 2020-08-12 North Spring 1 malaria_tot 46
## 5 Facility 2 2020-08-11 2020-08-12 North Bolo 2 malaria_rdt_0-4 11
## 6 Facility 2 2020-08-11 2020-08-12 North Bolo 2 malaria_rdt_5-14 10
## 7 Facility 2 2020-08-11 2020-08-12 North Bolo 2 malaria_rdt_15 5
## 8 Facility 2 2020-08-11 2020-08-12 North Bolo 2 malaria_tot 26
## 9 Facility 3 2020-08-11 2020-08-12 North Dingo 3 malaria_rdt_0-4 8
## 10 Facility 3 2020-08-11 2020-08-12 North Dingo 3 malaria_rdt_5-14 5
## # … with 12,142 more rowsAhora podemos pasar este nuevo conjunto de datos a ggplot2, y asignar la nueva columna count al eje-y y la nueva columna age_group al argumento fill = (el color interno de la columna). Esto mostrará los recuentos de malaria en un gráfico de barras apilado, por grupo de edad:
ggplot(data = df_long) +
geom_col(
mapping = aes(x = data_date, y = counts, fill = age_group),
width = 1
)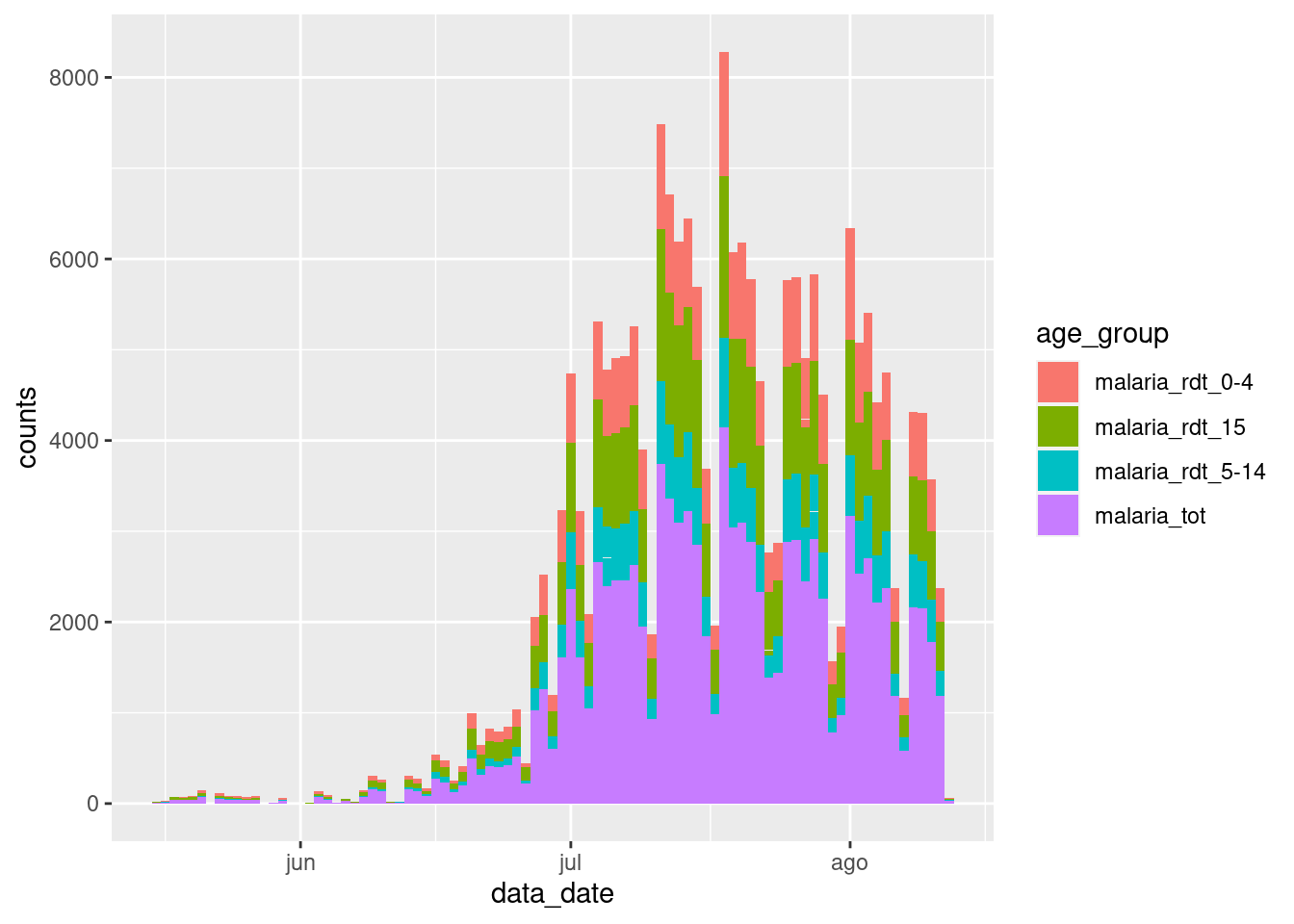
Examina esta nueva gráfica y compárala con la que hemos creado antes: ¿qué ha fallado?
Nos hemos encontrado con un problema común al manejar los datos de vigilancia: hemos incluido también los recuentos totales de la columna malaria_tot, por lo que la magnitud de cada barra en el gráfico es el doble de lo que debería ser.
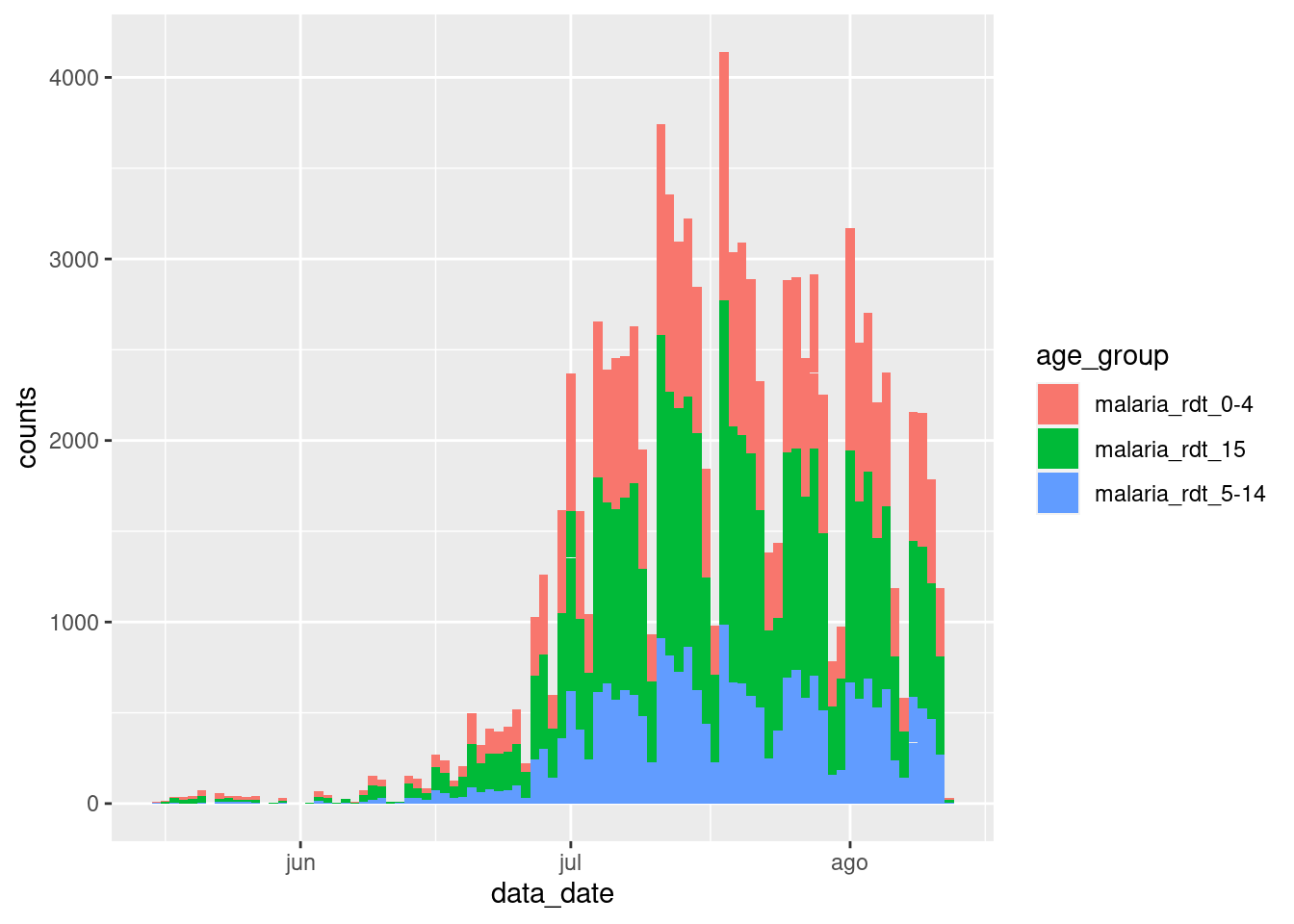
Podemos manejar esto de varias maneras. Podríamos simplemente filtrar estos totales en los datos antes de pasarlo a ggplot():
df_long %>%
filter(age_group != "malaria_tot") %>%
ggplot() +
geom_col(
aes(x = data_date, y = counts, fill = age_group),
width = 1
)
Como alternativa, podríamos haber excluido esta variable al ejecutar pivot_longer(), manteniéndola así en set de datos como una variable independiente. Observa cómo se “expanden” sus valores para llenar las nuevas filas.
count_data %>%
pivot_longer(
cols = `malaria_rdt_0-4`:malaria_rdt_15, # no incluye la columna de totales
names_to = "age_group",
values_to = "counts"
)## # A tibble: 9,114 × 9
## location_name data_date submitted_date Province District malaria_tot newid age_group counts
## <chr> <date> <date> <chr> <chr> <int> <int> <chr> <int>
## 1 Facility 1 2020-08-11 2020-08-12 North Spring 46 1 malaria_rdt_0-4 11
## 2 Facility 1 2020-08-11 2020-08-12 North Spring 46 1 malaria_rdt_5-14 12
## 3 Facility 1 2020-08-11 2020-08-12 North Spring 46 1 malaria_rdt_15 23
## 4 Facility 2 2020-08-11 2020-08-12 North Bolo 26 2 malaria_rdt_0-4 11
## 5 Facility 2 2020-08-11 2020-08-12 North Bolo 26 2 malaria_rdt_5-14 10
## 6 Facility 2 2020-08-11 2020-08-12 North Bolo 26 2 malaria_rdt_15 5
## 7 Facility 3 2020-08-11 2020-08-12 North Dingo 18 3 malaria_rdt_0-4 8
## 8 Facility 3 2020-08-11 2020-08-12 North Dingo 18 3 malaria_rdt_5-14 5
## 9 Facility 3 2020-08-11 2020-08-12 North Dingo 18 3 malaria_rdt_15 5
## 10 Facility 4 2020-08-11 2020-08-12 North Bolo 49 4 malaria_rdt_0-4 16
## # … with 9,104 more rowsPivotear datos de múltiples tipos
El ejemplo anterior funciona bien en situaciones en las que todas las columnas que se quieren “pivotar más” son del mismo tipo (carácter, numérico, lógico…).
Sin embargo, habrá muchos casos en los que, en el trabajo de campo, se trabaje con datos preparados por personas no especializadas y que sigan su propia lógica no estándar - como señaló Hadley Wickham (haciendo referencia a Tolstoi) en su artículo seminal sobre los principios de Tidy Data: “Como las familias, los conjuntos de datos ordenados son todos iguales, pero cada conjunto de datos desordenado es desordenado a su manera”.
Un problema particularmente común que encontrarás será la necesidad de pivotar columnas que contienen diferentes tipos de datos. Este pivote resultará en el almacenamiento de estos diferentes tipos de datos en una sola columna, lo cual no es una buena situación. Se pueden seguir varios enfoques para separar el desorden que esto crea, pero hay un paso importante que puedes seguir usando pivot_longer() para evitar crear tal situación tu mismo.
Tomemos una situación en la que ha habido una serie de observaciones en diferentes pasos de tiempo para cada uno de los tres elementos A, B y C. Ejemplos de estos elementos podrían ser individuos (por ejemplo, contactos de un caso de ébola que se rastrean cada día durante 21 días) o puestos de salud de aldeas remotas que se supervisan una vez al año para garantizar que siguen funcionando. Utilicemos el ejemplo del rastreo de contactos. Imaginemos que los datos se almacenan de la siguiente manera:
Como puede verse, los datos son un poco complicados. Cada fila almacena información sobre un elemento, pero con la serie temporal cada vez más alejada hacia la derecha a medida que avanza el tiempo. Además, los tipos de columnas alternan entre valores de fecha y caracteres.
Un ejemplo particularmente malo que encontró este autor fue el de los datos de vigilancia del cólera, en el que se añadieron 8 nuevas columnas de observaciones cada día en el transcurso de 4 años. El simple hecho de abrir el archivo de Excel en el que se almacenaban estos datos me llevó más de 10 minutos en mi ordenador portátil.
Para trabajar con estos datos, necesitamos transformar el dataframe a formato largo, pero manteniendo la separación entre una columna date y una columna de character (estado), para cada observación de cada elemento. Si no lo hacemos, podríamos terminar con una mezcla de tipos de variables en una sola columna (un gran “no-no” cuando se trata de gestión de datos y de datos ordenados):
df %>%
pivot_longer(
cols = -id,
names_to = c("observation")
)## # A tibble: 18 × 3
## id observation value
## <chr> <chr> <chr>
## 1 A obs1_date 2021-04-23
## 2 A obs1_status Healthy
## 3 A obs2_date 2021-04-24
## 4 A obs2_status Healthy
## 5 A obs3_date 2021-04-25
## 6 A obs3_status Unwell
## 7 B obs1_date 2021-04-23
## 8 B obs1_status Healthy
## 9 B obs2_date 2021-04-24
## 10 B obs2_status Healthy
## 11 B obs3_date 2021-04-25
## 12 B obs3_status Healthy
## 13 C obs1_date 2021-04-23
## 14 C obs1_status Missing
## 15 C obs2_date 2021-04-24
## 16 C obs2_status Healthy
## 17 C obs3_date 2021-04-25
## 18 C obs3_status HealthyArriba, nuestro pivote ha fusionado fechas y caracteres en una sola columna de value. R reaccionará convirtiendo toda la columna en tipo carácter, y se pierde la utilidad de las fechas.
Para evitar esta situación, podemos aprovechar la estructura sintáctica de los nombres de las columnas originales. Hay una estructura de nombres común, con el número de observación, un guión bajo, y luego “estado” o “fecha”. Podemos aprovechar esta sintaxis para mantener estos dos tipos de datos en columnas separadas después del pivote.
Para ello:
- Proporcionar un vector de caracteres al argumento names_to =, siendo el segundo elemento (
".value"). Este término especial indica que las columnas pivotadas se dividirán basándose en un carácter de su nombre… - También se debe proporcionar el carácter de “división” al argumento
names_sep =. En este caso, es el guión bajo “_“.
Así, la denominación y división de las nuevas columnas se basa en el guión bajo de los nombres de las variables existentes.
df_long <-
df %>%
pivot_longer(
cols = -id,
names_to = c("observation", ".value"),
names_sep = "_"
)
df_long## # A tibble: 9 × 4
## id observation date status
## <chr> <chr> <chr> <chr>
## 1 A obs1 2021-04-23 Healthy
## 2 A obs2 2021-04-24 Healthy
## 3 A obs3 2021-04-25 Unwell
## 4 B obs1 2021-04-23 Healthy
## 5 B obs2 2021-04-24 Healthy
## 6 B obs3 2021-04-25 Healthy
## 7 C obs1 2021-04-23 Missing
## 8 C obs2 2021-04-24 Healthy
## 9 C obs3 2021-04-25 HealthyToques finales:
Ten en cuenta que la columna de fecha es actualmente de tipo carácter - podemos convertirla fácilmente en tipo fecha utilizando las funciones mutate() y as_date() descritas en la página Trabajar con fechas.
También podemos convertir la columna de observation a un formato numeric eliminando el prefijo “obs” y convirtiendo a numérico. Podemos hacer esto con str_remove_all() del paquete stringr (véase la página Caracteres y cadenas).
df_long <-
df_long %>%
mutate(
date = date %>% lubridate::as_date(),
observation =
observation %>%
str_remove_all("obs") %>%
as.numeric()
)
df_long## # A tibble: 9 × 4
## id observation date status
## <chr> <dbl> <date> <chr>
## 1 A 1 2021-04-23 Healthy
## 2 A 2 2021-04-24 Healthy
## 3 A 3 2021-04-25 Unwell
## 4 B 1 2021-04-23 Healthy
## 5 B 2 2021-04-24 Healthy
## 6 B 3 2021-04-25 Healthy
## 7 C 1 2021-04-23 Missing
## 8 C 2 2021-04-24 Healthy
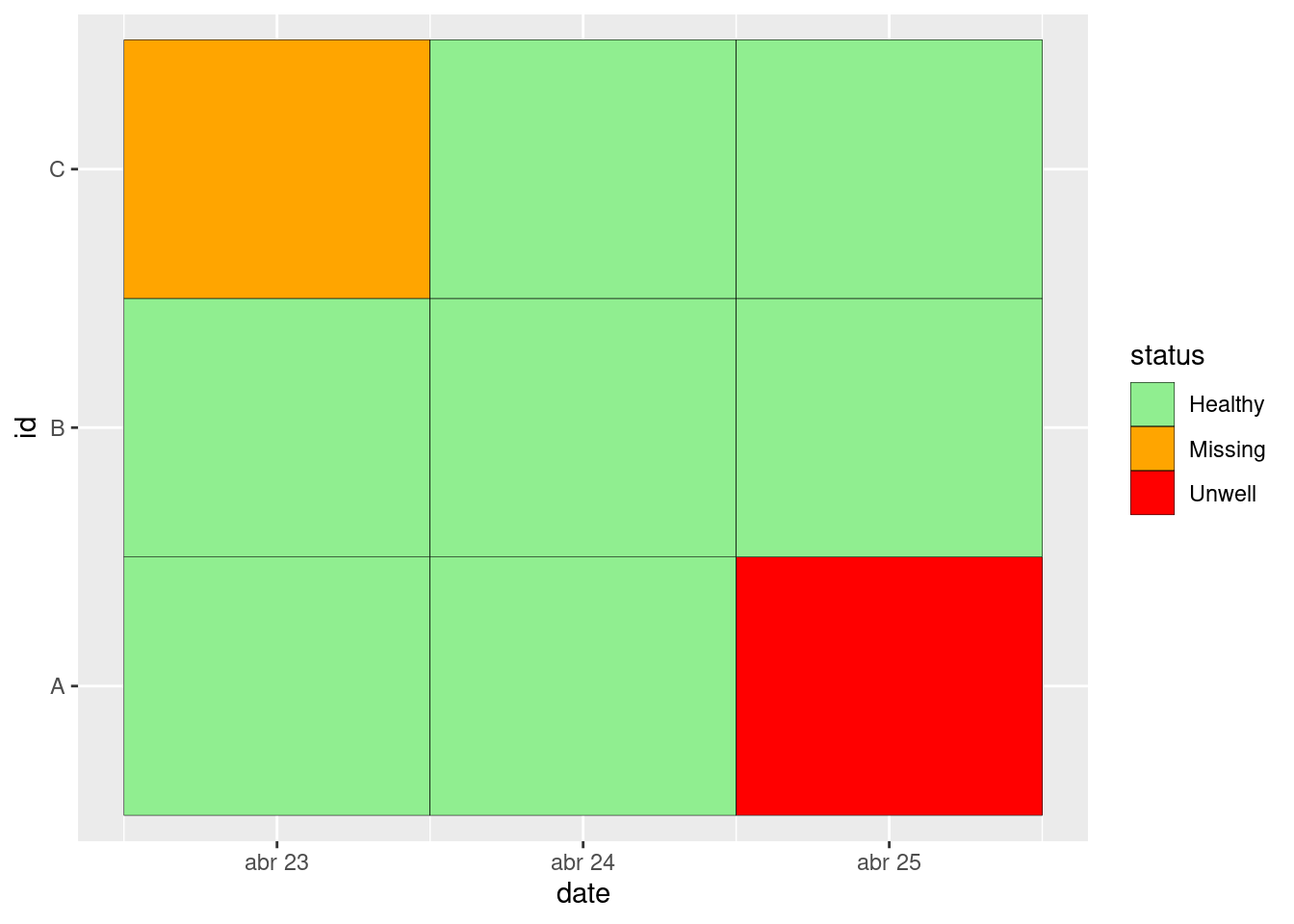
## 9 C 3 2021-04-25 HealthyY ahora, podemos empezar a trabajar con los datos en este formato, por ejemplo, trazando un mosaico de calor descriptivo:
ggplot(data = df_long, mapping = aes(x = date, y = id, fill = status)) +
geom_tile(colour = "black") +
scale_fill_manual(
values =
c("Healthy" = "lightgreen",
"Unwell" = "red",
"Missing" = "orange")
)
12.3 De largo a ancho
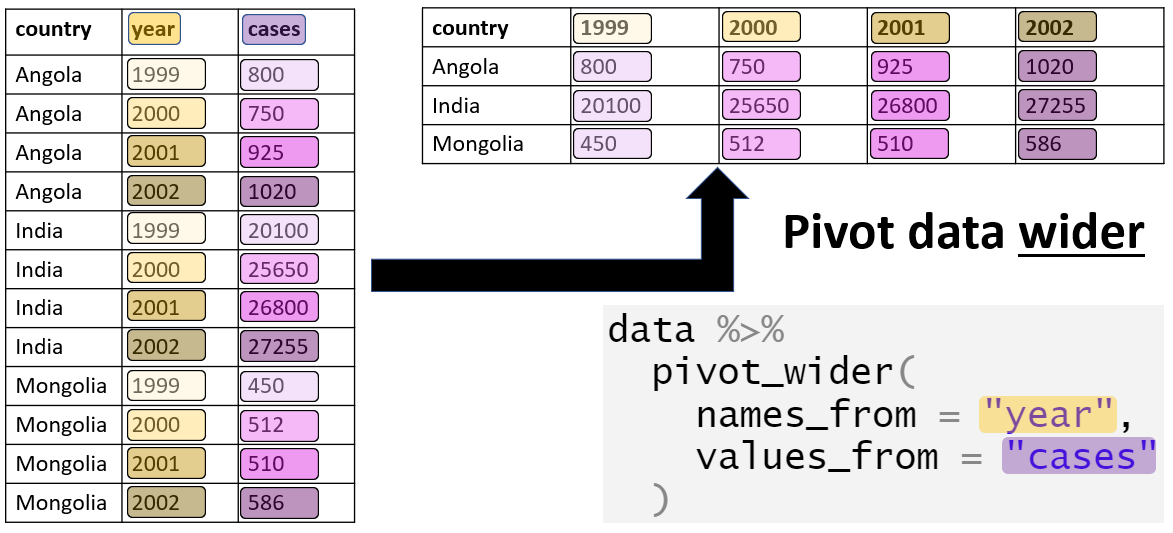
En algunos casos, es posible que queramos convertir unos datos a un formato ancho. Para ello, podemos utilizar la función pivot_wider().
Un caso de uso típico es cuando queremos transformar los resultados de un análisis en un formato que sea más digerible para el lector (como una tabla para su presentación). Por lo general, se trata de transformar unos datos en el que la información de un sujeto está repartida en varias filas en un formato en el que esa información se almacena en una sola fila.
Datos
Para esta sección de la página, utilizaremos la lista de casos (véase la sección Preparación), que contiene una fila por caso.
Aquí están las primeras 50 filas:
Supongamos que queremos conocer los recuentos de individuos en los diferentes grupos de edad, por género:
## age_cat gender n
## 1 0-4 f 640
## 2 0-4 m 416
## 3 0-4 <NA> 39
## 4 5-9 f 641
## 5 5-9 m 412
## 6 5-9 <NA> 42
## 7 10-14 f 518
## 8 10-14 m 383
## 9 10-14 <NA> 40
## 10 15-19 f 359
## 11 15-19 m 364
## 12 15-19 <NA> 20
## 13 20-29 f 468
## 14 20-29 m 575
## 15 20-29 <NA> 30
## 16 30-49 f 179
## 17 30-49 m 557
## 18 30-49 <NA> 18
## 19 50-69 f 2
## 20 50-69 m 91
## 21 50-69 <NA> 2
## 22 70+ m 5
## 23 70+ <NA> 1
## 24 <NA> <NA> 86Esto nos da un largo conjunto de datos que es genial para producir visualizaciones en ggplot2, pero no es ideal para la presentación en una tabla:
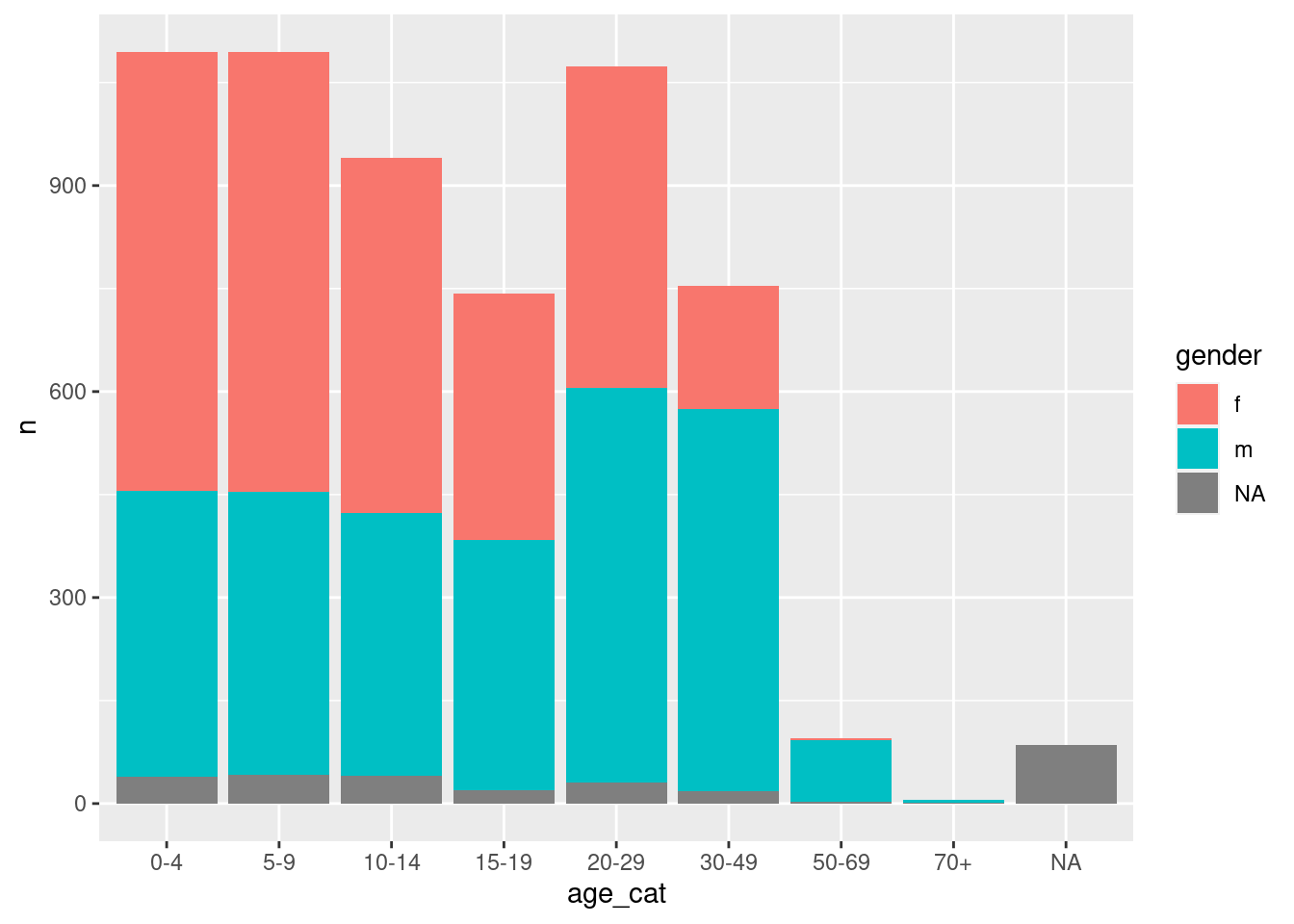
Pivote ancho
Por lo tanto, podemos utilizar pivot_wider() para transformar los datos en un formato mejor para incluirlos como tablas en nuestros informes.
El argumento names_from especifica la columna from que genera la columna nueva names, mientras que el argumento values_from especifica la columna from de la que tomar los values para rellenar las celdas. El argumento id_cols = es opcional, pero se puede proporcionar un vector de nombres de columnas que no deben ser pivotadas, y que por tanto identificarán cada fila.
table_wide <-
df_wide %>%
pivot_wider(
id_cols = age_cat,
names_from = gender,
values_from = n
)
table_wide## # A tibble: 9 × 4
## age_cat f m `NA`
## <fct> <int> <int> <int>
## 1 0-4 640 416 39
## 2 5-9 641 412 42
## 3 10-14 518 383 40
## 4 15-19 359 364 20
## 5 20-29 468 575 30
## 6 30-49 179 557 18
## 7 50-69 2 91 2
## 8 70+ NA 5 1
## 9 <NA> NA NA 86Esta tabla es mucho más fácil de leer y, por tanto, mejor para incluirla en nuestros informes. Se puede convertir en una tabla bonita con varios paquetes, como flextable y knitr. Este proceso se elabora en la página Tablas para presentaciones.
table_wide %>%
janitor::adorn_totals(c("row", "col")) %>% # adds row and column totals
knitr::kable() %>%
kableExtra::row_spec(row = 10, bold = TRUE) %>%
kableExtra::column_spec(column = 5, bold = TRUE) | age_cat | f | m | NA | Total |
|---|---|---|---|---|
| 0-4 | 640 | 416 | 39 | 1095 |
| 5-9 | 641 | 412 | 42 | 1095 |
| 10-14 | 518 | 383 | 40 | 941 |
| 15-19 | 359 | 364 | 20 | 743 |
| 20-29 | 468 | 575 | 30 | 1073 |
| 30-49 | 179 | 557 | 18 | 754 |
| 50-69 | 2 | 91 | 2 | 95 |
| 70+ | NA | 5 | 1 | 6 |
| NA | NA | NA | 86 | 86 |
| Total | 2807 | 2803 | 278 | 5888 |
12.4 Rellenar
En algunas situaciones después de pivotar, y más comúnmente después de unir con bind, nos quedan huecos en algunas celdas que nos gustaría rellenar.
Datos
Por ejemplo, toma dos conjuntos de datos, cada uno con observaciones para el número de medición, el nombre del centro y el recuento de casos en ese momento. Sin embargo, el segundo conjunto de datos también tiene la variable Year.
df1 <-
tibble::tribble(
~Measurement, ~Facility, ~Cases,
1, "Hosp 1", 66,
2, "Hosp 1", 26,
3, "Hosp 1", 8,
1, "Hosp 2", 71,
2, "Hosp 2", 62,
3, "Hosp 2", 70,
1, "Hosp 3", 47,
2, "Hosp 3", 70,
3, "Hosp 3", 38,
)
df1 ## # A tibble: 9 × 3
## Measurement Facility Cases
## <dbl> <chr> <dbl>
## 1 1 Hosp 1 66
## 2 2 Hosp 1 26
## 3 3 Hosp 1 8
## 4 1 Hosp 2 71
## 5 2 Hosp 2 62
## 6 3 Hosp 2 70
## 7 1 Hosp 3 47
## 8 2 Hosp 3 70
## 9 3 Hosp 3 38
df2 <-
tibble::tribble(
~Year, ~Measurement, ~Facility, ~Cases,
2000, 1, "Hosp 4", 82,
2001, 2, "Hosp 4", 87,
2002, 3, "Hosp 4", 46
)
df2## # A tibble: 3 × 4
## Year Measurement Facility Cases
## <dbl> <dbl> <chr> <dbl>
## 1 2000 1 Hosp 4 82
## 2 2001 2 Hosp 4 87
## 3 2002 3 Hosp 4 46Cuando realizamos un bind_rows() para unir los dos conjuntos de datos, la variable Year se rellena con NA para aquellas filas en las que no había información previa (es decir, el primer conjunto de datos):
## # A tibble: 12 × 4
## Measurement Facility Cases Year
## <dbl> <chr> <dbl> <dbl>
## 1 1 Hosp 1 66 NA
## 2 1 Hosp 2 71 NA
## 3 1 Hosp 3 47 NA
## 4 1 Hosp 4 82 2000
## 5 2 Hosp 1 26 NA
## 6 2 Hosp 2 62 NA
## 7 2 Hosp 3 70 NA
## 8 2 Hosp 4 87 2001
## 9 3 Hosp 1 8 NA
## 10 3 Hosp 2 70 NA
## 11 3 Hosp 3 38 NA
## 12 3 Hosp 4 46 2002
fill()
En este caso, Year es una variable útil para incluir, especialmente si queremos explorar las tendencias a lo largo del tiempo. Por lo tanto, utilizamos fill() para rellenar esas celdas vacías, especificando la columna a rellenar y la dirección (en este caso hacia arriba):
## # A tibble: 12 × 4
## Measurement Facility Cases Year
## <dbl> <chr> <dbl> <dbl>
## 1 1 Hosp 1 66 2000
## 2 1 Hosp 2 71 2000
## 3 1 Hosp 3 47 2000
## 4 1 Hosp 4 82 2000
## 5 2 Hosp 1 26 2001
## 6 2 Hosp 2 62 2001
## 7 2 Hosp 3 70 2001
## 8 2 Hosp 4 87 2001
## 9 3 Hosp 1 8 2002
## 10 3 Hosp 2 70 2002
## 11 3 Hosp 3 38 2002
## 12 3 Hosp 4 46 2002Alternativamente, podemos reordenar los datos para que tengamos que rellenar en sentido descendente:
## # A tibble: 12 × 4
## Measurement Facility Cases Year
## <dbl> <chr> <dbl> <dbl>
## 1 1 Hosp 4 82 2000
## 2 1 Hosp 3 47 NA
## 3 1 Hosp 2 71 NA
## 4 1 Hosp 1 66 NA
## 5 2 Hosp 4 87 2001
## 6 2 Hosp 3 70 NA
## 7 2 Hosp 2 62 NA
## 8 2 Hosp 1 26 NA
## 9 3 Hosp 4 46 2002
## 10 3 Hosp 3 38 NA
## 11 3 Hosp 2 70 NA
## 12 3 Hosp 1 8 NA## # A tibble: 12 × 4
## Measurement Facility Cases Year
## <dbl> <chr> <dbl> <dbl>
## 1 1 Hosp 4 82 2000
## 2 1 Hosp 3 47 2000
## 3 1 Hosp 2 71 2000
## 4 1 Hosp 1 66 2000
## 5 2 Hosp 4 87 2001
## 6 2 Hosp 3 70 2001
## 7 2 Hosp 2 62 2001
## 8 2 Hosp 1 26 2001
## 9 3 Hosp 4 46 2002
## 10 3 Hosp 3 38 2002
## 11 3 Hosp 2 70 2002
## 12 3 Hosp 1 8 2002Ahora tenemos unos datos útiles para representarlos gráficamente:
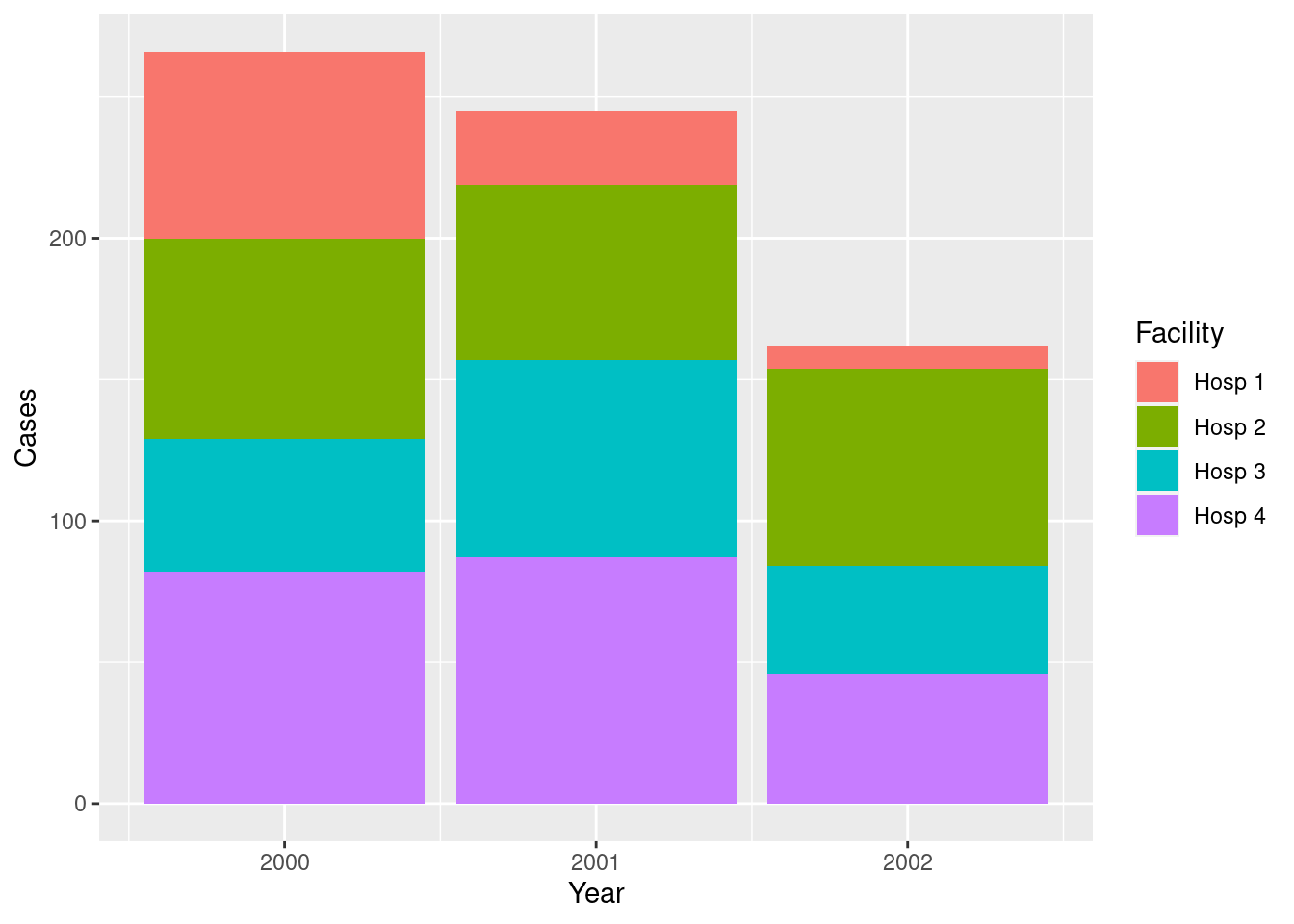
Pero es menos útil para presentarlo en una tabla, así que practiquemos la conversión de este largo y desordenado dataframe en un dataframe ancho y ordenado:
df_combined %>%
pivot_wider(
id_cols = c(Measurement, Facility),
names_from = "Year",
values_from = "Cases"
) %>%
arrange(Facility) %>%
janitor::adorn_totals(c("row", "col")) %>%
knitr::kable() %>%
kableExtra::row_spec(row = 5, bold = TRUE) %>%
kableExtra::column_spec(column = 5, bold = TRUE) | Measurement | Facility | 2000 | 2001 | 2002 | Total |
|---|---|---|---|---|---|
| 1 | Hosp 1 | 66 | NA | NA | 66 |
| 2 | Hosp 1 | NA | 26 | NA | 26 |
| 3 | Hosp 1 | NA | NA | 8 | 8 |
| 1 | Hosp 2 | 71 | NA | NA | 71 |
| 2 | Hosp 2 | NA | 62 | NA | 62 |
| 3 | Hosp 2 | NA | NA | 70 | 70 |
| 1 | Hosp 3 | 47 | NA | NA | 47 |
| 2 | Hosp 3 | NA | 70 | NA | 70 |
| 3 | Hosp 3 | NA | NA | 38 | 38 |
| 1 | Hosp 4 | 82 | NA | NA | 82 |
| 2 | Hosp 4 | NA | 87 | NA | 87 |
| 3 | Hosp 4 | NA | NA | 46 | 46 |
| Total |
|
266 | 245 | 162 | 673 |
N.B. En este caso, tuvimos que especificar que sólo se incluyeran las tres variables Facility, Year, y Cases, ya que la variable adicional Measurement interferiría en la creación de la tabla:
df_combined %>%
pivot_wider(
names_from = "Year",
values_from = "Cases"
) %>%
knitr::kable()| Measurement | Facility | 2000 | 2001 | 2002 |
|---|---|---|---|---|
| 1 | Hosp 4 | 82 | NA | NA |
| 1 | Hosp 3 | 47 | NA | NA |
| 1 | Hosp 2 | 71 | NA | NA |
| 1 | Hosp 1 | 66 | NA | NA |
| 2 | Hosp 4 | NA | 87 | NA |
| 2 | Hosp 3 | NA | 70 | NA |
| 2 | Hosp 2 | NA | 62 | NA |
| 2 | Hosp 1 | NA | 26 | NA |
| 3 | Hosp 4 | NA | NA | 46 |
| 3 | Hosp 3 | NA | NA | 38 |
| 3 | Hosp 2 | NA | NA | 70 |
| 3 | Hosp 1 | NA | NA | 8 |
12.5 Recursos
Aquí hay un tutorial útil