3 Fundamentos de R

Bienvenido.
Esta página repasa los aspectos esenciales de R. No pretende ser un tutorial exhaustivo, pero proporciona los fundamentos y puede ser útil para refrescar la memoria. La sección de Recursos para el aprendizaje enlaza con tutoriales más completos.
Partes de esta página han sido adaptadas con permiso del proyecto R4Epis.
Consulta la página sobre Transición a R para obtener consejos sobre cómo cambiar a R desde STATA, SAS o Excel.
3.1 ¿Por qué utilizar R?
Como se indica en el sitio web de R project, éste es un lenguaje de programación y un entorno para la computación estadística y gráficos. Es muy versátil, ampliable y dirigido por la comunidad.
Coste
El uso de R es gratuito. Hay una fuerte ética en la comunidad de material libre y de código abierto.
Reproducibilidad
La gestión y el análisis de los datos a través de un lenguaje de programación (en comparación con Excel u otra herramienta principalmente manual) mejora la reproducibilidad, facilita la detección de errores y alivia la carga de trabajo.
Comunidad
La comunidad de usuarios de R es enorme y colaborativa. Cada día se desarrollan nuevos paquetes y herramientas para abordar problemas cotidianos, que son examinados por la comunidad de usuarios. Por ejemplo, R-Ladies es una asociación mundial cuya misión es promover la diversidad de género en la comunidad de R, siendo una de las mayores asociaciones de usuarios de R. Es probable que tengas un grupo cerca.
3.2 Términos clave
RStudio - RStudio es una interfaz gráfica de usuario (GUI) para facilitar el uso de R. Lee más en la sección RStudio.
Objetos - Todo lo que se almacena en R - conjuntos de datos, variables, una lista de nombres de pueblos, un número total de población, incluso resultados como gráficos - son objetos a los que se les asigna un nombre y pueden ser referenciados en comandos posteriores. Lee más en la sección Objetos.
Funciones - Una función es una operación de código que acepta entradas y devuelve una salida transformada. Lee más en la sección Funciones.
Paquetes - Un paquete de R es un conjunto de funciones que se pueden compartir. Lee más en la sección Paquetes.
Scripts - Un script es un archivo que contiene una serie comandos. Lee más en la sección Scripts
3.3 Recursos para aprender
Recursos en RStudio
Documentación de ayuda
Busca en la pestaña “Help” de RStudio la documentación sobre los paquetes de R y funciones específicas. Esto está dentro del panel que también contiene Archivos, Gráficos y Paquetes (normalmente en el panel inferior derecho). También puedes escribir el nombre de un paquete o función en la consola de R después de un signo de interrogación para abrir la página de ayuda correspondiente. No incluyas paréntesis.
Por ejemplo: ?filter o ?diagrammeR.
Tutoriales interactivos
Hay varias formas de aprender R de forma interactiva dentro de RStudio.
El propio RStudio ofrece un Tutorial que se encuentra en el paquete de R learnr. Simplemente instala este paquete y abre un tutorial a través de la nueva pestaña “Tutorial” en el panel superior derecho de RStudio (que también contiene las pestañas Environment e History).
El paquete de R swirl ofrece cursos interactivos en la consola de R. Instala y carga este paquete, luego ejecuta el comando swirl() (paréntesis vacío) en la consola de R. Verás que aparecen indicaciones en la consola. Responde escribiendo en la misma. Te guiará a través de un curso de tu elección.
Hojas de referencia
Hay muchas “hojas de referencias o trucos” (Cheatsheets) en PDF disponibles en el sitio web de RStudio, por ejemplo:
- Factores con el paquete forcats
- Fechas y horarios con el paquete lubridate
- Cadenas con el paquete stringr
- Operaciones iterativas con el paquete purrr
- Importación de datos
- Transformación de datos con el paquete dplyr
- R Markdown (para crear documentos como PDF, Word, Powerpoint…)
- Shiny (para crear aplicaciones web interactivas)
- Visualización de datos con el paquete ggplot2
- Cartografía (GIS)
- Mapas interactivos con el paquete leaflet
- Python con R (paquete reticulate)
En este enlace puedes encontrar un recurso en línea, específicamente para los usuarios de Excel
R tiene una vibrante comunidad en Twitter en la que puedes aprender trucos, atajos y noticias: sigue estas cuentas:
- Síguenos! @epiRhandbook
- R Function A Day @rfuntionaday (Es un recurso increíble)
- R para ciencia de datos @rstats4ds
- RStudio @RStudio
- Trucos de RStudio @rstudiotips
- R-Bloggers @Rbloggers
- R-ladies @RLadiesGlobal
- Hadley Wickham @hadleywickham
También:
#epitwitter y #rstats
Recursos gratuitos en línea
Un texto definitivo es el libro R for Data Science de Garrett Grolemund y Hadley Wickham
El sitio web del proyecto R4Epis tiene como objetivo “desarrollar herramientas estandarizadas de limpieza de datos, análisis y elaboración de informes para cubrir los tipos comunes de brotes y estudios realizados en la población en un entorno de respuesta de emergencia de MSF”. Se pueden encontrar materiales de formación sobre los fundamentos de R, plantillas para informes de RMarkdown sobre brotes y encuestas, y tutoriales para ayudar a configurarlos.
Idiomas distintos del inglés
3.4 Instalación
R y RStudio
Cómo instalar R
Visita este sitio web https://www.r-project.org/ y descarga la última versión de R adecuada a tu ordenador.
Cómo instalar RStudio
Visita este sitio web https://rstudio.com/products/rstudio/download/ y descarga la última versión gratuita de RStudio para escritorio adecuada para tu ordenador.
Permisos
Ten en cuenta que debes instalar R y RStudio en una unidad donde tengas permisos de lectura y escritura. De lo contrario, la capacidad para instalar paquetes de R (algo frecuente) se verá afectada. Si tienes problemas, intenta abrir RStudio con el botón derecho en el icono y seleccionando “Ejecutar como administrador”. Puedes encontrar otros consejos en la página R en unidades de red.
Cómo actualizar R y RStudio
Tu versión de R se muestra al inicio de la consola de R. También puede ejecutar sessionInfo().
Para actualizar R, puedes ir al sitio web mencionado anteriormente y vuelva a instalar R. También puede utilizar el paquete installr (en Windows) ejecutando installr::updateR(). Esto abrirá cuadros de diálogo para ayudarle a descargar la última versión de R y actualizar sus paquetes a la nueva versión de R. Puedes encontrar más detalles en la documentación de installr.
Ten en cuenta que la versión antigua de R seguirá existiendo en tu ordenador. Puedes ejecutar temporalmente una versión anterior (una “instalación” más antigua) de R clicando en “Herramientas” -> “Opciones globales” en RStudio y eligiendo una versión de R. Esto puede ser útil si quieres utilizar un paquete que no ha sido actualizado para funcionar en la versión más reciente de R.
Para actualizar RStudio, puede ir a la página web anterior y volver a descargar RStudio. Otra opción es clicar en “Ayuda” -> “Buscar actualizaciones” dentro de RStudio, pero esto puede no mostrar las últimas actualizaciones.
Para ver qué versiones de R, RStudio o paquetes se utilizaron cuando se hizo este Manual, consulta la página de Notas técnicas y editoriales.
Otros programas que puedes necesitar instalar
- TinyTeX (para compilar un documento RMarkdown en PDF)
- Pandoc (para compilar documentos RMarkdown)
- RTools (para construir paquetes para R)
- phantomjs (para guardar imágenes fijas de redes animadas, como cadenas de transmisión)
TinyTex
TinyTex es una distribución LaTeX personalizada, útil cuando se trata de producir PDFs desde R. Ver https://yihui.org/tinytex/ para más información.
Para instalar TinyTex desde R:
install.packages('tinytex')
tinytex::install_tinytex()
# para desinstalar TinyTeX, ejecutar tinytex::uninstall_tinytex()Pandoc
Pandoc es un conversor de documentos, un software separado de R. Viene incluido con RStudio y no debería ser necesario descargarlo. Ayuda en el proceso de conversión de documentos Rmarkdown a distintos formatos como pdf o html y añade funcionalidades complejas.
RTools
RTools es una colección de software para construir paquetes para R
Se instala desde este sitio web:https://cran.r-project.org/bin/windows/Rtools/
phantomjs
Esto se utiliza a menudo para hacer “capturas de pantalla” de las páginas web. Por ejemplo, cuando se hace una cadena de transmisión con el paquete epicontacts, se produce un archivo HTML que es interactivo y dinámico. Si deseas una imagen estática, puede ser útil utilizar el paquete webshot para automatizar este proceso. Para ello se necesita el programa externo “phantomjs”. Puedes instalar phantomjs a través del paquete webshot con el comando webshot::install_phantomjs().
3.5 RStudio
Orientación de RStudio
Primero, abre RStudio. Como sus iconos pueden ser muy similares, asegúrate de que estás abriendo RStudio y no R.
Para que RStudio funcione, también debes tener instalado R en el ordenador (consulta las instrucciones de instalación más arriba).
RStudio es una interfaz (GUI) para facilitar el uso de R. Puedes pensar que R es el motor de un vehículo, que hace el trabajo crucial, y RStudio es la carrocería del vehículo (con asientos, accesorios, etc.) que te ayuda a usar el motor para avanzar. Puedes ver la hoja de trucos completa de la interfaz de usuario de RStudio (PDF) aquí
Por defecto, RStudio muestra cuatro paneles rectangulares.
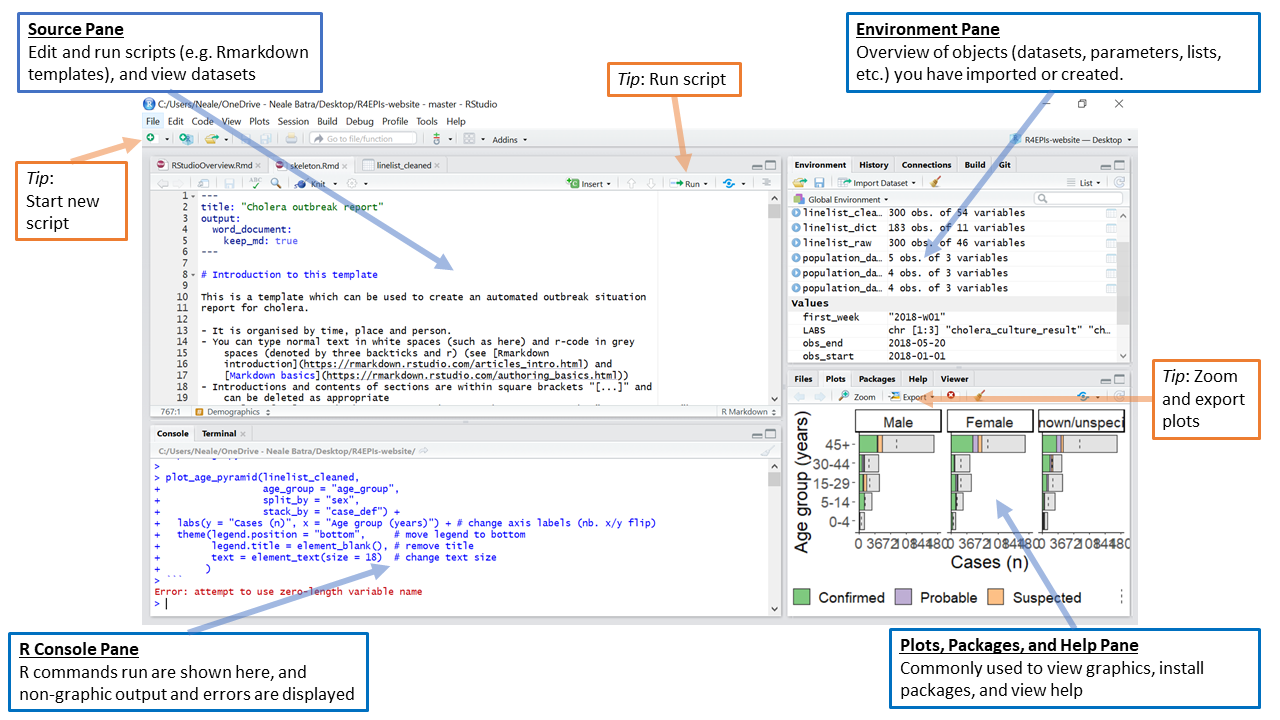
CONSEJO: Si tu RStudio sólo muestra un panel izquierdo es porque aún no tiene ningún script abierto.
El panel Source
Este panel de código Fuente u origen, por defecto en la parte superior izquierda, es un espacio para editar, ejecutar y guardar tus scripts. Los scripts contienen los comandos que desea ejecutar. Este panel también puede mostrar conjuntos de datos (data frames) para su visualización.
Para los usuarios de Stata, este panel es similar a las ventanas de Do-file y del Editor de Datos.
El panel Console
La consola de R es el hogar del “motor” de R es, por defecto, el panel izquierdo o inferior izquierdo en R Studio. Aquí es donde se ejecutan realmente los comandos y aparecen las salidas no gráficas y los mensajes de error/advertencia. Puedes introducir y ejecutar directamente comandos en la Consola de R, pero ten en cuenta que estos comandos no se guardan como cuando se ejecutan comandos desde un script.
Si estás familiarizado con Stata, la consola de R es como la ventana de comandos y también la ventana de resultados.
El panel Environment
Este panel de Entorno, por defecto en la parte superior derecha, se utiliza más a menudo para ver breves resúmenes de los objetos en el Entorno R en la sesión actual. Estos objetos pueden incluir conjuntos de datos importados, modificados o creados, parámetros que hayas definido (por ejemplo, una semana epi específica para el análisis), o vectores o listas que hayas definido durante el análisis (por ejemplo, nombres de regiones). Puedes clicar en la flecha situada junto al nombre de un dataframe para ver sus variables.
En Stata, esto es muy similar a la ventana del Gestor de Variables.
Este panel también contiene History donde puede ver los comandos ejecutados anteriormente. También tiene una pestaña “Tutorial” donde puedes completar tutoriales interactivos de R si tienes el paquete learnr instalado. También tiene una pestaña de “Conexiones” para las conexiones externas, y puede tener un panel “Git” si decides interactuar con Github.
Panel Files, Plots, Packages, Help, Viewer Este panel inferior derecho incluye varias pestañas importantes. La pestaña Files (Archivos) permite navegar por las carpetas y puede utilizarse para abrir o eliminar archivos. En la pestaña Plots (Gráficos), se mostrarán todos los gráficos, incluyendo los mapas. Las salidas interactivas o HTML se mostrarán en la pestaña Viewer (Visor). El panel Packages (Paquetes) permite ver, instalar, actualizar, eliminar, cargar/descargar paquetes de R y ver qué versión del paquete tiene. En la sección de paquetes más abajo se puede aprender más sobre los paquetes. Por último, en el panel de Ayuda (Help) se mostrará la documentación y los archivos de ayuda.
Este panel contiene los equivalentes en Stata de las ventanas Plots Manager y Project Manager.
Configuración de RStudio
Cambia la configuración y la apariencia de RStudio en el menú desplegable Tools (Herramientas), seleccionando Global Options (Opciones globales). Allí puede cambiar la configuración por defecto, incluyendo la apariencia/color de fondo.
Reiniciar
Si se cuelga R, se puede reiniciar yendo al menú Sesión y clicando en “Restart R” (Reiniciar R)“. Esto evita la molestia de cerrar y abrir RStudio. Al hacer esto, se eliminará todo el entorno de esa sesión de R.
Atajos de teclado
Algunos atajos de teclado muy útiles están abajo. Se pueden ver todos los atajos de teclado para Windows, Max y Linux en la segunda página de esta hoja de trucos de la interfaz de usuario de RStudio.
| Windows/Linux | Mac | Acción |
|---|---|---|
| Esc | Esc | Interrumpir el comando actual (útil si accidentalmente ejecutó un comando incompleto y no puede evitar ver “+” en la consola de R) |
| Ctrl+s | Cmd+s | Guardar (script) |
| Tab | Tab | Autocompletar |
| Ctrl + Enter | Cmd + Enter | Ejecutar la(s) línea(s) de código actual(es) |
| Ctrl + Mayús + C | Cmd + Shift + c | Comentar/descomentar las líneas resaltadas |
| Alt + * | Opción + * | Insertar <- |
| Ctrl + Shift + m | Cmd + Shift + m | Insertar %>% |
| Ctrl + l | Cmd + l | Limpiar la consola de R |
| Ctrl + Alt + b | Cmd + Opción + b | Ejecutar desde el inicio hasta la línea actual |
| Ctrl + Alt + t | Cmd + Opción + t | Ejecutar la sección de código actual (R Markdown) |
| Ctrl + Alt + i | Cmd + Shift + r | Insertar un trozo (chunk) de código (en R Markdown) |
| Ctrl + Alt + c | Cmd + Opción + c | Ejecutar el código chunk actual (R Markdown) |
| flechas arriba/abajo en la consola R | En el mismo | Recorrer los comandos ejecutados recientemente |
| Shift + flechas arriba/abajo en el script | En el mismo | Seleccionar varias líneas de código |
| Ctrl + f | Cmd + f | Buscar y reemplazar en el script actual |
| Ctrl + Mayús + f | Cmd + Shift + f | Buscar en archivos (buscar/reemplazar en muchos scripts) |
| Alt + l | Cmd + Opción + l | Plegar el código seleccionado |
| Shift + Alt + l | Cmd + Shift + Opción+l Desplegar el código seleccionado |
CONSEJO: Utiliza la tecla Tab cuando escribas para activar la función de autocompletar de RStudio. Esto puede evitar errores de ortografía. Pulsa el tabulador mientras escribes para que aparezca un menú desplegable de posibles funciones y objetos, basándose en lo que escrito hasta ese momento.
3.6 Funciones
Las funciones son la pieza principal en el uso de R. Las funciones son la forma de realizar tareas y operaciones. Muchas vienen instaladas con R, mientras muchas otras están disponibles para su descarga en paquetes (explicados en la sección de paquetes), ¡E incluso, puedes escribir tus propias funciones personalizadas.
Esta sección básica sobre las funciones explica:
- Qué es una función y cómo funciona
- Qué son los argumentos de una función
- Cómo obtener ayuda para entender una función
Una nota rápida sobre la sintaxis: En este manual, las funciones se escriben en código-texto con paréntesis abiertos, así: filter(). Como se explica en la sección de paquetes, las funciones se descargan dentro de los paquetes. En este manual, los nombres de los paquetes se escriben en negrita, como dplyr. A veces en el código de ejemplo puede ver el nombre de la función vinculado explícitamente al nombre de su paquete con dos dos puntos (::) como: dplyr::filter(). El propósito de esta vinculación se explica en la sección de paquetes.
Funciones simples
Una función es como una máquina que recibe entradas, realiza alguna acción con esas entradas y produce una salida. El resultado depende de la función.
Las funciones suelen operar sobre algún objeto colocado dentro de los paréntesis de la función. Por ejemplo, la función sqrt() calcula la raíz cuadrada de un número:
sqrt(49)## [1] 7El objeto proporcionado a una función también puede ser una columna de datos (véase la sección Objetos para conocer todos los tipos de objetos). Dado que R puede almacenar múltiples conjuntos de datos, tendrás que especificar tanto el set de datos como la columna. Una forma de hacerlo es utilizar la notación $ para vincular el nombre de los datos y el nombre de la columna (dataset$column). En el siguiente ejemplo, la función summary() se aplica a la columna numérica age en los datos linelist, y la salida es un resumen de los valores numéricos y faltantes de la columna.
# Muestra estadísticas resumen de la columna 'age' del dataset 'linelist'
summary(linelist$age)## Min. 1st Qu. Median Mean 3rd Qu. Max. NA's
## 0.00 6.00 13.00 16.07 23.00 84.00 86NOTA: Entre bastidores, una función representa un código adicional complejo que ha sido envuelto para el usuario en un comando sencillo.
Funciones con multiples argumentos
Las funciones suelen pedir varias entradas, llamadas argumentos, situadas dentro del paréntesis de la función, normalmente separadas por comas.
- Algunos argumentos son necesarios para que la función funcione correctamente, mientras otros son opcionales
- Los argumentos opcionales tienen una configuración por defecto
- Los argumentos pueden tomar caracteres, números, lógica (TRUE/FALSE) y otras entradas
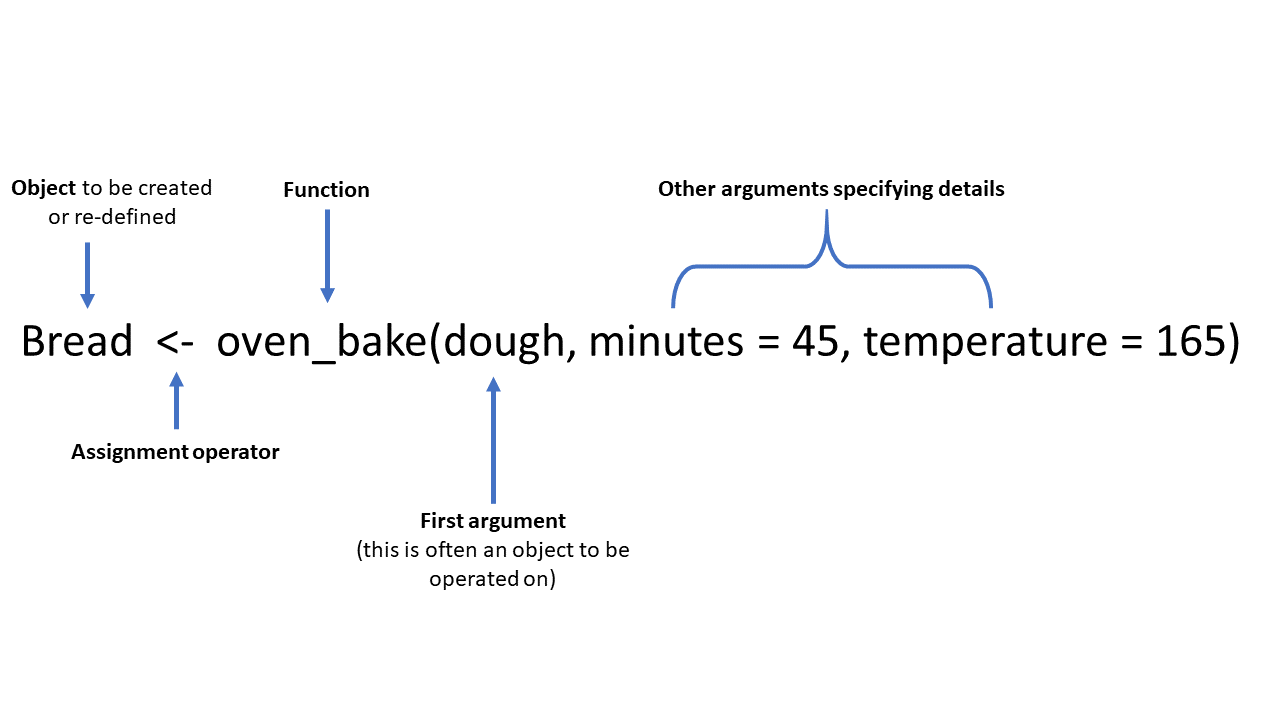
He aquí una divertida función ficticia, llamada oven_bake(), como ejemplo de una función típica (hacer en el horno). Toma un objeto de entrada (“input”) (por ejemplo, una base de datos o en este ejemplo “masa”) y realiza operaciones en él según lo especificado por los argumentos adicionales (minutes = y temperature =). La salida (“output”) puede ir a la consola, o guardarse como un objeto utilizando el operador de asignación <-.
En un ejemplo más realista, el comando age_pyramid() que aparece a continuación produce un gráfico de pirámide de edad basado en grupos de edad definidos y una columna de división binaria, como gender. La función recibe tres argumentos dentro de los paréntesis, separados por comas. Los valores suministrados a los argumentos establecen linelist como los datos (dataframe) a utilizar, age_cat5 como la columna a contar, y gender como la columna binaria a utilizar para dividir la pirámide por color según género.
# Crea una pirámide de edad
age_pyramid(data = linelist, age_group = "age_cat5", split_by = "gender")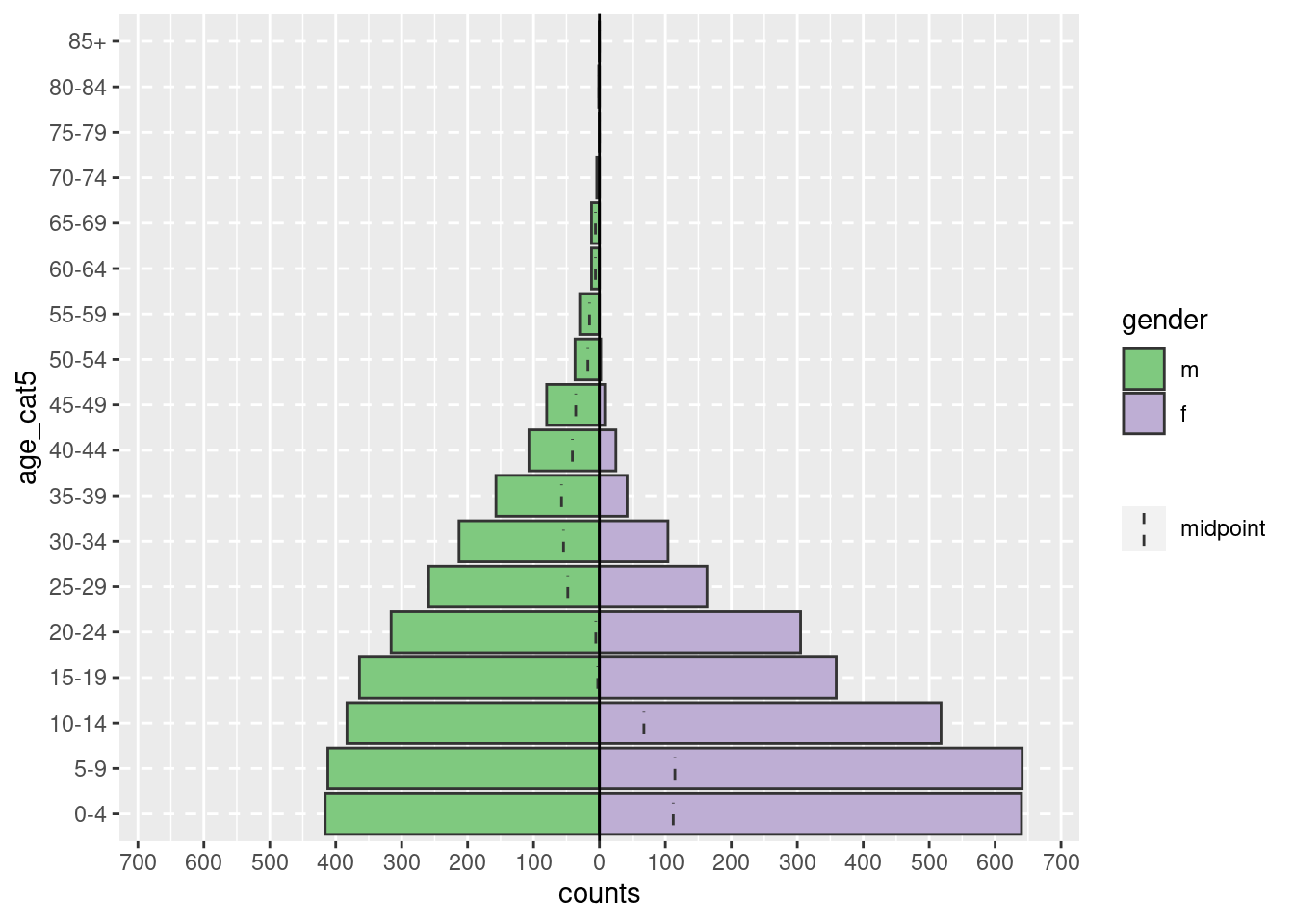
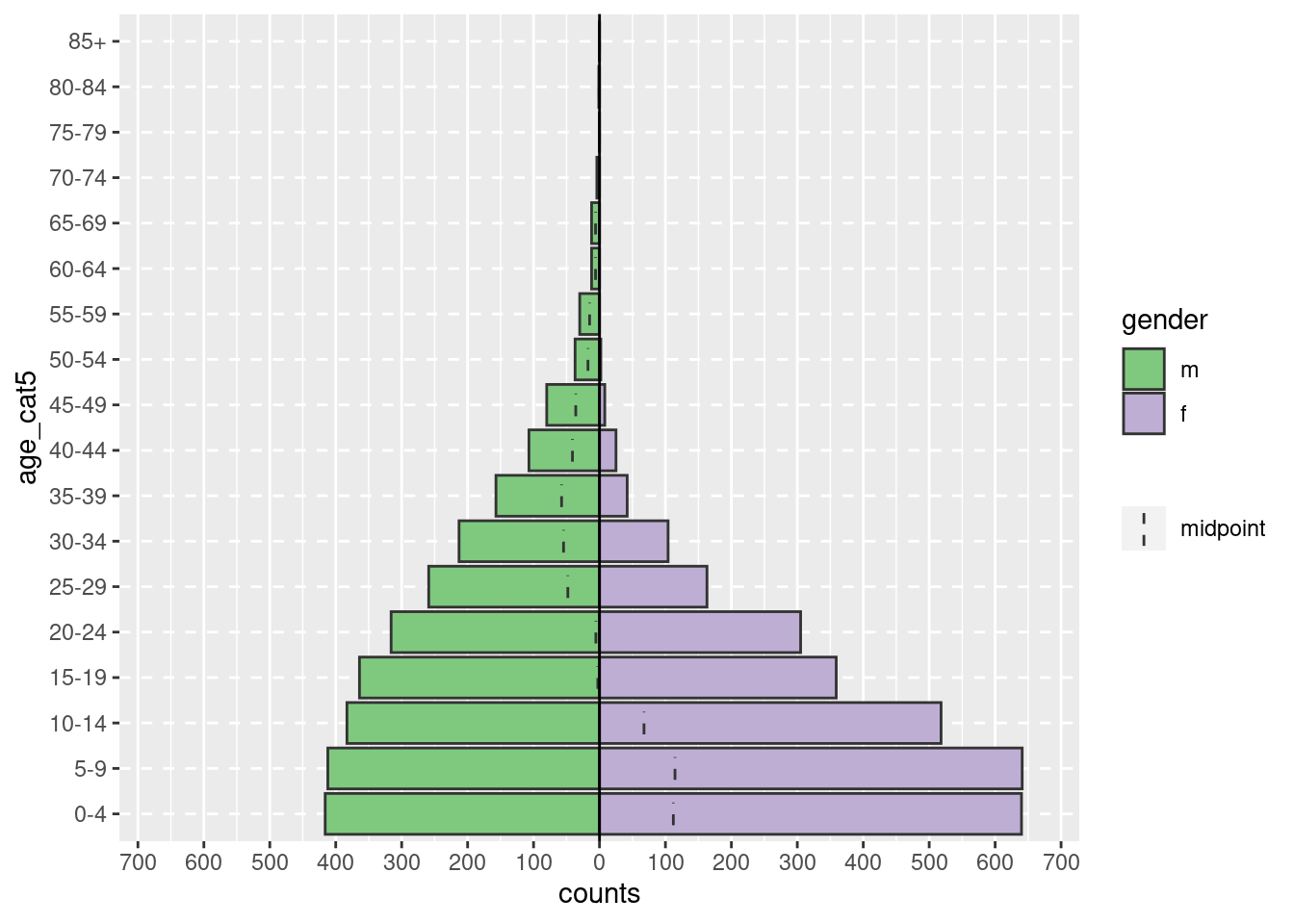
El comando anterior puede escribirse de forma equivalente a la de más abajo, con un estilo más largo con una nueva línea para cada argumento. Este estilo puede ser más fácil de leer, y más fácil de escribir “comentarios” con # para explicar cada segmento de código (¡comentar en el código es considerado una buena práctica!). Para ejecutar este comando más largo puedes seleccionar todo el texto y clicar en “Ejecutar”, o simplemente colocar el cursor en la primera línea y luego clicar las teclas Ctrl y Enter simultáneamente.
# Crea una pirámide de edad
age_pyramid(
data = linelist, # usa los datos de linelist
age_group = "age_cat5", # especifica la columna para los grupos de edad
split_by = "gender" # usa la columna gender para los dos lados de la pirámide
)
No es necesario especificar la primera mitad de una asignación de argumentos (por ejemplo, data =) si los argumentos se escriben en su orden específico (especificado en la documentación de la función). El código siguiente produce exactamente la misma pirámide que la anterior, porque la función espera ese orden de los argumentos: conjunto de datos, la variable para age_group, y la variable parasplit_by` .
# Esta orden produce exactamente el mismo gráfico que la anterior
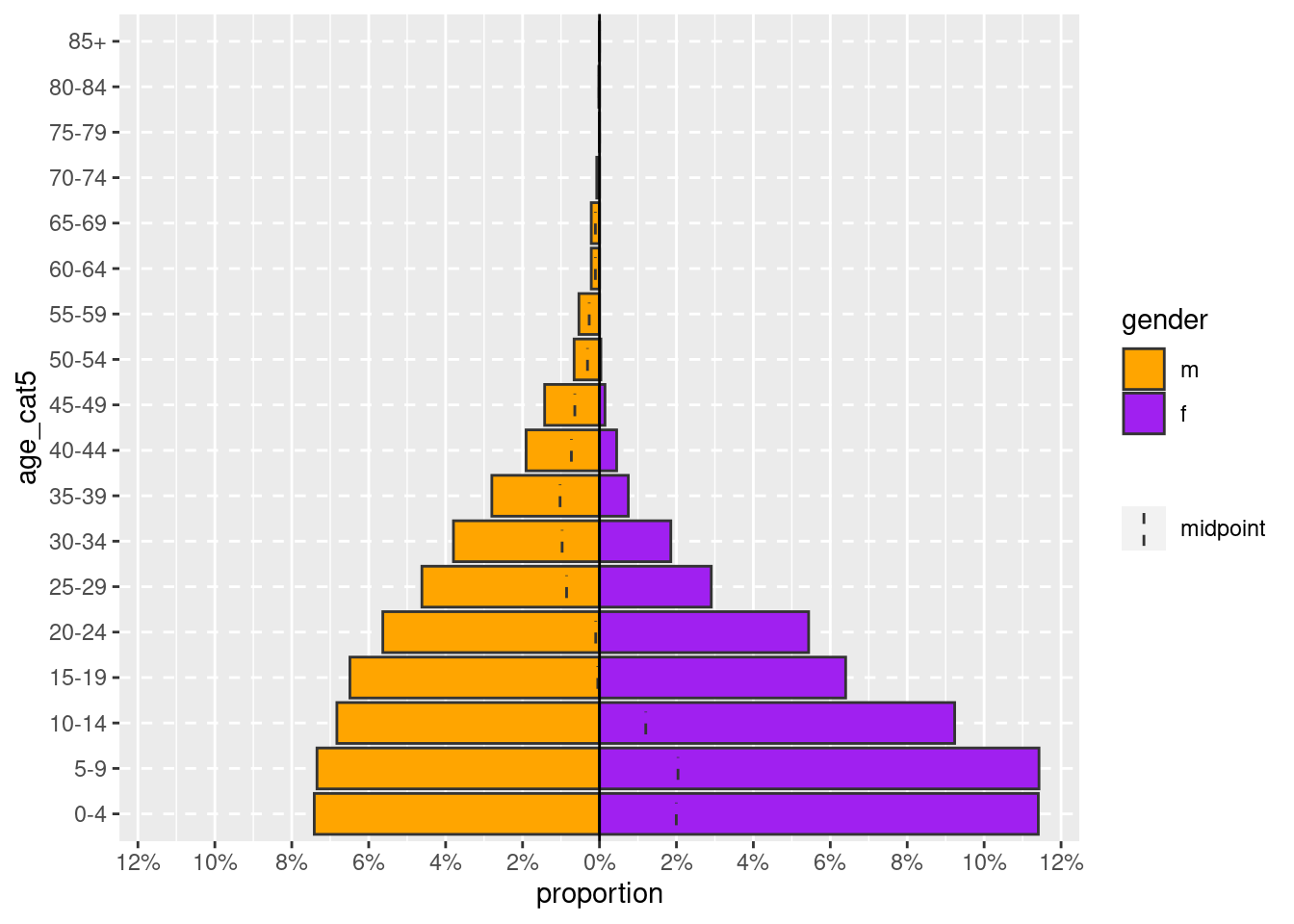
age_pyramid(linelist, "age_cat5", "gender")Un comando age_pyramid() más complejo podría incluir argumentos opcionales para:
- Mostrar proporciones en lugar de recuentos (estableciendo
proportional = TRUEcuando el valor por defecto esFALSE) - Especificar los dos colores a utilizar (
pal =es la abreviatura de “paleta” y se suministra con un vector de dos nombres de color. Para saber cómo se hace un vector con la funciónc()puedes consultar la página de objetos .
NOTA: En los argumentos en los que se especifican ambas partes del argumento (por ejemplo, proporcional = TRUE), no importa el orden de estos argumentos.
age_pyramid(
linelist, # usa los casos de linelist
"age_cat5", # columna de los grupos de edad
"gender", # dividido por género
proportional = TRUE, # porcentajes en vez de números absolutos
pal = c("orange", "purple") # colores
)
Escribir funciones
R es un lenguaje orientado a las funciones, por lo que deberías sentirte capacitado para escribir tus propias funciones. La creación de funciones aporta varias ventajas:
- Facilita la programación modular, es decir, la separación del código en partes independientes y manejables.
- Sustituye el repetitivo copiar y pegar, que puede dar lugar a errores
- Se puede dar a las piezas de código nombres fáciles de recordar
En la página Escribir funciones se trata en profundidad cómo escribir funciones.
3.7 Paquetes
Los paquetes contienen funciones.
Un paquete de R es un conjunto de código y documentación que se puede compartir y que contiene funciones predefinidas. Los usuarios de la comunidad R desarrollan paquetes todo el tiempo atendiendo a problemas específicos, ¡es probable que alguno pueda ayudarte en tu trabajo! En tu uso de R instalarás y utilizarás cientos de paquetes.
En la instalación, R contiene paquetes “base” y funciones que realizan tareas elementales comunes. Pero muchos usuarios de R crean funciones especializadas, que son verificadas por la comunidad de R y que puedes descargar como paquete para tu propio uso. En este manual, los nombres de los paquetes se escriben en negrita. Uno de los aspectos más desafiantes de R es que a menudo hay muchas funciones o paquetes donde elegir para una tarea determinada.
Instalar y cargar
Las funciones están contenidas en paquetes que pueden descargarse (“instalarse”) en tu ordenador desde Internet. Una vez descargado un paquete, se almacena en tu “librería”. Puedes acceder a las funciones que contiene durante una sesión de R “cargando” el paquete.
Piensa en R como tu librería personal: Cuando se descarga un paquete, tu librería adquiere un nuevo libro de funciones, pero cada vez que quieras utilizar una función de ese libro, debes tomar prestado (“cargar”) ese libro de tu librería.
En resumen: para utilizar las funciones disponibles en un paquete de R, hay que realizar dos pasos:
- El paquete debe ser instalado (una vez), y
- El paquete debe ser cargado (cada sesión de R)
Tu librería
Tu “librería” es en realidad una carpeta en tu ordenador, que contiene una carpeta para cada paquete que se ha instalado. Averigua dónde está instalado R en tu ordenador y, si es windows, busca una carpeta llamada “win-library”. Por ejemplo: R-library\4.0 (4.0 es la versión de R - tendrás una librería diferente para cada versión de R que haya descargado).
Puedes imprimir la ruta del archivo de tu librería introduciendo .libPaths() (paréntesis vacíos). Esto resulta especialmente importante si se trabaja con R en unidades de red.
Instalar desde CRAN
Lo habitual es que los usuarios de R descarguen paquetes de CRAN. CRAN (Comprehensive R Archive Network) es un almacén público online de paquetes de R que han sido publicados por los miembros de la comunidad R.
¿Te preocupan los virus y la seguridad al descargar un paquete de CRAN?. Lee este artículo sobre el tema.
Cómo instalar y cargar
En este manual, sugerimos utilizar el paquete pacman (abreviatura de “packages manager”). Ofrece una interesante función p_load() que instalará un paquete si es necesario y lo cargará para su uso en la sesión actual de R.
La sintaxis es bastante sencilla. Sólo hay que listar los nombres de los paquetes dentro de los paréntesis de p_load(), separados por comas. Este comando instalará los paquetes rio, tidyverse y here si aún no están instalados, y los cargará para su uso. Esto hace que el enfoque de p_load() sea conveniente y conciso si se comparten scripts con otros. Ten en cuenta que los nombres de los paquetes distinguen entre mayúsculas y minúsculas.
# Instala (si es necesario) y carga los paquetes a utilizar
pacman::p_load(rio, tidyverse, here)Fíjate que hemos utilizado la sintaxis pacman::p_load() que escribe explícitamente el nombre del paquete (pacman) antes del nombre de la función (p_load()), conectado por dos dos puntos ::. Esta sintaxis es útil porque también carga el paquete pacman (suponiendo que ya esté instalado).
Hay funciones de R base alternativas que verás a menudo. La función de R base para instalar un paquete es install.packages(). El nombre del paquete a instalar debe proporcionarse entre paréntesis entre comillas. Si deseas instalar varios paquetes en un solo comando, deben ser listados dentro de un vector de caracteres c().
Nota: este comando instala un paquete, pero no lo carga para utilizarlo en la sesión actual.
# instala un solo paquete con R base
install.packages("tidyverse")
# instala múltiples paquetes con R base
install.packages(c("tidyverse", "rio", "here"))La instalación también se puede realizar clicando en el panel “Packages” de RStudio, luego en “Install” y buscando el nombre del paquete deseado.
La función alternativa para cargar un paquete (después de haberlo instalado) es library(). Sólo puedes cargar un paquete a la vez (otra razón para usar p_load()). Se puede escribir el nombre del paquete con o sin comillas.
Para comprobar si un paquete está instalado y/o cargado, puedes mirar en la pestaña de Packages de RStudio. Si el paquete está instalado, se muestra allí con el número de versión. Si su casilla está marcada, está cargado para la sesión actual.
Instalar desde Github
A veces, necesitas instalar un paquete que aún no está disponible en CRAN. O tal vez el paquete está disponible en CRAN pero quieres la versión de desarrollo con nuevas características que aún no se ofrecen en la versión más estable publicada en CRAN. Éstas suelen estar alojadas en el sitio web github.com en un “repositorio” de código gratuito y de acceso público. Lee más sobre Github en la página del manual sobre Control de versiones y colaboración con Git y Github.
Para descargar los paquetes de R desde Github, puedes utilizar la función p_load_gh() de pacman, que instalará el paquete si es necesario, y lo cargará para utilizarlo en tu sesión actual de R. Las alternativas de instalación incluyen el uso de los paquetes remotes o devtools. Puedes leer más sobre todas las funciones de pacman en la documentación del paquete.
Para instalar desde Github, tienes que proporcionar más información. Debe proporcionar:
- El ID de Github del propietario del repositorio
- El nombre del repositorio que contiene el paquete
- (opcional) El nombre de la “rama” (versión de desarrollo específica) que quieras descargar
En los ejemplos siguientes, la primera palabra entre comillas es el ID de Github del propietario del repositorio, después de la barra es el nombre del repositorio (el nombre del paquete).
# instala/carga el paquete epicontacts desde su repositorio de Github
p_load_gh("reconhub/epicontacts")Si quieres instalar desde una “rama” (versión) distinta de la rama principal, añade el nombre de la rama tras una “@”, después del nombre del repositorio.
# instala el paquete epicontacts de la rama "timeline" desde Github
p_load_gh("reconhub/epicontacts@timeline")Si no hay diferencia entre la versión de Github y la versión en tu ordenador, no se realizará ninguna acción. Puedes “forzar” una reinstalación usando p_load_current_gh() con el argumento update = TRUE. Puedes leer más sobre pacman en esta viñeta online
Instalar desde ZIP o TAR
Puedes instalar el paquete desde una URL:
packageurl <- "https://cran.r-project.org/src/contrib/Archive/dsr/dsr_0.2.2.tar.gz"
install.packages(packageurl, repos=NULL, type="source")O bien, descargarlo en tu ordenador en un archivo comprimido:
Opción 1: utilizar install_local() del paquete remotes
remotes::install_local("~/Downloads/dplyr-master.zip")Opción 2: utilizando install.packages() de R bases, proporcionando la ruta del archivo ZIP y estableciendo type = "source" y repos = NULL.
install.packages("~/Downloads/dplyr-master.zip", repos=NULL, type="source")Sintaxis del código
Para mayor claridad en este manual, las funciones van a veces precedidas por el nombre de su paquete utilizando el símbolo :: de la siguiente manera: nombre_del_paquete::nombre_de_la_función()
Una vez cargado un paquete para una sesión, este estilo explícito no es necesario. Se puede utilizar simplemente nombre_de_la_funcion(). Sin embargo, escribir el nombre del paquete es útil cuando el nombre de una función es común y puede existir en varios paquetes (por ejemplo, plot()). Escribir el nombre del paquete también cargará el paquete si no está todavía cargado.
# Este comando usa el paquete "rio" y su función "import()" para importar un dataset
linelist <- rio::import("linelist.xlsx", which = "Sheet1")Ayuda sobre una función
Para leer más sobre una función, se puede buscar en la pestaña Ayuda de la parte inferior derecha de RStudio. También se puede ejecutar un comando como ?nombre_de_la_funcion (escribe el nombre de la función después de un signo de cerrar interrogación) y aparecerá la página de ayuda en la pestaña de ayuda. Por último, intenta buscar otros recursos en Internet.
Actualizar paquetes
Puedes actualizar los paquetes reinstalándolos. También puedes clicar en el botón verde “Update” en la pestaña de paquetes de RStudio para ver qué paquetes tienen nuevas versiones para instalar. Ten en cuenta que tu código antiguo puede necesitar ser actualizado si hay una revisión importante en el funcionamiento de una función.
Eliminar paquetes
Utiliza p_delete() de pacman, o remove.packages() de utils. Alternativamente, puedes buscar la carpeta que lo contiene en tu librería y borrarla manualmente.
Dependencias
Los paquetes a menudo dependen de otros paquetes para funcionar. Estos se llaman dependencias. Si una dependencia no está instalada, entonces el paquete que depende de ella también puede no instalarse.
Se pueden ver las dependencias de un paquete con p_depends(), y ver qué paquetes dependen de él con p_depends_reverse()
Funciones enmascaradas
No es raro que dos o más paquetes contengan el mismo nombre de función. Por ejemplo, el paquete dplyr tiene una función filter(), pero también la tiene el paquete stats. La función filter() por defecto depende del orden en que estos paquetes se cargan por primera vez en la sesión de R - el último será el predeterminado para el comando filter().
Puedes comprobar el orden en la Pestaña de Entorno de R Studio - clica en el desplegable de “Global Evironment” (Entorno Global) y mira el orden de los paquetes. Las funciones de los paquetes más bajos en esa lista desplegable enmascararán las funciones del mismo nombre en los paquetes que aparecen más arriba en la lista desplegable. Cuando se carga por primera vez un paquete, R advertirá en la consola si se está produciendo el enmascaramiento, pero esto puede ser fácil de pasar por alto.
Aquí hay formas de arreglar el enmascaramiento:
- Especifica el nombre del paquete en el comando. Por ejemplo, utiliza
dplyr::filter() - Reordena el orden de carga de los paquetes (por ejemplo, dentro de
p_load()), e iniciar una nueva sesión de R
Descargar
Para descargar (detach / unload) un paquete, utiliza este comando, con el nombre correcto del paquete y sólo dos puntos. Ten en cuenta que esto puede no resolver el enmascaramiento.
detach(package:PACKAGE_NAME_HERE, unload=TRUE)Instalar una versión anterior
Consulta esta guía para instalar una versión anterior de un paquete concreto.
Paquetes recomendados
Consulta la página de Paquetes recomendados para obtener una lista de paquetes que recomendamos para la epidemiología del día a día.
3.8 Scripts
Los scripts son una parte fundamental de la programación. Son documentos que contienen comandos (por ejemplo, funciones para crear y modificar datos, imprimir visualizaciones, etc.). Puedes guardar un script y volver a ejecutarlo más tarde. Almacenar y ejecutar tus comandos desde un script tiene muchas ventajas (frente a teclear los comandos uno a uno en la “línea de comandos” de la consola de R):
- Portabilidad: puedes compartir tu trabajo con otros enviándoles tus scripts
- Reproducibilidad: para que tú y los demás sepan exactamente lo que se ha hecho
- Control de versiones: para que puedas hacer un seguimiento de los cambios realizados por ti mismo o por tus colegas
- Comentarios/anotaciones: para explicar a tus compañeros lo que has hecho
Comentarios
En un script también puedes anotar (“comentar”) alrededor de su código R. Los comentarios son útiles para explicarte a tí mismo y a otros lectores lo que está haciendo. Puedes añadir un comentario escribiendo el símbolo de almohadilla (#) y el comentario después de él. El texto comentado aparecerá en un color diferente al del código R.
Cualquier código escrito después de la # no se ejecutará. Por lo tanto, colocar un # antes del código es también una forma útil de bloquear temporalmente una línea de código (“comentar”) si no quieres borrarla). Puedes comentar varias líneas a la vez resaltándolas y clicando Ctrl+Mayús+c (Cmd+Mayús+c en Mac).
# Un comentario puede ser una línea en sí mismo
# importar datos
linelist <- import("linelist_raw.xlsx") %>% # un comentario también puede venir después del código
# filter(age > 50) # También se puede utilizar para desactivar / quitar una línea de código
count()- Comenta lo que haces y por qué lo haces.
- Divide tu código en secciones lógicas
- Acompaña tu código con un texto describiendo paso a paso lo que está haciendo (por ejemplo, pasos numerados)
Estilo
Es importante ser consciente de tu estilo de codificación, especialmente si trabajas en equipo. Nosotros abogamos por la guía de estilo tidyverse . También hay paquetes como styler y lintr que te ayudan a ajustarte a este estilo.
Algunos puntos muy básicos para que tu código sea legible para los demás:
* Al nombrar los objetos, utiliza sólo letras minúsculas, números y guiones bajos _, por ejemplo, mis_datos.
* Utiliza espacios frecuentemene, incluso alrededor de los operadores, por ejemplo, n = 1 y edad_nueva <- edad_vieja + 3.
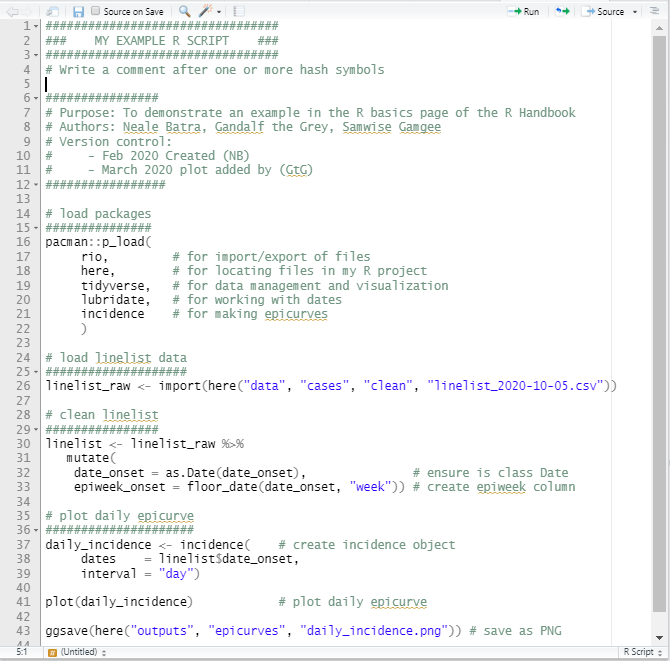
### Ejemplo de Script {.unnumbered}
A continuación se muestra un ejemplo de un breve script de R. ¡Recuerda!, cuanto mejor expliques brevemente el código con los comentarios, ¡más gustará a tus colegas!
R markdown
Un script de R markdown es un tipo de script de R en el que el propio script se convierte en un documento de salida (PDF, Word, HTML, Powerpoint, etc.). Se trata de herramientas increíblemente útiles y versátiles que suelen utilizarse para crear informes dinámicos y automatizados. ¡Hasta este sitio web y este manual se han hecho con scripts de R markdown!
Vale la pena señalar que los usuarios principiantes de R también pueden utilizar R Markdown - ¡no te dejes intimidar! Para saber más, consulta el capítulo del manual sobre Informes con R Markdown.
R notebooks
No hay ninguna diferencia entre escribir en Rmarkdown o R notebook. Sin embargo, la ejecución del documento difiere ligeramente. Consulta este sitio para obtener más detalles.
Shiny
Las aplicaciones/sitios web de Shiny están contenidas en un script, que debe llamarse app.R. Este archivo tiene tres componentes:
- Una interfaz de usuario (ui)
- Una función de servidor
- Una llamada a la función
shinyApp
Consulta la página del manual sobre Dashboards con Shiny, o este Tutorial de Shiny
Hace tiempo, el archivo anterior se dividía en dos archivos (ui.R y server.R)
Plegar código
Puedes contraer porciones de código para facilitar la lectura del script.
Para ello, crea una cabecera de texto con #, escribe tu cabecera y sigue con al menos 4 guiones (-), almohadillas (#) o iguales (=). Cuando hayas hecho esto, aparecerá una pequeña flecha en el “margen” de la izquierda (junto al número de fila). Puedes clicar en esta flecha y en el código de abajo hasta que la siguiente cabecera se pliegue y aparezca un icono de flecha doble en su lugar.
Para expandir el código, clica de nuevo en la flecha del margen o en el icono de la flecha doble. También hay atajos de teclado como se explica en la sección de RStudio de esta página.
Al crear cabeceras con #, también activarás el índice de contenidos en la parte inferior del script (véase más abajo) que puedes utilizar para navegar por el script. Se pueden crear subcabeceras añadiendo más símbolos #, por ejemplo # para las primarias, ## para las secundarias y ### para las terciarias.
A continuación se muestran dos versiones de un script de ejemplo. A la izquierda está el original con cabeceras comentadas. A la derecha, se han escrito cuatro guiones después de cada cabecera, haciéndolas plegables. Dos de ellas están plegadas, y se puede ver que la Tabla de Contenidos en la parte inferior muestra cada sección.
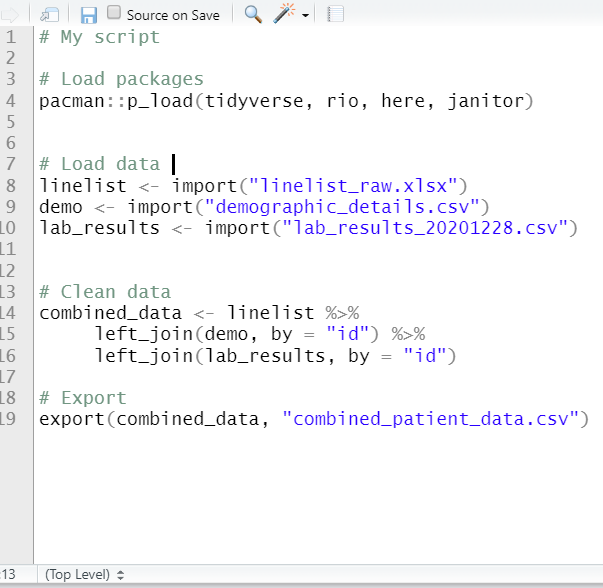
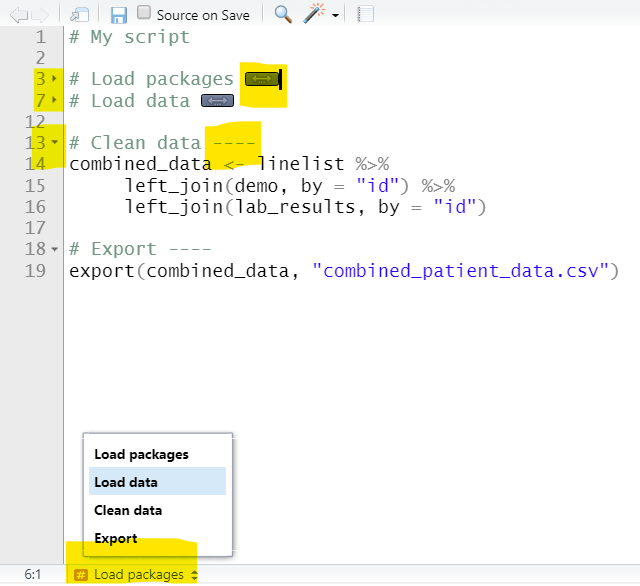
Otras áreas de código que son automáticamente elegibles para plegarlas son las zonas entre corchetes { } como las definiciones de funciones o los bloques condicionales (sentencias if else). Puedes leer más sobre el plegado de código en el sitio de RStudio.
3.9 Directorio de trabajo
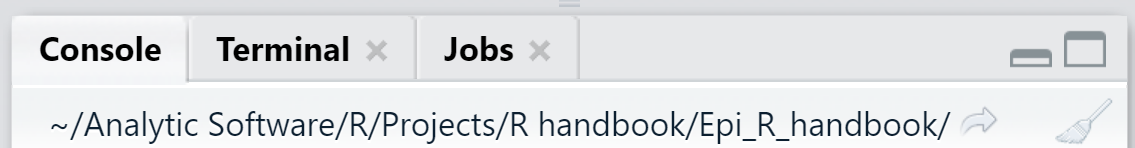
El directorio de trabajo (o Working Directory “WD”) es la ubicación de la carpeta raíz utilizada por R para su trabajo - donde R busca y guarda los archivos por defecto. Por defecto, guardará los nuevos archivos y resultados en esta ubicación, y buscará los archivos de datos para importar aquí también.
El directorio de trabajo aparece en texto gris en la parte superior de la consola de RStudio. También puedes imprimir el directorio de trabajo actual ejecutando getwd() (deja los paréntesis vacíos).
Enfoque recomendado
Consulta la página sobre proyectos de R para obtener detalles sobre nuestro enfoque recomendado para gestionar tu directorio de trabajo. Una forma común, eficiente y sin problemas de gestionar tu directorio de trabajo y las rutas de los archivos es combinar estos 3 elementos en un flujo de trabajo orientado a los proyectos de R:
- Un proyecto R para almacenar todos tus archivos (ver página sobre proyectos R)
- El paquete here para localizar los archivos (véase la página sobre importación y exportación)
- El paquete rio para importar/exportar archivos (véase la página sobre importación y exportación)
Mediante comandos
Hasta hace poco, a muchas personas que aprendían R se les enseñaba a comenzar sus scripts con un comando setwd(). En vez de esto, piensa mejor en un flujo de trabajo orientado al proyecto R y lee las razones para no usar setwd(). En resumen, tu trabajo se convierte en algo específico de tu ordenador, las rutas de archivo utilizadas para importar y exportar archivos se vuelven “frágiles”, y esto dificulta gravemente la colaboración y el uso de tu código en cualquier otro ordenador. Hay alternativas fáciles!
Como se ha indicado anteriormente, aunque no recomendamos este enfoque en la mayoría de las circunstancias, puedes utilizar el comando setwd() con la ruta del archivo de la carpeta deseada entre comillas, por ejemplo:
setwd("C:/Documents/R Files/My analysis")PELIGRO: Establecer un directorio de trabajo con setwd() puede ser “frágil” si la ruta del archivo es específica de un ordenador. En su lugar, utiliza rutas de archivos relativas a un directorio raíz del proyecto R (con el paquete here).
Manualmente
Para establecer el directorio de trabajo manualmente (el equivalente de apuntar y clicar en setwd()), clica en el menú desplegable Session y luego “Set Working Directory” (Establecer el directorio de trabajo) y entonces “Choose Directory” (Elegir el directorio). Esto establecerá el directorio de trabajo para esa sesión específica de R. Nota: si utilizas este enfoque, tendrás que hacerlo manualmente cada vez que abras RStudio.
Con un proyecto
Si se utiliza un proyecto R, el directorio de trabajo será por defecto la carpeta raíz del proyecto R que contiene el archivo “.rproj”. Esto se aplicará si clicas en abrir el proyecto en RStudio (el archivo con extensión “.rproj”).
Directorio en Rmarkdown
En un script de R markdown, el directorio de trabajo por defecto es la carpeta en la que se guarda el archivo Rmarkdown (.Rmd). Si se utiliza un proyecto R y el paquete here, esto no se aplica y el directorio de trabajo será here() como se explica en la página de proyectos R.
Si quieres cambiar el directorio de trabajo de un Rmarkdown independiente (no en un proyecto R), si utilizas setwd() sólo se aplicará a ese trozo de código específico. Para hacer el cambio para todos párrafos, edita el trozo de configuración para añadir el parámetro root.dir =, como se indica a continuación:
knitr::opts_knit$set(root.dir = 'desired/directorypath')Es mucho más fácil usar Rmarkdown dentro de un proyecto R y usar el paquete here.
Escribir la ruta completa
Quizás la fuente más común de frustración para un principiante de R (al menos en una máquina Windows) es escribir una ruta de archivo para importar o exportar datos. Hay una explicación exhaustiva sobre la mejor manera de introducir las rutas de los archivos en la página de importación y exportación, pero aquí hay algunos puntos clave:
Rutas rotas
A continuación se muestra un ejemplo de una ruta de archivo “absoluta” o de “dirección completa”. Es probable que se rompa si se utiliza en otro ordenador. Una excepción es si estás usando una unidad compartida de red.
C:/Nombre de usuario/Documento/Software analítico/R/Proyectos/Análisis2019/datos/Marzo2019.csvDirección de la barra
Si escribes una ruta de archivo, ten en cuenta la dirección de las barras inclinadas (/). Utiliza barras inclinadas (/) para separar los componentes (“data/provincial.csv”). Para los usuarios de Windows, la forma predeterminada en que se muestran las rutas de los archivos es con barras invertidas (\) - por lo que tendrás que cambiar la dirección de cada barra. Si utilizas el paquete here, tal y como se describe en la página de proyectos de R, la dirección de la barra no es un problema.
Rutas de archivo relativas
Generalmente recomendamos proporcionar rutas de archivo “relativas” - es decir, la ruta relativa a la raíz de tu proyecto R. Puedes hacer esto usando el paquete here como se explica en la página de proyectos R. Una ruta de archivo relativa podría ser así:
# Importa linelist csv de las subcarpetas data/linelist/clean/ del proyectoo R
linelist <- import(here("data", "clean", "linelists", "marin_country.csv"))Incluso si se utilizan rutas de archivo relativas dentro de un proyecto R, se pueden utilizar rutas absolutas para importar/exportar datos fuera del proyecto R.
3.10 Objetos
Todo en R es un objeto, y R es un lenguaje “orientado a objetos”. Estas secciones lo explicarán:
- Cómo crear objetos (
<-) - Tipos de objetos (por ejemplo, dataframe -conjunto de datos-, vectores…)
- Cómo acceder a las subpartes de los objetos (por ejemplo, las variables de unos datos)
- Tipos de objetos (por ejemplo, numéricos, lógicos, enteros, dobles, caracteres, factores)
Todo es un objeto
Esta sección está adaptada del proyecto R4Epis. Todo lo que se almacena en R -conjuntos de datos (dataframe), variables, una lista de nombres de pueblos, un número total de población, incluso resultados como gráficos- son objetos a los que se asigna un nombre y a los que se puede hacer referencia en comandos posteriores.
Un objeto existe cuando se le ha asignado un valor (véase la sección de asignación más adelante). Cuando se le asigna un valor, el objeto aparece en el Entorno -Environment- (en el panel superior derecho de RStudio). A partir de ese momento se puede operar con él, manipularlo, cambiarlo y redefinirlo.
Definición de objetos (<-)
Crea objetos asignándoles un valor con el operador <-.
Puedes pensar que el operador de asignación <- significa: “se define como”. Los comandos de asignación suelen seguir un orden estándar:
nombre_objeto <- valor (o proceso/cálculo que produce un valor)
Por ejemplo, es posible que desees registrar la semana de notificación epidemiológica actual como un objeto para referenciarlo posteriormente en el código. En este ejemplo, el objeto current_week se crea cuando se le asigna el valor "2018-W10" (las comillas hacen que sea un valor de tipo carácter). El objeto current_week aparecerá entonces en el panel de Environment de RStudio (arriba a la derecha) y podrá ser referenciado en comandos posteriores.
Ver los comandos de R y su salida en los cuadros de abajo.
current_week <- "2018-W10" # Este comando crea el objeto current_week asignándole un valor
current_week # Este comando muestra en la consola el valor actual del objeto current_week## [1] "2018-W10"NOTA: Observa que el [1] en la consola de resultados simplemente indica que estás viendo el primer elemento de resultados
ATENCIÓN: El valor de un objeto puede sobrescribirse en cualquier momento ejecutando un comando de asignación para redefinir su valor. Por lo tanto, el orden de ejecución de los comandos es muy importante.
El siguiente comando redefinirá el valor de current_week:
current_week <- "2018-W51" # asigna un NUEVO valor al objeto current_week
current_week # muestra en la consola el valor actual del objeto current_week## [1] "2018-W51"Signos de igualdad =
También verás signos de igualdad en el código R:
- Un doble signo de igualdad
==entre dos objetos o valores formula una pregunta lógica: “¿es esto igual a aquello?”. - También verás signos de igualdad dentro de las funciones que se utilizan para especificar los valores de los argumentos de la función (lee sobre ellos en las secciones siguientes), por ejemplo
max(edad, na.rm = TRUE). -
Puedes utilizar un único signo de igualdad
=en lugar de<-para crear y definir objetos, pero se desaconseja hacerlo. Puedes leer por qué se desaconseja aquí.
Conjuntos de datos (datasets)
Los conjuntos de datos también son objetos (normalmente “dataframes”) y se les debe asignar un nombre cuando se importan. En el código siguiente, se crea el objeto linelist y se le asigna el valor de un archivo CSV importado con la función import() del paquete rio.
# se crea linelist y se le asigna los valores del archivo CSV importado
linelist <- import("my_linelist.csv")Puedes obtener más información sobre la importación y la exportación de datos en la sección sobre importación y exportación.
ATENCIÓN: Una nota rápida sobre la denominación de los objetos:
- Los nombres de los objetos no deben contener espacios, debes utilizar el guión bajo (_) o un punto (.) en lugar de un espacio.
- Los nombres de los objetos distinguen entre mayúsculas y minúsculas (lo que significa que Dataset_A es diferente de dataset_A).
- Los nombres de los objetos deben empezar por una letra (no pueden empezar por un número como 1, 2 o 3).
Resultados
Los resutados, como las tablas y los gráficos proporcionan un ejemplo de cómo las salidas pueden guardarse como objetos, o simplemente mostrarse en la consola sin ser guardadas. Una tabla cruzada de género y resultado utilizando la función table() de R base puede mostrarse directamente en la consola de R (sin guardarse).
# solamente se muestra en la consola de R
table(linelist$gender, linelist$outcome)##
## Death Recover
## f 1227 953
## m 1228 950Pero la misma tabla puede guardarse como un objeto con nombre. Y luego, opcionalmente, se puede mostrar o imprimir.
# guarda la tabla
gen_out_table <- table(linelist$gender, linelist$outcome)
# la muestra en la consola (print)
gen_out_table##
## Death Recover
## f 1227 953
## m 1228 950Columnas
Las columnas de unos datos también son objetos y pueden definirse, sobrescribirse y crearse como se describe a continuación en la sección sobre Columnas.
Puedes utilizar el operador de asignación de R base para crear una nueva columna. A continuación, se crea la nueva columna bmi (Índice de masa corporal), y para cada fila el nuevo valor es el resultado de una operación matemática sobre el valor de la fila en las columnas wt_kg y ht_cm.
# crea la columna "bmi" utilizando sintaxis de R base
linelist$bmi <- linelist$wt_kg / (linelist$ht_cm/100)^2Sin embargo, en este manual, hacemos hincapié en un enfoque diferente para definir las columnas, que utiliza la función mutate() del paquete dplyr y la canalización con el operador pipe (%>%). La sintaxis es más fácil de leer y hay otras ventajas que se explican en la página sobre Limpieza de datos y funciones básicas. Puedes leer más sobre la canalización en dicha sección más abajo.
Estructura de los objetos
Los objetos pueden ser un solo dato (por ejemplo, mi_número <- 24), o pueden consistir en datos estructurados.
El gráfico siguiente está tomado de este tutorial de R en línea. Muestra algunas estructuras de datos comunes y sus nombres. No se incluyen en esta imagen los datos espaciales, de los que se habla en la página de fundamentos del SIG.
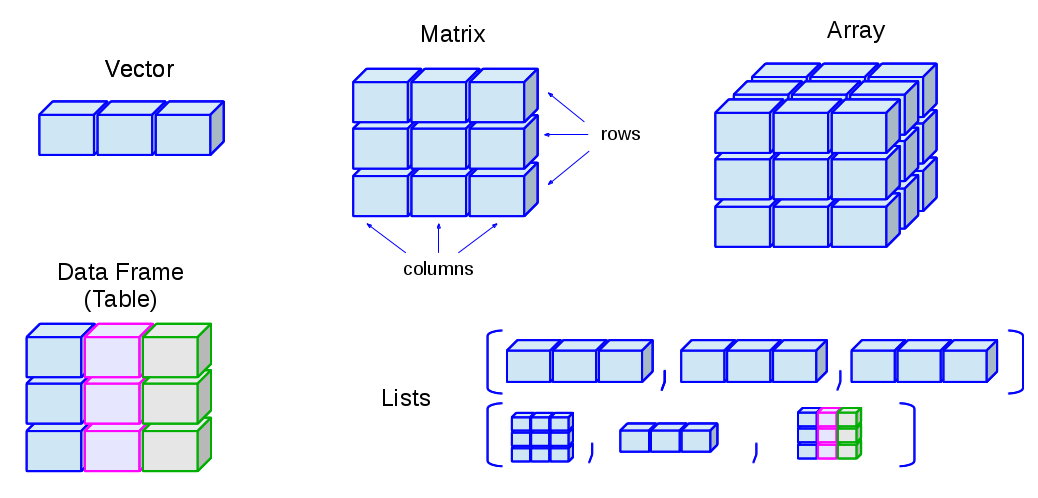
En epidemiología (y en particular en epidemiología de campo), lo habitual es encontrar dataframes (conjuntos de datos) y vectores:
| Estructura común | Explicación | Ejemplo |
|---|---|---|
| Vectores | Un contenedor para una secuencia de objetos singulares, todos del mismo tipo (por ejemplo, numérico, carácter). | Las variables (columnas) en los dataframes son vectores (por ejemplo, la columna de edad en años age_years). |
| Dataframes | Vectores (por ejemplo, columnas) unidos que tienen todos el mismo número de filas. |
linelist es un dataframe. |
Ten en cuenta que para crear un vector que “está solo” (no forma parte de un dataframe) se utiliza la función c() para combinar los diferentes elementos. Por ejemplo, si se crea un vector de colores de la escala de colores del gráfico: vector_de_colores <- c("azul", "rojo2", "naranja", "gris")
Tipos de objeto
Todos los objetos almacenados en R tienen un tipo (class) que indica a R cómo manejar el objeto. Hay muchos tipos de datos, pero los más comunes son:
| Tipo | Explicación | Ejemplos |
|---|---|---|
| Character | Son textos/palabras/frases “entre comillas”. No se pueden realizar operaciones matemáticas con estos objetos. | “Los objetos de carácter están entre comillas” |
| Integer | Números sólo enteros (sin decimales) | -5, 14, o 2000 |
| Numeric | Son números y pueden incluir decimales. Si están entre comillas se considerarán de tipo de caracteres. | 23.1 o 14 |
| Factor | Se trata de vectores que tienen un orden determinado o jerarquía de valores | Una variable de situación económica con valores ordenados |
| Date | Una vez que se le dice a R que ciertos datos son Fechas, estos datos pueden ser manipulados y mostrados de maneras especiales. Para más información, consulta la página sobre el trabajo con fechas. | 2018-04-12 o 15/3/1954 o miércoles 4 de enero de 1980 |
| Logical | Los valores deben ser uno de los dos valores especiales TRUE o FALSE (nótese que no son “TRUE” y “FALSE” entre comillas) | TRUE o FALSE |
| data.frame | Un dataframe es la forma en que R almacena un ** conjunto de datos típico**. Consiste en vectores (columnas) de datos unidos, que tienen todos el mismo número de observaciones (filas). | El set de datos AJS de ejemplo denominado linelist_raw contiene 68 variables con 300 observaciones (filas) cada una. |
| tibble | Los tibbles son una variación de los dataframe, la principal diferencia operativa es que muestran de forma más agradable en la consola (muestran las 10 primeras filas y sólo las columnas que caben en la pantalla) | Cualquier conjunto de datos, lista o matriz puede convertirse en un tibble con as_tibble()
|
| List | Una lista es como un vector, pero contiene otros objetos que pueden ser de otras tipos diferentes | Una lista puede contener un solo número, un dataframe de datos, un vector e incluso otra lista. |
Puedes comprobar el tipo de un objeto escribiendo su nombre en la función class(). Nota: puede hacer referencia a una columna específica dentro de unos datos utilizando la notación $ para separar el nombre de los datos y el nombre de la columna.
class(linelist) # debe ser de tipo dataframe o tibble## [1] "data.frame"
class(linelist$age) # debe ser de tipo numérico## [1] "numeric"
class(linelist$gender) # debe ser de tipo carácter## [1] "character"A veces, una columna puede ser convertida automáticamente por R en un tipo diferente. ¡Cuidado con esto! Por ejemplo, si tiene un vector o columna de números, pero se inserta un valor de carácter… toda la columna cambiará al tipo carácter.
num_vector <- c(1,2,3,4,5) # define vector como todos números
class(num_vector) # el vector es de tipo numérico## [1] "numeric"
num_vector[3] <- "three" # convierte el tercer elemento en de tipo carácter
class(num_vector) # el vector es ahora de tipo carácter class## [1] "character"Un ejemplo común de esto es cuando se manipula unos datos para imprimir una tabla - si se hace una fila de totales y se intenta pegar porcentajes en la misma celda como números (por ejemplo, 23 (40%)), toda la columna numérica de arriba se convertirá en carácter y ya no se podrá utilizar para cálculos matemáticos. A veces, tendrás que convertir objetos o columnas a otro tipo.
| Función | Acción |
|---|---|
| as.character() | Convierte al tipo de carácter |
| as.numeric() | Convierte al tipo numérico |
| as.integer() | Convierte al tipo entero |
| as.date() | Convierte al tipo de fecha - Nota: ver la sección de fechas para más detalles |
| factor() | Convierte en factor - Nota: la redefinición del orden de los niveles de valor requiere argumentos adicionales |
Asimismo, existen funciones de R base para comprobar si un objeto ES de un tipo específico, como is.numeric(), is.character(), is.double(), is.factor(), is.integer()
Aquí hay más material en línea sobre tipos y estructuras de datos en R.
Columnas/Variables ($)
Una columna en un dataframe es técnicamente un “vector” (véase la tabla anterior) - una serie de valores que deben ser todos del mismo tipo (ya sea carácter, numérico, lógico, etc).
Un vector puede existir independientemente de un dataframe, por ejemplo, un vector de nombres de columnas que se desea incluir como variables explicativas en un modelo. Para crear un vector “independiente”, utiliza la función c() como se indica a continuación:
# define el vector con valores de caracteres
explanatory_vars <- c("gender", "fever", "chills", "cough", "aches", "vomit")
# muestra los valores de este vector
explanatory_vars## [1] "gender" "fever" "chills" "cough" "aches" "vomit"Las columnas de un dataframe también son vectores y pueden ser llamadas, referenciadas, extraídas o creadas utilizando el símbolo $. El símbolo $ conecta el nombre de la columna con el nombre del dataframe. En este manual, tratamos de utilizar la palabra “columna” en lugar de “variable”.
# Recuperar la longitud del vector age_years
length(linelist$age) # (age es una columna del dataframe linelist)Al escribir el nombre del dataframe seguido de $ también verá un menú desplegable de todas las columnas del dataframe. Puedes desplazarte por ellas con la tecla de flecha, seleccionar una con la tecla Intro y evitar errores ortográficos.

CONSEJO AVANZADO: Algunos objetos más complejos (por ejemplo, una lista, o un objeto epicontacts) pueden tener múltiples niveles a los que se puede acceder a través de múltiples signos de dólar. Por ejemplo epicontacts$linelist$date_onset
Acceso/índice con corchetes ([ ])
Es posible que tengas que mirar sólo partes de los objetos, lo que también se llama “indexación”, que a menudo se hace utilizando los corchetes [ ]. El uso de $ en un dataframe para acceder a una columna también es un tipo de indexación.
my_vector <- c("a", "b", "c", "d", "e", "f") # definir el vector
my_vector[5] # mostrar el 5º elemento## [1] "e"Los corchetes también sirven para devolver partes específicas de un resultado, como la salida de una función summary():
# resumen completo
summary(linelist$age)## Min. 1st Qu. Median Mean 3rd Qu. Max. NA's
## 0.00 6.00 13.00 16.07 23.00 84.00 86
# Sólo el segundo elemento del resumen, nombre (usando solo conchetes simples)
summary(linelist$age)[2]## 1st Qu.
## 6
# Sólo el segundo elemento, sin nombre (usando dobles corchetes)
summary(linelist$age)[[2]]## [1] 6
# Extrae un elemento por nombre, sin mostrar el nombre
summary(linelist$age)[["Median"]]## [1] 13Los corchetes también funcionan en los dataframes para ver filas y columnas específicas. Puedes hacerlo utilizando la sintaxis dataframe[filas, columnas]:
# Ver una fila específica (2) del dataset, con todas las columnas (¡no olvides la coma!)
linelist[2,]
# Ver todas las filas, pero solo una columna
linelist[, "date_onset"]
# Ver los valores de la fila 2 y las columnas 5 hasta la 10
linelist[2, 5:10]
# Ver los valores desde la fila 2 y las columnas 5 hasta la 10 y 18
linelist[2, c(5:10, 18)]
# Ver las filas 2 a la 20 y sólo unas columnas especificadas
linelist[2:20, c("date_onset", "outcome", "age")]
# Ver filas y columnas basado en ciertos criterios
# *** ¡Observa que el nombre del dataframe también debe estar entre los criterios!
linelist[linelist$age > 25 , c("date_onset", "outcome", "age")]
# Usa View() para ver el resultado en el Visor de Rstudio (más fácil de leer)
# *** Fíjate que la "V" de la función View() está en mayúsculas
View(linelist[2:20, "date_onset"])
# Guarda como objeto nuevo
new_table <- linelist[2:20, c("date_onset")] Ten en cuenta que también puedes lograr la indexación de filas/columnas anterior en dataframes y tibbles utilizando sintaxis de dplyr (funciones filter() para filas, y select() para columnas). Puedes leer más sobre estas funciones básicas en la página de Limpieza de datos y funciones básicas.
Para filtrar en base al “número de fila”, puedes utilizar la función row_number() de dplyr (con paréntesis vacíos) como parte de una sentencia lógica de filtrado. A menudo se utiliza el operador %in% y un rango de números como parte de esa sentencia lógica, como se muestra a continuación. Para ver las primeras N filas, también puede utilizar la función especial head() de dplyr.
# Ver las primeras 100 filas
linelist %>% head(100)
# Mostrar sólo la fila 5
linelist %>% filter(row_number() == 5)
# Ver las filas 2 hasta la 20, y tres columnas específicas (No sonnecesarias las comillas pasa los nombres de columna)
linelist %>% filter(row_number() %in% 2:20) %>% select(date_onset, outcome, age)Cuando se indexa un objeto del tipo list, los corchetes simples siempre devuelven con el tipo list, incluso si sólo se devuelve un único objeto. Los corchetes dobles, sin embargo, pueden utilizarse para acceder a un solo elemento y devolver un tipo diferente al de la lista. Los corchetes también pueden escribirse uno tras otro, como se demuestra a continuación.
Esta explicación visual de la indexación de listas, con pimenteros, es divertida y útil.
# define demo list
my_list <- list(
# El primer elemento en la lista es un vector de carácter
hospitals = c("Central", "Empire", "Santa Anna"),
# El segundo elemento en la lista es un data frame de direcciones
addresses = data.frame(
street = c("145 Medical Way", "1048 Brown Ave", "999 El Camino"),
city = c("Andover", "Hamilton", "El Paso")
)
)Este es el aspecto de la lista cuando se muestra en la consola. Mira cómo hay dos elementos con nombre:
-
hospital, un vector de caracteres -
addresses, un data frame de direcciones
my_list## $hospitals
## [1] "Central" "Empire" "Santa Anna"
##
## $addresses
## street city
## 1 145 Medical Way Andover
## 2 1048 Brown Ave Hamilton
## 3 999 El Camino El PasoAhora extraeremos información, utilizando varios métodos:
my_list[1] # este devuelve el elemento de tipo "list" - también se muestra el nombre del elemento## $hospitals
## [1] "Central" "Empire" "Santa Anna"
my_list[[1]] # este devuelve sólo el vector de carácter (sin nombrarlo) ## [1] "Central" "Empire" "Santa Anna"
my_list[["hospitals"]] # también puedes indexar por nombre del elemento list## [1] "Central" "Empire" "Santa Anna"
my_list[[1]][3] # este devuelve el tercer elemento del vector de carácter "hospitals"## [1] "Santa Anna"
my_list[[2]][1] # este devuelve la primera columna ("street") del data frame address## street
## 1 145 Medical Way
## 2 1048 Brown Ave
## 3 999 El Camino
3.11 Piping (%>%)
Dos aproximaciones para trabajar con objetos:
-
Pipes/tidyverse - pipes envía un objeto de una función a otra función - énfasis en la acción, no en el objeto
- Definir objetos intermedios - se re-define un objeto una y otra vez - el énfasis está en el objeto
Pipes (%>%)
Hay dos enfoques generales para trabajar con objetos:
Pipes/tidyverse - los pipes envían un objeto de función a función - el énfasis está en la acción, no en el objeto
Definir objetos intermedios - un objeto se redefine una y otra vez - el énfasis está en el objeto
Pipes
Explicado de forma sencilla, el operador pipe (%>%) pasa un resultado intermedio de una función a la siguiente. Puedes pensar en él como si dijera “entonces”. Muchas funciones pueden enlazarse con %>%.
- Los pipes hacen hincapié en una secuencia de acciones, no en el objeto sobre el que se realizan las acciones
- Los pipes son mejores cuando hay que realizar una secuencia de acciones en un objeto
- Los pipes provienen del paquete magrittr, que se incluye automáticamente en los paquetes dplyr y tidyverse
- Los pipes pueden hacer que el código sea más limpio y fácil de leer, más intuitivo
Más información sobre este enfoque en la guía de estilo de tidyverse
He aquí un ejemplo para comparar, utilizando funciones ficticias para “hornear un pastel”. Primero, el método pipe:
# Un ejemplo falso de cómo hornear un pastel utilizando sintaxis con pipes
cake <- flour %>% # para definir pastel, empezar con harina, y luego...
add(eggs) %>% # añadir huevos
add(oil) %>% # añadir aceite
add(water) %>% # añadir agua
mix_together( # mezclarlos juntos
utensil = spoon,
minutes = 2) %>%
bake(degrees = 350, # hornear
system = "fahrenheit",
minutes = 35) %>%
let_cool() # dejar que se enfríeAquí hay otro enlace que describe la utilidad de Los pipes.
El uso de pipes no es una función base. Para usarlos, debe estar instalado y cargado el paquete magrittr (esto se hace normalmente cargando el paquete tidyverse o dplyr que lo incluyen). Puedes leer más sobre pipes en la documentación de magrittr.
Ten en cuenta que, al igual que otros comandos de R, Los pipes se pueden utilizar para mostrar sólo el resultado, o para guardar/re-guardar un objeto, dependiendo de si el operador de asignación <- está involucrado. Mira ambas cosas a continuación:
# Crear o sobreescribir objetos, definiéndolo como como recuentos agregados por categoría de edad (sin mostrarlo)
linelist_summary <- linelist %>%
count(age_cat)## age_cat n
## 1 0-4 1095
## 2 5-9 1095
## 3 10-14 941
## 4 15-19 743
## 5 20-29 1073
## 6 30-49 754
## 7 50-69 95
## 8 70+ 6
## 9 <NA> 86%<>%
Se trata de un “pipe de asignación” del paquete magrittr, que lleva un objeto hacia adelante y también redefine el objeto. Debe ser el primer operador pipe en la cadena. Es una forma abreviada. Los dos comandos siguientes son equivalentes:
Definir objetos intermedios
Este enfoque para cambiar objetos/dataframes puede ser mejor si:
- Necesitas manipular múltiples objetos
- Hay pasos intermedios que son significativos y merecen nombres de objetos separados
Riesgos:
- Crear nuevos objetos para cada paso significa crear muchos objetos. Si usas el equivocado, ¡puedes no darte cuenta!
- Nombrar todos los objetos puede ser confuso
- Los errores pueden no ser fácilmente detectables
O bien nombrar cada objeto intermedio, o sobrescribir el original, o combinar todas las funciones juntas. Todo ello conlleva sus propios riesgos.
A continuación se muestra el mismo ejemplo de “pastel” falso que el anterior, pero utilizando este estilo:
# Un ejemplo falso de cómo hornear un pastel definiendo objetos intermedios
batter_1 <- left_join(flour, eggs)
batter_2 <- left_join(batter_1, oil)
batter_3 <- left_join(batter_2, water)
batter_4 <- mix_together(object = batter_3, utensil = spoon, minutes = 2)
cake <- bake(batter_4, degrees = 350, system = "fahrenheit", minutes = 35)
cake <- let_cool(cake)Combinar todas las funciones juntas - esto es difícil de leer:
# un ejemplo de combinación/anidación de múltiples funciones - difícil de leer
cake <- let_cool(bake(mix_together(batter_3, utensil = spoon, minutes = 2), degrees = 350, system = "fahrenheit", minutes = 35))3.12 Operadores y funciones clave
Esta sección detalla los operadores en R, como por ejemplo
- Operadores de definición
- Operadores relacionales (menor que, igual a ..)
- Operadores lógicos (and, or…)
- Tratamiento de los valores faltantes
- Operadores y funciones matemáticas (+/-, >, sum(), median(), …)
- El operador %in%
Operadores de asignación
<-
El operador de asignación básico en R es <-. Como en nombre_objeto <- valor.
Este operador de asignación también se puede escribir como =. Aconsejamos de forma general el uso de <- .
También aconsejamos rodear tales operadores con espacios, para mejorar la legibilidad.
<<-
Si se escriben funciones, o se utiliza R de forma interactiva con scripts de origen, entonces puede ser necesario utilizar este operador de asignación <<- (de R base). Este operador se utiliza para definir un objeto en un entorno R superior “padre”. Mira esta referencia online.
%<>%
Se trata de un “pipe de asignación” del paquete magrittr, que canaliza un objeto hacia adelante y también redefine el objeto. Debe ser el primer operador de pipe en la cadena. Es una forma abreviada, como se muestra a continuación en dos ejemplos equivalentes:
Lo anterior equivale a lo siguiente:
linelist %<>% mutate(age_months = age_years * 12)%<+%
Se utiliza para añadir datos a los árboles filogenéticos con el paquete ggtree. Consulta la página sobre árboles filogenéticos o este libro de recursos en línea.
Operadores relacionales y lógicos
Los operadores relacionales comparan valores y se utilizan a menudo al definir nuevas variables y subconjuntos de datos. Estos son los operadores relacionales más comunes en R:
| Significado | Operador | Ejemplo | Ejemplo de resultado |
|---|---|---|---|
| Igual a | == |
"A" == "a" |
FALSE (R distingue entre mayúsculas y minúsculas) Ten en cuenta que == (doble igual) es diferente de = (simple igual), que actúa como el operador de asignación <-
|
| No es igual a | != |
2 != 0 |
TRUE |
| Mayor que | > |
4 > 2 |
TRUE |
| Menor que | < |
4 < 2 |
FALSE |
| Mayor que o igual a | >= |
6 >= 4 |
TRUE |
| Menor que o igual a | <= |
6 <= 4 |
FALSE |
| Falta el valor | is.na() |
is.na(7) |
FALSE (véase la página sobre Valores faltantes) |
| No falta el valor | !is.na() |
!is.na(7) |
TRUE |
Los operadores lógicos o booleanos, como AND y OR, suelen utilizarse para conectar operadores relacionales y crear criterios más complicados. Las sentencias complejas pueden requerir paréntesis () para la agrupación y el orden de aplicación.
| gnificado |Operado | r |
| ———-|——- | —————– |
D |&
|
|
|| (ba |
rra vertical) |
réntesis |( ) S |
e utiliza para agrupar criterios y aclarar el orden de las operaciones |
Por ejemplo, a continuación, tenemos un listado con dos variables que queremos utilizar para crear nuestra definición de caso, hep_e_rdt, un resultado de la prueba y other_cases_in_hh, que nos dirá si hay otros casos en el hogar. El comando siguiente utiliza la función case_when() para crear la nueva variable case_def tal que:
linelist_cleaned <- linelist %>%
mutate(case_def = case_when(
is.na(rdt_result) & is.na(other_case_in_home) ~ NA_character_,
rdt_result == "Positive" ~ "Confirmed",
rdt_result != "Positive" & other_cases_in_home == "Yes" ~ "Probable",
TRUE ~ "Suspected"
))| Criterios del ejemplo anterior | Valor resultante en la nueva variable “case_def” |
|---|---|
Si falta el valor de las variables rdt_result y other_cases_in_home
|
NA (valor faltante) |
Si el valor de rdt_result es “Positivo” |
“Confirmado” |
Si el valor de rdt_result NO es “Positivo” Y el valor de other_cases_in_home es “Si” |
“Probable” |
| Si no se cumple alguno de los criterios anteriores | “Sospechoso” |
Ten en cuenta que R distingue entre mayúsculas y minúsculas, por lo que “Positivo” es diferente de “positivo”…
Valores faltantes
En R, los valores faltantes (missing value) se representan con el valor especial NA (una palabra “reservada” para ello) (letras mayúsculas N y A - sin comillas). Si importas datos que registran los valores faltantes de otra manera (por ejemplo, 99, “Nulo”, o .), es posible que quieras re-codificar esos valores a NA. En la página de importación y exportación se explica cómo hacerlo.
Para comprobar si un valor es NA, utiliza la función especial is.na(), que devuelve TRUE o FALSE.
rdt_result <- c("Positive", "Suspected", "Positive", NA) # dos casos positivos, uno sospechoso y uno desconocido
is.na(rdt_result) # Comprueba si el valor de rdt_result es NA## [1] FALSE FALSE FALSE TRUEPuedes leer más sobre los valores faltantes, infinitos, NULL e imposibles en la página sobre Valores faltantes]. Aprende a convertir los valores faltantes al importar datos en la página sobre Importación y exportación.
Matemáticos y estadísticos
Todos los operadores y funciones de esta página están disponibles automáticamente en R base.
Operadores matemáticos
Se utilizan a menudo para realizar sumas, divisiones, crear nuevas columnas, etc. A continuación se muestran los operadores matemáticos más comunes en R. No es importante poner espacios alrededor de los operadores.
| Propósito | Ejemplo en R |
|---|---|
| suman | 2 + 3 |
| resta | 2 - 3 |
| multiplicación | 2 * 3 |
| división | 30 / 5 |
| exponente | 2^3 |
| orden de operaciones | ( ) |
Funciones matemáticas
| Propósito | Función** |
|---|---|
| redondeo | round(x, digits = n) |
| redondeo | janitor::round_half_up(x, digits = n) |
| redondeo arriba | ceiling(x) |
| redondeo abajo | floor(x) |
| valor absoluto | abs(x) |
| raiz cuadrada | sqrt(x) |
| exponente | exponent(x) |
| logaritmo natural | log(x) |
| log base 10 | log10(x) |
| log base 2 | log2(x) |
Nota: para round() los dígits = especifican el número de decimales. Utiliza signif() para redondear a un número de cifras significativas.
Notación científica
La probabilidad de que se utilice la notación científica depende del valor de la opción scipen.
De la documentación de ?options: scipen es una penalización que se aplica cuando se decide obtener valores numéricos en notación fija o exponencial. Los valores positivos se inclinan hacia la notación fija y los negativos hacia la notación científica: se preferirá la notación fija a menos que tenga más dígitos que ‘scipen’.
Si se establece en un número bajo (por ejemplo, 0) estará “activada” siempre. Para “desactivar” la notación científica en tu sesión de R, configúrela con un número muy alto, por ejemplo:
# desactiva la notación científica
options(scipen=999)Redondeo
PELIGRO: round() utiliza el “redondeo del banquero” que redondea hacia arriba desde un 0,5 sólo si el número superior es par. Utiliza round_half_up() de janitor para redondear consistentemente las mitades hacia arriba al número entero más cercano. Aquí tienes esta explicación
## [1] 2 4
janitor::round_half_up(c(2.5, 3.5))## [1] 3 4Funciones estadísticas
PRECAUCIÓN: Las funciones que aparecen a continuación incluyen por defecto los valores faltantes en los cálculos. Los valores faltantes darán como resultado una salida de NA, a menos que se especifique el argumento na.rm = TRUE. Esto se puede escribir de forma abreviada como na.rm = T.
| Objetivo | Función** |
|---|---|
| media (promedio) | mean(x, na.rm=T) |
| mediana | median(x, na.rm=T) |
| desviación estándar | sd(x, na.rm=T) |
| cuantiles* | quantile(x, probs) |
| suma | sum(x, na.rm=T) |
| valor mínimo | min(x, na.rm=T) |
| valor máximo | max(x, na.rm=T) |
| rango de valores numéricos | range(x, na.rm=T) |
| resumen** | summary(x) |
Notas:
*quantile():xes el vector numérico a examinar, yprobs =es un vector numérico con probabilidades entre 0 y 1.0, por ejemploc(0.5, 0.8, 0.85)**summary(): da un resumen estadístico sobre un vector numérico incluyendo la media, mediana y percentiles más comunes
PELIGRO: Si proporciona un vector de números a una de las funciones anteriores, asegúrese de envolver los números dentro de c().
# Si se suministran números sin procesar a una función, envolver con c()
mean(1, 6, 12, 10, 5, 0) # !!! INCORRECTO !!! ## [1] 1## [1] 5.666667Otras funciones útiles
| Objetivo | Función | Ejemplo** |
|---|---|---|
| crear una secuencia | seq(de, a, por) | seq(1, 10, 2) |
| repetir x, n veces | rep(x, nveces) |
rep(1:3, 2) or rep(c("a", "b", "c"), 3)
|
| subdividir un vector numérico | cut(x, n) | cut(linelist$age, 5) |
| tomar una muestra aleatoria | sample(x, tamaño) | sample(linelist$id, size = 5, replace = TRUE) |
%in%
Un operador muy útil para comparar valores, y para evaluar rápidamente si un valor está dentro de un vector o dataframe.
my_vector <- c("a", "b", "c", "d")
"a" %in% my_vector## [1] TRUE
"h" %in% my_vector## [1] FALSEPara preguntar si un valor no está %in% en un vector, pon un signo de exclamación (!) delante de la declaración lógica:
# para negar, pon una exclamación delante
!"a" %in% my_vector## [1] FALSE
!"h" %in% my_vector## [1] TRUE%in% es muy útil cuando se utiliza la función case_when() de dplyr. Se puede definir un vector previamente y referenciarlo después. Por ejemplo:
affirmative <- c("1", "Yes", "YES", "yes", "y", "Y", "oui", "Oui", "Si")
linelist <- linelist %>%
mutate(child_hospitaled = case_when(
hospitalized %in% affirmative & age < 18 ~ "Hospitalized Child",
TRUE ~ "Not"))Nota: Si quieres detectar una cadena parcial, quizás usando str_detect() de stringr, no aceptará un vector de caracteres como c("1", "Yes", "yes", "y"). En su lugar, se le debe dar una expresión regular - una cadena condensada con barras OR, como “1|Yes|yes|y”. Por ejemplo, str_detect(hospitalized, "1|Yes|yes|y"). Consulta la página sobre Caracteres y cadenas para obtener más información.
Puedes convertir un vector de caracteres en una expresión regular con este comando:
affirmative <- c("1", "Yes", "YES", "yes", "y", "Y", "oui", "Oui", "Si")
affirmative## [1] "1" "Yes" "YES" "yes" "y" "Y" "oui" "Oui" "Si"
# condense to
affirmative_str_search <- paste0(affirmative, collapse = "|") # opción con R base
affirmative_str_search <- str_c(affirmative, collapse = "|") # opción con el paquete stringr
affirmative_str_search## [1] "1|Yes|YES|yes|y|Y|oui|Oui|Si"3.13 Errores & avisos
En esta sección se explica:
- La diferencia entre errores y avisos (warnings)
- Consejos generales de sintaxis para escribir código R
- Código de asistencia
En las página de Errores comunes y de Obtener ayuda se pueden encontrar los errores y avisos más comunes, así como consejos para la resolución de problemas.
Error vs aviso
Cuando se ejecuta un comando, la Consola de R puede mostrarte mensajes de aviso o error en texto rojo.
Una aviso significa que R ha completado su comando, pero ha tenido que dar pasos adicionales o ha producido una salida inusual de la que deberías estar al tanto.
Un error significa que R no pudo completar su comando.
Busca pistas:
El mensaje de error/advertencia suele incluir un número de línea donde está el problema.
Si un objeto “es desconocido” o “no se encuentra”, quizás lo hayas escrito mal, hayas olvidado llamar a un paquete con library(), o hayas olvidado volver a ejecutar tu script después de hacer cambios.
Si todo lo demás falla, copia el mensaje de error en Google junto con algunos términos clave; lo más probable es que a alguien le haya pasado lo mismo y ¡ya haya resuelto el problema!.
Consejos generales de sintaxis
Algunas cosas que hay que recordar al escribir comandos en R, para evitar errores y advertencias:
- Cierra siempre los paréntesis - consejo: cuenta el número de paréntesis de apertura “(” y de cierre “)” de cada trozo de código
- Evita los espacios en los nombres de columnas y objetos. Utiliza barras bajas ( _ ) o puntos ( . ) en su lugar
- No olvides separar los argumentos de una función con comas
- R distingue entre mayúsculas y minúsculas, lo que significa que
Variable_Aes diferente devariable_A