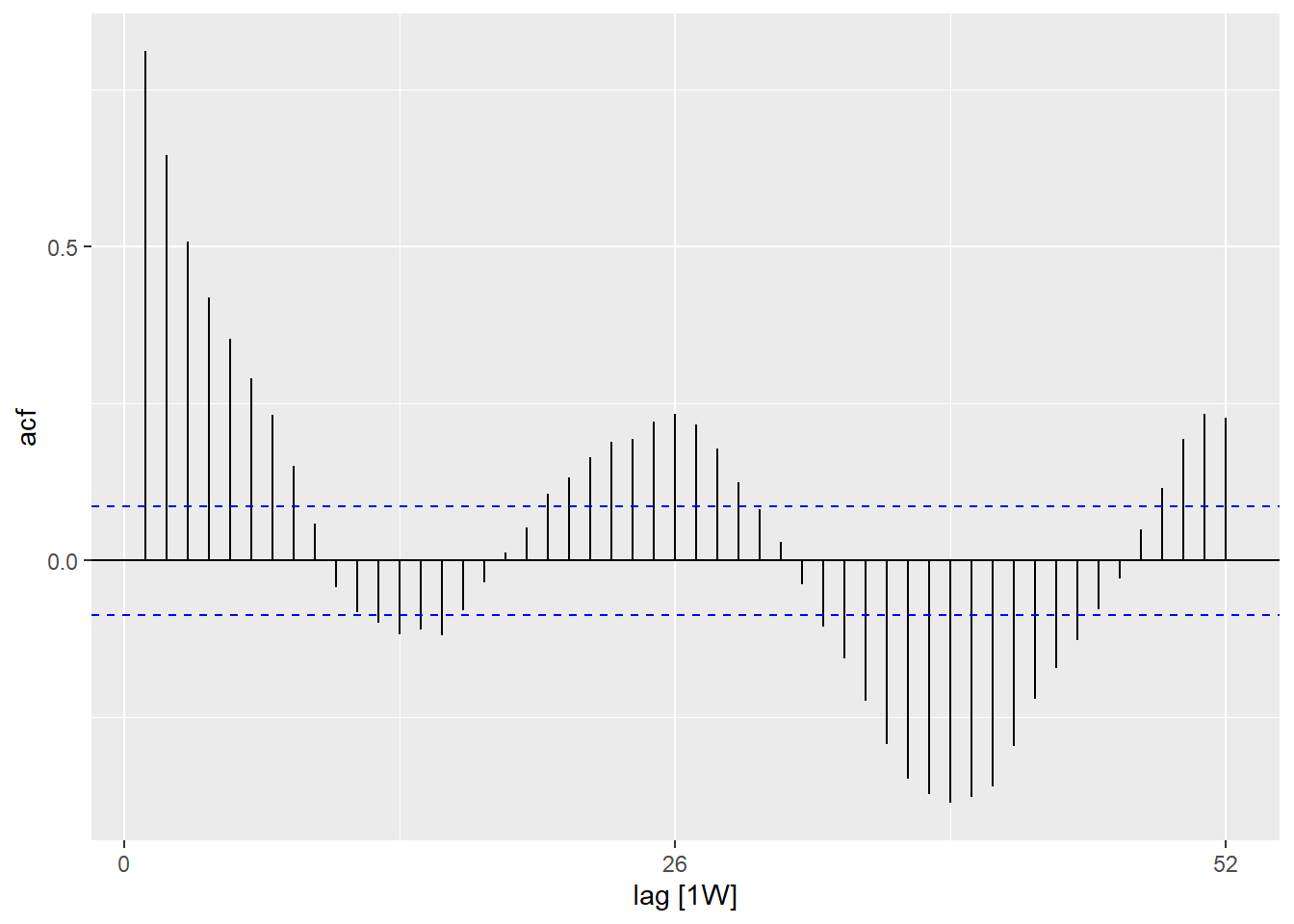pacman::p_load(rio, # Importar arquivos
here, # Localizar arquivos
tidyverse, # gerenciamento de dados + gráficos ggplot2
tsibble, # trabalhe com dados de séries temporais
slider, # para calcular médias móveis
imputeTS, # para preencher campos em branco
feasts, # para decomposição de séries temporais e autocorrelações
forecast, # ajusta senos e cosenos nos dados (nota: precisa ser carregado após pacote feasts)
trending, # ajusta e visualizada modelos
tmaptools, # para obter coordenadas geográficas (long/lat) baseado no nome dos locais
ecmwfr, # para interagir com o satélite copernicus CDS API
stars, # para ler arquivos em .nc (dados climáticos)
units, # para definir unidades de medida (dados climáticos)
yardstick, # para avaliar a acurácia do modelo
surveillance, # para detecção de aberrações
ncmeta # ler arquivos .nc
)23 Séries temporais e detecção de surtos
23.1 Visão geral do tópico
Esta página demonstra o uso de diferentes pacotes para analisar séries temporais. São utilizados, principalmente, os pacotes da família tidyverts , mas também o pacote RECON trending para aprimorar modelos mais apropriados para epidemiologia de doenças infecciosas.
Observe que, no exemplo abaixo, nós utilizamos um banco de dados do pacote surveillance sobre a Campylobacter na Alemanha (veja o capítulo sobre dados, deste manual para mais detalhes). Entretanto, se você quiser executar o mesmo código em um banco de dados com múltiplos países, ou outros estratos, então aqui está um exemplo de código para isto no repositório do r4epis no github.
Tópicos abordados incluem:
- Dados com séries temporais
- Análise descritiva
- Ajustando regressões
- Relação de duas séries temporais
- Detecção de surtos
- Séries temporais interrompidas
23.2 Preparação
Carregue os pacotes R
O código abaixo realiza o carregamento dos pacotes necessários para a análise dos dados. Neste manual, enfatizamos o uso da função p_load(), do pacman, que instala os pacotes, caso não estejam instalados, e os carrega no R para utilização. Também é possível carregar pacotes instalados utilizando a função library(), do R base. Para mais informações sobre os pacotes do R, veja a página Introdução ao R.
Carregue os dados no R
Você pode baixar todos os dados utilizados neste manual através das instruções na página de download do manual e dos dados.
Os dados utilizados nessa seção para fins de exemplificação são as contagens semanais de casos de campylobacter notificados na Alemanha entre 2001 e 2011. Você pode clicar aqui para baixar este arquivo de dados (.xlsx).
Este banco de dados é uma versão reduzida do banco de dados disponível no pacote surveillance. (para mais detalhes, carregue o pacote surveillance e veja ?campyDE)
Importe estes dados com a função import() do pacote rio (ele manipula muitos tipos de arquivos, como .xlsx, .csv, .rds - veja a página sobre Importar e exportar para mais detalhes).
# importe as contagens para o R
counts <- rio::import("campylobacter_germany.xlsx")As primeiras 10 linhas da contagem são mostradas abaixo.
Limpe os dados
O código abaixo garante que a coluna de datas esteja no formato apropriado. Nesta página, iremos utilizar o pacote tsibble com funções yearweek que serão usadas para criar uma variável com um calendário de semanas. Existem outras maneiras de fazer isso (veja a página trabalhando com datas para mais detalhes), entretanto, para séries temporais é melhor nos mantermos dentro de um único tipo de abordagem (tsibble).
## garanta que a coluna com datas estejam no formato apropriado
counts$date <- as.Date(counts$date)
## crie uma variável com semanas do calendário
## ajuste definições ISO de semanas iniciando na segunda
counts <- counts %>%
mutate(epiweek = yearweek(date, week_start = 1))Baixe (download) os dados climáticos
Na seção sobre a relação de duas séries temporais nesta página, nós iremos comparar contagens de casos de campylobacter com dados climáticos.
Dados climáticos de qualquer lugar do mundo podem ser baixados do Satélite Copernicus da União Européia Estas não são medições exatas, mas baseadas em um modelo (similar a interpolação). Entretanto, o benefício é a cobertura global a cada hora, bem como previsões.
Você pode baixar cada um desses arquivos de dados climáticos da página Download do manual e dos dados.
Para fins de demonstração, nós iremos mostrar códigos em R que utilizam o pacote ecmwfr para baixar esses dados do repositório do Copernicus. Você precisa criar uma conta gratuita para que isto funcione. O website do pacote tem um passo a passo útil de como fazer isto. Quando tiver as chaves API apropriadas, o código abaixo é um exemplo de como proceder. Você precisa substituir o X abaixo com o ID da sua conta. Você só pode baixar um ano de dados por vez. Do contrário, o tempo do servidor expira.
Se você não tiver certeza das coordenadas de uma localização que quer baixar os dados, é possível utilizar o pacote tmaptools para baixar as coordenadas de um mapa open street. Outra alternativa é o pacote photon, entretanto ele ainda não foi liberado no CRAN; algo interessante do
photon é que ele fornece mais dados contextuais caso existam diferentes resultados para o que se busca.
## recupere as coordenadas de localização
coords <- geocode_OSM("Germany", geometry = "point")
## baixe as long/lats juntas no formado para busca no ERA-5 (caixa de delimitação)
## (como só quer um único ponto, pode repetir as coordenadas)
request_coords <- str_glue_data(coords$coords, "{y}/{x}/{y}/{x}")
## Baixando dados modelados do satélite copernicus (reanálise ERA-5)
## https://cds.climate.copernicus.eu/cdsapp#!/software/app-era5-explorer?tab=app
## https://github.com/bluegreen-labs/ecmwfr
## configure uma chave para os dados climáticos
wf_set_key(user = "XXXXX",
key = "XXXXXXXXX-XXXX-XXXX-XXXX-XXXXXXXXXXX",
service = "cds")
## para cada ano de interesse, execute o código (do contrário o tempo do servidor expira)
for (i in 2002:2011) {
## monte uma requisição (*Query*)
## veja aqui para como fazer isso: https://bluegreen-labs.github.io/ecmwfr/articles/cds_vignette.html#the-request-syntax
## altere o pedido para uma lista utilizando o botão *addin* acima (python para lista)
## O alvo é o nome do arquivo de saída!!
request <- request <- list(
product_type = "reanalysis",
format = "netcdf",
variable = c("2m_temperature", "total_precipitation"),
year = c(i),
month = c("01", "02", "03", "04", "05", "06", "07", "08", "09", "10", "11", "12"),
day = c("01", "02", "03", "04", "05", "06", "07", "08", "09", "10", "11", "12",
"13", "14", "15", "16", "17", "18", "19", "20", "21", "22", "23", "24",
"25", "26", "27", "28", "29", "30", "31"),
time = c("00:00", "01:00", "02:00", "03:00", "04:00", "05:00", "06:00", "07:00",
"08:00", "09:00", "10:00", "11:00", "12:00", "13:00", "14:00", "15:00",
"16:00", "17:00", "18:00", "19:00", "20:00", "21:00", "22:00", "23:00"),
area = request_coords,
dataset_short_name = "reanalysis-era5-single-levels",
target = paste0("germany_weather", i, ".nc")
)
## baixe o aquivo e armazene-o no diretório de trabalho atual
file <- wf_request(user = "XXXXX", # ID do usuário (para autenticação)
request = request, # o pedido
transfer = TRUE, # baixe o arquivo
path = here::here("data", "weather")) ## diretório para salvar os dados
}Carregue os dados climáticos
Se você baixou os dados climáticos seguindo o nosso manual, ou se utilizou o código acima, agora você deve ter 10 anos de arquivos com dados climáticos “.nc” armazenados na mesma pasta do seu computador.
Utilize o código abaixo para importar estes arquivos no R com o pacote stars.
## defina o diretório para a pasta de clima
file_paths <- list.files(
here::here("data", "time_series", "weather"), # substitua com o seu próprio endereço de arquivos
full.names = TRUE)
## mantenha apenas os arquivos que tenham o nome de interesse atual
file_paths <- file_paths[str_detect(file_paths, "germany")]
## leia todos os arquivos como um objeto stars
data <- stars::read_stars(file_paths)t2m, tp,
t2m, tp,
t2m, tp,
t2m, tp,
t2m, tp,
t2m, tp,
t2m, tp,
t2m, tp,
t2m, tp,
t2m, tp, Uma vez que estes arquivos foram importados como o objeto data, nós iremos convertê-los para um quadro de dados (data frame).
## mude para um quadro de dados (*data frame*)
temp_data <- as_tibble(data) %>%
## adicione variáveis e corrija unidades
mutate(
## crie uma variável com um calendário semanal
epiweek = tsibble::yearweek(time),
## crie uma variável de data (início do calendário semanal)
date = as.Date(epiweek),
## mude a temperatura de kelvin para celsius
t2m = set_units(t2m, celsius),
## altere a precipitação de metros para milímetros
tp = set_units(tp, mm)) %>%
## agrupe por semana (mantenha a semana também)
group_by(epiweek, date) %>%
## obtenha a média por semana
summarise(t2m = as.numeric(mean(t2m)),
tp = as.numeric(mean(tp)))`summarise()` has grouped output by 'epiweek'. You can override using the
`.groups` argument.23.3 Dados de séries temporais
Existem diferentes pacotes para estruturar e manipular dados de séries temporais. Como dito, nós iremos focar nos pacotes da família tidyverts, e, assim, utilizar o pacote tsibble para definir nosso objeto de séries temporais. Definir os dados como um objeto de séries temporais significa que é muito mais fácil estruturar a nossa análise.
Para fazer isso, nós utilizamos a função tsibble() e especificamos o “index”. Por exemplo, a variável especificando a unidade de tempo de interesse. No nosso caso, esta é a variável epiweek.
Se nós tivéssemos dados com contagens semanais por províncias, por exemplo, também seríamos capazes de especificar a variável de agrupamento utilizando o argumento key =. Isto iria nos permitir realizar a análise para cada grupo.
## defina o objeto da série temporal
counts <- tsibble(counts, index = epiweek)Ao executar class(counts) nota-se que, além de ser um quadro de dados (data frame) organizado (“tbl_df”, “tbl”, “data.frame”), ele tem propriedades adicionais de um quadro de dados de uma série temporal (“tbl_ts”).
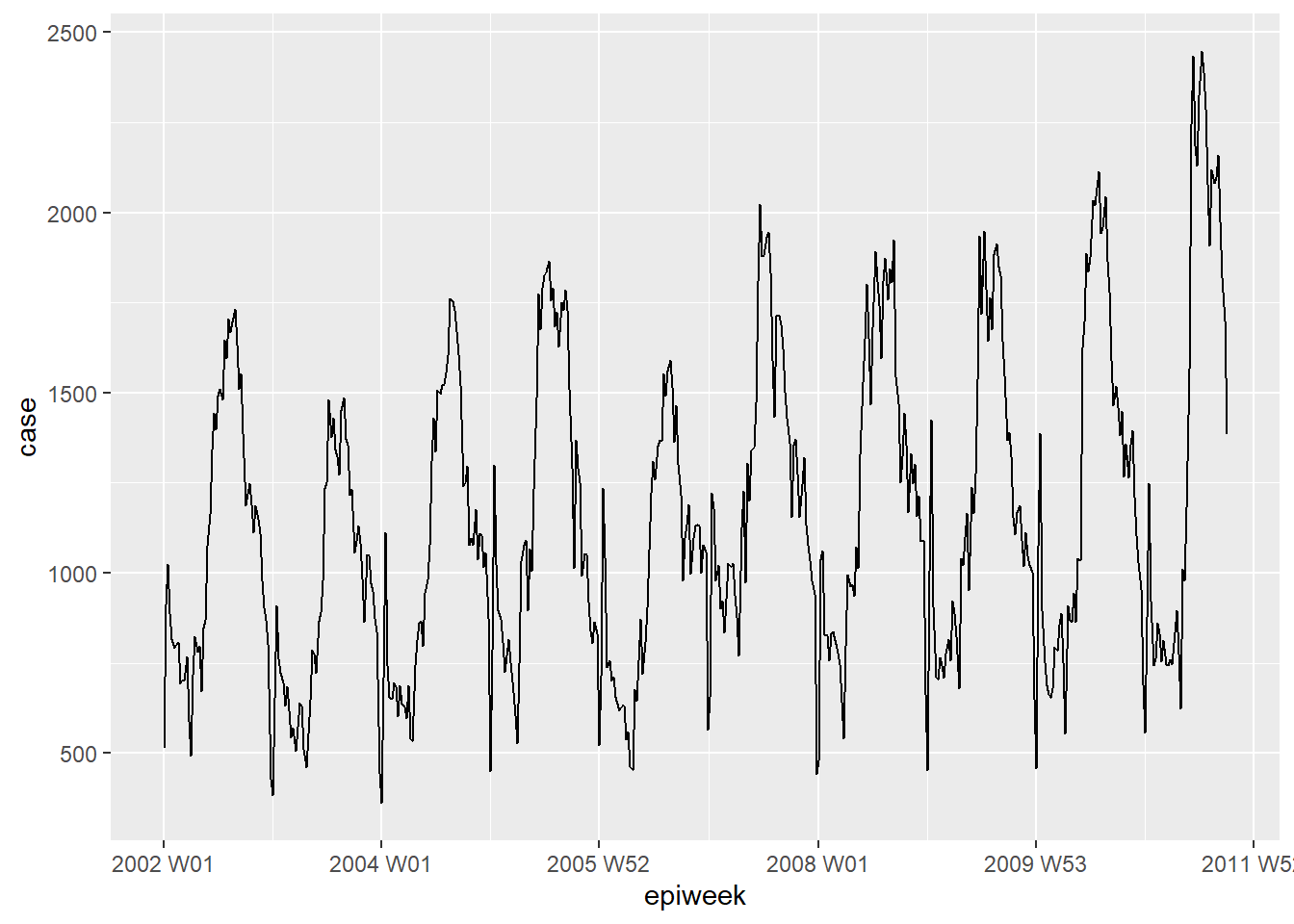
Você pode analisar rapidamente os seus dados ao utilizar o ggplot2. Nós vemos pelo gráfico que existe um padrão sazonal óbvio, e que não existem campos em branco. Entretanto, parece existir um erro para notificar os casos no começo de cada ano; o número de casos cai na última semana do ano, e então aumenta na primeira semana do ano seguinte.
## trace um gráfico de linha de casos por semana
ggplot(counts, aes(x = epiweek, y = case)) +
geom_line()PERIGO: Boa parte dos bancos de dados não são limpos como o deste exemplo. Você irá precisar checar por duplicatas e campos em branco como mostrado abaixo.
Dados duplicados
tsibble não permite observações duplicadas. Então, cada linha precisa ser única, ou única dentro de um grupo (variável key). O pacote tem algumas funções que ajudam a identificar duplicatas. Estes incluem a função are_duplicated(), que gera um vetor TRUE/FALSE caso a linha seja uma duplicata, e a função duplicates() que gera um quadro de dados com as linhas duplicadas.
Veja a página sobre Eliminando duplicidades para mais detalhes sobre como selecionar as linhas que você quer.
## obtenha um vetor de TRUE/FALSE caso as linhas sejam duplicatas
are_duplicated(counts, index = epiweek)
## obtenha um quadro de dados (*data frame*) das linhas duplicadas
duplicates(counts, index = epiweek)Campos em branco
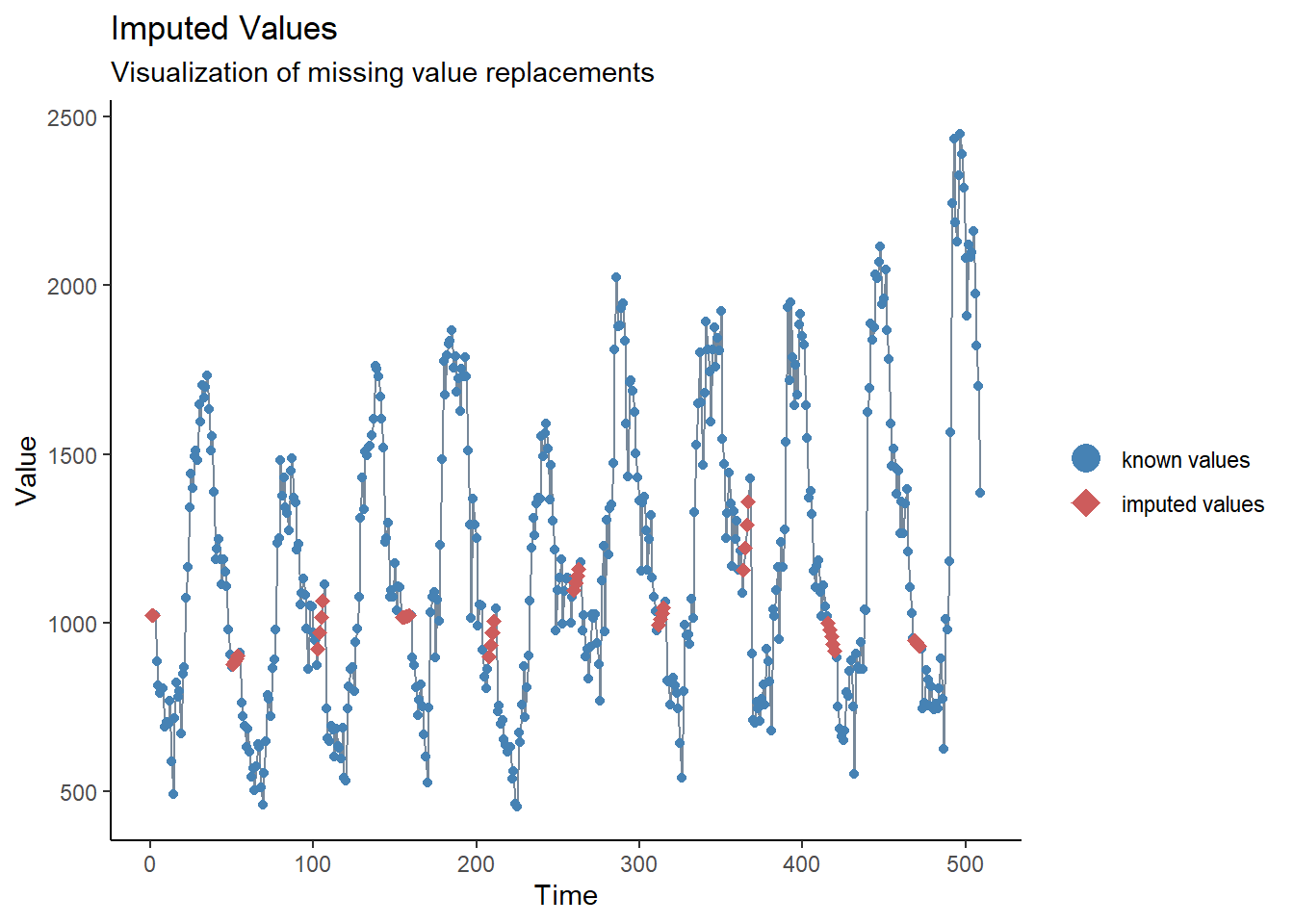
Por meio da breve inspeção demonstrada acima, nós vimos que não existem campos em branco em nossos dados, mas também notamos que existe um problema com atrasos de notificação próximo ao ano novo. Uma forma de abordar isto seria atribuir campos vazios a esses valores, e então inserir novos valores. A forma mais simples de iserção (input) em uma série temporal é desenhar uma linha reta entre o último valor preenchido e o próximo valor preenchido (entre os campos em branco). Para fazer isso, nós iremos utilizar a função na_interpolation() do pacote imputeTS.
Veja a página sobre Dados faltantes para mais informações sobre imputação.
Outra alternativa seria calcular a média móvel, para tentar suavizar esses problemas aparentes de notificação (veja a próxima seção, e a página sobre médias móveis).
## crie uma variálvel com campos em branco em vez das semanas com problemas na notificação
counts <- counts %>%
mutate(case_miss = if_else(
## se as semanas epidemilógicas contém 52, 53, 1 or 2
str_detect(epiweek, "W51|W52|W53|W01|W02"),
## então troque para missing
NA_real_,
## do contrário mantenha o valor do campo
case
))
## alternativamente interpole os campos em branco por uma tendência linear
## entre os dois pontos adjacentes mais próximos
counts <- counts %>%
mutate(case_int = imputeTS::na_interpolation(case_miss)
)
## para checar quais valores foram imputados comparados com os originais
ggplot_na_imputations(counts$case_miss, counts$case_int) +
## crie um gráfico tradicional (com eixos negros e fundo branco)
theme_classic()23.4 Análise descritiva
Médias móveis
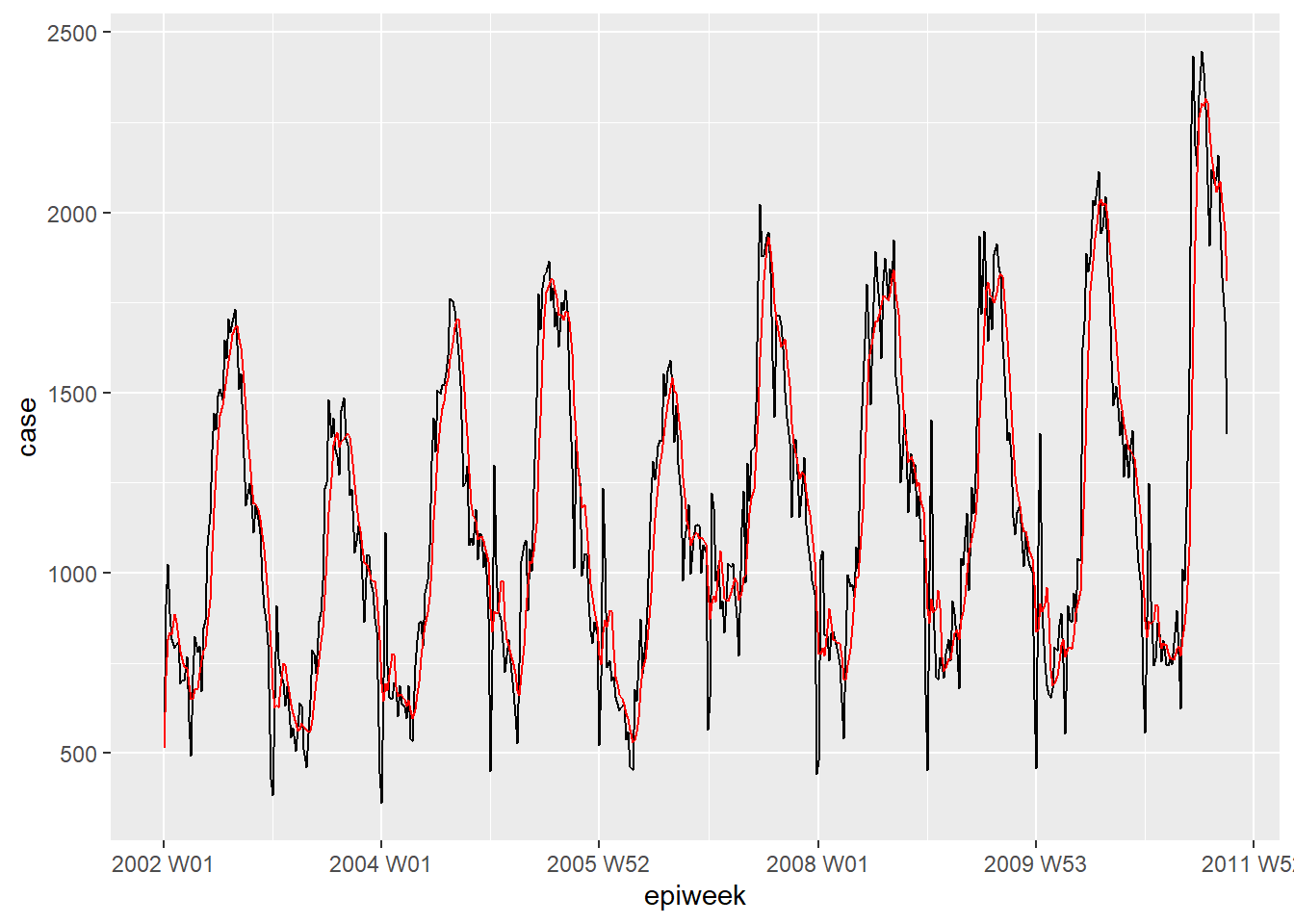
Se os dados apresentam muitos ruídos (contagens pulando para cima e para baixo), então pode ser útil calcular a média móvel. No exemplo abaixo, para cada semana nós calculamos o número médio de casos das quatro semanas anteriores. Isto suaviza os dados para torná-los mais fáceis de interpretar. No nosso caso, isto não traz grandes vantagens, então nós iremos manter os dados interpolados para mais análises. Veja a página sobre Médias móveis para mais detalhes.
## crie uma variável de média móvel (o que resolve o problema dos campos em branco)
counts <- counts %>%
## crie a variável ma_4w
## passe por cada linha da variável dos casos
mutate(ma_4wk = slider::slide_dbl(case,
## para cada linha, calcule a média
~ mean(.x, na.rm = TRUE),
## use as quatro semanas anteriores
.before = 4))
## faça uma visualização rápida da diferença
ggplot(counts, aes(x = epiweek)) +
geom_line(aes(y = case)) +
geom_line(aes(y = ma_4wk), colour = "red")Peridiocidade
Abaixo, nós definimos uma função customizada para criar um periodograma. Veja a página sobre Escrevendo funções para informações sobre como escrever funções no R.
Primeiro, a função é definida. Seus argumentos incluem um banco de dados com uma coluna counts, start_week = que é a primeira semana do banco de dados, um número para indicar quantos períodos por ano (ex.: 52, 12), e, por último, o estilo da saída da análise (veja detalhes no código abaixo).
## Argumentos da função
#####################
## x é um banco de dados
## counts é uma variável com dados contados ou com taxas em x
## start_week é a primeira semana do banco de dados
## period são quantas unidades em um ano
## output é caso você queira retornar um periodograma espectral, ou as semanas de pico
## "periodogram" ou "weeks"
# Defina a função
periodogram <- function(x,
counts,
start_week = c(2002, 1),
period = 52,
output = "weeks") {
## garanta que não é uma classe tsibble, filtre por projeto e mantenha apenas as colunas de interesse
prepare_data <- dplyr::as_tibble(x)
# prepare_data <- prepare_data[prepare_data[[strata]] == j, ]
prepare_data <- dplyr::select(prepare_data, {{counts}})
## crie uma zona intermediária de série temporal para permitir a utilização no spec.pgram
zoo_cases <- zoo::zooreg(prepare_data,
start = start_week, frequency = period)
## obtenha um periodograma espectral sem utilizar a transformação fast fourier
periodo <- spec.pgram(zoo_cases, fast = FALSE, plot = FALSE)
## retorne as semanas com picos de casos
periodo_weeks <- 1 / periodo$freq[order(-periodo$spec)] * period
if (output == "weeks") {
periodo_weeks
} else {
periodo
}
}
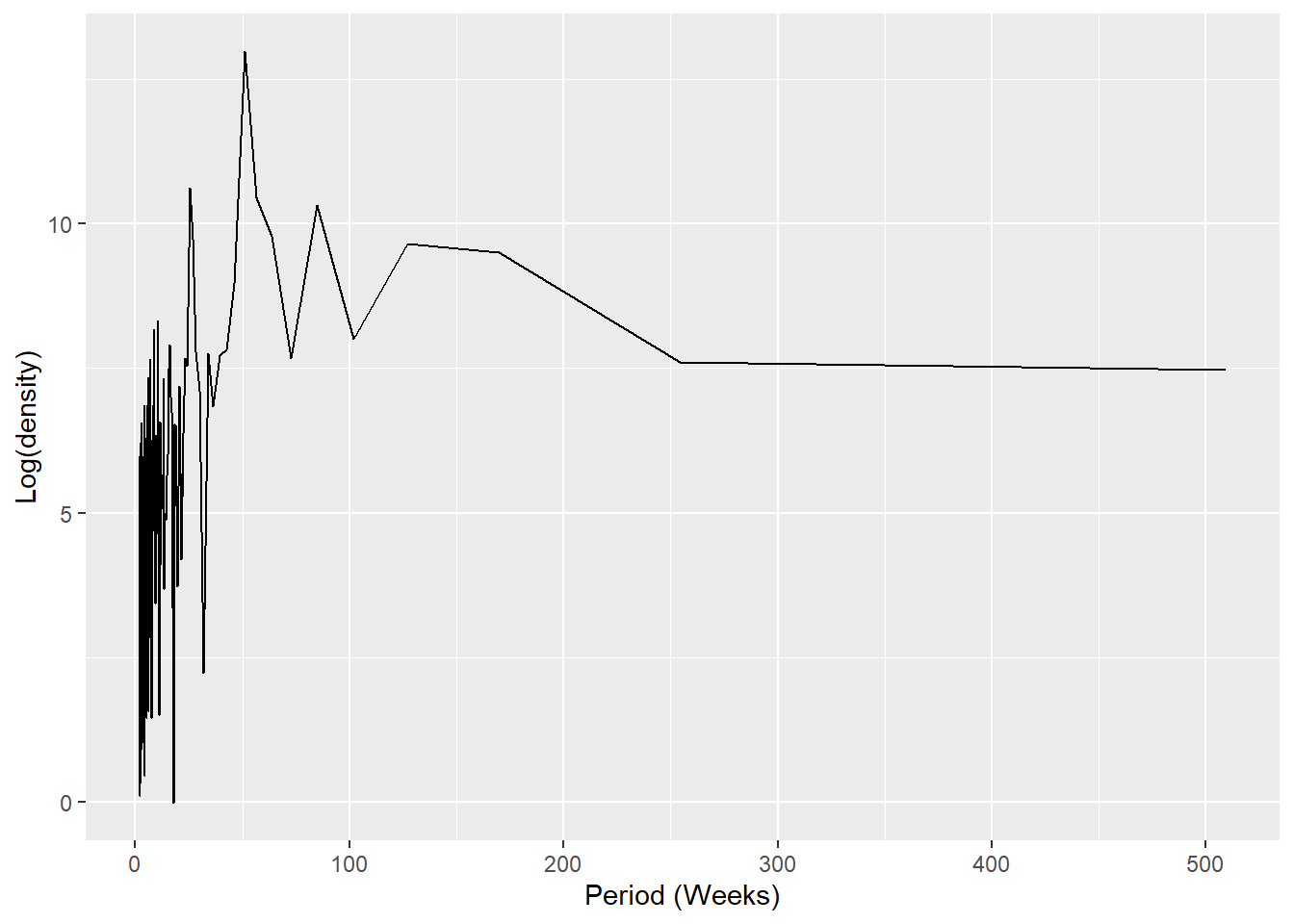
## obtenha um periodograma espectral para extrair semanas com as maiores frequências
## (checando para sazonalidade)
periodo <- periodogram(counts,
case_int,
start_week = c(2002, 1),
output = "periodogram")
## coloque o espectro e a frequência em um quadro de dados para plotagem
periodo <- data.frame(periodo$freq, periodo$spec)
## trace um periodograma mostrando a peridiocidade que ocorre mais frequentemente
ggplot(data = periodo,
aes(x = 1/(periodo.freq/52), y = log(periodo.spec))) +
geom_line() +
labs(x = "Period (Weeks)", y = "Log(density)")## obtenha as semanas em vetor em ordem ascendente
peak_weeks <- periodogram(counts,
case_int,
start_week = c(2002, 1),
output = "weeks")NOTA: É possível utilizar as semanas acima para adicioná-las aos termos de seno e coseno. Entretanto, nós iremos utilizar uma função para gerar esses termos (veja a seção sobre regressão abaixo)
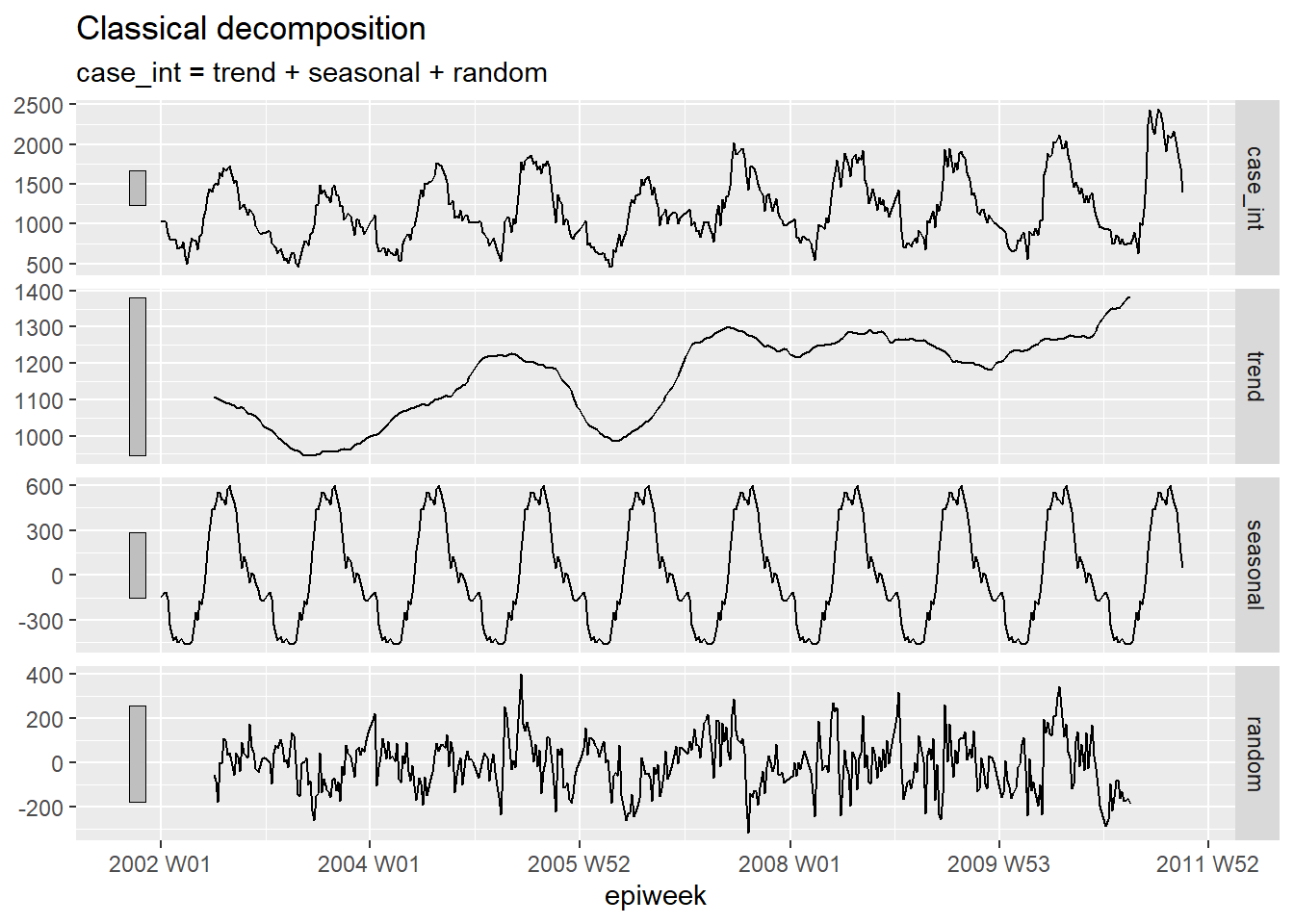
Decomposição
Para dividir a série temporal em diferentes partes, a decomposição clássica é utilizada, pois quando analisadas juntas, é possível verificar um padrão. Estas partes distintas são:
- O ciclo-tendência (a tendência dos dados no longo prazo)
- A sazonalidade (repetição dos padrões)
- O aleatório (o que resta após remover a tendência e a sazonalidade)
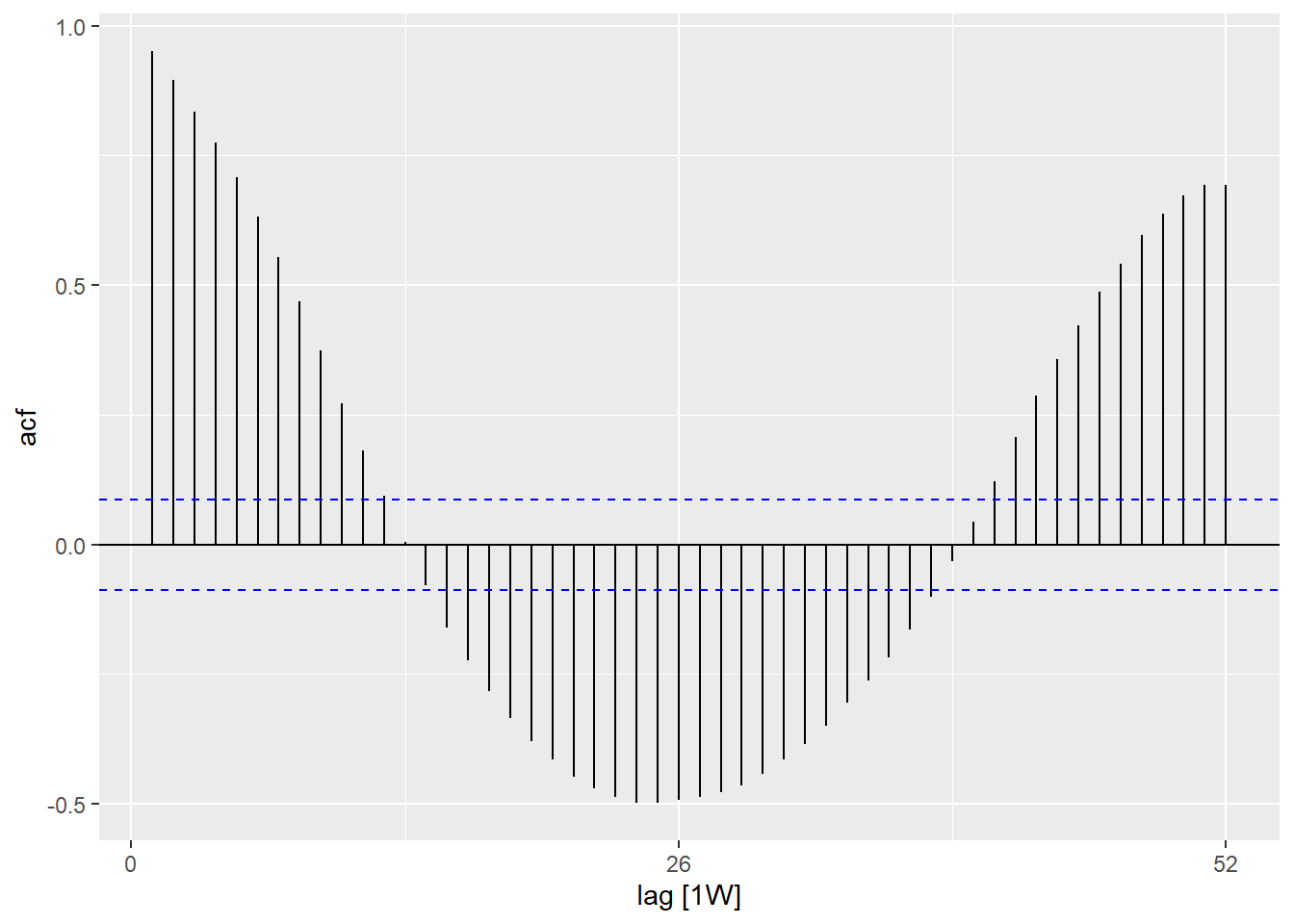
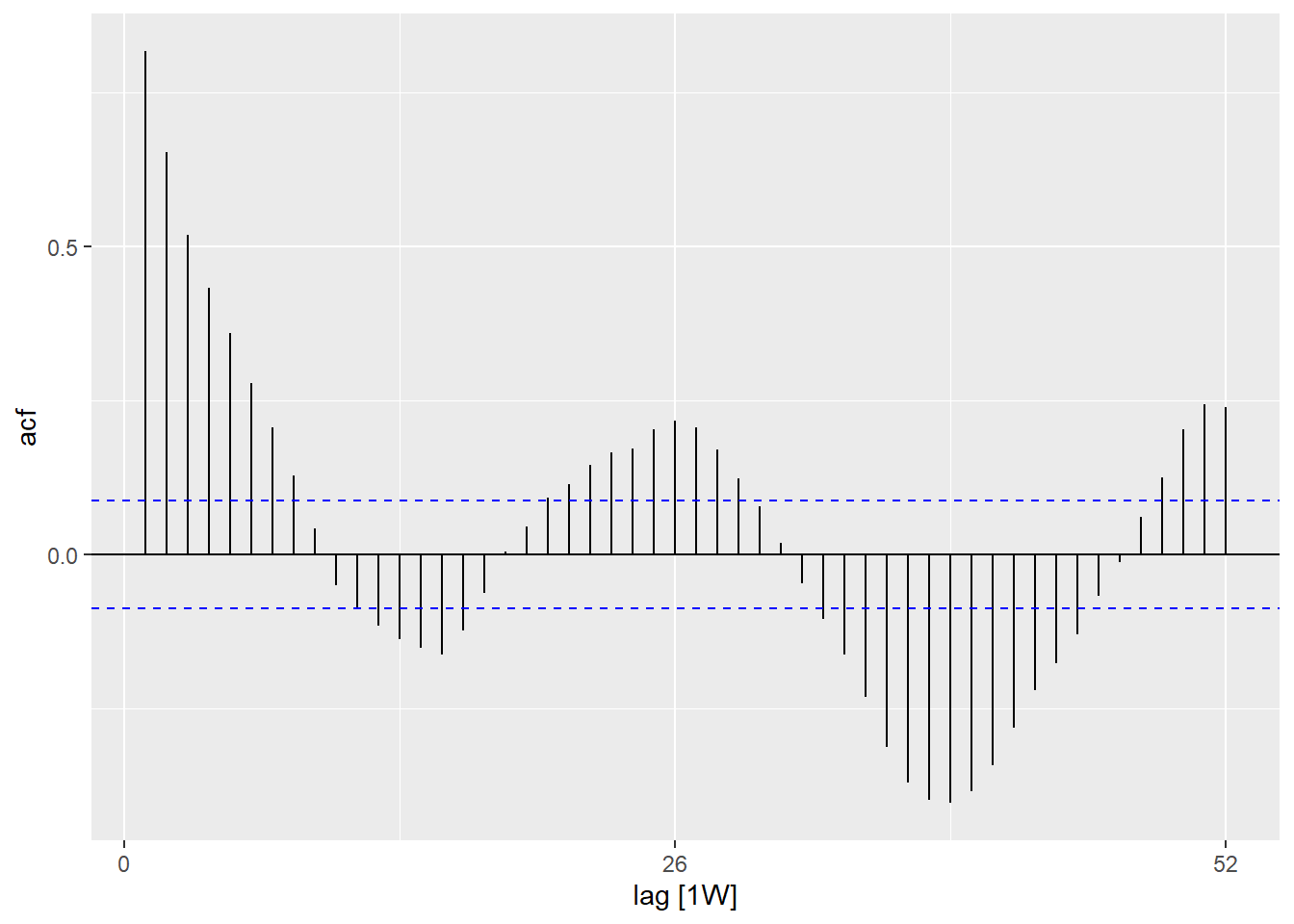
Autocorrelação
Autocorrelação mostra a relação entre as contagens de cada semana e das semanas anteriores a la (chamado de lags).
Ao utilizar a função ACF(), nós podemos gerar um gráfico que mostra o número de linhas em relação aos diferentes defasagens (lags). Quando o lag for 0 (x = 0), esta linha deve ser sempre 1 pois mostra a relação entre uma observação e ela mesma (não mostrado aqui). A primeira linha mostrada aqui (x = 1) mostra a relação entre cada observação e a última observação antes dela (lag de 1), a segunda mostra a relação entre cada observação e a penúltima observação anterior a ela (lag de 2) e assim por diante, até o lag de 52, que mostra a relação entre cada observação e a observação de 1 ano (52 semanas antes).
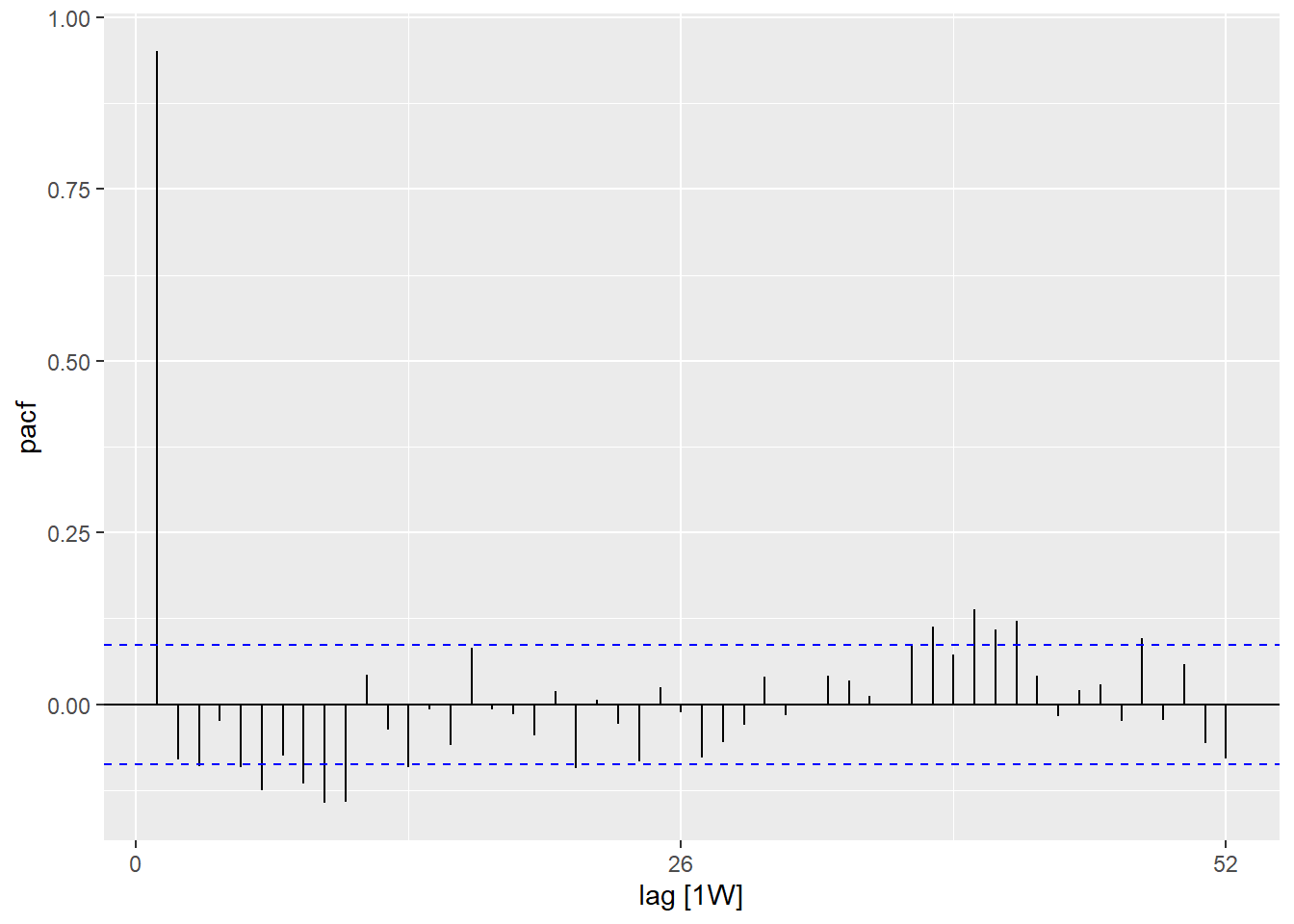
A função PACF() (para autocorrelação parcial) mostra o mesmo tipo de relação mas ajustado para todas as outras semanas no intervalo. Isto é menos útil para determinar a peridiocidade.
## utilizando os dados de contagem
counts %>%
## calcule a autocorrelação utilizando defasagens (lags) que cobrem anos completos
ACF(case_int, lag_max = 52) %>%
## mostre um gráfico
autoplot()## utilizando os dados de contagem
counts %>%
## calcule a autocorrelação parcial utilizando lags que cobrem anos completos
PACF(case_int, lag_max = 52) %>%
## mostre um gráfico
autoplot()Você pode testar formalmente a hipótese nula de independência em uma série temporal (i.e.: ausência de autocorrelação) utilizando o teste de Ljung-Box (no pacote stats). Um p-valor significantivo sugere que exista uma autocorrelação nos dados.
## avalia a existência de autocorrelação
Box.test(counts$case_int, type = "Ljung-Box")
Box-Ljung test
data: counts$case_int
X-squared = 462.65, df = 1, p-value < 2.2e-1623.5 Ajustando regressões
É possível ajustar uma vasta quantidade de diferentes regressões em uma série temporal. Entretanto, aqui nós iremos demonstrar como ajustar uma regressão binominal negativa - uma
vez que frequentemente esta é a mais apropriada para dados de contagem em doenças infecciosas.
Termos de Fourier
Termos de Fourier são os equivalentes de curvas seno e coseno. A diferença é que estes são ajustados com base na identificação da combinação mais apropriada das curvas para explicar seus dados.
O ajuste de apenas um termo de Fourier equivale a ajustar um seno e um coseno para a defasagem (lag) que ocorre mais frequentemente em seu periodograma (no nosso caso, de 52 semanas). Para isso, nós utilizamos a função fourier() do pacote forecast.
No código abaixo, a atribuição é feita utilizando o $, uma vez que fourier() retorna duas colunas (uma
para seno, outra para coseno) que são adicionadas aos dados como uma lista, chamada “fourier”, que pode então ser utilizada normalmente como uma variável na regressão.
## adicione os termos de Fourier utilizando as variáveis epiweek e case_int
counts$fourier <- select(counts, epiweek, case_int) %>%
fourier(K = 1)Binominal negativa
É possível ajustar regressões utilizando funções básicas do stats ou MASS (ex.: lm(), glm() e glm.nb()). Entretanto, nós iremos utilizar as funções do pacote trending, pois ele permite o cálculo apropriado dos intervalos de confiança e de predição (que de outra forma não estão disponíveis). A sintaxe é a mesma, em que você especifica uma variável para atribuir o resultado, um til (~), e então adiciona as variáveis de interesse separadas por um sinal de mais (+).
A outra diferença é que nós primeiro definimos o modelo e então o ajustamos fit() (ajustamos) para os dados. Isto é útil porque permite a comparação de diferentes modelos com a mesma sintaxe.
DICA: Se você quiser utilizar taxas, em vez de contagens, é possível incluir a variável população como um termo logaritmo de compensação, ao adicionar
offset(log(population). Para calcular uma taxa, você então precisaria ajustar a população para ser 1, antes de utilizar a função predict().
DICA: Para ajustar modelos mais complexos como ARIMA ou profeta (prophet), veja o pacote fable.
## escolha o modelo que você quer ajustar (binominal negativa)
model <- glm_nb_model(
## escolha o número de casos como resultado de interesse
case_int ~
## utilize a variável epiweek para levar em consideração a tendência
epiweek +
## utilize os termos de Fourier para levar em consideração a sazonalidade
fourier)
## ajuste seu modelo utilizando os dados de contagem
fitted_model <- trending::fit(model, data.frame(counts))
## calcule os intervalos de confiança e predição
observed <- predict(fitted_model, simulate_pi = FALSE)
estimate_res <- data.frame(observed$result)
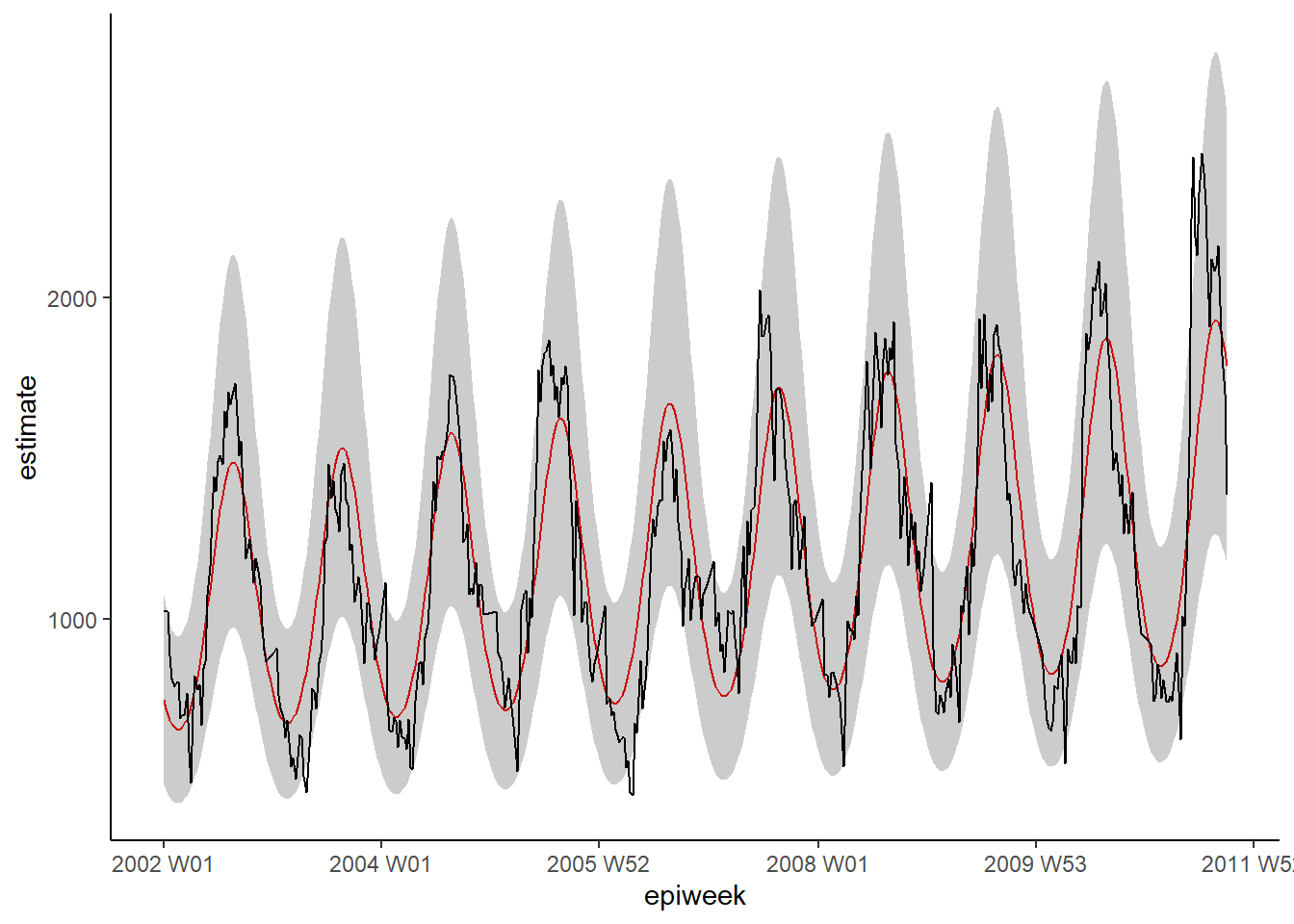
## faça um gráfico da sua regressão
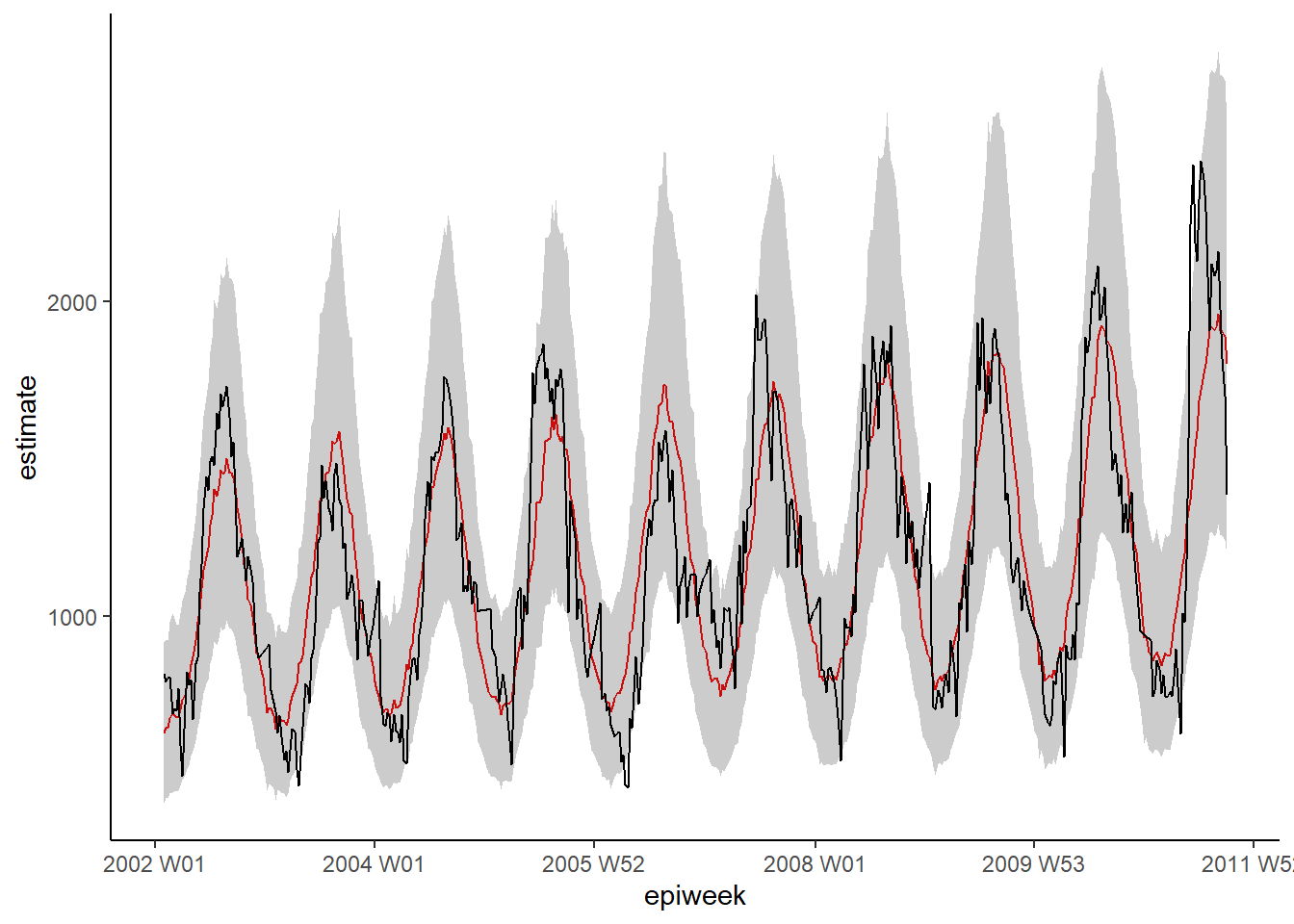
ggplot(data = estimate_res, aes(x = epiweek)) +
## adicione uma linha para a estimativa do modelo
geom_line(aes(y = estimate),
col = "Red") +
## adicione uma faixa sombreada dos intervalos de predição
geom_ribbon(aes(ymin = lower_pi,
ymax = upper_pi),
alpha = 0.25) +
## adicione uma linha para o número de casos
geom_line(aes(y = case_int),
col = "black") +
## faça um gráfico tradicional (com eixos pretos e fundo branco)
theme_classic()Resíduos
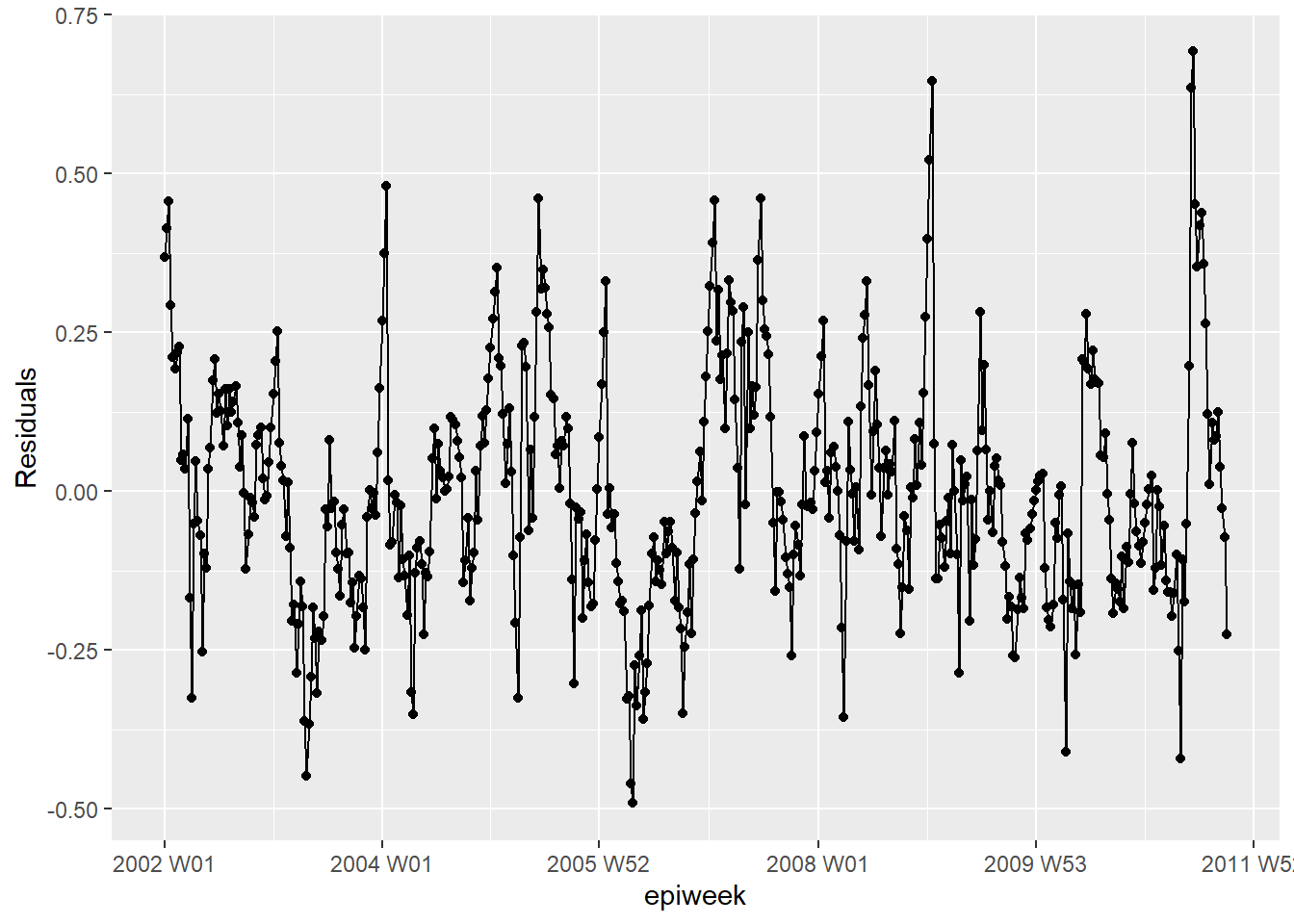
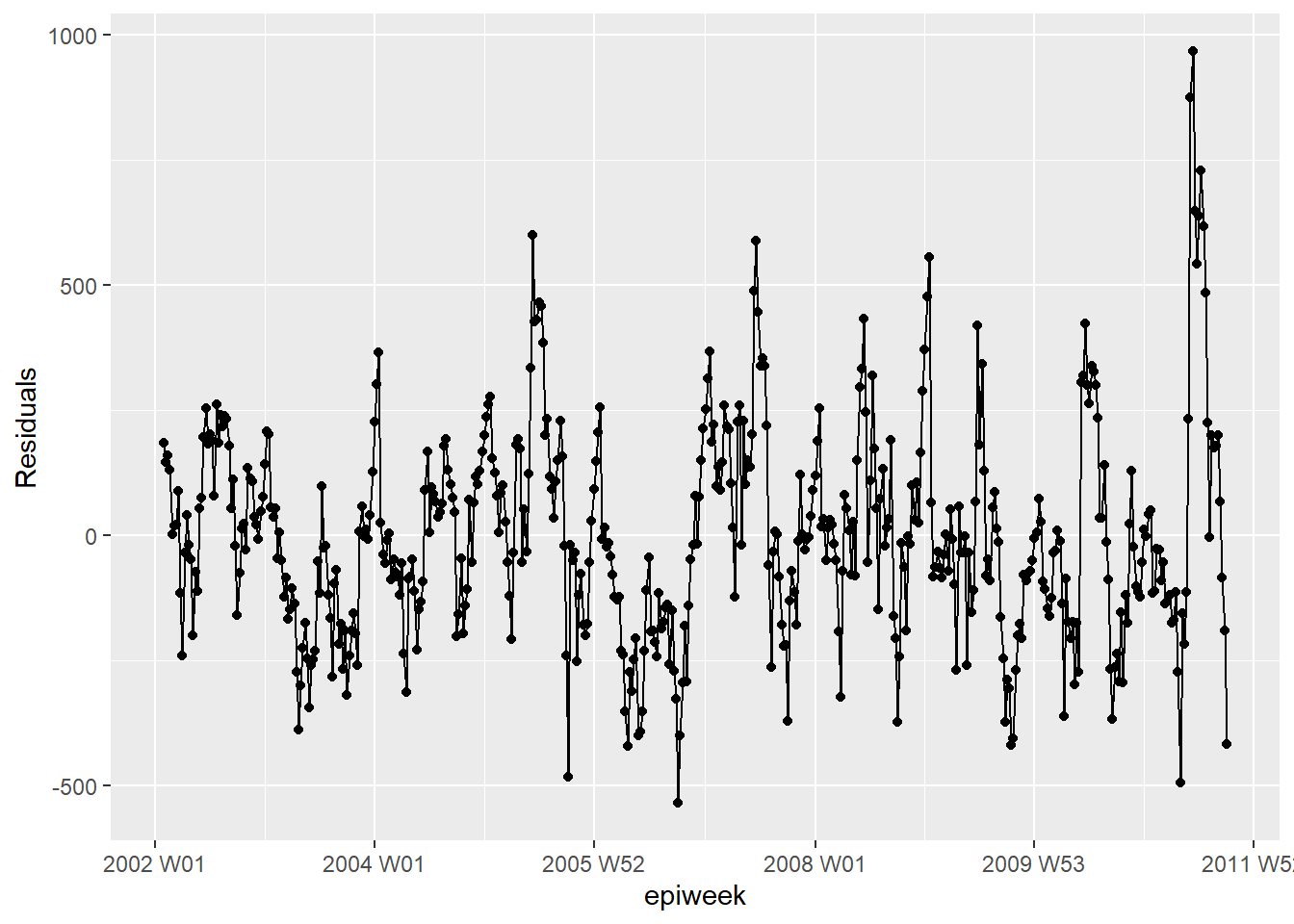
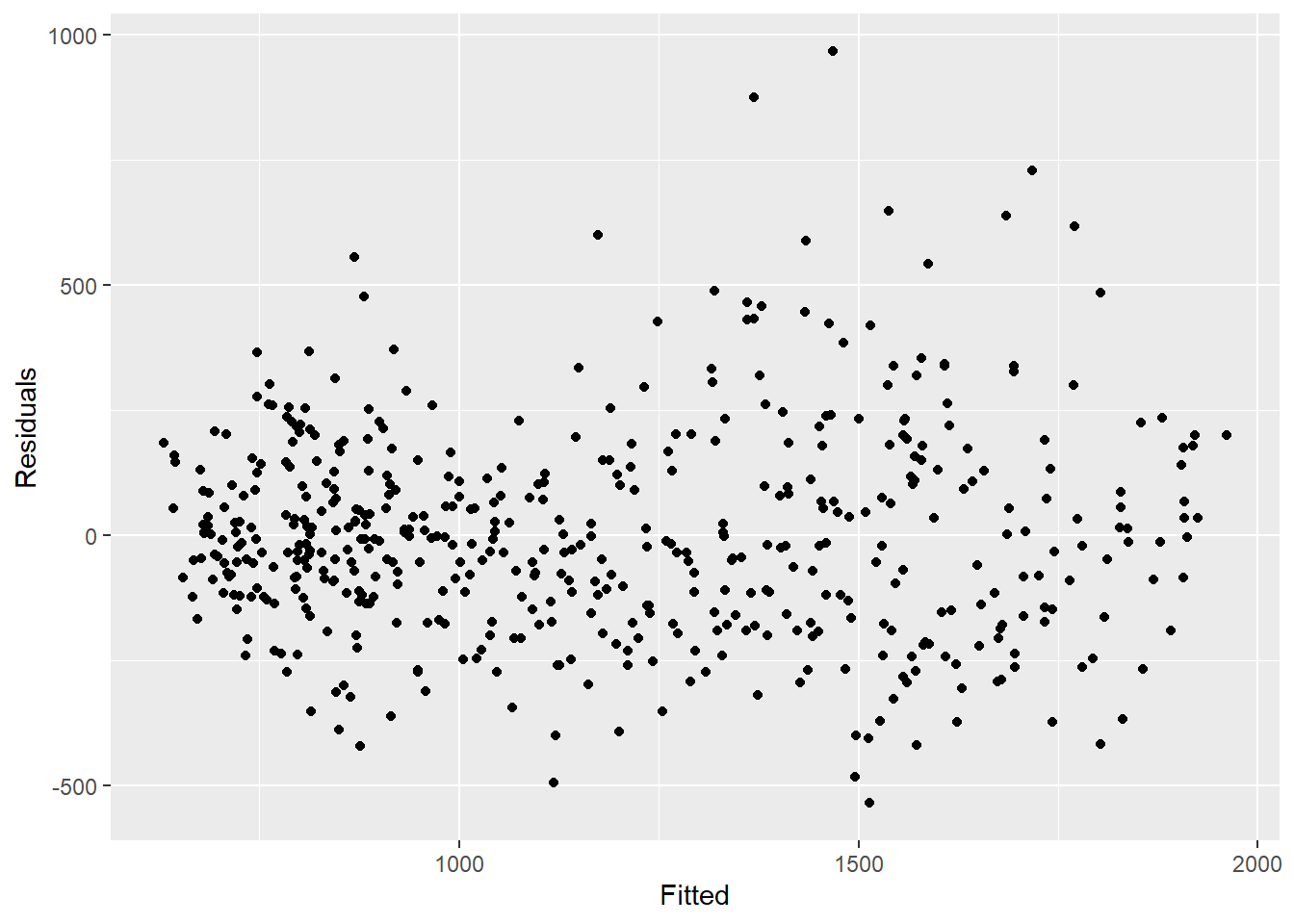
Para avaliar o quão bem nosso modelo se ajusta aos dados observados, precisamos checar os resíduos. Os resíduos são a diferença entre as contagens observadas e as contagens estimadas pelo modelo. Nós podemos calcular isto de forma simples, utilizando case_int - estimate, mas a função residuals() extrai isso diretamente da regressão para nós.
O que vemos abaixo é que não estamos explicando toda a variabilidade dos dados com o nosso modelo. Uma possibilidade seria ajustar mais termos de Fourier, e verificar a amplitude. Entretanto, para este exemplo, nós iremos deixar da forma que está. Os gráficos mostram que nosso modelo possui pior desempenho nos picos e nas baixas (quando as contagens estão nas maiores ou nas menores quantidades). Sendo assim, ele provavelmente irá subestimar as contagens observadas.
## calcule os resíduos
estimate_res <- estimate_res %>%
mutate(resid = fitted_model$result[[1]]$residuals)
## os resíduos são razoavelmente constantes ao longo do tempo (se não: surtos? mudanças na prática?)
estimate_res %>%
ggplot(aes(x = epiweek, y = resid)) +
geom_line() +
geom_point() +
labs(x = "epiweek", y = "Residuals")## existe autocorrelação nos resíduos (existe um padrão para os erros?)
estimate_res %>%
as_tsibble(index = epiweek) %>%
ACF(resid, lag_max = 52) %>%
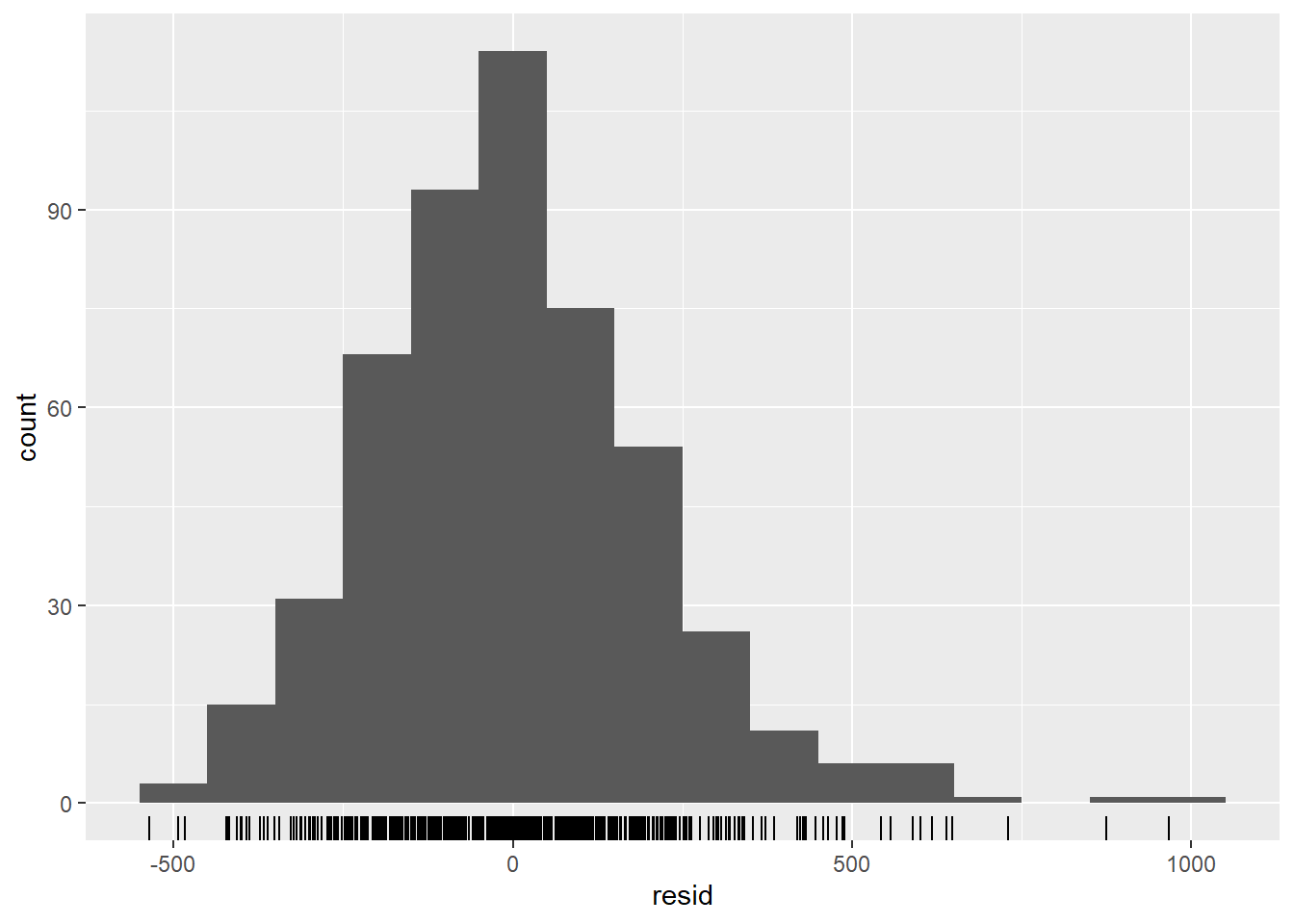
autoplot()## os resíduos estão distribuídos de forma normal (estão sendo sub ou super estimados?)
estimate_res %>%
ggplot(aes(x = resid)) +
geom_histogram(binwidth = 100) +
geom_rug() +
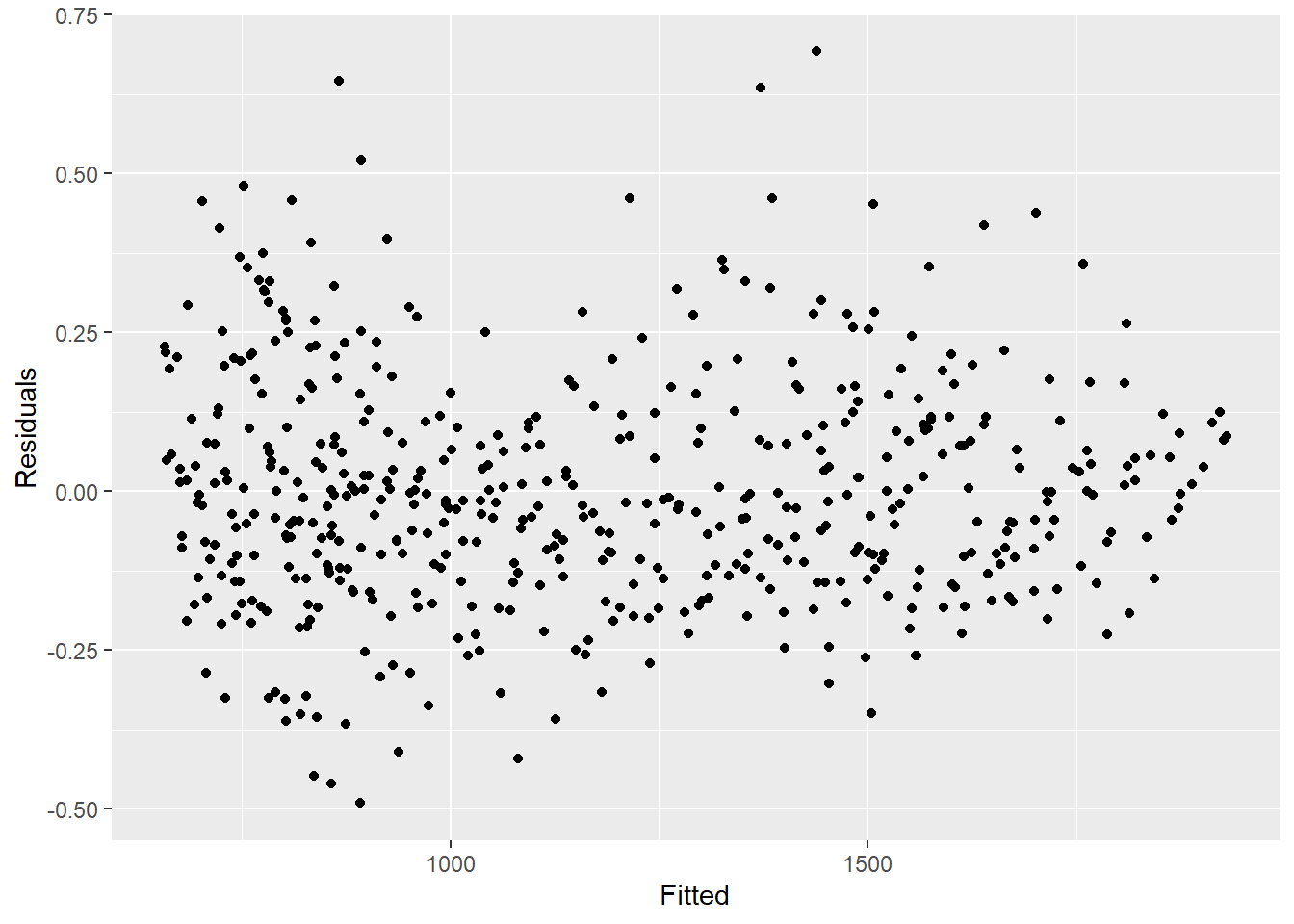
labs(y = "count") ## compare as contagens observadas com os resíduos
## não deve haver padrão
estimate_res %>%
ggplot(aes(x = estimate, y = resid)) +
geom_point() +
labs(x = "Fitted", y = "Residuals")## teste formalmente a autocorrelação dos resíduos
## H0 é que os resíduos são ruídos brancos (i.e.: aleatórios)
## teste de independência
## se p-valor for significantivo, então não é aleatório
Box.test(estimate_res$resid, type = "Ljung-Box")
Box-Ljung test
data: estimate_res$resid
X-squared = 336.25, df = 1, p-value < 2.2e-1623.6 Relação entre duas séries temporais
Aqui nós vemos como utilizar dados climáticos (especificamente a temperatura) para explicar a quantidade de casos de campylobacter.
Unindo bancos de dados
Nós podemos unir nossos bancos de dados utilizando a variável de semana. Para mais informações sobre junção de dados, veja no manual a seção sobre unindo dados.
## junção à esquerda, de forma que nós teremos apenas as linhas existentes na tabela de contagens
## exclua a variável date do temp_data (do contrário, ficará duplicada)
counts <- left_join(counts,
select(temp_data, -date),
by = "epiweek")Análise descritiva
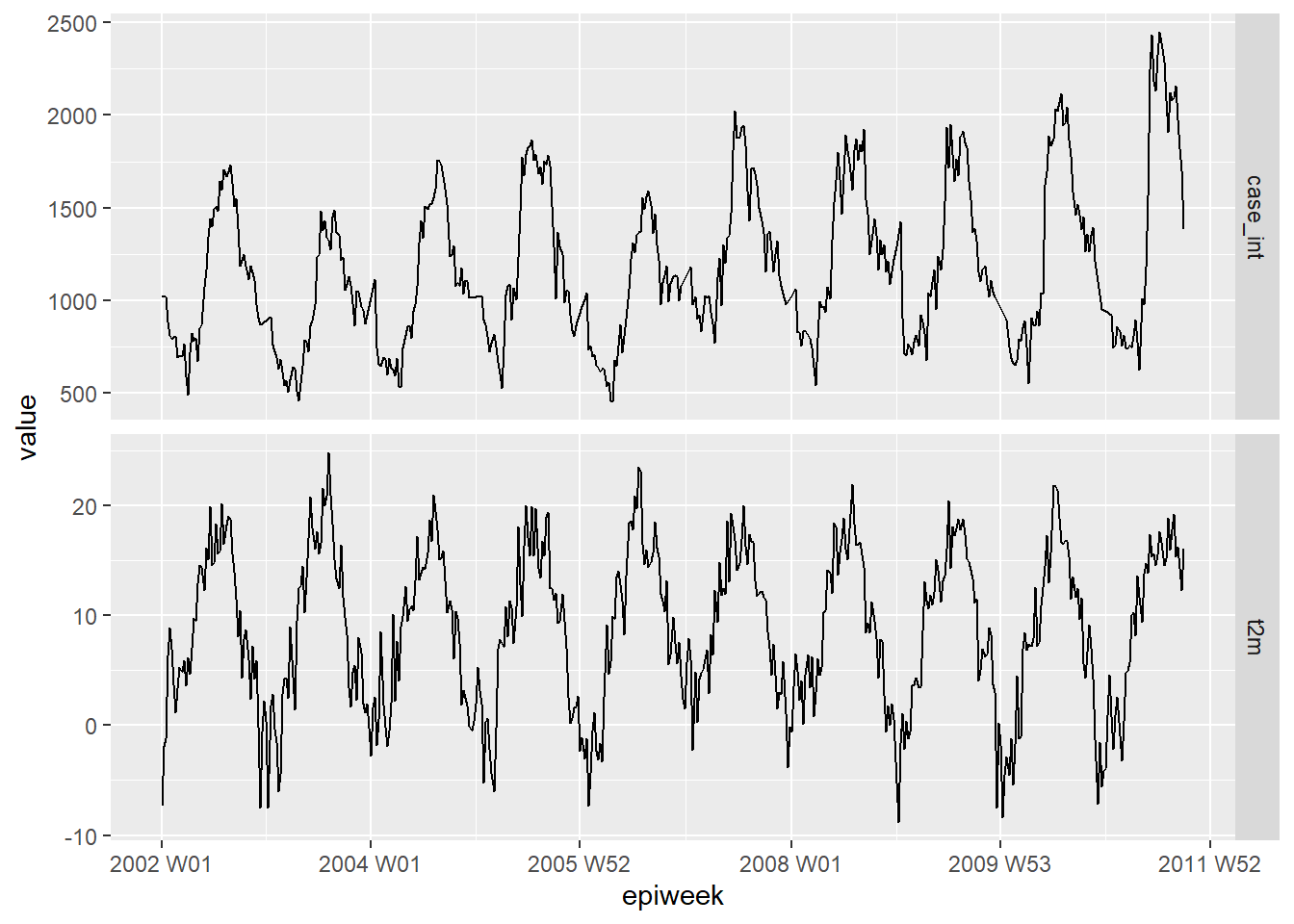
Primeiro, faça um gráfico de seus dados para ver se existe alguma relação óbvia. O gráfico abaixo mostra que existe uma relação clara na sazonalidade das duas variáveis, e que a temperatura pode aumentar algumas semanas antes da contagem de casos. Para mais sobre o pivoteamento dos dados, veja a seção sobre pivoteando dados neste manual.
counts %>%
## mantenha as variáveis de interesse
select(epiweek, case_int, t2m) %>%
## mude seus dados para o formato longo
pivot_longer(
## use a epiweek como "chave" para união
!epiweek,
## move o nome das colunas para a nova coluna "measure"
names_to = "measure",
## move os valores das células para a nova coluna "values"
values_to = "value") %>%
## crie um gráfico com os dados acima
## trace um gráifco com a epiweek no eixo x e os valores (contagens/temperatura) no eixo y
ggplot(aes(x = epiweek, y = value)) +
## crie um gráfico separado para temperaturas e contagens dos casos
## deixe a escala do eixo y livre
facet_grid(measure ~ ., scales = "free_y") +
## adiciona as informações como uma linha no gráfico
geom_line()Lags e correlação cruzada
Para formalmente testar quais semanas estão mais relacionadas com os casos e temperatura. Nós podemos utilizar a função de correlação cruzada (CCF()) do pacote feasts. Você pode também visualizar (em vez de utilizar arrange) os dados com a função autoplot().
counts %>%
## calcule a correlação cruzada entre as contagens e temperaturas interpoladas
CCF(case_int, t2m,
## ajuste o lag máximo para 52 semanas
lag_max = 52,
## retorne o coeficiente de correlação
type = "correlation") %>%
## organize pela ordem decrescente do coeficiente de correlação
## mostre as defasagens de tempo com maior associação
arrange(-ccf) %>%
## apenas mostre os dez maiores coeficientes
slice_head(n = 10)# A tsibble: 10 x 2 [1W]
lag ccf
<cf_lag> <dbl>
1 -4W 0.749
2 -5W 0.745
3 -3W 0.735
4 -6W 0.729
5 -2W 0.727
6 -7W 0.704
7 -1W 0.695
8 -8W 0.671
9 0W 0.649
10 47W 0.638A partir disso, nós vemos que uma defasagem (lag) de 4 semanas é o mais altamente correlacionado. Então, nós criamos uma variável de temperatura com esse lag para incluir em nossa regressão.
PERIGO: Observe que as quatro primeiras semanas dos nossos dados na variável de temperatura com lag estão em branco (NA) - como não existem quatro semanas anteriores para obter os dados. Para utilizar esses dados com a função predict(),
do pacote trending, nós precisamos usar o argumento simulate_pi = FALSE dentro de predict() nas etapas posteriores. Se nós quiséssemos utilizar a opção de simular, então precisaríamos excluir esses campos em branco, e salvar um novo conjunto de dados ao adicionar drop_na(t2m_lag4) ao código abaixo.
counts <- counts %>%
## crie uma nova variável para temperatura com lag de quatro semanas
mutate(t2m_lag4 = lag(t2m, n = 4))Binominal negativa com duas variáveis
Nós ajustamos uma regressão binominal negativa como feito anteriormente. Desta vez, adicionamos a variável temperatura com uma defasagem de tempo (lag) de quatro semanas.
CUIDADO: Observe o uso do argumento simulate_pi = FALSE dentro da função predict(). Isto se deve porque o comportamento padrão de trending é usar o pacote ciTools para estimar um intervalo de predição. Isto não funciona se existirem contagen NA, e também produz intervalos mais granulares. Veja ?trending::predict.trending_model_fit para mais detalhes.
## defina o modelo que você quer ajustar (binominal negativa)
model <- glm_nb_model(
## ajuste o número de casos como resultado de interesse
case_int ~
## use epiweek para levar em conta a tendência
epiweek +
## use os termos fourier para levar em conta a sazonalidade
fourier +
## use a temperatura com lag de quatro semanas
t2m_lag4
)
## ajuste seu modelo utilizando os dados de contagem
fitted_model <- trending::fit(model, counts)
## calcule os intervalos de confiança e de predição
observed <- predict(fitted_model, simulate_pi = FALSE)Para investigar os termos individual, nós podemos retomar a regressão binominal negativa original do formato trending utilizando a função get_model() e aplica-la na função tidy() function do pacote broom para obter estimativas exponencializadas e intervalos de confiança associados.
O que isso nos mostra é que a temperatura defasada, depois de controlar o modelo para tendência e sazonalidade, é similar a contagem dos casos (estimativa ~ 1) e significativamente associada com os casos. Isto sugere que esta possa ser uma boa variável para prever a quantidade de casos futura (conforme a previsão do tempo é disponibilizada).
fitted_model %>%
## extraia a regressão negativa binominal original
get_fitted_model() #%>% [[1]]
Call: glm.nb(formula = case_int ~ epiweek + fourier + t2m_lag4, data = counts,
init.theta = 32.80689607, link = log)
Coefficients:
(Intercept) epiweek fourierS1-52 fourierC1-52 t2m_lag4
5.825e+00 8.464e-05 -2.850e-01 -1.954e-01 6.672e-03
Degrees of Freedom: 504 Total (i.e. Null); 500 Residual
(4 observations deleted due to missingness)
Null Deviance: 2015
Residual Deviance: 508.2 AIC: 6784 ## obtenha um quadro de dados organizado dos resultados
# tidy(exponentiate = TRUE,
# conf.int = TRUE)Um inspeção visual rápida do modelo mostra que ele pode fazer um trabalho melhor em estimar a quantidade de casos observada.
estimate_res <- data.frame(observed$result)
## faça um gráfico da sua regressão
ggplot(data = estimate_res, aes(x = epiweek)) +
## adicione uma linha com a estimativa do modelo
geom_line(aes(y = estimate),
col = "Red") +
## adicione uma faixa sombreada dos intervalos de predição
geom_ribbon(aes(ymin = lower_pi,
ymax = upper_pi),
alpha = 0.25) +
## adicione uma linha com a quantidade de casos observada
geom_line(aes(y = case_int),
col = "black") +
## faça uma gráfico tradicional (com eixos pretos e fundo branco)
theme_classic()Resíduos
Aqui, novamente avaliaremos os resíduos para ver o quão bem o nosso modelo se ajusta aos dados observados. Os resultados e a interpretação são similares àqueles da regressão anterior, então pode ser mais viável continuar com o modelo mais simples, sem a temperatura.
## calcule os resíduos
estimate_res <- estimate_res %>%
mutate(resid = case_int - estimate)
## os resíduos são razoavelmente constantes ao longo do tempo (caso não: surtos? mudanças na prática?)
estimate_res %>%
ggplot(aes(x = epiweek, y = resid)) +
geom_line() +
geom_point() +
labs(x = "epiweek", y = "Residuals")## existe uma autocorrelação nos resíduos (existe um padrão para o erro?)
estimate_res %>%
as_tsibble(index = epiweek) %>%
ACF(resid, lag_max = 52) %>%
autoplot()## os resíduos são normalmente distribuídos (estão sub ou super estimando?)?
estimate_res %>%
ggplot(aes(x = resid)) +
geom_histogram(binwidth = 100) +
geom_rug() +
labs(y = "count") ## compare as contagens observadas com os seus resíduos
## também não deve haver padrão
estimate_res %>%
ggplot(aes(x = estimate, y = resid)) +
geom_point() +
labs(x = "Fitted", y = "Residuals")## formalmente avalie a autocorrelação dos resíduos
## H0 é que os resíduos são uma série de ruídos brancos (i.e.: aleatórios)
## teste de independência
## se o p-valor for signficativo, então os resíduos não são aleatórios
Box.test(estimate_res$resid, type = "Ljung-Box")
Box-Ljung test
data: estimate_res$resid
X-squared = 339.52, df = 1, p-value < 2.2e-1623.7 Detecção de surtos
Aqui, nós iremos demonstrar dois métodos similares para detecção de surtos. O primeiro tem como base as seções acima. Nós usamos o pacote trending para ajustar regressões para os anos anteriores, e então predizer o que esperar para o próximo ano. Se as contagens observadas estão acima do esperado, é possível que exista um surto. O segundo método é baseado em princípios similares, mas utiliza o pacote surveillance, que possui uma variedade de algoritmos diferentes para detecção de anomalias.
CUIDADO: Normalmente, você tem interesse no ano corrente (onde você apenas conhece contagens até a semana atual). Então, neste exemplo, nós fingiremos estar na semana 39 de 2011.
Pacote trending
Para este método, nós definimos uma base de referência (usualmente 5 anos de dados). Nós ajustamos uma regressão com esses dados, e então usamos isto para prever as estimativas para o próximo ano.
Data limite
É mais fácil definir suas datas em um local, e então utiliza-las no resto do seu código.
Aqui, nós definimos uma data de início (quando suas observações começaram) e uma data limite (o final do seu período de referência - e quando o período que nós queremos prever começa). ~Nós também definimos quantas semanas estão no nosso ano de interesse (o ano que iremos fazer previsões)~. E também definimos quantas semanas estão entre o limite do nosso período de referência, e a data final que estamos interessados em fazer previsões.
NOTA: Neste exemplo, nós fingimos estar no final de setembro de 2011 (“2011 W39”).
## defina data de início (quando as observações foram iniciadas)
start_date <- min(counts$epiweek)
## defina uma semana limite (final do período de referência, início do período de previsão)
cut_off <- yearweek("2010-12-31")
## defina a última data de interesse (i.e.: final da previsão)
end_date <- yearweek("2011-12-31")
## encontre quantas semanas no período (ano) de interesse
num_weeks <- as.numeric(end_date - cut_off)Adicione linhas
Para ser capaz de prever no formato tidyverse, nós precisamos ter o número correto de linhas em nosso banco de dados, i.e.: uma linha para cada semana até a data final (end_date) definida acima. O código abaixo permite adicionar essas linhas de acordo com uma variável de agrupamento - por exemplo, se nós tivéssemos múltiplos países em um conjunto de dados, poderíamos agrupar por país e então adicionar linhas apropriadas para cada um. A função group_by_key(), do pacote tsibble, nos permite fazer esse agrupamento e então passar os dados agrupados para as funções do dplyr, group_modify() e add_row(). Então, nós especificamos a sequência de semanas entre a primeira e a última semana atualmente disponíveis nos dados e a semana final.
## adicione semanas em branco até o final do ano
counts <- counts %>%
## agrupe por região
group_by_key() %>%
## para cada grupo, adicione linhas da maior semana epidemiológica até o final do ano
group_modify(~add_row(.,
epiweek = seq(max(.$epiweek) + 1,
end_date,
by = 1)))Termos de Fourier
Nós precisamos redefinir nossos termos de fourier - uma vez que queremos ajustá-los apenas ao período de referência, e então prever (extrapolar) esses termos para o próximo ano. Para fazer isso, nós precisamos combinar duas listas geradas da função fourier(); a primeira se trata dos dados que são a base de referência, e a segunda a previsão para o ano de interesse (ao definir o argumento h).
N.b. para unir linhas, nós temos que usar rbind() (em vez da função bind_rows do tidyverse) pois as colunas fourier estão em forma de lista (logo, não são nomeadas individualmente).
## defina os termos de fourier (senos e cosenos)
counts <- counts %>%
mutate(
## combine os termos de fourier para semanas antes e após a data limite de 2010
## (termos de fourier para 2011 e antes são estimados)
fourier = rbind(
## obtenha termos de fourier dos anos anteriores
fourier(
## mantenha apenas as linhas anteriores à 2011
filter(counts,
epiweek <= cut_off),
## inclua um conjunto de termos seno e coseno
K = 1
),
## preveja os termos de fourier para 2011 (utilizando os dados base de referência)
fourier(
## mantenha apenas as linhas que estejam dentro do período de interesse
filter(counts,
epiweek <= cut_off),
## inclua um conjunto de termos seno e coseno
K = 1,
## preveja as 52 semanas seguintes
h = num_weeks
)
)
)Divida os dados e ajuste a regressão
Agora nós precisamos dividir o nosso banco de dados entre o período de referência e o período a ser previsto. Isto é feito utilizando a função group_split() do pacote dplyr, após utilizar group_by(), e irá criar uma lista com dois conjuntos de dados, um para antes do período da previsão, e outro com esse período.
Então nós utilizamos a função pluck() do pacote purrr, para extrair o conjunto de dados da lista (equivalente de utilizar os colchetes quadrados, ex.: dat[[1]]), e então ajustar o nosso modelo para os dados de referência, e então aplicar a função predict() para as nossas datas de interesse após a data limite do período de referência.
Veja a página sobre Iteração, loops, e listas para aprender mais sobre o pacote purrr.
CUIDADO: Observe o uso do argumento simulate_pi = FALSE dentro de predict(). Isto é porque o comportamento padrão de trending é utilizar o pacote ciTools para estimar o intervalo de previsão. Entretanto, isto não funciona se existirem contagens de NA, além de produzir mais intervalos granulares. Veja ?trending::predict.trending_model_fit para mais detalhes.
# divida os dados para ajustes e previsões
dat <- counts %>%
group_by(epiweek <= cut_off) %>%
group_split()
## defina o modelo que você quer ajustar (binominal negativo)
model <- glm_nb_model(
## ajuste o número de casos como resultado de interesse da análise
case_int ~
## use as semanas epidemiológicas ('epiweek') para considerar a tendência
epiweek +
## use os termos de fourier para considerar a sazonalidade
fourier
)
# defina quais dados utilizar para ajustar o modelo e quais para predizer
fitting_data <- pluck(dat, 2)
pred_data <- pluck(dat, 1) %>%
select(case_int, epiweek, fourier)
# ajuste o modelo
fitted_model <- trending::fit(model, fitting_data)
# obtenha os intervalos de confiança e estimativas para os dados ajustados
observed <- fitted_model %>%
predict(simulate_pi = FALSE)
# faça as previsões com os dados a serem preditos
forecasts <- fitted_model %>%
predict(pred_data, simulate_pi = FALSE)
## combine os bancos de dados de referência e os dados preditos
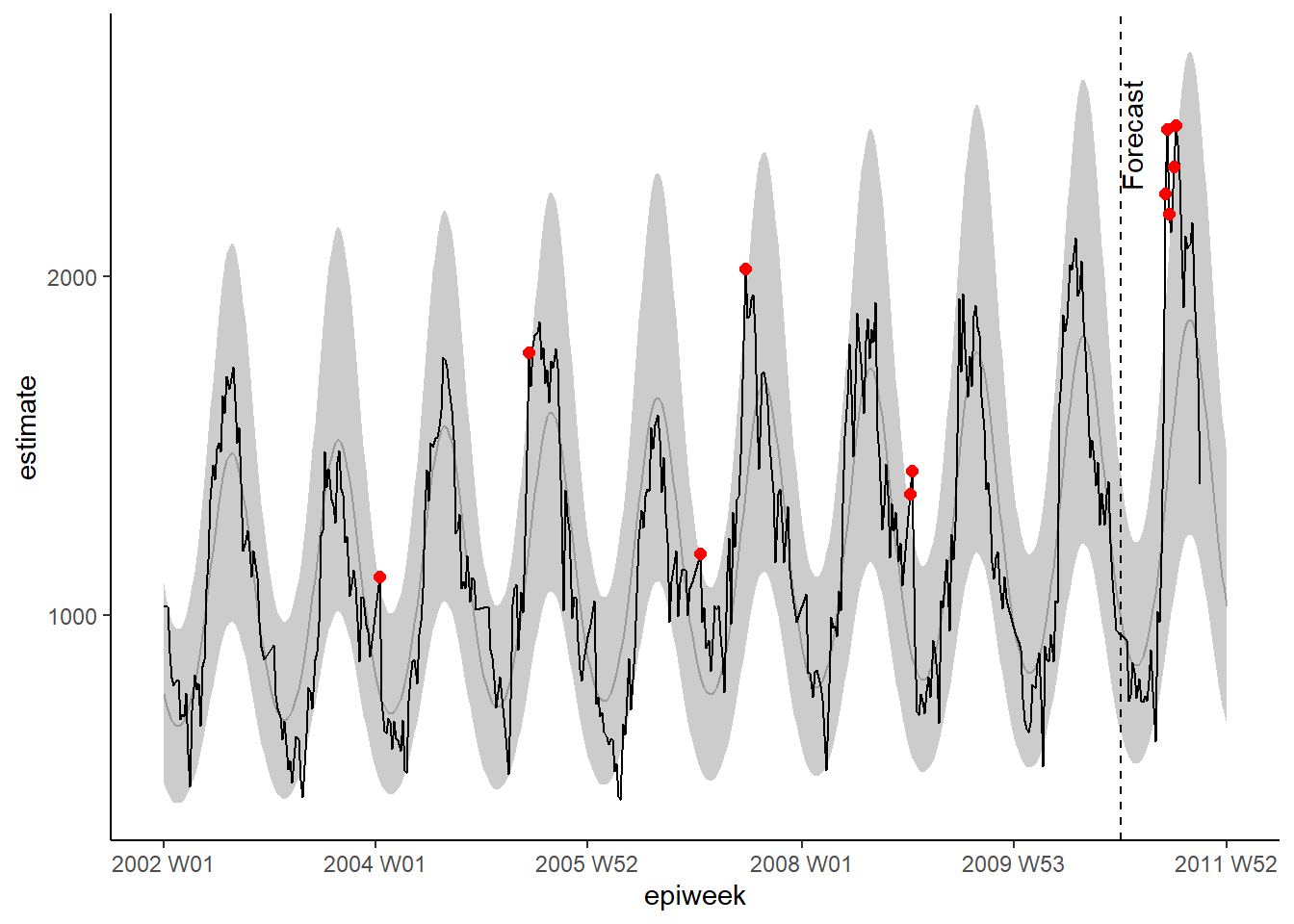
observed <- bind_rows(observed$result, forecasts$result)Como anteriormente, nós podemos visualizar nosso modelo com o ggplot. Nós destacamos os alertas de surtos com pontos vermelhos, para contagens observadas acima de 95% do intervalo de predição. Desta vez, nós também adicionamos uma linha vertical para indicar onde o período de previsão começa.
## faça gráficos da sua regressão
ggplot(data = observed, aes(x = epiweek)) +
## adicione uma linha para as estimativas do modelo
geom_line(aes(y = estimate),
col = "grey") +
## adicione uma faixa sombreada com os intervalos de predição
geom_ribbon(aes(ymin = lower_pi,
ymax = upper_pi),
alpha = 0.25) +
## adiciona uma linha para os as contagens de caso observadas
geom_line(aes(y = case_int),
col = "black") +
## crie pontos para as contagens observadas acima do esperado
geom_point(
data = filter(observed, case_int > upper_pi),
aes(y = case_int),
colour = "red",
size = 2) +
## adicione linha vertical e a nomeie para indicar onde a previsão dos casos começou
geom_vline(
xintercept = as.Date(cut_off),
linetype = "dashed") +
annotate(geom = "text",
label = "Forecast",
x = cut_off,
y = max(observed$upper_pi) - 250,
angle = 90,
vjust = 1
) +
## crie um gráfico tradicional (com eixos pretos e fundo branco)
theme_classic()Warning: Removed 13 rows containing missing values or values outside the scale range
(`geom_line()`).Validando a previsão do número de casos
Além de inspecionar os resíduos, é importante investigar o quão bom o seu modelo é em prever casos no futuro. Isto te dá uma ideia sobre o quão fidedignos os seus patamares para alertas são.
A forma tradicional de validar é verificar quão bem você consegue prever o último ano antes do ano atual (porque você ainda desconhece as contagens para o “ano corrente”). Por exemplo, em nosso conjunto de dados, nós iríamos utilizar os dados de 2002 a 2009 para prever 2010, e então ver o quão acuradas estas previsões são. A partir disto, reajustar o modelo para incluir dados de 2010 e usar para prever as contagens de 2011.
Como pode ser visto na figura abaixo, de Hyndman et al em “Princípios e práticas de previsão” .
 figura reproduzida com permissão dos autores
figura reproduzida com permissão dos autores
O ponto negativo desta abordagem é que você não está utilizando todos os dados disponíveis, além de não ser o modelo final o utilizado para realizar as previsões.
Uma alternativa é utilizar um método chamado de validação cruzada. Neste cenário, você passa por todos os dados disponíveis para ajustar diferentes modelos para prever um ano à frente. Você utiliza mais e mais dados em cada modelo, como visto na figura abaixo do mesmo [texto de Hyndman et al]((https://otexts.com/fpp3/). Por exemplo, o primeiro modelo utiliza dados de 2002 para prever 2003, o segundo usa dados de 2002 e 2003 para prever 2004, e assim por diante.  figura reproduzida com permissão dos autores
figura reproduzida com permissão dos autores
Abaixo, nós utilizamos a função map() do pacote purrr para passar por cada conjunto de dados. Nós então colocamos estimativas em um banco de dados, e unimos com o número de casos original, para utilizar o pacote yardstick para calcular medidas de acurácia. Nós calculamos quatro medidas, incluindo: We compute four measures including: erro quadrático médio (RMSE, do inglês Root mean squared error), erro médio absoluto
(MAE), erro médio absoluto em escala (MASE), erro médio percentual absoluto (MAPE).
CUIDADO: observe o uso do argumento simulate_pi = FALSE dentro de predict(). Isto é porque o comportamento padrão de trending é utilizar o pacote ciTools para estimar um intervalo de previsão. Isto não funciona caso existam contagens de NA, e também produz mais intervalos granulares. Veja ?trending::predict.trending_model_fit para detalhes.
## Validação cruzada: prevendo semana(s) à frente baseado na técnica da janela deslizante
## expanda seus dados, trabalhando com janelas de 52 semanas (antes + depois)
## para predizer as 52 semanas seguintes
## (cria correntes de observação mais e mais longas - mantém dados antigos)
## defina o tamanho da janela a ser trabalhada
roll_window <- 52
## define weeks ahead want to predict
weeks_ahead <- 52
## crie um conjunto de dados com dados repetindo e aumentando
## nomeie cada conjunto de dados com um id único
## utilize apenas os casos antes do ano de interesse (i.e.: 2011)
case_roll <- counts %>%
filter(epiweek < cut_off) %>%
## mantenha apenas as variáveis de semana e quantidade de casos
select(epiweek, case_int) %>%
## exclua as últimas x observações
## dependendo quantas semanas à frente estão sendo previstas
## (do contrário será uma previsão real para "desconhecida")
slice(1:(n() - weeks_ahead)) %>%
as_tsibble(index = epiweek) %>%
## role por cada semana após x janelas para para criar um ID de agrupamento
## de acordo com qual janela rolante especificar
stretch_tsibble(.init = roll_window, .step = 1) %>%
## exclua a primeira dupla - pois não existem casos "antes"
filter(.id > roll_window)
## para cada um dos conjuntos de dados, execute o código abaixo
forecasts <- purrr::map(unique(case_roll$.id),
function(i) {
## mantenha apenas a janela atual sendo ajustada no modelo
mini_data <- filter(case_roll, .id == i) %>%
as_tibble()
## crie um conjunto de dados vazio para fazer as previsões
forecast_data <- tibble(
epiweek = seq(max(mini_data$epiweek) + 1,
max(mini_data$epiweek) + weeks_ahead,
by = 1),
case_int = rep.int(NA, weeks_ahead),
.id = rep.int(i, weeks_ahead)
)
## adicione os dados previstos aos originais
mini_data <- bind_rows(mini_data, forecast_data)
## defina o limite de corte baseado nos últimos dados de contagem, com exceção dos dados em branco
cv_cut_off <- mini_data %>%
## mantenha apenas as linhas sem campos em branco
drop_na(case_int) %>%
## obtenha a última semana
summarise(max(epiweek)) %>%
## extraia os dados de forma que não estejam como quadro de dados
pull()
## crie a variável mini_data de volta como um tsibble
mini_data <- tsibble(mini_data, index = epiweek)
## defina os termos de fourier (sincos)
mini_data <- mini_data %>%
mutate(
## combine os termos de fourier para semanas antes e após a data limite
fourier = rbind(
## obtenha termos de fourier para anos anteriores
forecast::fourier(
## matenha as linhas até a data limite
filter(mini_data,
epiweek <= cv_cut_off),
## inclua um conjunto de termos seno e coseno
K = 1
),
## preveja os termos courier para o ano seguinte (utilizando os dados base de referências)
fourier(
## matenha apenas as linhas antes da data limite
filter(mini_data,
epiweek <= cv_cut_off),
## inclua um conjunto de termos seno e coseno
K = 1,
## preveja as 52 semanas à frente
h = weeks_ahead
)
)
)
# divida os dados para ajustar e prever
dat <- mini_data %>%
group_by(epiweek <= cv_cut_off) %>%
group_split()
## defina o modelo que você quer ajustar (negativo binominal)
model <- glm_nb_model(
## ajuste o número de casos como resultado de interesse
case_int ~
## utilize a epiweek para levar em consideração a tendência
epiweek +
## utilize os termos furier para levar em consideração a sazonalidade
fourier
)
# defina quais dados utilizar para ajustar e quais para prever
fitting_data <- pluck(dat, 2)
pred_data <- pluck(dat, 1)
# ajuste o modelo
fitted_model <- trending::fit(model, fitting_data)
# prejeva com os dados que você quer utilizar para prever
forecasts <- fitted_model %>%
predict(pred_data, simulate_pi = FALSE)
forecasts <- data.frame(forecasts$result[[1]]) %>%
## mantenha apenas a semana e os dados estimados
select(epiweek, estimate)
}
)
## transforme a lista gerada em um quadro de dados com todas as estimativas
forecasts <- bind_rows(forecasts)
## una as previsões com os dados observados
forecasts <- left_join(forecasts,
select(counts, epiweek, case_int),
by = "epiweek")
## utilize {yardstick} para calcular métricas
## RMSE: Erro quadrático médio
## MAE: Erro médio absoluto
## MASE: Erro médio absoluto escalonado
## MAPE: Erro médio absoluto percentual
model_metrics <- bind_rows(
## no seu conjunto de dados previstos, compare o observado com o estimado
rmse(forecasts, case_int, estimate),
mae( forecasts, case_int, estimate),
mase(forecasts, case_int, estimate),
mape(forecasts, case_int, estimate),
) %>%
## mantenha apenas o tipo métrico e o resultado
select(Metric = .metric,
Measure = .estimate) %>%
## crie no formato amplo de forma que seja possível unir as linhas posteriormente
pivot_wider(names_from = Metric, values_from = Measure)
## retorne as métricas do modelo
model_metrics# A tibble: 1 × 4
rmse mae mase mape
<dbl> <dbl> <dbl> <dbl>
1 252. 199. 1.96 17.3Pacote surveillance
Nesta seção, nós utilizamos o pacote surveillance para criar alertas de acordo com limites baseados em algorítmos de detecção de surtos. Existem diferentes métodos disponíveis no pacote. Entretanto, aqui nós iremos focar em duas opções. Para detalhes, veja esses artigos sobre a aplicação e teoria dos algorítmos utilizados.
A primeira opção utiliza o método Farrington melhorado. Isto ajusta uma glm binominal negativa (incluíndo tendências) e diminui o peso de surtos passados (pontos fora do normal) para criar um nível de limiar.
A segunda opção utiliza o método glrnb. Isto também ajusta uma glm binominal negativa mas inclui tendências e termos de fourier (então é a favorita aqui). A regressão é utilizada para calcular o “controle médio” (~valores ajustados) - ele então utiliza uma razão estatística generalizada de verosimilhança para avaliar se existe uma mudança na média de cada semana. Observe que o limiar para cada semana leva em conta semanas anteriores, então se as mudanças persistirem, um alarme será ativado. (Adicionalmente, observe que após cada alarme, o algoritmo é reinicilizado)
Para trabalhar com o pacote surveillance, nós primeiro precisamos definir um objeto de “séries temporais de vigilância” (utilizando a função sts()) para ajustar dentro do quadro.
## defina um objeto de série temporal de vigilância
## nb. você pode incluir um denominador com o objeto que contém a população (veja ?sts)
counts_sts <- sts(observed = counts$case_int[!is.na(counts$case_int)],
start = c(
## subconjunto para manter apenas o ano da variável start_date
as.numeric(str_sub(start_date, 1, 4)),
## subconjunto para manter apenas a semana da variável start_date
as.numeric(str_sub(start_date, 7, 8))),
## defina o tipo/frequência dos dados (neste caso, semanal)
freq = 52)
## defina o intervalo de semanas que você quer incluir (ex.: período de previsão)
## nb. o objto sts apenas contabiliza observações sem identificar uma semana ou
## ano a eles - assim, nós utilizamos nossos dados para definir as observações apropriadas
weekrange <- cut_off - start_dateMétodo Farrington
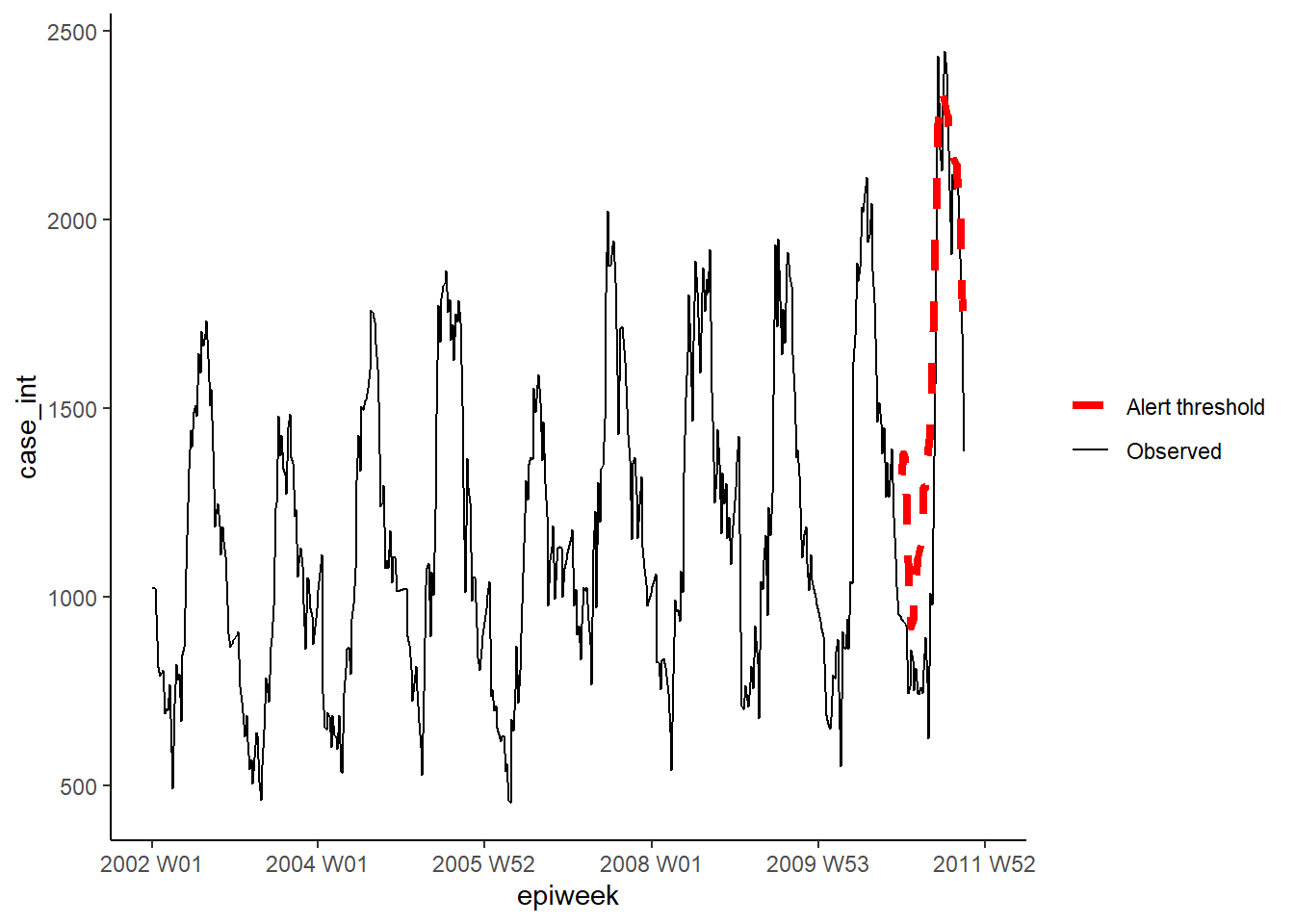
Nós então definimos cada um dos nossos parâmetros para o método Farrington em uma list (lista). Então, nós executamos o algoritmo utilizando a função farringtonFlexible() e então podemos extrair o limiar para um alerta utilizando farringtonmethod@upperbound para incluir isto em nosso banco de dados. Também é possível extrair um booleano TRUE/FALSE (VERDADEIRO/FALSO) para cada semana no caso do alerta ter sido ativado (semana com casos acima do limiar) utilizando farringtonmethod@alarm.
## defina o controle
ctrl <- list(
## defina qual período de tempo que quer utilizar o limiar (i.e.: 2011)
range = which(counts_sts@epoch > weekrange),
b = 9, ## quantos anos anteriores para considerar com os dados de referência
w = 2, ## tamanho da janela flutuante em semanas
weightsThreshold = 2.58, ## reponderando surtos passados (método melhorado de noufaily - método original sugere 1)
## pastWeeksNotIncluded = 3, ## utilize todas as semanas disponíveis (noufaily suggests drop 26)
trend = TRUE,
pThresholdTrend = 1, ## 0.05 normalmente, entretanto 1 é recomendado no método melhorado (i.e.: sempre mantenha)
thresholdMethod = "nbPlugin",
populationOffset = TRUE
)
## aplique o método flexível de farrington
farringtonmethod <- farringtonFlexible(counts_sts, ctrl)
## Crie uma nova variável no conjunto de dados original chamado "threshold"
## contendo o limite superior do farrington
## nb. isto é apenas para as semanas em 2011 (então é necessário subdividir as linhas)
counts[which(counts$epiweek >= cut_off &
!is.na(counts$case_int)),
"threshold"] <- farringtonmethod@upperboundNós podemos, então, visualizar os resultados no ggplot como feito anteriormente.
ggplot(counts, aes(x = epiweek)) +
## adicione as quantidades de casos observada como uma linha
geom_line(aes(y = case_int, colour = "Observed")) +
## adicione o limite superior do algoritmo de aberrações
geom_line(aes(y = threshold, colour = "Alert threshold"),
linetype = "dashed",
size = 1.5) +
## defina as cores
scale_colour_manual(values = c("Observed" = "black",
"Alert threshold" = "red")) +
## crie um gráfico tradicional (com eixos pretos e fundo branco)
theme_classic() +
## remova o título da legenda
theme(legend.title = element_blank())Método GLRNB
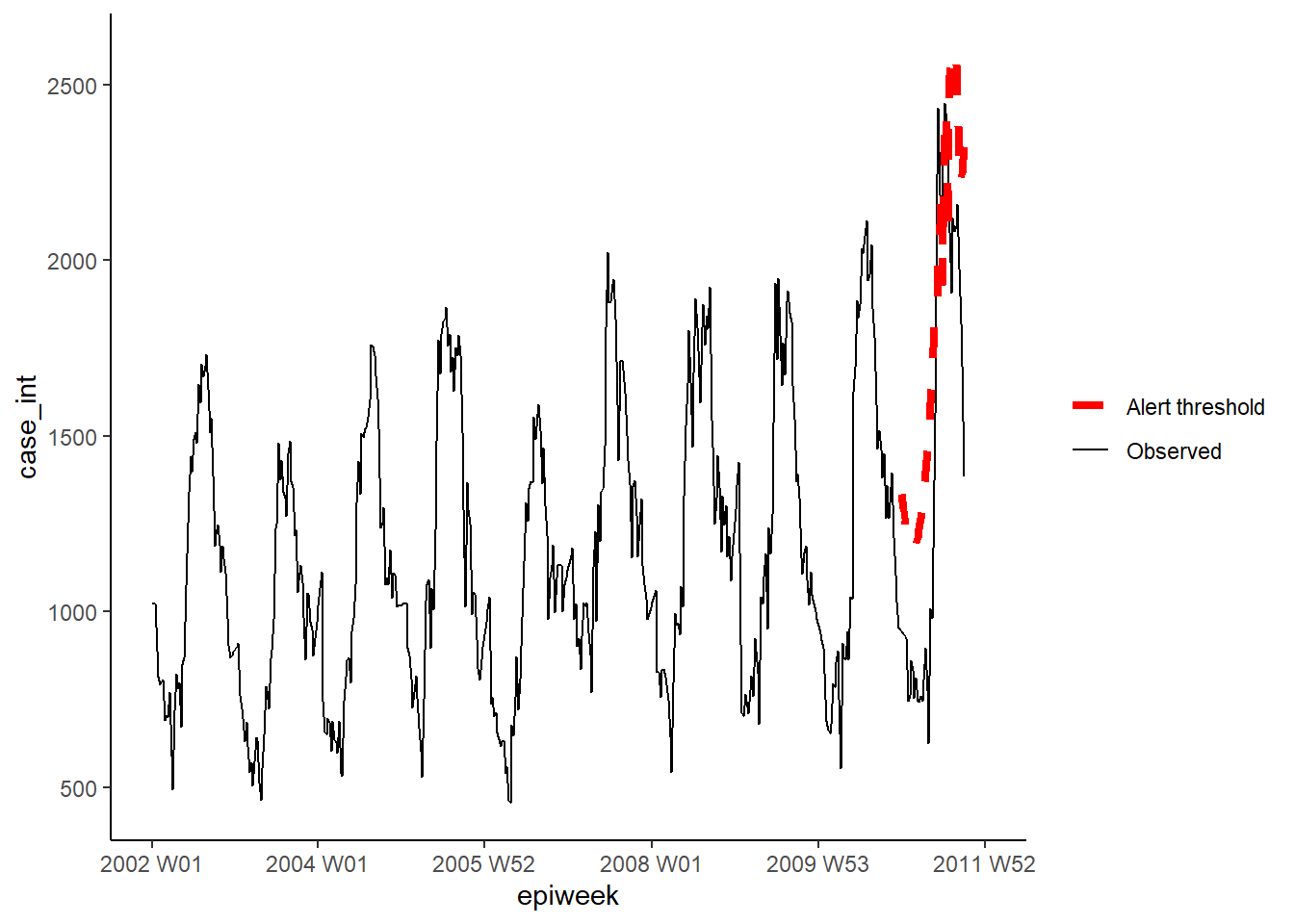
De forma similar, para o método GLRNB nós definimos cada um dos nossos parâmetros para em uma list (lista), então ajuste o algoritmo e extraia os limites superiores.
CUIDADO: Este método utiliza “força bruta” (similar ao bootstrapping) para calcular os limiares, então pode levar muito tempo!
Veja o tutorial sobre GLRNB para detalhes.
## defina as opções de controle
ctrl <- list(
## defina qual período de tempo que quer utilizar o limiar (i.e.: 2011)
range = which(counts_sts@epoch > weekrange),
mu0 = list(S = 1, ## número de termos de fourier (harmônicos) para incluir
trend = TRUE, ## incluir tendências ou não
refit = FALSE), ## reajustar o modelo após cada alarme ou não
## cARL = limiar para estatísticas GLR (arbitrário)
## 3 ~ meio-termo para minimizar os falsos positivos
## 1 ajusta para os 99% de intervalo de previsão do glm.nb - com mudanças após picos (limiar diminuído para alertas)
c.ARL = 2,
# theta = log(1.5), ## equivale a um aumento de 50% nos casos em um surto
ret = "cases" ## retorne os limites superiores como contagem de casos
)
## aplique o método glrnb
glrnbmethod <- glrnb(counts_sts, control = ctrl, verbose = FALSE)
## crie uma nova variável no conjunto de dados original chamada "threshold"
## contendo o limite superior do glrnb
## nb. isto é apenas para as semanas em 2011 (então é necessário subdividir as linhas)
counts[which(counts$epiweek >= cut_off &
!is.na(counts$case_int)),
"threshold_glrnb"] <- glrnbmethod@upperboundVisualize os resultados das análises como mostrado anteriormente.
ggplot(counts, aes(x = epiweek)) +
## adicione as quantidades de casos observadas como uma linha
geom_line(aes(y = case_int, colour = "Observed")) +
## adicione o limite superior do algoritmo de aberração
geom_line(aes(y = threshold_glrnb, colour = "Alert threshold"),
linetype = "dashed",
size = 1.5) +
## defina as cores
scale_colour_manual(values = c("Observed" = "black",
"Alert threshold" = "red")) +
## crie um gráfico tradicional (com eixos pretos e fundo branco)
theme_classic() +
## remova o título da legenda
theme(legend.title = element_blank())23.8 Séries temporais interrompidas
Séries temporais interrompidas (também chamadas de regressão segmentada ou análise de intervenção), são usadas frequentemente para avaliar o impacto de vacinas na incidência de doenças. Mas também pode ser utilizada para avaliar o impacto de uma variedade de intervenções ou introduções. Por exemplo, alterações nos procedimentos de um hopital, ou introdução de uma nova doença para a população. Neste exemplo, nós iremos pretender que uma nova cepa de Campylobacter foi introduzida na Alemanha no final de 2008, e ver se isso afeta o número de casos. Iremos utilizar a regressão binominal negativa novamente. Desta vez, a regressão será dividida em duas partes, uma antes da intervenção (ou introdução da nova cepa neste caso) e uma após (os períodos pré e pós intervenção/introdução). Isto nos permite calcular uma razão da taxa de incidência comparando os dois períodos de tempo. Explicar a equação pode clarear isso (se não, somente ignore!).
A regressão negativa binominal pode ser definida da seguinte forma:
\[\log(Y_t)= β_0 + β_1 \times t+ β_2 \times δ(t-t_0) + β_3\times(t-t_0 )^+ + log(pop_t) + e_t\]
Onde: \(Y_t\)é o número de casos observado naquele momento \(t\)
\(pop_t\) é o tamanho da população em 100,000s naquele momento \(t\) (não utilizado aqui)
\(t_0\) é o último ano do pré-período (incluindo o tempo de transição, se existir) \(δ(x\) é a função indicadora (será 0 se x≤0 e 1 se x>0)
\((x)^+\) é o operador de corte de acordo com o limite (é x se x>0 e 0 de outra forma)
\(e_t\) denota os resíduos Termos adicionais sobre tendência e sazonalidade podem ser adicionadas conforme necessário.
\(β_2 \times δ(t-t_0) + β_3\times(t-t_0 )^+\) é a parte linear generalizada do pós-período e é zero no pré-período. Isto significa que as estimativas \(β_2\) e \(β_3\) são efeitos da intervenção.
Nós precisamos recalcular os termos de fourier sem realizar a previsão aqui, uma vez que iremos utilizar todos os dados disponíveis para nós (i.e.: retrospectivamente). Adicionalmente, nós precisamos calcular os termos extras necessários para a regressão.
## adicione os termos de fourier utilizando as variáveis epiweek e case_int
counts$fourier <- select(counts, epiweek, case_int) %>%
as_tsibble(index = epiweek) %>%
fourier(K = 1)
## defina a semana de intervenção
intervention_week <- yearweek("2008-12-31")
## defina as variáveis para a regressão
counts <- counts %>%
mutate(
## corresponde ao t na fórmula
## contagem das semanas (provavelmente poderia também utilizar diretamente a variável epiweeks (semanas epidemiológicas))
# linear = row_number(epiweek),
## corresponde ao delta(t-t0) na fórmula
## período pré ou pós intervenção
intervention = as.numeric(epiweek >= intervention_week),
## corresponde a (t-t0)^+ na fórmula
## contagem de semenas após a intervenção
## (escolha o maior número entre 0 e o que resultar do cálculo)
time_post = pmax(0, epiweek - intervention_week + 1))Nós então utilizamos estes termos para ajustar uma regressão binominal negativa, e produz uma tabela com a mudança dos percentuais. O que este exemplo mostra é que não existe mudanças significativas.
CUIDADO: Observe o uso do argumento simulate_pi = FALSE dentro de predict(). Isto ocorre porque o comportamento padrão de trending é utilizar o pacote ciTools para estimar o intervalo de previsão. Isto não funciona se existirem contagens NA, e também produz mais intervalos granulares. Veja ?trending::predict.trending_model_fit para detalhes.
## defina o modelo que você quer ajustar (binominal negativo)
model <- glm_nb_model(
## defina o número de casos como resultado de interesse da análise
case_int ~
## utilize a epiweek para levar em consideração a tendência
epiweek +
## utilize os termos de fourier para levar em consideração a sazonalidade
fourier +
## adicione o clima nos pré- e pós- períodos
intervention +
## adicione o tempo após a intervenção
time_post
)
## ajuste seu modelo utilizando os dados de contagem
fitted_model <- trending::fit(model, counts)
## calcule os intervalos de confiança e de previsão
observed <- predict(fitted_model, simulate_pi = FALSE)## mostre as estimativas e mudanças percentuais em uma tabela
fitted_model %>%
## extraia a regressão original negativa binominal
get_result() %>%
## extrair o primeiro elemento da lista, consequência das alterações de pacotes de tendências.
simplify() %>%
first() %>%
## obtenha um quadro de dados organizado dos resultados
tidy(exponentiate = TRUE,
conf.int = TRUE) %>%
## mantenha apenas o valor da intervenção
filter(term == "intervention") %>%
## altere o IRR para diferença de porcentagem para estimativas e invervalos de confiança (CI)
mutate(
## para cada uma das colunas de interesse - crie uma uma nova coluna
across(
all_of(c("estimate", "conf.low", "conf.high")),
## aplique a fórmula para calcular a mudança de porcentagem
.f = function(i) 100 * (i - 1),
## adicione ao nome das novas colunas o sufixo "_perc"
.names = "{.col}_perc")
) %>%
## apenas mantenha (e renomeie) certas colunas
select("IRR" = estimate,
"95%CI low" = conf.low,
"95%CI high" = conf.high,
"Percentage change" = estimate_perc,
"95%CI low (perc)" = conf.low_perc,
"95%CI high (perc)" = conf.high_perc,
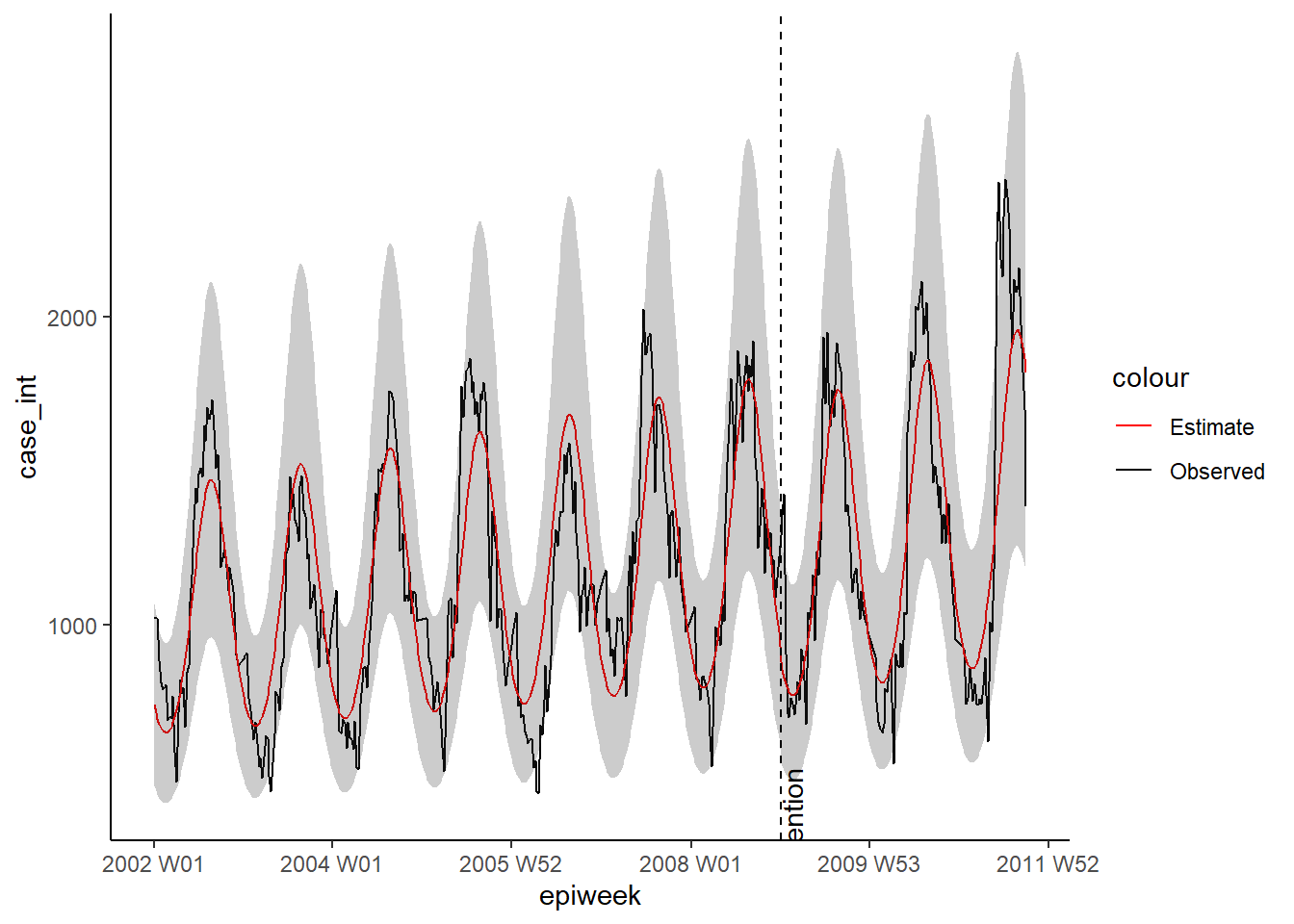
"p-value" = p.value)Como anteriormente, nós podemos visualizar os resultados da regressão.
estimate_res <- data.frame(observed$result)
ggplot(estimate_res, aes(x = epiweek)) +
## adicione as quantidades de casos observadas como uma linha
geom_line(aes(y = case_int, colour = "Observed")) +
## adicione uma linha para a estimativa do modelo
geom_line(aes(y = estimate, col = "Estimate")) +
## adicione uma banda para os intervalos de previsão
geom_ribbon(aes(ymin = lower_pi,
ymax = upper_pi),
alpha = 0.25) +
## adicione uma linha vertical e uma etiqueta para mostrar onde a previsão começou
geom_vline(
xintercept = as.Date(intervention_week),
linetype = "dashed") +
annotate(geom = "text",
label = "Intervention",
x = intervention_week,
y = max(observed$upper_pi),
angle = 90,
vjust = 1
) +
## defina as cores
scale_colour_manual(values = c("Observed" = "black",
"Estimate" = "red")) +
## faça um gráfico tradicional (com eixos pretos e fundo branco)
theme_classic()Warning: Unknown or uninitialised column: `upper_pi`.Warning in max(observed$upper_pi): no non-missing arguments to max; returning
-Inf23.9 Recursos
previsões: texto sobre princípios e práticas
Estudos de caso EPIET sobre séries temporais
Curso da Penn State Artigo sobre o pacote surveillance