8 Limpeza de dados e principais funções
Esta página demonstra passos comuns usados no processo de “limpeza” de um conjunto de dados, e também explica o uso de muitas funções essenciais de gerenciamento de dados.
Para demonstrar a limpeza de dados, esta página começa por importar um conjunto de dados brutos de uma lista de casos, e realiza passo-a-passo o processo de limpeza. No código R, isto se manifesta com uma cadeia “pipe”, a qual faz referência ao operador “pipe” %>%` que passa um conjunto de dados de uma operação para a próxima.
Funções essenciais
Este manual emfatiza a utilização das funções da família de pacotes tidyversedo R. As funções essenciais do R demonstradas nesta página estão listadas abaixo.
Muitas destas funções pertencem ao pacote do R dplyr, que fornece funções “verbo” para resolver desafios de manipulação de dados (o nome é uma referência a um “dataframe-plier (N.T.: plier significa alicate, fazendo uma analogia da funcionalidade dessa ferramenta sobre um dataframe). O pacote dplyr é parte da família de pacotes tidyverse do R (os quais também incluem ggplot2, tidyr, stringr, tibble, purrr, magrittr, e forcats entre outros).
| Função | Utilidade | Pacote | |
|---|---|---|---|
%>% |
“pipe” (passar) dados de uma função para a próxima | magrittr | ||
mutate() |
criar, transformar, e redefinir colunas | dplyr | |
select() |
manter, remover, selecionar, ou renomear colunas | dplyr | |
rename() |
renomear colunas | dplyr | |
clean_names() |
padronizar a síntaxe de nomes de colunas | janitor | |
as.character(), as.numeric(), as.Date(), etc. |
converter a classe de uma coluna | R base | |
across() |
transformar múltiplas colunas de uma vez | dplyr | |
| Funções tidyselect | use lógica para selecionar colunas | tidyselect | |
filter() |
manter certas linhas | dplyr | |
distinct() |
remover linhas duplicadas (duplicidades) | dplyr | |
rowwise() |
operações por/em cada linha | dplyr | |
add_row() |
adicionar linhas manualmente | tibble | |
arrange() |
ordenar linhas | dplyr | |
recode() |
recodificar valores em uma coluna | dplyr | |
case_when() |
recodificar valores em uma coluna usando critérios lógicos mais complexos | dplyr | |
replace_na(), na_if(), coalesce() |
funções especiais de recodificação | tidyr | |
age_categories() and cut() |
criar grupos categóricos de uma coluna numérica | epikit e R base | |
match_df() |
recodificar/limpar valores usando um dicionário de dados | matchmaker | |
which() |
aplicar critérios lógicos; retorna índices | R base | |
Se quiser ver como estas funções se comparam aos comandos Stata ou SAS, consulte a página em Transição para o R.
Você poderá encontrar uma estrutura alternativa de gestão de dados a partir do pacote do R data.table com operadores como := e utilização frequente de colchetes [ ]. Estas abordagem e sintaxe são brevemente explicadas na página Tabela de dados.
Nomenclatura
Neste manual, utilizamos os termos “colunas” e “linhas” em vez de “variáveis” e “observações”. Como explicado neste manual sobre “dados arrumados”, a maioria dos conjuntos de dados estatísticos epidemiológicos consistem estruturalmente em linhas, colunas e valores.
Variáveis contêm os valores que medem o mesmo atributo subjacente (como o faixa-etária, desfecho, ou data de início). Observações contêm todos os valores medidos na mesma unidade (por exemplo, uma pessoa, local, ou amostra de laboratório). Portanto, estes aspectos podem ser mais difíceis de definir de forma tangível.
Em um conjunto de dados “arrumados”, cada coluna é uma variável, cada linha é uma observação, e cada célula é um único valor. Contudo, alguns conjuntos de dados que você encontra não se enquadrarão neste molde - um conjunto de dados de formato “amplo” (wide) pode ter uma variável dividida em várias colunas (ver um exemplo na página Pivotando Dados). Da mesma forma, as observações podem ser divididas em várias linhas.
A maior parte deste manual trata da gestão e transformação de dados, referindo assim à estrutura concreta dos dados de linhas e colunas é mais relevante do que utilizar os termos observações/variáveis, que são mais abstratos. As exceções ocorrem principalmente em páginas sobre análise de dados, onde se verá mais referências a variáveis e observações.
8.1 Conduta de limpeza
Esta página prossegue através das típicas etapas de limpeza, adicionando-as sequencialmente a uma cadeia de pipe de limpeza.
Na análise epidemiológica e processamento de dados, as etapas de limpeza são frequentemente executadas sequencialmente, ligadas entre si. No R, isto manifesta-se frequentemente como um “pipeline” de limpeza, onde o conjunto de dados bruto é passado ou “canalizado” de uma etapa de limpeza para outra.
Tais cadeias utilizam funções “verbo” dplyr e o operador pipe ‘%>%’ do magrittr. Esta pipe começa com os dados “brutos” (“linelist_raw.xlsx”) e termina com um data frame de R “limpo” (linelist) que pode ser utilizada, guardada, exportada, etc.
Em uma cadeia de limpeza, a ordem das etapas é importante. As etapas de limpeza podem incluir:
- Importação de dados
- Nomes de colunas limpos ou alterados
- Remoção de duplicidades
- Criação e transformação de colunas (por exemplo, re-codificação ou normalização de valores)
- Linhas filtradas ou adicionadas
8.2 Carregar pacotes
Este pedaço de código mostra o carregamento das pacotes necessários para as análises. Neste manual, damos ênfase a p_load() do pacman, que instala o pacote se necessário e carrega-o para uso. Você pode também carregar pacotes instalados com library() do R base. Veja a página do Introdução ao R para mais informações do pacotes do R.
pacman::p_load(
rio, # importação de dados
here, # caminhos de arquicos relacionados
janitor, # limpeza de dados e tabelas
lubridate, # trabalhando com datas
matchmaker, # limpeza baseada no dicionário
epikit, # funções de age_categories()
tidyverse, # manejo e visualização de dados
skimr
)8.3 Importação de dados
Importação
Aqui nós importamos o arquivo de Excel linelist de casos “brutos” usando a função import() do pacote rio. O pacote rio trata de forma flexível muitos tipos de arquivos (por ex. .xlsx, .csv, .tsv, .rds. Ver a página em Importar e exportar para mais informações e dicas sobre situações não usuais (por exemplo, saltar linhas, definir valores em falta, importar Google sheets, etc.)
Se quiser acompanhar, clique para descarregar a linelist “bruta” (como arquivo .xlsx).
Se o seu conjunto de dados for grande e demorar muito tempo a importar, pode ser útil que o comando de importação seja separado da cadeia pipe e que o “bruto” seja guardado como um arquivo distinto. Isto também permite uma comparação fácil entre a versão original e a versão limpa.
Abaixo, importamos o arquivo bruto do Excel e o salvamos como o dataframe linelist_raw. Presumimos que o arquivo esteja localizado no diretório de trabalho ou na raiz do projeto R e, portanto, nenhuma subpasta é especificada no caminho do arquivo.
linelist_raw <- import("linelist_raw.xlsx")Você pode visualizar as primeiras 50 linhas do quadro de dados abaixo. Nota: a função contida no pacote R base head(n) permite que você visualize apenas as primeiras n linhas no console do R.
Revisão
Você pode usar a função skim() do pacote skimr para obter uma visão geral de todo o dataframe (ver página em Tabelas descritivas para mais informações). As colunas são resumidas por classe/tipo, como caractere, numérico. Nota: “POSIXct” é um tipo de classe de data bruta (ver Trabalhando com datas).
skimr::skim(linelist_raw)| Name | linelist_raw |
| Number of rows | 6611 |
| Number of columns | 28 |
| _______________________ | |
| Column type frequency: | |
| character | 17 |
| numeric | 8 |
| POSIXct | 3 |
| ________________________ | |
| Group variables | None |
Variable type: character
| skim_variable | n_missing | complete_rate | min | max | empty | n_unique | whitespace |
|---|---|---|---|---|---|---|---|
| case_id | 137 | 0.98 | 6 | 6 | 0 | 5888 | 0 |
| date onset | 293 | 0.96 | 10 | 10 | 0 | 580 | 0 |
| outcome | 1500 | 0.77 | 5 | 7 | 0 | 2 | 0 |
| gender | 324 | 0.95 | 1 | 1 | 0 | 2 | 0 |
| hospital | 1512 | 0.77 | 5 | 36 | 0 | 13 | 0 |
| infector | 2323 | 0.65 | 6 | 6 | 0 | 2697 | 0 |
| source | 2323 | 0.65 | 5 | 7 | 0 | 2 | 0 |
| age | 107 | 0.98 | 1 | 2 | 0 | 75 | 0 |
| age_unit | 7 | 1.00 | 5 | 6 | 0 | 2 | 0 |
| fever | 258 | 0.96 | 2 | 3 | 0 | 2 | 0 |
| chills | 258 | 0.96 | 2 | 3 | 0 | 2 | 0 |
| cough | 258 | 0.96 | 2 | 3 | 0 | 2 | 0 |
| aches | 258 | 0.96 | 2 | 3 | 0 | 2 | 0 |
| vomit | 258 | 0.96 | 2 | 3 | 0 | 2 | 0 |
| time_admission | 844 | 0.87 | 5 | 5 | 0 | 1091 | 0 |
| merged_header | 0 | 1.00 | 1 | 1 | 0 | 1 | 0 |
| …28 | 0 | 1.00 | 1 | 1 | 0 | 1 | 0 |
Variable type: numeric
| skim_variable | n_missing | complete_rate | mean | sd | p0 | p25 | p50 | p75 | p100 | hist |
|---|---|---|---|---|---|---|---|---|---|---|
| generation | 7 | 1.00 | 16.60 | 5.71 | 0.00 | 13.00 | 16.00 | 20.00 | 37.00 | ▁▆▇▂▁ |
| lon | 7 | 1.00 | -13.23 | 0.02 | -13.27 | -13.25 | -13.23 | -13.22 | -13.21 | ▅▃▃▅▇ |
| lat | 7 | 1.00 | 8.47 | 0.01 | 8.45 | 8.46 | 8.47 | 8.48 | 8.49 | ▅▇▇▇▆ |
| row_num | 0 | 1.00 | 3240.91 | 1857.83 | 1.00 | 1647.50 | 3241.00 | 4836.50 | 6481.00 | ▇▇▇▇▇ |
| wt_kg | 7 | 1.00 | 52.69 | 18.59 | -11.00 | 41.00 | 54.00 | 66.00 | 111.00 | ▁▃▇▅▁ |
| ht_cm | 7 | 1.00 | 125.25 | 49.57 | 4.00 | 91.00 | 130.00 | 159.00 | 295.00 | ▂▅▇▂▁ |
| ct_blood | 7 | 1.00 | 21.26 | 1.67 | 16.00 | 20.00 | 22.00 | 22.00 | 26.00 | ▁▃▇▃▁ |
| temp | 158 | 0.98 | 38.60 | 0.95 | 35.20 | 38.30 | 38.80 | 39.20 | 40.80 | ▁▂▂▇▁ |
Variable type: POSIXct
| skim_variable | n_missing | complete_rate | min | max | median | n_unique |
|---|---|---|---|---|---|---|
| infection date | 2322 | 0.65 | 2012-04-09 | 2015-04-27 | 2014-10-04 | 538 |
| hosp date | 7 | 1.00 | 2012-04-20 | 2015-04-30 | 2014-10-15 | 570 |
| date_of_outcome | 1068 | 0.84 | 2012-05-14 | 2015-06-04 | 2014-10-26 | 575 |
8.4 Nomes de colunas
No R, os nomes das colunas são o “cabeçalho” ou valor do “topo” de uma coluna. Eles são usados para referir a colunas em código, e serve como um rotulo por omissão nas figuras.
Outros softwares estatísticos como SAS e STATA utilizam “etiquetas” que coexistem como versões impressas mais longas dos nomes das colunas mais curtas. Embora o R ofereça a possibilidade de adicionar etiquetas de coluna aos dados, isto não é enfatizado na maioria das práticas. Para que os nomes das colunas sejam “amigáveis para impressão” de figuras, normalmente se ajusta sua exibição dentro dos próprios comandos de criação dos gráficos e tabelas (por exemplo, títulos de eixos ou legendas de um gráfico, ou cabeçalhos de coluna em uma tabela impressa - veja a seção de escalas da página de dicas ggplot e páginas de Tabelas para apresentação). Se você quiser atribuir etiquetas de coluna nos dados, leia mais online aqui e aqui.
Como os nomes de colunas no R são usados muito frequentemente, então eles devem ter uma sintaxe “limpa”. Nós sugerimos o seguinte:
- Nomes curtos
- Sem espaços (substituir com sublinhados _ )
- Sem caracteres especiais (&, #, <, >, …)
- Nomenclatura de estilo similar (por exemplo, todas as colunas de datas nomeadas como data_início, data_relato, data_morte…)
Os nomes das colunas de linelist_raw são “printados” abaixo utilizando names() do R base. Podemos ver isso inicialmente:
- Alguns nomes contêm espaços (por exemplo a data de infeccção
infecction date)
- Diferentes padrões de nomes são utilizados para datas (data de início vs data de infecção,
date onsetvs.infecction date)
- Deve ter havido um cabeçalho combinado nas duas últimas colunas no .xlsx. Sabemos disso porque o nome de duas colunas combinadas (“merged_header”) foi atribuído pelo R à primeira coluna, e à segunda coluna foi atribuído um nome de espaço reservado “…28” (pois estava então vazia e é a 28ª coluna).
names(linelist_raw) [1] "case_id" "generation" "infection date" "date onset"
[5] "hosp date" "date_of_outcome" "outcome" "gender"
[9] "hospital" "lon" "lat" "infector"
[13] "source" "age" "age_unit" "row_num"
[17] "wt_kg" "ht_cm" "ct_blood" "fever"
[21] "chills" "cough" "aches" "vomit"
[25] "temp" "time_admission" "merged_header" "...28" NOTA: Para referenciar um nome de coluna que inclua espaços, rodeie o nome com o acentro grave (N.T. chamado vulgarmente de “crase”), por exemplo: linelist$` '\x60infection date\x60'`. Note que no seu teclado, o acento grave (`) é diferente das aspas simples/apóstrofe (’).
Limpeza automática
A função clean_names() do pacote janitor padroniza os nomes das colunas e os torna únicos ao fazer o seguinte:
- Converte todos os nomes para consistir apenas em sublinhados, números e letras
- Os caracteres acentuados são transliterados para ASCII (por exemplo, german o com umlaut torna-se “o”, “enye” espanhol torna-se “n”)
- A preferência de capitalização para os nomes das novas colunas pode ser especificada utilizando o argumento
case =argumento (“snake” é padrão, as alternativas incluem “sentence”, “title”, “small_camel”…)
- Você pode especificar substituições específicas de nomes fornecendo um vetor para o argumento
replace =argumento (por exemplo,replace = c(onset = "date_of_onset"))
- Aqui está uma vinheta online.
A seguir, a linha de limpeza começa utilizando clean_names() na linelist bruta.
# pipe do conjunto de dados brutos através da função clean_names(), atribuindo o resultado como "linelist"
linelist <- linelist_raw %>%
janitor::clean_names()
# Veja os novos nomes das colunas
names(linelist) [1] "case_id" "generation" "infection_date" "date_onset"
[5] "hosp_date" "date_of_outcome" "outcome" "gender"
[9] "hospital" "lon" "lat" "infector"
[13] "source" "age" "age_unit" "row_num"
[17] "wt_kg" "ht_cm" "ct_blood" "fever"
[21] "chills" "cough" "aches" "vomit"
[25] "temp" "time_admission" "merged_header" "x28" NOTA: O nome da última coluna “…28” foi mudado para “x28”.
Limpeza manual de nomes
A renomeação manual das colunas é frequentemente necessária, mesmo após a etapa de padronização acima. Abaixo, a renomeação é realizada utilizando a função rename() do pacote dplyr, como parte de uma cadeia pipe. A função rename() utiliza o estilo NOVO = VELHO - o novo nome da coluna é dado antes do nome da coluna antiga.
Abaixo, um comando de renomeação é adicionado a linha de pipe limpa. Espaços foram adicionados estrategicamente para alinhar o código para facilitar a leitura.
# CADEIA 'PIPE' DE LIMPEZA
# (inicia com dados brutos e encadeia funções com o pipe até a limpeza dos dados)
##################################################################################
linelist <- linelist_raw %>%
# padronização da sintaxe do nome de coluna
janitor::clean_names() %>%
# colunas renomeadas manualmente
# NOVO nome # nome VELHO
rename(date_infection = infection_date,
date_hospitalisation = hosp_date,
date_outcome = date_of_outcome)Agora você pode ver que os nomes das colunas está sendo mudado:
[1] "case_id" "generation" "date_infection"
[4] "date_onset" "date_hospitalisation" "date_outcome"
[7] "outcome" "gender" "hospital"
[10] "lon" "lat" "infector"
[13] "source" "age" "age_unit"
[16] "row_num" "wt_kg" "ht_cm"
[19] "ct_blood" "fever" "chills"
[22] "cough" "aches" "vomit"
[25] "temp" "time_admission" "merged_header"
[28] "x28" Renomear a posição da coluna
Você pode também renomear pela posição da coluna, ao invés do nome da coluna, por exemplo:
rename(newNameForFirstColumn = 1,
newNameForSecondColumn = 2)Renomear via select() e summarise()
Como um atalho, você também pode renomear colunas dentro das funções dplyr select() e summarise(). select() é utilizada para manter apenas certas colunas (e é abordada mais adiante nesta página). summarise() é coberta nas páginas Agrupamento de dados e Tabelas descritivas. Estas funções também utilizam o formato novo_nome = antigo_nome. Aqui está um exemplo:
linelist_raw %>%
select(# NOVO nome # nome VELHO
date_infection = `infection date`, # renomear e MANTER APENAS estas colunas
date_hospitalisation = `hosp date`)Outros desafios
Nomes de colunas Excel vazias
O R não pode ter colunas de conjuntos de dados que não tenham nomes de colunas (cabeçalhos). Portanto, se você importar um conjunto de dados Excel com dados mas sem cabeçalhos de colunas, o R preencherá os cabeçalhos com nomes como “…1” ou “…2”. O número representa o número da coluna (por exemplo, se a 4ª coluna do conjunto de dados não tiver cabeçalho, então R lhe dará o nome “…4”).
Você pode limpar estes nomes manualmente referenciando o número de posição (ver exemplo acima), ou seu nome atribuído (linelist_raw$...1).
Nomes de colunas e células de Excel combinado
As células combinadas em um arquivo Excel são uma ocorrência comum no recebimento de dados. Como explicado em Transição para R, células combinadas podem ser agradáveis para a leitura humana de dados, mas não são “dados arrumados” e causam muitos problemas para a leitura de dados pela máquina. O R não pode acomodar células combinadas.
Lembre as pessoas que fazem a entrada de dados que dados legíveis por humanos não é o mesmo que dados legíveis por máquinas. Esforce-se para treinar os usuários sobre os princípios de dados arrumados. Se possível, tente mudar os procedimentos para que os dados cheguem em um formato ordenado sem células combinadas.
- Cada variável deve ter sua própria coluna.
- Cada observação deve ter sua própria linha.
- Cada valor deve ter sua própria célula.
Ao utilizar a função import() do rio, o valor em uma célula mesclada será atribuído à primeira célula e as células subsequentes estarão vazias.
Uma solução para lidar com células mescladas é importar os dados com a função readWorkbook() do pacote openxlsx. Defina o argumento fillMergedCells = TRUE. Isto dá o valor em uma célula mesclada a todas as células dentro da faixa mesclada.
linelist_raw <- openxlsx::readWorkbook("linelist_raw.xlsx", fillMergedCells = TRUE)ATENÇÃO: Se os nomes das colunas forem mesclados com readWorkbook(), você terminará com nomes de colunas duplicadas, que você precisará corrigir manualmente - o R não funciona bem com nomes de colunas duplicadas! Você pode renomeá-los referenciando sua posição (por exemplo, coluna 5), como explicado na seção sobre limpeza manual de nomes de colunas
8.5 Selecionar ou reordenar colunas
Utilize select() do dplyr para selecionar as colunas que você deseja reter e especificar sua ordem no quadro de dados,
CUIDADO: Nos exemplos abaixo, o quadro de dados linelist é modificado com select() e exibido, mas não salvo. Isto é para fins de demonstração. Os nomes das colunas modificadas são exibidos ao fornecer banco de dados com o pipe para função names().
Aqui estão TODOS os nomes que a linelist tem até este ponto da cadeia de limpeza:
names(linelist) [1] "case_id" "generation" "date_infection"
[4] "date_onset" "date_hospitalisation" "date_outcome"
[7] "outcome" "gender" "hospital"
[10] "lon" "lat" "infector"
[13] "source" "age" "age_unit"
[16] "row_num" "wt_kg" "ht_cm"
[19] "ct_blood" "fever" "chills"
[22] "cough" "aches" "vomit"
[25] "temp" "time_admission" "merged_header"
[28] "x28" Manter colunas
Selecione apenas as colunas que você quer manter
Coloque seus nomes no comando select(), sem aspas. Eles aparecerão no data frame na ordem que você fornecer. Observe que se você incluir uma coluna que não existe, o R retornará um erro (veja o uso de any_of() abaixo se você não quiser nenhum erro nesta situação).
# O conjunto de dados linelist é canalisado através do comando select(), e names() "printam" apenas os nomes das colunas
linelist %>%
select(case_id, date_onset, date_hospitalisation, fever) %>%
names() # exibir os nomes das colunas[1] "case_id" "date_onset" "date_hospitalisation"
[4] "fever" Funções auxiliares “tidyselect”
Estas funções de ajuda existem para facilitar a especificação de colunas para manter, descartar ou transformar. Elas são do pacote tidyselect, que está incluído no tidyverse e está subjacente à forma como as colunas são selecionadas nas funções dplyr.
Por exemplo, se você quiser reordenar as colunas, everything() é uma função útil para significar “todas as outras colunas ainda não mencionadas”. O comando abaixo move as colunas date_onset e date_hospitalisation para o início (esquerda) do conjunto de dados, mas mantém todas as outras colunas depois. Note que everything() é escrito com parênteses vazios:
# mova date_onset e date_hospitalisation para o início
linelist %>%
select(date_onset, date_hospitalisation, everything()) %>%
names() [1] "date_onset" "date_hospitalisation" "case_id"
[4] "generation" "date_infection" "date_outcome"
[7] "outcome" "gender" "hospital"
[10] "lon" "lat" "infector"
[13] "source" "age" "age_unit"
[16] "row_num" "wt_kg" "ht_cm"
[19] "ct_blood" "fever" "chills"
[22] "cough" "aches" "vomit"
[25] "temp" "time_admission" "merged_header"
[28] "x28" Aqui estão outras funções auxiliares “tidyselect” que também trabalham dentro das funções dplyr como select(), across(), e summarise():
everything()- todas as outras colunas não mencionadas
last_col()- a última coluna
where()- aplica uma função a todas as colunas e seleciona aquelas para as quais a função retorna TRUE (verdadeiro)
contains()- colunas contendo uma cadeia de caracteres- exemplo:
select(contains("time"))
- exemplo:
starts_with()- corresponde a um prefixo especificado- exemplo:
select(starts_with("date_"))
- exemplo:
ends_with()- corresponde a um sufixo especificado- exemplo:
select(ends_with("_post"))
- exemplo:
matches()- para aplicar uma expressão regular (regex)- exemplo:
select(matches("[pt]al"))
- exemplo:
num_range()- uma faixa numérica como x01, x02, x03
any_of()- faz a correspondência SE a coluna existir, mas não retorna nenhum erro se não for encontrada- exemplo:
select(any_of(date_onset, date_death, cardiac_arrest))
- exemplo:
Além disso, utilizar operadores normais como c() para listar várias colunas, : para colunas consecutivas, ! para o oposto, & para AND, e | para OR.
Utilize where() para especificar critérios lógicos para as colunas. Se fornecer uma função dentro de where(), não inclua os parênteses vazios da função. O comando abaixo seleciona colunas que são de classe Numérica.
# selecione colunas que são a classe numérica
linelist %>%
select(where(is.numeric)) %>%
names()[1] "generation" "lon" "lat" "row_num" "wt_kg"
[6] "ht_cm" "ct_blood" "temp" Utilize contains() para selecionar apenas colunas nas quais o nome da coluna contém uma cadeia de caracteres especificada. Os ends_with() e starts_with() fornecem mais nuances.
# selecione colunas contendo certos caracteres
linelist %>%
select(contains("date")) %>%
names()[1] "date_infection" "date_onset" "date_hospitalisation"
[4] "date_outcome" A função matches() funciona de forma semelhante a contains() mas pode ser fornecida uma expressão regular (ver página em Caracteres e strings), tais como múltiplos strings separados por “barras de OU” dentro dos parênteses:
# procurado por combinações de caracteres múltiplos
linelist %>%
select(matches("onset|hosp|fev")) %>% # note o símbolo OR "|"
names()[1] "date_onset" "date_hospitalisation" "hospital"
[4] "fever" CUIDADO: Se um nome de coluna que você fornecer especificamente não existir nos dados, ele pode retornar um erro e parar seu código. Considere utilizar any_of() para citar colunas que podem ou não existir, especialmente úteis em seleções negativas (remoção).
Apenas uma destas colunas existe, mas nenhum erro é produzido e o código continua sem parar sua cadeia de limpeza.
linelist %>%
select(any_of(c("date_onset", "village_origin", "village_detection", "village_residence", "village_travel"))) %>%
names()[1] "date_onset"Remova colunas
Indicar quais colunas devem ser removidas colocando um símbolo menos “-” na frente do nome da coluna (por exemplo,select(-outcome)), ou um vetor de nomes de colunas (como abaixo). Todas as outras colunas serão retidas.
linelist %>%
select(-c(date_onset, fever:vomit)) %>% # remova date_onset e todas as colunas de febre a vômito
names() [1] "case_id" "generation" "date_infection"
[4] "date_hospitalisation" "date_outcome" "outcome"
[7] "gender" "hospital" "lon"
[10] "lat" "infector" "source"
[13] "age" "age_unit" "row_num"
[16] "wt_kg" "ht_cm" "ct_blood"
[19] "temp" "time_admission" "merged_header"
[22] "x28" Você também pode remover uma coluna utilizando a síntaxe do R base, definindo-a como NULL. Por exemplo:
linelist$date_onset <- NULL # elimina a coluna com a síntaxe da base do R Autonomia
select() também pode ser utilizado como um comando independente (não em uma cadeia de pipe). Neste caso, o primeiro argumento é o dataframe original a ser operado.
# Criar uma nova linelist com colunas relacionadas a id e idade
linelist_age <- select(linelist, case_id, contains("age"))
# exiba os nomes das colunas
names(linelist_age)[1] "case_id" "age" "age_unit"Acrescente à cadeia de pipes
Na linelist_raw, há algumas colunas que não precisamos: row_num, merged_header, e x28. Nós as removemos com um comando select() na cadeia pipe limpa:
# CADEIA 'PIPE' DE LIMPEZA
#(inicia com dados brutos e encadeia funções com o pipe até a limpeza dos dados)
##################################################################################
# inicie a cadeia pipe de limpeza
###########################
linelist <- linelist_raw %>%
# padronize a sintaxe do nome da coluna
janitor::clean_names() %>%
# renomeie manualmente colunas
# NOVO nome # nome VELHO
rename(date_infection = infection_date,
date_hospitalisation = hosp_date,
date_outcome = date_of_outcome) %>%
# ACIMA ESTÃO OS PASSOS DE LIMPEZA JÁ DISCUTIDOS
#####################################################
# remova coluna
select(-c(row_num, merged_header, x28))8.6 Eliminação de duplicidades
Consulte a página do manual de Eliminação de duplicidades para obter opções extensivas sobre como de-duplicar os dados. Apenas um exemplo muito simples de eliminação de duplicidades é apresentado aqui.
O pacotedplyr oferece a função distinct() function. Esta função examina cada linha e reduz o data frame para apenas as linhas únicas. Isto é, ele remove as linhas que são 100% duplicadas.
Ao avaliar linhas duplicadas, ela leva em conta uma gama de colunas - por padrão ele considera todas as colunas. Como mostrado na página de desduplicação, é possível ajustar este intervalo de colunas para que a singularidade das linhas só seja avaliada em relação a determinadas colunas.
Neste exemplo simples, basta adicionar o comando vazio distinct() para a cadeia de pipe. Isto garante que não haja linhas que sejam 100% duplicatas de outras linhas (avaliadas em todas as colunas).
Começamos com nrow(linelist) de linhas em linelist.
linelist <- linelist %>%
distinct()Após a eliminação de duplicidades, existem nrow(linelist) linhas. Qualquer linha removida teria sido 100% duplicada de outras linhas.
Abaixo, o comando distinct() é adicionado à cadeia de pipe de limpeza:
# CADEIA 'PIPE' DE LIMPEZA
#(inicia com dados brutos e encadeia funções com o pipe até a limpeza dos dados)
##################################################################################
# começa a limpeza da cadeia pipe
###########################
linelist <- linelist_raw %>%
# padronizar a sintaxe do nome da coluna
janitor::clean_names() %>%
# re-nomear colunas manualmente
# NOVO nome # nome VELHO
rename(date_infection = infection_date,
date_hospitalisation = hosp_date,
date_outcome = date_of_outcome) %>%
# remova a coluna
select(-c(row_num, merged_header, x28)) %>%
# ACIMA ESTÃO OS PASSOS DE LIMPEZA JÁ DISCUTIDOS
#####################################################
# desduplicar
distinct()8.7 Criação de coluna e transformação
Nós recomendamos usar a função dplyr mutate() para adicionar uma nova coluna, ou para modificar uma existente.
Abaixo está um exemplo de criação de uma nova coluna com mutate(). A sintaxe é: mutate(novo_nome_de_coluna = valor ou transformação)
No Stata, isto é similar ao comando generate, mas o mutate() do R pode também ser usado para modificar uma coluna existente.
Novas colunas
O comando mutate() mais básico para criar uma nova coluna deve parecer como este. Ele cria uma nova coluna nova_coluna onde o valor em cada linha é 10.
linelist <- linelist %>%
mutate(nova_coluna = 10)Você também pode referenciar valores em outras colunas, para realizar cálculos. Abaixo, uma nova coluna bmi é criada para manter o Índice de Massa Corporal (IMC, Body Mass Index em inglês) para cada caso - como calculado utilizando a fórmula IMC = kg/m^2, utilizando coluna ht_cm e coluna wt_kg.
linelist <- linelist %>%
mutate(bmi = wt_kg / (ht_cm/100)^2) #bmi = IMC em portugêsSe forem criadas várias colunas novas, separe cada uma delas com uma vírgula e uma nova linha. Abaixo estão exemplos de novas colunas, incluindo aquelas que consistem de valores de outras colunas combinadas utilizando str_glue() do pacote stringr (veja a página em Caracteres e strings.
new_col_demo <- linelist %>%
mutate(
new_var_dup = case_id, # nova coluna = duplicata/copia outra coluna existente
new_var_static = 7, # nova coluna = todos os valores o mesmo
new_var_static = new_var_static + 5, # você pode sobrescrever uma coluna, e ele pode ser um cálculo de outras variáveis
new_var_paste = stringr::str_glue("{hospital} on ({date_hospitalisation})") # nova coluna = colando juntos valores de outras colunas
) %>%
select(case_id, hospital, date_hospitalisation, contains("new")) # mostra apenas nova coluna, para demonstrar objetivosRevise as novas colunas. Para demonstrar os objetivos, apenas as novas colunas e as colunas usadas para criá-las são mostradas:
DICA: Uma variação de mutate() é a função transmute(). Esta função adiciona uma nova coluna apenas como mutate(), mas também remove todas outras colunas que você não menciona dentro destes parênteses.
# ESCONDIDO DO LEITOR
# remove novas colunas de demonstração criadas acima
# linelist <- linelist %>%
# select(-contains("new_var"))Converter a classe de coluna
Colunas contendo valores que são datas, números ou valores lógicos (TRUE/FALSE, ou seja, verdadeiro/falso) só se comportarão como esperado se forem corretamente classificadas. Há uma diferença entre “2” da classe caracteres e 2 da classe numérica!
Há maneiras de definir a classe da coluna durante os comandos de importação, mas isso é muitas vezes complicado. Veja a seção Introdução ao R sobre classes de objetos para saber mais sobre a conversão da classe de objetos e colunas.
Primeiro, vamos fazer algumas verificações em colunas importantes para ver se são a classe correta. Também vimos isso no início, quando corremos skim().
Atualmente, a classe da coluna age é caractere. Para realizar análises quantitativas, precisamos que estes números sejam reconhecidos como numéricos!
class(linelist$age)[1] "character"A classe da coluna date_onset também é caractere! Para realizar análises, estas datas devem ser reconhecidas como datas!
class(linelist$date_onset)[1] "character"Para resolver isto, utilize a capacidade de mutate() de redefinir uma coluna com uma transformação. Nós definimos a coluna como ela mesma, mas convertida em uma classe diferente. Aqui está um exemplo básico, convertendo ou assegurando que a coluna ‘age’ seja de classe numérica:
linelist <- linelist %>%
mutate(age = as.numeric(age))Em um modo similar, você pode usar as.character() e as.logical(). Para converter para a classe Fator, você pode usar factor() do R base ou as_factor() do forcats. Leia mais sobre isso na página Fatores.
Você deve ter cuidado ao converter para a classe Data. Vários métodos são explicados na página Trabalhando com datas. Normalmente, os valores de data bruta devem estar todos no mesmo formato para que a conversão funcione corretamente (por exemplo “MM/DD/YYYY”, ou “DD MM YYYY”). Após a conversão para a classe Data, verifique seus dados para confirmar que cada valor foi convertido corretamente.
Dados agrupados
Se o seu dataframe já estiver agrupado (veja a página Dados agrupados), mutate() pode se comportar de forma diferente do que se o dataframe não estiver agrupado. Qualquer função resumida, como mean(), median(), max(), etc. será calculada por grupo, não por todas as linhas.
# idade normalizada pela média de TODAS as linhas
linelist %>%
mutate(age_norm = age / mean(age, na.rm=T))
# idade normalizada pela média do grupo do hospital
linelist %>%
group_by(hospital) %>%
mutate(age_norm = age / mean(age, na.rm=T))Leia mais sobre a utilização de mutate () em dataframes agrupados nesta documentaçao do mutate tidyverse.
Transformar múltiplas colunas
Muitas vezes para escrever um código conciso você quer aplicar a mesma transformação a várias colunas ao mesmo tempo. Uma transformação pode ser aplicada a múltiplas colunas ao mesmo tempo utilizando a função across() do pacote dplyr (ambém contida no pacote tidyverse). across() pode ser utilizada com qualquer função dplyr, mas é comumente utilizada dentro de select(), mutate(), filter(), ou summarise(). Veja como é aplicado a summarise() na página de Tabelas Descritivas.
Especifique as colunas para o argumento .cols = e a(s) função(ões) a ser(em) aplicada(s) a .fns =. Quaisquer argumentos adicionais a serem fornecidos à função .fns' podem ser incluídos após uma vírgula, ainda dentro deacross()`.
Seleção de coluna across()
Especifique as colunas para o argumento .cols =. Você pode nomeá-las individualmente, ou usar as funções do helper do “tidyselect”. Especifique a função para .fns =`. Observe que utilizando o modo de função demonstrado abaixo, a função é escrita sem seus parênteses ( ).
Aqui a transformação as.character() é aplicada a colunas específicas nomeadas dentro de across().
linelist <- linelist %>%
mutate(across(.cols = c(temp, ht_cm, wt_kg), .fns = as.character))As funções auxiliares “tidyselect” estão disponíveis para ajudá-lo a especificar colunas. Elas são detalhadas acima na seção sobre Seleção e reordenação de colunas, e eles incluem: everything(), last_col(), where(), starts_with(), ends_with(), contains(), matches(), num_range() e any_of().
Aqui está um exemplo de como se pode mudar todas as colunas para a classe de caracteres:
# para mudar todas as colunas para a classe caractere
linelist <- linelist %>%
mutate(across(.cols = everything(), .fns = as.character))Converter em caracteres todas as colunas onde o nome contém a string “data” (note a colocação de vírgulas e parênteses):
# para mudar todas as colunas para classe de caracteres
linelist <- linelist %>%
mutate(across(.cols = contains("date"), .fns = as.character))Abaixo, um exemplo de transformação de colunas que atualmente são da classe POSIXct (uma classe de data/hora bruta que mostra marcações de hora) - em outras palavras, onde a função is.POSIXct() avalia para TRUE. Em seguida, queremos aplicar a função as.Date() a estas colunas para convertê-las em uma classe normal Date.
linelist <- linelist %>%
mutate(across(.cols = where(is.POSIXct), .fns = as.Date))- Note que dentro de
across()também utilizamos a funçãowhere()comois.POSIXctestá avaliando para TRUE ou FALSE.
- Note que
is.POSIXct()é do pacote lubridate. Outras funções “is” similares comois.character(),is.numeric(), eis.logical()são do R base.
Funçõesacross()
Você pode ler a documentação com ?across' para obter detalhes sobre como fornecer funções paraacross()`. Alguns pontos resumidos: há várias maneiras de especificar a(s) função(ões) a ser(em) executada(s) em uma coluna e você pode até mesmo definir suas próprias funções:
Você pode fornecer apenas o nome da função (por ex.,
meanouas.character)
Você pode fornecer a função no estilo purrr (por ex.,
~ mean(.x, na.rm = TRUE)) (veja esta página de Iterações, Listas e Loops)
Você pode especificar múltiplas funções fornecendo uma lista (por exemplo,
list(mean = mean, n_miss = ~ sum(is.na(.x))).Se você fornecer múltiplas funções, múltiplas colunas transformadas serão retornadas por coluna de entrada, com nomes únicos no formato
col_fn. Você pode ajustar como as novas colunas são nomeadas com o argumento.names =usando a sintaxe glue (veja a página de Caracteres e strings) onde{.col}e{.fn}são are abreviaturas para a coluna de entrada e função.
Aqui estão alguns recursos on-line sobre a utilização de across(): creator Hadley Wickham’s thoughts/rationale
coalesce()
Esta função dplyr encontra o primeiro valor não-faltante em cada posição. Ela “preenche” os valores ausentes com o primeiro valor disponível em uma ordem especificada por você.
Aqui está um exemplo fora do contexto de um data frame: Digamos que você tem dois vetores, um contendo a vila de detecção do paciente e outro contendo a vila de residência do paciente. Você pode usar a coalesce para escolher o primeiro valor não-faltante para cada índice:
village_detection <- c("a", "b", NA, NA)
village_residence <- c("a", "c", "a", "d")
village <- coalesce(village_detection, village_residence)
village # imprima[1] "a" "b" "a" "d"Isto funciona da mesma forma se você fornecer colunas de data frame: para cada linha, a função atribuirá o novo valor de coluna com o primeiro valor não descartado nas colunas que você forneceu (na ordem fornecida).
linelist <- linelist %>%
mutate(village = coalesce(village_detection, village_residence))Este é um exemplo de uma operação “em linha”. Para cálculos mais complicados em linha, veja a seção abaixo sobre cálculos em linha.
Matemática cumulativa
Se você quiser que uma coluna reflita a soma cumulativa/média/mínimo/máximo etc para avaliar as linhas em um dataframe até este ponto, usa as seguintes funções:
cumsum() retorna a soma cumulativa, como mostrado abaixo:
sum(c(2,4,15,10)) # retorna apenas um número[1] 31cumsum(c(2,4,15,10)) # retorna a soma cumulativa em cada etapa[1] 2 6 21 31Isto pode ser usado em um dataframe ao fazer uma nova coluna. Por exemplo, para calcular o número cumulativo de casos por dia em um surto, considere um código como este:
cumulative_case_counts <- linelist %>% # inicia com o caso linelist
count(date_onset) %>% # conta as linhas por dia, como coluna 'n'
mutate(cumulative_cases = cumsum(n)) # nova coluna, da soma cumulativa em cada linhaAbaixo estão as primeiras 10 linhas:
head(cumulative_case_counts, 10) date_onset n cumulative_cases
1 2012-04-15 1 1
2 2012-05-05 1 2
3 2012-05-08 1 3
4 2012-05-31 1 4
5 2012-06-02 1 5
6 2012-06-07 1 6
7 2012-06-14 1 7
8 2012-06-21 1 8
9 2012-06-24 1 9
10 2012-06-25 1 10Veja a página sobre Curvas epidêmicas para saber como plotar a incidência acumulada com a epicurva.
Veja também:
cumsum(), cummean(), cummin(), cummax(), cumany(), cumall()
Usando o R base
Para definir uma nova coluna (ou redefinir uma coluna) utilizando o R base, escreva o nome do data frame, conectado com $, para a nova coluna (ou a colunas a ser modificada). Use o operador de atribuição <- para definir o(s) novo(s) valor(es). Relembre que quando usar o R base você deve especificar o nome do data frame antes do nome da coluna toda vez (por ex., dataframe$column). Aqui é um exemplo de criação da coluna bmi usando o R base:
linelist$bmi = linelist$wt_kg / (linelist$ht_cm / 100) ^ 2)Adicionar a cadeia pipe
Abaixo, uma nova coluna é adicionada à cadeia pipe e algumas classes são convertidas.
# CADEIA 'PIPE' DE LIMPEZA
#(começa com dados brutos e faz pipes através das etapas de limpeza)
##################################################################################
# inicie a cadeia pipe limpa
###########################
linelist <- linelist_raw %>%
# padronizar a sintaxe do nome da coluna
janitor::clean_names() %>%
# re-nomear colunas manualmente
# NOVO nome # nome VELHO
rename(date_infection = infection_date,
date_hospitalisation = hosp_date,
date_outcome = date_of_outcome) %>%
# remova coluna
select(-c(row_num, merged_header, x28)) %>%
# remova duplicidade
distinct() %>%
# ACIMA SÃO PASSOS DE LIMPEZA A MONTANTE JÁ DISCUTIDOS
###################################################
# adicione nova coluna
mutate(bmi = wt_kg / (ht_cm/100)^2) %>%
# converta a classe das colunas
mutate(across(contains("date"), as.Date),
generation = as.numeric(generation),
age = as.numeric(age)) 8.8 Re-codificar valores
Aqui estão alguns cenários onde você precisa recodificar (mudar) os valores:
- para editar um valor específico (por exemplo, uma data com um ano ou formato incorreto)
- para reconciliar valores não soletrados da mesma forma
- para criar uma nova coluna de valores categóricos
- para criar uma nova coluna de categorias numéricas (por exemplo, categorias de idade)
Valores específicos
Para alterar valores manualmente, você pode utilizar a função recode() dentro da função mutate().
Imagine que haja uma data sem sentido nos dados (por exemplo, “2014-14-15”): você poderia fixar a data manualmente nos dados da fonte bruta, ou, você poderia escrever a alteração no pipe de limpeza via mutate() e recode(). Esta última é mais transparente e reprodutível para qualquer outra pessoa que procure entender ou repetir sua análise.
# corrigir valores incorretos # valor velho # valor novo
linelist <- linelist %>%
mutate(date_onset = recode(date_onset, "2014-14-15" = "2014-04-15"))A linha mutate() acima pode ser lida como: “mude a coluna date_onset para igualar a coluna date_onset re-codificada para que o VALOR VELHO seja alterado para o NOVO VALOR”. Note que este padrão (VELHO = NOVO) para recode() é o oposto da maioria dos padrões de R (novo = velho). A comunidade de desenvolvedores do R está trabalhando na revisão deste padrão.
Aqui está outro exemplo de recodificação de múltiplos valores dentro de uma coluna.
Na linelist os valores dna coluna “hospital” devem ser limpos. Há várias grafias diferentes e muitos valores em falta.
table(linelist$hospital, useNA = "always") # imprimir tabela de todos os valores únicos, incluindo os que faltam
Central Hopital Central Hospital
11 457
Hospital A Hospital B
290 289
Military Hopital Military Hospital
32 798
Mitylira Hopital Mitylira Hospital
1 79
Other Port Hopital
907 48
Port Hospital St. Mark's Maternity Hospital (SMMH)
1756 417
St. Marks Maternity Hopital (SMMH) <NA>
11 1512 O comando recode() abaixo re-define a coluna “hospital” como a coluna atual “hospital”, mas com a recodificação especificada muda. Não esqueça as vírgulas depois de cada uma!
linelist <- linelist %>%
mutate(hospital = recode(hospital,
# for reference: OLD = NEW
"Mitylira Hopital" = "Military Hospital",
"Mitylira Hospital" = "Military Hospital",
"Military Hopital" = "Military Hospital",
"Port Hopital" = "Port Hospital",
"Central Hopital" = "Central Hospital",
"other" = "Other",
"St. Marks Maternity Hopital (SMMH)" = "St. Mark's Maternity Hospital (SMMH)"
))Agora vemos que as grafias na coluna ‘hospitalar’ foram corrigidas e consolidadas:
table(linelist$hospital, useNA = "always")
Central Hospital Hospital A
468 290
Hospital B Military Hospital
289 910
Other Port Hospital
907 1804
St. Mark's Maternity Hospital (SMMH) <NA>
428 1512 [DICA: O número de espaços antes e depois de um sinal de igual não importa. Torne seu código mais fácil de ler, alinhando-o = para todas ou para a maioria das linhas. Além disso, considere acrescentar uma linha de comentários em destaque para esclarecer aos futuros leitores qual lado é VELHO e qual lado é NOVO.] {style=“color: darkgreen;”}
DICA: Às vezes existe um valor de caractere em branco em um conjunto de dados (não reconhecido como o valor de R por ausente - NA). Você pode referenciar este valor com duas aspas sem espaço intermediário
Pela lógica
A seguir demonstramos como recodificar valores em uma coluna usando lógica e condições:
- Utilizando
replace(),ifelse()eif_else()para uma lógica simples - Utilizando
case_when()para uma lógica mais complexa
Lógica simples
replace()
Para re-codificar com critérios lógicos simples, você pode utilizar replace() dentro de mutate(). replace() é uma função do R base. Utilize uma condição lógica para especificar as linhas a serem alteradas . A sintaxe geral é:
mutate(col_to_change = replace(col_to_change, critério para linhas, new value)).
Uma situação comum para utilizar replace() é alterar apenas um valor em uma linha, utilizando um identificador único de linha. Abaixo, o gênero é alterado para “Female” (Feminino) na linha onde a coluna case_id é “2195”.
# Exemplo: mudar o gênero de uma observação específica para "Female" (Feminino).
linelist <- linelist %>%
mutate(gender = replace(gender, case_id == "2195", "Female"))O comando equivalente utilizando a síntaxe do R base e colchetes indexadores [ ] está abaixo. Ele é lido como “Alterar o valor da coluna gender do dataframe linelist (para as linhas onde a coluna case_id do linelist tem o valor de ‘2195’) para ‘Female’”.
linelist$gender[linelist$case_id == "2195"] <- "Female"ifelse() e if_else()
Outra ferramenta de lógica simples é ifelse() e seu parceiro if_else(). Entretanto, na maioria dos casos para re-codificação é mais claro utilizar case_when() (detalhado abaixo). Estes comandos “if else” são versões simplificadas de uma declaração de programação if e else. A sintaxe geral é:
ifelse(condição, valor para retornar se a condição avlia se TRUE, valor para retornar se a condição avaliada é FALSE)
Abaixo, a coluna definida source_known é definida. Seu valor em uma determinada linha é definido como “conhecido” se o valor da linha na coluna source não estiver faltando. Se o valor em source está faltando, então o valor em source_known está definido como “desconhecido”.
linelist <- linelist %>%
mutate(source_known = ifelse(!is.na(source), "known", "unknown"))if_else() é uma versão especial de dplyr que manipula datas. Observe que se o valor ‘verdadeiro’ é uma data, o valor ‘falso’ também deve qualificar uma data, portanto, utilizando o valor especial NA_real_ em vez de apenas NA.
# Criar uma coluna de data de morte, que é NA, se o paciente não tiver morrido.
linelist <- linelist %>%
mutate(date_death = if_else(outcome == "Death", date_outcome, NA_real_))Evitar a junção de muitos comandos ifelse… use case_when() ao invés disso! case_when() é muito mais fácil de ler e você cometerá menos erros.
Fora do contexto de um data frame, se você quiser que um objeto utilizado em seu código mude seu valor, considere utilizar switch() do R base.
Lógica complexa
Use ocase_when() do dplyr se você estiver recodificando em muitos grupos novos, ou se você precisar utilizar declarações lógicas complexas para recodificar valores. Esta função avalia cada linha no data frame, avalia se as linhas atendem aos critérios especificados e atribui o novo valor correto.
Os comandos case_when() consistem de declarações que têm um Lado Direito (RHS, de Right-Hand Side em inglês) e um Lado Esquerdo (LHS, Left-Hand Side em inglês) separados por um “til” ~. Os critérios lógicos estão no lado esquerdo e os valores de ajuste estão no lado direito de cada afirmação. As afirmações são separadas por vírgulas.
Por exemplo, aqui utilizamos as colunas age e age_unit para criar uma coluna age_years:
linelist <- linelist %>%
mutate(age_years = case_when(
age_unit == "years" ~ age, # se a idade for dada em anos
age_unit == "months" ~ age/12, # se a idade for dada em meses
is.na(age_unit) ~ age)) # se falta a unidade de idade, suponha anos)
# qualquer outra circunstância, atribui NA (falta)Como cada linha dos dados é avaliado, os critérios são aplicados/avaliados na ordem em que as declaraçõe case_when() são escritos - de cima para baixo. Se o critério de cima avaliar TRUE para uma dada linha, o valor RHS é atribuído, e os critérios restantes não são sequer testados para essa linha. Assim, é melhor escrever primeiro os critérios mais específicos, e por último os mais gerais.
Nesta linha, em sua declaração final, coloque TRUE no lado esquerdo, que captará qualquer linha que não atenda a nenhum dos critérios anteriores. Ao lado direito desta declaração pode ser atribuído um valor como “checar!” ou perda.
PERIGO: Valores no lado direito devem ser todos na mesma classe - seja numérico, caractere, data, lógico, etc. Para atribuir valores em falta (NA), você talvez necessite usar as variações especiais de NA como NA_character_, NA_real_ (para numérico ou POSIX), e as.Date(NA). Leia mais em Trabalhando com datas.
Valores faltantes
Abaixo estão as funções especiais para manipular valores ausentes no contexto da limpeza de dados.
Veja a página em Campos em branco/faltantes para dicas mais detahadas sobre como identificas e tratar os valores ausentes. Por exemplo, a função is.na()que logicamente testa a falta de dados.
replace_na()
Para alterar valores ausentes (NA) para um valor específico, como “Ausente”, use a função dplyr replace_na() dentro de mutate(). Observe que isto é utilizado da mesma forma que a recode acima - o nome da variável deve ser repetida dentro de replace_na().
linelist <- linelist %>%
mutate(hospital = replace_na(hospital, "Ausente"))fct_explicit_na()
Esta é uma função do pacote forcats. O pacote forcats manipula colunas de classe Fator. Fatores são a forma do R manipular valores ordenados tais como c("Primeiro", "Segundo", "Terceiro") ou para definir a ordem em que os valores (por exemplo, hospitais) aparecem em tabelas e gráficos. Veja a página de Fatores.
Se seus dados forem de classe Fator e você tentar converter NA para “Ausente” utilizando replace_na(), você receberá este erro: invalid factor level, NA generated. Você tentou adicionar “Ausente” como um valor, quando este não foi definido como um nível possível do fator, e ele foi rejeitado.
A maneira mais fácil de resolver isto é utilizar a função de forcats fct_explicit_na() que converte uma coluna para a classe fator, e converte os valores NA para o caractere “(Missing)” (Ausente/Faltante).
linelist %>%
mutate(hospital = fct_explicit_na(hospital))Uma alternativa mais lenta seria adicionar o nível do fator utilizando fct_expand() e então converter os valores ausentes.
na_if()
Para converter um valor específico para NA, use na_if() do dplyr. O comando abaixo executa a operação oposta de replace_na(). No exemplo abaixo, quaisquer valores de “Ausente” na coluna hospital são convertidas para NA.
linelist <- linelist %>%
mutate(hospital = na_if(hospital, "Missing"))Observe: na_if() não pode ser usado para critérios lógicos (por ex., “todos os valores > 99”) - use replace() ou case_when() para isso:
# Converta temperaturas acima de 40 para NA
linelist <- linelist %>%
mutate(temp = replace(temp, temp > 40, NA))
# Converta as datas de início antes de 1 de janeiro de 2000 em ausente
linelist <- linelist %>%
mutate(date_onset = replace(date_onset, date_onset > as.Date("2000-01-01"), NA))Dicionário de limpeza
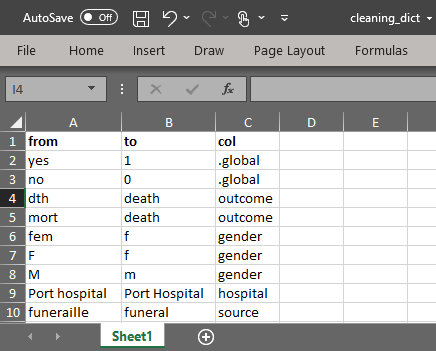
Utilize o pacote matchmaker do R e sua função match_df() para limpar um data frame com um dicionário de limpeza.
Criar um dicionário de limpeza com 3 colunas:
- Uma coluna “de” (o valor incorreto)
- Uma coluna “para” (o valor correto)
- Uma coluna especificando a coluna para as mudanças a serem aplicadas (ou “.global” para aplicar a todas as colunas)
- Uma coluna “de” (o valor incorreto)
Note: As entradas do dicionário .global serão substituídas por entradas de dicionário específicas de coluna
- Importar o arquivo do dicionário para R. Este exemplo pode ser baixado através das instruções na página Baixar manual e dados.
cleaning_dict <- import("cleaning_dict.csv")- Passe a linelist bruta para
match_df(), especificando paradictionary =o data frame do dicionário de limpeza. O argumentofrom =deve ter o nome da coluna que ontem os valores “antigos”, o argumentoby =deve ser a coluna do dicionário que contem os valores “novos”, e o terceiro argumento lista a coluna sobre a qual fazer a mudança.
Leia mais sobre em package documentation digitando ?match_df no console. Note que essa função pode demorar um tempo longo de execução em uma base de dados grande.
linelist <- linelist %>% # sua base de dados
matchmaker::match_df(
dictionary = cleaning_dict, # nome do dicionário
from = "from", # coluna com as variáveis a serem substituidas (o padrão é a col 1)
to = "to", # coluna ocm os valores finais (padrão é col 2)
by = "col" # coluna com nome de colunas (padrão é col 3)
)Agora role para a direita para ver como os valores mudaram - particularmente gender (de minúsculas para maiúsculas), e todas as colunas de sintomas foram transformadas de sim/não para 1/0.
Observe que os nomes das colunas no dicionário de limpeza devem corresponder aos nomes neste ponto do seu script de limpeza. Veja esta referência online para o pacote linelist para obter mais detalhes.
Adicione a cadeia pipe
Abaixo, algumas novas colunas e transformações de colunas são adicionadas à cadeia pipe.
# CADEIA 'PIPE' DE LIMPEZA
# inicia com dados brutos e canaliza-os através de etapas de limpeza)
##################################################################################
# inicie a cadeia pipe de limpeza
###########################
linelist <- linelist_raw %>%
# padronize a sintaxe do nome da coluna
janitor::clean_names() %>%
# renomeie as colunas manualmente
# NOVO nome # nome VELHO
rename(date_infection = infection_date,
date_hospitalisation = hosp_date,
date_outcome = date_of_outcome) %>%
# remova coluna
select(-c(row_num, merged_header, x28)) %>%
# remova duplicidade
distinct() %>%
# adicione colunas
mutate(bmi = wt_kg / (ht_cm/100)^2) %>%
# converta classe de colunas
mutate(across(contains("date"), as.Date),
generation = as.numeric(generation),
age = as.numeric(age)) %>%
# adicione coluna: demora para internação
mutate(days_onset_hosp = as.numeric(date_hospitalisation - date_onset)) %>%
# ACIMA ESTÃO AS ETAPAS DE LIMPEZA A MONTANTE JÁ DISCUTIDAS
###################################################
# limpe valores da coluna do hospital
mutate(hospital = recode(hospital,
# OLD = NEW
"Mitylira Hopital" = "Military Hospital",
"Mitylira Hospital" = "Military Hospital",
"Military Hopital" = "Military Hospital",
"Port Hopital" = "Port Hospital",
"Central Hopital" = "Central Hospital",
"other" = "Other",
"St. Marks Maternity Hopital (SMMH)" = "St. Mark's Maternity Hospital (SMMH)"
)) %>%
mutate(hospital = replace_na(hospital, "Missing")) %>%
# crie a coluna age_years (de age e age_unit)
mutate(age_years = case_when(
age_unit == "years" ~ age,
age_unit == "months" ~ age/12,
is.na(age_unit) ~ age,
TRUE ~ NA_real_))8.9 Categorias numéricas
Aqui descrevemos algumas abordagens especiais para criar categorias a partir de colunas numéricas. Exemplos comuns incluem categorias de idade, grupos de valores de laboratório, etc. Aqui vamos discutir:
age_categories(), do pacote epikit
cut(), do R base
case_when()
- quebras de quantis com
quantile()entile()
Distribuição de comentários
Para este exemplo, nós vamos criar uma coluna age_cat usando a coluna age_years.
# Verifique a classe da variável da linelist age
class(linelist$age_years)[1] "numeric"Primeiro, examine a distribuição de seus dados, para fazer os pontos de corte apropriados. Veja a página sobre Básico do ggplot.
summary(linelist$age_years, na.rm=T) Min. 1st Qu. Median Mean 3rd Qu. Max. NA's
0.00 6.00 13.00 16.04 23.00 84.00 107 CUIDADO: Às vezes, variáveis numéricas serão importadas como classe “character”. Isso ocorre se houver caracteres não numéricos em alguns dos valores, por exemplo, uma entrada de “2 meses” para idade ou (dependendo das configurações locais do R) se uma vírgula for usada na casa decimal (por exemplo, “4,5” para significar quatro anos e meio)..
age_categories()
Com o pacote epikit, você pode usar a função age_categories() fpara categorizar e rotular facilmente colunas numéricas (observação: esta função também pode ser aplicada a variáveis numéricas que não sejam de idade). Como bônus, a coluna de saída é automaticamente um fator ordenado.
Aqui estão as entradas necessárias:
- Um vetor numérico (coluna)
- O argumento
breakers =- fornece um vetor numérico de pontos de interrupção para os novos grupos
Primeiro, o exemplo mais simples:
# Exemplo simples
################
pacman::p_load(epikit) # carregue pacote
linelist <- linelist %>%
mutate(
age_cat = age_categories( # crie nova coluna
age_years, # coluna numérica para fazer grupos de
breakers = c(0, 5, 10, 15, 20, # pontos de quebra
30, 40, 50, 60, 70)))
# show table
table(linelist$age_cat, useNA = "always")
0-4 5-9 10-14 15-19 20-29 30-39 40-49 50-59 60-69 70+ <NA>
1227 1223 1048 827 1216 597 251 78 27 7 107 Os valores de quebra que você especifica são, por padrão, os limites inferiores - ou seja, eles são incluídos no grupo “superior” / os grupos são “abertos” no lado inferior/esquerdo. Conforme mostrado abaixo, você pode adicionar 1 a cada valor de quebra para obter grupos abertos na parte superior/direita.
# Incluir extremidades superiores para as mesmas categorias
############################################
linelist <- linelist %>%
mutate(
age_cat = age_categories(
age_years,
breakers = c(0, 6, 11, 16, 21, 31, 41, 51, 61, 71)))
# Mostre tabela
table(linelist$age_cat, useNA = "always")
0-5 6-10 11-15 16-20 21-30 31-40 41-50 51-60 61-70 71+ <NA>
1469 1195 1040 770 1149 547 231 70 24 6 107 Você pode ajustar como os rótulos são exibidos com separator =. O padrão é “-”
Você pode ajustar como os números superiores são tratados, com o argumento ceiling =. Para definir um limite superior, defina ceiling = TRUE. Neste uso, o valor de quebra mais alto fornecido é um “teto” e uma categoria “XX+” não é criada. Quaisquer valores acima do valor de quebra mais alto (ou para upper =, se definido) são categorizados como NA. Abaixo está um exemplo com ceiling = TRUE, para que não haja categoria de XX+ e valores acima de 70 (o maior valor de quebra) sejam atribuídos como NA.
# Com teto definido para TRUE
##########################
linelist <- linelist %>%
mutate(
age_cat = age_categories(
age_years,
breakers = c(0, 5, 10, 15, 20, 30, 40, 50, 60, 70),
ceiling = TRUE)) # 70 is ceiling, all above become NA
# Mostre a tabela
table(linelist$age_cat, useNA = "always")
0-4 5-9 10-14 15-19 20-29 30-39 40-49 50-59 60-70 <NA>
1227 1223 1048 827 1216 597 251 78 28 113 Alternativamente, em vez de breakers =, você pode fornecer todos de lower =, upper =, e by =:
lower =O número mais baixo que você quer considerar - o padrão é 0
upper =O número mais alto que você quer considerar
by =O número de anos entre grupos
linelist <- linelist %>%
mutate(
age_cat = age_categories(
age_years,
lower = 0,
upper = 100,
by = 10))
# Mostre a tabela
table(linelist$age_cat, useNA = "always")
0-9 10-19 20-29 30-39 40-49 50-59 60-69 70-79 80-89 90-99 100+ <NA>
2450 1875 1216 597 251 78 27 6 1 0 0 107 Veja a página de Ajuda da função para mais detalhes (digite ?age_categories no console do R).
cut()
cut() é uma alternativa do R base para age_categories(), mas eu acho que você verá porque age_categories() foi desenvolvida para simplificar este processo. Algumas diferenças notáveis da age_categories() são:
- Você não precisa instalar/carregar outro pacote
- Você pode especificar se os grupos estão abertos/fechados à direita/esquerda
- Você mesmo deve fornecer etiquetas precisas
- Se você quiser 0 incluído no grupo mais baixo, você deve especificar isso
A sintaxe básica dentro de cut() é fornecer primeiro a coluna numérica a ser cortada (age_years), e então o argumento breaks, que é um vetor numérico c() de pontos de quebra. Usando cut(), a coluna resultante é um fator ordenado.
Por padrão, a categorização ocorre de forma que o lado direito/superior seja “aberto” e inclusivo (e o lado esquerdo/inferior seja “fechado” ou exclusivo). Este é o comportamento oposto da função age_categories(). Os rótulos padrão usam a notação “(A, B]”, o que significa que A não está incluído, mas B sim. Inverta esse comportamento fornecendo o argumento right = TRUE.
Assim, por padrão, os valores “0” são excluídos do grupo mais baixo e categorizados como NA! Os valores “0” podem ser bebês codificados como 0, então tenha cuidado! Para alterar isso, adicione o argumento include.lowest = TRUE para que qualquer valor “0” seja incluído no grupo mais baixo. O rótulo gerado automaticamente para a categoria mais baixa será “[A],B]”. Observe que se você incluir o argumento include.lowest = TRUE e right = TRUE, a inclusão extrema agora será aplicada ao valor e categoria do ponto de quebra mais alto, não ao mais baixo.
Você pode fornecer um vetor de rótulos personalizados usando o argumento labels =. Como estes são escritos manualmente, tenha muito cuidado para garantir que eles sejam precisos! Verifique seu trabalho usando tabulação cruzada, conforme descrito abaixo.
Um exemplo de cut() aplicado a age_years para fazer a nova variável age_cat está abaixo:
# Crie a nova variável, pelo corte da variável numérica age
# quebra inferior é excluída, mas quebra superior é incluída em cada categoria
linelist <- linelist %>%
mutate(
age_cat = cut(
age_years,
breaks = c(0, 5, 10, 15, 20,
30, 50, 70, 100),
include.lowest = TRUE # inclua 0 no grupo mais baixo
))
# tabule o número de observaçõs por grupo
table(linelist$age_cat, useNA = "always")
[0,5] (5,10] (10,15] (15,20] (20,30] (30,50] (50,70] (70,100]
1469 1195 1040 770 1149 778 94 6
<NA>
107 Verifique seu trabalho!!! Verifique se cada valor de idade foi atribuído à categoria correta fazendo uma tabulação cruzada das colunas numéricas e de categoria. Examine a atribuição de valores de limite (por exemplo, 15, se as categorias vizinhas forem 10-15 e 16-20).
# Tabulação cruzada das colunas numéricas e de categoria.
table("Numeric Values" = linelist$age_years, # nomes especificados na tabela para maior clareza.
"Categories" = linelist$age_cat,
useNA = "always") # não esqueça de examinar os valores NA Categories
Numeric Values [0,5] (5,10] (10,15] (15,20] (20,30] (30,50] (50,70]
0 136 0 0 0 0 0 0
0.0833333333333333 1 0 0 0 0 0 0
0.25 2 0 0 0 0 0 0
0.333333333333333 6 0 0 0 0 0 0
0.416666666666667 1 0 0 0 0 0 0
0.5 6 0 0 0 0 0 0
0.583333333333333 3 0 0 0 0 0 0
0.666666666666667 3 0 0 0 0 0 0
0.75 3 0 0 0 0 0 0
0.833333333333333 1 0 0 0 0 0 0
0.916666666666667 1 0 0 0 0 0 0
1 275 0 0 0 0 0 0
1.5 2 0 0 0 0 0 0
2 308 0 0 0 0 0 0
3 246 0 0 0 0 0 0
4 233 0 0 0 0 0 0
5 242 0 0 0 0 0 0
6 0 241 0 0 0 0 0
7 0 256 0 0 0 0 0
8 0 239 0 0 0 0 0
9 0 245 0 0 0 0 0
10 0 214 0 0 0 0 0
11 0 0 220 0 0 0 0
12 0 0 224 0 0 0 0
13 0 0 191 0 0 0 0
14 0 0 199 0 0 0 0
15 0 0 206 0 0 0 0
16 0 0 0 186 0 0 0
17 0 0 0 164 0 0 0
18 0 0 0 141 0 0 0
19 0 0 0 130 0 0 0
20 0 0 0 149 0 0 0
21 0 0 0 0 158 0 0
22 0 0 0 0 149 0 0
23 0 0 0 0 125 0 0
24 0 0 0 0 144 0 0
25 0 0 0 0 107 0 0
26 0 0 0 0 100 0 0
27 0 0 0 0 117 0 0
28 0 0 0 0 85 0 0
29 0 0 0 0 82 0 0
30 0 0 0 0 82 0 0
31 0 0 0 0 0 68 0
32 0 0 0 0 0 84 0
33 0 0 0 0 0 78 0
34 0 0 0 0 0 58 0
35 0 0 0 0 0 58 0
36 0 0 0 0 0 33 0
37 0 0 0 0 0 46 0
38 0 0 0 0 0 45 0
39 0 0 0 0 0 45 0
40 0 0 0 0 0 32 0
41 0 0 0 0 0 34 0
42 0 0 0 0 0 26 0
43 0 0 0 0 0 31 0
44 0 0 0 0 0 24 0
45 0 0 0 0 0 27 0
46 0 0 0 0 0 25 0
47 0 0 0 0 0 16 0
48 0 0 0 0 0 21 0
49 0 0 0 0 0 15 0
50 0 0 0 0 0 12 0
51 0 0 0 0 0 0 13
52 0 0 0 0 0 0 7
53 0 0 0 0 0 0 4
54 0 0 0 0 0 0 6
55 0 0 0 0 0 0 9
56 0 0 0 0 0 0 7
57 0 0 0 0 0 0 9
58 0 0 0 0 0 0 6
59 0 0 0 0 0 0 5
60 0 0 0 0 0 0 4
61 0 0 0 0 0 0 2
62 0 0 0 0 0 0 1
63 0 0 0 0 0 0 5
64 0 0 0 0 0 0 1
65 0 0 0 0 0 0 5
66 0 0 0 0 0 0 3
67 0 0 0 0 0 0 2
68 0 0 0 0 0 0 1
69 0 0 0 0 0 0 3
70 0 0 0 0 0 0 1
72 0 0 0 0 0 0 0
73 0 0 0 0 0 0 0
76 0 0 0 0 0 0 0
84 0 0 0 0 0 0 0
<NA> 0 0 0 0 0 0 0
Categories
Numeric Values (70,100] <NA>
0 0 0
0.0833333333333333 0 0
0.25 0 0
0.333333333333333 0 0
0.416666666666667 0 0
0.5 0 0
0.583333333333333 0 0
0.666666666666667 0 0
0.75 0 0
0.833333333333333 0 0
0.916666666666667 0 0
1 0 0
1.5 0 0
2 0 0
3 0 0
4 0 0
5 0 0
6 0 0
7 0 0
8 0 0
9 0 0
10 0 0
11 0 0
12 0 0
13 0 0
14 0 0
15 0 0
16 0 0
17 0 0
18 0 0
19 0 0
20 0 0
21 0 0
22 0 0
23 0 0
24 0 0
25 0 0
26 0 0
27 0 0
28 0 0
29 0 0
30 0 0
31 0 0
32 0 0
33 0 0
34 0 0
35 0 0
36 0 0
37 0 0
38 0 0
39 0 0
40 0 0
41 0 0
42 0 0
43 0 0
44 0 0
45 0 0
46 0 0
47 0 0
48 0 0
49 0 0
50 0 0
51 0 0
52 0 0
53 0 0
54 0 0
55 0 0
56 0 0
57 0 0
58 0 0
59 0 0
60 0 0
61 0 0
62 0 0
63 0 0
64 0 0
65 0 0
66 0 0
67 0 0
68 0 0
69 0 0
70 0 0
72 1 0
73 3 0
76 1 0
84 1 0
<NA> 0 107Rotule novamente os valores NA
Você pode querer atribuir a valores NA um rótulo tal como “Ausente”. Como a nova coluna é de classe Fator (valores restritos), você não pode simplesmente mudá-la com replace_na(), pois este valor será rejeitado. Em vez disso, utilize fct_explicit_na() de forcats, como explicado na página Fatores.
linelist <- linelist %>%
# cut() cria age_cat, automaticamente da classe Fator
mutate(age_cat = cut(
age_years,
breaks = c(0, 5, 10, 15, 20, 30, 50, 70, 100),
right = FALSE,
include.lowest = TRUE,
labels = c("0-4", "5-9", "10-14", "15-19", "20-29", "30-49", "50-69", "70-100")),
# tornar explícitos os valores ausentes
age_cat = fct_explicit_na(
age_cat,
na_level = "Idade Ausente") # você pode especificar o rótulo
) Warning: There was 1 warning in `mutate()`.
ℹ In argument: `age_cat = fct_explicit_na(age_cat, na_level = "Idade
Ausente")`.
Caused by warning:
! `fct_explicit_na()` was deprecated in forcats 1.0.0.
ℹ Please use `fct_na_value_to_level()` instead.# tabela para ver contagens
table(linelist$age_cat, useNA = "always")
0-4 5-9 10-14 15-19 20-29
1227 1223 1048 827 1216
30-49 50-69 70-100 Idade Ausente <NA>
848 105 7 107 0 Fazer quebras e rótulos rapidamente
Para uma maneira rápida de fazer quebras e rotular vetores, use algo como abaixo. Veja a página Introdução do R para referências em seq() e rep().
# Fazer pontos de quebra de 0 a 90 por 5
age_seq = seq(from = 0, to = 90, by = 5)
age_seq
# Fazer etiquetas para as categorias acima, assumindo as configurações padrão de cut()
age_labels = paste0(age_seq + 1, "-", age_seq + 5)
age_labels
# verificar se ambos os vetores têm o mesmo comprimento
length(age_seq) == length(age_labels)Leia mais sobre cut() em sua página de Ajuda entrando em ?cut no console R
Quebrar por quantil
No entendimento comum, “quantis” ou “percentis” normalmente se referem a um valor abaixo do qual uma proporção de valores está contida. Por exemplo, o 95º percentil de idades dessa linelist seria a idade abaixo da qual estão 95% dos valores de idade.
No entanto, na fala comum, “quartis” e “decis” também podem se referir aos grupos de dados igualmente divididos em 4 ou 10 grupos (observe que haverá mais um ponto de quebra do que grupo).
Para obter pontos de quebra de quantil, você pode usar quantile() do pacote stats do R base. Você fornece um vetor numérico (por exemplo, uma coluna em um conjunto de dados) e um vetor de valores numéricos de probabilidade variando de 0 a 1,0. Os pontos de interrupção são retornados como um vetor numérico. Explore os detalhes das metodologias estatísticas inserindo ?quantile.
- Se o seu vetor numérico de entrada tiver algum valor ausente, é melhor definir
na.rm = TRUE
- Defina
names = FALSEpara obter um vetor numérico sem nome
quantile(linelist$age_years, # especifique o vetor numérico para trabalhar nele
probs = c(0, .25, .50, .75, .90, .95), # especifique os percentis que você quer
na.rm = TRUE) # ignore os valores ausentes 0% 25% 50% 75% 90% 95%
0 6 13 23 33 41 Você pode usar os resultados de quantile() como pontos de quebra em age_categories() ou cut(). Abaixo nós criamos uma nova coluna deciles usando cut() onde as quebras são definidas usando quantiles() em age_years. Abaixo, nós exibimos os resultados usando tabyl() do janitor para que você possa ver as porcentagens (consulte a página Tabelas descritivas ). Observe como eles não são exatamente 10% em cada grupo.
linelist %>% # inicie com linelist
mutate(deciles = cut(age_years, # crie a nova coluna decile como cut() da coluna age_years
breaks = quantile( # defina as quebras usando quantile()
age_years, # opere em age_years
probs = seq(0, 1, by = 0.1), # de 0.0 a 1.0 a cada 0.1
na.rm = TRUE), # ignore os valores ausentes
include.lowest = TRUE)) %>% # para cut() incluir idade 0
janitor::tabyl(deciles) # pipe para a tabela ser exibida deciles n percent valid_percent
[0,2] 748 0.11319613 0.11505922
(2,5] 721 0.10911017 0.11090601
(5,7] 497 0.07521186 0.07644978
(7,10] 698 0.10562954 0.10736810
(10,13] 635 0.09609564 0.09767728
(13,17] 755 0.11425545 0.11613598
(17,21] 578 0.08746973 0.08890940
(21,26] 625 0.09458232 0.09613906
(26,33] 596 0.09019370 0.09167820
(33,84] 648 0.09806295 0.09967697
<NA> 107 0.01619249 NAGrupos de tamanho uniforme
Outra ferramenta para fazer grupos numéricos é a função ntile() do dplyr , que tenta dividir seus dados em n grupos de tamanho uniforme - mas esteja ciente de que diferente com quantile() o mesmo valor pode aparecer em mais de um grupo. Forneça o vetor numérico e, em seguida, o número de grupos. Os valores na nova coluna criada são apenas “números” do grupo (por exemplo, 1 a 10), não o intervalo de valores em si, como ao usar cut().
# faça grupos com ntile()
ntile_data <- linelist %>%
mutate(even_groups = ntile(age_years, 10))
# faça uma tabela de contagem e proporções por grupo
ntile_table <- ntile_data %>%
janitor::tabyl(even_groups)
# fixe os valores mín/máx para demonstrar as faixas
ntile_ranges <- ntile_data %>%
group_by(even_groups) %>%
summarise(
min = min(age_years, na.rm=T),
max = max(age_years, na.rm=T)
)Warning: There were 2 warnings in `summarise()`.
The first warning was:
ℹ In argument: `min = min(age_years, na.rm = T)`.
ℹ In group 11: `even_groups = NA`.
Caused by warning in `min()`:
! no non-missing arguments to min; returning Inf
ℹ Run `dplyr::last_dplyr_warnings()` to see the 1 remaining warning.# combine e "printe" - note que valores estão presentes em múltiplos grupos
left_join(ntile_table, ntile_ranges, by = "even_groups") even_groups n percent valid_percent min max
1 651 0.09851695 0.10013844 0 2
2 650 0.09836562 0.09998462 2 5
3 650 0.09836562 0.09998462 5 7
4 650 0.09836562 0.09998462 7 10
5 650 0.09836562 0.09998462 10 13
6 650 0.09836562 0.09998462 13 17
7 650 0.09836562 0.09998462 17 21
8 650 0.09836562 0.09998462 21 26
9 650 0.09836562 0.09998462 26 33
10 650 0.09836562 0.09998462 33 84
NA 107 0.01619249 NA Inf -Infcase_when()
É possível utilizar a função case_when() do dplyr para criar categorias a partir de uma coluna numérica, mas é mais fácil utilizar age_categories() do epikit ou cut() porque estas criarão automaticamente um fator ordenado.
Se utilizar case_when(), por favor, revise o uso apropriado conforme descrito anteriormente na seção recodificar valores desta página. Esteja ciente também que todos os valores do lado direito devem ser da mesma classe. Assim, se você quiser NA no lado direito, você deve escrever “Ausente” ou utilizar o valor NA especial NA_character_.
Adicionar à cadeia pipe
Abaixo, é adicionado o código para criar duas colunas de idade categóricas à cadeia pipe de limpeza:
# CADEIA DE LIMPEZA 'PIPE'
# (inicie com dados brutos e canalize-os através de etapas de limpeza)
##################################################################################
# inicie a cadeia pipe de limpeza
###########################
linelist <- linelist_raw %>%
# padronize a sintaxe do nome da coluna
janitor::clean_names() %>%
# renomeie as colunas manualmente
# NOVO nome # nome VELHO
rename(date_infection = infection_date,
date_hospitalisation = hosp_date,
date_outcome = date_of_outcome) %>%
# remova coluna
select(-c(row_num, merged_header, x28)) %>%
# remova as duplicidades
distinct() %>%
# adicione coluna
mutate(bmi = wt_kg / (ht_cm/100)^2) %>%
# converta classe de colunas
mutate(across(contains("date"), as.Date),
generation = as.numeric(generation),
age = as.numeric(age)) %>%
# adicione coluna: demora para internação
mutate(days_onset_hosp = as.numeric(date_hospitalisation - date_onset)) %>%
# limpe valores da coluna do hospital
mutate(hospital = recode(hospital,
# VELHO = NOVO
"Mitylira Hopital" = "Military Hospital",
"Mitylira Hospital" = "Military Hospital",
"Military Hopital" = "Military Hospital",
"Port Hopital" = "Port Hospital",
"Central Hopital" = "Central Hospital",
"other" = "Other",
"St. Marks Maternity Hopital (SMMH)" = "St. Mark's Maternity Hospital (SMMH)"
)) %>%
mutate(hospital = replace_na(hospital, "Ausente")) %>%
# crie a coluna age_years (de age e age_unit)
mutate(age_years = case_when(
age_unit == "years" ~ age,
age_unit == "months" ~ age/12,
is.na(age_unit) ~ age,
TRUE ~ NA_real_)) %>%
# ACIMA ESTÃO AS ETAPAS DE LIMPEZA A MONTANTE JÁ DISCUTIDAS
###################################################
mutate(
# categorias de idade:personalizado
age_cat = epikit::age_categories(age_years, breakers = c(0, 5, 10, 15, 20, 30, 50, 70)),
# categorias etárias: 0 a 85 por 5s
age_cat5 = epikit::age_categories(age_years, breakers = seq(0, 85, 5)))8.10 Adicionar linhas
Uma-por-uma
Adicionar linhas uma-por-uma manualmente é tediosa mas pode ser feito com add_row() do dplyr. Relembre que cada coluna deve conter valores de apenas uma classe (seja caracter, numérico, lógico, etc.). Então adicionar uma linha reque detalhes para manter isso.
linelist <- linelist %>%
add_row(row_num = 666,
case_id = "abc",
generation = 4,
`infection date` = as.Date("2020-10-10"),
.before = 2)Utilize .before e .after. para especificar a colocação da linha que você deseja adicionar. .before = 3colocará a nova linha antes da 3ª linha atual. O comportamento padrão é adicionar a linha ao final. As colunas não especificadas serão deixadas vazias (NA).
O novo número da linha pode parecer estranho (“…23”), mas os números das linhas preexistentes mudaram. Portanto, se utilizar o comando duas vezes, examine/teste a inserção cuidadosamente.
Se uma classe que você fornece estiver desligada, você verá um erro como este:
Erro: Não é possível combinar ..1$infection date <date> e ..2$infection date <character>. (ao inserir uma linha com um valor de data, lembre-se de embrulhar a data na função as.Date() como as.Date("2020-10-10")).
Colar linhas
Para combinar conjuntos de dados unindo as linhas de um dataframe ao fundo de outro dataframe, pode-se utilizar bind_rows() do dplyr. Isto é explicado com mais detalhes na página Juntando dados.
8.11 Filtrar linhas
Uma etapa típica de limpeza depois de limpar as colunas e recodificar os valores é filtrar o dataframe para linhas específicas utilizando o verbo filter() do dplyr.
Dentro de filter(), especificar a lógica que deve ser TRUE para que uma linha do conjunto de dados seja mantida. Abaixo mostramos como filtrar linhas com base em condições lógicas simples e complexas.
Filtro simples
Este exemplo simples redefine o dataframe linelist como ela mesma, tendo filtrado as linhas para atender a uma condição lógica. Apenas as linhas onde a declaração lógica dentro dos parênteses avaliada comoTRUE são mantidas.
Neste exemplo, a afirmação lógica é gender == "f", que é perguntar se o valor na colunagender é igual a “f” (sensível a maiúsculas e minúsculas).
Antes do filtro ser aplicado, o número de linhas da linelist é nrow(linelist).
linelist <- linelist %>%
filter(gender == "f") # mantenha apenas as linhas onde o gênero é igual a "f"Depois que o filtro é aplicado, o número de linhas da linelist é linelist %>% filter(gender == "f") %>% nrow().
Filtrar valores faltantes
É bastante comum querer filtrar as linhas que possuem valores ausentes. Resista à tentação de escrever filter(!is.na(column) & !is.na(column)) e ao invés disso use a função tidyr que é customizada para este propósito: drop_na(). Se executado com parênteses vazios, ele remove linhas com qualquer valores ausentes. Alternativamente, você pode fornecer nomes de colunas específicas a serem avaliadas quanto à ausência, ou usar as funções auxiliares “tidyselect” descritas acima.
linelist %>%
drop_na(case_id, age_years) # elimila as linhas com valores faltates nas colunas case_id ou age_yearsVeja a página sobre Campos em branco/faltantes para várias técnicas para analisar e manipular valores faltantes nos seus dados.
Filtrar pelo número da linha
Em um quadro de dados ou tibble, cada linha terá geralmente um “número de linha” que (quando visto no R Viewer) aparece à esquerda da primeira coluna. Não é em si uma coluna verdadeira nos dados, mas pode ser utilizada em uma declaração filter().
Para filtrar com base no “número de linha”, é possível utilizar a função dplyr com o row_number() com parênteses abertos como parte de uma instrução de filtragem lógica. Muitas vezes você utilizará o operador %in% e um range de números como parte dessa instrução lógica, como mostrado abaixo. Para ver as primeiras N fileiras , você também pode utilizar a função especial head()l do pacote dplyr .
# Veja as primeiras 100 linhas
linelist %>% head(100) # ou use tail() tvara ver as últimas n linhas
# Mostre apenas 5 linhas
linelist %>% filter(row_number() == 5)
# Veja linha 2 a 20 e especifique 3 colunas
linelist %>% filter(row_number() %in% 2:20) %>% select(date_onset, outcome, age)Você também pode converter os números das linhas para uma coluna verdadeira, canalizando seu data Frame para a função tibble rownames_to_column() (não coloque nada entre parênteses).
Filtro complexo
Sentenças lógicas mais complexas podes ser contruídas usando parênteses ( ), OU os operadores |, !, %in%, e AND &. Um exemplo pode ser visto abaixo:
Nota: Você pode utilizar o ! operador em frente a um critério lógico para negá-lo. Por exemplo, !is.na(column) avalia para verdadeiro se o valor da coluna não é faltante. Da mesma forma, !column %in% c("a", "b", "c") avalia se o valor da coluna não está contido no vetor.
Examine os dados
Abaixo está um simples comando de uma linha para criar um histograma de datas de início. Veja que um segundo surto menor de 2012-2013 também está incluído neste conjunto de dados brutos. Para nossas análises, queremos remover as entradas deste surto anterior.
Como os filtros lidam com valores numericos e datas faltantes
Podemos simplesmente filtrar por ‘date_onset’ para as linhas após junho de 2013? Cuidado! Aplicando o código filter(date_onset > as.Date("2013-06-01"))) removeria qualquer linha na última epidemia com uma data de início ausente!
PERIGO: Filtrar para maior que (>) ou menor que (<) uma data ou número pode remover qualquer linha com valores ausentes (NA)! Isto porque NA é tratado como infinitamente grande e pequeno.
(Veja a página em Trabalhando com datas para mais informações sobre como trabalhar com datas e o pacote lubridate)
Desenhe o filtro
Examine a tabulação cruzada para ter certeza que excluímos somente as linhas corretas:
table(Hospital = linelist$hospital, # nome do hospital
YearOnset = lubridate::year(linelist$date_onset), # ano de inicio dos sintomas (date_onset)
useNA = "always") # mostrar valores faltantes YearOnset
Hospital 2012 2013 2014 2015 <NA>
Ausente 0 0 1117 318 77
Central Hospital 0 0 351 99 18
Hospital A 229 46 0 0 15
Hospital B 227 47 0 0 15
Military Hospital 0 0 676 200 34
Other 0 0 684 177 46
Port Hospital 9 1 1372 347 75
St. Mark's Maternity Hospital (SMMH) 0 0 322 93 13
<NA> 0 0 0 0 0Que outros critérios podemos filtrar para remover o primeiro surto (em 2012 e 2013) do conjunto de dados? Vemos isso:
- A primeira epidemia em 2012 & 2013 ocorreu no Hospital A, Hospital B, e que também houve 10 casos no Port Hospital.
- Os Hospitais A e B não tiveram casos na segunda epidemia, mas o Port Hospital teve.
Queremos excluir:
- A
nrow(linelist %>% filter(hospital %in% c("Hospital A", "Hospital B") | date_onset < as.Date("2013-06-01")))filas com início em 2012 e 2013 no hospital A, B, ou no Porto.- Excluir
nrow(linelist %>% filter(date_onset < as.Date("2013-06-01")))filas com início em 2012 e 2013 - Excluir
nrow(linelist %>% filter(hospital %em% c('Hospital A', 'Hospital B') & is.na(date_onset)))linhas dos Hospitais A & B com datas de início ausentes
- Fazer não excluir
nrow(linelist %>% filter(!hospital %in% c('Hospital A', 'Hospital B') & is.na(date_onset)))outras linhas com datas de início ausentes.
- Excluir
Começamos com uma lineliste de nrow(linelist){\i1}. Aqui está nossa declaração de filtro:
linelist <- linelist %>%
# mantém linhas onde o início é depois de 1 de Junho OU o início dos sintomas está faltante E o hospital é OUTRO que não A nem B
filter(date_onset > as.Date("2013-06-01") | (is.na(date_onset) & !hospital %in% c("Hospital A", "Hospital B")))
nrow(linelist)[1] 6019Quando refazemos a tabulação cruzada, vemos que os Hospitais A e B são completamente removidos, e os 10 casos de hospitais portuários de 2012 e 2013 são removidos, e todos os outros valores são os mesmos - exatamente como queríamos.
table(Hospital = linelist$hospital, # nome do hospital
YearOnset = lubridate::year(linelist$date_onset), # ano de início dos sintomas
useNA = "always") # mostrar valores faltantes YearOnset
Hospital 2014 2015 <NA>
Ausente 1117 318 77
Central Hospital 351 99 18
Military Hospital 676 200 34
Other 684 177 46
Port Hospital 1372 347 75
St. Mark's Maternity Hospital (SMMH) 322 93 13
<NA> 0 0 0Declarações múltiplas podem ser incluídas dentro de um comando de filtro (separadas por vírgulas), ou você pode sempre encadear com um %>% para um comando filter() separado para maior clareza.
Nota: alguns leitores podem notar que seria mais fácil filtrar apenas por date_hospitalisation porque é 100% completo sem valores ausentes. Isto é verdade. Mas o date_onset é utilizado com o propósito didático de demonstrar um filtro complexo.
Autônomo/Independente
Um filtro também pode ser feito como um comando autônomo (não parte de uma cadeia de pipes (%>%)). Como outros verbos dplyr, neste caso o primeiro argumento deve ser o próprio conjunto de dados.
# dataframe <- filter(dataframe, condition(s) for rows to keep)
linelist <- filter(linelist, !is.na(case_id))Você também pode usar o R base para criar um subconjunto dos dados, usando colchetes que refletem as [linhas, colunas] do que você deseja reter.
# dataframe <- dataframe[row conditions, column conditions] (se deixar a entrada em branco significa "todas as linhas ou todas as colunas")
linelist <- linelist[!is.na(case_id), ]Revisar rapidamente registros
Muitas vezes você quer rever rapidamente alguns registros, para apenas algumas colunas. A função do R base View() exibirá um dataframe para visualização em seu RStudio.
Veja a lineliste no RStudio:
View(linelist)Aqui estão dois exemplos de visualização de células específicas (linhas específicas, e colunas específicas):
Com funções dplyr filter() e select():
Dentro de View(), encadeie o conjunto de dados com o pipe (%>%) com a função filter() para manter certas linhas, e depois com a função select() para manter certas colunas. Por exemplo, para rever as datas de início e hospitalização de 3 casos específicos:
View(linelist %>%
filter(case_id %in% c("11f8ea", "76b97a", "47a5f5")) %>%
select(date_onset, date_hospitalisation))Você consegue os mesmos resultados com a seguinte sintaxe do R base , usando colchetes [ ] para subdividir o que você quer ver.
View(linelist[linelist$case_id %in% c("11f8ea", "76b97a", "47a5f5"), c("date_onset", "date_hospitalisation")])Adiciotar a uma cadeia usando o pipe (%>%)
# CADEIA DE LIMPEZA 'PIPE' (inicie com dados brutos e canalize-os através de etapas de limpeza)
##################################################################################
# inicie a cadeia pipe de limpeza
###########################
linelist <- linelist_raw %>%
# padronize a sintaxe do nome da coluna
janitor::clean_names() %>%
# renomeie as colunas manualmente
# NOVO nome # nome VELHO
rename(date_infection = infection_date,
date_hospitalisation = hosp_date,
date_outcome = date_of_outcome) %>%
# remova coluna
select(-c(row_num, merged_header, x28)) %>%
# remova duplicidades
distinct() %>%
# adicione colunas
mutate(bmi = wt_kg / (ht_cm/100)^2) %>%
# converta classe de colunas
mutate(across(contains("date"), as.Date),
generation = as.numeric(generation),
age = as.numeric(age)) %>%
# adicione coluna: demora para internação
mutate(days_onset_hosp = as.numeric(date_hospitalisation - date_onset)) %>%
# limpe valores da coluna do hospital
mutate(hospital = recode(hospital,
# VELHO = NOVO
"Mitylira Hopital" = "Military Hospital",
"Mitylira Hospital" = "Military Hospital",
"Military Hopital" = "Military Hospital",
"Port Hopital" = "Port Hospital",
"Central Hopital" = "Central Hospital",
"other" = "Other",
"St. Marks Maternity Hopital (SMMH)" = "St. Mark's Maternity Hospital (SMMH)"
)) %>%
mutate(hospital = replace_na(hospital, "Ausente")) %>%
# crie a coluna age_years (de age e age_unit)
mutate(age_years = case_when(
age_unit == "years" ~ age,
age_unit == "months" ~ age/12,
is.na(age_unit) ~ age,
TRUE ~ NA_real_)) %>%
mutate(
# categorias de idade: personalizado
age_cat = epikit::age_categories(age_years, breakers = c(0, 5, 10, 15, 20, 30, 50, 70)),
# categorias de idade: 0 a 85 por 5s
age_cat5 = epikit::age_categories(age_years, breakers = seq(0, 85, 5))) %>%
# ACIMA ESTÃO AS ETAPAS DE LIMPEZA A MONTANTE JÁ DISCUTIDAS
###################################################
filter(
# keep only rows where case_id is not missing
!is.na(case_id),
# also filter to keep only the second outbreak
date_onset > as.Date("2013-06-01") | (is.na(date_onset) & !hospital %in% c("Hospital A", "Hospital B")))8.12 Cálculo por linha
Se você quiser realizar um cálculo dentro de uma linha, você pode utilizar rowwise() a partir de dplyr. Veja esta vinheta online em cálculos em linha.
Por exemplo, este código se aplica rowwise() e então cria uma nova coluna que soma o número das colunas de sintomas especificados que têm o valor “yes” (sim), para cada linha da lista de linhas. As colunas são especificadas dentro de sum() pelo nome dentro de um vetor c(). A coluna rowwise() é essencialmente um tipo especial de group_by(), portanto é melhor utilizar ungroup() quando você terminar (página em Agrupar dados).
linelist %>%
rowwise() %>%
mutate(num_symptoms = sum(c(fever, chills, cough, aches, vomit) == "yes")) %>%
ungroup() %>%
select(fever, chills, cough, aches, vomit, num_symptoms) # para mostrar# A tibble: 5,888 × 6
fever chills cough aches vomit num_symptoms
<chr> <chr> <chr> <chr> <chr> <int>
1 no no yes no yes 2
2 <NA> <NA> <NA> <NA> <NA> NA
3 <NA> <NA> <NA> <NA> <NA> NA
4 no no no no no 0
5 no no yes no yes 2
6 no no yes no yes 2
7 <NA> <NA> <NA> <NA> <NA> NA
8 no no yes no yes 2
9 no no yes no yes 2
10 no no yes no no 1
# ℹ 5,878 more rowsAo especificar a coluna a ser avaliada, talvez você queira utilizar as funções de ajuda “tidyselect” descritas na seção select() desta página. Você só tem que fazer um ajuste (porque não está utilizando-as dentro de uma função dplyr como select() ou summarise()).
Coloque o critério de especificação de coluna dentro da função dplyr c_across(). Isto porque c_across (documentação) é projetado para trabalhar com rowwise() especificamente. Por exemplo, o seguinte código:
- Aplica-se
rowwise()assim a seguinte operação (sum()) é aplicada dentro de cada linha (não somando colunas inteiras)
- Cria nova coluna
num_NA_dates, definida para cada linha como o número de colunas (com nome contendo “data”) para as quaisis.na()avaliadas para VERDADEIRO (estão faltando dados).
- grupo()
ungroup()para remover os efeitos derowwise()para as etapas subseqüentes.
linelist %>%
rowwise() %>%
mutate(num_NA_dates = sum(is.na(c_across(contains("date"))))) %>%
ungroup() %>%
select(num_NA_dates, contains("date")) # para mostrar# A tibble: 5,888 × 5
num_NA_dates date_infection date_onset date_hospitalisation date_outcome
<int> <date> <date> <date> <date>
1 1 2014-05-08 2014-05-13 2014-05-15 NA
2 1 NA 2014-05-13 2014-05-14 2014-05-18
3 1 NA 2014-05-16 2014-05-18 2014-05-30
4 1 2014-05-04 2014-05-18 2014-05-20 NA
5 0 2014-05-18 2014-05-21 2014-05-22 2014-05-29
6 0 2014-05-03 2014-05-22 2014-05-23 2014-05-24
7 0 2014-05-22 2014-05-27 2014-05-29 2014-06-01
8 0 2014-05-28 2014-06-02 2014-06-03 2014-06-07
9 1 NA 2014-06-05 2014-06-06 2014-06-18
10 1 NA 2014-06-05 2014-06-07 2014-06-09
# ℹ 5,878 more rowsVocê também poderia fornecer outras funções, tais como max() para obter a data mais recente ou mais recente para cada linha:
linelist %>%
rowwise() %>%
mutate(latest_date = max(c_across(contains("date")), na.rm=T)) %>%
ungroup() %>%
select(latest_date, contains("date")) # para mostrar # A tibble: 5,888 × 5
latest_date date_infection date_onset date_hospitalisation date_outcome
<date> <date> <date> <date> <date>
1 2014-05-15 2014-05-08 2014-05-13 2014-05-15 NA
2 2014-05-18 NA 2014-05-13 2014-05-14 2014-05-18
3 2014-05-30 NA 2014-05-16 2014-05-18 2014-05-30
4 2014-05-20 2014-05-04 2014-05-18 2014-05-20 NA
5 2014-05-29 2014-05-18 2014-05-21 2014-05-22 2014-05-29
6 2014-05-24 2014-05-03 2014-05-22 2014-05-23 2014-05-24
7 2014-06-01 2014-05-22 2014-05-27 2014-05-29 2014-06-01
8 2014-06-07 2014-05-28 2014-06-02 2014-06-03 2014-06-07
9 2014-06-18 NA 2014-06-05 2014-06-06 2014-06-18
10 2014-06-09 NA 2014-06-05 2014-06-07 2014-06-09
# ℹ 5,878 more rows8.13 Organizar e ordenar
Utilize a função dplyr arrange() para ordenar ou ordenar as linhas por valores de coluna.
Simplesmente liste as colunas na ordem em que elas devem ser ordenadas. Especifique .by_group = TRUE se você quiser que a ordenação ocorra primeiro por quaisquer grupos aplicados aos dados (ver página em Agrupar dados).
Por padrão, a coluna será ordenada em ordem “ascendente” (que se aplica às colunas numéricas e também às colunas de caracteres). Você pode ordenar uma variável em ordem “decrescente”, envolvendo-a com desc().
A ordenação de dados com arrange() é particularmente útil ao fazer Tabelas para apresentação, utilizando slice() para pegar as linhas “top” por grupo, ou definir a ordem de nível de fator por ordem de aparência.
Por exemplo, para ordenar as linhas de nossa linelist por hospital, depois por date_onset em ordem decrescente, nós utilizaríamos:
linelist %>%
arrange(hospital, desc(date_onset))