28 Introdução ao GIS
28.1 Visão Geral
Os aspectos especiais dos seus dados podem trazer vários insights à situação de um surto, e pode responder perguntas como:
- Onde estão os hotspots da doença?
- Como os hotspots mudaram ao longo do tempo?
- Como é o acesso às unidades de saúdo? Melhorias são necessárias?
O foco dessa página sobre GIS é atender às necessidades dos epidemiologistas na resposta aos surtos. Iremos explorar métodos básicos de visualização de dados espaciais utilizando os pacotes tmap e ggplot2. Também vamos caminhar por métodos básicos de gerenciamento e consulta de dados espaciais com o pacote sf. Por fim, vamos dar uma olhada em conceitos de estatística espacial tais como relações espaciais, autocorrelação espacial e regressão espacial utilizando o pacote spdep.
28.2 Terminologia
Abaixo introduziremos a terminologia principal. Para uma introdução mais profunda ao GIS e à análise espacial, sugerimos que você revise algum dos tutoriais maiores ou cursos listados na seção de Referências.
Geographic Information System (GIS) (Sistema de Informação Geográfica) - Um GIS é um framework ou ambiente para reunir, gerenciar, analisar e visualizar dados espaciais.
Softwares GIS
Alguns Softwares GIS mais conhecidos permitem interação do tipo “apontar-e-clicar” (point-and-click) para o desenvolvimento de mapas e análises espaciais. Essas ferramentas vêm com as vantagens de não precisar aprender código e com a facilidade de selecionar e posicionar manualmente ícones e outras características em um mapa. Aqui temos dois dos mais conhecidos:
ArcGIS - Um software GIS comercial desenvolvido pela empresa ESRI, que é muito famoso mas bastante caro.
QGIS - Um software GIS gratuito e open-source que pode fazer praticamente qualquer coisa que o ArcGIS também pode. Você pode baixar o QGIS aqui
Utilizar o R como um GIS pode parecer mais intimidador no início, pois em vez do “apontar-e-clicar”, ele tem uma interface de linha de comando (você precisa escrever código para chegar no resultado esperado). No entanto, essa é uma grande vantagem se você precisa produzir mapas repetitivamente ou criar análises que sejam reprodutíveis.
Dados espaciais
As duas formas primordiais de dados espaciais utilizados no GIS são dados vetoriais e rasterizados.
Dados Vetoriais - O formato de dados espaciais mais comum utilizado no GIS. Dados vetoriais compreendem as propriedades geométricas de vértices e trajetos (paths). Os dados vetoriais espaciais também podem se subdividir em três tipos amplamente utilizados:
Pontos - Um ponto consiste em um par ordenado (x,y) representando um local específico em um sistema de coordenadas. Pontos são a forma mais básica de dados espaciais e podem ser utilizados para representar um caso (casa de um paciente) ou um local (hospital) em um mapa.
Linhas - Uma linha é composta de dois pontos conectados. Linhas possuem um comprimento, e podem ser utilizadas para representar coisas como rodovias ou rios.
Polígonos - Um polígono é composto de pelo menos três segmentos de linha conectados por pontos. As propriedades de um Polígono são comprimento (o perímetro da área) bem como a própria medida da área. Polígonos podem ser utilizado para demarcar uma área (uma vila) ou uma estrutura (a área de um hospita ).
Dados Rasterizados - Um formato alternativo para dados espaciais, dados rasterizados são uma matriz de células (por exemplo, pixels) em que cada célula contém informações como altura, temperatura, inclinação, cobertura florestal, etc. Essas geralmente são fotos aéreas, imagens de satélite, etc. Os “Rasters” também podem ser utilizados como “mapas base” sob dados vetoriais.
Visualizando dados espaciais
Para representar visualmente os dados espaciais em um mapa, os softwares GIS exigem que você forneça informação suficiente sobre onde posicionar cada característica em relação umas às outras. Se você estiver utilizando dados vetoriais, o que vai acontecer na maioria dos casos, essas informações normalmente estarão armazenadas em um “shapefile”.
Shapefiles - Um shapefile é um formato de dado comum para armazenar dados espaciais “vetoriais” que consistem de linhas, pontos ou polígonos. Um único shapefile na verdade é um conjunto de pelo menos três arquivos - .shp, .shx, e .dbf. Todos esses sub-componentes devem estar presente em um mesmo diretório (pasta) para que o shapefile possa ser lido. Esses arquivos associados podem ser comprimidos em uma pasta ZIP para ser enviado por email ou baixado de um site.
O shapefile vai conter informação sobre as propriedades propriamente ditas, bem como onde encontrá-las na superfície da Terra. Isso é importante pois apesar da Terra ser um globo, os mapas são tipicamente bidimensionais; escolhas sobre como “achatar” os dados espaciais podem ter grande impactos no visual e na interpretação do mapa produzido.
Coordinate Reference Systems (CRS) (Sistemas de Referências de Coordenadas) - Um SRC é um sistema baseado em coordenadas utilizado para localizar propriedades geográficas na superfície da Terra. Ele possui alguns componentes principais:
Sistema de Coordenadas - Existem muitos tipos diferentes de sistemas de coordenadas, então certifique-se de qual dos tipos suas coordenadas fazem parte. Graus de latitude/longitude são comuns, mas você pode também ver coordenadas UTM.
Unidades - Saiba qual são as unidades para o seu sistema de coordenadas (exemplo: graus decimais, metros)
Datum - Uma versão modelada específica da Terra. Essa versão vem sendo revisada ao longo dos anos, então certifique-se de que as camadas do seu mapa utilizam o mesmo datum.
Projeção - Uma referência à equação matemática que foi utilizada para projetar a verdadeiramente redonda Terra em uma superfície plana (mapa).
Lembre-se que você pode resumir dados espaciais sem utilizar as ferramentas de mapas mostradas abaixo. Às vezes, uma simples tabela por dados geográficos (distritos, países, etc.) é todo o necessário!
28.3 Iniciando com GIS
Existem alguns itens importantes que você precisará ter ou pensar a respeito para fazer um mapa. Esses incluem:
- Uma base de dados – pode ser em formato de dados espaciais (tais como shapefiles, como mostrado acima) ou pode não ser em formato espacial (apenas um csv).
- Se sua base de dados não estiver em um formato espacial você também vai precisar de uma base de dados de referência. Dados de referência consistem nas representações espaciais de dados e seus atributos relacionados, que deverão incluir material contendo o local e informações sobre o endereço de propriedades específicas.
- Se você estiver trabalhando com fronteiras geográficas pré-definidas (por examplo, regiões administrativas), shapefiles de referências geralmente estão disponíveis gratuitamente para download nos sites das agências governamentais ou organizações de compartilhamento de dados. Se estiver em dúvida, um bom jeito de começar é buscar no Google por “[região] shapefile”
- Se você tiver informação sobre endereços, mas não tiver latitude e longitude, talvez você precisa utilizar um motor de geocodificação (geocoding engine) para coletar os dados de referências espaciais para seus regristros.
- Uma ideia sobre como você quer apresentar a informação em suas bases para seu público alvo. Existem muitos tipos diferentes de mapas, e é importante pensar sobre qual se encaixa melhor à suas necessidades.
Tipos de mapas para visualizar seus dados
Mapa coroplético (Choropleth map) - um tipo de mapa temático onde cores, sombreamento ou padrões são utilizados para representar regiões geográficas de acordo com seus valores de um atributo. Por exemplo, um valor maior pode ser indicado por uma cor mais escura que um valor menor. Esse tipo de mapa é particularmente útil quando estamos visualizando uma variável e como seu valor muda através de regiões definidas ou áreas geopolíticas.
Mapa de calor de densidade de casos (Case density heatmap) - um tipo de mapa temático em que as cores são utilizada para representar a intensidade de um valor, no entanto, ele não utiliza regiões definidas ou fronteiras geopolíticas para agrupar os dados. Esse tipo de mapa é tipicamente utilizado para mostrar ‘hot spots’ ou áreas com uma alta densidade ou concentração de pontos.
Mapa de densidade de pontos (Dot density map) - um tipo de mapa temático que usa pontos para reprensentar valores de atributos nos seus dados. Esse tipo de mapa é melhor utilizado para visualizar o espalhamento dos dados e permitir um “escaneamento” visual de agrupamentos (clusters).
Mapa de símbolos proporcionais (mapa de símbolos graduados) (Proportional symbols map (graduated symbols map)) - um mapa temático similar ao mapa de coroplético mas, em vez de utilizar cores para indicar o valor de um atributo, ele utiliza um símbolo (geralmente um círculo) de acordo com o valor. Por exemplo, um valor maior pode ser indicado por um símbolo maior que um valor menor. Esse tipo de mapa é melhor utilizado quando se quer visualizar o tamanho ou quantidade dos seus dados através de regiões geográficas.
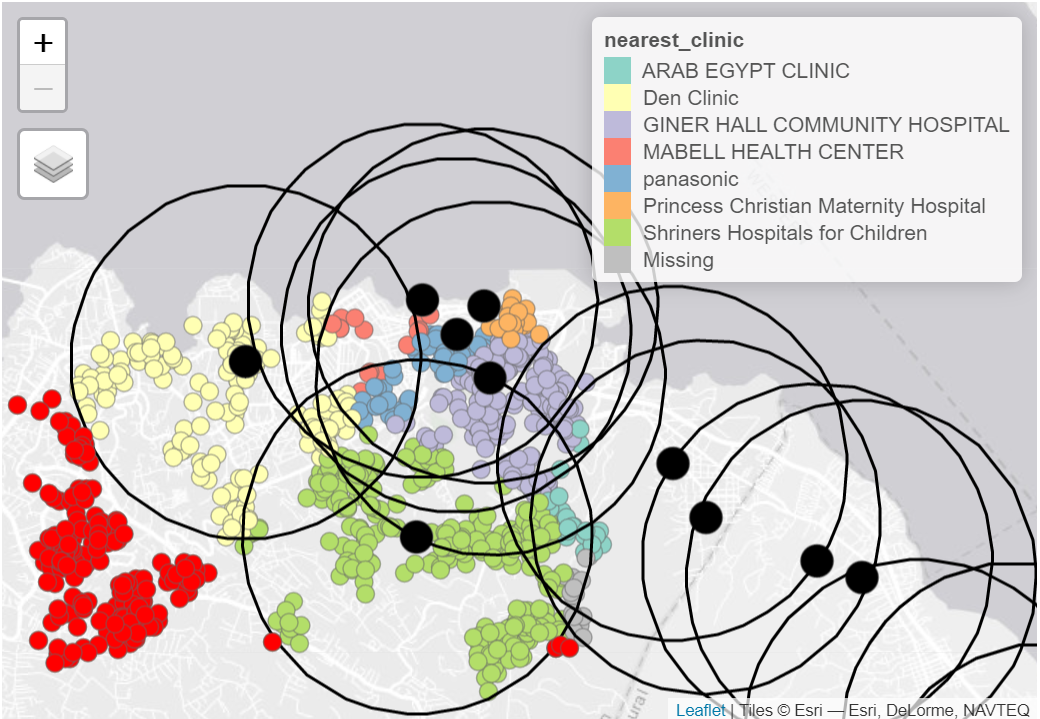
Você também pode combinar vários tipos diferentes de visualização para mostrar padrões geográficos complexos. Por exemplo, os casos (pontos) no mapa abaixo estão coloridos de acordo com a unidade de saúde mais próxima (veja a legenda). Os círculos maiores mostram áreas de alcance das unidades de saúde a partir de um certo raio e os pontos vermelho-vivo mostram os casos fora do alcance de qualquer uma das unidades de saúde mostradas:
Nota: O foco principal dessa página sobre GIS é baseado no contexo de uma resposta de campo a um surto. Por isso, os conteúdos da página irão cobrir o básico de manipulação, visualização e análise de dados espaciais.
28.4 Preparação
Carregue os pacotes R
O código abaixo realiza o carregamento dos pacotes necessários para a análise dos dados. Neste manual, enfatizamos o uso da função p_load(), do pacman, que instala os pacotes, caso não estejam instalados, e os carrega no R para utilização. Também é possível carregar pacotes instalados utilizando a função library(), do R base. Para mais informações sobre os pacotes do R, veja a página Introdução ao R.
pacman::p_load(
rio, # para importar os dados
here, # para localizar arquivos
tidyverse, # para limpar, manipular e gerar gráficos com os dados (inclui o pacote ggplot2)
sf, # para gerenciar dados espaciais utilizando o formato Simple Feature
stars, # para produzir mapas simples, funciona tanto para mapas interativos quanto estáticos
janitor, # para limpar nomes das colunas
terra, #dependencia tmap
tmap,
OpenStreetMap, # para adicionar o mapa-base do OpenStreeMap ao mapa do ggplot
tmaptools,
spdep
) Você pode ter uma visão geral de todos os pacotes do R que lidam com dados espaciais em CRAN “Spatial Task View”.
Amostra de dados dos casos
Para fins de demonstração, iremos trabalhar com uma amostra aleatória de 1000 casos do dataframe da linelist de uma epidemia simulada de Ebola (computacionalmente, trabalhar com menos casos é mais fácil para mostrar nesse handbook). Se você quiser acompanhar a análise, clique aqui para baixar a linelist “limpa” (como um arquivo .rds).
Uma vez que estamos tomando uma amostra aleatória dos casos, seus resultados podem parecer ligeiramente diferentes do que está demonstrado aqui quando você rodar os códigos por conta própria.
Importe os dados com a função import() do pacote rio (ela lida com vários tipos de arquivo como .xlsx, .csv, .rds - veja a página Importar and exportar para mais detalhes).
# importa a linelist de casos limpa
linelist <- rio::import(here::here("data", "case_linelists", "linelist_cleaned.rds")) Next we select a random sample of 1000 rows using sample() from base R.
# gera 1000 números aleatórios de linhas, a partir do número de linhas da linelist
sample_rows <- sample(nrow(linelist), 1000)
# gera um subconjunto da linelist para manter apenas as linhas da amostra, e todas as colunas
linelist <- linelist[sample_rows,]Queremos converter essa linelist, que possui a classe dataframe, para um objeto da classe “sf” (spatial features). Dado que a linelist possui duas colunas “lon” e “lat” representando a longitude e latitude da casa de cada caso, isso será fácil.
Utilizamos o pacote sf (spatial features) e sua função st_as_sf() para criar um novo objeto que chamaremos de linelist_sf. Esse novo objeto parece essencialmente a mesma coisa que a linelist, mas as colunas lon e lat foram designadas como colunas de coordenadas, e um sistema de referência de coordenadas (SRC) foi atribuído para quando os pontos forem mostrados. O número 4326 identifica nossas coordenadas de acordo com o World Geodetic System 1984 (WGS84) - que é o padrão para coordenadas de GPS.
# Cria um objeto do tipo sf
linelist_sf <- linelist %>%
sf::st_as_sf(coords = c("lon", "lat"), crs = 4326)O dataframe da linelist original tem esta forma. Nesta demonstração, iremos utilizar apenas a coluna date_onset e geometry (que foi construída a partir dos campos de longitude e latitude acima e são as últimas colunas no dataframe).
DT::datatable(head(linelist_sf, 10), rownames = FALSE, options = list(pageLength = 5, scrollX=T), class = 'white-space: nowrap' )Shapefiles das fronteiras administrativas
Serra Leoa: Shapefiles das fronteiras administrativas
De antemão, já baixamos todas as fronteiras administrativas de Serra Leoa do site da Humanitarian Data Exchange (HDX) que você encontra aqui. Caso prefira, você pode baixar esse e todos os outros dados de exemplos para esse manual através de nosso pacote de R, conforme explicado na página Download dos dados no manual.
Agora faremos o seguinte para salvar no R o shapefile do nível administrativo 3
- Importar o shapefile
- Limpar o nome das colunas
- Filtar linhas para manter apenas as áreas de interesse
Para importar um shapefile usamos a função read_sf() do pacote sf. À ela é fornecido o caminho do arquivo através da função here(). - em nosso caso o arquivo está em nosso projeto R, nas subpastas “data”, “gis”, e “shp”, com o nome de arquivo “sle_adm3.shp” (veja as páginas Importar e exportar e Projetos R para mais informações). Você precisará fornecer seu próprio caminho de arquivo.
Em seguida usamos a função clean_names() do pacote janitor para padronizar os nomes das colunas do shapefile. Também utilizamos filter() para manter apenas as linhas com a área administrativa 2 (admin2name) chamada “Western Area Urban” ou “Western Area Rural”.
# nível ADM3 limpo
sle_adm3 <- sle_adm3_raw %>%
janitor::clean_names() %>% # padronizar o nome das colunas
filter(admin2name %in% c("Western Area Urban", "Western Area Rural")) # filtrar para manter certas áreasAbaixo, podemos ver como fica o shapefile depois da importação e limpeza. Role para a direita para ver como há colunas com nível administrativo 0 (país), nível administrativo 1, nível administrativo 2, e finalmente nível administrativo 3. Cada nível tem um nome composto de letras e um identificador único chamado “pcode”. O pcode expande com cada nível administrativo que aumenta ex: SL (Serra Leoa) -> SL04 (Oeste) -> SL0410 (Area Rural do Oeste) -> SL040101 (Koya Rural).
Dados populacionais
Serra Leoa: População por ADM3
Novamente, esses dados podem ser baixadados do site da HDX (link aqui) ou pelo pacote epirhandbook explicado nessa página. Usamos import() para carregar o arquivo .csv. Também passamos o arquivo importado para clean_names() para padronizar a sintaxe dos nomes das colunas.
# População por nível ADM3
sle_adm3_pop <- import(here("data", "gis", "population", "sle_admpop_adm3_2020.csv")) %>%
janitor::clean_names()Essa é a aparência do arquivo de população. Role para a direita para ver como cada jurisdição tem colunas com a população masculina (male), feminina (female), e a população total, e as subdivisões nas colunas columns por grupo de idade.
Unidades de Saúde
Serra Leoa: Dados das Unidades de Saúde do OpenStreetMap
Novamente, baixamos os locais das unidades de saúde do HDX aqui ou por meio das instruções na página manual de Download e dados.
Nós importamos o shapefile dos pontos das unidades com read_sf(), e novamente limpamos os nomes das colunas, e depois, filtramos para manter apenas os pontos marcados com “hospital”, clínica (“clinic”), ou doutores (“doctors”).
# shapefile das unidades de saúde do OSM (open street map)
sle_hf <- sf::read_sf(here("data", "gis", "shp", "sle_hf.shp")) %>%
janitor::clean_names() %>%
filter(amenity %in% c("hospital", "clinic", "doctors"))Aqui está o dataframe resultante - role para a direita para ver o nome da unidade e as coordenadas de geometria (geometry).
28.5 Criando gráficos das coordenadas
A forma mais fácil de criar um gráfico de coordenadas X-Y (longitude/latitude, pontos), nessa situação dos casos, é desenhenhá-los como pontos diretamente do objeto linelist_sf que criamos na seção de preparação.
O pacote tmap oferece algumas capacidades simples de fazer mapas tanto de forma estática ( modo “plot”) quanto interativa ( modo de visualização - “view”) com apenas algumas linhas de código. A sintaxe do tmap é parecida com a do ggplot2, de forma que os comandos são somados uns aos outros com o operador +. Veja mais detalhes nessa vignette.
- Defina o modo do tmap. Neste caso iremos utilizar o modo “plot”, que produz saídas estáticas.
tmap_mode("plot") # escolha entre "view" ou "plot"Abaixo, os pontos são projetados sozinhos. A função tm_shape() é disponibilizada com os objetos linelist_sf. Em seguida, adicionamos pontos via tm_dots(), especificando o tamanho e a cor. Pelo fato de linelist_sf ser um objeto do tipo sf, nós já temos as duas colunas designadas que contêm as coordenadas de latitude e longitude e o sistema de referência de coordenadas (CRS):
Sozinhos os pontos não nos dizem muita coisa. Então devemos também projetar as fronteiras administrativas:
Novamente, utilizaremos tm_shape() (veja a documentação) mas em vez de fornecer o shapefile com os pontos dos casos, forneceremos o shapefile das fronteiras administrativas (polygons).
Com o argumento bbox = (bbox significa “bounding box”) podemos especificar as coordenadas das fronteiras. Primeiro mostramos o mapa sem bbox, e depois com ele.
# Apenas as fronteiras administrativas (polígonos)
tm_shape(sle_adm3) + # shapefile das fronteiras administrativas
tm_polygons(col = "#F7F7F7") + # exibir os polígonos em cinza claro
tm_borders(col = "#000000", # exibir as bordas com cor e peso de linha
lwd = 2) +
tm_text("admin3name") # coluna de texto para mostrar para cada polígono
# Mesmo que acima, mas com zoom à partir do bounding box
tm_shape(sle_adm3,
bbox = c(-13.3, 8.43, # vértice (do bounding box)
-13.2, 8.5)) + # vértice (do bounding box)
tm_polygons(col = "#F7F7F7") +
tm_borders(col = "#000000", lwd = 2) +
tm_text("admin3name")E agora ambos, pontos e polígonos juntos:
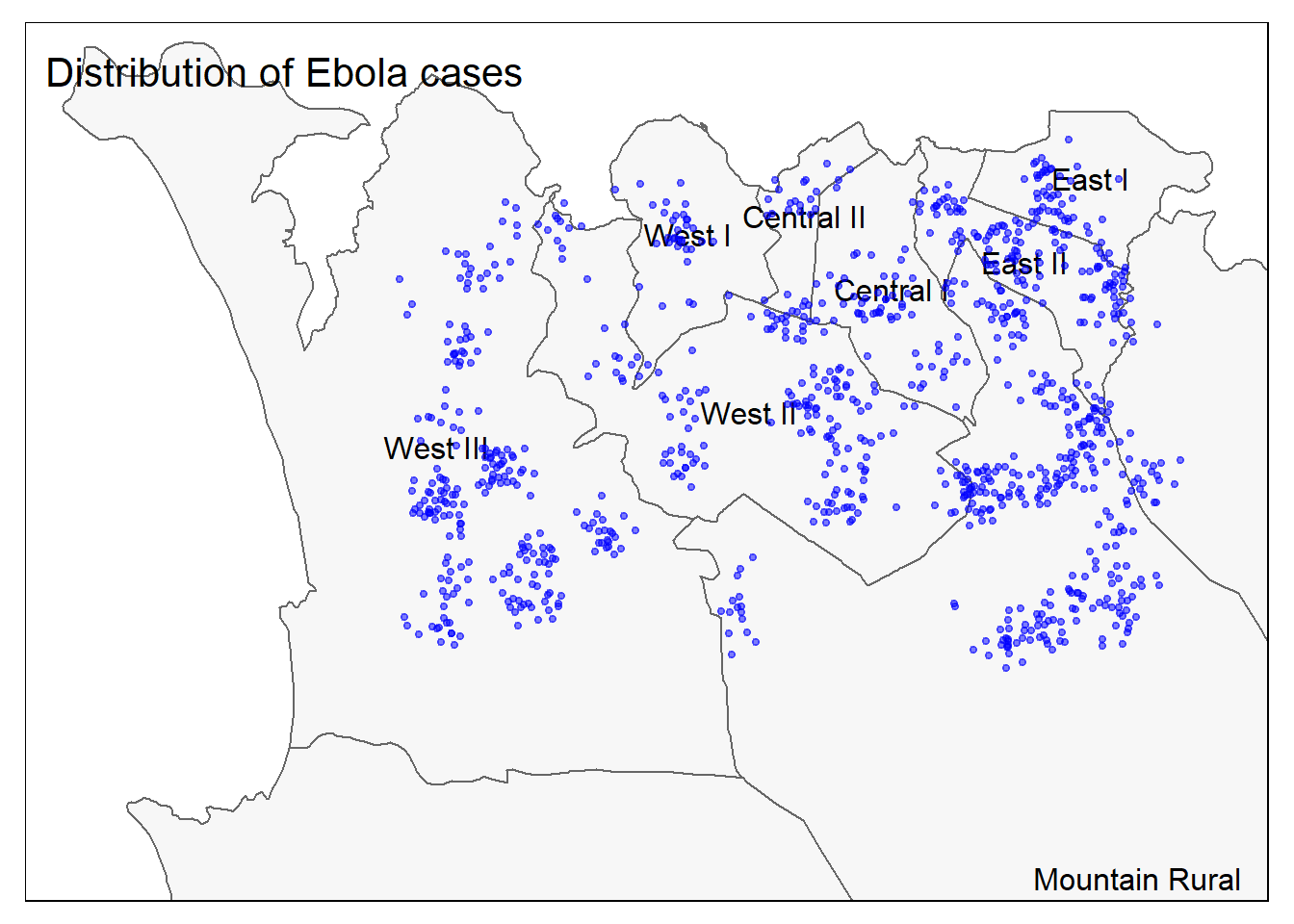
# Todos juntos
tm_shape(sle_adm3, bbox = c(-13.3, 8.43, -13.2, 8.5)) + #
tm_polygons(col = "#F7F7F7") +
tm_borders(col = "#000000", lwd = 2) +
tm_text("admin3name")+
tm_shape(linelist_sf) +
tm_dots(size=0.08, col='blue', alpha = 0.5) +
tm_layout(title = "Distribution of Ebola cases") # Dá título ao gráfico Para ler uma boa comparação das diferentes opções de criação de mapas no R, leia esse blog post.
28.6 “Joins” Espaciais
Você deve estar familiarizado com o conceito de fazer join dos dados de uma base em outra. Vários métodos são discutidos na página Juntando dados desse manual. Um “join espacial” tem um propósito similar, mas lidando com os relacionamentos espaciais dos dados. Em vez de utilizar valores comuns às colunas para parear corretamente as observações, você pode utilizar as relações espaciais deles, tais como o fato de uma característica estar contida em outra, ou de um ponto ser o vizinho mais próximo de outro, ou contido na área de influência (buffer) de um certo raio de outro, etc.
O pacote sf oferece vários métodos para fazer joins espaciais. Veja mais na documentação sobre o métodos st_join e outros tipos de join espaciais nessa referência.
Pontos no polígono
Relaciona unidades administrativas aos casos
Aqui temos um dilema interessante: a linelist dos casos não contém nenhuma informação sobre as unidades administrativas dos casos. Apesar do ideal ser coletar esse tipo de informação nas fases iniciais da coleta de dados, nós também podemos relacionar as unidades administrativas aos casos individuais baseado em seus relacionamentos espaciais (ex: pontos que intersectam com um polígono).
Abaixo, vamos fazer a intersecção espacial dos locais de nossos casos (pontos) com as fronteiras ADM3 (polígonos):
- Comece com a linelist (pontos)
- Faça o join espacial com as fronteiras, defina o tipo do join em “st_intersects”
- Utilize
select()para manter apenas algumas das novas colunas das fronteiras administrativas
linelist_adm <- linelist_sf %>% # faça join do arquivo com as fronteiras administrativas com o da linelist, baseado na intersecção espacial
sf::st_join(sle_adm3, join = st_intersects)Todas as colunas de sle_adms foram adicionadas à linelist! Agora cada caso possui colunas detalhando os níveis administativos onde eles se encontram. Nesse exemplo, queremos manter apenas duas das novas colunas (admin level 3), então usamos select() nos nomes das colunas antigas e apenas nas duas novas que temos interesse:
linelist_adm <- linelist_sf %>% # faça join do arquivo com as fronteiras administrativas com o da linelist, baseado na intersecção espacial
sf::st_join(sle_adm3, join = st_intersects) %>%
# Mantém os nomes das colunas antigas e as duas novas de interessa das áreas administrativas
select(names(linelist_sf), admin3name, admin3pcod)Você pode ver abaixo, apenas para fins de conferência, os primeiros dez casos e as jurisdições de seus níveis administrativos 3 (ADM3) que foram anexadas baseados em onde o ponto intersectou espacialmente com as formas dos polígonos.
# Agora você verá os nomes AD3 anexados a cada caso.
linelist_adm %>% select(case_id, admin3name, admin3pcod)Simple feature collection with 1000 features and 3 fields
Geometry type: POINT
Dimension: XY
Bounding box: xmin: -13.27074 ymin: 8.447887 xmax: -13.20522 ymax: 8.491748
Geodetic CRS: WGS 84
First 10 features:
case_id admin3name admin3pcod geometry
3693 9c3676 East III SL040205 POINT (-13.2103 8.466647)
3521 5fbeea West III SL040208 POINT (-13.26599 8.473993)
4363 8e0af5 West II SL040207 POINT (-13.2459 8.468553)
3159 a509e6 Central I SL040201 POINT (-13.23163 8.478547)
3544 f5bc7b West III SL040208 POINT (-13.25092 8.45933)
1472 9fcf61 West III SL040208 POINT (-13.26314 8.465306)
341 8968ee West II SL040207 POINT (-13.24839 8.465598)
3802 bc6562 Mountain Rural SL040102 POINT (-13.21879 8.463824)
4230 a5b60c West II SL040207 POINT (-13.2487 8.470481)
128 6a007d West II SL040207 POINT (-13.23488 8.467028)Agora podemos descrever nossos casos por unidade administrativa - algo que não era possível antes do join espacial!
# Cria um novo dataframe contendo as contagens de casos por unidade administrativa
case_adm3 <- linelist_adm %>% # inicia com a lineliste contendo as novas colunas administrativas
as_tibble() %>% # converte para tibble para melhor exibição
filter(!is.na(admin3pcod))%>%
group_by(admin3pcod, admin3name) %>% # agrupa por unidade administrativa, por nome e pcode
summarise(cases = n()) %>% # resume e conta as linhas
arrange(desc(cases)) # organiza em ordem descentente
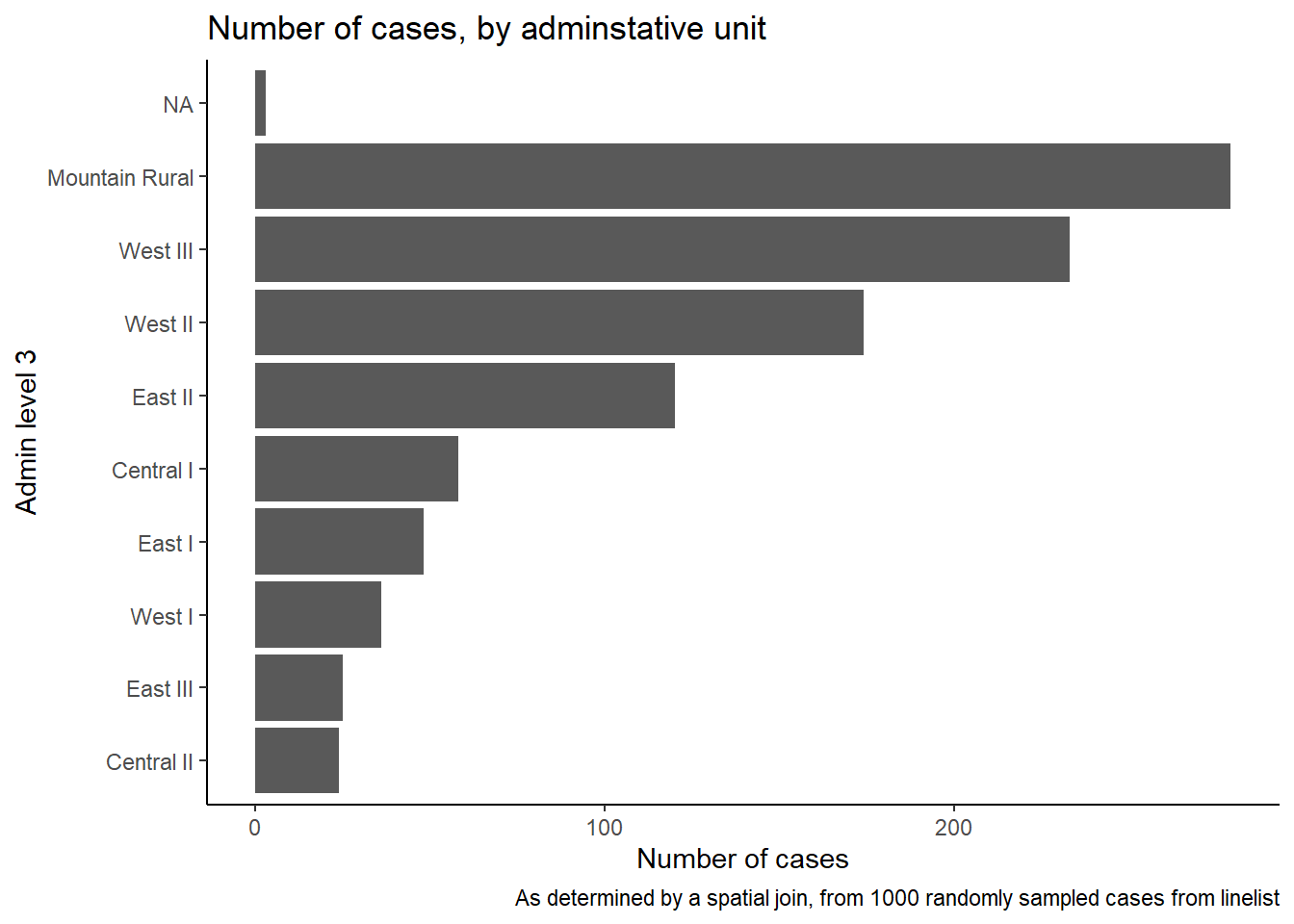
case_adm3# A tibble: 9 × 3
# Groups: admin3pcod [9]
admin3pcod admin3name cases
<chr> <chr> <int>
1 SL040102 Mountain Rural 279
2 SL040208 West III 233
3 SL040207 West II 174
4 SL040204 East II 120
5 SL040201 Central I 58
6 SL040203 East I 48
7 SL040206 West I 36
8 SL040205 East III 25
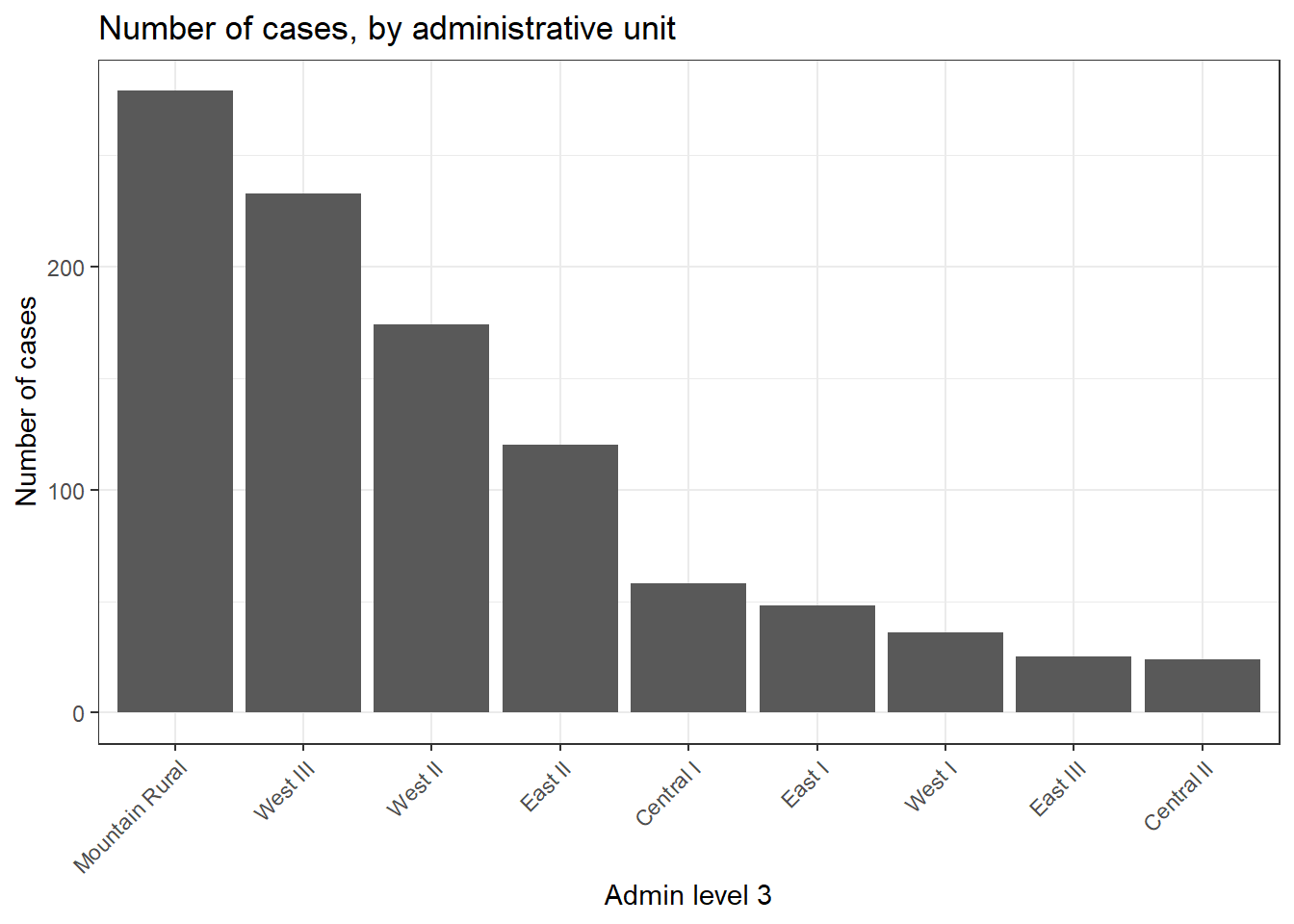
9 SL040202 Central II 24Também podemos criar um gráfico de barras com as contagens dos casos por unidade administrativa.
Nesse exemplo, iniciamos o ggplot() com linelist_adm, de forma que podemos aplicar as funções de fatores como fct_infreq() que ordena as barras por frequência (veja a página de Fatores para dicas).
ggplot(
data = linelist_adm, # inicia com linelist contendo informações das unidades administrativas
mapping = aes(
x = fct_rev(fct_infreq(admin3name))))+ # eixo x são unidades administrativas, ordenadas por frequência (reversa)
geom_bar()+ # cria as barras, altura é o número de linhas
coord_flip()+ # inverte os eixos X e Y para facilitar a leitura das unidades administrativas
theme_classic()+ # simplifica a cor de fundo do gráfico
labs( # títulos e rótulos
x = "Admin level 3",
y = "Number of cases",
title = "Number of cases, by adminstative unit",
caption = "As determined by a spatial join, from 1000 randomly sampled cases from linelist"
)Vizinho mais próximo
Achando a unidade de saúde / área de influência mais próxima
Pode ser útil saber onde as unidades de saúde estão localizadas em relação aos hotspots das doenças.
Podemos utilizar o método de join st_nearest_feature da função st_join() (do pacote sf) para visualizar as unidades de saude mais próximas aos casos individuais.
- Iniciamos com o shapefile da linelist:
linelist_sf
- Fazemos o join espacial com
sle_hf, que possui os locais das unidades de saúde e clínicas (pontos)
# Unidade de saúde mais próxima a cada caso
linelist_sf_hf <- linelist_sf %>% # inicia com o shapefile da linelist
st_join(sle_hf, join = st_nearest_feature) %>% # dados da clínica mais próxima com join nos dados dos casos
select(case_id, osm_id, name, amenity) %>% # mantém as colunas de interesse, incluindo id, nome, tipo, e geometria da unidade de saúde
rename("nearest_clinic" = "name") # renomeia para uma maior clarezaPodemos ver abaixo (primeiras 50 linhas) que cada caso agora possui dados sobre as clínicas/hospitais mais próximos
Podemos ver que “Den Clinic” é a unidade de saúde mais próxima de cerca de 30% dos casos.
# Conta os casos por unidade de saúde
hf_catchment <- linelist_sf_hf %>% # inicia com a linelist incluindo os dados das clínicas mais próximas
as.data.frame() %>% # converte o shapefile em dataframe
count(nearest_clinic, # conta linhas por nome (da clínica)
name = "case_n") %>% # define a nova coluna de contagens como "case_n"
arrange(desc(case_n)) # organiza em ordem descrescente
hf_catchment # imprime no console nearest_clinic case_n
1 Den Clinic 355
2 Shriners Hospitals for Children 326
3 GINER HALL COMMUNITY HOSPITAL 173
4 panasonic 59
5 ARAB EGYPT CLINIC 29
6 Princess Christian Maternity Hospital 29
7 MABELL HEALTH CENTER 17
8 <NA> 12Para visualizar os resultados, podemos utilizar tmap - dessa vez, no modo interativo para facilitar a visualização
tmap_mode("view") # define o modo do tmap para interativo
# gera o gráfico dos pontos dos casos e clínicas
tm_shape(linelist_sf_hf) + # gera o gráfico dos casos
tm_dots(size=0.08, # colore os casos pelas clínicas mais próximas
col='nearest_clinic') +
tm_shape(sle_hf) + # adiciona as clínicas ao gráfico como grandes pontos pretos
tm_dots(size=0.3, col='black', alpha = 0.4) +
tm_text("name") + # sobrepõe com o nome
tm_view(set.view = c(-13.2284, 8.4699, 13), # ajusta o zoom (centra as coordenadas, e define o zoom)
set.zoom.limits = c(13,14))+
tm_layout(title = "Cases, colored by nearest clinic")Áreas de Influência (Buffers)
Também podemos explorar quantos casos estão localizados em uma distância a pé de 2.5km (~30 min) da unidade de saúde mais próxima.
Nota: Para cálculos de distância mais precisos, é melhor re-projetar seu objeto sf ao sistema de projeção do local respectivo, tal como UTM (Terra projetada sobre uma superfície plana). Nesse exemplo, para fins de simplicidade vamos nos ater ao sistema de coordenadas do Sistema Geodésico Mundial (WGS84) (Terra representada em uma superfície esférica / redonda, portanto, as unidades estão em graus decimais). Utilizaremos uma conversão geral de: 1 grau decimal = ~111km.
Veja mais informações sobre projeções de mapas e sistemas de coordenadas nesse artigo da esri. Esse blog fala sobre diferentes tipos de projeções de mapas e como cada uma delas pode ser escolhida dependendo da área de interesse e o contexto do seu mapa / análise.
Primeiro, crie uma área de influência circular com um raio de ~2.5km ao redor de cada unidade de saúde. Isso é feito com a função st_buffer() do pacote tmap. Pelo fato das unidades do mapa estarem em lat/long em graus decimais, é assim que “0.02” será interpretado. Se o sistema de coordenadas do seu mapa for em metros, o número deve ser fornecido em metros.
sle_hf_2k <- sle_hf %>%
st_buffer(dist=0.02) # graus decimais representando aprox. 2.5km Abaixo criamos o gráfico das zonas de influência propriamente ditas, com:
tmap_mode("plot")
# Cria as zonas de influência circulares
tm_shape(sle_hf_2k) +
tm_borders(col = "black", lwd = 2)+
tm_shape(sle_hf) + # adiciona as clínicas ao gráfico como grandes pontos vermelhos
tm_dots(size=0.3, col='black') Segundo, intersectamos essas áreas de influência com os casos (pontos) utilizando st_join() e com o tipo de join st_intersects. Ou seja, join dos dados das áreas de influência com os pontos que eles intersectam.
# Intersecta os casos com as áreas de influência
linelist_sf_hf_2k <- linelist_sf_hf %>%
st_join(sle_hf_2k, join = st_intersects, left = TRUE) %>%
filter(osm_id.x==osm_id.y | is.na(osm_id.y)) %>%
select(case_id, osm_id.x, nearest_clinic, amenity.x, osm_id.y)Agora podemos contar os resultados: nrow(linelist_sf_hf_2k[is.na(linelist_sf_hf_2k$osm_id.y), ]) dos 1000 casos não intersectam com nenhuma área de influência (esses valores estão faltando), e então residem a mais de 30 min a pé da da unidade de saúde mais próxima.
# Casos que não intesectaram com nenhuma das áreas de influência das unidades de saúde
linelist_sf_hf_2k %>%
filter(is.na(osm_id.y)) %>%
nrow()[1] 1000Podemos visualizar os resultados de forma que os casos que não intersectam apareçam em vermelho.
tmap_mode("view")
# Primeiro mostra os casos como pontos
tm_shape(linelist_sf_hf) +
tm_dots(size=0.08, col='nearest_clinic') +
# adiciona as clínicas como grandes pontos pretos
tm_shape(sle_hf) +
tm_dots(size=0.3, col='black')+
# Depois sobrepõe as áreas de influência das unidades de saúde como polilinhas
tm_shape(sle_hf_2k) +
tm_borders(col = "black", lwd = 2) +
# Destaca os casos que não são parte de nenhuma área de influência das unidades de saúde
# como pontos vermelhos
tm_shape(linelist_sf_hf_2k %>% filter(is.na(osm_id.y))) +
tm_dots(size=0.1, col='red') +
tm_view(set.view = c(-13.2284,8.4699, 13), set.zoom.limits = c(13,14))+
# adiciona título
tm_layout(title = "Cases by clinic catchment area")Outros joins espaciais
Valores alternativos para o argumento join incluem (a partir da documentação)
- st_contains_properly
- st_contains
- st_covered_by
- st_covers
- st_crosses
- st_disjoint
- st_equals_exact
- st_equals
- st_is_within_distance
- st_nearest_feature
- st_overlaps
- st_touches
- st_within
28.7 Mapas coropléticos
Mapas coropléticos podem ser úteis para visualizar seus dados por áreas pré-definidas, geralmente unidades administrativas ou de saúde. Nas respostas aos surtos isso pode ajudar a direcionar a alocação de recursos para áreas específicas com altas taxas de incidência, por exemplo.
Agora que já temos os nomes das unidades administrativas associadas a todos os casos (veja a seção sobre joins espaciais, acima), podemos começar a mapear as contagens dos casos por área (mapas coropléticos)
Uma vez que também temos dados de população por ADM3, podemos adicionar essas informações à tabela case_adm3 criada anteriormente.
Vamos começar com o dataframe case_adm3 criado no passo anterior, que é uma tabela resumo com cada unidade administrativa e seus números de casos.
- Os dados populacionais
sle_adm3_popsão obtidos utilizandoleft_join()do pacote dplyr com base nos valores comuns ao longo das colunasadmin3pcodno dataframecase_adm3, e colunaadm_pcodeno dataframesle_adm3_pop. Veja a página Juntando dados).
select()é aplicado ao novo dataframe, para manter apenas as colunas úteis -totalé a população total- Casos por 10,000 é calculado como uma nova coluna com
mutate()
# Adiciona dados populacionais e calcula casos por 10mil
case_adm3 <- case_adm3 %>%
left_join(sle_adm3_pop, # adiciona colunas da base 'pop'
by = c("admin3pcod" = "adm3_pcode")) %>% # faz join baseado nos valores comum ao longo dessas duas colunas
select(names(case_adm3), total) %>% # mantém apenas as colunas importantes, incluindo população total
mutate(case_10kpop = round(cases/total * 10000, 3)) # cria uma nova coluna com taxa de casos por 10000, arredondando para 3 casas decimais
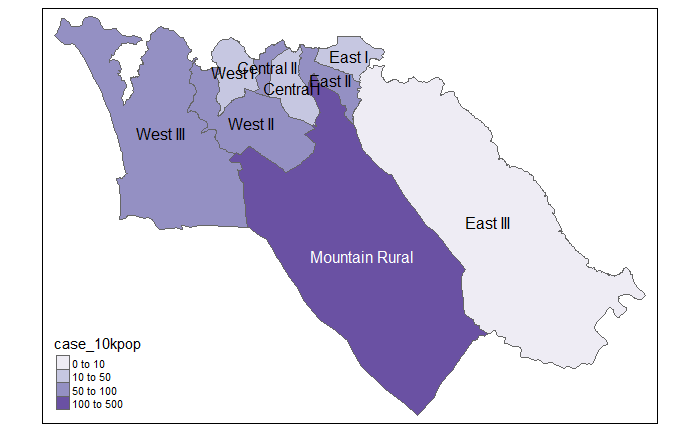
case_adm3 # imprime no console para visualização# A tibble: 9 × 5
# Groups: admin3pcod [9]
admin3pcod admin3name cases total case_10kpop
<chr> <chr> <int> <int> <dbl>
1 SL040102 Mountain Rural 279 33993 82.1
2 SL040208 West III 233 210252 11.1
3 SL040207 West II 174 145109 12.0
4 SL040204 East II 120 99821 12.0
5 SL040201 Central I 58 69683 8.32
6 SL040203 East I 48 68284 7.03
7 SL040206 West I 36 60186 5.98
8 SL040205 East III 25 500134 0.5
9 SL040202 Central II 24 23874 10.1 Faz join dessa tabela com os polígonos do shapefile de ADM3 para poder mapeá-los
case_adm3_sf <- case_adm3 %>% # inicia com casos e taxas por unidade administrativa
left_join(sle_adm3, by="admin3pcod") %>% # faz join dos dados do shapefile por coluna comum
select(objectid, admin3pcod, # mantém apenas algumas colunas de interesse
admin3name = admin3name.x, # limpa o nome de uma coluna
admin2name, admin1name,
cases, total, case_10kpop,
geometry) %>% # mantém a geometria de forma que os polígonos possam ser adicionados ao gráfico
st_as_sf() # converte para shapefileMapeando os resultados
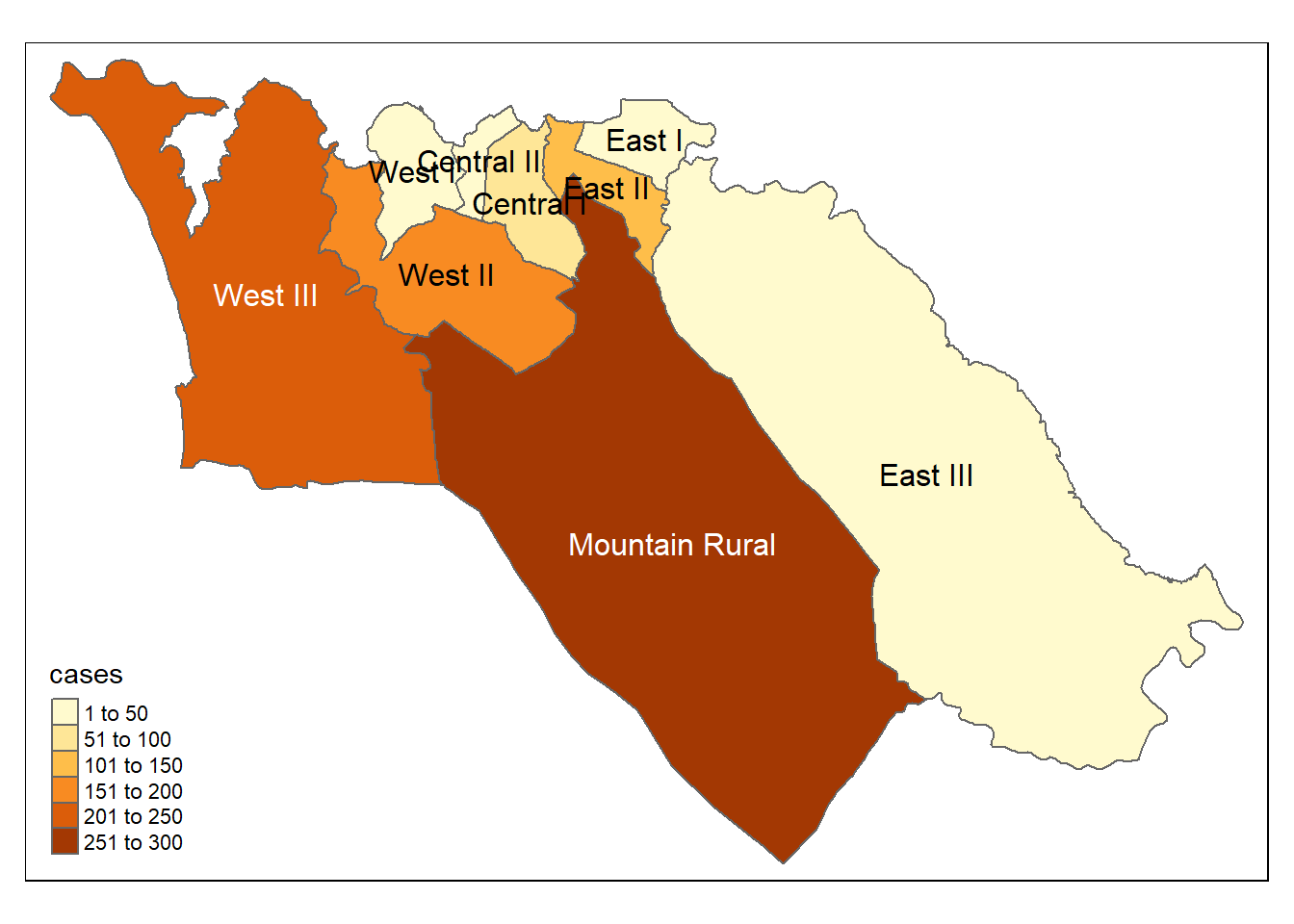
# modo do tmap
tmap_mode("plot") # visualiza o mapa estático
# adiciona os polígonos
tm_shape(case_adm3_sf) +
tm_polygons("cases") + # colore pela coluna de número de casos
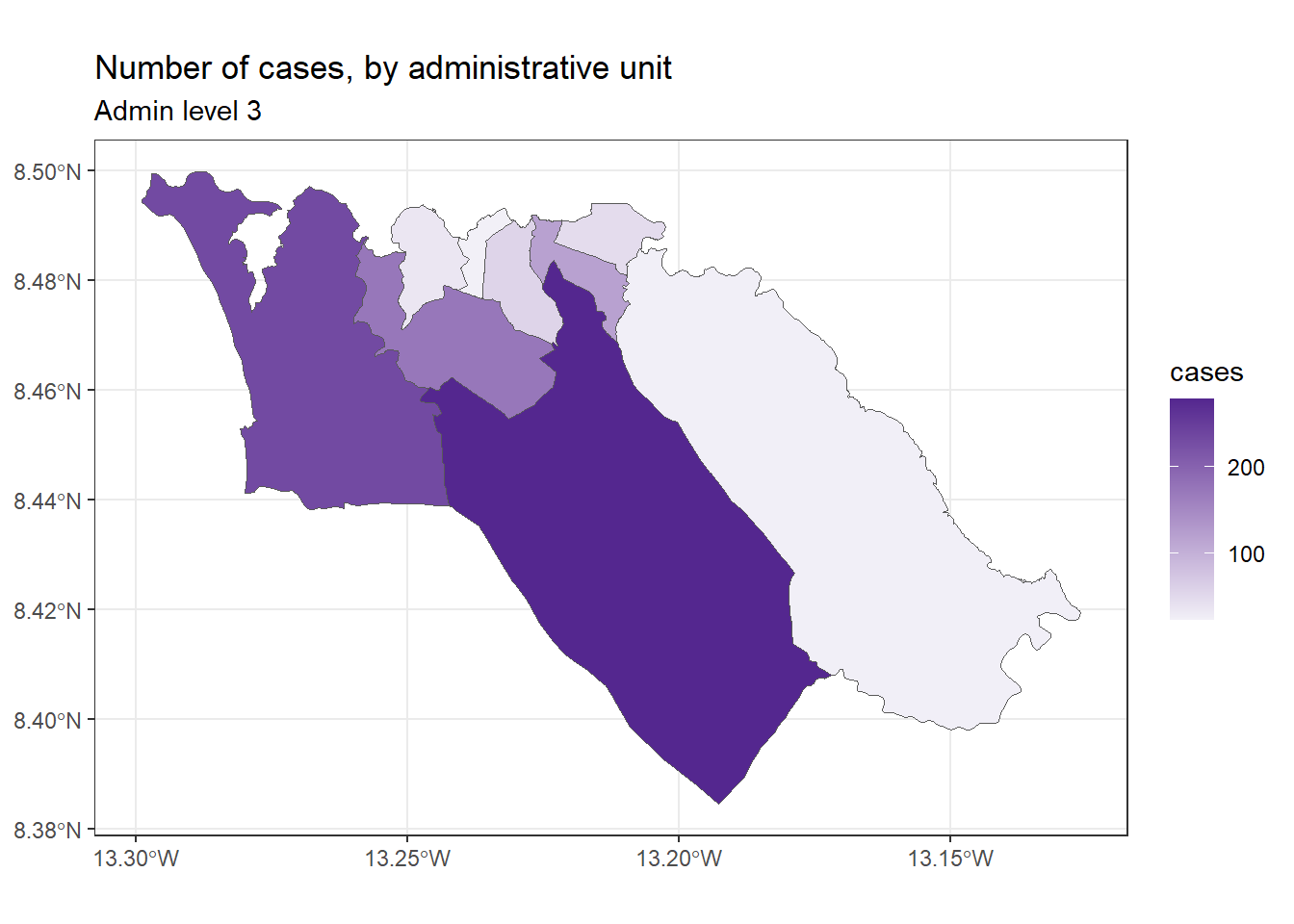
tm_text("admin3name") # nome exibidoTambém podemos mapear as taxas de incidência
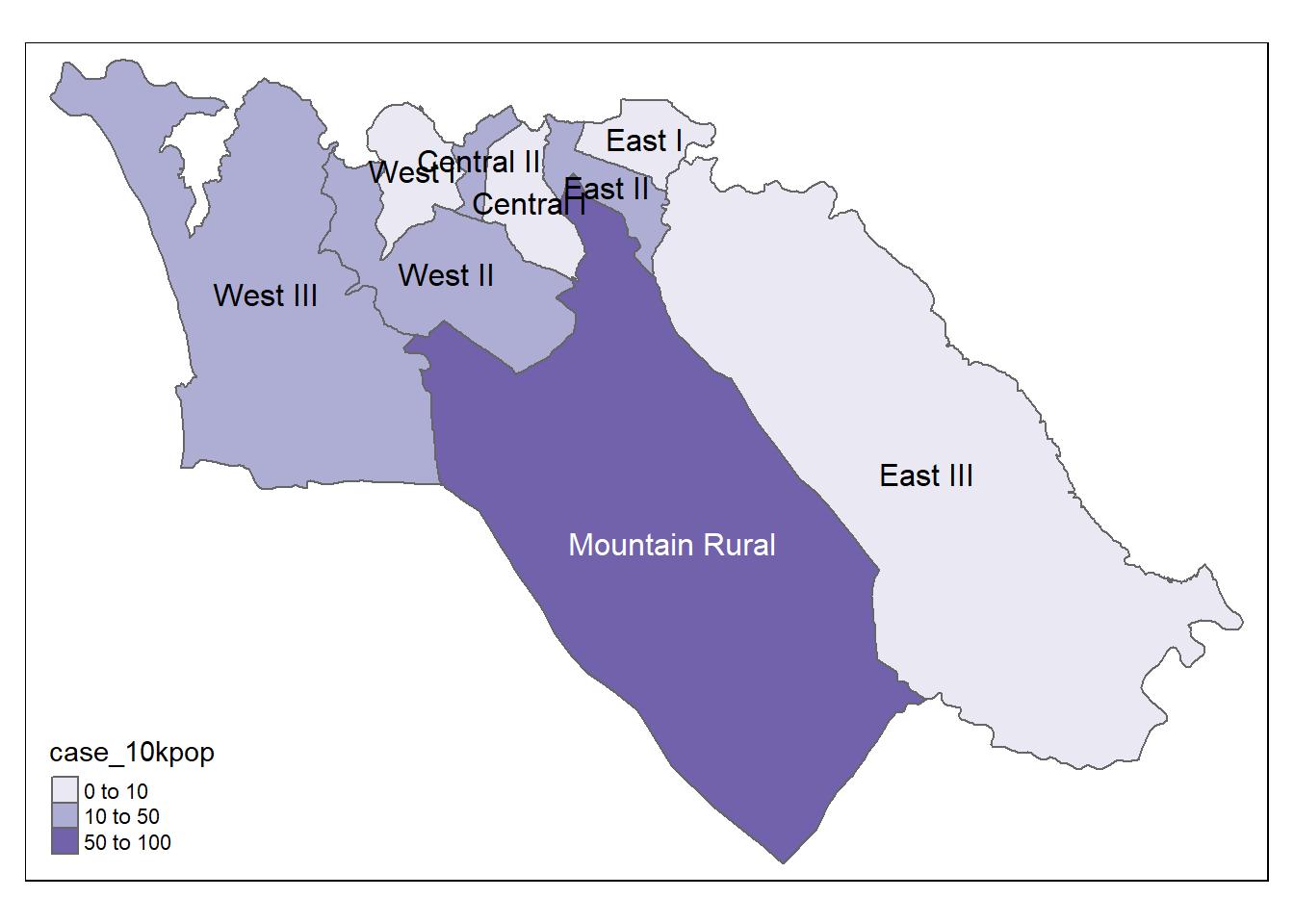
# Casos por 10mil
tmap_mode("plot") # modo de visualização estática
# gráfico
tm_shape(case_adm3_sf) + # adiciona polígonos ao mapa
tm_polygons("case_10kpop", # colore pelas colunas contendo a taxa de casos
breaks=c(0, 10, 50, 100), # define os pontos de quebra para as cores
palette = "Purples" # utiliza uma paleta de cor roxa
) +
tm_text("admin3name") # exibe o texto28.8 Criando mapas com ggplot2
Se você já está familiarizado em usar o ggplot2, você pode usar esse pacote para criar mapas estáticos de seus dados. A função geom_sf() vai desenhar diferentes objetos baseados em quais características (pontos, linhas ou polígonos) estão em seus dados. Por exemplo, você pode usar geom_sf() em um ggplot() utilizando dados sf com geometria de polígonos para criar um mapa coroplético.
Para ilustrar como isso funciona, podemos iniciar com os polígonos do shapefile de ADM3 que utilizamos anteriormente. Lembre-se que esses são regiões de Níveis Administrativos 3 em Serra Leoa:
sle_adm3Simple feature collection with 12 features and 19 fields
Geometry type: MULTIPOLYGON
Dimension: XY
Bounding box: xmin: -13.29894 ymin: 8.094272 xmax: -12.91333 ymax: 8.499809
Geodetic CRS: WGS 84
# A tibble: 12 × 20
objectid admin3name admin3pcod admin3ref_n admin2name admin2pcod admin1name
* <dbl> <chr> <chr> <chr> <chr> <chr> <chr>
1 155 Koya Rural SL040101 Koya Rural Western A… SL0401 Western
2 156 Mountain Ru… SL040102 Mountain R… Western A… SL0401 Western
3 157 Waterloo Ru… SL040103 Waterloo R… Western A… SL0401 Western
4 158 York Rural SL040104 York Rural Western A… SL0401 Western
5 159 Central I SL040201 Central I Western A… SL0402 Western
6 160 East I SL040203 East I Western A… SL0402 Western
7 161 East II SL040204 East II Western A… SL0402 Western
8 162 Central II SL040202 Central II Western A… SL0402 Western
9 163 West III SL040208 West III Western A… SL0402 Western
10 164 West I SL040206 West I Western A… SL0402 Western
11 165 West II SL040207 West II Western A… SL0402 Western
12 167 East III SL040205 East III Western A… SL0402 Western
# ℹ 13 more variables: admin1pcod <chr>, admin0name <chr>, admin0pcod <chr>,
# date <date>, valid_on <date>, valid_to <date>, shape_leng <dbl>,
# shape_area <dbl>, rowcacode0 <chr>, rowcacode1 <chr>, rowcacode2 <chr>,
# rowcacode3 <chr>, geometry <MULTIPOLYGON [°]>Podemos utilizar a função left_join() do pacote dplyr para adicionar dados que queremos mapear ao objeto shapefile. Nesse caso, vamos utilizar o dataframe case_adm3 que criamos anterioremente para resumir a contagem de casos por regiões administrativas; no entanto, podemos utilizar essa mesma abordagem para mapear qualquer dado armazenado no dataframe.
sle_adm3_dat <- sle_adm3 %>%
inner_join(case_adm3, by = "admin3pcod") # inner join = mantém apenas se existir em ambos os objetos
select(sle_adm3_dat, admin3name.x, cases) # imprime as variáveis selecionadas no consoleSimple feature collection with 9 features and 2 fields
Geometry type: MULTIPOLYGON
Dimension: XY
Bounding box: xmin: -13.29894 ymin: 8.384533 xmax: -13.12612 ymax: 8.499809
Geodetic CRS: WGS 84
# A tibble: 9 × 3
admin3name.x cases geometry
<chr> <int> <MULTIPOLYGON [°]>
1 Mountain Rural 279 (((-13.21496 8.474341, -13.21479 8.474289, -13.21465 8.4…
2 Central I 58 (((-13.22646 8.489716, -13.22648 8.48955, -13.22644 8.48…
3 East I 48 (((-13.2129 8.494033, -13.21076 8.494026, -13.21013 8.49…
4 East II 120 (((-13.22653 8.491883, -13.22647 8.491853, -13.22642 8.4…
5 Central II 24 (((-13.23154 8.491768, -13.23141 8.491566, -13.23144 8.4…
6 West III 233 (((-13.28529 8.497354, -13.28456 8.496497, -13.28403 8.4…
7 West I 36 (((-13.24677 8.493453, -13.24669 8.493285, -13.2464 8.49…
8 West II 174 (((-13.25698 8.485518, -13.25685 8.485501, -13.25668 8.4…
9 East III 25 (((-13.20465 8.485758, -13.20461 8.485698, -13.20449 8.4…Para fazer um gráfico de colunas com contagem de casos por região, utilizando ggplot2, podemos então utilizar geom_col() dessa forma:
ggplot(data=sle_adm3_dat) +
geom_col(aes(x=fct_reorder(admin3name.x, cases, .desc=T), # reordena o eixo x 'cases' de forma decrescente
y=cases)) + # eixo y é o número de casos por região
theme_bw() +
labs( # define o texto da figura
title="Number of cases, by administrative unit",
x="Admin level 3",
y="Number of cases"
) +
guides(x=guide_axis(angle=45)) # angula os rótulos do eixo x para 45 graus para caber melhorSe quisermos utilizar o ggplot2 para fazer um mapa coroplético da contagem dos casos, podemos utilizar uma sintaxe parecida para chamar a função geom_sf():
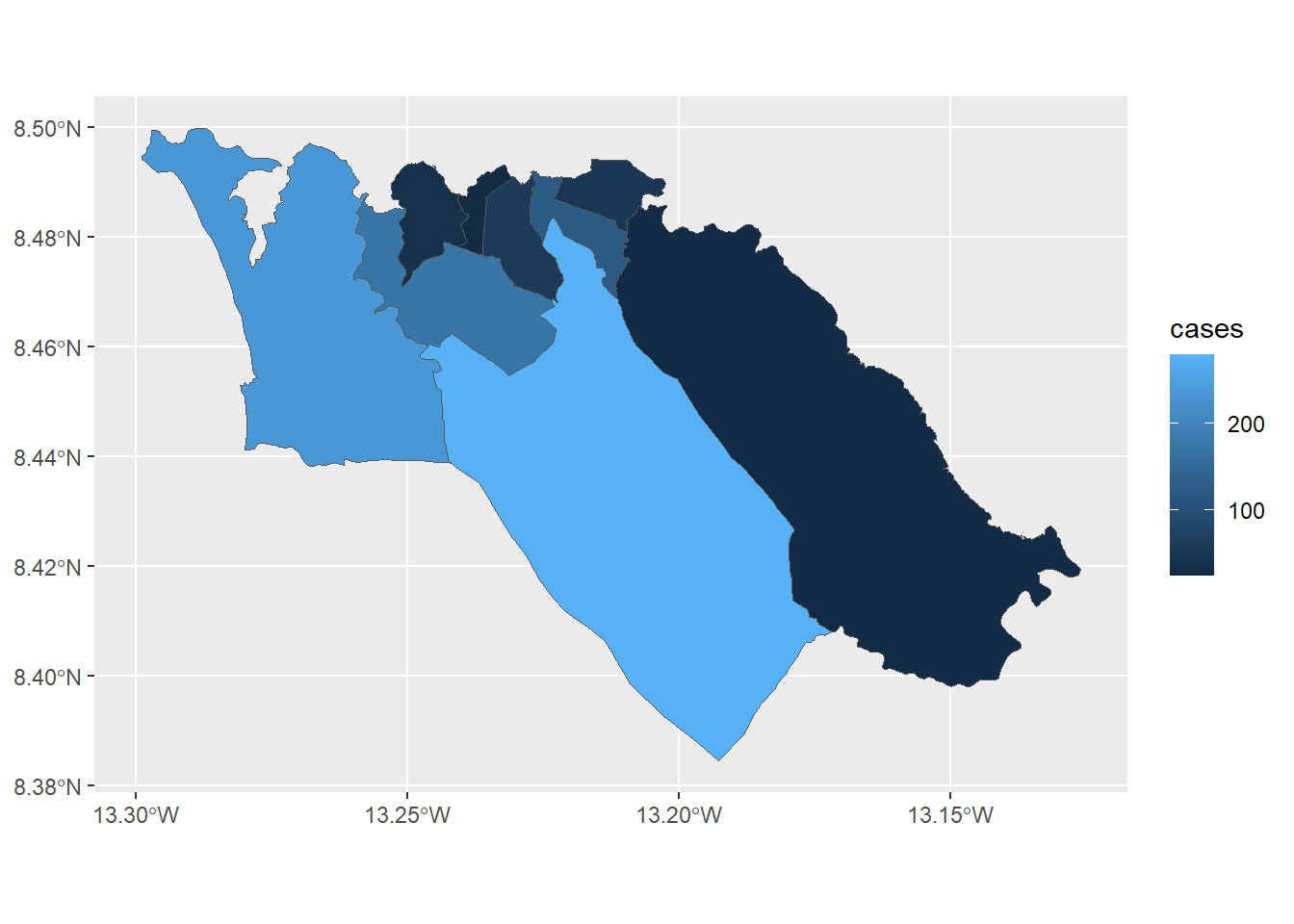
ggplot(data=sle_adm3_dat) +
geom_sf(aes(fill=cases)) # define a cor de preenchimento (fill) para variar de acordo com a variável de contagem dos casos Podemos customizar a aparência de nosso mapa utilizando a gramática que é consistente no ggplot2, por exemplo:
ggplot(data=sle_adm3_dat) +
geom_sf(aes(fill=cases)) +
scale_fill_continuous(high="#54278f", low="#f2f0f7") + # muda a cor do gradiente
theme_bw() +
labs(title = "Number of cases, by administrative unit", # define o texto da figura
subtitle = "Admin level 3"
)Para usuários de R que estão confortáveis em trabalhar com ggplot2, a função geom_sf() oferece uma implementação simples e direta que é adequada para visualizações básicas de mapas. Para aprender mais, leia a vinheta do geom_sf() ou o livro do ggplot2.
28.9 Mapas Base (Basemaps)
OpenStreetMap
Descrevemos abaixo como gerar um mapa base (basemap) para um mapa do ggplot2 utilizando funcionalidades do OpenStreetMap. Métodos alternativos incluem utilizar o ggmap que requer um cadastro gratuito via Google (detalhes).
OpenStreetMap é um projeto colaborativo para criar um mapa do mundo editável e gratuito. Os dados de geolocalização fundamentais (ex: localidades de cidades, estradas, características naturais, aeroportos, escolas, hospitais, etc) são considerados as saídas (outputs) principais do projeto.
Primeiro carregamos o pacote OpenStreetMap, de onde pegaremos nosso mapa base.
Depois, criamos o objeto map, o qual definimos utilizando a função openmap() do pacote OpenStreetMap (documentação). Passaremos os seguintes argumentos para função:
upperLeftelowerRightdois pares de coordenadas especificando os limites do tile do mapa base- Nesse caso inserimos o max e min das linhas da linelist, para que o mapa responda dinamicamente aos dados
zoom =(se não for preenchido será determinado automaticamente)
type =qual tipo de mapa base - listamos várias possibilidades aqui e o código está utilizando a primeira delas ([1]) “osm”
mergeTiles =optamos por TRUE para que os tiles base sejam mesclados em um só
# carrega o pacote
pacman::p_load(OpenStreetMap)
# Ajusta o mapa base pelo intervalo das coordenadas de lat/long. Define o tipo do tile
map <- openmap(
upperLeft = c(max(linelist$lat, na.rm=T), max(linelist$lon, na.rm=T)), # limites do tile do mapa base
lowerRight = c(min(linelist$lat, na.rm=T), min(linelist$lon, na.rm=T)),
zoom = NULL,
type = c("osm", "stamen-toner", "stamen-terrain", "stamen-watercolor", "esri","esri-topo")[1])Se executarmos esse mapa base nesse momento, utilizando autoplot.OpenStreetMap() do pacote OpenStreetMap, você verá que as unidades nos eixos não são coordenadas de latitude/longitude. Ele está utilizando um sistema de coordenadas diferente. Para mostrar corretamente as residências dos casos (que estão armazenados em lat/long), isso precisa ser modificado.
Assim, nossa intenção é converter o mapa para latitude/longitude com a função openproj() do pacote OpenStreetMap. Nós passamos para a função o mapa base map e também o Sistema de Referência de Coordenadas (SRC) que queremos. Fazemos isso passando a string do tipo “proj.4” para a projeção WGS 1984, mas você pode passar o SRC de outras formas também. (veja esta página para compreender melhor do que se trata uma string do tipo proj.4)
# Projeção WGS84
map_latlon <- openproj(map, projection = "+proj=longlat +ellps=WGS84 +datum=WGS84 +no_defs")Agora, quando executamos o mapa, vemos que ao longo dos eixos estão as coordenadas de latitude e longitude. O sistema de coordenadas foi convertido. Agora todos os nossos casos serão mostrados corretamente se sobrepostos ao mapa!
# Gera o mapa. É necessário usar "autoplot" para funcionar com o ggplot
autoplot.OpenStreetMap(map_latlon)28.10 Mapas de calor de densidade com contornos
Abaixo descrevemos como gerar um mapa de calor de densidade com contornos para os dados dos casos, sobre um mapa base, a partir de uma linelist (uma linha por caso).
- Cria o tile do mapa base a partir do OpenStreetMap, como descrito acima.
- Adiciona os casos da
linelistutilizando as colunas de latitude e longitude - Converte os pontos para um mapa de calor de densidade com
stat_density_2d()do ggplot2,
Quando temos um mapa base com coordenadas lat/long, podemos sobrepor nossos casos utilizando as coordenadas lat/long de suas residências.
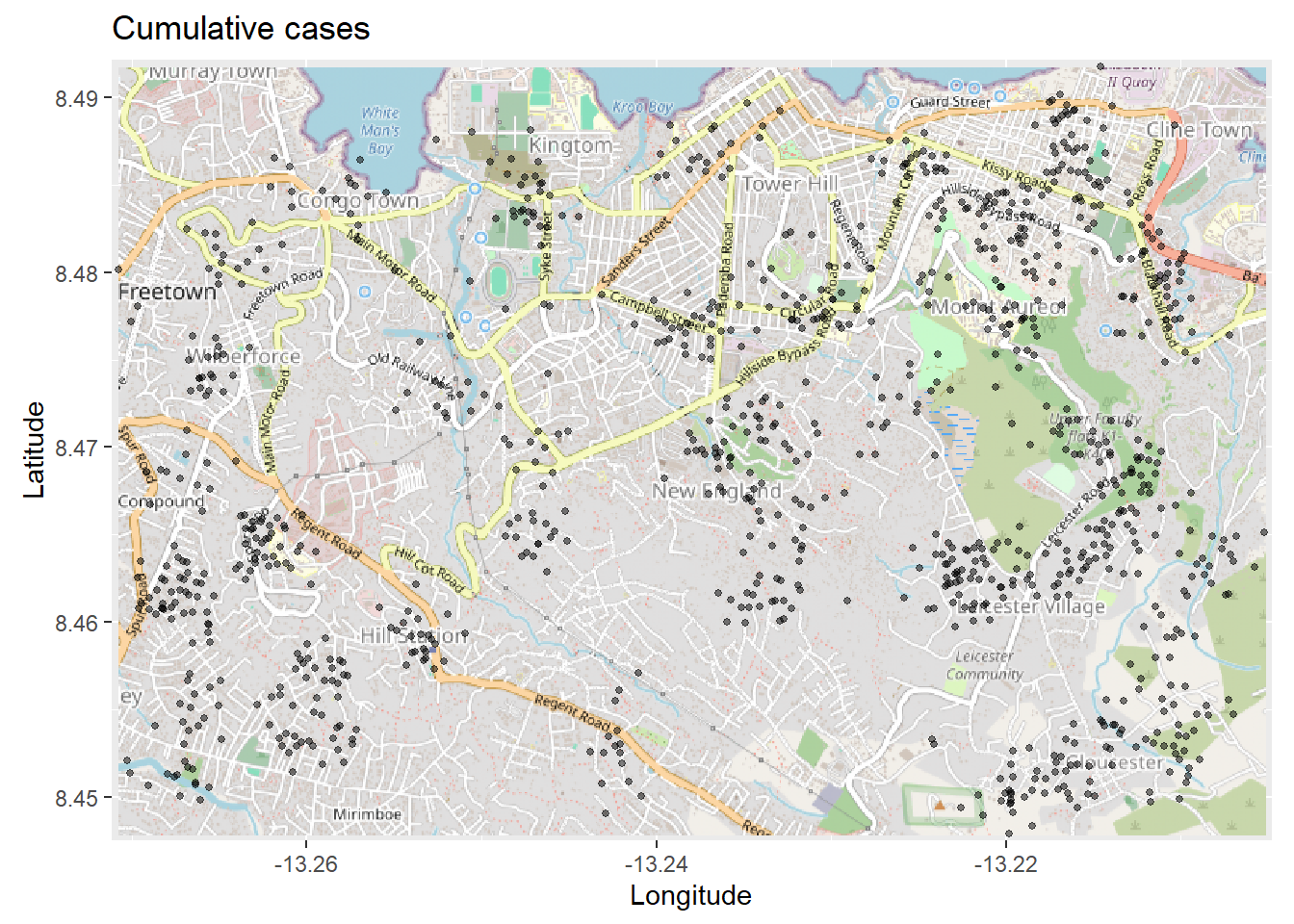
Utilizando a função autoplot.OpenStreetMap() para criar o mapa base, as funções do ggplot2 podem facilmente sobrepor camadas sobre ele, como mostrado com geom_point() abaixo:
# Gera o mapa. É necessário usar "autoplot" para funcionar com o ggplot
autoplot.OpenStreetMap(map_latlon)+ # inicia com o mapa base
geom_point( # adiciona os pontos xy a partir das colunas lo e lat da linelist
data = linelist,
aes(x = lon, y = lat),
size = 1,
alpha = 0.5,
show.legend = FALSE) + # remove a legenda completamente
labs(x = "Longitude", # títulos e rótulos
y = "Latitude",
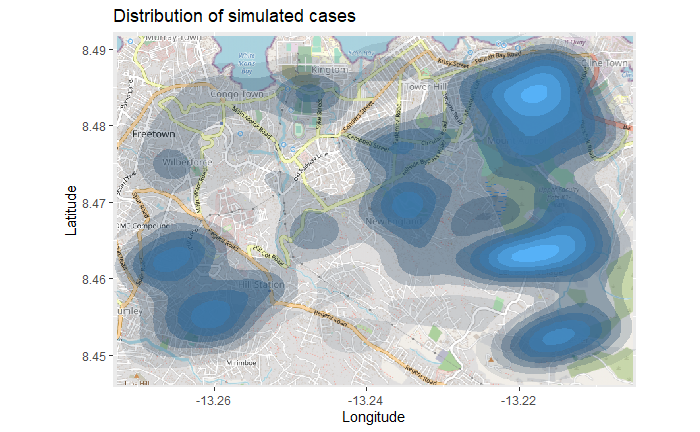
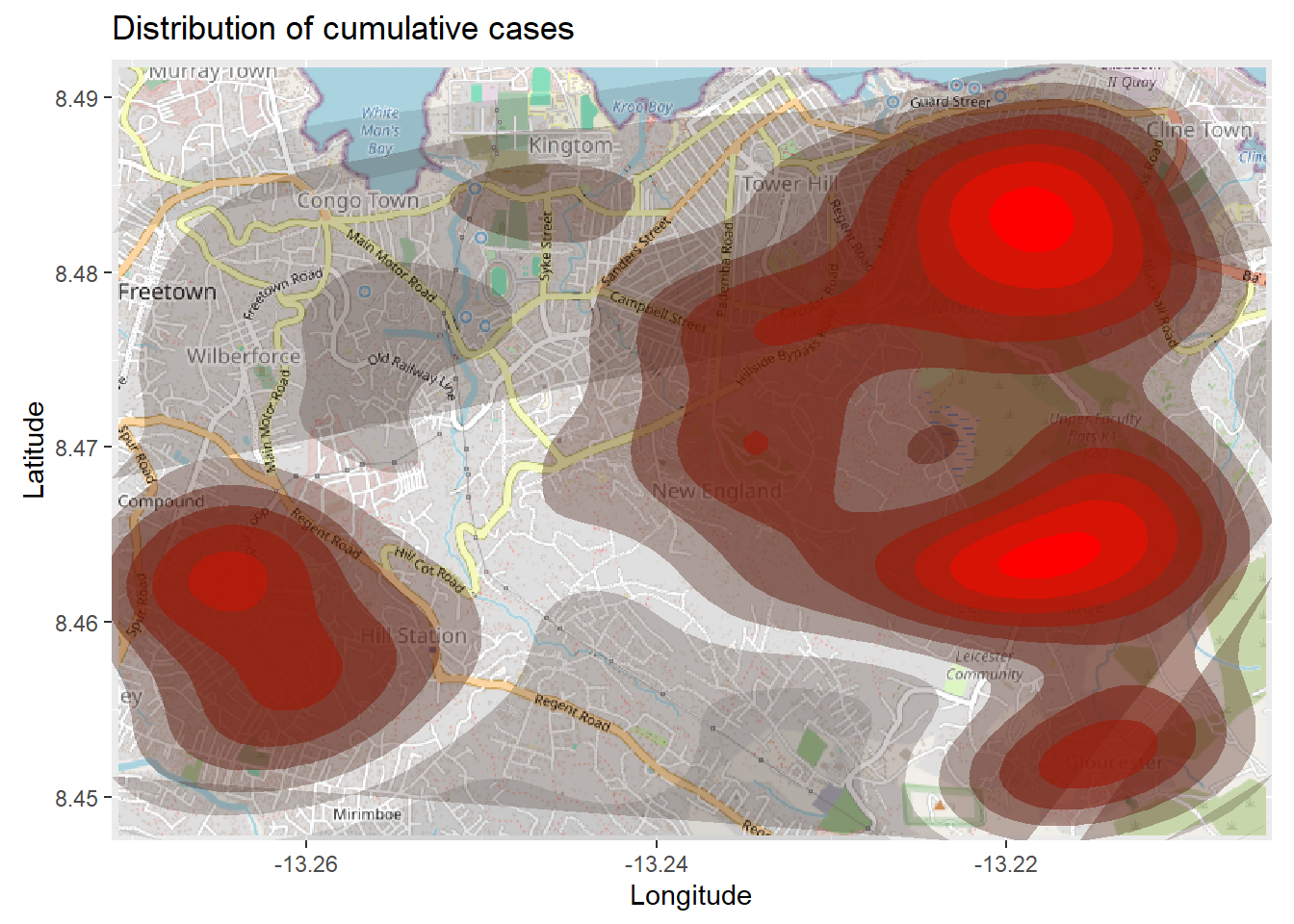
title = "Cumulative cases")O mapa acima pode ser difícil de interpretar, principalmente pelos pontos se soprepondo. Então, você pode, ao invés, gerar um mapa de densidade 2d utilizando a função stat_density_2d() do ggplot2. Você ainda estará utilizando as coordenadas lat/lon da linelist, mas uma estimativa de densidade por kernel 2D será calculada e os resultados serão mostrados como linhas de contorno - como em um mapa topográfico. Leia a documentação aqui.
# inicia com um mapa base
autoplot.OpenStreetMap(map_latlon)+
# adiciona o mapa de densidade
ggplot2::stat_density_2d(
data = linelist,
aes(
x = lon,
y = lat,
fill = ..level..,
alpha = ..level..),
bins = 10,
geom = "polygon",
contour_var = "count",
show.legend = F) +
# especifica a escala de cores
scale_fill_gradient(low = "black", high = "red")+
# rótulos
labs(x = "Longitude",
y = "Latitude",
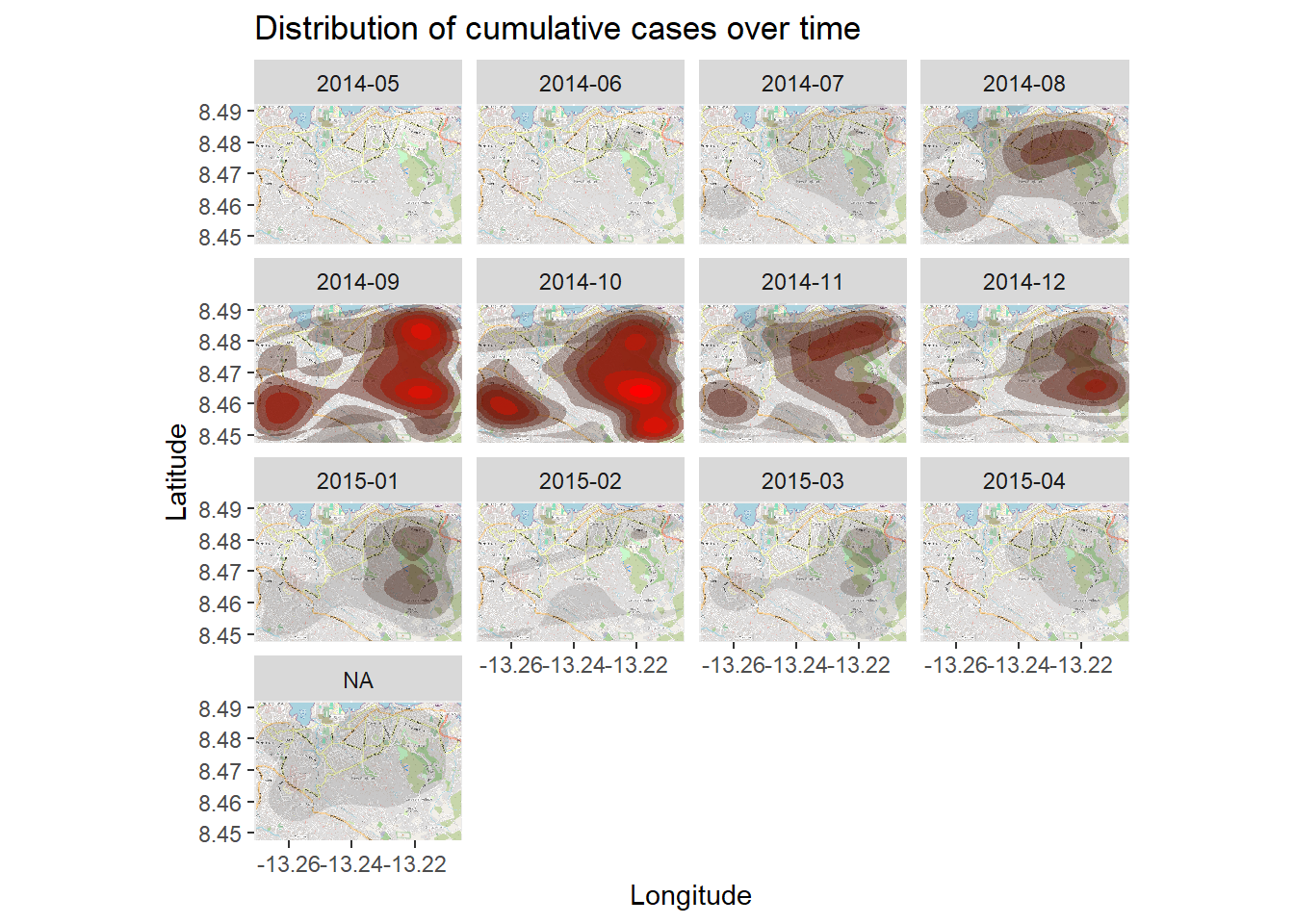
title = "Distribution of cumulative cases")Mapa de calor de séries temporais
O mapa de calor de densidade acima mostra os casos cumulativos. Podemos investigar o surto ao longo do tempo e espaço dividindo o mapa de calor em facetas baseadas no mês de início dos sintomas (symptom onset), derivado da linelist.
Começamos com a linelist, criando uma nova coluna com o Ano (Year) e Mês (Month) do início. A função format() do R base modifica a forma como a data é exibida. Nesse caso, queremos o formato “YYYY-MM”.
# Extrai o mês de início
linelist <- linelist %>%
mutate(date_onset_ym = format(date_onset, "%Y-%m"))
# Investiga os valores
table(linelist$date_onset_ym, useNA = "always")
2014-05 2014-06 2014-07 2014-08 2014-09 2014-10 2014-11 2014-12 2015-01 2015-02
10 19 42 97 180 181 127 104 64 50
2015-03 2015-04 <NA>
45 35 46 Agora, nós simplesmente adicionamos as facetas via ggplot2 ao mapa de calor de densidade. Aplicamos a função facet_wrap(), utilizando a nova coluna como linhas. Nós definimos o número de colunas das facetas para 4, para fins de clareza.
# pacotes
pacman::p_load(OpenStreetMap, tidyverse)
# inicia com o mapa base
autoplot.OpenStreetMap(map_latlon)+
# adiciona o mapa de densidade
ggplot2::stat_density_2d(
data = linelist,
aes(
x = lon,
y = lat,
fill = ..level..,
alpha = ..level..),
bins = 10,
geom = "polygon",
contour_var = "count",
show.legend = F) +
# especifica a escala de cores
scale_fill_gradient(low = "black", high = "red")+
# rótulos
labs(x = "Longitude",
y = "Latitude",
title = "Distribution of cumulative cases over time")+
# gera as facetas do gráfico por mês-ano de início
facet_wrap(~ date_onset_ym, ncol = 4) 28.11 Estatística espacial
Grande parte de nossa discussão até aqui estava focada na visualização de dados espaciais. Em alguns casos, você também pode estar interessado em estatística espacial para quantificar as relações espaciais entre os atributotos de seu dados. Essa seção vai passar uma visão global sobre alguns conceitos chave da estatística espacial, e sugerir alguns recursos que podem explorados caso você deseje fazer uma análise espacial mais aprofundada.
Relações espaciais
Antes de calcularmos qualquer estatística espacial, precisamos especificar as relações entre as características de nossos dados. Existem várias formas de conceituar relações espaciais, mas uma forma simples e um modelo comumente aplicado é utilizar o conceito de adjacência - especificamente, o fato de esperarmos uma relação geográfica entre áreas que compartilham uma fronteira ou são vizinhos uns dos outros.
Podemos quantificar relações de adjacências entre polígonos de regiões administrativas da base que viemos utilizando (sle_adm3) com o pacote spdep. Iremos especificar contiguidade do tipo queen (rainha), o que significa que as regiões serão vizinhas se compartilharem pelo menos um ponto ao longo de suas fronteiras. O método alternativo seria contiguidade do tipo rook (torre), que requer que as regiões compartilhem uma aresta - em nosso caso, com polígonos irregulares, a diferença é trivial, mas em alguns casos a escolha entre queen e rook pode fazer diferença. (Nota do tradutor: Os termos queen (rainha) e rook (torre) correspondem à peças do xadrez e o tipo de vizinhança que representam descritas acima estão relacionadas aos movimentos que fazem no referido jogo: na diagonal para rainha, ou seja apenas com um ponto de contato com a casa vizinha, e para frente ou para os lados com a torre, ou seja com uma aresta -a lateral do quadrado- completa de contato com a casa vizinha).
sle_nb <- spdep::poly2nb(sle_adm3_dat, queen=T) # cria vizinhos
sle_adjmat <- spdep::nb2mat(sle_nb) # cria uma matriz resumindo os relacionamentos entre vizinhos
sle_listw <- spdep::nb2listw(sle_nb) # cria um um objeto listw (lista de pesos (weights) ) -- iremos precisar disso posteriormente
sle_nbNeighbour list object:
Number of regions: 9
Number of nonzero links: 30
Percentage nonzero weights: 37.03704
Average number of links: 3.333333 round(sle_adjmat, digits = 2) [,1] [,2] [,3] [,4] [,5] [,6] [,7] [,8] [,9]
1 0.00 0.20 0.00 0.20 0.00 0.2 0.00 0.20 0.20
2 0.25 0.00 0.00 0.25 0.25 0.0 0.00 0.25 0.00
3 0.00 0.00 0.00 0.50 0.00 0.0 0.00 0.00 0.50
4 0.25 0.25 0.25 0.00 0.00 0.0 0.00 0.00 0.25
5 0.00 0.33 0.00 0.00 0.00 0.0 0.33 0.33 0.00
6 0.50 0.00 0.00 0.00 0.00 0.0 0.00 0.50 0.00
7 0.00 0.00 0.00 0.00 0.50 0.0 0.00 0.50 0.00
8 0.20 0.20 0.00 0.00 0.20 0.2 0.20 0.00 0.00
9 0.33 0.00 0.33 0.33 0.00 0.0 0.00 0.00 0.00
attr(,"call")
spdep::nb2mat(neighbours = sle_nb)A matriz exibida acima mostra as relações entre as 9 regiões de nossa base sle_adm3. Um score de 0 indica duas regiões que não são vizinhas, enquanto quaisquer outros valores diferentes de zero indicam uma relação de vizinhança. Os valores na matriz estão escalados de forma que a linha de cada região tenha um peso total de 1.
Uma melhor forma de visualizar essas relações de vizinhança é gerando um gráfico para elas:
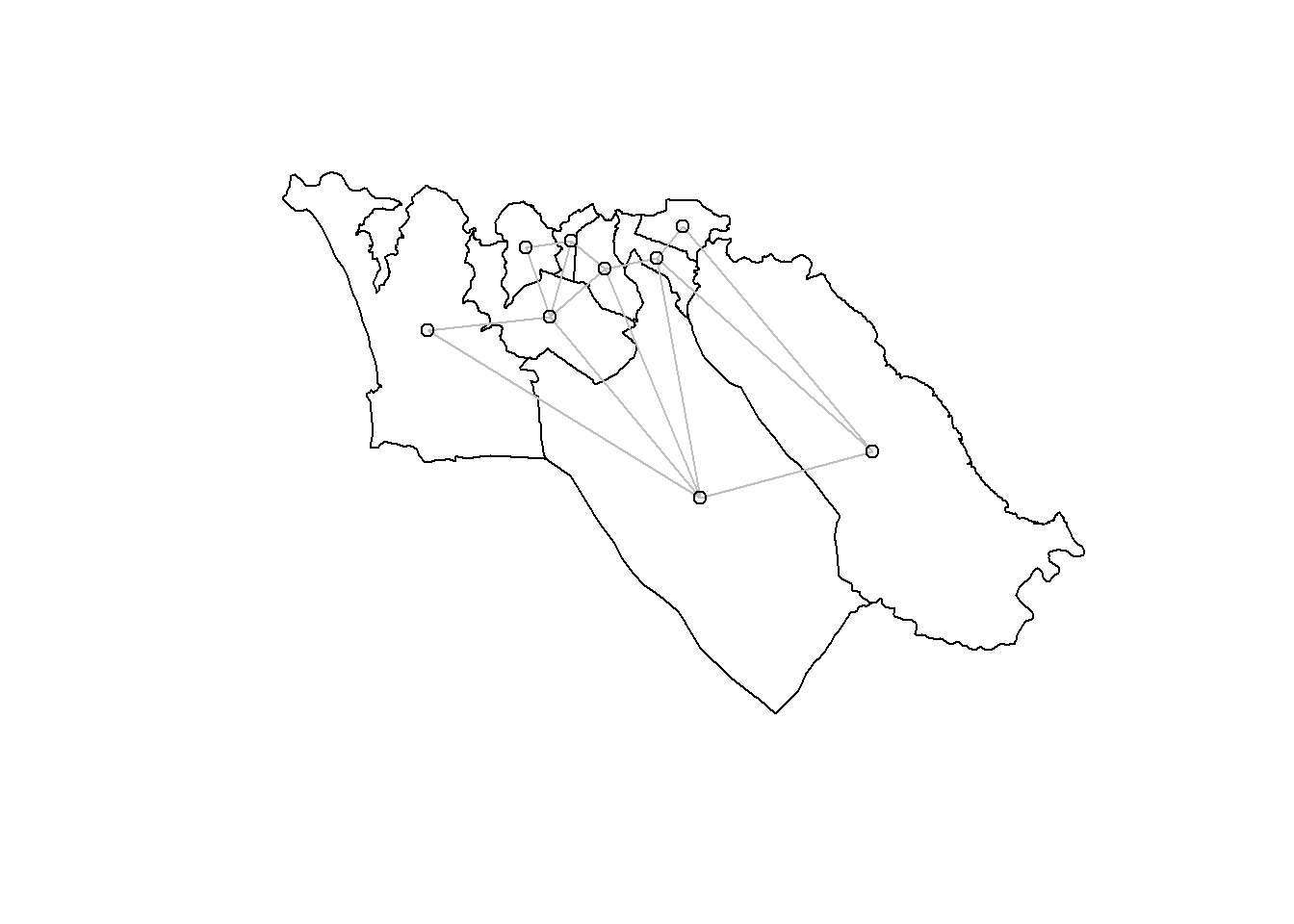
plot(sle_adm3_dat$geometry) + # cria gráficos com as fronteiras das regiões
spdep::plot.nb(sle_nb,as(sle_adm3_dat, 'Spatial'), col='grey', add=T) # adiciona relações de vizinhançaUtilizamos uma abordagem de adjacência para identificar polígonos vizinhos; os vizinhos identificados também podem ser chamados de vizinhos baseados em contiguidade (contiguity-based neighbors). Mas, essa é apenas um das formas de escolher quais regiões são esperadas de possuírem uma relação geográfica. As abordagens alternativas mais comuns para identificar relações geográficas geram vizinhos baseados em distância (distance-based neighbors); basicamente, eles são:
- K-vizinhos mais próximos (K-nearest neighbors) - Baseados nas distâncias entre centróides (centros geograficamente-pesados dos polígonos de cada região), seleciona as n regiões mais próximas como vizinhas. Um limite de proximidade de máxima-distância também pode ser especificado. Em spdep, você pode usar
knearneigh()(veja documentação). - Vizinhos por limite de distância - Seleciona todos os vizinhos contidos em um limite de distância. Em spdep, essa relação de vizinhos pode ser identificada utilizando a função
dnearneigh()(veja documentação).
Autocorrelação espacial
A conhecida primeira lei da geografia de Tobler diz que “todas as coisas estão relacionadas com todas as outras, mas coisas próximas estão mais relacionadas do que coisas distantes.” Na epidemiologia, isso geralmente significa que o risco de um certo desfecho de saúde em uma dada região é mais similar ao de suas regiões vizinhas que daquelas regiões mais longínquas. Esse conceito foi formalizado como autocorrelação espacial - a propridedade estatística em que características geográficas com valores similares são agregados no espaço. Medidas estatísticas de autocorrelação espacial podem ser utilizadas para quantificar a extensão da agregação espacial em seus dados, localizar onde a agregação ocorre, e identificar padrões compartilhados da autocorrelação espacial entre diferentes variáveis dos seus dados. Essa seção te dá uma visão global de algumas medidas de autocorrelação espacial mais comuns e como calculá-las no R.
I de Moran - Esta é uma estatística resumo global da correlação entre o valor de uma varíavel em uma região e os valores da mesma variável em regiões vizinhas. O I de Moran geralmente varia entre -1 e 1. O valor 0 indica que não há nenhum padrão de correlação espacial, enquanto valores próximos a 1 ou -1 indicam uma autocorrelação espacial mais forte (valores similares próximos de si) ou dispersão espacial (valores dissimilares próximoas de si), respectivamente.
Por exemplo, podemos calcular o I de Moran para quantificar a autocorrelação espacial nos casos de Ebola que mapeamos mais cedo (lembre-se, esse é um subset dos casos do dataframe da linelist da epidemia simulada). O pacote spdep tem uma função moran.test, que pode fazer esse cálculo para nós:
moran_i <-spdep::moran.test(sle_adm3_dat$cases, # vetor numérico com a variável de interesse
listw=sle_listw) # objeto listw resumindo as relações entre vizinhos
moran_i # imprime os resultados do teste do I de Moran
Moran I test under randomisation
data: sle_adm3_dat$cases
weights: sle_listw
Moran I statistic standard deviate = 1.6653, p-value = 0.04793
alternative hypothesis: greater
sample estimates:
Moran I statistic Expectation Variance
0.22479823 -0.12500000 0.04412088 A saída da função moran.test() nos mostra um valor do I de Moran de round(moran_i$estimate[1],2). Isso indica a presença de autocorrelação espacial em nossos dados - especificamente, que regiões com números parecidos de casos de Ebola estão potencialmente próximos entre si. O p-valor fornecido por moran.test() é gerado por comparação à expectativa, sob a hipótese nula, de não haver autocorrelação espacial, e pode ser utilizado caso você precise mostrar os resultados de um teste de hipóteses formal.
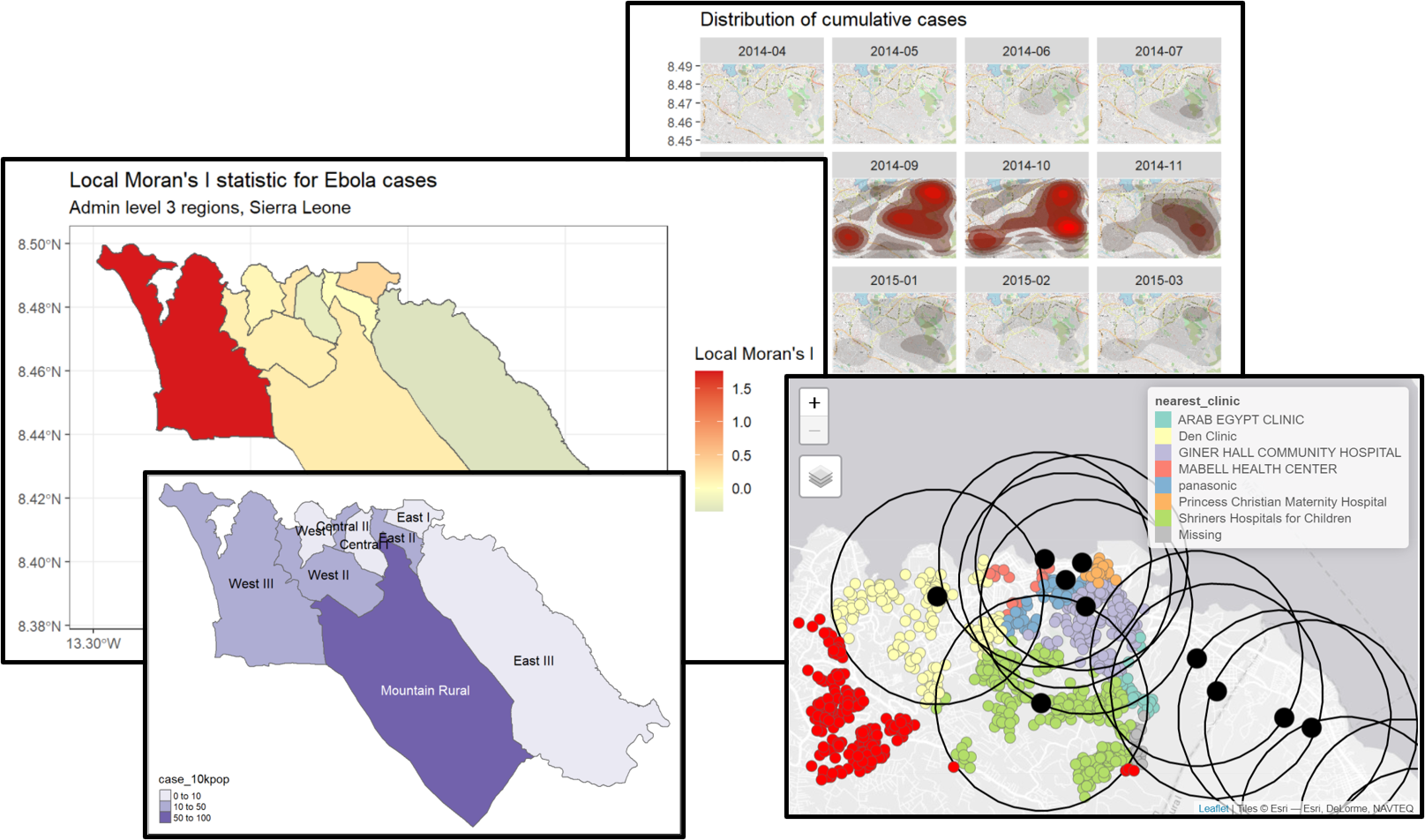
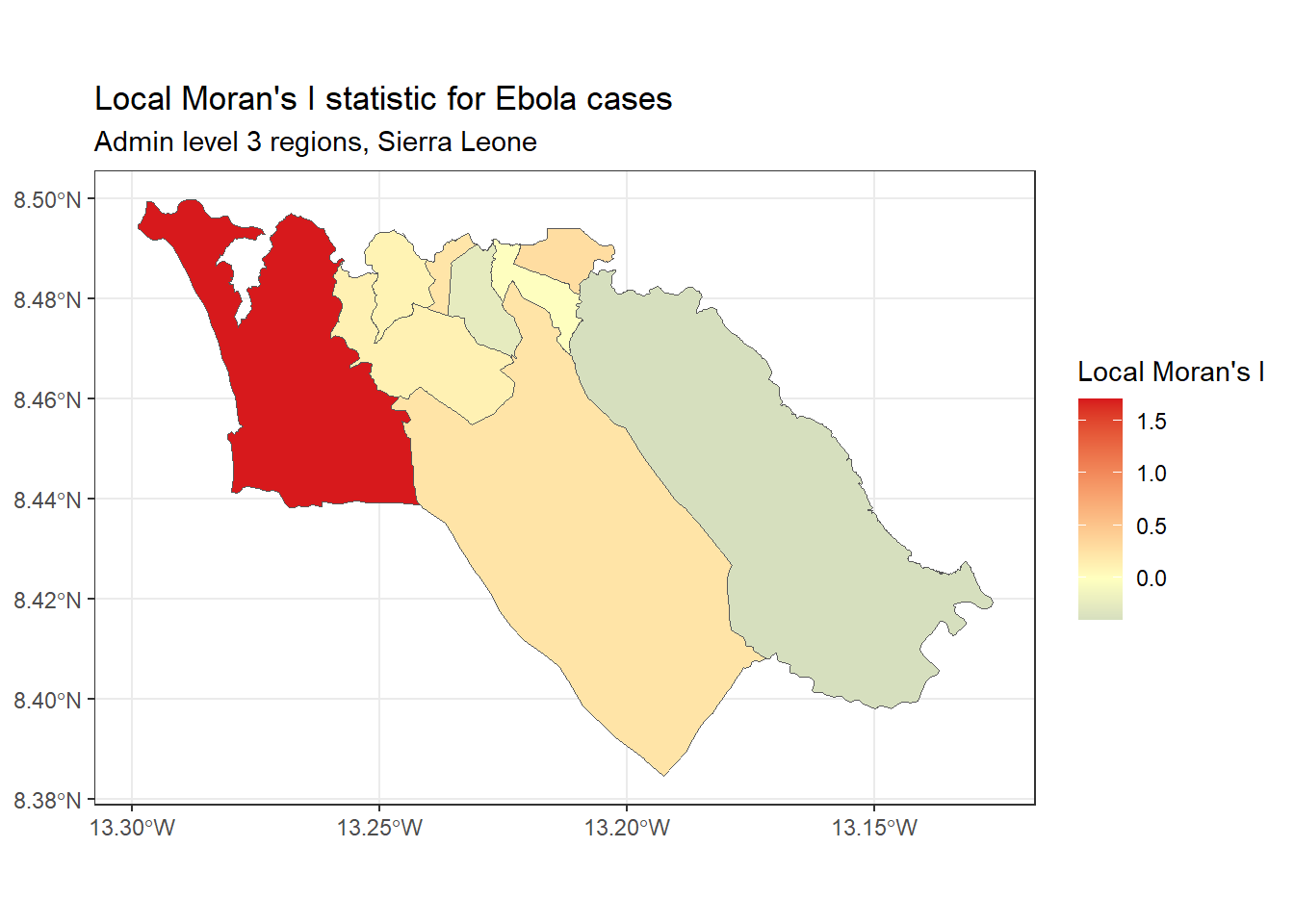
I de Moran Local - Podemos decompor o I de Moran (global) calculado acima para identificar uma autocorrelação espacial localizada; ou seja, para identificar agrupamentos específicos em nossos dados. Essa estatística, que as vezes é chamada de Indicador Local de Associação Espacial Local Indicator of Spatial Association (LISA), resume a extensão da autocorrelação espacial sobre cada região individual. Ela pode ser útil para achar regiões “quentes” ou “frias” no mapa.
Para mostrar um exemplo, podemos calcular e mapear I’s de Moran locais para as contagens de casos de Ebola utilizados acima, com a função local_moran() do pacote spdep:
# calcula o I de Moran local
local_moran <- spdep::localmoran(
sle_adm3_dat$cases, # variável de interesse
listw=sle_listw # objeto listw com pesos para os vizinhos
)
# faz join dos resultados aos dados do sf (shapefile)
sle_adm3_dat<- cbind(sle_adm3_dat, local_moran)
# gera o mapa
ggplot(data=sle_adm3_dat) +
geom_sf(aes(fill=Ii)) +
theme_bw() +
scale_fill_gradient2(low="#2c7bb6", mid="#ffffbf", high="#d7191c",
name="Local Moran's I") +
labs(title="Local Moran's I statistic for Ebola cases",
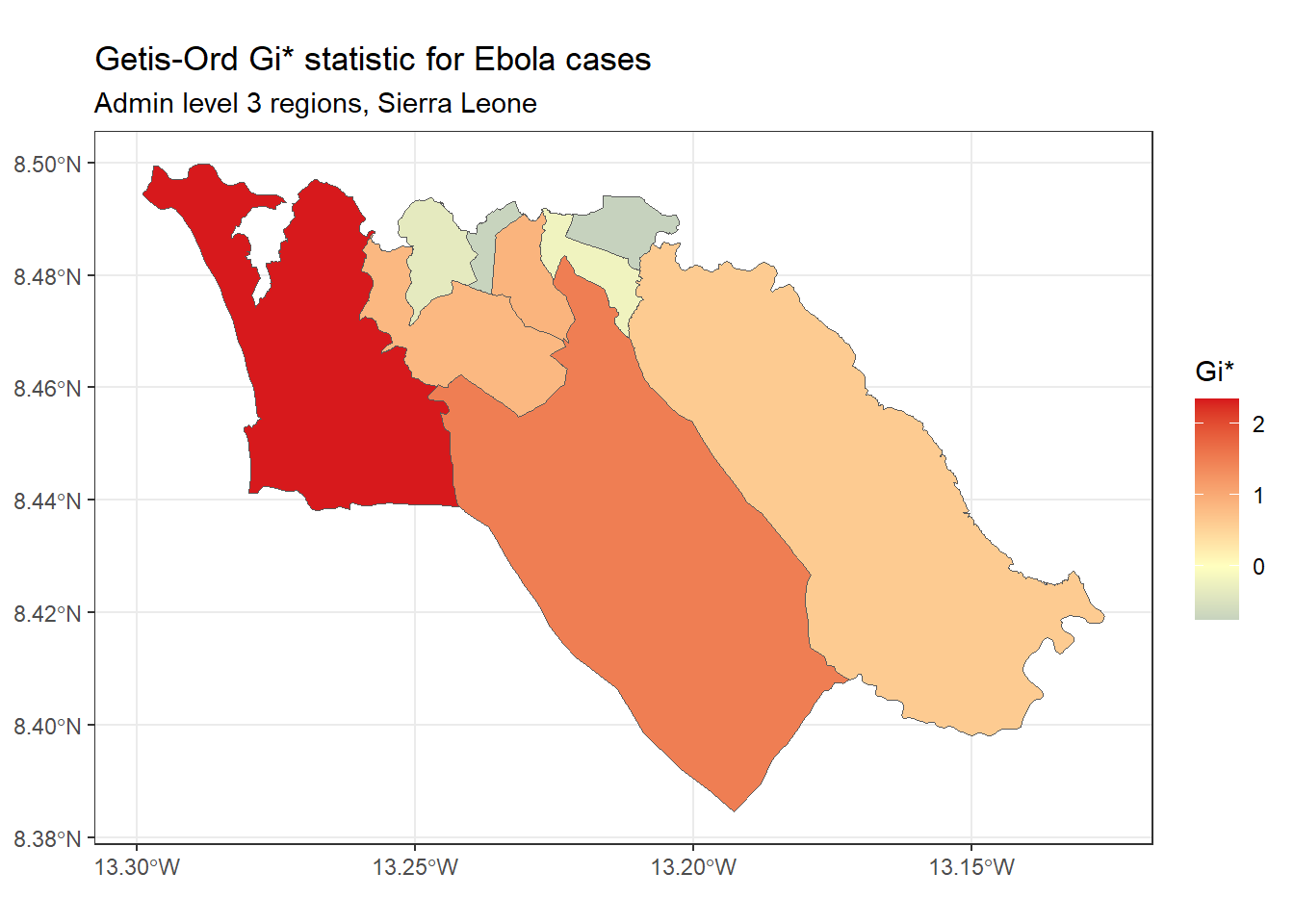
subtitle="Admin level 3 regions, Sierra Leone")Gi (ou G) de Getis-Ord - Essa é outra estatística comumente utilizada para análises de hotspots; em grande parte, a popularidade dessa estatística está relacionada à sua utilização na ferramenta de análise de hotspots no ArcGIS. Isso é baseado na premissa de que tipicamente, a diferença entre os valores de uma variável entre regiões vizinhas deve seguir uma distribuição normal. Ela usa a abordagem do z-score para identificar regiões que possuem valores de uma dada variável que sejam significativamente mais altas (hot spot) ou significativamente mais baixas (cold spot), comparados aos seus vizinhos.
Podemos calcular e mapear a estatística Gi* utilizando a função localG() do spdep:
# Faz uma análise G
getis_ord <- spdep::localG(
sle_adm3_dat$cases,
sle_listw
)
# faz join dos resultados aos dados do sf
sle_adm3_dat$getis_ord <- as.numeric(getis_ord)
# gera o mapa
ggplot(data=sle_adm3_dat) +
geom_sf(aes(fill=getis_ord)) +
theme_bw() +
scale_fill_gradient2(low="#2c7bb6", mid="#ffffbf", high="#d7191c",
name="Gi*") +
labs(title="Getis-Ord Gi* statistic for Ebola cases",
subtitle="Admin level 3 regions, Sierra Leone")Como você pode ver, o mapa de Gi* de Getis-Ord tem um visual ligeiramente diferente do mapa de I de Moran Local produzido anteriormente. Isso demonstra que o método utilizado para calcular essas duas estatísticas são ligeiramente diferentes; aquele que você vai utilizar depende do seu caso de uso específico e sua pergunta experimental de interesse.
Test L de Lee - Esse é um teste estatístico para correlação espacial bivariada. Ele permite que você teste se um padrão espacial para uma dada variável x é similar ao padrão espacial de outra variável y, cuja hipótese é que seja espacialmente relacionada com x.
Para dar um exemplo, vamos testar se um padrão espacial dos casos de Ebola da epidemia simulada é correlacionado com o padrão espacial da população. Para iniciar, precisamos de uma variável population em nossa base sle_adm3. Podemos utilizar a variável total do dataframe sle_adm3_pop que carregamos anteriormente.
sle_adm3_dat <- sle_adm3_dat %>%
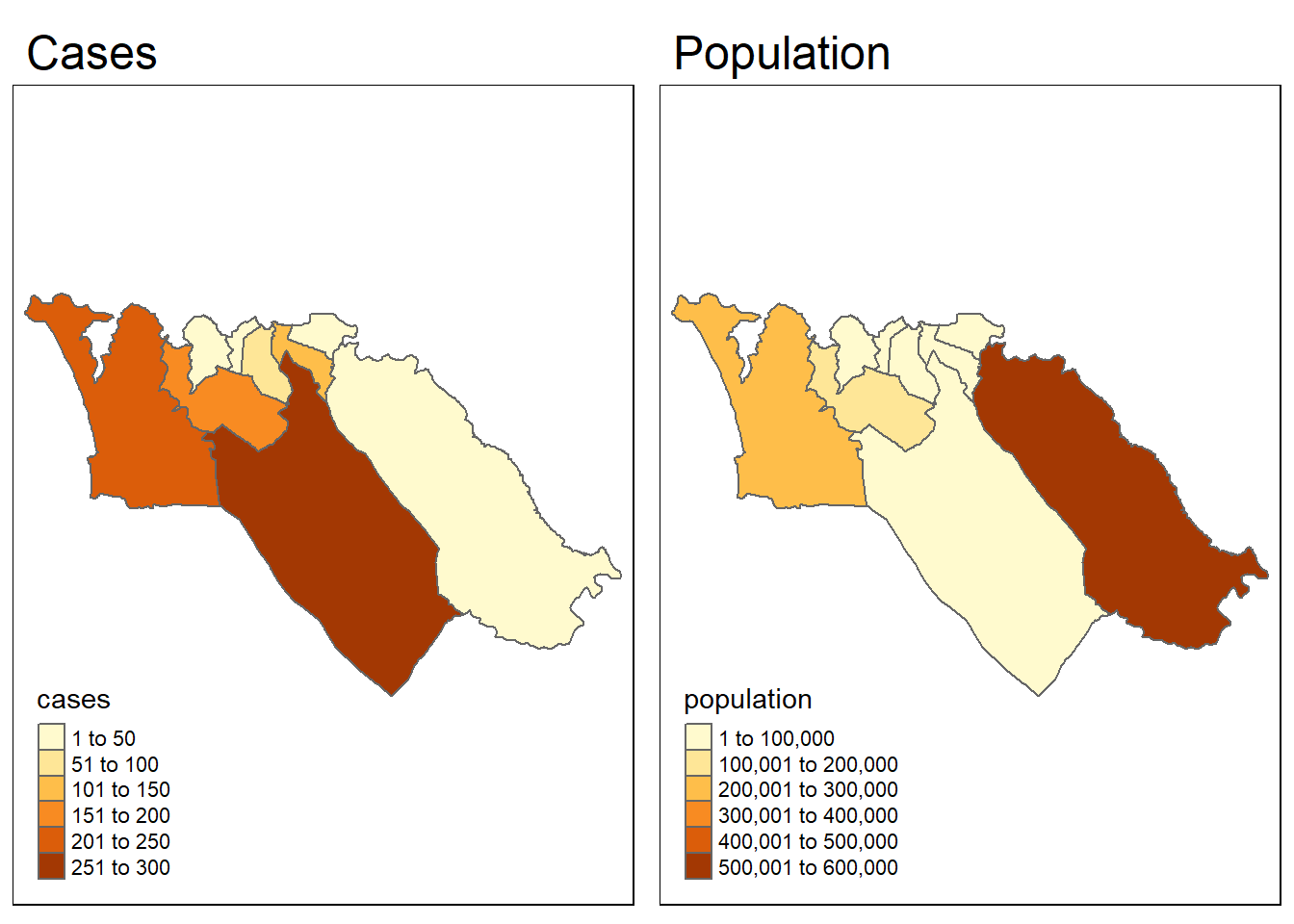
rename(population = total) # renomeia 'total' para 'population'Podemos visualizar rapidamente os padrões espaciais das duas variáveis lado a lado, para ver se eles se parecem:
tmap_mode("plot")
cases_map <- tm_shape(sle_adm3_dat) + tm_polygons("cases") + tm_layout(main.title="Cases")
pop_map <- tm_shape(sle_adm3_dat) + tm_polygons("population") + tm_layout(main.title="Population")
tmap_arrange(cases_map, pop_map, ncol=2) # organiza em facetas 2x1 Visualmente, os padrões parecem dissimilares. Podemos utilizar a função lee.test() do pacote spdep para testar estatisticamente se o padrão de autocorrelação espacial nas duas variáveis é relacionado. A estística L será próxima de 0 se não houver correlação entre os padrões, e próxima de 1 se tiver uma forte correlação positiva (ou seja, os padrões são similares), e próximo de -1 se houver uma forma correlação negativa (ou seja, os padrões são inversos).
lee_test <- spdep::lee.test(
x=sle_adm3_dat$cases, # variável 1 para comparar
y=sle_adm3_dat$population, # variável 2 para comparar
listw=sle_listw # objeto listw com peso dos vizinhos
)
lee_test
Lee's L statistic randomisation
data: sle_adm3_dat$cases , sle_adm3_dat$population
weights: sle_listw
Lee's L statistic standard deviate = -0.86785, p-value = 0.8073
alternative hypothesis: greater
sample estimates:
Lee's L statistic Expectation Variance
-0.13219674 -0.03580398 0.01233658 A saída acima mostra que a estatística L de Lee para as nossas duas variáveis é round(lee_test$estimate[1],2), o que indica uma fraca correlação negativa. Isso confirma nossa avaliação visual de que o padrão dos casos e população não estão relacionados entre si, e traz evidência de que o padrão espacial dos casos não é estritamente um resultado da densidade populacional em áreas de alto risco.
A estatística L de Lee pode ser útil para fazer esses tipos de inferência sobre a relação entre variáveis espacialmente distribuídas; no entanto, para descrever a natureza do relacionamento entre duas variáveis com mais detalhes, ou ajustar para fatores de confusão, técnicas de regressão espacial serão necessárias. Essas são descritas brevemente na seção seguinte.
Regressão espacial
Talvez você queira fazer inferências estatísticas a respeito dos relacionamentos entre variáveis nos seus dados espaciais. Nesses casos, pode ser útil considerar técnicas de regressão espacial - ou seja, abordagens para regressão que consideram explicitamente a organização espacial das unidades dos seus dados. Algumas razões que podem fazer você considerar modelos de regressão espacial, em vez de modelos de regressão padrão tais como GLMs, incluem:
- Modelos padrão de regressão assumem que os resíduos são independentes uns dos outros. Na presença de fortes autocorrelações espaciais, os resíduos de um modelo padrão de regressão têm a potencialidade de estarem espacialmente autocorrelacionados também, portanto, violando essa premissa. Isso pode levar a problemas com a interpretação dos resultados do modelo, o que para o caso, um modelo espacial seria mais adequado.
- Modelos de regressão também assumem, tipicamente, que o efeito de uma variável x é constante sobre todas as observações. No caso de heterogeneidade espacial, os efeitos que queremos estimar podem variar sobre o espaço, e podemos estar interessados em quantificar essas diferenças. Nesse caso, modelos de regressão espacial oferecem mais flexibilidade para estimar e interpretar os efeitos.
Os detalhes das abordagens de regressão espacial estão além do escopo desse manual. Essa seção vai, ao invés, passar uma visão global dos modelos de regressão espacial mais comuns e seus usos, e sugerir referências que podem ser úteis caso você queira explorar essa área mais a fundo.
Modelos de Erro Espacial - Esses modelos assumem que os termos de erro ao longo de unidades espaciais são correlacionados, sendo esse o caso, os dados violariam as premissas de um modelo OLS padrão. Modelos de Erro Espacial também podem ser chamados de modelos autorregressivos simultâneos (simultaneous autoregressive (SAR) models). Eles podem ser ajustados utilizando a função errorsarlm() do pacote spatialreg (funções de regressão espacial que costumavam fazer parte do pacote spdep).
Modelos de Defasagem Espacial (spacial lag models) - Esses modelos assumem que a variável dependente para uma região i é influenciada não apenas pelo valor das variáveis independentes em i, mas também pelos valores daquelas variáveis em regiões vizinhas a i. Assim como os modelos de erro espacial, modelos de defasagem espacial também são descritos algumas vezes como modelos autorregressivos simultâneos (simultaneous autoregressive (SAR) models). Eles podem ser ajustados utilizando a função lagsarlm() do pacote spatialreg.
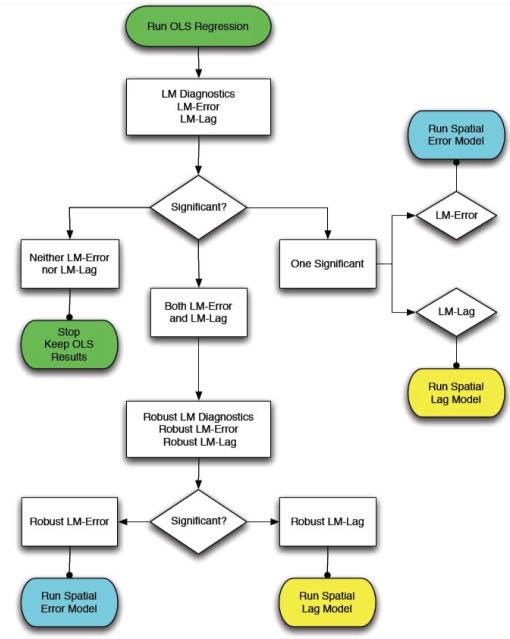
O pacote spdep contém vários testes diagnóstico úteis para decidir entre os modelos OLS padrão, de defasagem espacial ou de erro espacial. Esses testes, chamados diagnósticos de Multiplicador de (Lagrange Multiplier diagnostics), podem ser utilizados para identificar o tipo de dependência espacial em seus dados e escolher qual modelo é mais apropriado. A função lm.LMtests() pode ser utilizada para calcular todos os testes de Multiplicador de Lagrange. Anselin (1988) também demonstra uma ferramenta de fluxogramas para decidir qual modelo de regressão espacial a ser utilizado baseado nos resultados dos testes de Multiplicador de Lagrange:
Modelos Hierárquicos Bayesianos - Abordagens Bayesianas são comumente utilizadas para algumas aplicação em análise espacial, principalmente para mapeamento de doenças. Elas são preferidas em ocasiões onde os dados dos casos estão esparçamente distribuídas (por exemplo, no caso de um desfecho raro) ou com muito “ruído” estatístico, de forma que elas podem ser utilizadas para gerar estimativas “suavizadas” do risco de doenças ao considerar processos espaciais subjacentes que estavam latentes. Isso pode aumentar a qualidade das estimativas. Elas também permitem ao investigador a pre-especificação (via escolha de uma priori) de complexos padrões de correlação espaciais que podem existir nos dados, o que pode considerar variações espaço-dependente e -independente tanto nas variáveis independentes quanto nas dependentes. No R, Modelos Hierarquicos Bayesianos podem ser ajustados utilizando o pacote CARbayes (veja documentação) ou R-INLA (veja website e livro). R também pode ser utilizado para chamar softwares externos que fazem estimativas Bayesianas, tais como JAGS ou WinBUGS.
28.12 Recursos
- Pacote Simple Features vignette
- Pacote tmap vignette
- ggmap: Visualização Espacial com ggplot2
- Introdução à produção de mapas com R, visão geral de diferentes pacotes
- Dados Espaciais no R (EarthLab course)
- Análise de Dados Espaciais Aplicada com R livro
- SpatialEpiApp - um Shiny app que pode ser baixado como um pacote R, permitindo que você insira seus próprios dados e faça a criação de mapas, análises de agrupamento (clusters) e estatísticas espaciais.
- Introdução à Economoetria Espacial com R workshop