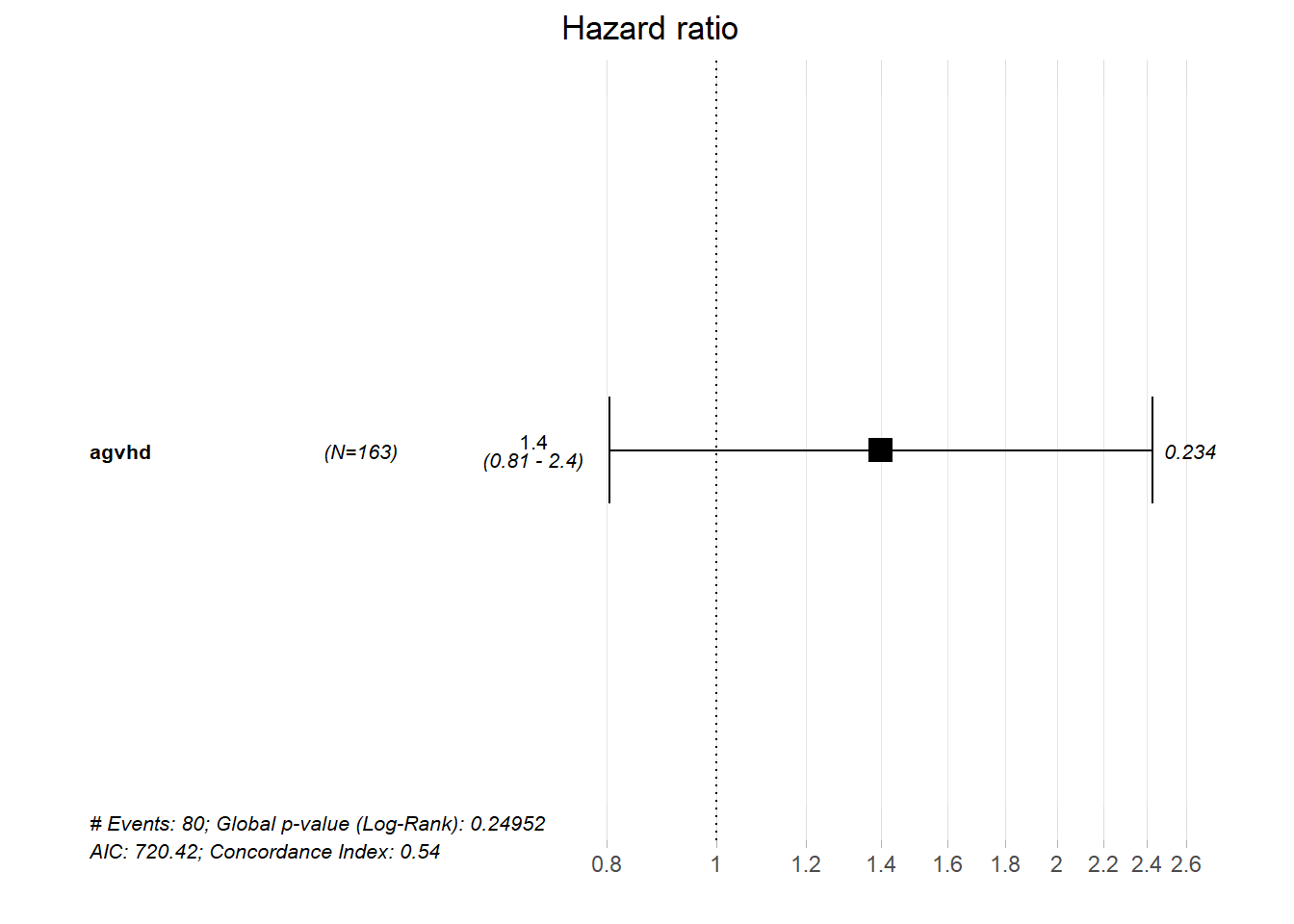27 Análise de sobrevivência
27.1 Visão Geral
A análise de sobrevivência centra-se na descrição, para um determinado indivíduo ou grupo de indivíduos, de um ponto definido de evento chamado a falha (ocorrência de uma doença, cura de uma doença, óbito, recaída após resposta ao tratamento…) que ocorre após um período de tempo chamado tempo de falha (ou tempo de seguimento em estudos baseados em coorte/população) durante o qual os indivíduos são observados. Para determinar o tempo de falha, é então necessário definir um tempo de origem (que pode ser a data de inclusão, a data de diagnóstico…).
O alvo de inferência para a análise de sobrevivência é então o tempo entre uma origem e um evento. Na investigação médica actual, é amplamente utilizado em estudos clínicos para avaliar o efeito de um tratamento, por exemplo, ou na epidemiologia do câncer para avaliar uma grande variedade de medidas de sobrevivência ao câncer.
É normalmente expressa através da probabilidade de sobrevivência que é a probabilidade de o evento de interesse não ter ocorrido por uma duração t.
Censura: A censura ocorre quando no final do seguimento, alguns dos indivíduos não tiveram o evento de interesse, e assim o seu verdadeiro tempo para o evento é desconhecido. Aqui focaremos principalmente na censura correta, mas para mais detalhes sobre a censura e a análise de sobrevivência em geral, é possível ver referências.
27.2 Preparação
Carregar Pacotes
Para realizar análises de sobrevivência em R, um dos pacotes mais utilizados é o pacote survival. Primeiro o instalamos e depois o carregamos, bem como os outros pacotes que serão utilizados nesta secção:
Neste manual enfatizamos p_load() do pacman, que instala o pacote se necessário e carrega-o para utilização. Pode também carregar os pacotes instalados com library() do R base . Veja a página em Introdução ao R para mais informações sobre os pacotes R.
Esta página explora análises de sobrevivência usando a linelist usada na maioria das páginas anteriores e na qual aplicamos algumas alterações para termos dados de sobrevivência adequados.
Importar conjunto de dados
Importamos o conjunto de dados de casos de uma epidemia simulada de Ebola. Se quiser acompanhar, clique para fazer o download da linelist “limpa” (as .rds file). Importe os dados com a função import() do pacote rio (suporta muitos formatos de arquivos .xlsx, .csv, .rds - veja a página Importar e exportar para mais detalhes).
# importar linelist
linelist_case_data <- rio::import("linelist_cleaned.rds")Gestão e transformação de dados
Em suma, os dados de sobrevivência podem ser descritos como tendo as três características seguintes:
- a variável ou resposta dependente é o tempo de espera até à ocorrência de um evento bem definido,
- as observações são censuradas, no sentido de que para algumas unidades o evento de interesse não ocorreu no momento em que os dados são analisados, e
- existem preditores ou variáveis explicativas cujo efeito sobre o tempo de espera desejamos avaliar ou controlar.
Assim, criaremos diferentes variáveis necessárias para respeitar essa estrutura e executar a análise de sobrevivência.
Definimos:
- um novo data frame
linelist_survpara esta análise - o nosso evento de interesse como sendo “óbito” (nossa probabilidade de sobrevivência será a probabilidade de estar vivo um certo tempo após o tempo de origem),
- o tempo de seguimento (
futime) como o tempo entre o tempo de início e o tempo do resultado em dias, - pacientes censurados como aqueles que recuperaram ou para os quais o resultado final não é conhecido, ou seja, o evento “morte” não foi observado (
evento=0).
CUIDADO: Uma vez que num estudo de coorte real, a informação sobre a hora de origem e o fim do seguimento é conhecida, dado que são observados indivíduos, removeremos as observações onde a data de início ou a data do desfecho é desconhecida. Também os casos em que a data de início é posterior à data do resultado serão removidos, uma vez que são considerados errados
DICA: Dado que filtrar para maior que (>) ou menor que (<) uma data pode remover linhas sem valores, aplicar o filtro nas datas erradas também removerá as linhas sem datas.
Depois utilizamos case_when() para criar uma coluna age_cat_small na qual existem apenas 3 categorias de idade.
#criar um novo dado chamado linelist_surv a partir do linelist_case_data
linelist_surv <- linelist_case_data %>%
dplyr::filter(
# remover observações com datas de início ou desfecho erradas ou vazias
date_outcome > date_onset) %>%
dplyr::mutate(
# criar o evento var que é 1 se o paciente morreu e 0 se ele foi censurado correctamente
event = ifelse(is.na(outcome) | outcome == "Recover", 0, 1),
# criar o var sobre o tempo de seguimento em dias
futime = as.double(date_outcome - date_onset),
# criar uma nova variável de categoria de idade com apenas 3 níveis de estratos
age_cat_small = dplyr::case_when(
age_years < 5 ~ "0-4",
age_years >= 5 & age_years < 20 ~ "5-19",
age_years >= 20 ~ "20+"),
# passo anterior criou age_cat_small var como caractere
# agora o converte em fator e especifica os níveis.
# note que os valores NA continuam a ser NA e não são colocados num nível "desconhecido", por exemplo,
# uma vez que nas análises seguintes tem de ser removidas.
age_cat_small = fct_relevel(age_cat_small, "0-4", "5-19", "20+")
)DICA: Podemos verificar as novas colunas que criamos fazendo um resumo sobre o futime e uma tabulação cruzada entre o evento e o resultado de onde foi criado. Para além desta verificação, é um bom hábito comunicar o tempo médio de seguimento ao interpretar os resultados da análise de sobrevivência.
summary(linelist_surv$futime) Min. 1st Qu. Median Mean 3rd Qu. Max.
1.00 6.00 10.00 11.98 16.00 64.00 # tabulação cruzada das novas variáveis modificadas "event" e "outcome" (desfecho)
# para garantir que o código fazia o que se pretendia
linelist_surv %>%
tabyl(outcome, event) outcome 0 1
Death 0 1952
Recover 1547 0
<NA> 1040 0Agora fazemos uma tabulação cruzada da nova variável “age_cat_small” e da antiga coluna “age_cat” para garantir atribuições corretas
linelist_surv %>%
tabyl(age_cat_small, age_cat) age_cat_small 0-4 5-9 10-14 15-19 20-29 30-49 50-69 70+ NA_
0-4 834 0 0 0 0 0 0 0 0
5-19 0 852 717 575 0 0 0 0 0
20+ 0 0 0 0 862 554 69 5 0
<NA> 0 0 0 0 0 0 0 0 71Agora revisamos as 10 primeiras observações dos dados da linelist_surv, olhando para variáveis específicas (incluindo aquelas recentemente criadas).
linelist_surv %>%
select(case_id, age_cat_small, date_onset, date_outcome, outcome, event, futime) %>%
head(10) case_id age_cat_small date_onset date_outcome outcome event futime
1 8689b7 0-4 2014-05-13 2014-05-18 Recover 0 5
2 11f8ea 20+ 2014-05-16 2014-05-30 Recover 0 14
3 893f25 0-4 2014-05-21 2014-05-29 Recover 0 8
4 be99c8 5-19 2014-05-22 2014-05-24 Recover 0 2
5 07e3e8 5-19 2014-05-27 2014-06-01 Recover 0 5
6 369449 0-4 2014-06-02 2014-06-07 Death 1 5
7 f393b4 20+ 2014-06-05 2014-06-18 Recover 0 13
8 1389ca 20+ 2014-06-05 2014-06-09 Death 1 4
9 2978ac 5-19 2014-06-06 2014-06-15 Death 1 9
10 fc15ef 5-19 2014-06-16 2014-07-09 Recover 0 23Também podemos fazer tabulações cruzadas entre as colunas age_cat_small e gender para obter mais detalhes acercada distribuição dessas novas colunas por gênero. Usamos tabyl() e a função adorn do pacote janitor, como descrito na página Tabelas descritivas.
linelist_surv %>%
tabyl(gender, age_cat_small, show_na = F) %>%
adorn_totals(where = "both") %>%
adorn_percentages() %>%
adorn_pct_formatting() %>%
adorn_ns(position = "front") gender 0-4 5-19 20+ Total
f 482 (22.4%) 1,184 (54.9%) 490 (22.7%) 2,156 (100.0%)
m 325 (15.0%) 880 (40.6%) 960 (44.3%) 2,165 (100.0%)
Total 807 (18.7%) 2,064 (47.8%) 1,450 (33.6%) 4,321 (100.0%)27.3 Noções básicas de análise de sobrevivência
Construir um objeto do tipo sobrevivente
Utilizaremos primeiro a função Surv() do pacote survival para construir um objeto de sobrevivência a partir das colunas de tempo e evento seguintes.
O resultado de tal passo é produzir um objeto do tipo Surv que condensa a informação do tempo e se o evento de interesse (óbito) foi observado. Este objeto acabará por ser utilizado no lado direito das fórmulas do modelo subsequente (ver documentação).
# Use a sintaxe Suv() para dados censurados
survobj <- Surv(time = linelist_surv$futime,
event = linelist_surv$event)Para rever, aqui estão as primeiras 10 linhas dos dados da linelist_surv, visualizando apenas algumas colunas importantes.
linelist_surv %>%
select(case_id, date_onset, date_outcome, futime, outcome, event) %>%
head(10) case_id date_onset date_outcome futime outcome event
1 8689b7 2014-05-13 2014-05-18 5 Recover 0
2 11f8ea 2014-05-16 2014-05-30 14 Recover 0
3 893f25 2014-05-21 2014-05-29 8 Recover 0
4 be99c8 2014-05-22 2014-05-24 2 Recover 0
5 07e3e8 2014-05-27 2014-06-01 5 Recover 0
6 369449 2014-06-02 2014-06-07 5 Death 1
7 f393b4 2014-06-05 2014-06-18 13 Recover 0
8 1389ca 2014-06-05 2014-06-09 4 Death 1
9 2978ac 2014-06-06 2014-06-15 9 Death 1
10 fc15ef 2014-06-16 2014-07-09 23 Recover 0E aqui estão os primeiros 10 elementos do survobj. Imprime essencialmente como um vetor de tempo de seguimento, com “+” para representar se uma observação foi bem censurada. Veja como os números se alinham acima e abaixo.
#imprime os 50 primeiros elementos do vetor para ver como ele se apresenta
head(survobj, 10) [1] 5+ 14+ 8+ 2+ 5+ 5 13+ 4 9 23+Rodando análises iniciais
Iniciamos então a nossa análise utilizando a função survfit() para produzir um objeto survfit, que se ajusta aos cálculos padrões de estimativas da curva de sobrevivência global (marginal) Kaplan Meier (KM) , que são na verdade uma função com saltos em tempos de eventos observados. O objeto final survfit contém uma ou mais curvas de sobrevivência e são criadas usando o objeto Surv como uma resposta variável na fórmula do modelo.
NOTA: A estimativa de Kaplan-Meier é uma estimativa não paramétrica da máxima probabilidade (MLE na sigla em inglês) da função de sobrevivência. . (ver recursos para mais informações)
O resumo deste objeto survfit dará o que se chama uma tabela de vida. Para cada passo do seguimento (tempo) em que um evento aconteceu (em ordem ascendente):
- o número de pessoas que estavam em risco de desenvolver o evento (pessoas que ainda não tinham o evento nem foram censuradas:
n.risk) - aqueles que desenvolveram o evento (
n.event) - e do acima exposto: a probabilidade de não desenvolver o evento (probabilidade de não morrer, ou de sobreviver depois desse tempo específico)
- finalmente, o erro padrão e o intervalo de confiança para essa probabilidade são derivados e exibidos
Encaixamos as estimativas KM usando a fórmula em que o objeto anteriormente Surv “survobj” é a variável de resposta. “~ 1” precisa que executamos o modelo para a sobrevivência global.
# encaixa nas estimativas KM usando uma fórmula onde o objeto Surv "sobrevivente" é a variável de resposta.
# "~ 1" significa que executamos o modelo para a sobrevivência global
linelistsurv_fit <- survival::survfit(survobj ~ 1)
# imprime o resumo para mais detalhes
summary(linelistsurv_fit)Call: survfit(formula = survobj ~ 1)
time n.risk n.event survival std.err lower 95% CI upper 95% CI
1 4539 30 0.993 0.00120 0.991 0.996
2 4500 69 0.978 0.00217 0.974 0.982
3 4394 149 0.945 0.00340 0.938 0.952
4 4176 194 0.901 0.00447 0.892 0.910
5 3899 214 0.852 0.00535 0.841 0.862
6 3592 210 0.802 0.00604 0.790 0.814
7 3223 179 0.757 0.00656 0.745 0.770
8 2899 167 0.714 0.00700 0.700 0.728
9 2593 145 0.674 0.00735 0.660 0.688
10 2311 109 0.642 0.00761 0.627 0.657
11 2081 119 0.605 0.00788 0.590 0.621
12 1843 89 0.576 0.00809 0.560 0.592
13 1608 55 0.556 0.00823 0.540 0.573
14 1448 43 0.540 0.00837 0.524 0.556
15 1296 31 0.527 0.00848 0.511 0.544
16 1152 48 0.505 0.00870 0.488 0.522
17 1002 29 0.490 0.00886 0.473 0.508
18 898 21 0.479 0.00900 0.462 0.497
19 798 7 0.475 0.00906 0.457 0.493
20 705 4 0.472 0.00911 0.454 0.490
21 626 13 0.462 0.00932 0.444 0.481
22 546 8 0.455 0.00948 0.437 0.474
23 481 5 0.451 0.00962 0.432 0.470
24 436 4 0.447 0.00975 0.428 0.466
25 378 4 0.442 0.00993 0.423 0.462
26 336 3 0.438 0.01010 0.419 0.458
27 297 1 0.436 0.01017 0.417 0.457
29 235 1 0.435 0.01030 0.415 0.455
38 73 1 0.429 0.01175 0.406 0.452Enquanto usamos summary() podemos adicionar a opção times e especificar certos tempos em que queremos ver informações de sobrevivência.
#imprime o resumo de tempos específicos
summary(linelistsurv_fit, times = c(5,10,20,30,60))Call: survfit(formula = survobj ~ 1)
time n.risk n.event survival std.err lower 95% CI upper 95% CI
5 3899 656 0.852 0.00535 0.841 0.862
10 2311 810 0.642 0.00761 0.627 0.657
20 705 446 0.472 0.00911 0.454 0.490
30 210 39 0.435 0.01030 0.415 0.455
60 2 1 0.429 0.01175 0.406 0.452Podemos também utilizar a função print(). O argumento print.rmean = TRUE é utilizado para obter o tempo médio de sobrevivência e o seu erro padrão (SE na sigla em inglês).
NOTA: O tempo médio de sobrevivência restrito (RMST na sigla em inglês) é uma medida de sobrevivência específica cada vez mais utilizada na análise de sobrevivência ao câncer e que é frequentemente definida como a área sob a curva de sobrevivência, dado que observamos pacientes até ao tempo restrito T (mais detalhes na seção Recursos).
# imprimir objeto linelistsurv_fit com tempo médio de sobrevivência e sua SE.
print(linelistsurv_fit, print.rmean = TRUE)Call: survfit(formula = survobj ~ 1)
n events rmean* se(rmean) median 0.95LCL 0.95UCL
[1,] 4539 1952 33.1 0.539 17 16 18
* restricted mean with upper limit = 64 DICA: Podemos criar o objeto surv diretamente na função survfit() e economizar uma linha de código. Ficará então: linelistsurv_quick <- survfit(Surv(futime, event) ~ 1, data=linelist_surv).
Risco acumulado
Além da função summary(), também podemos utilizar a função str() que dá mais detalhes sobre a estrutura do objeto survfit(). É uma lista de 16 elementos.
Entre estes elementos é um importante: cumhaz, que é um vetor numérico. Este pode ser traçado para permitir mostrar o risco cumulativo, sendo o risco o índice instantâneo de ocorrência de eventos (ver referências).
str(linelistsurv_fit)List of 16
$ n : int 4539
$ time : num [1:59] 1 2 3 4 5 6 7 8 9 10 ...
$ n.risk : num [1:59] 4539 4500 4394 4176 3899 ...
$ n.event : num [1:59] 30 69 149 194 214 210 179 167 145 109 ...
$ n.censor : num [1:59] 9 37 69 83 93 159 145 139 137 121 ...
$ surv : num [1:59] 0.993 0.978 0.945 0.901 0.852 ...
$ std.err : num [1:59] 0.00121 0.00222 0.00359 0.00496 0.00628 ...
$ cumhaz : num [1:59] 0.00661 0.02194 0.05585 0.10231 0.15719 ...
$ std.chaz : num [1:59] 0.00121 0.00221 0.00355 0.00487 0.00615 ...
$ type : chr "right"
$ logse : logi TRUE
$ conf.int : num 0.95
$ conf.type: chr "log"
$ lower : num [1:59] 0.991 0.974 0.938 0.892 0.841 ...
$ upper : num [1:59] 0.996 0.982 0.952 0.91 0.862 ...
$ call : language survfit(formula = survobj ~ 1)
- attr(*, "class")= chr "survfit"Traçando curvas Kaplan-Meir
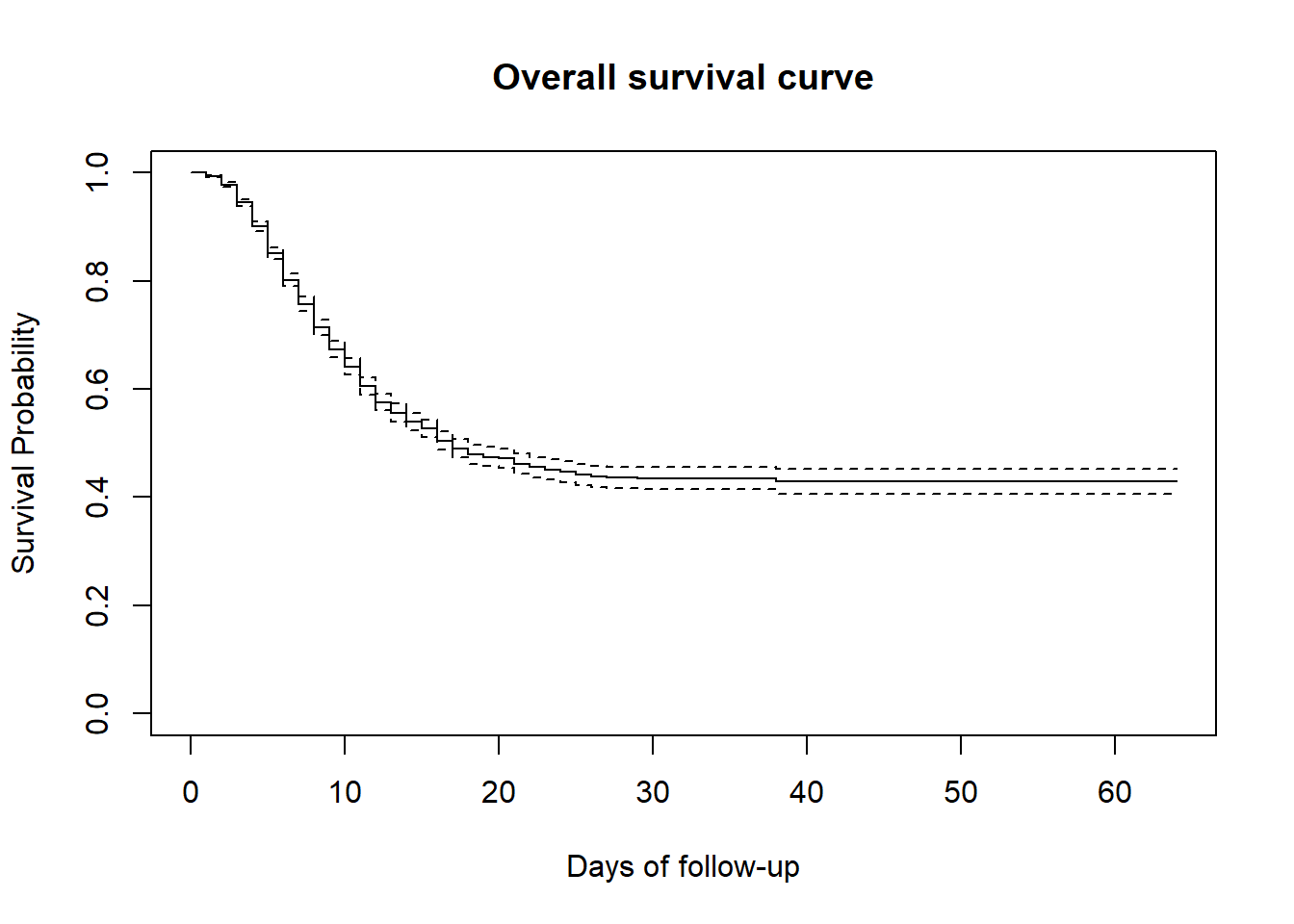
Uma vez encaixadas as estimativas KM, podemos visualizar a probabilidade de estarmos vivos durante um determinado tempo utilizando a função básica plot() que desenha a “curva Kaplan-Meier”. Em outras palavras, a curva abaixo é uma ilustração convencional da experiência de sobrevivência em todo o grupo de pacientes.
Podemos verificar rapidamente o tempo de seguimento mínimo e máximo na curva.
Uma maneira fácil de interpretar é dizer que no tempo zero, todos os participantes ainda estão vivos e a probabilidade de sobrevivência é então de 100%. Esta probabilidade diminui com o tempo, à medida que os pacientes morrem. A proporção de participantes que sobrevivem nos últimos 60 dias de seguimento é de cerca de 40%.
plot(linelistsurv_fit,
xlab = "Days of follow-up", # nome eixo X
ylab="Survival Probability", # nome eixo Y
main= "Overall survival curve" # título da figura
)O intervalo de confiança das estimativas de sobrevivência do KM também são traçados por padrão e podem ser descartados adicionando a opção conf.int = FALSE ao comando plot().
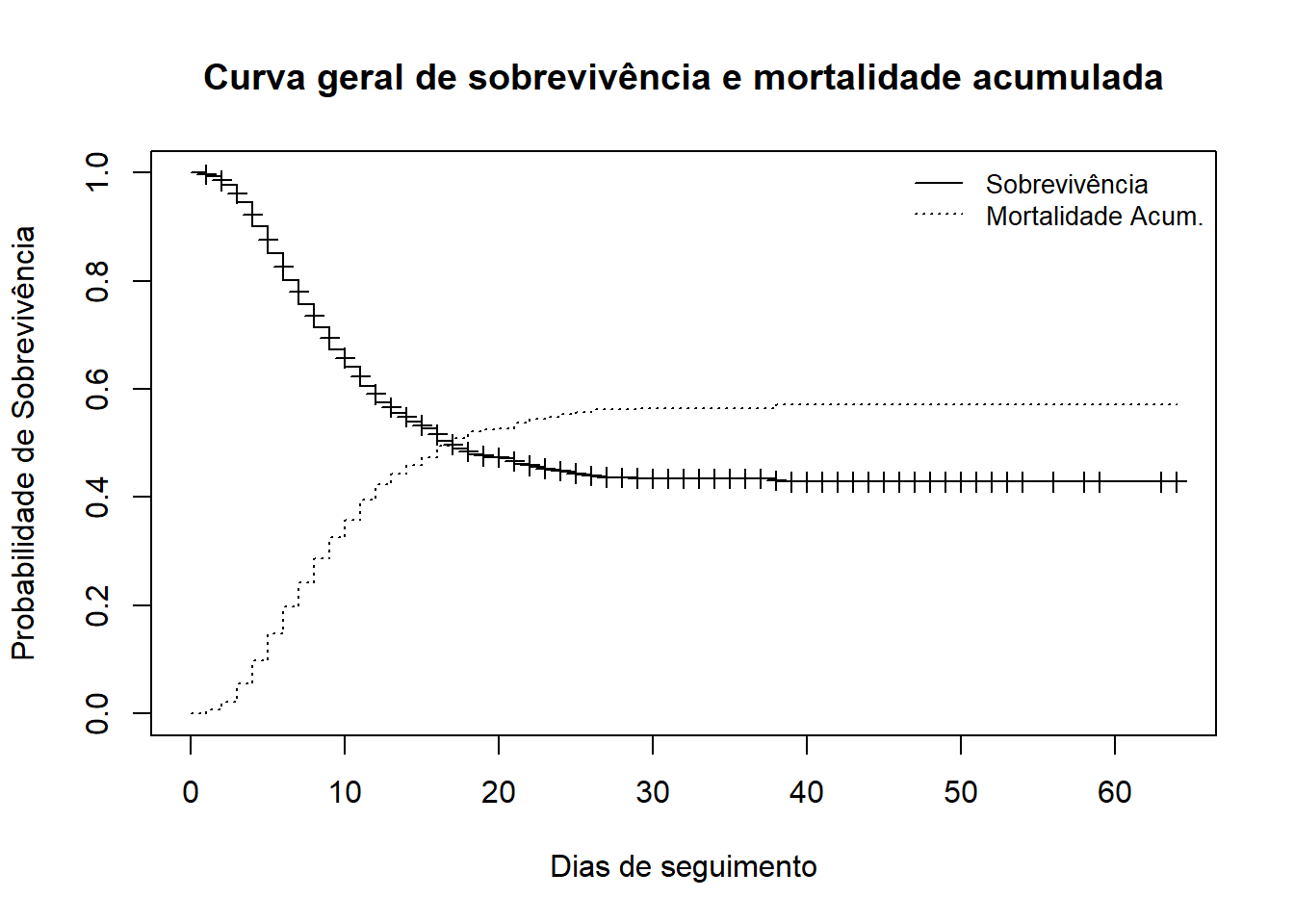
Uma vez que o evento de interesse é “óbito”, desenhar uma curva descrevendo os complementos das proporções de sobrevivência levará a desenhar as proporções de mortalidade acumulada. Isto pode ser feito com lines(), o que acrescenta informação a uma parcela existente.
# gráfico original
plot(
linelistsurv_fit,
xlab = "Dias de seguimento",
ylab = "Probabilidade de Sobrevivência",
mark.time = TRUE, # marcar eventos na curva: um "+" é impresso em cada evento
conf.int = FALSE, # não traçar o intervalo de confiança
main = "Curva geral de sobrevivência e mortalidade acumulada"
)
# desenhar uma curva adicional ao gráfico anterior
lines(
linelistsurv_fit,
lty = 3, # usar tipo de linha diferente para maior clareza
fun = "event", # desenhar os eventos cumulativos em vez da sobrevivência
mark.time = FALSE,
conf.int = FALSE
)
# adiciona legenda ao gráfico
legend(
"topright", # posição da legenda
legend = c("Sobrevivência", "Mortalidade Acum."), # texto da legenda
lty = c(1, 3), # tipos de linha a serem usados na legenda
cex = .85, # parâmetros que definem o tamanho do texto da legenda
bty = "n" # nenhum tipo de caixa a ser desenhada para a legenda
)27.4 Comparação de curvas de sobrevivência
Para comparar a sobrevivência dentro de diferentes grupos dos nossos participantes ou pacientes observados, podemos ter de olhar primeiro para as suas respectivas curvas de sobrevivência e depois fazer testes para avaliar a diferença entre grupos independentes. Esta comparação pode dizer respeito a grupos baseados no sexo, idade, tratamento, comorbidade…
Teste de classificação de registo
O teste de log-rank é um teste popular que compara toda a experiência de sobrevivência entre dois ou mais grupos independentes e pode ser pensado como um teste para verificar se as curvas de sobrevivência são idênticas (sobreposição) ou não (hipótese nula de não haver diferença na sobrevivência entre os grupos). A função survdiff() do pacote survival permite executar o teste de log-rank quando especificamos rho = 0 (que é o padrão). Os resultados do teste dão uma estatística de qui-quadrado juntamente com um p-valor, uma vez que a estatística de classificação logarítmica é distribuída aproximadamente como uma estatística de teste de qui-quadrado.
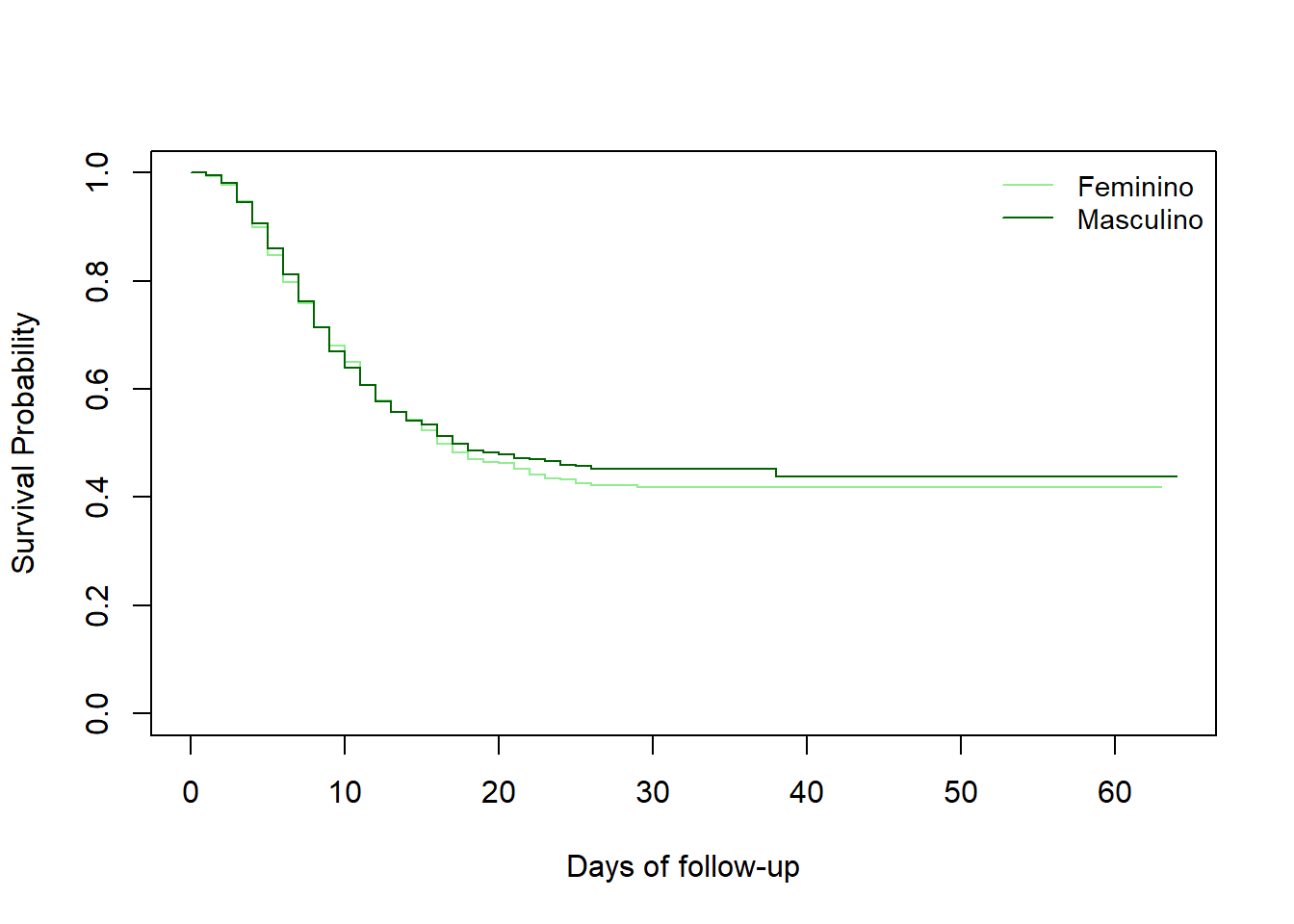
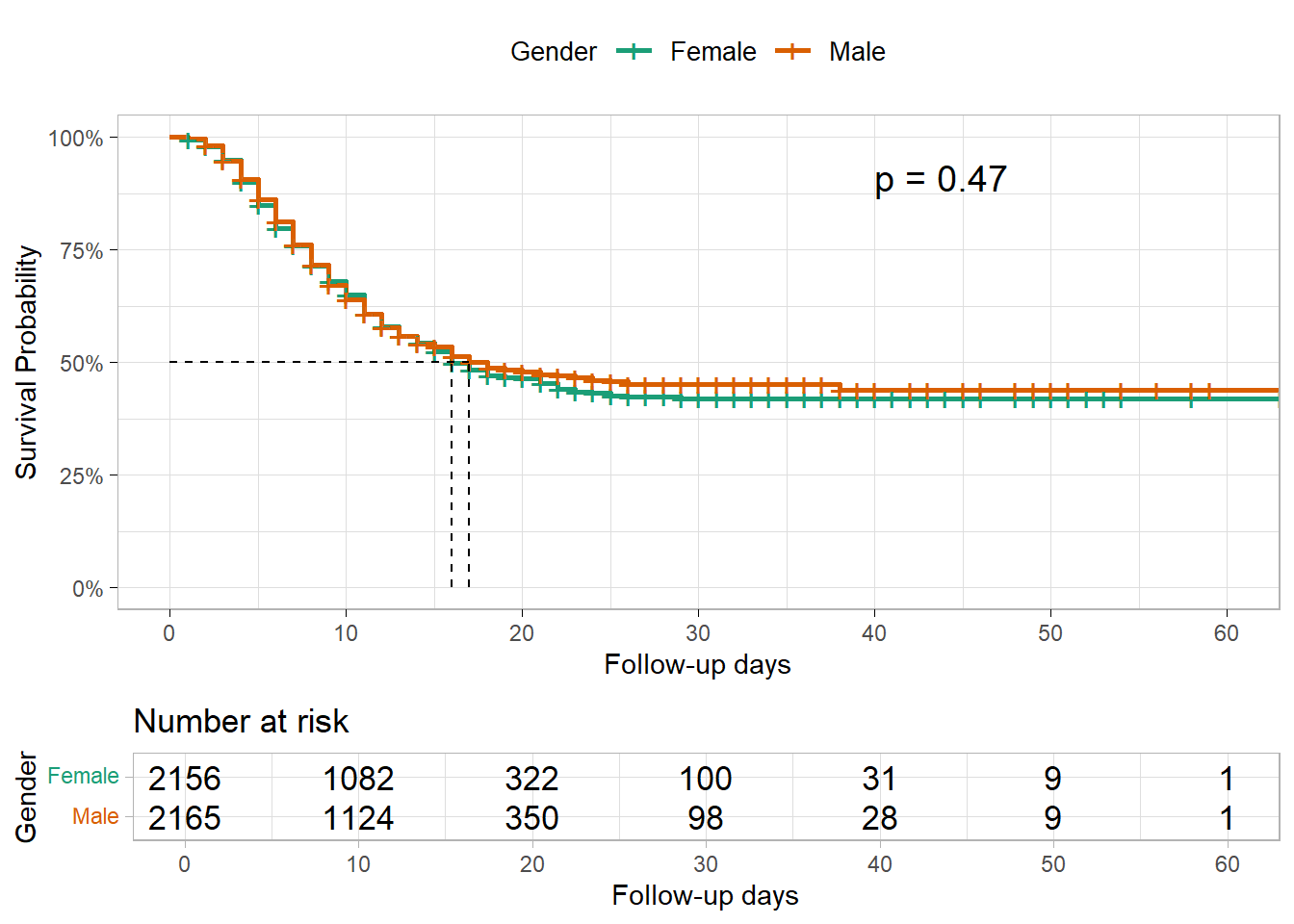
Tentamos primeiro comparar as curvas de sobrevivência por grupo de gênero. Para tal, tentamos primeiro visualizá-la (verificar se as duas curvas de sobrevivência estão sobrepostas). Um novo objeto survfit será criado com uma fórmula ligeiramente diferente. Depois será criado o objeto survdiff.
Ao fornecer ~ gender como o lado direito da fórmula, deixamos de traçar a sobrevivência global e passamos a fazê-lo por gênero.
# criar um novo objeto do tipo survfit baseado no gênero
linelistsurv_fit_sex <- survfit(Surv(futime, event) ~ gender, data = linelist_surv)Agora podemos traçar as curvas de sobrevivência por gênero. Veja a ordem dos níveis de estratos na coluna do gênero antes de definir as suas cores e legendas.
# padrão de cor
col_sex <- c("lightgreen", "darkgreen")
# cria a impressão
plot(
linelistsurv_fit_sex,
col = col_sex,
xlab = "Days of follow-up",
ylab = "Survival Probability")
# adiciona legenda
legend(
"topright",
legend = c("Feminino","Masculino"),
col = col_sex,
lty = 1,
cex = .9,
bty = "n")E agora podemos calcular o teste da diferença entre as curvas de sobrevivência usando survdiff ()
# calcula o teste da diferença entre as curvas de sobrevivência
survival::survdiff(
Surv(futime, event) ~ gender,
data = linelist_surv
)Call:
survival::survdiff(formula = Surv(futime, event) ~ gender, data = linelist_surv)
n=4321, 218 observations deleted due to missingness.
N Observed Expected (O-E)^2/E (O-E)^2/V
gender=f 2156 924 909 0.255 0.524
gender=m 2165 929 944 0.245 0.524
Chisq= 0.5 on 1 degrees of freedom, p= 0.5 Vemos que a curva de sobrevivência das mulheres e a dos homens se sobrepõem e o teste do log-rank não dá provas de uma diferença de sobrevivência entre mulheres e homens.
Alguns outros pacotes de R permitem ilustrar curvas de sobrevivência para diferentes grupos e testar a diferença de uma só vez. Utilizando a função ggsurvplot() do pacote survminer, podemos também incluir na nossa curva as tabelas de risco impressas para cada grupo, bem como o valor p do teste de log-rank.
[CUIDADO: funções survminer requerem que se especifique o objeto de sobrevivência e novamente os dados utilizados para encaixar o objeto de sobrevivência. Lembre-se de fazer isto para evitar mensagens de erro não específicas].{style=“color: orange;”}
survminer::ggsurvplot(
linelistsurv_fit_sex,
data = linelist_surv, # novamente especificar o dado usado para encaixar linelistsurv_fit_sex
conf.int = FALSE, # não mostrar o intervalo de confiança da estimativa KM
surv.scale = "percent", # apresentar probabilidades no eixo Y em %
break.time.by = 10, # apresentar o eixo de tempo com um incremento de 10 dias
xlab = "Follow-up days",
ylab = "Survival Probability",
pval = T, # adicionar o p-valor do teste Log-rank
pval.coord = c(40,.91), # adicionar o p-valor às coordenadas especificadas
risk.table = T, # imprimir a tabela de risco no fundo
legend.title = "Gender", # características da legenda
legend.labs = c("Female","Male"),
font.legend = 10,
palette = "Dark2", # especificar paleta de cores
surv.median.line = "hv", # desenhar linhas horizontais e verticais para a mediana de sobrevivência
ggtheme = theme_light() # simplificar o plano de fundo da impressão
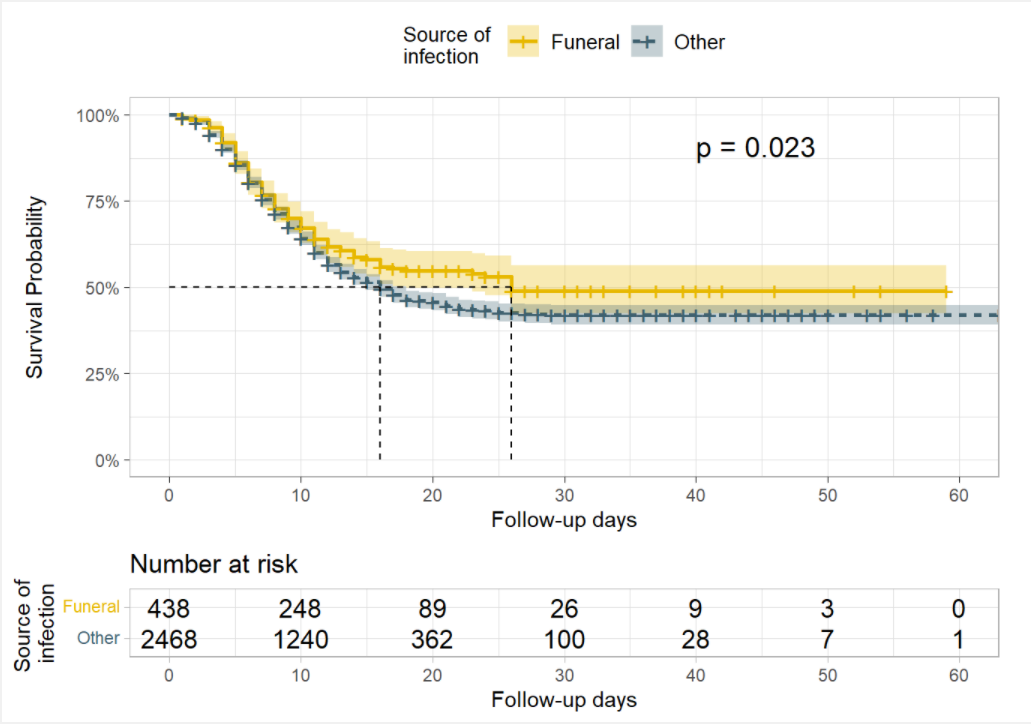
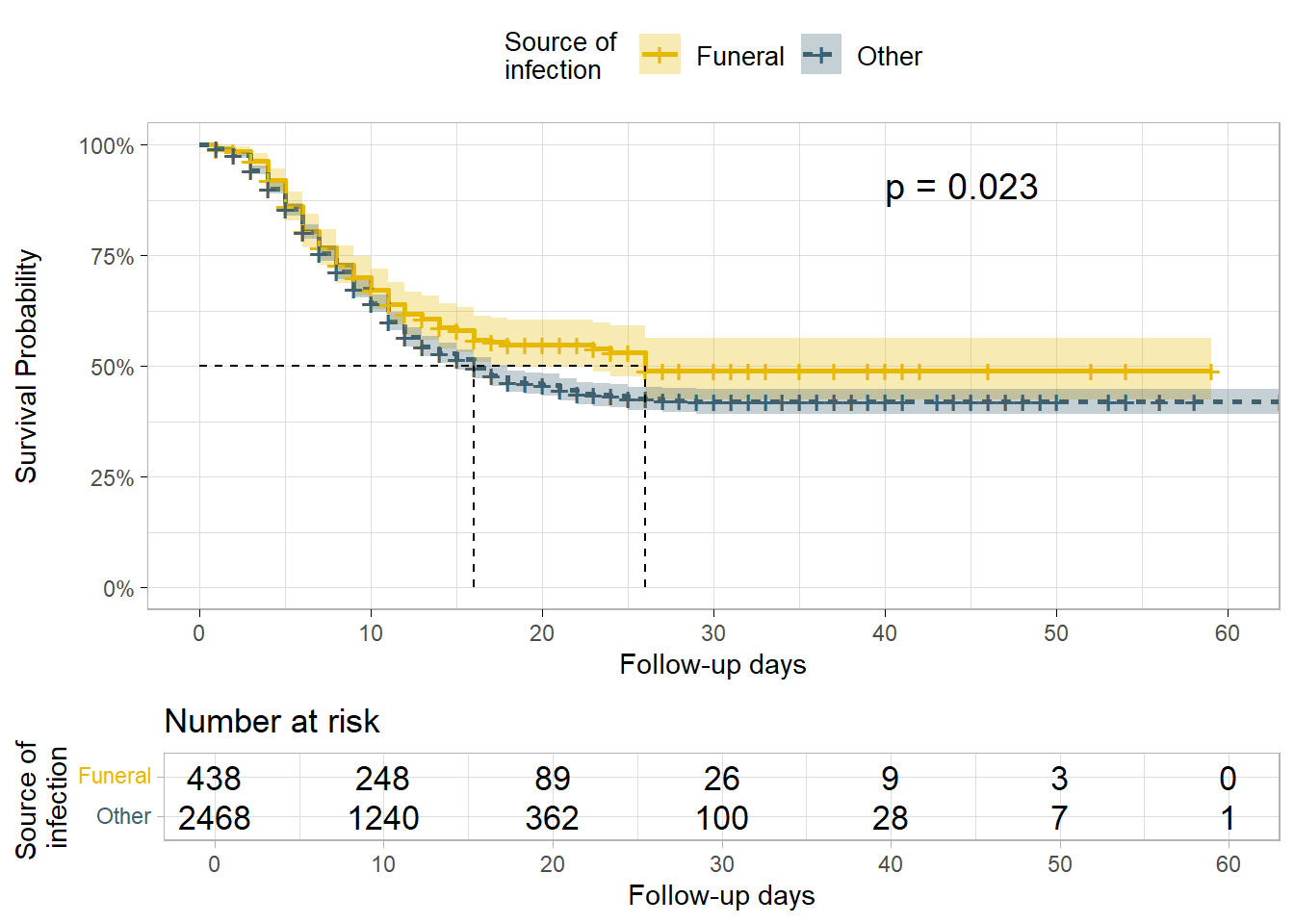
)Também podemos querer testar diferenças na sobrevivência pela fonte de infecção (fonte de contaminação).
Neste caso, o teste de Log rank dá provas suficientes de uma diferença nas probabilidades de sobrevivência em alpha= 0,005. As probabilidades de sobrevivência de pacientes que foram infectados em funerais são superiores às probabilidades de sobrevivência de pacientes que foram infectados em outros locais, sugerindo um benefício de sobrevivência.
linelistsurv_fit_source <- survfit(
Surv(futime, event) ~ source,
data = linelist_surv
)
# impressão
ggsurvplot(
linelistsurv_fit_source,
data = linelist_surv,
size = 1, linetype = "strata", # tipos de linhas
conf.int = T,
surv.scale = "percent",
break.time.by = 10,
xlab = "Follow-up days",
ylab= "Survival Probability",
pval = T,
pval.coord = c(40,.91),
risk.table = T,
legend.title = "Source of \ninfection",
legend.labs = c("Funeral", "Other"),
font.legend = 10,
palette = c("#E7B800","#3E606F"),
surv.median.line = "hv",
ggtheme = theme_light()
)Warning in geom_segment(aes(x = 0, y = max(y2), xend = max(x1), yend = max(y2)), : All aesthetics have length 1, but the data has 2 rows.
ℹ Did you mean to use `annotate()`?
All aesthetics have length 1, but the data has 2 rows.
ℹ Did you mean to use `annotate()`?
All aesthetics have length 1, but the data has 2 rows.
ℹ Did you mean to use `annotate()`?
All aesthetics have length 1, but the data has 2 rows.
ℹ Did you mean to use `annotate()`?27.5 Análise de regressão de Cox
O modelo de riscos proporcionais de Cox é uma das técnicas de regressão mais populares para a análise de sobrevivência. Outros modelos também podem ser utilizados, uma vez que o modelo Cox requer importantes pressupostos que precisam ser verificados para uma utilização adequada, tal como o pressuposto dos riscos proporcionais: ver referências.
Num modelo de riscos proporcionais de Cox, a medida do efeito é a taxa de risco ou razão de riscos(HR na sigla Hazard Ratio em inglês), que é o risco de fracasso (ou o risco de morte no nosso exemplo), dado que o participante sobreviveu até um tempo específico. Normalmente, estamos interessados em comparar grupos independentes com respeito aos seus perigos, e utilizamos uma taxa de risco, que é análoga a uma taxa de probabilidade no estabelecimento de uma análise de regressão logística múltipla. A função cox.ph() do pacote survival é utilizada para se adequar ao modelo. A função cox.zph() do pacote survival pode ser utilizada para testar a hipótese de riscos proporcionais para um ajuste do modelo de regressão Cox.
NOTA: A probabilidade deve estar no intervalo de 0 a 1. No entanto, o perigo representa o número esperado de eventos por uma unidade de tempo.
- Se a razão de perigo para um preditor for próxima de 1, então esse preditor não afeta a sobrevivência,
- se o HR for inferior a 1, então o preditor é protetor (ou seja, associado a uma melhor sobrevivência),
- e se a FC for superior a 1, então o preditor está associado ao aumento do risco (ou diminuição da sobrevivência).
Ajuste de um modelo Cox
Podemos primeiro ajustar um modelo para avaliar o efeito da idade e do gênero na sobrevivência. Ao imprimir apenas o modelo, temos a informação sobre:
- os coeficientes de regressão estimados “coef” que quantificam a associação entre os preditores e o resultado,
- a sua exponencial (para interpretabilidade,
exp(coef)) que produz a razão de riscos HR, - o seu erro padrão
se(coef), - o z-score: quantos erros padrão é o coeficiente estimado a partir de 0,
- e o p-valor: a probabilidade de o coeficiente estimado poder ser 0.
A função summary() aplicada ao objeto modelo Cox dá mais informações, tais como o intervalo de confiança do HR estimado e os diferentes resultados do teste.
O efeito da primeira covariável “gênero” é apresentado na primeira linha. O genderm (masculino) é mostrado indicando que o primeiro nível de estratos (“f”), ou seja, o grupo feminino, é o grupo de referência para as comparações dentro do gênero. Assim, a interpretação do parâmetro de teste é a dos homens em comparação com a das mulheres. O p-valor indica que não havia evidências suficientes de um efeito do gênero sobre o risco esperado ou de uma associação entre o gênero e a mortalidade por todas as causas.
A mesma falta de evidências é notada no que diz respeito ao grupo etário.
# ajustando o modelo cox
linelistsurv_cox_sexage <- survival::coxph(
Surv(futime, event) ~ gender + age_cat_small,
data = linelist_surv
)
#imprimindo o modelo ajustado
linelistsurv_cox_sexageCall:
survival::coxph(formula = Surv(futime, event) ~ gender + age_cat_small,
data = linelist_surv)
coef exp(coef) se(coef) z p
genderm -0.03149 0.96900 0.04767 -0.661 0.509
age_cat_small5-19 0.09400 1.09856 0.06454 1.456 0.145
age_cat_small20+ 0.05032 1.05161 0.06953 0.724 0.469
Likelihood ratio test=2.8 on 3 df, p=0.4243
n= 4321, number of events= 1853
(218 observations deleted due to missingness)#resumo do modelo
summary(linelistsurv_cox_sexage)Call:
survival::coxph(formula = Surv(futime, event) ~ gender + age_cat_small,
data = linelist_surv)
n= 4321, number of events= 1853
(218 observations deleted due to missingness)
coef exp(coef) se(coef) z Pr(>|z|)
genderm -0.03149 0.96900 0.04767 -0.661 0.509
age_cat_small5-19 0.09400 1.09856 0.06454 1.456 0.145
age_cat_small20+ 0.05032 1.05161 0.06953 0.724 0.469
exp(coef) exp(-coef) lower .95 upper .95
genderm 0.969 1.0320 0.8826 1.064
age_cat_small5-19 1.099 0.9103 0.9680 1.247
age_cat_small20+ 1.052 0.9509 0.9176 1.205
Concordance= 0.514 (se = 0.007 )
Likelihood ratio test= 2.8 on 3 df, p=0.4
Wald test = 2.78 on 3 df, p=0.4
Score (logrank) test = 2.78 on 3 df, p=0.4Foi interessante executar o modelo e olhar para os resultados, mas um primeiro olhar para verificar se os pressupostos de riscos proporcionais são respeitados poderia ajudar a poupar tempo.
test_ph_sexage <- survival::cox.zph(linelistsurv_cox_sexage)
test_ph_sexage chisq df p
gender 0.454 1 0.50
age_cat_small 0.838 2 0.66
GLOBAL 1.399 3 0.71NOTA: Um segundo argumento da função chamado method pode ser especificado ao calcular o modelo cox, que determina como os empates são tratados. O padrão é “efron”, e as outras opções são “breslow” e “exact”.
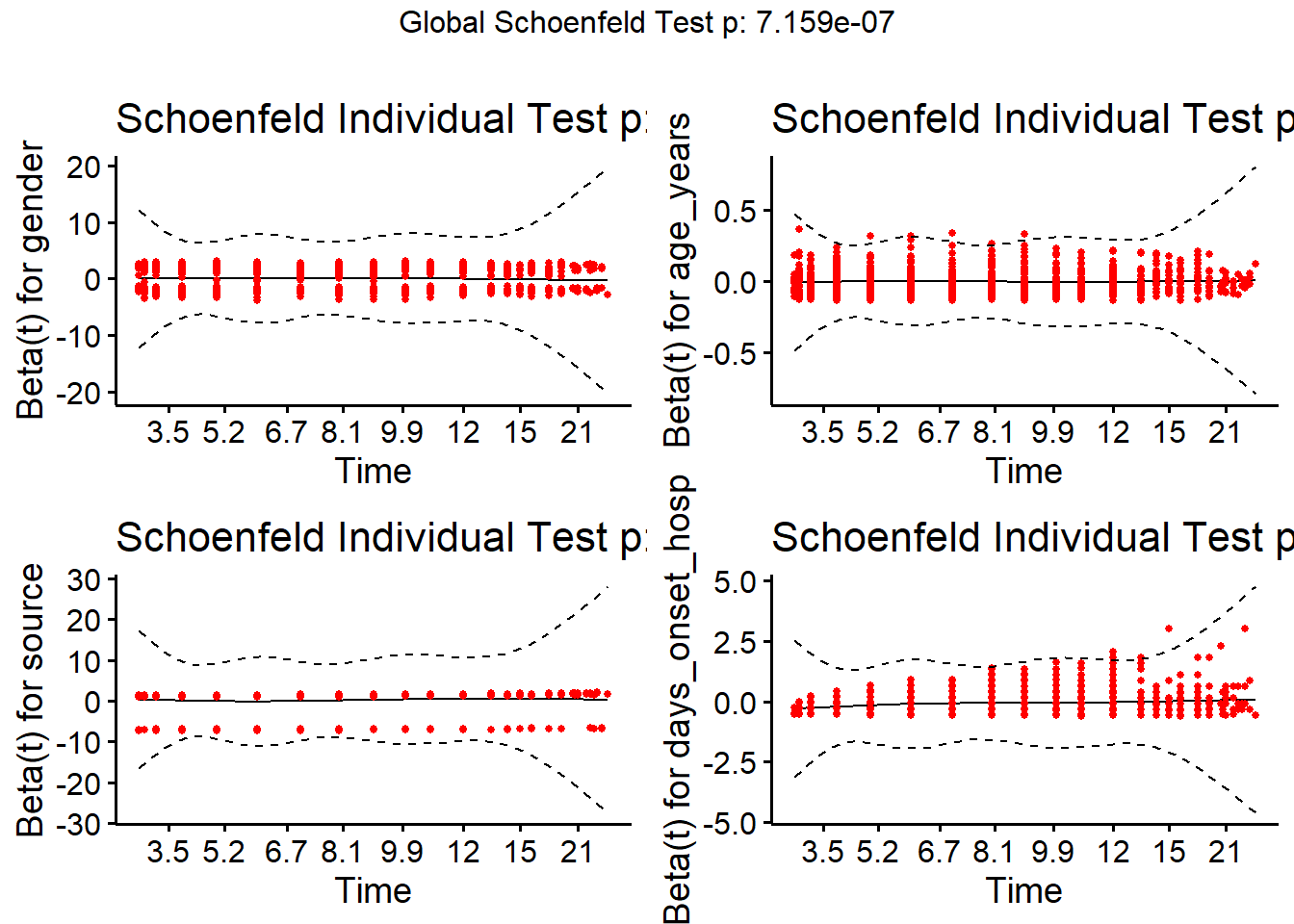
Num outro modelo, adicionamos mais fatores de risco, tais como a fonte da infecção e o número de dias entre a data de início e a admissão. Desta vez, verificamos primeiro a hipótese de riscos proporcionais antes de avançarmos.
Neste modelo, incluímos um preditor contínuo (days_onset_hosp). Neste caso, interpretamos as estimativas dos parâmetros como o aumento do registo esperado do risco relativo para cada aumento de uma unidade no preditor, mantendo constantes outros preditores. Primeiro verificamos a suposição de perigos proporcionais.
#cria o modelo
linelistsurv_cox <- coxph(
Surv(futime, event) ~ gender + age_years+ source + days_onset_hosp,
data = linelist_surv
)
#testa o modelo de risco proporcional
linelistsurv_ph_test <- cox.zph(linelistsurv_cox)
linelistsurv_ph_test chisq df p
gender 0.45062 1 0.50
age_years 0.00199 1 0.96
source 1.79622 1 0.18
days_onset_hosp 31.66167 1 1.8e-08
GLOBAL 34.08502 4 7.2e-07A verificação gráfica desta hipótese pode ser realizada com a função ggcoxzph() do pacote survminer.
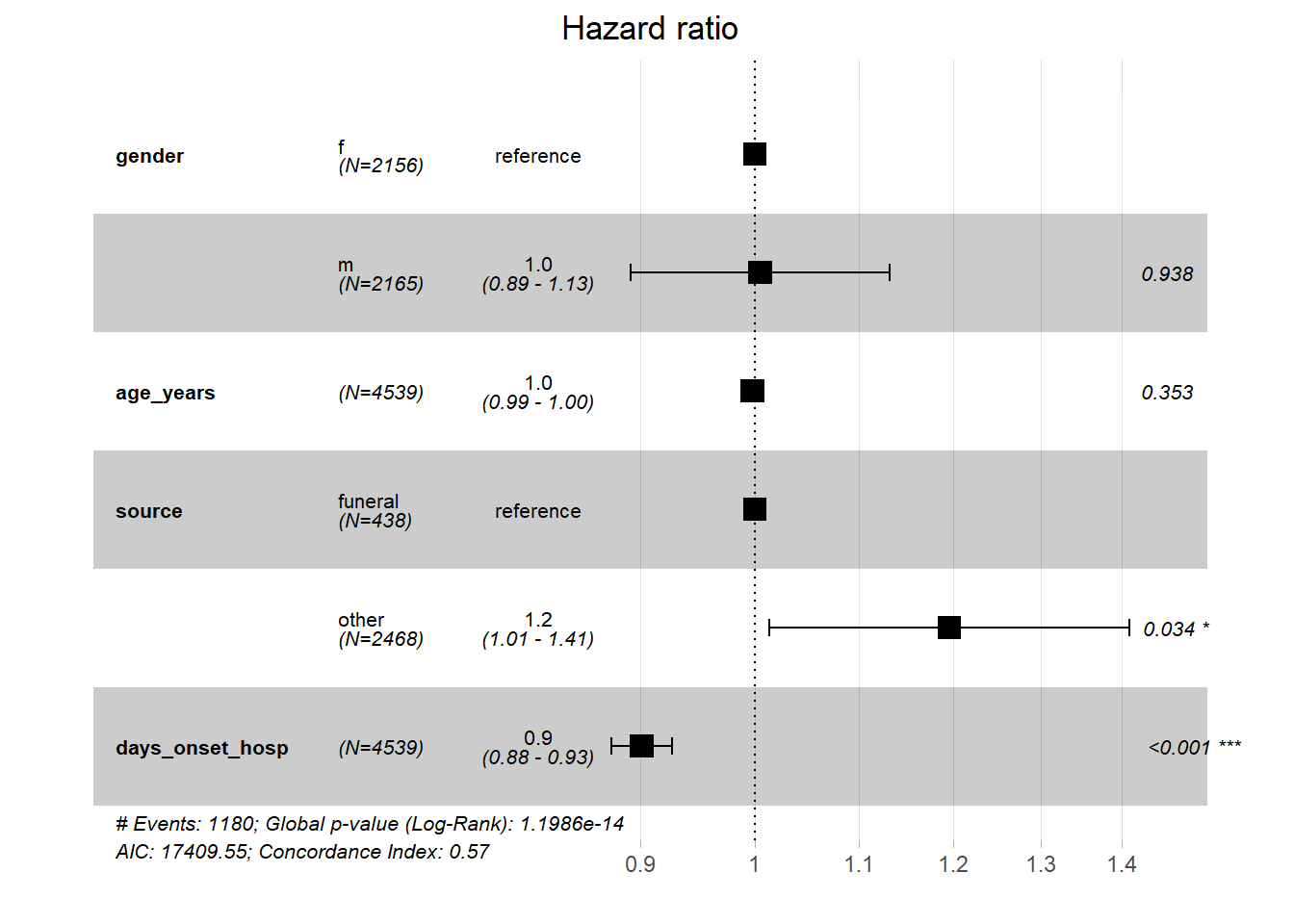
Os resultados do modelo indicam a existência de uma associação negativa entre o início da duração da admissão e a mortalidade por todas as causas. O risco esperado é 0,9 vezes menor numa pessoa que é admitida um dia mais tarde do que outra, mantendo constante o gênero. Ou, numa explicação mais direta, um aumento de uma unidade na duração do início da admissão está associado a uma diminuição de 10,7% (coef *100) no risco de morte.
Os resultados mostram também uma associação positiva entre a fonte da infecção e a mortalidade por todas as causas. Ou seja, há um aumento do risco de morte (1,21x) para os pacientes que têm outra fonte de infecção.
#resumo do modelo
summary(linelistsurv_cox)Call:
coxph(formula = Surv(futime, event) ~ gender + age_years + source +
days_onset_hosp, data = linelist_surv)
n= 2772, number of events= 1180
(1767 observations deleted due to missingness)
coef exp(coef) se(coef) z Pr(>|z|)
genderm 0.004710 1.004721 0.060827 0.077 0.9383
age_years -0.002249 0.997753 0.002421 -0.929 0.3528
sourceother 0.178393 1.195295 0.084291 2.116 0.0343 *
days_onset_hosp -0.104063 0.901169 0.014245 -7.305 2.77e-13 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
exp(coef) exp(-coef) lower .95 upper .95
genderm 1.0047 0.9953 0.8918 1.1319
age_years 0.9978 1.0023 0.9930 1.0025
sourceother 1.1953 0.8366 1.0133 1.4100
days_onset_hosp 0.9012 1.1097 0.8764 0.9267
Concordance= 0.566 (se = 0.009 )
Likelihood ratio test= 71.31 on 4 df, p=1e-14
Wald test = 59.22 on 4 df, p=4e-12
Score (logrank) test = 59.54 on 4 df, p=4e-12Podemos verificar essa relação com a tabela:
linelist_case_data %>%
tabyl(days_onset_hosp, outcome) %>%
adorn_percentages() %>%
adorn_pct_formatting() days_onset_hosp Death Recover NA_
0 44.3% 31.4% 24.3%
1 46.6% 32.2% 21.2%
2 43.0% 32.8% 24.2%
3 45.0% 32.3% 22.7%
4 41.5% 38.3% 20.2%
5 40.0% 36.2% 23.8%
6 32.2% 48.7% 19.1%
7 31.8% 38.6% 29.5%
8 29.8% 38.6% 31.6%
9 30.3% 51.5% 18.2%
10 16.7% 58.3% 25.0%
11 36.4% 45.5% 18.2%
12 18.8% 62.5% 18.8%
13 10.0% 60.0% 30.0%
14 10.0% 50.0% 40.0%
15 28.6% 42.9% 28.6%
16 20.0% 80.0% 0.0%
17 0.0% 100.0% 0.0%
18 0.0% 100.0% 0.0%
22 0.0% 100.0% 0.0%
NA 52.7% 31.2% 16.0%Teríamos de considerar e investigar porque é que esta associação existe nos dados. Uma explicação possível poderia ser que os pacientes que vivem o tempo suficiente para serem admitidos mais tarde, tem uma doença menos grave. Outra explicação talvez mais provável é que, uma vez que utilizámos um conjunto de dados falso simulado, este padrão não reflete a realidade!
Gráficos em floresta (Forest plots)
Podemos visualizar os resultados do modelo Cox usando “forest plots” com a função ggforest() do pacote survminer.
27.6 Covariáveis dependentes do tempo em modelos de sobrevivência
Algumas das seções seguintes foram adaptadas com permissão de uma excelente introdução à análise de sobrevivência em R pela Dra. Emily Zabor
Na última seção usamos a regressão Cox para examinar associações entre covariáveis de interesse e resultados de sobrevivência. Mas estas análises dependem da covariância ser medida na linha de base, ou seja, antes do tempo de seguimento do evento começar.
O que acontece se estiver interessado em uma covariável que é medida após o início do tempo de seguimento? Ou, e se uma dessas covariáveis possa mudar ao longo do tempo?
Por exemplo, talvez esteja trabalhando com dados clínicos onde repetiu medidas de valores laboratoriais hospitalares que podem mudar ao longo do tempo. Este é um exemplo de uma Covariável Dependente do Tempo. Para resolver este problema é necessário uma configuração especial, mas felizmente o modelo cox é muito flexível e este tipo de dados também pode ser modelado com ferramentas do pacote survival.
Configuração de covariável dependente do tempo
A análise de covariáveis dependentes do tempo em R requer a configuração de um conjunto de dados especial. Se estiver interessado, ver o artigo mais detalhado sobre este assunto do autor do pacote survival Using Time Dependent Covariates and Time Dependent Coefficients in the Cox Model.
Para tal, utilizaremos um novo conjunto de dados do pacote SemiCompRisks' chamadoBMT’, que inclui dados sobre 137 pacientes de transplante de medula óssea. As variáveis em que nos vamos concentrar são:
T1- tempo (em dias) até à morte ou último seguimento
delta1- indicador de óbito; 1-Óbito, 0-Vivos
TA- tempo (em dias) até à doença aguda de enxerto-versus-hospedeiro
deltaA- indicador de doença aguda de enxerto-versus-hospedeiro;- 1 - desenvolveu a doença aguda de enxerto-versus-hospedeiro
- 0 - Nunca desenvolveu doença aguda de enxerto-versus-hospedeiro
- 1 - desenvolveu a doença aguda de enxerto-versus-hospedeiro
Vamos carregar este conjunto de dados a partir do pacote survival utilizando o comando do R base data(), que pode ser utilizado para carregar dados que já estão incluídos num pacote R que é carregado. A moldura de dados BMT irá aparecer no seu ambiente R.
data(BMT, package = "SemiCompRisks")Adicionar identificador único de doente
Não existe uma coluna de ID única nos dados BMT, que é necessária para criar o tipo de conjunto de dados que desejamos. Assim, utilizamos a função rowid_to_column() do pacote tidyverse para criar uma nova coluna de id chamada my_id (adiciona coluna no início do Data Frame com ids sequenciais de linha, começando em 1). Nomeamos o data frame como bmt.
bmt <- rowid_to_column(BMT, "my_id")O conjunto de dados tem agora este aspecto:
Expandir as linhas de pacientes
A seguir, utilizaremos a função tmerge() com as funções auxiliares event() e tdc() helper para criar o conjunto de dados reestruturado. Neste caso, cada paciente pode ter no máximo duas linhas, dependendo se desenvolveu doença aguda de enxerto-versus-hospedeiro durante o período de coleta de dados. Vamos chamar o nosso novo indicador para o desenvolvimento da doença aguda de enxerto-versus-hospedeiro agvhd.
tmerge()cria um longo conjunto de dados com múltiplos intervalos de tempo para os diferentes valores covariados para cada pacienteevent()cria o novo indicador de evento para combinar com os intervalos de tempo recém-criadostdc()cria a coluna covariada dependente do tempo,agvhd, para combinar com os intervalos de tempo recém-criados
td_dat <-
tmerge(
data1 = bmt %>% select(my_id, T1, delta1),
data2 = bmt %>% select(my_id, T1, delta1, TA, deltaA),
id = my_id,
death = event(T1, delta1),
agvhd = tdc(TA)
)Para ver o que foi feito, vamos olhar os dados dos primeiros 5 pacientes.
As variáveis de interesse do dado original terão essa aparência:
bmt %>%
select(my_id, T1, delta1, TA, deltaA) %>%
filter(my_id %in% seq(1, 5)) my_id T1 delta1 TA deltaA
1 1 2081 0 67 1
2 2 1602 0 1602 0
3 3 1496 0 1496 0
4 4 1462 0 70 1
5 5 1433 0 1433 0O novo conjunto de dados para esses pacientes terá essa aparência:
td_dat %>%
filter(my_id %in% seq(1, 5)) my_id T1 delta1 tstart tstop death agvhd
1 1 2081 0 0 67 0 0
2 1 2081 0 67 2081 0 1
3 2 1602 0 0 1602 0 0
4 3 1496 0 0 1496 0 0
5 4 1462 0 0 70 0 0
6 4 1462 0 70 1462 0 1
7 5 1433 0 0 1433 0 0Agora alguns dos nossos pacientes têm duas linhas no conjunto de dados correspondentes a intervalos em que têm um valor diferente da nossa nova variável, agvhd. Por exemplo, o Paciente 1 tem agora duas linhas com um valor agvhd de zero do tempo 0 ao tempo 67, e um valor de 1 do tempo 67 ao tempo 2081.
Regressão Cox com covariáveis dependentes do tempo
Agora que remodelamos os nossos dados e acrescentamos a nova variável dependente do tempo aghvd , vamos encaixar um modelo simples de regressão de uma única variável cox. Podemos utilizar a mesma função coxph() como antes, só precisamos alterar a nossa função Surv() para especificar tanto o tempo de início como o tempo de paragem para cada intervalo utilizando os argumentos time1 = e time2 =.
bmt_td_model = coxph(
Surv(time = tstart, time2 = tstop, event = death) ~ agvhd,
data = td_dat
)
summary(bmt_td_model)Call:
coxph(formula = Surv(time = tstart, time2 = tstop, event = death) ~
agvhd, data = td_dat)
n= 163, number of events= 80
coef exp(coef) se(coef) z Pr(>|z|)
agvhd 0.3351 1.3980 0.2815 1.19 0.234
exp(coef) exp(-coef) lower .95 upper .95
agvhd 1.398 0.7153 0.8052 2.427
Concordance= 0.535 (se = 0.024 )
Likelihood ratio test= 1.33 on 1 df, p=0.2
Wald test = 1.42 on 1 df, p=0.2
Score (logrank) test = 1.43 on 1 df, p=0.2Novamente vamos visualizar o resultado do nosso modelo cox usando a função ggforest() do pacote survminer.:
Como se pode ver pela “forest plot”, intervalo de confiança e p-valor, não parece haver forte associação entre ocorrência de óbito e a doença aguda do enxerto-versus-hospedeiro no contexto do nosso modelo simples.
27.7 Referências
Survival Analysis Part I: Basic concepts and first analyses
Survival analysis in infectious disease research: Describing events in time
Chapter on advanced survival models Princeton
Using Time Dependent Covariates and Time Dependent Coefficients in the Cox Model
Cheatsheet (cola) de análise de sobrevivência