3 Introdução ao R
Bem-vindo!
Esta página analisa o essencial de R. Ela não pretende ser um tutorial abrangente, mas fornece o básico e pode ser útil para refrescar a sua memória. A secção Recurso para a aprendizagem tem links para a tutoriais mais abrangentes.
Partes desta página foram adaptadas com a permissão do projecto R4Epis.
Ver a página em Transição para R para dicas sobre como mudar para R de STATA, SAS, ou Excel.
3.1 Porque usar o R?
Segundo a definição do site R project, R é uma linguagem de programação e também um ambiente para computação estatística e gráfica. É altamente versátil, extensível, e orientado para a comunidade.
Custo
R é de utilização livre! Há uma política forte na comunidade para que o material seja gratuito e de código aberto.
Reprodutibilidade
A prática de conduzir a análise e a gestão dos dados por meio de uma linguagem de programação (em comparação com o Excel ou outra dessas ferramentas que requerem cliques manuais) melhora a reprodutibilidade, torna a detecção de erros mais fácil e alivia sua carga de trabalho.
Comunidade
A comunidade de usuários de R é enorme e colaborativa. Novos pacotes e ferramentas para resolver problemas da vida real são desenvolvidos diariamente e recebe críticas e contribuições da comunidade de utilizadores. Como um exemplo, R-Ladies é uma organização mundial cuja missão é promover a diversidade de gênero na comunidade R, e é uma das maiores organizações de utilizadores de R. É provável que tenha uma divisão perto de você!
3.2 Termos chave
RStudio - RStudio é uma Interface Gráfica de Utilizador (GUI) para uma utilização mais fácil da linguagem R. Leia mais na secção RStudio.
Objetos - Tudo o que se armazena em R - conjuntos de dados, variáveis, uma lista de nomes de cidades, um número total da população, mesmo saídas como gráficos - são objetos aos quais é atribuído um nome e podem ser referenciados em comandos posteriores. Ler mais na secção Objectos.
Funções - Uma função é uma operação de código que aceita entradas e devolve uma saída transformada. Leia mais na secção Funções.
Pacotes - Um pacote R é um pacote partilhável de funções. Leia mais na secção Pacotes
Scripts/Códigos - Um script é um arquivo que contém as linhas de comando que escreveu. Leia mais na secção Scripts
3.3 Recursos para a aprendizagem
Recursos dentro do RStudio
Documentação de ajuda
Pesquisar na aba “Ajuda” do RStudio pela documentação sobre pacotes do R e funções específicas. Isto está dentro do painel que também contém “Arquivos”, “Gráficos” e “Pacotes” (tipicamente no painel inferior direito). Como atalho, também se pode digitar o nome de um pacote ou função no console do R após um ponto de interrogação para abrir a página de Ajuda relevante. Não inclui parênteses.
Por exemplo: ?filter ou ?diagrammeR.
Tutoriais interativos
Há várias maneiras de aprender R interactivamente dentro do RStudio.
O próprio RStudio oferece um painel Tutorial que é alimentado pelo pacote do R learnr. Basta instalar este pacote e abrir um tutorial através da nova aba “Tutorial” no painel superior direito do RStudio, que também contém os separadores Environment (Ambiente) e History (Histórico).
O pacote do R swirl oferece cursos interativos no Console do R. Instale e carregue este pacote, depois execute o comando swirl() (parênteses vazios) no Console do R. Você verá avisos aparecerem no Console. Responda digitando no Console. Ele irá guiá-lo através de um curso à sua escolha.
Página de dicas
Há várias “cheatsheets” (“colinhas”) em pdf disponíveis no website do RStudio, por exemplo:
- Fatores com o pacote forcats
- Datas e horas com o pacote lubridate
- Caracteres com o pacote stringr
- Operações iterativas com o pacote purrr
- Importação de dados
- Folha de dica para transformação de dadas com o pacote dplyr
- R Markdown (para criar documentos como PDF, Word, Powerpoint…)
- Shiny (para construir aplicativos de web interativos)
- Visualização de dados com o pacote ggplot2
- Cartografia (GIS)
- Pacote leaflet (mapas interativos)
- Python com R (pacote reticulate)
Este é um recurso online do R especificamente para usuários do Excel
R tem uma vibrante comunidade no twitter onde você pode aprender dicas, atalhos e notícias, siga estas contas:
- Siga nos! @epiRhandbook
- R Function A Day (Uma função por dia) @rfuntionaday é um recurso incrível
- R for Data Science (R para Ciência de Dados) @rstats4ds
- RStudio @RStudio
- Dicas do RStudio@rstudiotips
- R-Bloggers @Rbloggers
- R-ladies @RLadiesGlobal
- Hadley Wickham @hadleywickham
Também:
#epitwitter e #rstats
Recursos online gratuitos
Um texto definitivo é o livro R for Data Science de Garrett Grolemund e Hadley Wickham
O site do projeto R4Epis tem como objetivo “desenvolver ferramentas padronizadas de limpeza, análise e relatório de dados para cobrir tipos comuns de surtos e pesquisas populacionais que seriam conduzidas em um ambiente de resposta a emergências de MSF”. Você pode encontrar materiais de treinamento básicos do R, modelos para relatórios do RMarkdown sobre surtos e pesquisas e tutoriais para ajudá-lo a configurá-los.
Idiomas além do inglês
Materiais do RStudio em Espanhol
3.4 Instalação
R e R Studio
Como instalar o R
Visite o site https://www.r-project.org/ e faça o download da versão mais recente do R que seja adequada para o seu computador.
Como instalar o RStudio
Visite esse website https://rstudio.com/products/rstudio/download/ e faça o download da versão gratuita mais recente para Desktop do RStudio que seja adequada para seu computador.
Permissões
Note que você deve instalar R e RStudio em um drive onde você tenha permissões de leitura e escrita. Caso contrário, sua capacidade de instalar pacotes R (um problema de ocorrência freqüente) será impactada. Se você encontrar problemas, tente abrir o RStudio clicando com o botão direito do mouse no ícone e selecionando “Executar como administrador”. Outras dicas podem ser encontradas na página R em unidades de rede.
Como atualizar o R e o RStudio
Sua versão de R é exibida (“printada”) no o Console R na inicialização. Você também pode executar o sessionInfo().
Para atualizar o R, vá para o site mencionado acima e reinstale o R. Alternativamente, você pode utilizar o pacote installr (no Windows) executando installr::updateR(). Isto abrirá caixas de diálogo para ajudá-lo a baixar a última versão R e atualizar seus pacotes para a nova versão R. Mais detalhes podem ser encontrados na installr documentação.
Esteja ciente de que a antiga versão R ainda existirá em seu computador. Você pode executar temporariamente uma versão antiga (antiga “instalação”) do R clicando em “Tools” (Ferramentas) -> “Global Options” (Opções Globais) no RStudio e escolhendo uma versão R. Isto pode ser útil se você quiser usar um pacote que não tenha sido atualizado para funcionar na versão mais nova do R.
Para atualizar o RStudio, você pode ir ao site acima e fazer o download novamente do RStudio. Outra opção é clicar em “Help” (Ajuda) -> “Check for Updates” (Verificar Atualizações) dentro do RStudio, mas isto pode não mostrar as últimas atualizações.
Para ver quais versões do R, RStudio, ou pacotes foram usados quando este Manual foi feito, veja a página em Notas editoriais e técnicas.
Outros softwares você pode precisar instalar
- TinyTeX (para compilaar documentos de RMarkdown em PDF)
- Pandoc (para compilar documentos de RMarkdown)
- RTools (para construir pacotes em R)
- phantomjs (para salvar imagens ou redes animadas, como redes de transmissão)
TinyTex
TinyTex é uma distribuição do LaTeX customizada, útil quando for tentar produzir PDFs no R. Veja https://yihui.org/tinytex/ para mais informações.
Para instalar o TinyTex no R:
install.packages('tinytex')
tinytex::install_tinytex()
# para desisntalar o TinyTeX, corra tinytex::uninstall_tinytex()Pandoc
Sua versão de R é impressa no o Console R na inicialização. Você também pode executar o Pandoc é um conversor de documentos, um software separado do R. Ele vem junto com o RStudio , provavelmente não precisará ser baixado. Ele ajuda no processo de conversão de documentos Rmarkdown para formatos como .pdf e adição de funcionalidades complexas.
RTools
RTools é uma coleção de softwares para construção de pacotes para R.
Instale desse website: https://cran.r-project.org/bin/windows/Rtools/
phantomjs
Esta ferramenta é freqüentemente usada para tirar “fotografias da tela” de páginas da web. Por exemplo, quando você cria uma cadeia de transmissão com o pacote epicontacts, um arquivo HTML é produzido e ele é interativo e dinâmico. Se você quiser uma imagem estática, pode ser útil usar o pacote webshot para automatizar este processo. Isto exigirá o programa externo “phantomjs”. Você pode instalar o phantomjs através do pacote webshot com o comando webshot::install_phantomjs().
3.5 RStudio
Se orientando no RStudio
Primeiramente, abra o RStudio. Como os ícones deles podem ser muito parecidos, certifique-se de que você está abrindo mesmo o RStudio e não R.
Para que o RStudio funcione, você também deve ter R instalado no computador (veja acima as instruções de instalação).
RStudio é uma interface (GUI) para facilitar o uso de R. Você pode pensar em R como sendo o motor de um veículo, fazendo o trabalho crucial, e RStudio como a carroceria do veículo (com assentos, acessórios, etc.) que ajuda você realmente a usar o motor para seguir em frente! Você pode ver a ficha completa da interface de usuário do RStudio (PDF) aqui
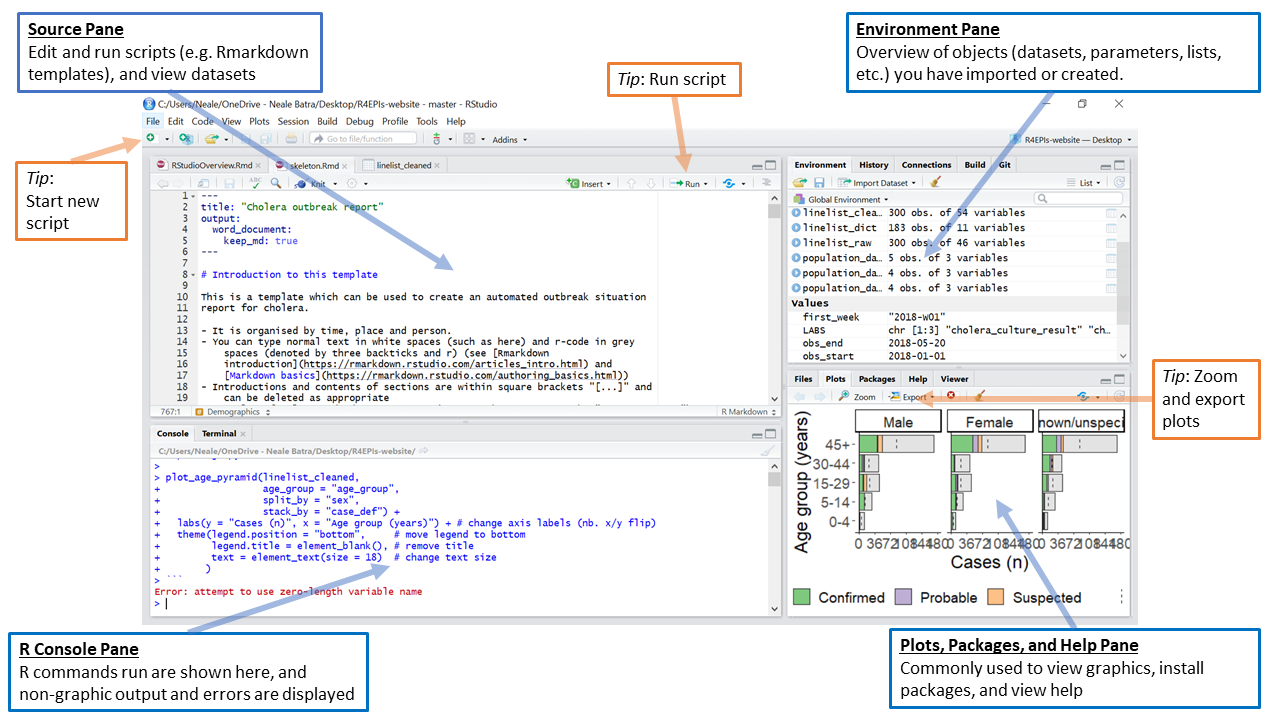
Por padrão, o RStudio exibe quatro painéis retangulares.
DICA: Se o seu RStudio mostrar apenas um painel esquerdo, é porque não há nenhum script aberto ainda
O Painel Fonte (Source) Este painel, por padrão localizado na parte superior esquerda, é um espaço onde é possível editar, executar e salvar seus scripts. Os scripts contêm os comandos que você deseja executar. Este painel também pode exibir conjuntos de dados (quadros de dados) para visualização.
Para usuários da Stata, este painel é similar às janelas de seu Do-file e Data Editor.
O Painel do Console R
O Console R, ou simplesmente Console, é por padrão o painel esquerdo ou inferior esquerdo do RStudio. Ele é a casa do “motor” de R. É aqui que os comandos são realmente executados e as saídas não gráficas e as mensagens de erro/aviso aparecem. Você pode digitar e executar comandos diretamente no Console, mas perceba que estes comandos não são salvos como são ao executar comandos de um script.
Se você está familiarizado com Stata, o Console R é como a Janela de Comando e também a Janela de Resultados.
O Painel Ambiente (Environment)
Este painel, por padrão na parte superior direita, é mais freqüentemente usado para ver resumos breves de objetos no Ambiente R na sessão atual. Estes objetos podem incluir conjuntos de dados importados, modificados ou criados, parâmetros que você definiu (por exemplo, uma semana epidemiológica específica para a análise), ou vetores ou listas que você definiu durante a análise (por exemplo, nomes de regiões). Você pode clicar na seta ao lado de um nome de data frame para ver suas variáveis.
No Stata, isto é mais parecido com a janela Gerenciador de Variáveis.
Este painel também contém a aba History onde você pode ver comandos que você pode ver anteriormente. Tem também uma aba “Tutorial” onde você pode completar tutoriais R interativos se você tiver o pacote learnr instalado. Tem também um painel “Conections” para conexões externas, e pode ter um painel “Git” se você optar por fazer interface com o Github.
Paineis: Gráficos (Plots), Visualizador (Viewer), Pacotes (Packages), Ajuda (Help)
O painel inferior-direito inclui várias abas importantes. Gráficos típicos, incluindo mapas, serão exibidos no painel Plo. Saídas interativas ou HTML serão exibidas no painel do Visualizador (Viewer). O painel de Ajuda (Help) pode exibir documentação e arquivos de ajuda. O painel Arquivos (Files) é um navegador que pode ser usado para abrir ou excluir arquivos. O painel Pacotes (Packages) permite ver, instalar, atualizar, excluir, carregar/descarregar pacotes R, e ver qual versão do pacote você tem. Para saber mais sobre pacotes, veja a seção pacotes abaixo.
Este painel contém os equivalentes Stata das janelas Plots Manager e Project Manager.
Configurações do RStudio
Altere as configurações e a aparência do RStudio no menu suspenso Ferramentas (Tools), selecionando Opções Globais (Global Options). Lá você pode alterar as configurações padrão, incluindo a cor da aparência/fundo.
Restart (Reiniciar)
Se seu R travar, você pode reiniciar o R indo ao menu Sessão (Session) e clicando em “Reiniciar R” (Restart R). Isto evita o incômodo de fechar e abrir o RStudio. Tudo em seu ambiente R será removido quando você fizer isso.
Atalhos no teclado
Alguns atalhos de teclado muito úteis estão abaixo. Veja todos os atalhos de teclado para Windows, Mac e Linux na segunda página deste RStudio cheatsheet da interface do usuário.
| Windows/Linux | Mac | Ação |
|---|---|---|
| Esc | Esc | Interrompe comando atual (útil se você acidentalmente executou um comando incompleto e não pode escapar de ver o “+” no console R) |
| Ctrl+s | Cmd+s | Salva (script) |
| Tab | Tab | Auto-completa |
| Ctrl + Enter | Cmd + Enter | Roda a(s) linha(s) atual(is)/seleção de código |
| Ctrl + Shift + C | Cmd + Shift + c | comenta/descomenta as linhas selecionadas |
| Alt + - | Option + - | Insere <- |
| Ctrl + Shift + m | Cmd + Shift + m | Insere %>% |
| Ctrl + l | Cmd + l | Limpa o console |
| Ctrl + Alt + b | Cmd + Option + b | Roda do início até a linha atual |
| Ctrl + Alt + t | Cmd + Option + t | Roda a seção de código atual (R Markdown) |
| Ctrl + Alt + i | Cmd + Shift + r | Insere chunk de código (no R Markdown) |
| Ctrl + Alt + c | Cmd + Option + c | Roda o chunk atual (R Markdown) |
| up/down arrows in R console | Same | Alterna entre os comandos executados recentemente |
| Shift + up/down arrows in script | Same | Seleciona múltiplas linhas de código |
| Ctrl + f | Cmd + f | Procura e substitui no script atual |
| Ctrl + Shift + f | Cmd + Shift + f | Encontra em arquivos (pesquisa/substitui em vários script) |
| Alt + l | Cmd + Option + l | Oculta código atual |
| Shift + Alt + l | Cmd + Shift + Option+l | Exibe código sele |
DICA: Use sua tecla Tab ao digitar para ativar a funcionalidade de auto-completar do RStudio. Isto pode evitar erros ortográficos. Pressione Tab enquanto digita para produzir um menu suspenso de funções e objetos prováveis, com base no que você digitou até agora.
3.6 Funções
As funções estão no cerne do uso de R. As funções são a forma como você executa tarefas e operações. Muitas funções vêm instaladas com R, muitas mais estão disponíveis para download em packages (explicado na seção pacotes), e você pode até mesmo escrever suas próprias funções personalizadas!
Esta seção básica sobre funções explica:
- O que é uma função e como funcionam
- O que são os argumentos/parâmetros de uma função
- Como conseguir ajuda para compreender uma função
Uma nota rápida sobre a sintaxe: Neste manual, as funções são escritas em texto de código com parênteses abertos, como este: filter(). Como explicado na seção pacotes, as funções são baixadas dentro de pacotes. Neste manual, os nomes dos pacotes são escritos em negrito, como dplyr. Às vezes, no código de exemplo, você pode ver o nome da função ligado explicitamente ao nome de seu pacote com um par de dois-pontos (::) como este: dplyr::filter(). O propósito desta ligação é explicado na seção de pacotes.
Funções simples
Uma função é como uma máquina que recebe entradas, faz alguma ação com essas entradas e produz uma saída. O que é a saída depende da função.
Funções normalmente operam sobre algum objeto colocado dentro dos parênteses da função. Por exemplo, a função sqrt() calcula a raiz quadrada de um número:
sqrt(49)[1] 7O objeto fornecido a uma função também pode ser uma coluna em um conjunto de dados (veja a seção Objetos para detalhes sobre todos os tipos de objetos). Como R pode armazenar vários conjuntos de dados, será necessário especificar tanto o conjunto de dados quanto a coluna. Uma maneira de fazer isso é utilizar a notação $ para ligar o nome do conjunto de dados e o nome da coluna (dataset$coluna). No exemplo abaixo, a função summary() é aplicada à coluna numérica age (idade) no conjunto de dados linelist, e a saída é um resumo dos valores numéricos e valores ausentes da coluna.
#Imprimir estatísticas resumidas da coluna 'age' (idade) no conjunto de dados 'linelist'.
summary(linelist$age) Min. 1st Qu. Median Mean 3rd Qu. Max. NA's
0.00 6.00 13.00 16.07 23.00 84.00 86 NOTA: Nos bastidores, uma função representa um código adicional complexo que foi condensado para o usuário em um comando fácil.
Funções com argumentos múltiplos
As funções frequentemente pedem várias entradas, chamadas argumentos, ou parâmetros, localizadas dentro dos parênteses da função, geralmente separadas por vírgulas.
- Alguns argumentos são necessários para que a função funcione corretamente, outros são opcionais
- Argumentos opcionais têm configurações padrão
- Os argumentos podem ter caráter, numérico, lógico (TRUE/FALSE)(verdadeiro/falso), e outros inputs
Aqui está uma função ficcional divertida, chamada oven_bake() ( do inglês: assar no forno), como um exemplo de uma função típica. Ela pega um objeto de entrada (por exemplo, um conjunto de dados, ou neste exemplo “dough” - massa) e realiza operações sobre ele como especificado por argumentos adicionais (minutes = e temperature =). A saída pode ser impressa para o console, ou salva como um objeto utilizando o operador de atribuição <-.
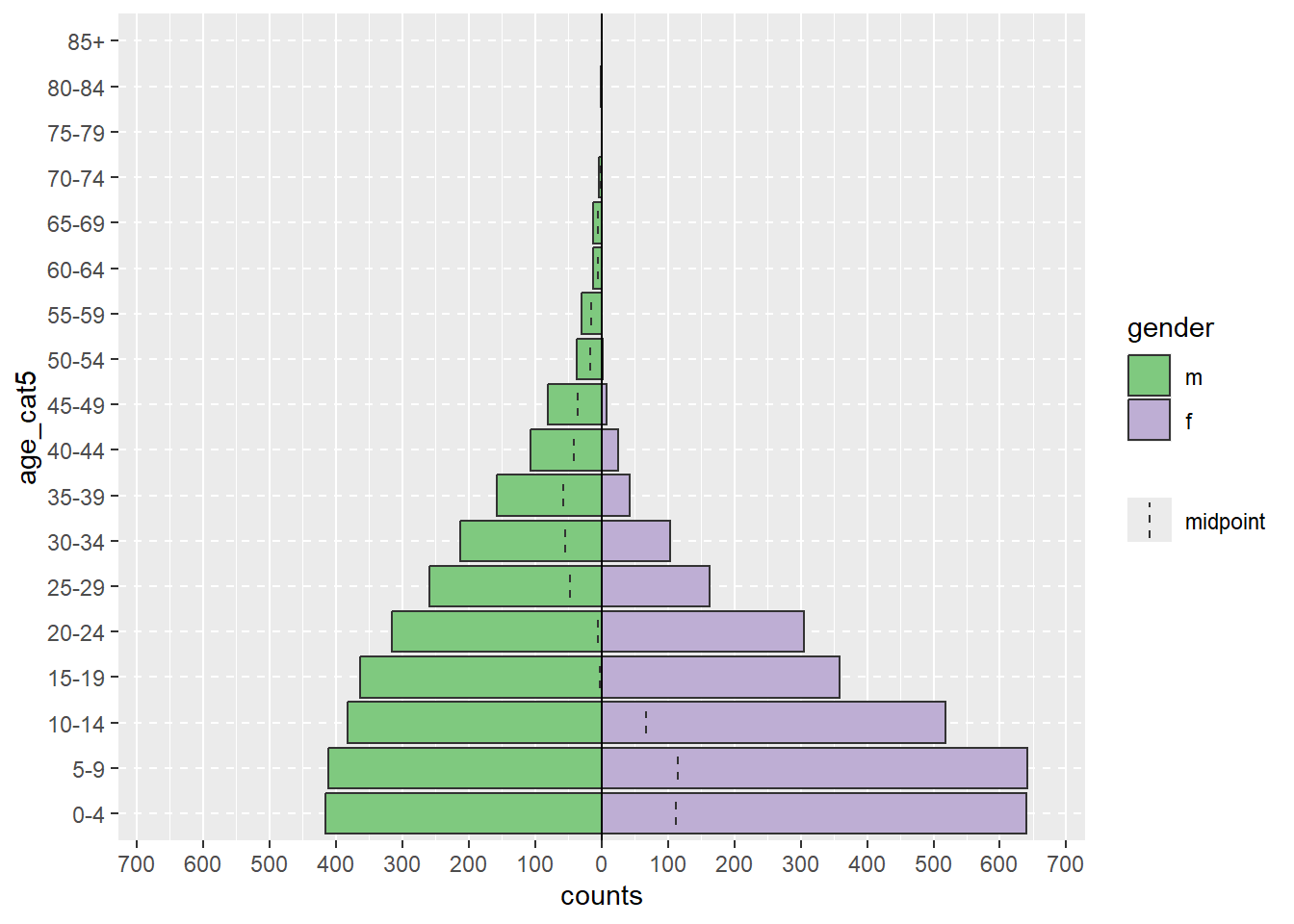
Em um exemplo mais realístico, o comando age_pyramid() abaixo produz uma pirâmide etária baseado em faixas-etárias e uma coluna divisória binária, como gender (gênero). A função recebe três argumentos dentro dos parênteses, separados por vírgulas. Os valores fornecidos aos argumentos estabelecem linelist como o quadro de dados a utilizar, age_cat5 como a coluna a contar, e gender como a coluna binária a utilizar para dividir a pirâmide por cor.
# Crie uma pirâmide etária
age_pyramid(data = linelist, age_group = "age_cat5", split_by = "gender")O comando acima pode ser escrito de forma equivalente ao abaixo, em um estilo mais longo com uma nova linha para cada argumento. Este estilo pode ser mais fácil de ler, e mais fácil de escrever “comentários” com # para explicar cada parte (comentar extensivamente é uma boa prática!). Para executar este comando mais longo, você pode destacar todo o comando e clicar em “Run”, ou simplesmente colocar o cursor na primeira linha e, em seguida, pressionar simultaneamente as teclas Ctrl e Enter.
# Crie uma pirâmide etária
age_pyramid(
data = linelist, # Use a linelist de casos
age_group = "age_cat5", # disponibilize a coluna de faixa-etária
split_by = "gender" #usee a coluna de gênero para criar dois lados da pirâmide
)A primeira metade de uma atribuição de argumentos (por exemplo, dados =) não precisa ser especificada se os argumentos forem escritos em uma ordem específica (especificada na documentação da função). O código abaixo produz exatamente a mesma pirâmide que o acima, porque a função espera a ordem dos argumentos: quadro de dados, variável age_group, variável split_by.
# Esse comando retorna o mesmo gráfico gerado acima
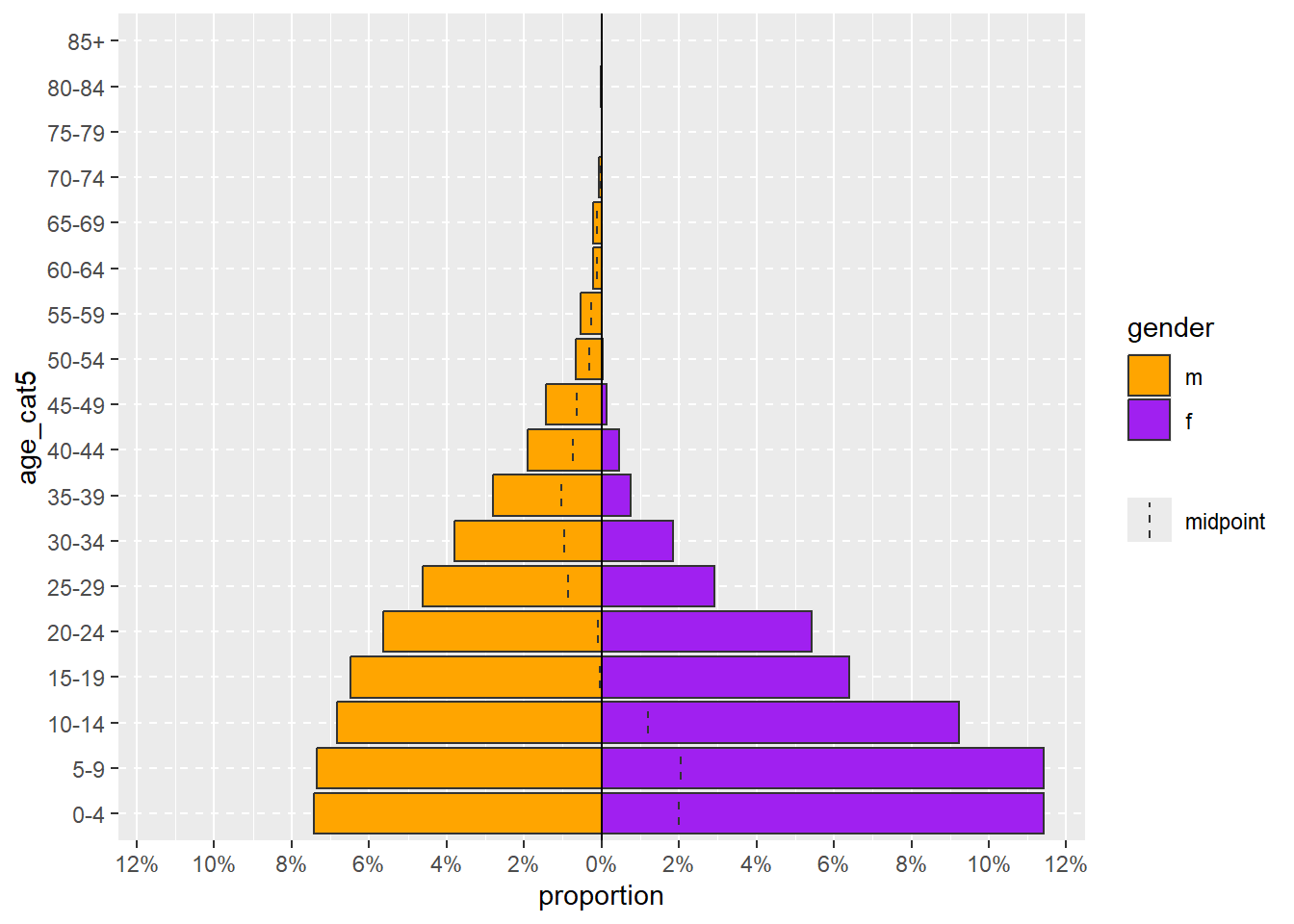
age_pyramid(linelist, "age_cat5", "gender")Um comando mais complexo de age_pyramid() poderia incluir os argumentos opcionais para:
- Mostrar proporções em vez de contagens (definir
proportional = TRUEquando o padrão éFALSE) - Especifique as duas cores a serem utilizadas (
pal =é a abreviação de “paleta” e é fornecido com um vetor de dois nomes de cores. Veja a página objetos para saber como a funçãoc()faz um vetor)
NOTA: Para argumentos que você especificar com ambas as partes do argumento (por exemplo, proportional = TRUE), sua ordem entre todos os argumentos não importa.
Escrevendo funções
R é uma linguagem que é orientada em torno de funções, portanto você deve se sentir capacitado para escrever suas próprias funções. A criação de funções traz várias vantagens:
- Para facilitar a programação modular - a separação do código em peças independentes e gerenciáveis
- Substituir o processo repetitivo de copia e cola, que pode ser propenso a erros.
- Dar nomes memoráveis aos pedaços de código
Como escrever uma função é abordado em profundidade na página Escrevendo funções.
3.7 Pacotes
Pacotes contém funções.
Um pacote R é um pacote compartilhável de código e documentação que contém funções pré-definidas. Os usuários da comunidade R desenvolvem pacotes o tempo todo, atendendo a problemas específicos, e é provável que alguém possa ajudar com seu trabalho inclusive! Você com certeza irá instalar e usar centenas de pacotes em seu uso de R.
Na instalação, o R contém pacotes de “base” e funções que executam tarefas elementares e comuns. Mas muitos usuários do R criam funções especializadas, que são verificadas pela comunidade R e que você pode baixar como um package (pacote) para seu próprio uso. Neste manual, os nomes dos pacotes estão escritos em negrito. Um dos aspectos mais desafiadores do R é que muitas vezes há muitas funções ou pacotes a serem escolhidos para complementar uma determinada tarefa.
Instalar e carregar
As funções estão contidas nos pacotes que podem ser baixados (“instalados”) para seu computador a partir da Internet. Uma vez que um pacote é baixado, ele é armazenado em sua “biblioteca” (do inglês, library). Você pode então acessar as funções que ele contém durante sua sessão R atual “carregando” este pacote.
Pense em R como sua biblioteca pessoal: Quando você baixa um pacote, sua biblioteca ganha um novo livro de funções, mas cada vez que você quiser usar uma função naquele livro, você deve pegar emprestado (“carregar”) aquele livro de sua biblioteca.
Em resumo: para usar as funções disponíveis em um pacote R, duas etapas devem ser implementadas:
O pacote precisa ser instalado (uma vez), e
O pacote precisa ser carregado (a cada sessão de R que abrir)
Sua biblioteca
Sua “biblioteca” é na verdade uma pasta em seu computador, contendo uma subpasta para cada pacote que foi instalado. Descubra onde R está instalado em seu computador, e procure uma pasta chamada “win-library”. Por exemplo: R\win-library\4.0 (a 4.0 é a versão R - você terá uma biblioteca diferente para cada versão R que você baixou).
Você pode exibir (“printar”) o caminho do arquivo para sua biblioteca digitando .libPaths() (parênteses vazios). Isto se torna especialmente importante se você trabalhar com R em unidades de rede.
Instalar do CRAN
Na maioria das vezes, os usuários R baixam pacotes da CRAN. CRAN (Comprehensive R Archive Network) é um repositório público on-line de pacotes R que foram publicados por membros da comunidade R.
Você está preocupado com vírus e segurança ao fazer o download de um pacote da CRAN? Leia este artigo sobre o assunto.
Como instalar e carregar
Neste manual, sugerimos o uso do pacote pacman (abreviação para “package manager” que significa “gerenciador de pacotes” em inglês). Ele oferece uma função conveniente p_load() que instalará um pacote se necessário e o carregará para utilização na sessão R atual.
A sintaxe é bastante simples. Basta listar os nomes dos pacotes dentro dos parênteses p_load(), separados por vírgulas. Este comando instalará os pacotes rio, tidyverse, e here se ainda não estiverem instalados, e os carregará para utilização. Isto torna a abordagem p_load() conveniente e concisa se compartilhar scripts com outros. Observe que os nomes dos pacotes são sensíveis a maiúsculas e minúsculas.
# Instala (se necessário) e os carrega para o uso
pacman::p_load(rio, tidyverse, here)Note que utilizamos a sintaxe pacman::p_load() que escreve explicitamente o nome do pacote (pacman) antes do nome da função (p_load()), conectado por duas colunas ::. Esta sintaxe é útil porque também carrega o pacote pacman (assumindo que já esteja instalado).
Existem funções alternativas do R base que você verá com freqüência. A função do R base para instalar um pacote é install.packages(). O nome do pacote a ser instalado deve ser fornecido entre parênteses em aspas. Se você quiser instalar vários pacotes em um comando, eles devem ser listados dentro de um vetor de caracteres c().
Nota: este comando instala um pacote, mas não o carrega para utilização na sessão atual.
# essa função disponível no R base instala um único pacote
install.packages("tidyverse")
# install multiple packages with base R
install.packages(c("tidyverse", "rio", "here"))A instalação também pode ser feita clicando e apontando para o painel “Pacotes” do RStudio e clicando em “Instalar” e procurando pelo nome do pacote desejado.
A função do R base para carregar um pacote para utilização (após ter sido instalado) é library(). Ela pode carregar apenas um pacote de cada vez (outro motivo para utilizar p_load()). Você pode fornecer o nome do pacote com ou sem aspas.
# com o R base, você pode carregar os pacotes dessa forma
library(tidyverse)
library(rio)
library(here)Para verificar se um pacote está instalado e/ou carregado, você pode visualizar o painel de Pacotes no RStudio. Se o pacote estiver instalado, ele é mostrado lá com o número da versão. Se sua caixa for marcada, ela é carregada para a sessão atual.
Instalar a partir do Github.
Às vezes, você precisa instalar um pacote que ainda não está disponível na CRAN. Ou talvez o pacote esteja disponível na CRAN, mas você quer a versão em desenvolvimento com novos recursos ainda não oferecidos na versão publicada mais estável da CRAN. Estes são frequentemente hospedados no site github.com em um “repositório” de código gratuito e voltado para o público. Leia mais sobre Github na página do manual Controle de versão e colaboração com Git e Github.
Para baixar os pacotes R do Github, você pode utilizar a função p_load_gh() do pacman, que instalará o pacote se necessário, e o carregará para utilização em sua sessão R atual. As alternativas para instalar incluem a utilização dos pacotes remotes ou devtools. Leia mais sobre todas as funções pacman na documentação do pacote.
Para instalar a partir do Github, você precisa fornecer mais informações. Você tem que fornecer:
A identificação do proprietário do repositório Github
O nome do repositório que contém o pacote
(opcional) O nome do “ramo” (o “branch” da versão de desenvolvimento específico) que você deseja baixar
Nos exemplos abaixo, a primeira palavra entre aspas representa a ID do Github do proprietário do repositório, após a barra é o nome do repositório (o nome do pacote).
# instala/carrega o pacote epicontacts do seu repositório Github
p_load_gh("reconhub/epicontacts")Se você quiser instalar de um “branch” (versão) diferente da principal, adicione o nome do “branch” após um “@”, após o nome do repositório.
# instale o "branch"(ramo) "timeline" do pacte epicontacts do Github
p_load_gh("reconhub/epicontacts@timeline")Se não houver diferença entre a versão do Github e a versão em seu computador, nenhuma ação será tomada. Você pode “forçar” uma reinstalação utilizando p_load_current_gh() com o argumento update = TRUE. Leia mais sobre pacman nesta vinheta online
Instale a partir de ZIP ou TAR
Você poderia instalar um pacote de um endereço URL:
packageurl <- "https://cran.r-project.org/src/contrib/Archive/dsr/dsr_0.2.2.tar.gz"
install.packages(packageurl, repos=NULL, type="source")Ou, faça o download dele para seu computador em um arquivo comprimido (“zipado”):
Opção 1: usandoinstall_local() do pacote remotes
remotes::install_local("~/Downloads/dplyr-master.zip")Opção 2: usando install.packages() do R base, fornecendo o caminho do arquivo comprimido e configurando os parâmetros type = "source e repos = NULL.
install.packages("~/Downloads/dplyr-master.zip", repos=NULL, type="source")Sintaxe do código
For clarity in this handbook, functions are sometimes preceded by the name of their package using the :: symbol in the following way: package_name::function_name()
Once a package is loaded for a session, this explicit style is not necessary. One can just use function_name(). However writing the package name is useful when a function name is common and may exist in multiple packages (e.g. plot()). Writing the package name will also load the package if it is not already loaded.
# Este comando usa o pacote "rio" e sua função "import()" para importar uma base de dados.
linelist <- rio::import("linelist.xlsx", which = "Sheet1")Auxílio para as funções
Para maior clareza neste manual, as funções são algumas vezes precedidas pelo nome de seu pacote utilizando o símbolo :: da seguinte forma: nome_do_pacote::nome_da_função()
Uma vez carregado um pacote para uma sessão, este estilo explícito não é mais necessário. Pode-se simplesmente utilizar nome_da_função(). Entretanto, escrever o nome do pacote é útil quando um nome de função é muito comum e pode existir em vários pacotes (por exemplo, plot()). Escrever o nome do pacote também irá carregar o pacote se ele ainda não estiver carregado.
Atualizando pacotes
Você pode atualizar os pacotes, reinstalando-os. Você também pode clicar no botão verde “Atualizar” em seu painel de Pacotes RStudio para ver quais pacotes têm novas versões para instalar. Esteja ciente de que seu código antigo pode precisar ser atualizado se houver uma grande revisão de como uma função funciona!
Apagar pacotes
Utilize p_delete() de pacman, ou remove.packages() do R base. Alternativamente, procure a pasta que contém sua biblioteca e exclua manualmente a pasta.
Dependências
Os pacotes muitas vezes dependem de outros pacotes para funcionar. Estes são chamados de dependências. Se uma dependência falhar na instalação, então o pacote dependendo dela também pode falhar na instalação.
Veja as dependências de um pacote com p_depends(), e veja quais pacotes dependem dele com p_depends_reverse().
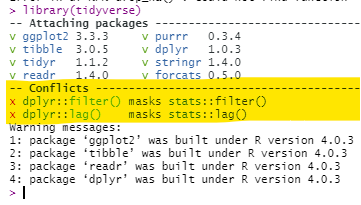
Funções mascaradas
Não é raro que dois ou mais pacotes contenham o mesmo nome de função. Por exemplo, o pacote dplyr tem uma função filter(), mas o pacote stats também. A função padrão filter() depende da ordem em que estes pacotes são carregados primeiro na sessão R - a última será o padrão para o comando filter().
Você pode verificar a ordem em seu painel Environment (Ambiente) do R Studio - clique no menu suspenso para Global Environment (“Ambiente Global”) e veja a ordem dos pacotes. Funções de pacotes mais abaixo nessa lista suspensa mascararão funções com o mesmo nome em pacotes que aparecem mais altos na lista suspensa. Ao carregar um pacote pela primeira vez, R avisará no console se estiver ocorrendo mascaramento, mas isto pode ser fácil de perder.
- Especifique o nome do pacote no comando. Por exemplo, utilize
dplyr::filter() - Reorganize a ordem na qual os pacotes são carregados (por exemplo, dentro de
p_load()), e inicie uma nova sessão R.
Desprender / descarregar
Para separar (descarregar) um pacote, use este comando, com o nome correto do pacote e apenas um “dois pontos”. Note que isto pode não resolver o mascaramento.
detach(package:PACKAGE_NAME_HERE, unload=TRUE)Instalar uma versão mais antiga
Veja este guia para instalar uma versão mais antiga de um pacote em particular.
Pacotes sugeridos
Veja a página em Pacotes sugeridos para uma listagem de pacotes que recomendamos para o dia-a-dia em epidemiologia.
3.8 Scripts
Os scripts (que significa “roteiro” em inglês) são uma parte fundamental da programação. Eles são documentos que contêm seus comandos (por exemplo, funções para criar e modificar conjuntos de dados, visualizações de impressão, etc.). Você pode salvar um script e executá-lo novamente mais tarde. Há muitas vantagens em armazenar e executar seus comandos a partir de um script (versus digitar comandos um a um na “linha de comando” do console R):
- Portabilidade - você pode compartilhar seu trabalho com outros enviando-lhes seus scripts
- Reprodutibilidade - para que você e outros saibam exatamente o que você fez
- Controle de versão - para que você possa acompanhar as mudanças feitas por você ou colegas
- Comentando/anotando - para explicar a seus colegas o que você tem feito
Comentando
Em um script você também pode anotar (“fazer comentários”) ao longo do seu código R. Comentar é útil para explicar a si mesmo e aos outros leitores o que você está fazendo. Você pode adicionar um comentário digitando o símbolo “hashtag” (#) e escrevendo seu comentário depois dele. O texto comentado aparecerá em uma cor diferente da do código R.
Qualquer código escrito após o # não será executado. Portanto, colocar um # antes do código também é uma maneira útil de bloquear temporariamente uma linha de código (“comentar fora”) se você não quiser apagá-lo). Você pode fazer isso em várias linhas ao mesmo tempo, selecionando-as e pressionando Ctrl+Shift+c (Cmd+Shift+c no Mac).
# Um comentário pode ser uma linha por si só
# importar dados
linelist <- import("linelist_raw.xlsx") %>% # também pode ser após o código
# filter(age > 50) # também pode ser usado para desativar uma linha de código
count()- Comente sobre o que você está fazendo e sobre por que você está fazendo.
- Divida seu código em seções lógicas…
- Acompanhe seu código com uma descrição passo a passo em texto do que você está fazendo (por exemplo, passos numerados)
Estilo
É importante estar consciente de seu estilo de codificação - especialmente se estiver trabalhando em equipe. Defendemos o uso do tidyverse guia de estilo. Há também pacotes como styler e lintr que o ajudam a se adequar a este estilo.
Alguns pontos muito básicos para tornar seu código legível para outros:
* Ao nomear objetos, utilize apenas letras minúsculas, números e sublinhados _, por exemplo my_data. * Utilize espaços frequentes, inclusive ao redor dos operadores, por exemplo n = 1 e age_new <- age_old + 3
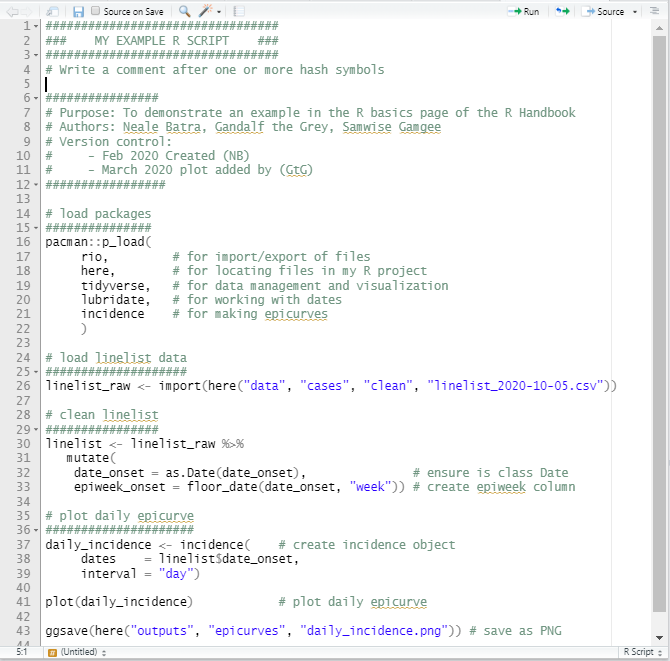
Exemplo de Script
Abaixo está um exemplo de um pequeno script R. Lembre-se, quanto melhor você explicar sucintamente seu código nos comentários, mais seus colegas vão gostar de você!
R markdown
Um script R markdown é um tipo de script R no qual o script em si resulta um documento de saída (PDF, Word, HTML, Powerpoint, etc.). Estas são ferramentas incrivelmente úteis e versáteis, freqüentemente usadas para criar relatórios dinâmicos e automatizados. Mesmo este website e manual são produzidos com um script R markdown!
Vale a pena notar que usuários iniciantes de R também podem usar R Markdown - não se intimidem! Para saber mais, consulte a página do manual nos documentos Relatórios com R Markdown.
Notebooks R
Não há diferença entre escrever em um Rmarkdown versus um caderno R. Entretanto, a execução do documento difere ligeiramente. Consulte este site para obter mais detalhes.
3.8.1 Shiny
Os aplicativos/websites shiny estão contidas em um script, que deve ser chamado de app.R. Este arquivo tem três componentes:
- Uma interface de usuário (ui)
- Uma função de servidor
- Uma chamada para a função
shinyApp.
Veja a página do manual em Dashboards com Shiny, ou este tutorial online: Tutorial com shiny
Nos tempos mais antigos, o arquivo acima era dividido em dois arquivos (ui.R e server.R)
Código dobrável
Você pode colapsar/dobrar (do inglês folding) porções de código para facilitar a leitura de seu roteiro.
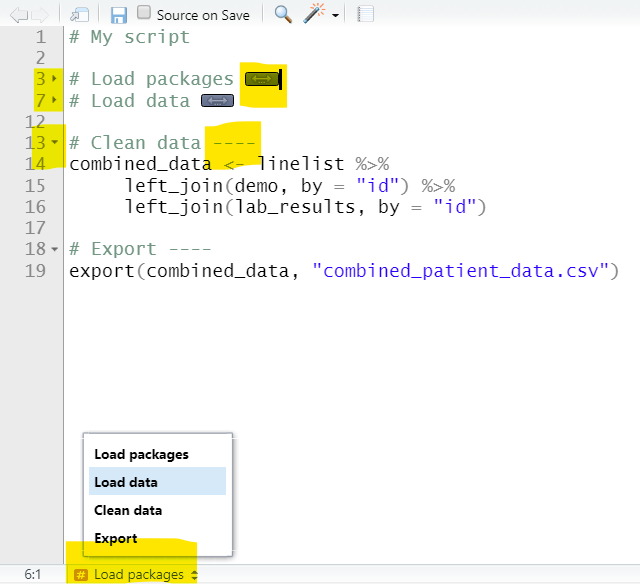
Para isso, crie um cabeçalho de texto com #, escreva seu cabeçalho, e siga-o com pelo menos 4 traços (-), hashes (#) ou igual a (=). Quando você tiver feito isto, uma pequena seta aparecerá na “sarjeta” à esquerda (pelo número da linha). Você pode clicar nesta seta e o código abaixo até o próximo cabeçalho cair e um ícone de seta dupla aparecerá em seu lugar.
Para expandir o código, clique novamente na seta na sarjeta, ou no ícone de duas fileiras. Há também atalhos de teclado como explicado na seção RStudio desta página.
Ao criar cabeçalhos com #, você também ativará o Índice na parte inferior de seu script (veja abaixo) que você pode usar para navegar em seu script. Você pode criar subtítulos adicionando mais # símbolos, por exemplo, # para primário, # # para secundário e ### para terciário.
Abaixo estão duas versões de um script de exemplo. À esquerda está o original com os cabeçalhos comentados. À direita, quatro traços foram escritos após cada cabeçalho, tornando-os colapsáveis. Dois deles foram colapsados, e você pode ver que a Tabela de Conteúdos na parte inferior agora mostra cada seção.
Outras áreas de código que são automaticamente elegíveis para fazer esse “dobramento” incluem as regiões entre chaves { } tais como definições de funções ou blocos condicionais ( declarações “if else”). Pode ler mais sobre dobramento de código no RStudio site.
3.9 Diretório de trabalho
O diretório de trabalho é o local da pasta raiz usada por R para seu trabalho - onde R procura e salva arquivos por padrão. Por padrão, ele salvará novos arquivos e saídas para este local, e procurará por arquivos para importar (por exemplo, conjuntos de dados) também aqui.
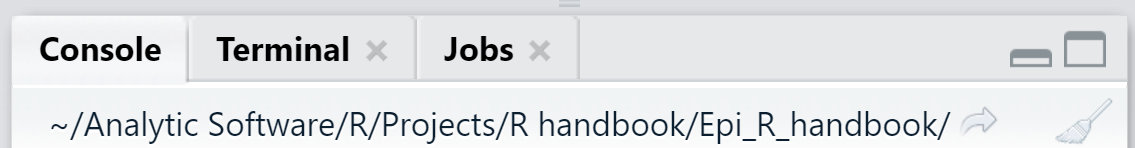
O diretório de trabalho aparece em texto cinza na parte superior do painel do RStudio Console. Você também pode imprimir o diretório de trabalho atual executando getwd() (deixe os parênteses vazios).
Abordagem recomendada
Veja a página sobre projetos R para obter detalhes sobre nossa abordagem recomendada para gerenciar seu diretório de trabalho. Uma maneira comum, eficiente e sem problemas para gerenciar seu diretório de trabalho e caminhos de arquivos é combinar estes 3 elementos em um fluxo de trabalho orientado segundo projeto R
- Um projeto R para armazenar todos os seus arquivos (ver página em projetos R)
- O pacote here para localização de arquivos (ver página em Importação e exportação)
- O pacote rio para arquivos de importação/exportação (ver página em Importação e exportação)
Definido por comando
Até recentemente, muitas pessoas aprendendo R eram ensinadas a começar seus roteiros com um comando setwd(). Em vez disso, considere a utilização de um fluxo de trabalho orientados segundo projeto R e leia as razões para não utilizar setwd(). Em resumo, seu trabalho torna-se específico para seu computador, os caminhos de arquivos utilizados para importar e exportar arquivos tornam-se ” frágeis”, e isso dificulta seriamente a colaboração e a utilização de seu código em qualquer outro computador. Existem alternativas fáceis!
Como observado acima, embora não recomendamos esta abordagem na maioria das circunstâncias, você pode utilizar o comando setwd() com o caminho do arquivo da pasta desejada escrito entre aspas, como no exemplo abaixo:
setwd("C:/Documents/R Files/My analysis")PERIGO: Definir um diretório de trabalho com setwd() pode ser “frágil” se o caminho do arquivo for específico para um computador. Em vez disso, use caminhos de arquivo relativos a um diretório raiz do R Project (com o pacote here).
Definindo manualmente
Para definir o diretório de trabalho manualmente (que seria o equivalente de apontar e clicar de setwd()), clique no menu suspenso Sessão (Session) e vá para “Definir diretório de trabalho” (Set Workig Directory) e depois “Escolher diretório” (Choose Directory). Isso definirá o diretório de trabalho para essa sessão específica do R. Nota: se estiver usando esta abordagem, você terá que fazer isso manualmente toda vez que abrir o RStudio.
Dentro de um projeto R
Se estiver usando um projeto R, o diretório de trabalho será padronizado para a pasta raiz do projeto R que contém o arquivo “.rproj”. Isso se aplicará se você abrir o RStudio clicando em abrir o Projeto R (o arquivo com extensão “.rproj”).
Diretório de trabalho em R markdown
Em um script R markdown, o diretório de trabalho padrão é a pasta na qual o arquivo Rmarkdown (.Rmd) é salvo. Se estiver usando um projeto R e um pacote here, isso não se aplica e o diretório de trabalho será here() conforme explicado na página projetos R.
Se você quiser alterar o diretório de trabalho de um R markdown autônomo (não em um projeto R), se você usar setwd(), isso se aplicará apenas a esse trecho de código específico. Para fazer a alteração para todos os trechos de código em um markdown R, edite o trecho de configuração para adicionar o parâmetro root.dir =, como abaixo:
knitr::opts_knit$set(root.dir = 'desired/directorypath')É muito mais fácil usar apenas o markdown do R dentro de um projeto R e usar o pacote here.
Fornecendo o caminho dos arquivos
Talvez a fonte mais comum de frustração para um iniciante em R (pelo menos em uma máquina Windows) seja digitar um caminho de arquivo para importar ou exportar dados. Há uma explicação completa de como melhor inserir caminhos de arquivo na página Importar e exportar, mas aqui estão alguns pontos-chave:
Caminhos quebrados
Abaixo está um exemplo de um caminho de arquivo “absoluto” ou “endereço completo”. Eles provavelmente quebrarão se forem usados por outro computador. Uma exceção é se você estiver usando uma unidade compartilhada/de rede.
C:/Users/Name/Document/Analytic Software/R/Projects/Analysis2019/data/March2019.csv Direção da barra
Se digitar um caminho de arquivo, observe a direção das barras. Use barras normais (/) para separar os componentes (“data/provincial.csv”). Para usuários do Windows, a maneira padrão de exibição dos caminhos de arquivo é com barras invertidas (ou contra-barra) (\) - portanto, você precisará alterar a direção de cada barra. Se você usar o pacote here conforme descrito na página projetos R, a direção da barra não será um problema.
Caminhos relativos
Geralmente, recomendamos fornecer caminhos de arquivo “relativos” - ou seja, o caminho relativo à raiz do seu projeto R. Você pode fazer isso usando o pacote here conforme explicado na página projetos R. Um caminho de arquivo relativo pode ser assim:
# Import csv linelist from the data/linelist/clean/ sub-folders of an R project
linelist <- import(here("data", "clean", "linelists", "marin_country.csv"))Mesmo usando caminhos de arquivo relativos em um projeto R, você ainda pode usar caminhos absolutos para importar/exportar dados fora do seu projeto R.
3.10 Objetos
Tudo em R é um objeto, e R é uma linguagem “orientada a objetos”. Estas seções explicarão:
- Como criar objetos (
<-) - Tipos de objetos (por exemplo, quadros de dados, vetores..)
- Como acessar subpartes de objetos (por exemplo, variáveis em um conjunto de dados)
- Classes de objetos (por exemplo, numérico, lógico, inteiro, duplo, caractere, fator)
Tudo é um objeto
Esta seção é adaptada do projeto R4Epis.
Tudo o que você armazena no R - conjuntos de dados, variáveis, uma lista de nomes de vilarejos, um número total da população, até saídas como gráficos - são objetos que recebem um nome e podem ser referenciados em comandos posteriores.
Um objeto existe quando você atribui um valor a ele (consulte a seção de atribuição abaixo). Quando lhe é atribuído um valor, o objeto aparece no Ambiente (Environment) (veja o painel superior direito do RStudio). Ele pode então ser operado, manipulado, alterado e redefinido.
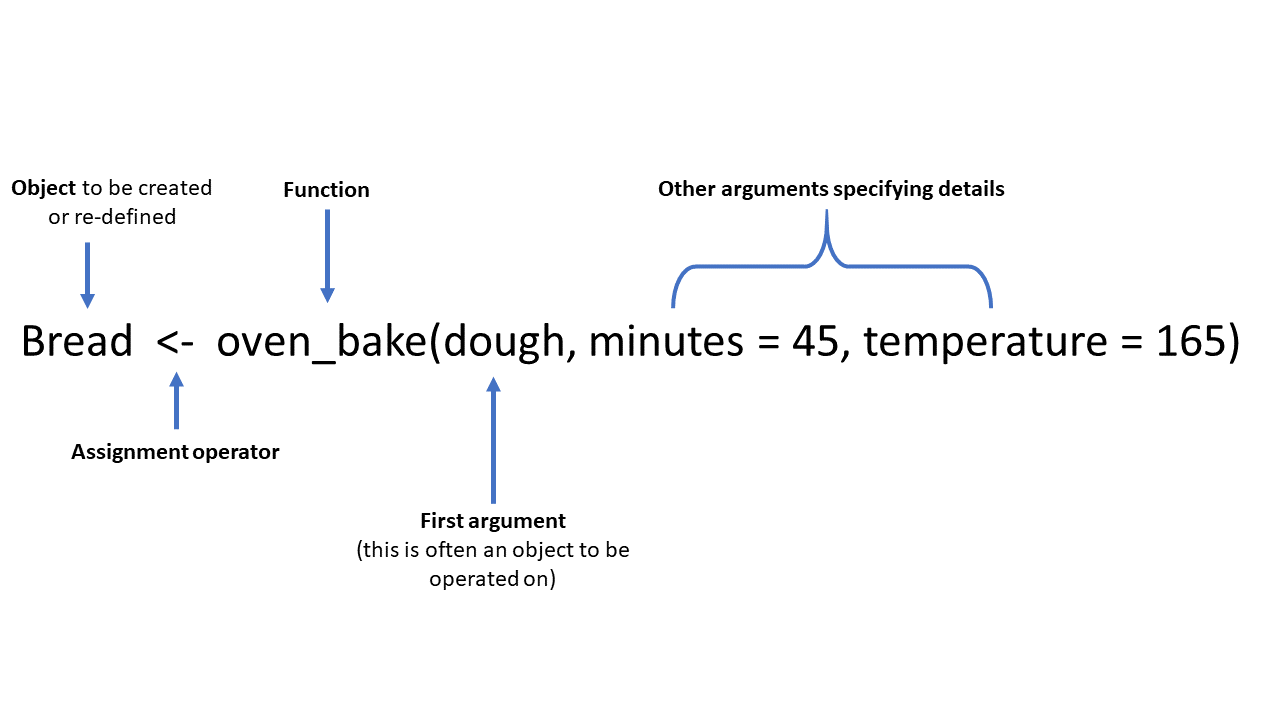
Definindo objetos (<-)
Crie objetos atribuindo-lhes um valor com o operador <-.
Você pode pensar no operador de atribuição <- como as palavras “é definido como”. Os comandos de atribuição geralmente seguem uma ordem padrão:
nome_do_objeto <- valor (ou processo/cálculo que produz um valor)
Por exemplo, você pode querer registrar a semana do relatório epidemiológico atual como um objeto para referência no código posterior. Neste exemplo, o objeto semana_atual é criado quando é atribuído o valor "2018-W10" (as aspas fazem disso um valor de caractere). O objeto semana_atual aparecerá no painel RStudio Environment (canto superior direito) e poderá ser referenciado em comandos posteriores.
Veja os comandos R e sua saída nas caixas abaixo.
semana_atual <- "2018-W10" # esse comando cria o objeto semana_atual ao atribuir a ele um valor
semana_atual # esse comento exibe ("printa") o valor atual do objeto semana_atual no Console [1] "2018-W10"NOTA: Observe que o [1] na saída do console R está simplesmente indicando que você está visualizando o primeiro item da saída
CUIDADO: O valor de um objeto pode ser sobrescrito a qualquer momento executando um comando de atribuição para redefinir seu valor. Assim, a ordem dos comandos executados é muito importante..
O comando a seguir irá redefinir o valor de semana_atual:
semana_atual <- "2018-W51" # atribui um NOVO valor para o objeto semana_atual
semana_atual # Exibe ("printa") o valor atual do objeto semana_atual no console [1] "2018-W51"Sinal de igual =
Você também verá sinais de igual no código R:
- Um sinal de igual duplo
==entre dois objetos ou valores faz uma pergunta lógica: “isso é igual a isso?”.
- Você também verá sinais de igual dentro de funções usadas para especificar valores de argumentos de função (leia sobre isso nas seções abaixo), por exemplo
max(age, na.rm = TRUE).
- Você pode usar um único sinal de igual
=no lugar de<-para criar e definir objetos, mas isso é desencorajado. Você pode ler sobre por que isso é desencorajado aqui.
Bases de dados
Os conjuntos de dados também são objetos (geralmente “dataframes”) e devem receber nomes quando são importados. No código abaixo, o objeto linelist é criado e atribuído o valor de um arquivo CSV importado com o pacote rio e sua função import().
# o objeto linelist é criado e a ele é atribuído o valor do arquivo CSV importado
linelist <- import("my_linelist.csv")Você pode ler mais sobre como importar e exportar conjuntos de dados na seção [Importar e exportar](#importing.
CUIDADO: Uma nota rápida sobre a nomeação de objetos:
- Os nomes dos objetos não devem conter espaços, mas você deve usar sublinhado (_) ou um ponto (.) em vez de um espaço.
- Os nomes dos objetos diferenciam maiúsculas de minúsculas (o que significa que Dataset_A é diferente de dataset_A).
- Os nomes dos objetos devem começar com uma letra (não pode começar com um número como 1, 2 ou 3).
Saídas (Outputs)
Saídas como tabelas e gráficos fornecem um exemplo de como as saídas podem ser salvas como objetos ou apenas exibidas (“printadas” no console) sem serem salvas. Uma tabulação cruzada de gênero e resultado usando a função do R base table() pode ser exibida diretamente no console R (sem ser salva).
# exibe apenas no console R
table(linelist$gender, linelist$outcome)
Death Recover
f 1227 953
m 1228 950Mas a mesma tabela pode ser salva como um objeto nomeado. Então, opcionalmente, pode ser printado.
# salvar
gen_out_table <- table(linelist$gender, linelist$outcome)
# printar
gen_out_table
Death Recover
f 1227 953
m 1228 950Colunas
As colunas em um conjunto de dados também são objetos e podem ser definidas, sobrescritas e criadas conforme descrito abaixo na seção Colunas.
Você pode usar o operador de atribuição do R base para criar uma nova coluna. Abaixo, a nova coluna bmi (IMC - Índice de Massa Corporal, do inglês Body Mass Index) é criada, e para cada linha o novo valor é resultado de uma operação matemática sobre o valor da linha nas colunas wt_kg e ht_cm.
# criar uma nova colna bmi (que é o valor de IMC) usando a sintaxe do R base
linelist$bmi <- linelist$wt_kg / (linelist$ht_cm/100)^2No entanto, neste manual, enfatizamos uma abordagem diferente para definir colunas, que usa a função mutate() do pacote dplyr e piping com o operador pipe (%>%). A sintaxe é mais fácil de ler e há outras vantagens explicadas na página em Limpeza de dados e principais funções. Você pode ler mais sobre tubulação na seção Tubulação abaixo.
# criar uma nova colna bmi (que é o valor de IMC) usando a sintaxe do dplyr
linelist <- linelist %>%
mutate(bmi = wt_kg / (ht_cm/100)^2)Estrutura de um objeto
Os objetos podem ser um único dado (por exemplo, meu_numero <- 24) ou podem consistir em dados estruturados.
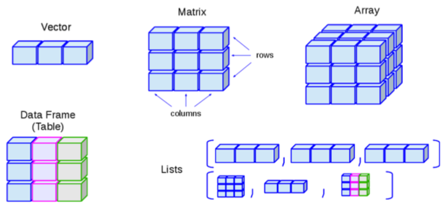
O gráfico abaixo é emprestado de este tutorial R online. Ele mostra algumas estruturas de dados comuns e seus nomes. Não estão incluídos nesta imagem os dados espaciais, que são discutidos na página Noções básicas de GIS.
Em epidemiologia (e particularmente epidemiologia de campo), você encontrará mais comumente dataframes e vetores:
| Estrutura comum | Explicação | Exemplo |
|---|---|---|
| Vetores | Um conteiner para uma sequência de objetos singulares, de uma mesma classe (es: numérico, caractere). | “Variáveis” (colunas) em data frames são vetores (ex: a coluna age_years). |
| Data Frames | Vetores (ex: colunas) que estão unidas e têm todas o mesmo número de linhas. | linelist é data frame. |
|
Observe que para criar um vetor que “independente” (não faz parte de um data frame) a função c() é usada para combinar os diferentes elementos. Por exemplo, se estiver criando um vetor de cores para uma paleta de cores de um gráfico: vector_de_cores <- c("blue", "red2", "orange", "grey")
Classe de objetos
Todos os objetos armazenados em R possuem uma classe que informa ao R como lidar com o objeto. Existem muitas classes possíveis, mas as mais comuns incluem:
| Classe | Explicação | Exemplos |
|---|---|---|
| Character | Caracteres, esses são textos/palavras/frases “dentro de aspas”. Nenhuma operação matemática pode ser realizada com esses objetos. | “Objetos do tipo caractere ficam entre aspas” |
| Integer | Números inteiros (sem decimais) | -5, 14, or 2000 |
| Numeric | Números que podem incluir decimais. Se estiverem dentro de aspas, serão considerados como caractere | 23.1 or 14 |
| Factor | Fatores, são vatores que tem uma ordem específica ou hierarquia de valores | Uma variável de status econômico com valores ordenados |
| Date | Uma vez que o R é informado que um certo grupo de dados corresponde a Data, esses valores só podem ser apresentados e manipulados de algumas maneiras especiais. Veja a página sobre Trabalhando com datas para mais informações | 2018-04-12 ou 15/3/1954 ou Qua 4 Jan 1980 |
| Logical | Valores lógicos que precisam um dos dois valores especiais TRUE (verdadeiro) ou FALSE (note que eles não não “TRUE” e “FALSE” entre aspas) | TRUE ou FALSE |
| data.frame | Uma data frame é como o R armazena uma base de dados típica. Consiste de vetores (colunas) de dados unidos, que tenha o mesmo número de observações (linhas). | O exemplo AJS nomeado linelist_raw contém 68 variáveis com 300 observações (linhas) cada. | |
|
| tibble | tibble é uma variação do data frame, a principal diferença operacional é que eles printam melhor no console (exibem as primeiras 10 linhas e apenas as colunas que cabem na tela) | Qualquer data frame, lista ou matriz pode ser convertido em um tibble com as_tibble() |
| list | Uma lista é como um vetor, mas contém outros objetos que podem ser de outras classes diferentes | Uma lista pode conter um único número, um dataframe, um vetor e até outra lista dentro dele! |
Você pode testar a classe de um objeto fornecendo seu nome para a função class(). Nota: você pode fazer referência a uma coluna específica dentro de um conjunto de dados utilizando a notação $ para separar o nome do conjunto de dados e o nome da coluna.
class(linelist) # a classe deve ser uma data frame ou tibble[1] "data.frame"class(linelist$age) # classe deve ser numérica [1] "numeric"class(linelist$gender) # classe deve ser caractere [1] "character"Às vezes, uma coluna será convertida automaticamente para uma classe diferente pelo R. Cuidado com isso! Por exemplo, se você tiver um vetor ou coluna de números, mas um valor de caractere for inserido… a coluna inteira mudará para caractere de classe.
num_vector <- c(1,2,3,4,5) # define um vetor só de números
class(num_vector) # este vetor é da classe numérico[1] "numeric"num_vector[3] <- "three" # converte o terceiro elemento para um caractere
class(num_vector) # o vetor agora é classe caractere[1] "character"Um exemplo comum disso é quando se manipula um data frame para exibir uma tabela - se você fizer uma linha total e tentar colar/colar porcentagens na mesma célula dos números (por exemplo, 23 (40%)), toda a coluna numérica acima será convertida em caractere e não poderá mais ser utilizada para cálculos matemáticos.** Algumas vezes, será necessário converter objetos ou colunas em outra classe.**
| Função | Ação |
|---|---|
as.character() |
Converte para a classe caractere |
as.numeric() |
Converte para a classe numérica |
as.integer() |
Converte para a classe inteiro |
as.Date() |
Converte para a classe Data - Nota: Veja a seção sobre datas para detalhes |
factor() |
Converte para a classe fator - Nota: Redefinir a ordem dos níveis requer argumentos extras |
Da mesma forma, existem as funções do R base para verificar se um objeto é de uma classe específica, como is.numeric(), is.character(), is.double(), is.factor(), is.integer()
Aqui está mais material on-line sobre classes e estruturas de dados em R.
Colunas/Variáveis ($)
Uma coluna em um data frame é tecnicamente um “vetor” (ver tabela acima) - uma série de valores que devem ser todos da mesma classe (tanto caracter, numérico, lógico, etc.).
Um vetor pode existir independentemente de um data frame, por exemplo, um vetor de nomes de colunas que você deseja incluir como variáveis explicativas em um modelo. Para criar um vetor independente, utilize a função c() como abaixo:
# define o vetor independente de entradas dp tipo caractere
explanatory_vars <- c("gender", "fever", "chills", "cough", "aches", "vomit")
# printa os valores desse vetor nomeado
explanatory_vars[1] "gender" "fever" "chills" "cough" "aches" "vomit" As colunas em uma data frame também são vetores e podem ser chamadas, referenciadas, extraídas ou criadas utilizando o símbolo $. O símbolo $ liga o nome da coluna ao nome de sua moldura de dados. Neste manual, tentamos utilizar a palavra “coluna” em vez de “variável”.
# Obter o comprimento do vetor age
length(linelist$age) # (age é uma coluna no data frame linelist)Ao digitar o nome do data frame seguido de $ você verá também um menu suspenso de todas as colunas data frame. Você pode percorrê-las usando sua tecla de seta, selecionar uma com sua tecla Enter e evitar erros ortográficos!
DICA AVANÇADA: Alguns objetos mais complexos (por exemplo, uma lista, ou um objeto ‘epicontacts’) podem ter múltiplos níveis que podem ser acessados através de múltiplos sinais de dólar. Por exemplo epicontacts$linelist$date_onset
Accessar/indexar com colchetes ([ ])
Talvez seja necessário visualizar partes de objetos, também chamadas de “indexação”, o que muitas vezes é feito utilizando os colchetes [ ]. Utilizar $ em um data frame para acessar uma coluna é também um tipo de indexação.
my_vector <- c("a", "b", "c", "d", "e", "f") # define o vetor
my_vector[5] # printa o quinto elemento[1] "e"Os colchetes também funcionam para retornar partes específicas de uma saída retornada, tais como a saída de uma função summary():
# Todo o resumo
summary(linelist$age) Min. 1st Qu. Median Mean 3rd Qu. Max. NA's
0.00 6.00 13.00 16.07 23.00 84.00 86 # Apenas o segundo elemento,sem nome, usando colchetes simples
summary(linelist$age)[2]1st Qu.
6 # Apenas o segundo elemento,sem nome, usando colchetes duplos
# Extrais um elemento pelo nome, sem mostrar o nome aparecer no console
summary(linelist$age)[["Median"]][1] 13Os colchetes também trabalham em data frames para visualizar linhas e colunas específicas. Você pode fazer isso utilizando a sintaxe dataframe[linhas, colunas]:
# Visualizar uma linha específica (2) de uma base de dados, com todas as colunas (não esqueça a vírgula!)
linelist[2,]
# Vert todas as linhas, mas só uma coluna
linelist[, "date_onset"]
# Ver valores da linha 2, e as colunas 5 a 10
linelist[2, 5:10]
# VVer valores da linha 2, e as colunas 5 a 10 e a 18
linelist[2, c(5:10, 18)]
# Ver valores da linha 2 a 20 , e colunas específicas
linelist[2:20, c("date_onset", "outcome", "age")]
#Ver linhas e colunas baseado em critérios
# *** Noque que o dataframe precira ainda ser nomeado no critério!
linelist[linelist$age > 25 , c("date_onset", "outcome", "age")]
# Use View() para ver as saídas no Painel Visualizador do RStudio Viewer (mais fácil de ler)
# *** Note o "V" maúsculo na função View()
View(linelist[2:20, "date_onset"])
# Salve como um novo objeto
new_table <- linelist[2:20, c("date_onset")] Observe que você também pode alcançar a indexação de linha/coluna acima em dataframes e tibbles utilizando dplyr sintaxe (funções filter() para linhas, e select() para colunas). Leia mais sobre estas funções centrais na página Dados de limpeza e principais funções.
Para filtrar com base no “número da linha”, você pode utilizar a função dplyr row_number() com parênteses abertos como parte de uma instrução de filtragem lógica. Muitas vezes você utilizará o operador %in% e uma faixa de números como parte dessa declaração lógica, como mostrado abaixo. Para ver as primeiras N fileiras , você também pode utilizar a função especial dplyr head().
# Visualizar as primeiras 100 linhas
linelist %>% head(100)
# Show row 5 only
linelist %>% filter(row_number() == 5)
# View rows 2 through 20, and three specific columns (note no quotes necessary on column names)
linelist %>% filter(row_number() %in% 2:20) %>% select(date_onset, outcome, age)Ao indexar um objeto de classe lista, parênteses simples sempre retornam coma classe lista, mesmo que apenas um único objeto seja retornado. Entretanto, colchetes duplos podem ser usados para acessar um único elemento e retornar uma classe diferente da lista.
Os parênteses também podem ser escritos um após o outro, como demonstrado abaixo.
Esta explicação visual da indexação de listas, com pimenteiros é bem-humorada e útil.
# define uma lista demo
my_list <- list(
# Primeiro elemento na lista é um vetor do tipo caractere
hospitals = c("Central", "Empire", "Santa Anna"),
# segundo elemento na lista é um dataframe com endereços
addresses = data.frame(
street = c("145 Medical Way", "1048 Brown Ave", "999 El Camino"),
city = c("Andover", "Hamilton", "El Paso")
)
)Veja como fica a lista quando exibida no console. Veja como há dois elementos nomeados:
hospitals,, um vetor de caracteres.addresses, um quadro de dados de endereços
my_list$hospitals
[1] "Central" "Empire" "Santa Anna"
$addresses
street city
1 145 Medical Way Andover
2 1048 Brown Ave Hamilton
3 999 El Camino El PasoAgora nos extraímos usando vários métodos:
my_list[1] # esse retorna o elemanto da classe "lista" - o nome do elemento ainda é exibido $hospitals
[1] "Central" "Empire" "Santa Anna"my_list[[1]] # este retorna apenas o vetor de caracter (sem nome) [1] "Central" "Empire" "Santa Anna"my_list[["hospitals"]] # você também pode indexar pelo nome do elemento da lista [1] "Central" "Empire" "Santa Anna"my_list[[1]][3] # Isto retorna o terceiro elemento do vetor de caracteres "hospitals" [1] "Santa Anna"my_list[[2]][1] # Isto retorna a primeira coluna ("street") da data frame de endereços street
1 145 Medical Way
2 1048 Brown Ave
3 999 El CaminoRemover objetos
Você pode remover objetos individuais de seu ambiente R colocando o nome na função rm() (sem aspas):
rm(object_name)Você pode reomver todos os objetos (limpar o ambiente de trabalho) ao executar:
rm(list = ls(all = TRUE))3.11 Piping (Encadeamento, %>%)
Duas abordagens gerais para trabalhar com objetos são:
- Pipes/tidyverse - pipes send an object from function to function - emphasis is on the action, not the object
- Definir objetos intermediários - um objeto é redefinido repetidamente - a ênfase está no objeto.
Pipes
Explicado de forma simples, o operador do pipe (%>%) passa uma saída intermediária de uma função para a próxima.
Você pode pensar nisso como dizendo “então”. Muitas funções podem ser ligadas com %>%.
- Piping enfatiza uma seqüência de ações, não o objeto sobre o qual as ações estão sendo realizadas
- Pipes são melhores quando uma seqüência de ações deve ser executada em um único objeto.
- Os pipes vêm do pacote magrittr, que é automaticamente incluído nos pacotes dplyr e tidyverse
- Os pipes podem tornar o código mais limpo e fácil de ler, mais intuitivo
Leia mais sobre esta abordagem no tidyverse guia de estilo
Aqui está um exemplo falso para comparação, usando funções fictícias para “assar um bolo”. Primeiro, o método do pipe:
# Um exemplo falso de como assar um bolo usando a sintaxe do pipe
cake <- flour %>% #para definir o bolo, comece com farinha, e então ...
add(eggs) %>% # adicione ovos
add(oil) %>% # adicione óleo
add(water) %>% # adicione água
mix_together( # misture
utensil = spoon,
minutes = 2) %>%
bake(degrees = 350, # asse
system = "fahrenheit",
minutes = 35) %>%
let_cool() # deixe esfriarAqui está outro link descrevendo a utilidade dos pipes.
O pipe não é uma função do R base. Para utilizar o pipe, o pacote magrittr deve ser instalado e carregado (isto é normalmente feito carregando os pacotes tidyverse ou dplyr que o inclui). Você pode ler mais sobre pipe na documentação magrittr.
Observe que, assim como outros comandos R, os pipes podem ser utilizados apenas para exibir o resultado, ou para salvar/reservar um objeto, dependendo se o operador de atribuição <- está envolvido. Veja os dois abaixo:
# Criar ou sobrescrever um objeto, definindo como contagens agregadas por faixa-etária (não "printado")
linelist_summary <- linelist %>%
count(age_cat)# Printa a tabela de contagens no console, mas nao a salva
linelist %>%
count(age_cat) age_cat n
1 0-4 1095
2 5-9 1095
3 10-14 941
4 15-19 743
5 20-29 1073
6 30-49 754
7 50-69 95
8 70+ 6
9 <NA> 86%<>%
Este é um “tubo de atribuição” do pacote magrittr, que encadeia um objeto para frente e também re-define o objeto. Deve ser o primeiro operador pipe da cadeia. É um “atalho”. Os dois comandos abaixo são equivalentes:
linelist <- linelist %>%
filter(age > 50)
linelist %<>% filter(age > 50)Definir objetos intermediários
Esta abordagem para mudar objetos/dataframes pode ser melhor se:
- Você precisa manipular vários objetos…
- Há etapas intermediárias que são significativas e merecem nomes de objetos separados
Riscos:
- Criar novos objetos para cada etapa significa criar muitos objetos. Se você usar o errado, talvez não se dê conta disso!
- Nomear todos os objetos pode ser confuso…
- Os erros podem não ser facilmente detectáveis
Ou nomear cada objeto intermediário, ou sobrescrever o original, ou combinar todas as funções em conjunto. Todos vêm com seus próprios riscos.
Abaixo está o mesmo exemplo falso de “bolo” como acima, mas usando este estilo:
# um exemplo falso de como assar um bolo usado este método (definindo objetos intermediários)
batter_1 <- left_join(flour, eggs)
batter_2 <- left_join(batter_1, oil)
batter_3 <- left_join(batter_2, water)
batter_4 <- mix_together(object = batter_3, utensil = spoon, minutes = 2)
cake <- bake(batter_4, degrees = 350, system = "fahrenheit", minutes = 35)
cake <- let_cool(cake)Combine todas as funções em uma só - isso dificulta a leitura):
# um exemplo de combinação/aninhamento de múltiplas funções juntas - difícil de ler
cake <- let_cool(bake(mix_together(batter_3, utensil = spoon, minutes = 2), degrees = 350, system = "fahrenheit", minutes = 35))3.12 Principais operadores e funções
Esta seção detalha os operadores em R, como por exemplo:
- Operadores de definição
- Operadores relacionais (menos do que, igual a…)
- Operadores lógicos (e, ou…)
- Lidando com valores faltantes…
- Operadores matemáticos e funções (+/-, >, sum(), median(), …)
- O operador
%in%
Operador de atribuição
<-
O operador de atribuição básica em R é <-. De tal forma que nome_objeto <- valor.
Este operador de atribuição também pode ser escrito como =. Aconselhamos o uso de <- para uso geral em R.
Aconselhamos também o uso de <- para uso geral em R. Aconselhamos também o uso de espaços em torno de tais operadores, para facilitar a leitura.
<<-
Se você estiver Escrevendo funções, ou utilizando R de forma interativa com scripts de origem, então você pode precisar utilizar este operador de atribuição <<- (do R base). Este operador é utilizado para definir um objeto em um ambiente R ‘pai’ superior. Veja isto referência on-line.
%<>%
Este é um “pipe de atribuição” do pacote magrittr, que canaliza um objeto para frente e também re-define o objeto. Deve ser o primeiro operador pipe da cadeia. É a abreviação, como mostrado abaixo em dois exemplos equivalentes:
linelist <- linelist %>%
mutate(age_months = age_years * 12)O código acima é equivalente ao abaixo:
linelist %<>% mutate(age_months = age_years * 12)%<+%
Isso é usado para adicionar dados a árvores filogenéticas com o pacote ggtree. Veja a página em Árvores Filogenéticas ou este livro de recursos online.
Operadores relacionais e lógicos
Os operadores relacionais comparam valores e são freqüentemente utilizados na definição de novas variáveis e subconjuntos de conjuntos de dados. Aqui estão os operadores relacionais comuns em R:
| Significado | Operador | Examplo | Resultado do exemplo |
|---|---|---|---|
| Igual a | == |
"A" == "a" |
FALSE (porque o R é sensível a letras maiúscula/minúsculas) Note que == (iguais duplos) é diferente de = (símbolo de igual normal), que age como o operador de atribuição <- |
| Não igual (diferente) | != |
2 != 0 |
TRUE |
| Maior que | > |
4 > 2 |
TRUE |
| Menor que | < |
4 < 2 |
FALSE |
| Maior ou igual a | >= |
6 >= 4 |
TRUE |
| Menor ou igual a | <= |
6 <= 4 |
FALSE |
| Valor está faltante | is.na() |
is.na(7) |
FALSE (veja página em Dados faltantes) |
| Valor não faltante | !is.na() |
!is.na(7) |
TRUE |
Operadores lógicos, como AND e OR, geralmente são usados para conectar operadores relacionais e criar critérios mais complicados. Instruções complexas podem exigir parênteses ( ) para agrupamento e ordem de aplicação.
| Significado | Operador |
|---|---|
| AND | & |
| OR | | (barra vertical) |
| Parentheses | ( ) Usado para agrupar critérios e clarificar a ordem das operações |
Por exemplo, abaixo, temos uma linelist com duas variáveis que queremos utilizar para criar nossa definição de caso, hep_e_rdt, um resultado de teste e other_cases_in_hh, que nos dirá se há outros casos na casa. O comando abaixo utiliza a função case_when() para criar a nova variável case_def de tal forma que:
linelist_cleaned <- linelist %>%
mutate(case_def = case_when(
is.na(rdt_result) & is.na(other_case_in_home) ~ NA_character_,
rdt_result == "Positive" ~ "Confirmed",
rdt_result != "Positive" & other_cases_in_home == "Yes" ~ "Probable",
TRUE ~ "Suspected"
))| Critérios no exemplo acima | Valor da nova variável “case_def” |
|---|---|
Se o valor das variáveis rdt_result e other_cases_in_home estiverem faltando |
NA (faltante) |
Se o valor em rdt_result for “Positive” |
Confirmed |
If the value in rdt_result is NOT “Positive” AND the value in other_cases_in_home is “Yes” |
Probable |
| If one of the above criteria are not met | Suspected |
Note that R is case-sensitive, so “Positive” is different than “positive”…
Valores faltantes
Em R, os valores ausentes são representados pelo valor especial NA (um valor “reservado”) (letras maiúsculas N e A - não entre aspas). Se você importar dados que registram dados ausentes de outra maneira (por exemplo, 99, “Missing” ou .), convém recodificar esses valores para NA. Como fazer isso é abordado na página Importação e exportação.
Para testar se um valor é NA, use a função especial is.na(), que retorna TRUE ou FALSE.
rdt_result <- c("Positive", "Suspected", "Positive", NA) # dois casos positivos, um suspeito e um desconhecido
is.na(rdt_result) # Testa se o valor de rdt_result é NA[1] FALSE FALSE FALSE TRUELeia mais sobre valores ausentes, infinitos, NULL e impossíveis na página em Campos em branco/faltantes. Saiba como converter valores ausentes ao importar dados na página em Importação e exportação.
Matemática e estatística
Todos os operadores e funções nesta página estão disponíveis automaticamente usando o pacote R base.
Operadores mmatemáticos
Estes são frequentemente usados para realizar adição, divisão, para criar novas colunas, etc. Abaixo estão os operadores matemáticos comuns em R. Colocar ou não os espaços ao redor dos operadores não é importante.
| Propósito | Exemplo no R |
|---|---|
| adição | 2 + 3 |
| subtração | 2 - 3 |
| multiplicação | 2 \* 3 |
| divisão | 30 / 5 |
| potência | 2\^3 |
| ordem das operações | ( ) |
Funções matemáticas
| Propósito | Função |
|---|---|
| arredondamento | round(x, digits = n) |
| arredondamento | janitor::round_half_up(x, digits = n) |
| teto (arredondar para cima) | ceiling(x) |
| chão (arredondar para baixo) | floor(x) |
| valor obsoluto (módulo) | abs(x) |
| raiz quadrada | sqrt(x) |
| exponencial | exponent(x) |
| logaritmo natural/neperiano | log(x) |
| logaritmo na base 10 | log10(x) |
| logaritmo na base 2 | log2(x) |
Nota: para round() o digits = especifica o número de casas decimais colocadas. Use signif() para arredondar para um número de algarismos significativos.
Notação científica
A probabilidade de notação científica ser usada depende do valor da opção scipen.
Da documentação de ?options: scipen é uma penalidade a ser aplicada ao decidir imprimir valores numéricos em notação fixa ou exponencial. Os valores positivos tendem para a notação fixa e os negativos para a notação científica: a notação fixa será preferida, a menos que seja mais do que os dígitos ‘scipen’ mais largos.
Se estiver definido para um número baixo (por exemplo, 0), ele estará sempre “ligado”. Para “desativar” a notação científica em sua sessão R, defina-a como um número muito alto, por exemplo:
# desligar a notação científica
options(scipen=999)Arredondando
PERIGO: A função round() usa “arredondamento do banqueiro” que arredonda de .5 somente se o número superior for par. Use round_half_up() do janitor para arredondar consistentemente metades para o número inteiro mais próximo. Veja esta explicação
# use a função de arredondamento adequanda para o seu trabalho
round(c(2.5, 3.5))[1] 2 4janitor::round_half_up(c(2.5, 3.5))[1] 3 4Funções estatísticas
CUIDADO: As funções abaixo incluirão, por padrão, valores ausentes nos cálculos. Valores ausentes resultarão em uma saída de NA, a menos que o argumento na.rm = TRUE seja especificado. Isso pode ser escrito de forma abreviada como na.rm = T.
| Objetivo | Função |
|---|---|
| média aritmética | mean(x, na.rm=T) |
| mediana | median(x, na.rm=T) |
| desvio padrão | sd(x, na.rm=T) |
| quantis* | quantile(x, probs) |
| soma | sum(x, na.rm=T) |
| valor mínimo | min(x, na.rm=T) |
| valor máximo | max(x, na.rm=T) |
| range de valores numéricos | range(x, na.rm=T) |
| resumo** | summary(x) |
Notas:
- *
quantile():xé o vetor numérico a ser examinado eprobs =é um vetor numérico com probabilidades entre 0 e 1,0, por exemplo,c(0.5 , 0.8 , 0,.85) - **
summary(): fornece um resumo em um vetor numérico incluindo média, mediana e percentis comuns
PERIGO: Ao fornecer um vetor de números para uma das funções acima, certifique-se de concatenar os números dentro de c() .
# Se fornecer números brutos para uma função, concatene-os antes com c()
mean(1, 6, 12, 10, 5, 0) # !!! INCORRETO !!! [1] 1mean(c(1, 6, 12, 10, 5, 0)) # CORRETO[1] 5.666667Outras funções úteis
| Objetivo | Função | Examplo |
|---|---|---|
| criar uma sequência de números | seq(from, to, by) | seq(1, 10, 2) |
| repetir x, n vezes | rep(x, ntimes) | rep(1:3, 2) or rep(c("a", "b", "c"), 3) |
| subdividir um vetor numérico | cut(x, n) | cut(linelist$age, 5) |
| pegar uma amostra aleatória | sample(x, size) | sample(linelist$id, size = 5, replace = TRUE) |
%in%
Um operador muito útil para combinar valores e avaliar rapidamente se um valor está dentro de um vetor ou dataframe.
meu_vetor <- c("a", "b", "c", "d")"a" %in% meu_vetor[1] TRUE"h" %in% meu_vetor[1] FALSEPara perguntar se um valor não está em (%in%) é um vetor, coloque um ponto de exclamação (!) na frente da instrução lógica:
# para negar, coloque a exclamação na frente
!"a" %in% meu_vetor[1] FALSE!"h" %in% meu_vetor[1] TRUE%in% é muito útil ao usar a função dplyr case_when(). Você pode definir um vetor anteriormente e depois referenciá-lo. Por exemplo:
affirmative <- c("1", "Yes", "YES", "yes", "y", "Y", "oui", "Oui", "Si")
linelist <- linelist %>%
mutate(child_hospitaled = case_when(
hospitalized %in% affirmative & age < 18 ~ "Hospitalized Child",
TRUE ~ "Not"))Note: If you want to detect a partial string, perhaps using str_detect() from stringr, it will not accept a character vector like c("1", "Yes", "yes", "y"). Instead, it must be given a regular expression - one condensed string with OR bars, such as “1|Yes|yes|y”. For example, str_detect(hospitalized, "1|Yes|yes|y"). See the page on Caracteres e strings for more information.
You can convert a character vector to a named regular expression with this command:
affirmative <- c("1", "Yes", "YES", "yes", "y", "Y", "oui", "Oui", "Si")
affirmative[1] "1" "Yes" "YES" "yes" "y" "Y" "oui" "Oui" "Si" # condensar
affirmative_str_search <- paste0(affirmative, collapse = "|") # opção com R base
affirmative_str_search <- str_c(affirmative, collapse = "|") # opção co pacote stringr
affirmative_str_search[1] "1|Yes|YES|yes|y|Y|oui|Oui|Si"3.13 Erros & avisos
Esta seção explica:
- A diferença entre erros e avisos
- Dicas gerais de sintaxe para escrever código R
- Ajudas para o código.
Erros e avisos comuns e dicas de solução de problemas podem ser encontrados na página em Erros e ajuda.
Erros versus Avisos
Quando um comando é executado, o Console R pode mostrar mensagens de aviso ou erro em texto vermelho.
Um aviso significa que o R concluiu seu comando, mas teve que executar etapas adicionais ou produziu uma saída incomum da qual você deve estar ciente.
Um erro significa que o R não conseguiu completar seu comando.
Procurar pistas:
A mensagem de erro/aviso geralmente inclui um número de linha para o problema.
Se um objeto “é desconhecido” ou “não encontrado”, talvez você o tenha escrito incorretamente, esquecido de chamar um pacote com library() ou esquecido de executar novamente seu script após fazer alterações.
Se tudo mais falhar, copie a mensagem de erro no Google junto com alguns termos-chave: é provável que alguém já tenha trabalhado com isso!
Dicas gerais de sintaxe
Algumas coisas para lembrar ao escrever comandos em R, para evitar erros e avisos:
- Sempre feche os parênteses - dica: conte o número de abertura “(” e fechamento de parênteses “)” para cada pedaço de código
- Evite espaços em nomes de colunas e objetos. Use sublinhado ( _ ) ou pontos ( . )
- Acompanhe e lembre-se de separar os argumentos de uma função com vírgulas
- R diferencia maiúsculas de minúsculas, o que significa que
Variable_Aé diferente devariable_A
Ajudas de código
Qualquer script (RMarkdown ou outro) dará pistas quando você cometer um erro. Por exemplo, se você esqueceu de escrever uma vírgula onde for necessário, ou de fechar um parêntese, o RStudio irá levantar uma bandeira nessa linha, no lado direito do script, para avisá-lo.