35 Diagramas e gráficos
Esta página abrange o código para produzir:
Diagramas de fluxo usando DiagrammeR e a linguagem DOT
Diagramas aluviais/Sankey
Cronogramas de eventos
## Preparação
Carregar pacotes
Este chunk mostra o carregamento dos pacotes necessárias para as análises. Neste manual damos ênfase à função p_load() do pacote pacman, que instala o pacote se necessário e carrega-o para utilização. Outra opção é carregar os pacotes instalados utilizando library() a partir de R base. Ver a página em Introdução ao R para mais informações sobre pacotes R.
pacman::p_load(
DiagrammeR, # para diagrama de fluxo
networkD3, # para diagrama aluvial/Sankey
tidyverse) # exploração (gestão) e visualização de dadosImportar dados
A maior parte do conteúdo desta página não requer um conjunto de dados. No entanto, na seção do diagrama de Sankey, usaremos a lista de casos de uma simulação de epidemia de Ebola. Se você deseja acompanhar esta parte, clique no link <ahref=‘https://github.com/appliedepi/epirhandbook_eng/raw/master/data/case_linelists/linelist_cleaned.rdsclass='download-button’> para baixar (como arquivo.rds). Importe os dados com import() função do pacote rio (ele lida com muitos tipos de arquivo como .xlsx, .csv, .rds - veja a página Importar e exportar para detalhes
# importar a lista de casos
linelist <- import("linelist_cleaned.rds")Para ver as primeiras 50 linhas do banco utilize o codigo a seguir
35.1 Diagramas de fluxos
Pode-se usar o pacote R DiagrammeR para criar gráficos ou gráficos de fluxo. Esses podem ser estáticos, ou podem ser ajustados de forma dinâmica, a partir das mudanças ou alterações do conjunto de dados utilizado.
Ferramentas
A função grViz() é utilizada para criar um diagrama “Graphviz”. Esta função aceita uma entrada de cadeia de caracteres contendo instruções para produção do diagrama. Dentro dessa cadeia, as instruções são escritas numa linguagem diferente, chamada DOT - mas muito fácil de aprender a estrutura básica necessária.
Estrutura básica
- Abra as instruções
grViz(" - Especifique a direcionalidade e o nome do gráfico, e abra colchetes, por exemplo
digraph my_flow_chart { - Declaração do gráfico (layout, direção de classificação)
- Declarações de nós (criar os nós)
- Declarações Bordas (fornece links entre os nós)
- Feche as instruções
}")
Exemplos simples
A seguir será usado dois exemplos simples
Um exemplo mínimo:
# plotar o mínimo
DiagrammeR::grViz("digraph {
graph[layout = dot, rankdir = LR]
a
b
c
a -> b -> c
}")Um exemplo mais aplicado ao contexto de saúde pública:
grViz(" # Todas as instruções estão dentro de uma grande cadeia de caracteres
digraph surveillance_diagram { # 'digraph' significa 'grafico direcional', depois o nome do gráfico
# declaração gráfica
#################
graph [layout = dot,
rankdir = TB,
overlap = true,
fontsize = 10]
# nos(nodea)
#######
node [shape = circle, # forma (shape) = circulo
fixedsize = true
width = 1.3] # Largura dos círculos
Primary # nome dos nós
Secondary
Tertiary
# edges
#######
Primary -> Secondary [label = ' case transfer']
Secondary -> Tertiary [label = ' case transfer']
}
")Sintaxe
Sintaxe básica
Os nomes de nós, ou declarações de borda, podem ser separados por espaços, ponto e vírgula ou novas linhas.
Direção da classificação
Um gráfico pode ser reorientado para se mover da esquerda para a direita, ajustando o rankdir argumento dentro da instrução do gráfico. O padrão é TB (da cigla em inglês top-to-bottom - de cima para baixo), mas pode ser LR (da cigla em inglês left-to-right - da esquerda para a direita), RL (da cigla em inglês right-to-left - da direita para esquerda) ou BT (da cigla em inglês bottom-to-top - baixo para cima).
Nomes dos nós
Os nomes dos nós podem ser palavras únicas, como no exemplo simples acima. Para usar nomes com várias palavras ou caracteres especiais (por exemplo, parênteses, travessões), coloque o nome do nó entre aspas simples (’ ’). Pode ser mais fácil ter um nome de nó curto e atribuir um rótulo label, como mostrado abaixo entre colchetes [ ]. Se você quiser ter uma nova linha no nome do nó, deve fazê-lo por meio de um rótulo - use \n rótulo do nó entre aspas simples, conforme mostrado abaixo.
Subgrupos
Dentro das declarações de borda, podem ser criados subgrupos em ambos os lados da borda com chaves ({ }). A borda aplica-se então a todos os nós do parêntese - é uma abreviação.
Layouts
- dot (set
rankdirto either TB, LR, RL, BT, ) - neato
- twopi
- circo
Nós - atributos editáveis
label(Rótulo - texto, entre aspas simples, se houver várias palavras)fillcolor(Cor do preenchimento - muitas cores possíveis)fontcolor(Cor da fonte - muitas cores possíveis)alpha(Alfa - transparência 0-1)shape(Formato - ellipse, oval, diamond, egg, plaintext, point, square, triangle)style(Estilo)sides(Tamanhos)peripheries(Margens)fixedsize(Tamanho fixo - h x w)height(Altura)width(Largura)distortion(Distorção)penwidth(Largura/espessura da linha - largura da borda da forma)x(deslocamento para a esquerda e direita - left/righty(ddeslocamento para cima e para baixo - up/downfontname(Nome da fonte)fontsize(Tamanho da fonte)icon(Ícone)
Bordas - atributos editáveis
arrowsize(Tamanho da seta)arrowhead(Ponta da seta - normal, box, crow, curve, diamond, dot, inv, none, tee, vee)arrowtail(Base da seta)dir(Direção)style(Estilo “dashed”, para tracejadas …)color(Cor)alpha(Alfa)headport(texto na ponta da seta)tailport(texto na da base da seta)fontname(Nome da fonte)fontsize(Tamanho da fonte)fontcolor(Cor da fonte)penwidth(Largura da linha)minlen(Comprimento minímo)
Nomes das cores: usa-se valores hexadecimais ou os nomes de cores ‘X11’, consulte detalhes X11
Exemplos complexos
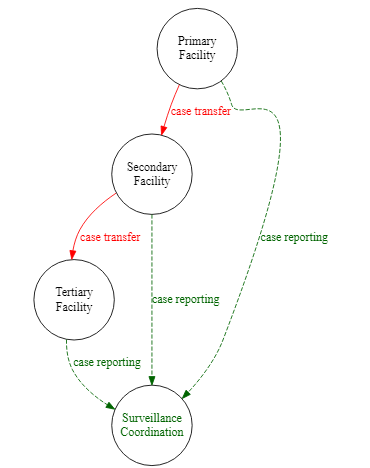
O exemplo abaixo, expande o diagrama de vigilância, adicionando nomes de nós complexos, de bordas agrupadas, com cores e estilo
DiagrammeR::grViz(" # Todas as instruções estão dentro de uma grande cadeia de caracteres
digraph surveillance_diagram { # 'digraph' significa 'gráfico direcional', depois vem o nome do gráfico
# Declaração gráfica
#################
graph [layout = dot,
rankdir = TB, # layout top-to-bottom (de cima para baixo)
fontsize = 10]
# nós (em círculos)
#################
node [shape = circle, # shape = circulos
fixedsize = true
width = 1.3]
Primary [label = 'Primary\nFacility']
Secondary [label = 'Secondary\nFacility']
Tertiary [label = 'Tertiary\nFacility']
SC [label = 'Surveillance\nCoordination',
fontcolor = darkgreen]
# Bordas/margens
#######
Primary -> Secondary [label = ' case transfer',
fontcolor = red,
color = red]
Secondary -> Tertiary [label = ' case transfer',
fontcolor = red,
color = red]
# Bordas agrugapas
{Primary Secondary Tertiary} -> SC [label = 'case reporting',
fontcolor = darkgreen,
color = darkgreen,
style = dashed]
}
")Clusters de sub-graficos
Para agrupar nós em caixas de clusters, coloque-os dentro do mesmo subgráfico nomeado (subgraph name {}). Para ter cada subgráfico identificado dentro de uma caixa delimitadora, comece o nome do subgráfico com “cluster”, como exemplificado com as 4 caixas abaixo
DiagrammeR::grViz(" # Todas as instruções estão dentro de uma grande cadeia de caracteres
digraph surveillance_diagram { # 'digraph' significa 'gráfico direcional', depois vem o nome do gráfico
# Demonstração gráfica
#################
graph [layout = dot,
rankdir = TB,
overlap = true,
fontsize = 10]
# nós (círculos)
#################
node [shape = circle, # shape = círculos
fixedsize = true
width = 1.3] # largura dos círculos
subgraph cluster_passive {
Primary [label = 'Primary\nFacility']
Secondary [label = 'Secondary\nFacility']
Tertiary [label = 'Tertiary\nFacility']
SC [label = 'Surveillance\nCoordination',
fontcolor = darkgreen]
}
# nós (boxes)
###############
node [shape = box, # formas dos nós (Box - Caixa)
fontname = Helvetica] # fonte do texto no nó
subgraph cluster_active {
Active [label = 'Active\nSurveillance']
HCF_active [label = 'HCF\nActive Search']
}
subgraph cluster_EBD {
EBS [label = 'Event-Based\nSurveillance (EBS)']
'Social Media'
Radio
}
subgraph cluster_CBS {
CBS [label = 'Community-Based\nSurveillance (CBS)']
RECOs
}
# Bordas
#######
{Primary Secondary Tertiary} -> SC [label = 'case reporting']
Primary -> Secondary [label = 'case transfer',
fontcolor = red]
Secondary -> Tertiary [label = 'case transfer',
fontcolor = red]
HCF_active -> Active
{'Social Media' Radio} -> EBS
RECOs -> CBS
}
")
Formas dos nós
O exemplo abaixo, emprestado deste tutoriall, mostra exemplos de formas de nós (nodes) aplicadas e uma abreviatura para conexões seriais de borda
DiagrammeR::grViz("digraph {
graph [layout = dot, rankdir = LR]
# definir os estilos globais dos nós. Podemos anulá-los em caixa, se essa for a opção escolhida.
node [shape = rectangle, style = filled, fillcolor = Linen]
data1 [label = 'Dataset 1', shape = folder, fillcolor = Beige]
data2 [label = 'Dataset 2', shape = folder, fillcolor = Beige]
process [label = 'Process \n Data']
statistical [label = 'Statistical \n Analysis']
results [label= 'Results']
# Definições de bordas com as identificações dos nós
{data1 data2} -> process -> statistical -> results
}")Saídas
Como lidar e salvar resultados
- As saídas aparecerão no painel Viewer do RStudio, por padrão no canto inferior direito ao lado de Files, Plots, Packages e Help.
- Para exportar, você pode “Salvar como imagem” ou “Copiar para a área de transferência” do Visualizador. O gráfico se ajustará ao tamanho especificado.
Figuras parametrizadas
Aqui temos uma citação do tutorial: https://mikeyharper.uk/flowcharts-in-r-using-diagrammer/
“Figuras parametrizadas: um grande benefício de projetar figuras dentro de R é que somos capazes de conectar as figuras diretamente com nossa análise, lendo os valores de R diretamente em nossos fluxogramas. Por exemplo, suponha que você tenha criado um processo de filtragem que remove valores após cada estágio de um processo, você pode ter uma figura mostrando o número de valores restantes no conjunto de dados após cada estágio de seu processo. Para fazer isso, você pode usar o símbolo @@X diretamente na figura e, em seguida, referir-se a ele no rodapé do gráfico usando [X]:, onde X é um índice numérico único.”
Recomenda-se rever este este tutorial se a parametrização é algo que você esteja interessado
35.2 Diagramas Aluviais/Sankey
Carregar pacotes
Esta parte de código mostra o carregamento de pacotes necessários para as análises. Neste manual enfatiza-se o p_load() do pacman, que instala o pacote se necessário e carrega-o para uso. Você também pode carregar pacotes instalados usando a biblioteca library() a partir da base R. Consulte a página sobre o [Introdução ao] (#basics) para obter mais informações sobre os pacotes R.
Carregou-se o pacote networkD3 para a construção do diagrama, e o pacote tidyverse para as etapas de preparação dos dados.
pacman::p_load(
networkD3,
tidyverse)Mapeamento do conjunto de dados
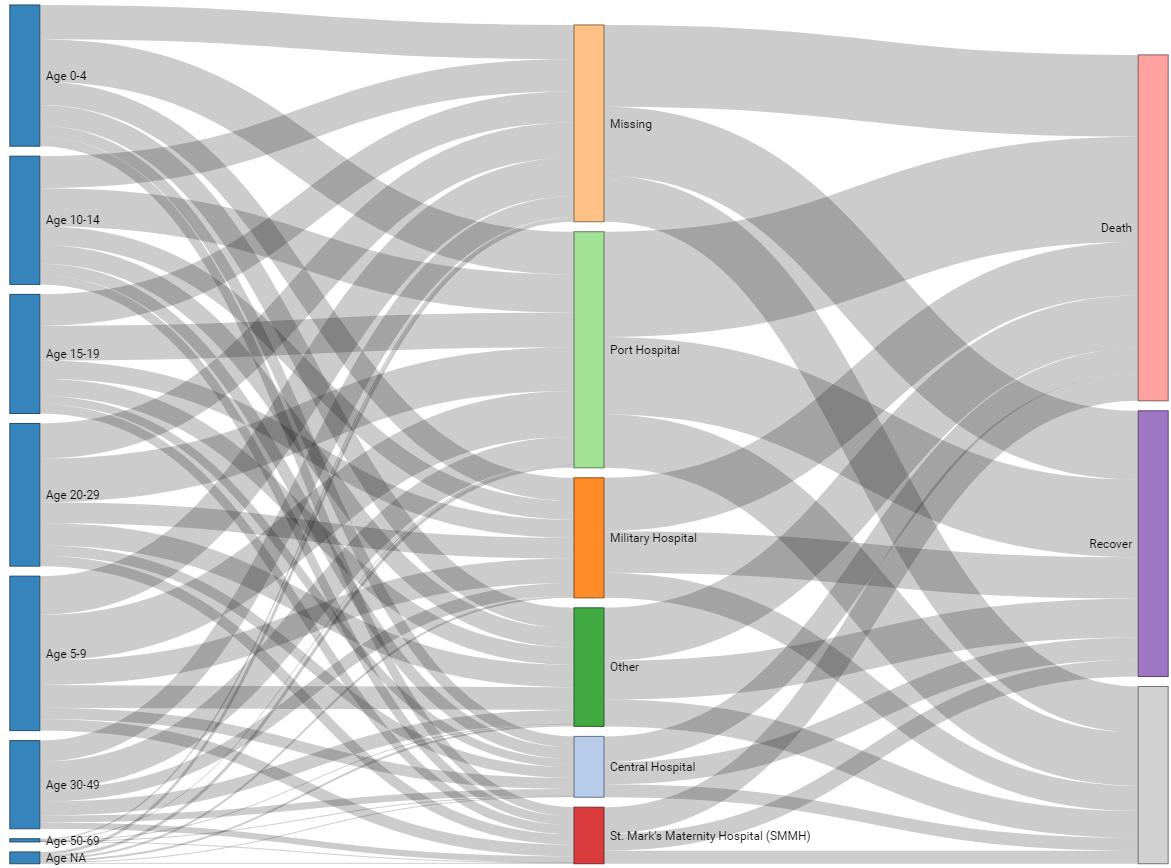
Mapeamento das ligações de um conjunto de dados. Abaixo é apresentado a utilização deste pacote sobre o caso linelist. O tutorial está disponível online para consulta. Tutorial.
Inicia-se obtendo a contagem de casos para cada categoria etária e a combinação por hospital. Os valores sem categoria de idade são removidos para maior clareza. Também foi realizado uma nova rotulagem (renomeadas), o hospital como fonte (source) e categorias de idades age_cat como alvo (target). Estes serão os dois lados do diagrama aluvial.
# Contar por hospital e categoria de idade
links <- linelist %>%
drop_na(age_cat) %>%
select(hospital, age_cat) %>%
count(hospital, age_cat) %>%
rename(source = hospital,
target = age_cat)O conjunto de dados tem agora este aspecto:
Agora cria-se um Data Frame, de todos os nós do diagrama, sob a coluna name. Isso consiste em todos os valores para hospital e age_cat. Observe que garantiu-se que todos eles pertecem a classe Character antes de combiná-los. E ajustou-se as colunas de ID para que fossem números em vez de rótulos:
# O nome único para os nós
nodes <- data.frame(
name=c(as.character(links$source), as.character(links$target)) %>%
unique()
)
nodes # Exibir name
1 Ausente
2 Central Hospital
3 Military Hospital
4 Other
5 Port Hospital
6 St. Mark's Maternity Hospital (SMMH)
7 0-4
8 5-9
9 10-14
10 15-19
11 20-29
12 30-49
13 50-69
14 70+Na sequência editou-se o Data Frame links, criado anteriormente com o count(). Duas novas colunas númericas então foram adicionadas IDsource e IDtarget que irão refletir/criar os linques entre os nós. Essas colunas manterão os números das linhas (posição) dos nós tanto de origem como o do destino. O 1 é subtraído, para que estes números de posição comecem em 0 (não em 1).
# corresponder a números e não a nomes
links$IDsource <- match(links$source, nodes$name)-1
links$IDtarget <- match(links$target, nodes$name)-1O conjunto de dados Links agora tem este aspecto:
Agora, hora de traçar o diagrama Sankey com sankeyNetwork(). Você pode ler mais sobre cada argumento correndo ?sankeyNetwork no console. Note que, a menos que defina iterations = 0 a ordem dos seus nós pode não ser a esperada.
# traçar o diagrama
######
p <- sankeyNetwork(
Links = links,
Nodes = nodes,
Source = "IDsource",
Target = "IDtarget",
Value = "n",
NodeID = "name",
units = "TWh",
fontSize = 12,
nodeWidth = 30,
iterations = 0) # assegurar que a ordem dos nós é como nos dados
pAqui está um exemplo em que o resultado do paciente também é incluído. Na etapa de preparação dos dados, é necessário calcular as contagens de casos entre a idade e o hospital, e separadamente entre o hospital e o resultado, e depois ligar todas estas contagens juntamente com bind_rows().
# conta por hospital e categoria de idade
age_hosp_links <- linelist %>%
drop_na(age_cat) %>%
select(hospital, age_cat) %>%
count(hospital, age_cat) %>%
rename(source = age_cat, # renomear
target = hospital)
hosp_out_links <- linelist %>%
drop_na(age_cat) %>%
select(hospital, outcome) %>%
count(hospital, outcome) %>%
rename(source = hospital, # renomear
target = outcome)
# combinar links
links <- bind_rows(age_hosp_links, hosp_out_links)
# Nome único para os nós
nodes <- data.frame(
name=c(as.character(links$source), as.character(links$target)) %>%
unique()
)
# Criar números de identificação
links$IDsource <- match(links$source, nodes$name)-1
links$IDtarget <- match(links$target, nodes$name)-1
# exibir
######
p <- sankeyNetwork(Links = links,
Nodes = nodes,
Source = "IDsource",
Target = "IDtarget",
Value = "n",
NodeID = "name",
units = "TWh",
fontSize = 12,
nodeWidth = 30,
iterations = 0)
p35.3 Cronograma de eventos
Para fazer uma linha do tempo mostrando eventos específicos, você pode usar o pacote vistime.
Veja vignette
# Carregando o pacote
pacman::p_load(vistime, # criando a linha do tempo
plotly # para visualização interativa
)Aqui está um conjunto de dados de eventos para começar
p <- vistime(data) # aplicar o vistime
library(plotly)
# passo 1: transformar em lista
pp <- plotly_build(p)
# passo 2: Tamanho do marcador
for(i in 1:length(pp$x$data)){
if(pp$x$data[[i]]$mode == "markers") pp$x$data[[i]]$marker$size <- 10
}
# passo 3: tamanho do texto
for(i in 1:length(pp$x$data)){
if(pp$x$data[[i]]$mode == "text") pp$x$data[[i]]$textfont$size <- 10
}
# passo 4: posição do texto
for(i in 1:length(pp$x$data)){
if(pp$x$data[[i]]$mode == "text") pp$x$data[[i]]$textposition <- "right"
}
#exibir
pp35.4 Gráficos cíclicos direcionados - DAGs
Você pode construir um DAG manualmente usando o pacote DiagammeR e a linguagem DOT conforme descrito acima.
Uma outra alternativa é usar os pacotes como ggdag e daggity
35.5 Recursos
Muito do que foi dito acima em relação à linguagem DOT é adaptado do tutorial nesta página
Outro tutorial mais aprofundado tutorial on DiagammeR
Consulte esta página Sankey diagrams