20 Campos em branco/faltantes
Está página irá abordar como:
- Avaliar a quantidade de campos em branco
- Filtrar linhas com variáveis em branco
- Traçar um gráfico da quantidade de campos em branco ao longo do tempo
- Modificar como os valores
NAsão mostrados nos gráficos - Realizar a atribuição de valores aos campos em branco: MCAR, MAR, MNAR
20.1 Preparando o ambiente R
Carregue os pacotes
O código abaixo realiza o carregamento dos pacotes necessários para a análise dos dados. Neste manual, enfatizamos o uso da função p_load(), do pacman, que instala os pacotes, caso não estejam instalados, e os carrega no R para utilização. Também é possível carregar pacotes instalados utilizando a função library(), do R base. Para mais informações sobre os pacotes do R, veja a página Introdução ao R.
pacman::p_load(
rio, # importar/exportar
tidyverse, # gerenciamento e visualização dos dados
naniar, # avaliar e visualizar campos em branco
mice # atribuir valores aos campos em branco
)Importe os dados
Nós iremos importar o banco de dados de casos de uma simulação de epidemia de Ebola. Se você quiser acompanhar os passos abaixo, clique aqui para fazer o download do banco de dados ‘limpo’ (como arquivo .rds). Importe seus dados utilizando a função import() do pacote rio (esta função importa muitos tipos de arquivos, como .xlsx, .rds, .csv - veja a página Importar e exportar para detalhes).
# importe os dados no R
linelist <- import("linelist_cleaned.rds")As primeiras 50 linhas do banco são mostradas abaixo
Padronize os dados dos campos em branco durante a importação
Ao importar os seus dados, esteja ciente de valores que deveriam ser classificados como campos em branco. Por exemplo, 99, 999, “Missing”, células em branco (““), ou céluas com um espaço em branco (” “). Você pode converter esses tipos de campos em branco (e outros) para NA (formato de campos em branco no R) ainda no código usado para importar os dados. Veja a seção sobre como importar os dados na página Campos em branco para mais detalhes, uma vez que a sintaxe muda de acordo com o tipo de arquivo.
20.2 Campos em branco no R
Abaixo, nós exploramos as formas como os campos em branco são mostrados e analisados no R, assim como valores e funções adjacentes.
NA
No R, campos em branco são representados por um valor especial e reservado - NA. Notar que é escrito sem as aspas. Já “NA”, com aspas, é diferente, sendo apenas um caractere normal no R (além de ser parte da letra de Hey Jude dos Beatles).
Os campos em branco nos seus dados podem estar representados com outras formas, como “99”, ou “Missing”, ou “Unknown” - ou até como um caractere vazio ““, parecido com um campo em branco, ou um espaço simples” “. Esteja ciente disto, e verifique a possibilidade de converte-los para NA durante a importação ou durante a limpeza dos dados, com a função na_if().
Durante a limpeza dos dados, você também pode realizar o oposto - alterando todos os NA para “Missing”, ou algo similar, utilizando a função replace_na() ou fct_explicit_na(), para valores da classe Factor.
Versões de NA
Para boa parte dos casos, NA representa os campos em branco e tudo funciona bem. Entretanto, em algumas circunstâncias você pode precisar de variações do NA, específicas para uma classe de objeto (caractere, numérico, etc). Isto será raro, mas esteja ciente dessa possibilidade. O cenário típico para isso ocorre ao criar uma nova coluna com a função case_when(), do pacote dplyr. Como descrito na página sobre Limpeza dos dados e principais funções, esta função verifica cada linha do banco de dados, avalia se cumprem regras lógicas (lado direito do código), e atribuem um novo valor correto (lado esquerdo do código). Importante: todos os valores no lado direito da fórmula precisam ser da mesma classe.
linelist <- linelist %>%
# Cria uma nova coluna chamada "age_years", utilizando a coluna "age"
mutate(age_years = case_when(
age_unit == "years" ~ age, # se a idade (age) é dada em anos, este valor é mantido
age_unit == "months" ~ age/12, # se a idade é dada em meses, o valor é dividido por 12
is.na(age_unit) ~ age, # se a unidade da idade não é informada, assume-se que sejam em anos
TRUE ~ NA_real_)) # para qualquer outra circunstância, o valor de campo em branco é atribuídoSe você quiser NA no lado direito, é necessário especificar uma das opções especiais da NA, listadas abaixo. Para valores em caracteres, utilize “Missing” em vez de NA, ou NA_character_. Se todos os valores são numéricos, utilize a opção NA_real_. Se todos os valores são formato de datas ou lógicos, utilize NA.
NA- utilize para datas ou valores lógicos TRUE/FALSENA_character_- utilize para caracteresNA_real_- utilize para valores numéricos
Novamente, é improvável que você encontre essas variações, a não ser que você esteja utilizando a função case_when()para criar uma nova coluna. Veja a documentação do R sobre NA para mais informações.
NULL
NULL é outro valor reservado (especial) no R. É a representação lógica de uma declaração que não é nem verdadeira (true), tampouco falsa (false). Ele é gerado por expressões ou funções em que os valores são indefinidos. Geralemnte, não atribua NULL como um valor, exceto ao escrever funções ou, talvez, uma [aplicação em shiny][Dashboards com Shiny] para gerar NULL em cenários específicos.
Para avaliar se é um valor NULL, é só utilizar a função is.null(), e conversões podem ser feitas com a função as.null().
Veja essa postagem detalhando as diferenças entre NULL e NA.
NaN
Valores impossíveis são representados pelo valor especial NaN. Um exemplo disso é quando você força o R a dividir 0 por 0. Você pode verificar se algo é impossível com a função is.nan(). Você também pode encontrar funções complementares, incluindo is.infinite() e is.finite().
Inf
Inf representa um valor infinito, como o obtido ao dividir um número por 0.
Um exemplo de como isto pode impactar seu trabalho: digamos que você possua um vetor/coluna z que contém estes valores: z <- c(1, 22, NA, Inf, NaN, 5)
Se você utilizar a função max() nesta coluna para encontrar o maior valor, é possível utilizar o atributo na.rm = TRUE para remover o NA da análise, mas os valores Inf e NaN continuarão e Inf será o resultado desta análise. Para resolver isto, você pode utilizar colchetes quadrados [ ] e a função is.finite() para analisar apenas o subconjunto de dados com apenas valores finitos: max(z[is.finite(z)]).
z <- c(1, 22, NA, Inf, NaN, 5)
max(z) # retorna NA
max(z, na.rm=T) # retorna Inf
max(z[is.finite(z)]) # retorna 22Exemplos
| Comando R | Resultado |
|---|---|
5 / 0 |
Inf |
0 / 0 |
NaN |
5 / NA |
NA |
5 / Inf |0|NA|Inf| "logical"class(NaN)| "numeric"class(Inf)| "numeric"class(NULL)` |
“NULL” |
“NAs introduzidos forçadamente” (“NAs introduced by coercion”) é uma mensagem de aviso comum. Isto pode acontecer se você tentar realizar uma conversão ilegal, como inserir um valor do tipo caractere em um vetor numérico.
as.numeric(c("10", "20", "thirty", "40"))Warning: NAs introduced by coercion[1] 10 20 NA 40NULL é ignorado em um vetor.
my_vector <- c(25, NA, 10, NULL) # defina
my_vector # print[1] 25 NA 10Variação de um número resulta em NA.
var(22)[1] NA20.3 Funções úteis
Abaixo são elencadas funções úteis do R base para avaliar e trabalhar com campos em branco:
is.na() e !is.na()
Utilize a função is.na() para identificar campos em branco, ou utilize o oposto desta função (com ! na frente) para identificar campos preenchidos. Ambas funções retornam um valor lógico (TRUE ou FALSE). Lembre que você pode somar (sum()) o vetor resultante para contar a quantidade de TRUE, ex.: sum(is.na(linelist$date_outcome)).
my_vector <- c(1, 4, 56, NA, 5, NA, 22)
is.na(my_vector)[1] FALSE FALSE FALSE TRUE FALSE TRUE FALSE!is.na(my_vector)[1] TRUE TRUE TRUE FALSE TRUE FALSE TRUEsum(is.na(my_vector))[1] 2na.omit()
Esta função, se aplicada em um conjunto de dados, irá remover linhas com qualquer campo em branco. Esta função também é do pacote R base.
Se aplicada em um vetor, os valores NA neste vetor serão removidos. Por exemplo:
na.omit(my_vector)[1] 1 4 56 5 22
attr(,"na.action")
[1] 4 6
attr(,"class")
[1] "omit"drop_na()
Esta é uma função do pacote tidyr, que é útil em um pipeline de limpeza de dados. Se executada com os parênteses vazios, ela remove linhas com qualquer um dos campos esteja em brancos. Se o nome das colunas são especificados em parênteses, linhas com campos em branco apenas nestas colunas serão excluídas. Você pode também utilizar a sintaxe do “tidyselect” para especificar as colunas.
linelist %>%
drop_na(case_id, date_onset, age) # exclui linhas com campos em branco em alguma dessas colunasna.rm = TRUE
Quando você executa uma função matemática, como max(), min(), sum() ou mean(), se existir quaisquer valores NA presentes, o resultado da análise será NA. Este comportamento padrão é intencional, de forma que você seja alertado no caso de seus dados estarem em branco.
Você pode evitar isto ao remover os campos em branco dos cálculos. Para fazer isto, inclua o argumento na.rm = TRUE (“na.rm” significa “remova os NA”).
my_vector <- c(1, 4, 56, NA, 5, NA, 22)
mean(my_vector) [1] NAmean(my_vector, na.rm = TRUE)[1] 17.620.4 Avalie os campos em branco no conjunto de dados
Você pode utilizar o pacote naniar para avaliar e visualizar os campos em brancos no seu conjunto de dados presente no objeto linelist.
# instale e/ou carregue o pacote
pacman::p_load(naniar)Quantificando os campos em branco
Para encontrar o percentual da quantidade de campos em branco, utilize a função pct_miss(). Utilize n_miss() para obter o número absoluto de campos em branco.
# percentual de TODOS os campos em branco no banco de dados
pct_miss(linelist)[1] 6.688745As duas funções abaixo retornam o percentual de linhas com qualquer campo em branco, ou que estão com todos os campos preenchidos, respectivamente. Lembre que NA significa perdido/em branco, e que "" ou " " não serão considerados campos em branco.
# Percentual de linhas com algum campo em branco
pct_miss_case(linelist) # utilize n_complete() para obter quantidades absolutas[1] 69.12364# Percentual de linhas com todos os campos preenchidos (sem campos em branco)
pct_complete_case(linelist) # utilize n_complete() para obter quantidades absolutas[1] 30.87636Visualizando as quantidades de campos em branco
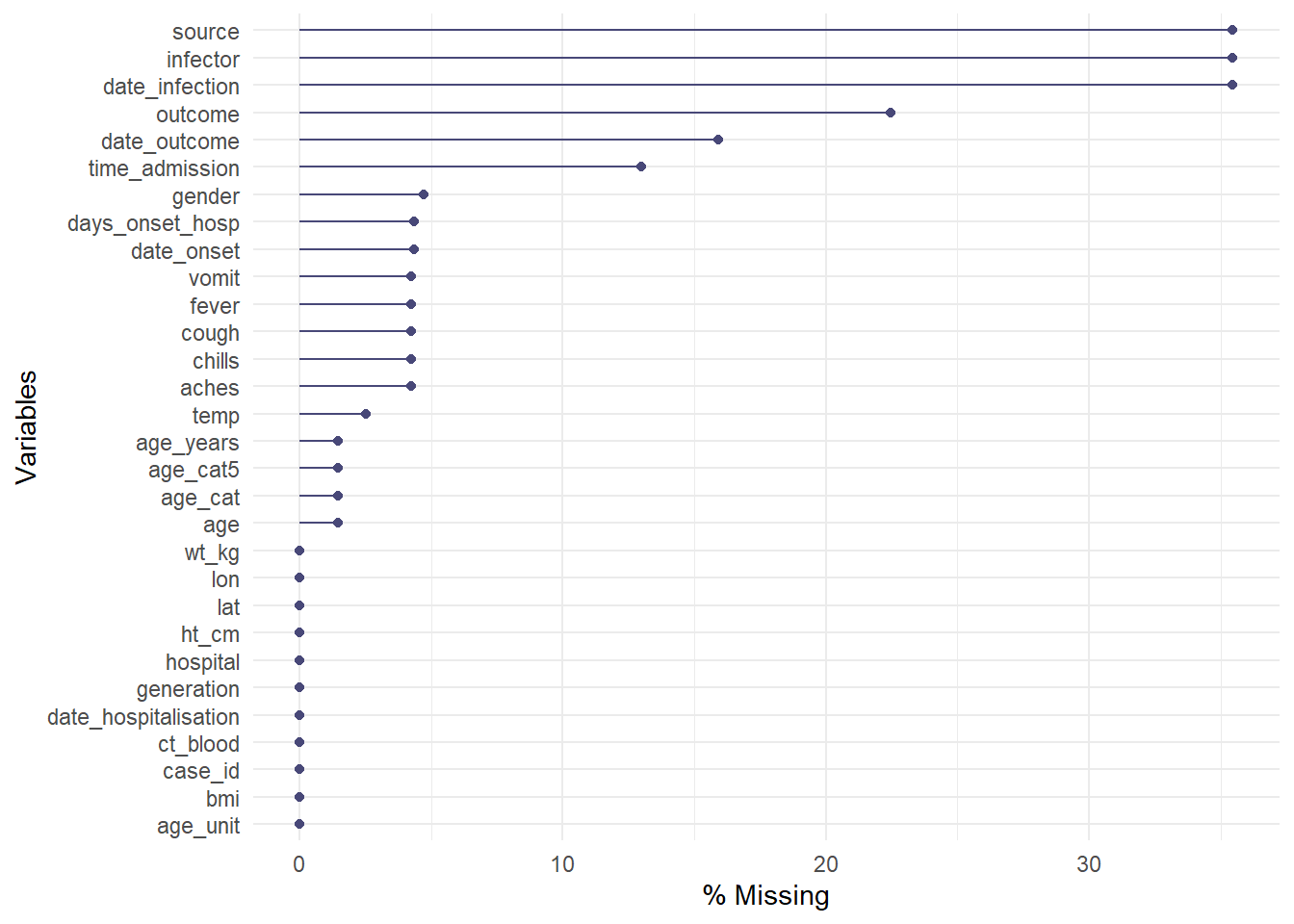
A função gg_miss_var() irá gerar o número absoluto (ou %) de campos em branco em cada coluna. Seguem alguns detalhes:
- Você pode adicionar um nome de coluna (sem as aspas) ao atributo
facet =para visualizar o gráfico por grupos - Por padrão, contagens absolutas são mostradas no lugar dos percentuais. Altere isso com o atributo
show_pct = TRUE
- Você pode adicionar etiquetas aos eixos e títulos utilizando
+ labs(...), como noggplot()
Aqui, os dados são encadeados %>% (do inglês pipe) na função. O atributo facet = também é utilizado para dividir os dados.
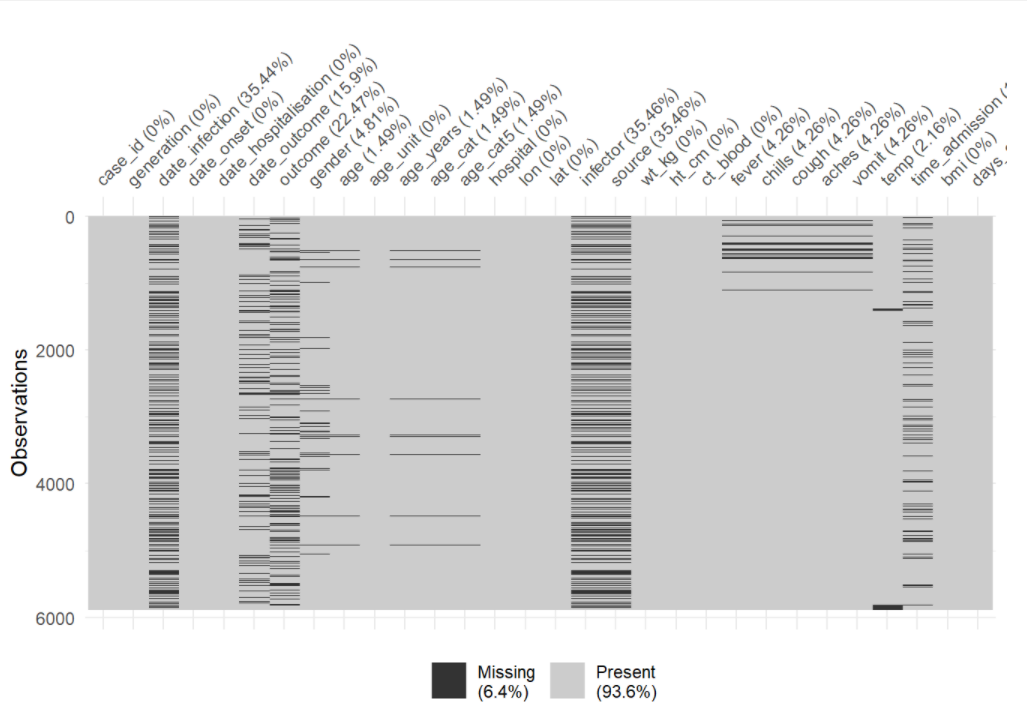
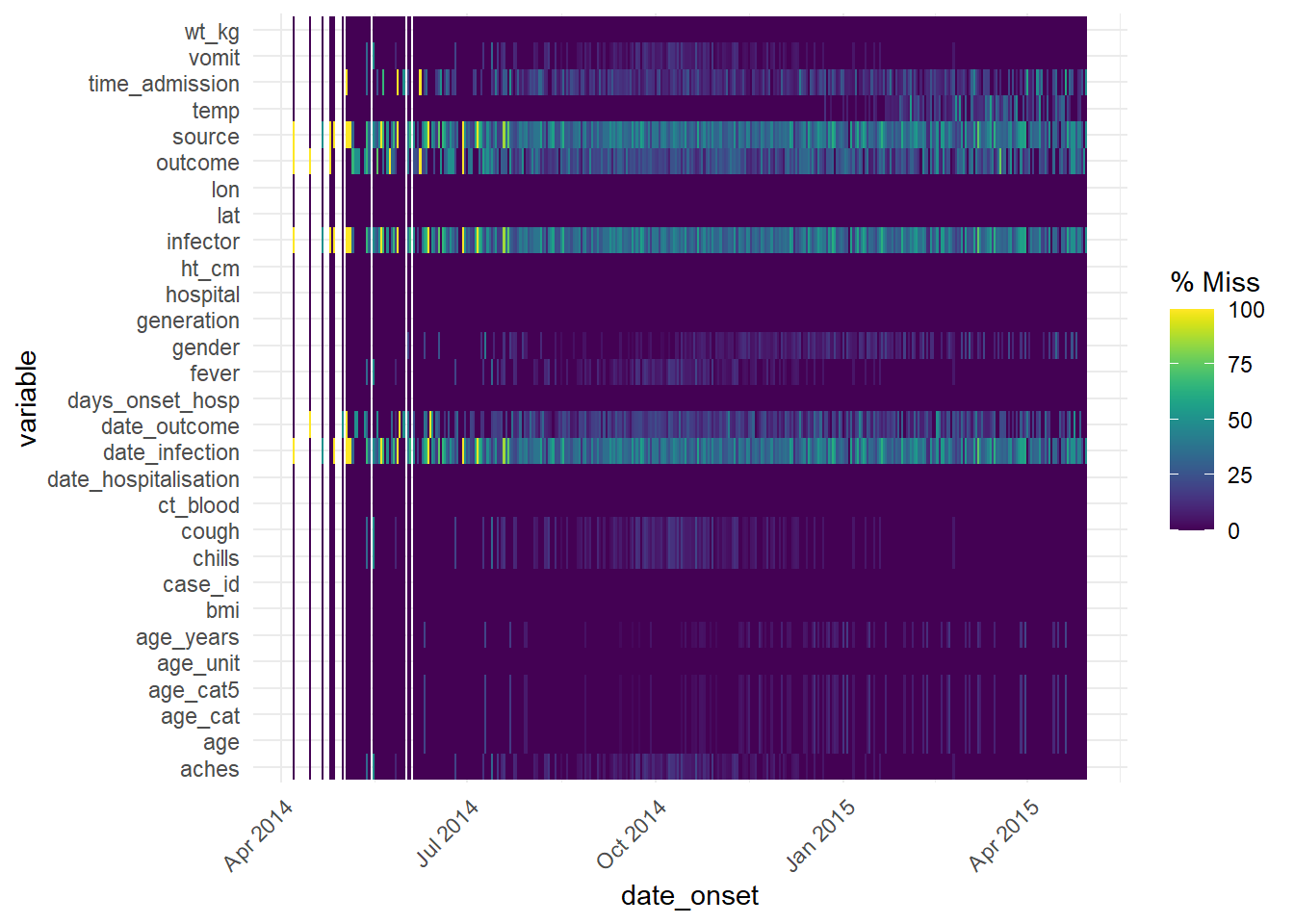
Você pode utilizar a função vis_miss() para visualizar os dados como um mapa de calor, mostrando se algum campo está em branco ou não. Também é possível utilizar o select() para escolher colunas específicas do banco de dados, e trabalhar apenas com elas.
Explore e visualize as relações entre os campos em branco
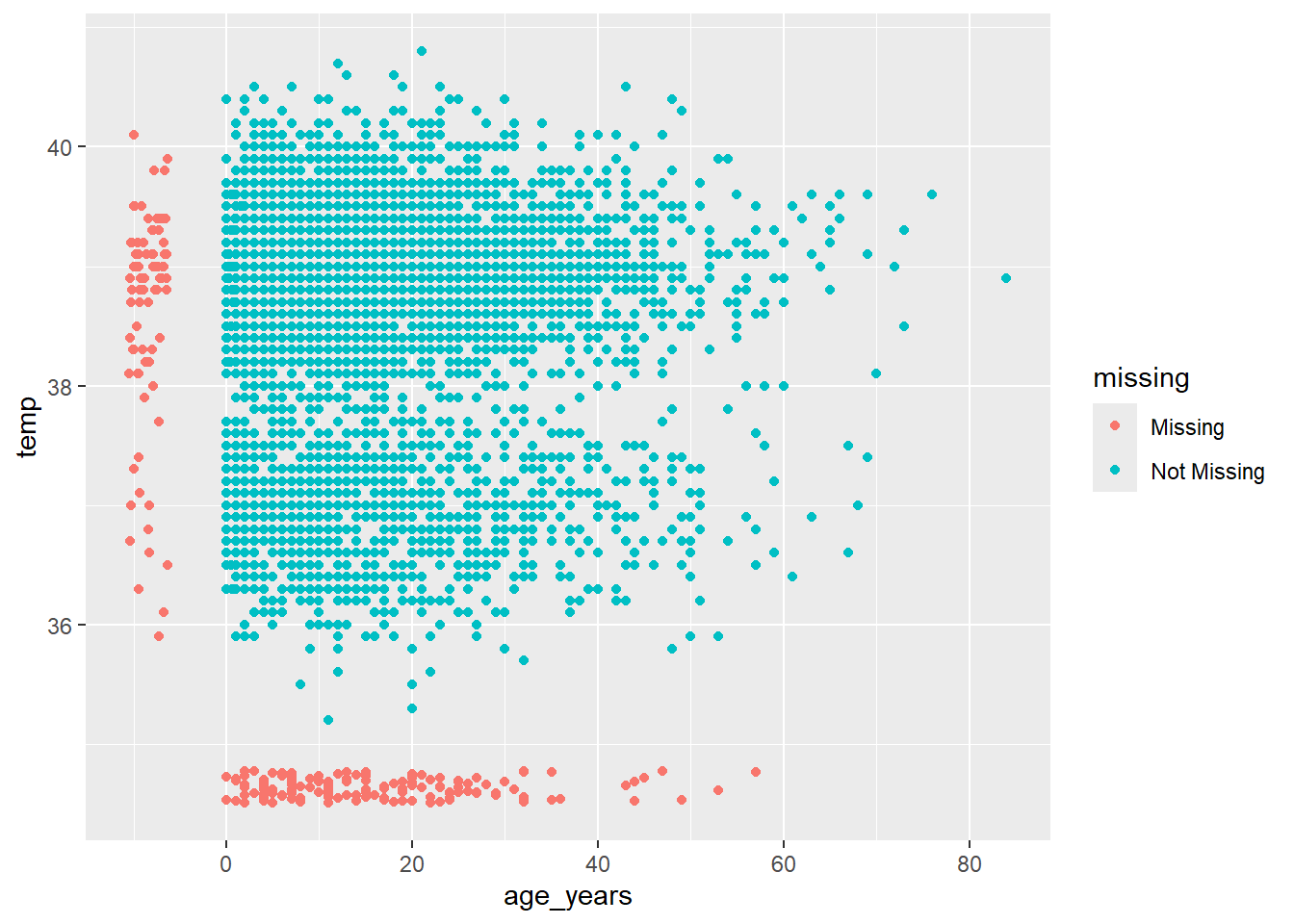
Como você visualiza algo que não existe??? Por padrão, o ggplot() remove pontos sem valores dos gráficos.
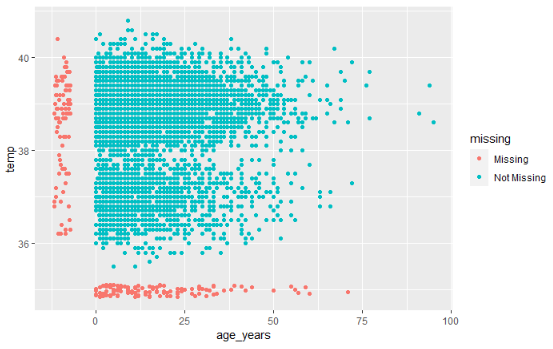
O pacote naniar oferece uma solução com a função geom_miss_point(). Ao criar um gráfico de dispersão de duas colunas, uma delas é construída sem os valores em branco, e a outra com estes pontos, onde valores 10% menores do que o menor valor da coluna são atribuídos a estes campos em branco, que são então coloridos de forma distinta dos demais pontos.
No gráfico de dispersão abaixo, os pontos vermelhos são os que foram adicionados, estando presentes em uma coluna, mas não na outra. Isto permite visualizar a distribuição de campos em branco em relação aos campos preenchidos.
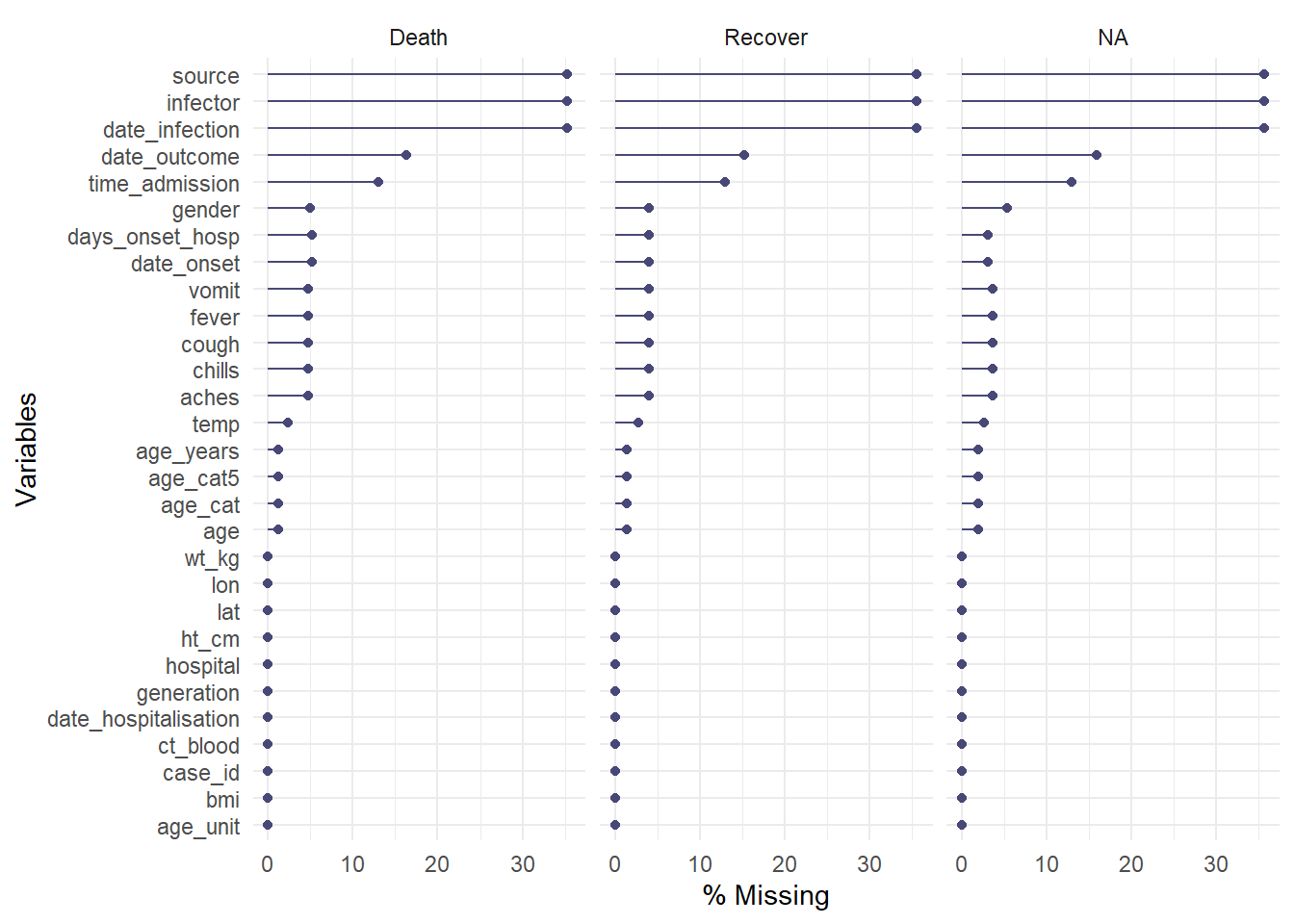
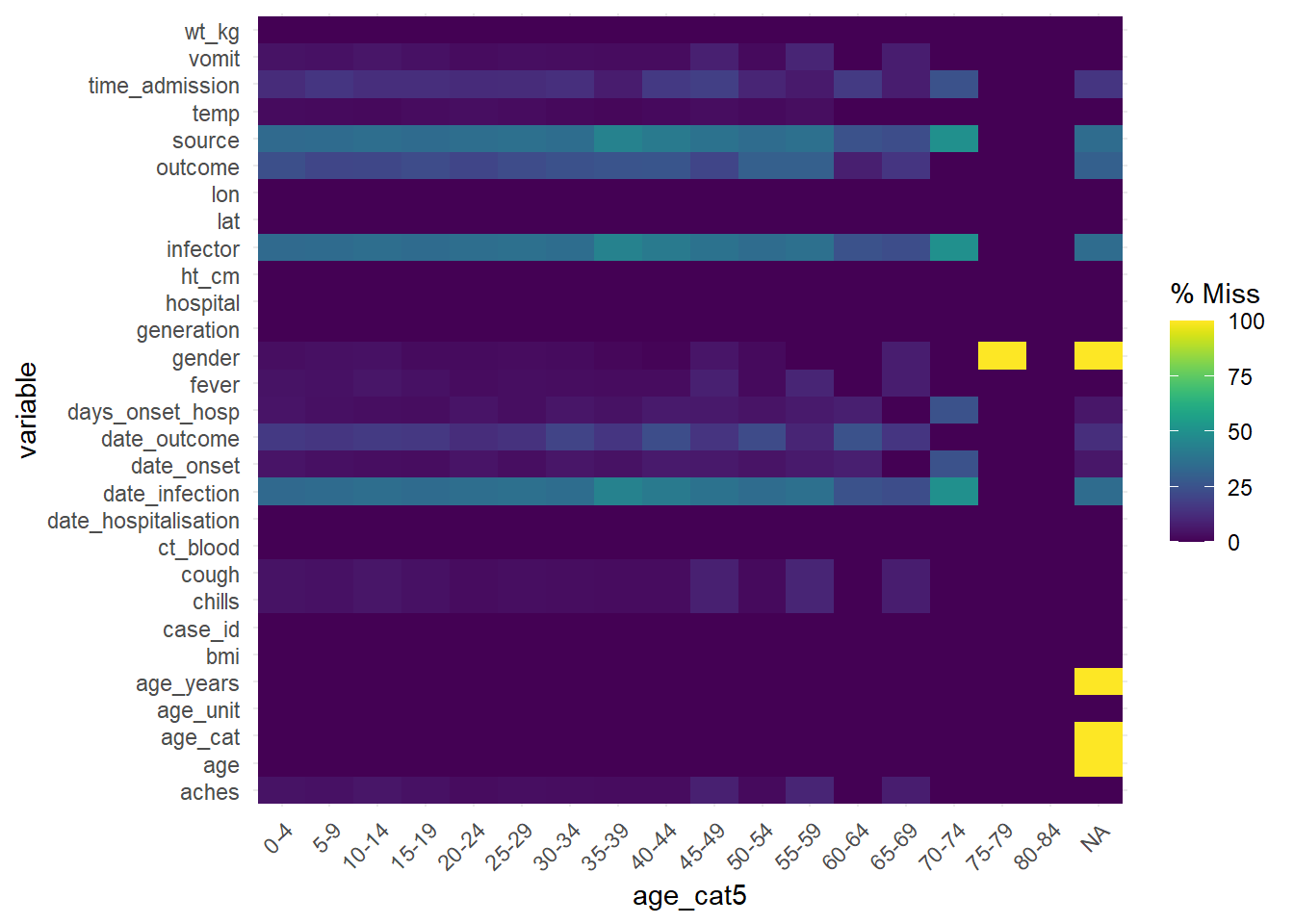
Para analisar os campos em branco nos dados, estratificados por outra coluna, utilize a função gg_miss_fct(), que gera um mapa de calor com o percentual de campos em brancos no banco de dados utilizando uma coluna da classe factor/categórica (ou por datas):
gg_miss_fct(linelist, age_cat5)Warning: There was 1 warning in `mutate()`.
ℹ In argument: `age_cat5 = (function (x) ...`.
Caused by warning:
! `fct_explicit_na()` was deprecated in forcats 1.0.0.
ℹ Please use `fct_na_value_to_level()` instead.
ℹ The deprecated feature was likely used in the naniar package.
Please report the issue at <https://github.com/njtierney/naniar/issues>.Esta função também pode ser utilizada com uma coluna com datas, para visualizar como a quantidade de campos em branco alterou de acordo com o tempo:
Colunas Sombra “Shadow”
Outra forma de visualizar os campos em branco em uma coluna, de acordo com os valores de uma segunda coluna, é utilizando o “shadow” que o pacote naniar consegue criar. A função bind_shadow() cria uma nova coluna com os valores binários NA/not NA para cada coluna existente, e então conecta todas essas colunas novas ao banco de dados original utilizando o sufixo “_NA” em seus nomes. Isto duplica o número de colunas:
shadowed_linelist <- linelist %>%
bind_shadow()
names(shadowed_linelist) [1] "case_id" "generation"
[3] "date_infection" "date_onset"
[5] "date_hospitalisation" "date_outcome"
[7] "outcome" "gender"
[9] "age" "age_unit"
[11] "age_years" "age_cat"
[13] "age_cat5" "hospital"
[15] "lon" "lat"
[17] "infector" "source"
[19] "wt_kg" "ht_cm"
[21] "ct_blood" "fever"
[23] "chills" "cough"
[25] "aches" "vomit"
[27] "temp" "time_admission"
[29] "bmi" "days_onset_hosp"
[31] "case_id_NA" "generation_NA"
[33] "date_infection_NA" "date_onset_NA"
[35] "date_hospitalisation_NA" "date_outcome_NA"
[37] "outcome_NA" "gender_NA"
[39] "age_NA" "age_unit_NA"
[41] "age_years_NA" "age_cat_NA"
[43] "age_cat5_NA" "hospital_NA"
[45] "lon_NA" "lat_NA"
[47] "infector_NA" "source_NA"
[49] "wt_kg_NA" "ht_cm_NA"
[51] "ct_blood_NA" "fever_NA"
[53] "chills_NA" "cough_NA"
[55] "aches_NA" "vomit_NA"
[57] "temp_NA" "time_admission_NA"
[59] "bmi_NA" "days_onset_hosp_NA" Estas colunas “shadow” podem ser utilizadas para traçar um gráfico da proporção de campos em branco, em relação à qualquer outra coluna.
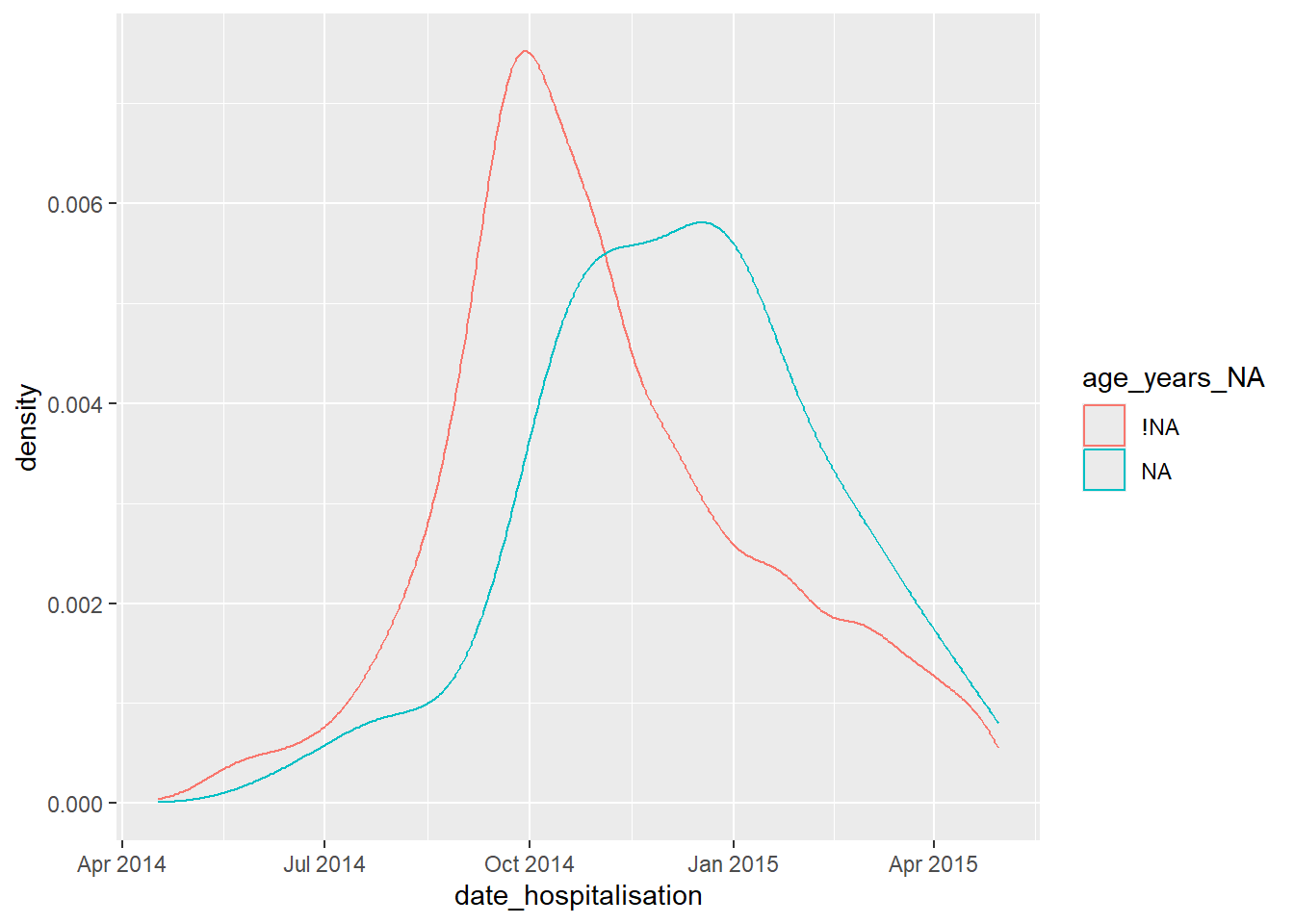
Por exemplo, o gráfico abaixo mostra a proporção de campos em branco na coluna days_onset_hosp (número de dias entre o início dos sintomas e a hospitalização), de acordo com o campo em date_hospitalisation. Essencialmente, você está criando um gráfico da densidade da coluna no eixo x, mas estratificando os resultados (color =) pela coluna “shadow” de interesse. Esta análise funciona melhor se o eixo x é uma coluna numérica ou com dados cronológicos.
ggplot(data = shadowed_linelist, # banco de dados com as colunas "shadow"
mapping = aes(x = date_hospitalisation, # colunas numéricas ou cronológicas
colour = age_years_NA)) + # coluna "shadow" de interesse
geom_density() # adição das curvas de densidadeVocê pode também utilizar as colunas “shadow” para estratificar um resumo estatístico, como mostrado abaixo:
linelist %>%
bind_shadow() %>% # cria as colunas "shadow"
group_by(date_outcome_NA) %>% # coluna "shadow" escolhida para estratificar
summarise(across(
.cols = age_years, # variável de interesse para realizar os cálculos
.fns = list("mean" = mean, # estatísticas calculadas
"sd" = sd,
"var" = var,
"min" = min,
"max" = max),
na.rm = TRUE)) # outros argumentos para o cálculo das estatísticasWarning: There was 1 warning in `summarise()`.
ℹ In argument: `across(...)`.
ℹ In group 1: `date_outcome_NA = !NA`.
Caused by warning:
! The `...` argument of `across()` is deprecated as of dplyr 1.1.0.
Supply arguments directly to `.fns` through an anonymous function instead.
# Previously
across(a:b, mean, na.rm = TRUE)
# Now
across(a:b, \(x) mean(x, na.rm = TRUE))# A tibble: 2 × 6
date_outcome_NA age_years_mean age_years_sd age_years_var age_years_min
<fct> <dbl> <dbl> <dbl> <dbl>
1 !NA 16.0 12.6 158. 0
2 NA 16.2 12.9 167. 0
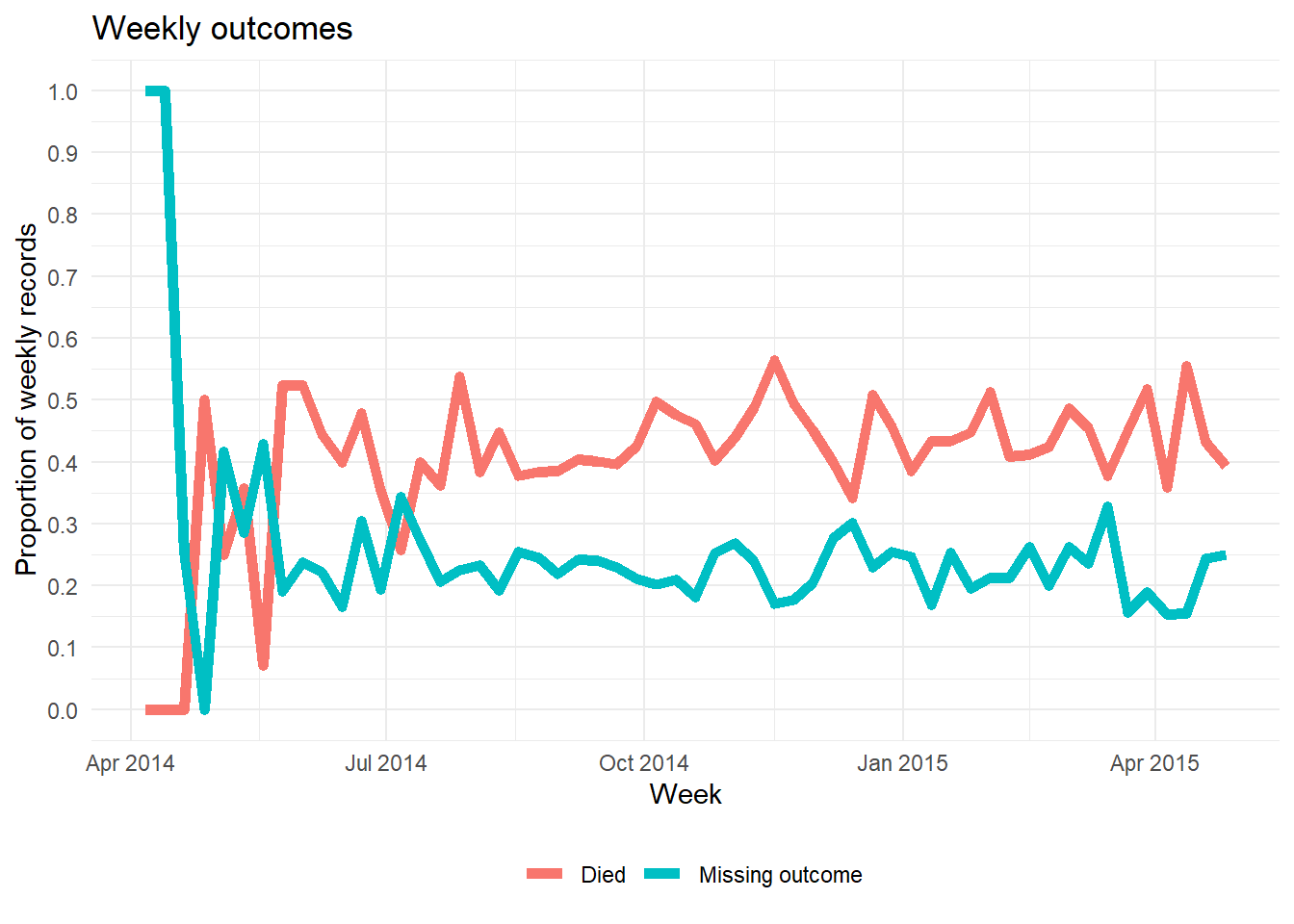
# ℹ 1 more variable: age_years_max <dbl>Uma forma alternativa para traçar um gráfico da proporção de campos em branco em uma coluna de acordo com o tempo é mostrada abaixo. Esta forma não involve o pacote naniar. Este exemplo mostra a porcentagem de observações semanais que estão em branco.
- Agregue os dados em uma unidade de tempo útil (dias, semanas, etc.), resumindo a proporção de observações com
NA(e/ou qualquer outro valor de interesse) - Faça um gáfico da proporção de campos em brancos como uma linha, utilizando o
ggplot()
Abaixo, nós trabalhamos com a linelist, adicionamos uma nova coluna contendo a semana, então agrupamos os dados por semana, e calculamos o percentual de registros em branco de acordo com a semana. (nota: se você quisesse obter a % por 7 dias, o cálculo seria sutilmente diferente).
outcome_missing <- linelist %>%
mutate(week = lubridate::floor_date(date_onset, "week")) %>% # crie uma coluna com as semanas
group_by(week) %>% # agrupe as linhas por semana
summarise( # faça o resumo por cada semana
n_obs = n(), # número absoluto de registros
outcome_missing = sum(is.na(outcome) | outcome == ""), # número de registros com campos em branco
outcome_p_miss = outcome_missing / n_obs, # proporção de registros em branco
outcome_dead = sum(outcome == "Death", na.rm=T), # número de registros com evolução para óbito
outcome_p_dead = outcome_dead / n_obs) %>% # proporção de registros com evolução para óbito
tidyr::pivot_longer(-week, names_to = "statistic") %>% # para utilizar o ggplot, altere todas as colunas, exceto a coluna com as semanas, para o formato longo
filter(stringr::str_detect(statistic, "_p_")) # mantenha apenas os valores proporcionaisEntão nóstraçamos um gráfico das proporções dos campos em branco como uma linha, de acordo com a semana. A página sobre básico do ggplot pode ser utilizada se você não tiver familiaridade com o pacote ggplot2 de visuzalização de dados.
ggplot(data = outcome_missing)+
geom_line(
mapping = aes(x = week, y = value, group = statistic, color = statistic),
size = 2,
stat = "identity")+
labs(title = "Weekly outcomes",
x = "Week",
y = "Proportion of weekly records") +
scale_color_discrete(
name = "",
labels = c("Died", "Missing outcome"))+
scale_y_continuous(breaks = c(seq(0,1,0.1)))+
theme_minimal()+
theme(legend.position = "bottom")20.5 Utilizando dados com campos em branco
Exclua linhas com campos em branco
Para rapidamente excluir linhas com valores em branco, utilize a função drop_na(), do pacote dplyr.
O objeto linelist original possui nrow(linelist) linhas. O número ajustado de linhas, após exclusão das que possuíam campos em branco, é mostrado abaixo:
linelist %>%
drop_na() %>% # excluí linhas com QUALQUER campo em branco
nrow()[1] 1818Você pode especificar para excluir linhas com campos em branco apenas em colunas específicas:
linelist %>%
drop_na(date_onset) %>% # exclua as linhas com campos em branco na coluna date_onset
nrow()[1] 5632Você pode listar as colunas uma após a outra, ou utilizar as funções auxiliares do “tidyselect”:
linelist %>%
drop_na(contains("date")) %>% # exclua linhas com campos em branco em qualquer coluna que contenha "date" no nome
nrow()[1] 3029Trabalhando com NA no ggplot()
Frequentemente, é sábio mostrar a quantidade de valores excluídos na confeção de um gráfico em sua legenda. Abaixo é um exemplo:
No ggplot(), você pode adicionar etiquetas com labs(), e dentro da função utilizar o atributo caption =. Neste atributo, você pode utilizar a função str_glue(), do pacote stringr, para unir os valores em uma senteça, de forma que eles são ajustados automaticamente. Um exemplo é mostrado abaixo:
- Observe o uso do
\npara adicionar uma nova linha - Observe que, caso colunas múltiplas contribuam para a exclusão dos valores do gráfico (ex.: age ou sex se estes interferirem no gráfico), então você precisa filtrar por estas colunas assim como calcular o número de registros não mostrados de forma correta.
labs(
title = "",
y = "",
x = "",
caption = stringr::str_glue(
"n = {nrow(central_data)} do Hospital Central;
{nrow(central_data %>% filter(is.na(date_onset)))} registros sem as datas de início dos sintomas não são mostrados.")) As vezes, pode ser mais fácil salvar o texto em um objeto em comandos anteriores a função ggplot(), e, simplesmente, referenciar o objeto criado dentro da função str_glue().
NA na classe factors
Se a sua coluna de interesse for um fator (da classe factors), utilize a função fct_explicit_na(), do pacote forcats, para converter os valores do tipo NA para valores do tipo caractere. Veja mais detalhes na página Fatores. Por padrão, o novo valor atribuído é “(Missing)”, mas pode ser ajustado através do argumento na_level =.
pacman::p_load(forcats) # carregue o pacote
linelist <- linelist %>%
mutate(gender = fct_explicit_na(gender, na_level = "Missing"))
levels(linelist$gender)[1] "f" "m" "Missing"20.6 Imputação de dados nos campos em branco
As vezes, ao analisar os dados, será importante “preencher as lacunas” e atribuir valores nos campos vazios. Mesmo que você possa analisar os dados após remover todos os campos em branco, isto pode causar diversos problemas. Aqui estão dois exemplos:
Ao remover todas as observações com campos em branco, ou variáveis com uma quantidade elevada de dados em branco, você pode reduzir seu poder de amostra ou capacidade para realizar algumas análises. Por exemplo, como descobrimos anteriormente, apenas uma pequena fração das observações no nosso banco de dados no linelist não possui campos em branco em todas as variáveis. Se nós removessemos a maioria dessas linhas, estaríamos perdendo muita informação! Também vimos que boa parte das nossas variáveis possui alguma quantidade de dados em branco – assim, para boa parte da análise, provavelmente não é razoável excluir todas as variáveis que possuem muitos campos vazios.
Dependendo do motivo de seus dados estarem em branco, realizar a análise apenas de dados completos pode levar a resultados enviesados ou incorretos. Por exemplo, anteriormente, nós descobrimos que estamos sem dados de alguns pacientes no que tange à presença de sintomas importantes, como febre e tosse. Mas, como uma possibilidade, talvez essa informação não foi registrada para pessoas que, obviamente, não estavam muito doentes. Neste caso, se nós apenas removermos essas observações, estaremos excluindo algumas das pessoas mais saudáveis do nosso banco de dados, o que iria enviesar nossos resultados.
É importante pensar sobre o porque seus dados podem estar em branco, além de avaliar a quantidade de campos em branco. Fazer isto pode ajudá-lo a decidir o quão importante será atribuir valores nos campos em branco, e qual o melhor método de imputação para a sua situação.
Tipos de dados em branco
Aqui estão três tipos gerais de dados em branco:
Dados faltantes completamente de forma aleatória (MCAR, do inglês Missing Comnpletely at Random). Isto significa que não existe relação entre os dados em branco e qualquer outra variável dos seus dados. A probabilidade dos dados estarem em branco são as mesmas para todos os casos. Isto é uma situação rara. Mas, se você tiver uma forte razão para acreditar que seus dados são do tipo MCAR, analisar apenas os dados completos sem atribuir valores não irá enviesar seus resultados (apesar de que você pode perder algum poder de amostra). [A fazer: considere discutir testes estatísticos para MCAR]
Dados faltantes aleatoriamente (MAR, do inglês Missing at Random). Este nome é, na verdade, um pouco incorreto, uma vez que dados do tipo MAR não estão perdidos de forma alearória, e sim de forma sistemática e previsível, baseado em outras informações que você tem. Por exemplo, talvez cada observação em branco do seu banco de dados para febre não foi registrada porque assumiram que todos os pacientes com calafrios e dores estavam com febre, e, então, suas temperaturas não foram medidas. Se verdade, nós poderíamos facilmente predizer que cada observação em branco em que o paciente tivesse calafrios e dores, ele também teve febre, e utilizar essa informação para atribuir dados. Na prática, existe um espectro de possibilidades. Talvez, se um paciente sem a temperatura medida tivesse tanto calafrios quanto dores, ele provavelmente também teria febre, mas nem sempre. Isto ainda é previsível, embora não seja perfeitamente previsível. Este é um tipo comum de perda de dados.
Dados faltantes de forma não aleatória (MNAR, do inglês Missing not at Random). As vezes, também chamado de Não perdidos aleatoriamente (NMAR). Esta situação considera que a probabilidade de um campo estar em branco NÃO é sistemática ou previsível utilizando as outras informações que temos, mas também não foram perdidos de forma aleatória. Assim, os dados foram perdidos por razões desconhecidas ou por motivos que você não tem informações sobre. Por exemplo, em nosso banco de dados, talvez as informações sobre as idades estejam em branco porque alguns pacientes muito idosos ou não sabiam a idade, ou recusaram informar a idade. Nesta situação, os dados perdidos de idade estão relacionados à idade diretamente (e, assim, não são perdas aleatórias), e não são dados previsíveis através de outras informações que temos. MNAR é complexo e, frequentemente, a melhor forma de trabalhar com isso é tentar coletar mais dados ou informações sobre o porque os dados estão faltando, em vez de atribuir valores.
Concluindo, geralmente, atribuir valores em dados MCAR é simples, enquanto em dados MNAR é desafiador, senão impossível. Muitos dos métodos de imputação de valores assumem dados MAR.
Pacotes úteis
Alguns pacotes úteis para imputar dados perdidos são Mmisc, missForest (que utiliza o modelo de florestas aleatórias para imputar dados perdidos), e mice (Imputação Multivariada por Equações em Cadeia). Para essa seção, nós iremos utilizar apenas o pacote mice, que implementa uma variedade de técnicas. O mantenedor do pacote mice publicou um livro online com mais detalhes sobre como imputar dados perdidos (https://stefvanbuuren.name/fimd/).
Segue o código para carregar o pacote mice:
pacman::p_load(mice)Imputação por média
As vezes, se você está realizando uma análise simples, ou possui uma forte razão para pensar que pode assumir os dados perdidos como MCAR, é possível simplesmente atribuir a média daquela variável nos campos em branco. Talvez possamos assumir que a perda de medições de temperatura em nosso banco de dados foi MCAR ou apenas valores normais. Aqui está o código para criar uma nova variável que substitui os valores faltantes pela temperatura média do nosso banco de dados. Entretanto, em muitas situações, substituir dados com o valor médio pode gerar resultados enviesados, então seja cuidadoso.
linelist <- linelist %>%
mutate(temp_replace_na_with_mean = replace_na(temp, mean(temp, na.rm = T)))Você pode também realizar um processo similar para substituir dados categóricos por um valor específico. Para o nosso banco de dados, imagine que você soubesse que todas observações com um campo em branco na variável outcome (evolução clínica, que pode ser “Death” ou “Recover”) foram de pessoas que evoluíram para óbito (nota: isto não é verdade para o nosso banco de dados):
linelist <- linelist %>%
mutate(outcome_replace_na_with_death = replace_na(outcome, "Death"))Imputação por regressão
Outro método de certa forma mais avançado para imputar valores é utilizar algum tipo de modelo estatístico para prever o que um valor perdido é. Aqui está um exemplo onde os valores preditos são criados para todos os campos sem a temperatura, mas com os campos de age (idade) e fever (febre) preenchidos, utilizando estas variáveis como preditoras em uma regressão linear simples. Na prática, você iria querer utilizar um modelo melhor do que este, que é mais simples.
simple_temperature_model_fit <- lm(temp ~ fever + age_years, data = linelist)
#utilizando o nosso simples modelo de temperatura para predizer valores de temperatura apenas para os campos em branco
predictions_for_missing_temps <- predict(simple_temperature_model_fit,
newdata = linelist %>% filter(is.na(temp))) Ou, utilizando a mesma abordagem de modelagem com o pacote mice para imputar valores de temperatura nos campos em branco:
model_dataset <- linelist %>%
select(temp, fever, age_years)
temp_imputed <- mice(model_dataset,
method = "norm.predict",
seed = 1,
m = 1,
print = F)Warning: Number of logged events: 1temp_imputed_values <- temp_imputed$imp$tempEste é o mesmo tipo de abordagem feita por alguns métodos mais avançados, como utilizando o pacote missForest para substituir os campos em branco pelos valores preditos. Neste caso, o modelo de predição utilizado é o de florestas aleatórias (random forest) em vez de uma regressão linear. Você pode utilizar outros tipos de modelos para fazer isso. Entretanto, enquanto esta abordagem funciona bem com dados MCAR, você deve ser cuidadoso se acredita que seus dados perdidos sejam do tipo MAR ou MNAR. A qualidade da imputação irá depender no quão bom o seu modelo de predição é, e, mesmo com um modelo muito bom, a variedade dos dados imputados pode ser subestimada.
LOCF e BOCF
Última observação levada adiante (LOCF, do inglês “Last observation carried forward”) e observação de base levada adiante (BOCF, do inglês “baseline observation carried forward”) são métodos de imputação para séries temporais/dados longitudinais. A ideia é utilizar o último valor observado para atribuir nos campos em branco. Quando valores múltiplos são perdidos sucessivamente, o método busca pelo último valor observado.
A função fill(), do pacote tidyr, pode ser utilizada para realizar imputação por LOCF e BOCF (entretanto, outros pacotes como HMISC, zoo, e data.table também incluem métodos para fazer isto). Para mostrar a sintaxe do fill(), nós iremos criar um simples banco de dados com série temporal contendo o número de casos de uma doença para cada quadrimestre dos anos 2000 e 2001. Entretanto, os valores para os quadrimestres após Q1 estão faltando, então nós iremos imputá-los. A sintaxe do fill() também é demonstrada na página sobre Pivoteamento dos dados.
#criando um banco de dados simples
disease <- tibble::tribble(
~quarter, ~year, ~cases,
"Q1", 2000, 66013,
"Q2", NA, 69182,
"Q3", NA, 53175,
"Q4", NA, 21001,
"Q1", 2001, 46036,
"Q2", NA, 58842,
"Q3", NA, 44568,
"Q4", NA, 50197)
#imputando os valores perdidos dos anos
disease %>% fill(year)# A tibble: 8 × 3
quarter year cases
<chr> <dbl> <dbl>
1 Q1 2000 66013
2 Q2 2000 69182
3 Q3 2000 53175
4 Q4 2000 21001
5 Q1 2001 46036
6 Q2 2001 58842
7 Q3 2001 44568
8 Q4 2001 50197Nota: tenha certeza de que seus dados estão ordenados corretamente antes de utilizar a função fill(). Por padrão, fill() irá preencher de cima para baixo, mas você também pode imputar valores em direções diferentes ao mudar o parâmetro .direction. Nós podemos criar um banco de dados similar onde os valores dos anos estão registrados apenas no ano final, e ausentes para quadrimestres anteriores:
#criando um banco de dados sutilmente diferente
disease <- tibble::tribble(
~quarter, ~year, ~cases,
"Q1", NA, 66013,
"Q2", NA, 69182,
"Q3", NA, 53175,
"Q4", 2000, 21001,
"Q1", NA, 46036,
"Q2", NA, 58842,
"Q3", NA, 44568,
"Q4", 2001, 50197)
#impute os anos nos campos em branco na direção para 'cima' ("up"):
disease %>% fill(year, .direction = "up")# A tibble: 8 × 3
quarter year cases
<chr> <dbl> <dbl>
1 Q1 2000 66013
2 Q2 2000 69182
3 Q3 2000 53175
4 Q4 2000 21001
5 Q1 2001 46036
6 Q2 2001 58842
7 Q3 2001 44568
8 Q4 2001 50197Neste exemplo, LOCF e BOCF são claramente os métodos corretos para se fazer, mas, em situações mais complicadas, pode ser mais difícil decidir se estes métodos são apropriados. Por exemplo, você pode ter valores em branco dos dados laboratoriais de um paciente do hospital após o primeiro dia. As vezes, isto quer dizer que os resultados laboratoriais não mudaram após o primeiro dia…mas também pode significar que o paciente se recuperou e seus valores seriam muito diferentes em relação ao primeiro dia! Utilize estes métodos com cautela.
Imputação múltipla
O livro online que mencionamos anteriormente, do autor do pacote mice (https://stefvanbuuren.name/fimd/), contém uma explicação detalhada da imputação múltipla e porque você gostaria de utilizá-la. Mas aqui está uma explicação básica do método:
Quando você faz uma imputação múltipla, múltiplos bancos de dados com valores plausíveis imputados nos campos em branco são criados (dependendo dos seus dados de pesquisa, você pode querer criar mais ou menos desses bancos de dados, mas o pacote mice produz 5 bancos de dados por padrão). A diferença é que, ao invés de um valor único e específico, cada valor imputado é retirado de uma distribuição estimada (então inclui alguma aleatoridade). Como resultado, cada um desses bancos de dados terá valores imputados ligeiramente diferentes (entretanto, os dados que não estavam em branco continuarão os mesmos nos diferentes bancos de dados). Você irá utilizar algum tipo de modelo preditivo para realizar a imputação em cada um dos novos bancos de dados (o pacote mice possui muitas opções para cada método de predição, incluíndo Correspondência Média Preditiva, regressão logística, e florestas aleatórias), mas o pacote mice cuida de muitos dos detalhes da modelagem.
Então, assim que você tiver criado estes novos bancos de dados com os valores imputados, é possível aplicar quaisquer modelos estatísticos ou análises que estava planejando realizar em cada um dos bancos, e então unir os resultados destes modelos. Isto funciona muito bem para reduzir o enviesamento dos resultados nas situações de MCAR e MAR, e, frequentemente, os resultados são mais acurados.
Aqui está um exemplo da aplicação da Imputação Múltipla para predizer as temperaturas em nosso banco de dados do linelist, utilizando as variáveis idade (age) e status da febre (fever) (nosso simples model_dataset anterior):
# imputando valores perdidos para todas as variáveis em nosso model_dataset, criando 10 novos bancos de dados com valores imputados
multiple_imputation = mice(
model_dataset,
seed = 1,
m = 10,
print = FALSE) Warning: Number of logged events: 1model_fit <- with(multiple_imputation, lm(temp ~ age_years + fever))
base::summary(mice::pool(model_fit)) term estimate std.error statistic df p.value
1 (Intercept) 3.703143e+01 0.0270863456 1.367162e+03 26.83673 1.583113e-66
2 age_years 3.867829e-05 0.0006090202 6.350905e-02 171.44363 9.494351e-01
3 feveryes 1.978044e+00 0.0193587115 1.021785e+02 176.51325 5.666771e-159Aqui nós utilizamos o método padrão do mice para imputação, que é a Correspondência Média Preditiva. Nós, então, utilizamos os bancos de dados gerados para, separadamente, estimar resultados com regressões lineares simples, e então uni-los. Existem muitos detalhes que não discutimos e muitas configurações que podem ser ajustadas durante o processo de Imputação Múltipla utilizando o pacote mice. Por exemplo, você nem sempre terá dados numéricos e pode precisar usar outros métodos de imputação (você ainda pode utilizar o pacote mice para muitos outros tipos de dados e métodos). Mas, para uma análise mais robusta quando os campos em branco são uma preocupação significativa, o método de Imputação Múltipla é uma boa solução que, quase sempre, não é muito mais trabalhosa do que realizar uma análise completa dos casos.
20.7 Recursos
Manual sobre o pacote naniar
Galeria com a visualização dos valores perdidos
Livro online sobre imputação múltipla no R, escrito pelo mantenedor do pacote mice