# instale a última versão do pacote do livro do Epi R
pacman::p_install_gh("appliedepi/epirhandbook")2 Baixe o livro e os dados
2.1 Baixe o livro offline
Você pode baixar a versão offline deste livro como um arquivo HTML para que você possa ver o arquivo em seu navegador, mesmo se você não tiver mais acesso à Internet. Se você está considerando o uso offline do livro do Epi R, aqui estão algumas coisas a serem consideradas:
- Quando você abre o arquivo, pode levar um ou dois minutos para as imagens e o índice serem carregados
- O livro off-line tem um layout ligeiramente diferente - uma página muito longa com Índice à esquerda. Para pesquisar termos específicos, use Ctrl+f (Cmd+f)
- Consulte a página Pacotes sugeridos para ajudá-lo a instalar os pacotes R apropriados antes que você perca a conectividade com a Internet
- Instale nosso pacote R epirhandbook que contém todos os dados de exemplo (processo de instalação descrito abaixo)
Existem duas maneiras de baixar o livro:
Use o link de download
Para acesso rápido, clique com o botão direito neste link e selecione “Salvar link como”.
Se estiver em um Mac, use Cmd+clique. Se estiver em um celular, pressione e segure o link e selecione “Salvar link”. O livro será baixado para o seu dispositivo. Se uma tela com código HTML bruto for exibida, certifique-se de seguir as instruções acima ou tente a Opção 2.
Use nosso pacote R
Oferecemos um pacote R denominado epirhandbook. Inclui uma função download_book () que baixa o arquivo do livro de nosso repositório Github para o seu computador.
Este pacote também contém uma função get_data() que baixa todos os dados de exemplo para o seu computador.
Execute o seguinte código para instalar nosso pacote R epirhandbook do repositório Github applyepi. Este pacote não está no CRAN, então use a função especial p_install_gh() para instalá-lo do Github.
Agora, carregue o pacote para uso em sua sessão R atual:
# carregue o pacote para uso
pacman::p_load(epirhandbook)Em seguida, execute a função do pacote download_book() (com parênteses vazios) para baixar o livro para o seu computador. Supondo que você esteja no RStudio, uma janela aparecerá permitindo que você selecione um local para salvar.
# baixe o livro offline para o seu computador
download_book()2.2 Baixe os dados para acompanhar
Para acompanhar as páginas do livro, você pode baixar os dados e resultados de exemplo.
Use nosso pacote R
A abordagem mais fácil para baixar todos os dados é instalar nosso pacote R epirhandbook. Ele contém uma função get_data() que salva todos os dados de exemplo em uma pasta de sua escolha em seu computador.
Para instalar nosso pacote R epirhandbook, execute o seguinte código. Este pacote não está no CRAN, então use a função p_install_gh() para instalá-lo. A entrada faz referência à nossa organização Github (“appliedepi”) e o pacote epirhandbook.
# instale a última versão do pacote do livro do Epi R
pacman::p_install_gh("appliedepi/epirhandbook")Agora, carregue o pacote para uso em sua sessão R atual:
# carregue o pacote para uso
pacman::p_load(epirhandbook)A seguir, use a função do pacote get_data() para baixar os dados de exemplo para o seu computador. Execute get_data("all") para obter todos os dados de exemplo ou forneça um nome de arquivo específico e extensão entre aspas para recuperar apenas um arquivo.
Os dados já foram baixados com o pacote e simplesmente precisam ser transferidos para uma pasta em seu computador. Uma janela pop-up aparecerá, permitindo que você selecione um local para salvar a pasta. Sugerimos que você crie uma nova pasta de “dados”, pois há cerca de 30 arquivos (incluindo dados de exemplo e saídas de exemplo).
# baixe todos os dados de exemplo em uma pasta em seu computador
get_data("all")
# baixe apenas os dados de exemplo da lista de linha em uma pasta em seu computador
get_data(file = "linelist_cleaned.rds")# baixe um arquivo específico em uma pasta em seu computador
get_data("linelist_cleaned.rds")Depois de usar get_data() para salvar um arquivo em seu computador, você ainda precisará importá-lo para R. Consulte a página Importar e exportar para obter detalhes.
Se desejar, você pode revisar todos os dados usados neste livro na pasta “data” de nosso repositório Github.
Baixe um por um
Esta opção envolve o download dos dados arquivo por arquivo de nosso repositório Github por meio de um link ou de um comando R específico para o arquivo. Alguns tipos de arquivo permitem um botão de download, enquanto outros podem ser baixados por meio de um comando R.
“Linelist” de casos
Este é um surto fictício de Ebola, expandido pela equipe do livro a partir do conjunto de dados de prática ebola_sim no pacote outbreaks.
- Clique para baixar a linelist “bruta” (.xlsx) . A linelist do caso “bruta” é uma planilha do Excel com dados confusos. Use-o para acompanhar a página Limpeza de dados e funções principais.
Se você quiser acompanhar, clique para baixar o linelist “limpo” (clean) (as .rds file). Use este arquivo para todas as outras páginas deste livro que usam a lista de linha. Um arquivo .rds é um tipo de arquivo específico de R que preserva classes de coluna. Isso garante que você terá apenas uma limpeza mínima para fazer após importar os dados para R.
Outros arquivos relacionados:
Se você quiser acompanhar, clique para baixar o linelist “limpo” (clean) (as .rds file).
- Parte da página de limpeza usa um “dicionário de limpeza” (arquivo .csv). Você pode carregá-lo diretamente no R executando os seguintes comandos:
pacman::p_load(rio) # instalar / carregar o pacote rio
# importe o arquivo diretamente do Github
cleaning_dict <- import("https://github.com/appliedepi/epirhandbook_eng/raw/master/data/case_linelists/cleaning_dict.csv")Dados de contagem de malária
Esses dados são contagens fictícias de casos de malária por faixa etária, serviço e dia. Um arquivo .rds é um tipo de arquivo específico de R que preserva classes de coluna. Isso garante que você terá apenas uma limpeza mínima para fazer após importar os dados para R.
Clique para fazer o download os dados de contagem de malária (arquivo .rds)
Dados em escala Likert
Estes são dados fictícios de uma pesquisa no estilo Likert, usados na página Pirâmides demográficas e escalas Likert. Você pode carregar esses dados diretamente no R executando os seguintes comandos:
pacman::p_load(rio) # instalar / carregar o pacote rio
# importe o arquivo diretamente do Github
likert_data <- import("https://raw.githubusercontent.com/appliedepi/epirhandbook_eng/master/data/likert_data.csv")Painéis com flexdashboard
Abaixo estão os links para o arquivo associado à página em Painéis (Dashboards) com R Markdown:
Rastreamento de contato
A página Rastreamento de contato demonstra a análise dos dados de rastreamento de contato, usando dados de exemplo de [Go.Data] (https://github.com/WorldHealthOrganization/godata/tree/master/analytics/r-reporting). Os dados usados na página podem ser baixados como arquivos .rds clicando nos seguintes links:
Clique para fazer o download os dados de investigação do caso (arquivo .rds)
Clique para fazer o download os dados de registro do contato (arquivo .rds)
Clique para fazer o download os dados de acompanhamento do contato (arquivo .rds)
NOTA: Dados de rastreamento de contato estruturado de outro software (por exemplo, KoBo, DHIS2 Tracker, CommCare) podem parecer diferentes. Se desejar contribuir com dados de amostra ou conteúdo alternativo para esta página, entre em contato.
DICA: Se você estiver implantando Go.Data e quiser se conectar à API da sua instância, consulte a página Importar e exportar (seção API) e a Go.Data Community of Practice.
Sobre o GIS
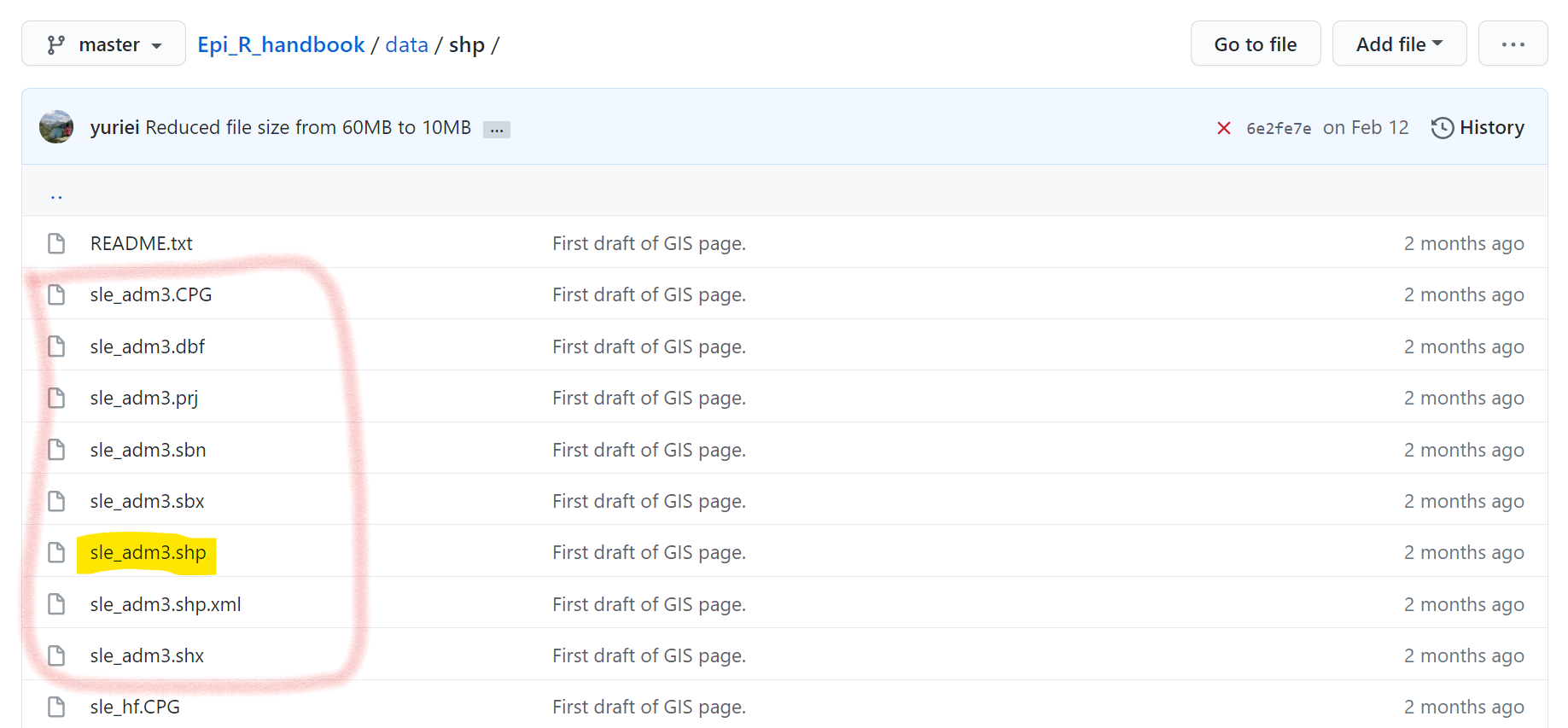
Os shapefiles têm muitos arquivos de subcomponentes, cada um com uma extensão de arquivo diferente. Um arquivo terá a extensão “.shp”, mas outros podem ter “.dbf”, “.prj”, etc.
A página GIS básico fornece links para o site Humanitarian Data Exchange onde você pode baixar os shapefiles diretamente como arquivos compactados.
Por exemplo, os dados dos pontos das unidades de saúde podem ser baixados aqui. Download “hotosm_sierra_leone_health_facilities_points_shp.zip”. Depois de salvar em seu computador, “descompacte” a pasta. Você verá vários arquivos com extensões diferentes (por exemplo, “.shp”, “.prj”, “.shx”) - todos eles devem ser salvos na mesma pasta em seu computador. Então, para importar para o R, forneça o caminho do arquivo e o nome do arquivo “.shp” para st_read() do pacote sf (conforme descrito na página Introdução ao GIS).
Se você seguir a Opção 1 para baixar todos os dados de exemplo (por meio de nosso pacote R epirhandbook), todos os shapefiles serão incluídos.
Alternativamente, você pode baixar os shapefiles da pasta “data” do R Handbook Github (veja a subpasta “gis”). No entanto, esteja ciente de que você precisará baixar cada subarquivo individualmente para o seu computador. No Github, clique em cada arquivo individualmente e baixe-os clicando no botão “Baixar”. Abaixo, você pode ver como o arquivo de forma “sle_adm3” consiste em muitos arquivos - cada um dos quais precisaria ser baixado do Github.
2.2.0.1 Árvores filogenéticas
Veja a página sobre Árvores filogenéticas. Arquivo Newick da árvore filogenética construída a partir do sequenciamento do genoma completo de 299 amostras de Shigella sonnei e dados de amostra correspondentes (convertidos em um arquivo de texto). As amostras belgas e os dados resultantes são gentilmente fornecidos pelo NRC belga para Salmonella e Shigella no âmbito de um projeto conduzido por um bolsista ECDC EUPHEM, e também serão publicados em um manuscrito. Os dados internacionais estão disponíveis abertamente em bases de dados públicas (NCBI) e foram publicados previamente.
- Para baixar o arquivo da árvore filogenética “Shigella_tree.txt”, clique com o botão direito neste link (Cmd + clique para Mac) e selecione “Salvar link como”.
- Para baixar o “sample_data_Shigella_tree.csv” com informações adicionais sobre cada amostra, clique com o botão direito neste link (Cmd + clique para Mac) e selecione “Salvar link como”.
- Para ver a nova árvore de subconjunto criada, clique com o botão direito neste link (Cmd + clique para Mac) e selecione “Salvar link como”. O arquivo .txt será baixado para o seu computador.
Você pode então importar os arquivos .txt com read.tree() do pacote ape, conforme explicado na página.
ape::read.tree("Shigella_tree.txt")Padronização
Consulte a página sobre Taxas padronizadas. Você pode carregar os dados diretamente de nosso repositório Github na Internet em sua sessão R com os seguintes comandos:
# instalar / carregar o pacote rio
pacman::p_load(rio)
)
# País A
)
# importar dados demográficos para o país A diretamente do Github
A_demo <- import("https://github.com/appliedepi/epirhandbook_eng/raw/master/data/standardization/country_demographics.csv")
# importação de mortes para o país A diretamente do Github
A_deaths <- import("https://github.com/appliedepi/epirhandbook_eng/raw/master/data/standardization/deaths_countryA.csv")
)
# País B
)
# importar dados demográficos para o país B diretamente do Github
B_demo <- import("https://github.com/appliedepi/epirhandbook_eng/raw/master/data/standardization/country_demographics_2.csv")
# importação de mortes para o país B diretamente do Github
B_deaths <- import("https://github.com/appliedepi/epirhandbook_eng/raw/master/data/standardization/deaths_countryB.csv")
)
# População Referência
)
# importar dados demográficos para o país B diretamente do Github
standard_pop_data <- import("https://github.com/appliedepi/epirhandbook_eng/raw/master/data/standardization/world_standard_population_by_sex.csv")Séries temporais e detecção de surto
Consulte a página em Séries temporais e detecção de surto. Usamos casos de Campylobacter relatados na Alemanha 2002-2011, conforme disponível no pacote surveillance R. (nb. este conjunto de dados foi adaptado do original, em que 3 meses de dados foram excluídos do final de 2011 para fins de demonstração).
Clique para fazer o download Campylobacter na Alemanha (.xlsx)
Também usamos dados climáticos da Alemanha 2002-2011 (temperatura em graus Celsius e queda de chuva em milímetros). Eles foram baixados do conjunto de dados de reanálise do satélite Copernicus da UE usando o pacote ecmwfr. Você precisará baixar tudo isso e importá-los com stars::read_stars() conforme explicado na página da série temporal.
Clique para fazer o download Alemanha meteorologia 2002 (arquivo .nc)
Clique para fazer o download Alemanha meteorologia 2003 (arquivo .nc)
Clique para fazer o download Alemanha meteorologia 2004 (arquivo .nc)
Clique para fazer o download Clima Alemanha 2005 (arquivo .nc)
Clique para fazer o download Clima Alemanha 2006 (arquivo .nc)
Clique para fazer o download Clima Alemanha 2007 (arquivo .nc)
Clique para fazer o download Clima Alemanha 2008 (arquivo .nc)
Clique para fazer o download Clima Alemanha 2009 (arquivo .nc)
Clique para fazer o download Clima Alemanha 2010 (arquivo .nc)
Clique para fazer o download Clima Alemanha 2011 (arquivo .nc)
Análise da pesquisa
Para a página análise da pesquisa, usamos dados fictícios de pesquisa de mortalidade baseados em modelos de pesquisa OCA do MSF. Esses dados fictícios foram gerados como parte do projeto “R4Epis”.
Clique para fazer o download Dados de pesquisa fictícios (.xlsx)
Clique para fazer o download Dicionário fictício de dados de pesquisa (.xlsx)
Clique para fazer o download Dados fictícios da população de pesquisas (.xlsx)
Shiny
A página em Painéis com Shiny demonstra a construção de um aplicativo simples para exibir dados da malária.
Para baixar os arquivos R que produzem o aplicativo Shiny:
Você pode clique aqui para baixar o arquivo facility_count_data.rds que contém dados de malária para o aplicativo Shiny. Observe que pode ser necessário armazená-lo em uma pasta “data” para que os caminhos de arquivo here() funcionem corretamente.
Você pode clique aqui para baixar o arquivo global.R que deve ser executado antes da abertura do aplicativo, conforme explicado na página.
Você pode clique aqui para baixar o arquivo plot_epicurve.R que é fornecido pela global.R. Observe que pode ser necessário armazená-lo em uma pasta “funcs” para que os caminhos de arquivo here() funcionem corretamente.