36 Análise de Combinações
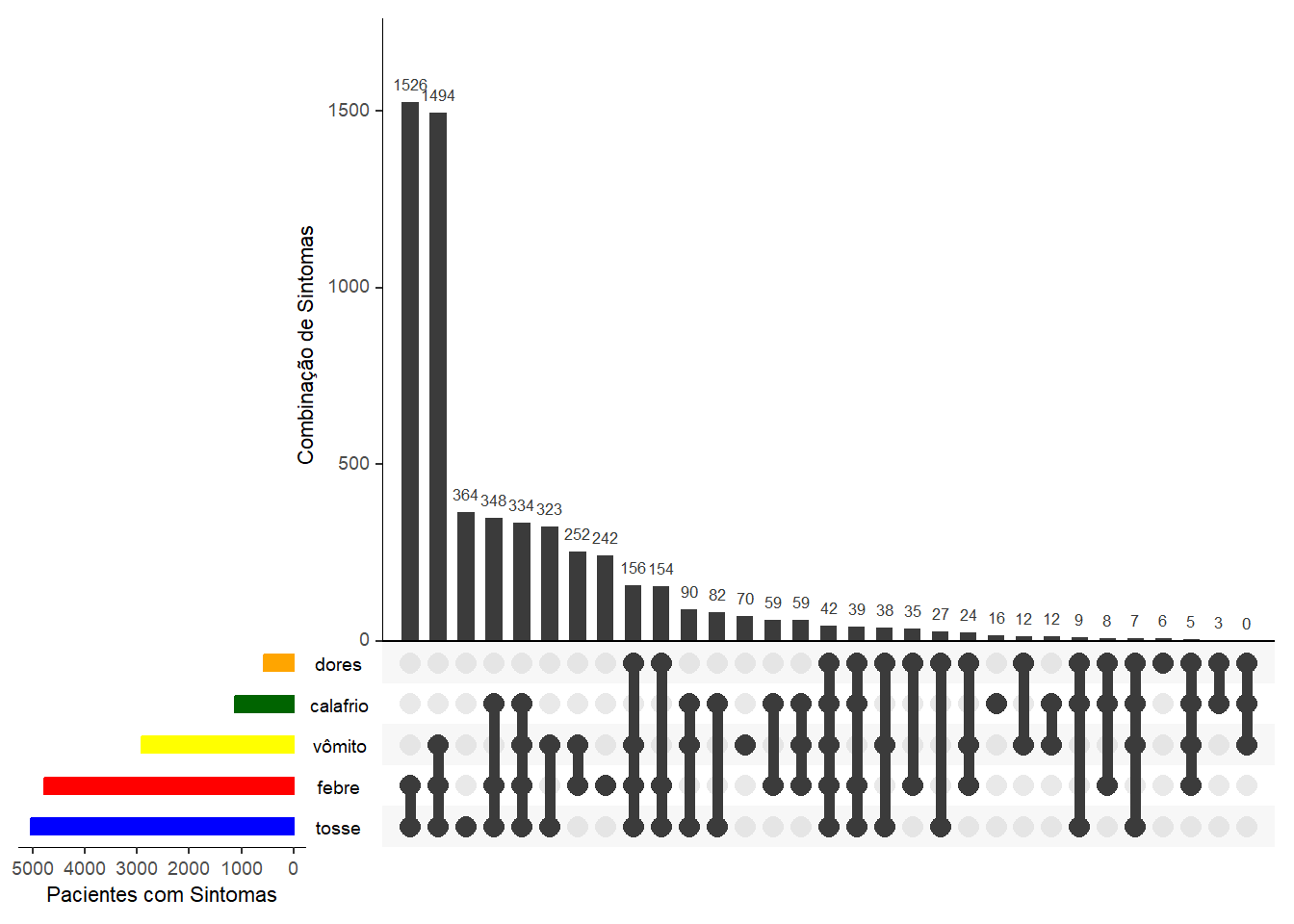
Essa análise gera um gráfico de frequência de diferentes combinações de valores e respostas. Nesse exemplo, geramos o gráfico da frequência casos exibiram várias combinações de sintomas.
Esse tipo de análise também é chamada de:
- “Análise de respostas múltiplas” (“Multiple response analysis”)
- “Análise de Conjuntos” (“Sets analysis”)
- “Análise de Combinações” (“Combinations analysis”)
No gráfico de exemplo acima são mostrados cinco sintomas. Abaixo de cada barra vertical há uma linha e pontos indicando a combinação de sintomas refletidos pela barra de cima. À esquerda, as barras horizontais refletem a frequência de cada sintoma individualmente.
O primeiro método que mostraremos utiliza o pacote ggupset, e o segundo utiliza o pacote UpSetR.
36.1 Preparação
Carregue os pacotes R
O código abaixo realiza o carregamento dos pacotes necessários para a análise dos dados. Neste manual, enfatizamos o uso da função p_load(), do pacman, que instala os pacotes, caso não estejam instalados, e os carrega no R para utilização. Também é possível carregar pacotes instalados utilizando a função library(), do R base. Para mais informações sobre os pacotes do R, veja a página Introdução ao R.
pacman::p_load(
tidyverse, # manupulação de dados e visualização
UpSetR, # pacote especial para gráficos de combinção
ggupset) # pacote especial para gráficos de combinção Importe os dados
Para iniciar, importaremos a linelist dos casos de uma epidemia simulada do Ebola. Se você quiser acompanhar, clique para baixar a linelist “limpa” (como um arquivo .rds). Importe os dados com a função import() do pacote rio (a função suporta vários tipos de arquivo como .xlsx, .csv, .rds - cheque a página Importar e exportar para mais detalhes).
# importe a linelit de casos
linelist_sym <- import("linelist_cleaned.rds")Essa linelist inclui cinco variáveis do tipo “sim/não” (“sim/no”) a respeito dos sintomas relatados. Nós vamos precisar transformar essas variáveis um pouco para poder utilizar o pacote ggupset para fazer nosso gráfico. Confira os dados (role para a direita para ver as variáveis dos sintomas).
Reformate os valores
Para estarmos alinhados com o formato esperado pelo pacote ggupset vamos converter os “sim” e “não” para o real nome do sintoma utilizando a função case_when() do pacote dplyr. Caso seja “não”, definiremos o valor como vazio, então os valores serão NA ou o nome do sintoma.
# criar uma coluna com todos os sintomas separados por ponto-vírgula
linelist_sym_1 <- linelist_sym %>%
rename(febre=fever, # só traduzindo
calafrio=chills,
tosse=cough,
dores=aches,
vômito = vomit) %>%
# converte os valores de "yes" e "no" no nome do sintoma
mutate(
febre = case_when(
febre == "yes" ~ "febre", #se o valor era "sim", o novo valor é "febre"
TRUE ~ NA_character_), # se o valor era diferente de "sim", o novo valor é NA
calafrio = case_when(
calafrio == "yes" ~ "calafrio",
TRUE ~ NA_character_),
tosse = case_when(
tosse == "yes" ~ "tosse",
TRUE ~ NA_character_),
dores = case_when(
dores == "yes" ~ "dores",
TRUE ~ NA_character_),
vômito = case_when(
vômito == "yes" ~ "vômito",
TRUE ~ NA_character_)
)Agora nós geramos duas colunas finais:
- Concatenando (unindo) todos os sintomas do paciente (uma coluna de caracteres)
- Convertendo a coluna acima para a classe list, para que ela possa ser passada ao pacote ggupset para fazer o gráfico.
Veja a página de Caracteres e strings para aprender mais sobre a função unite() do pacote stringr
linelist_sym_1 <- linelist_sym_1 %>%
unite(col = "all_symptoms",
c(febre, calafrio, tosse, dores, vômito),
sep = "; ",
remove = TRUE,
na.rm = TRUE) %>%
mutate(
# faz uma cópia de da coluna all_symptoms, mas na classe lista (que é requisito do ggupset() no próximo passo)
all_symptoms_list = as.list(strsplit(all_symptoms, "; "))
)Veja a nova tabela. Note as duas colunas na extremidade da direta - os valores combinados, e a lista
36.2 ggupset
Carregue o pacote
pacman::p_load(ggupset)Crie o gráfico. Vamos começar com um ggplot() e a função geom_bar(), mas depois vamos adicionar a função scale_x_upset() do pacote ggupset.
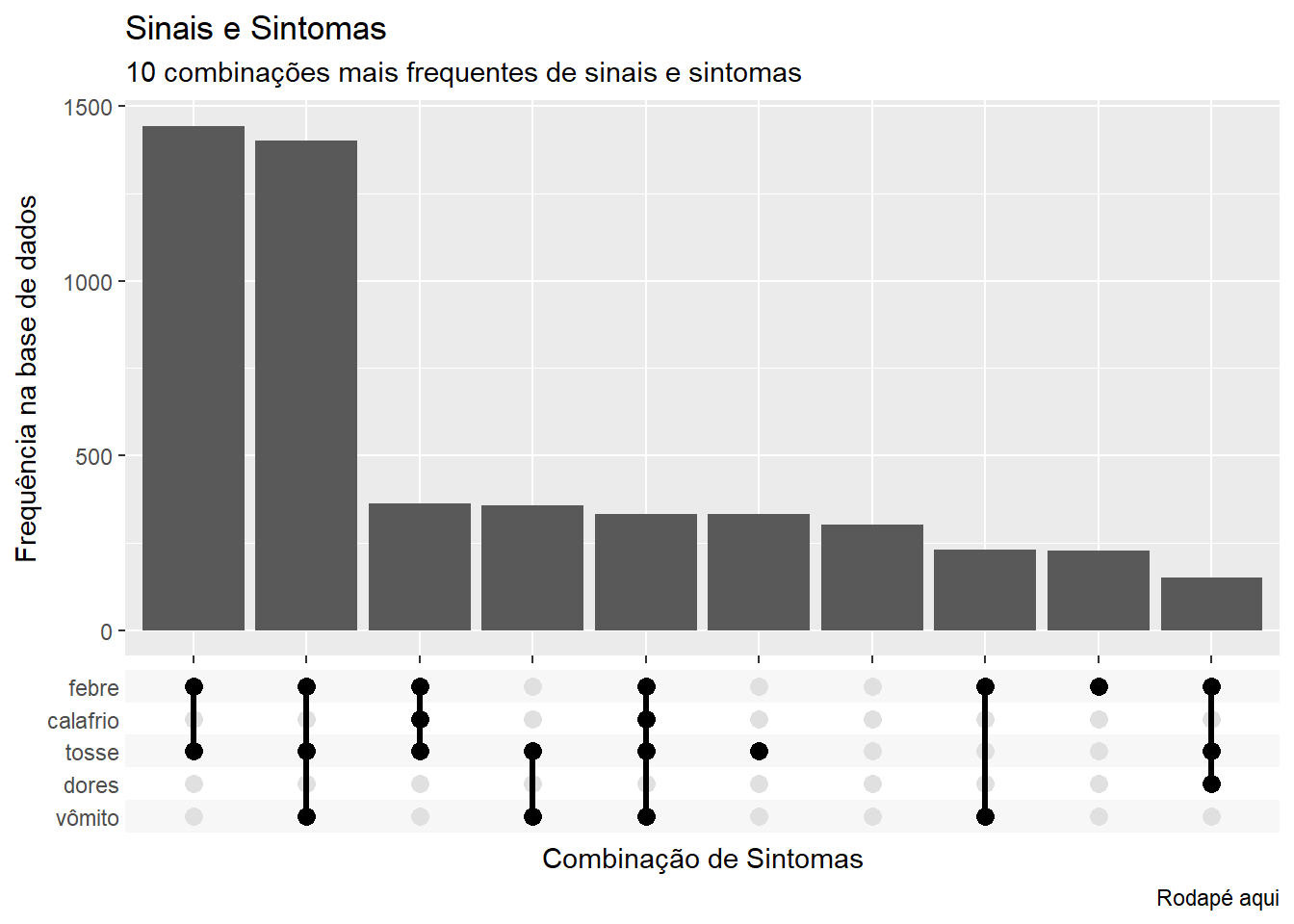
ggplot(
data = linelist_sym_1,
mapping = aes(x = all_symptoms_list)) +
geom_bar() +
scale_x_upset(
reverse = FALSE,
n_intersections = 10,
sets = c("febre", "calafrio", "tosse", "dores", "vômito"))+
labs(
title = "Sinais e Sintomas",
subtitle = "10 combinações mais frequentes de sinais e sintomas",
caption = "Rodapé aqui",
x = "Combinação de Sintomas",
y = "Frequência na base de dados")Mais informações sobre ggupset podem ser vistas online ou offline na documentação do pacote na aba de Ajuda do seu RStudio após digitar ?ggupset no console.
36.3 UpSetR
O pacote UpSetR permite mais personalizações do gráfico, mas pode ser mais difícil de executar.
Carregue o pacote
pacman::p_load(UpSetR)Limpando os dados
Precisamos converter os valores dos sintomas da linelist para 1 / 0.
# Make using upSetR
linelist_sym_2 <- linelist_sym %>%
rename(febre=fever, # só traduzindo
calafrio=chills,
tosse=cough,
dores=aches,
vômito = vomit) %>% # só traduzindo
# converte os valores de "sim" e "não" nos nomes dos sintomas
mutate(
febre = case_when(
febre == "yes" ~ 1, # se o valor antigo era "yes", então o novo valor é 1
TRUE ~ 0), # se o valor era qualquer coisa diferente de "yes", o novo valor é 0
calafrio = case_when(
calafrio == "yes" ~ 1,
TRUE ~ 0),
tosse = case_when(
tosse == "yes" ~ 1,
TRUE ~ 0),
dores = case_when(
dores == "yes" ~ 1,
TRUE ~ 0),
vômito = case_when(
vômito == "yes" ~ 1,
TRUE ~ 0)
)Agora vamos fazer o gráfico utilizando a função upset() - utilizando apenas as colunas de sintomas. Você deve definir quais “conjuntos” serão comparados (passe os nomes das colunas de sintomas). Ou então, utilize nsets = e order.by = "freq" para mostrar apenas as maiores X combinações.
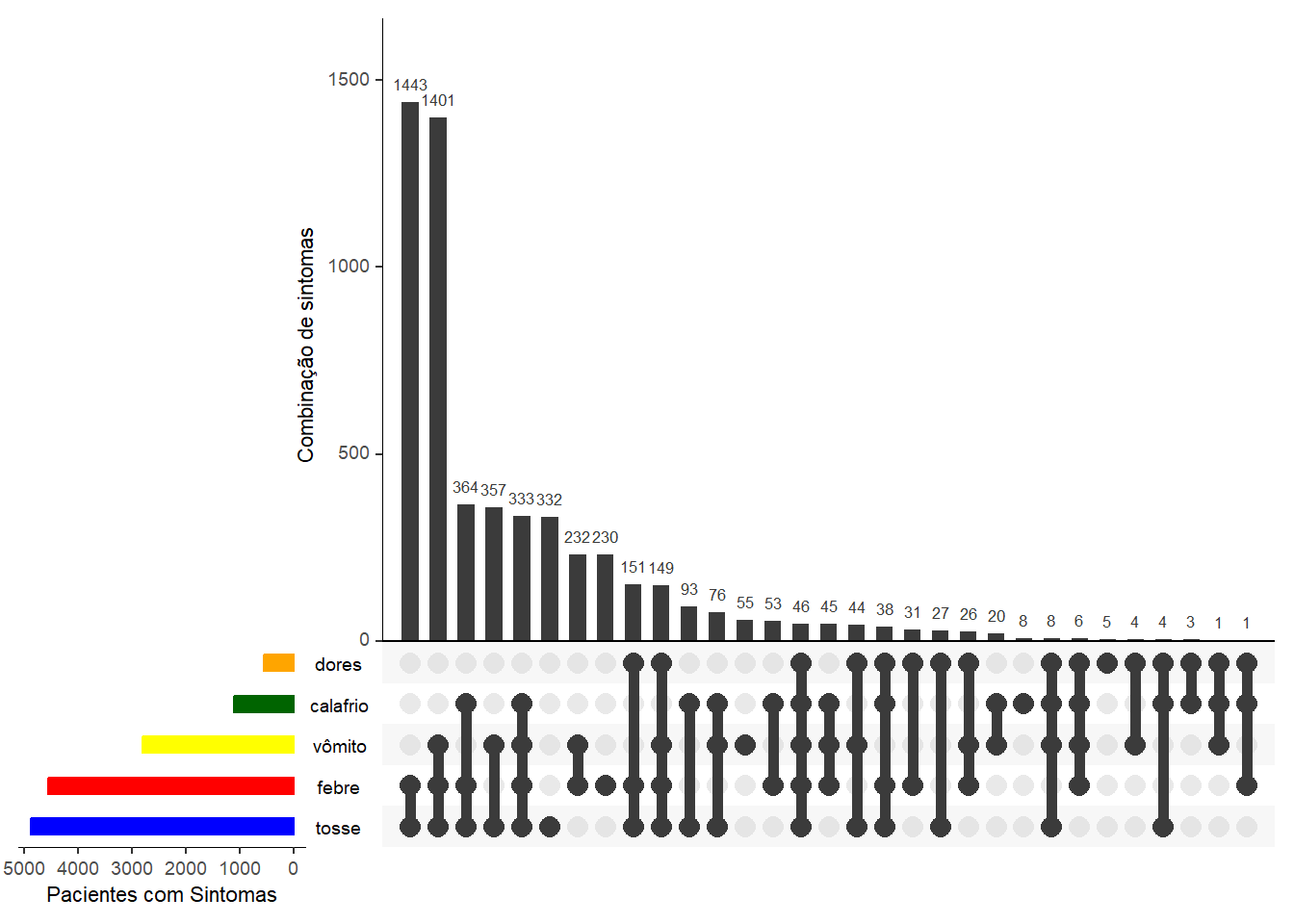
# Make the plot
UpSetR::upset(
select(linelist_sym_2, febre, calafrio, tosse, dores, vômito),
sets = c("febre", "calafrio", "tosse", "dores", "vômito"),
order.by = "freq",
sets.bar.color = c("blue", "red", "yellow", "darkgreen", "orange"), # cores opcionais
empty.intersections = "on",
# nsets = 3,
number.angles = 0,
point.size = 3.5,
line.size = 2,
mainbar.y.label = "Combinação de sintomas",
sets.x.label = "Pacientes com Sintomas")36.4 Recursos
Uma versão em Shiny App - você pode fazer upload de seus próprios dados