12 Verilerin pivotlanması

Verileri yönetirken, pivotlama aşağıdaki iki süreçten birini tarif eder:
- Daha kapsamlı bir tablonun verilerini özetleyen istatistik tabloları olan pivot tabloların oluşturulması
- Bir tablonun uzun formattan geniş formata dönüştürülmesi veya tam tersi.
Bu sayfada, ikinci tanıma odaklanacağız. İlk tanım, veri analizinde çok önemli bir adımdır ancak [Verileri gruplandırma] ve Tanımlayıcı tablolar sayfalarında ele alınmıştır.
Bu sayfada veri biçimleri tartışılmaktadır. Her değişkenin kendi sütununa, her gözlemin kendi satırına ve her değerin kendi hücresine sahip olduğu “düzenli-tidy veri” felsefesi akılda tutulmalıdır. Bu konu hakkında daha fazla bilgiyi Veri Bilimi için R çevrimiçi bölümünde bulabilirsiniz.
12.1 Hazırlık
Paketlerin yüklenmesi
Bu kod bloğu, analizler için gerekli olan paketlerin yüklenmesini gösterir. Bu el kitabında, gereken durumlarda paketi kuran ve kullanım için yükleyen pacman’ın p_load() fonksiyonunun kullanımı vurgulanmıştır. Ayrıca, R tabanından library() ile kurulu paketleri yükleyebilirsiniz. R paketleri hakkında daha fazla bilgi için [R temelleri] sayfasına bakın.
pacman::p_load(
rio, # Dosyanın içe aktarımı
here, # Dosyanın konumu
tidyverse) # veri yönetimi + ggplot2 grafik paketiSıtma hastalığı için sayım verisi
Bu sayfada, kuruluş ve yaş grubuna göre günlük sıtma vakalarını içeren kurgusal bir veri setini inceleyeceğiz. Adımları takip etmek isterseniz, veri setini indirmek için buraya tıklayın (.rds dosyası formatında). Verileri rio paketinden import() fonksiyonuyla içe aktarın (.xlsx, .csv, .rds gibi birçok dosya türünü işler - ayrıntılar için İçe Aktarma ve Dışa Aktarma sayfasına bakın).
# Verinin içeri aktarımı
count_data <- import("malaria_facility_count_data.rds")İlk 50 satır aşağıda gösterilmiştir.
Satır listesi verisi
Bu sayfanın sonraki bölümünde, simüle edilmiş bir Ebola salgını vakalarını içeren veri setini de kullanacağız. Devam etmek istiyorsanız, “temiz” satır listesini (.rds dosyası olarak) indirmek için tıklayın. Verilerinizi rio paketinden import() işleviyle içe aktarın (.xlsx, .rds, .csv gibi birçok dosya türünü kabul eder - ayrıntılar için İçe Aktarma ve Dışa Aktarma sayfasına bakın).
# Verinin içeri aktarımı
linelist <- import("linelist_cleaned.xlsx")12.2 Genişten uzuna
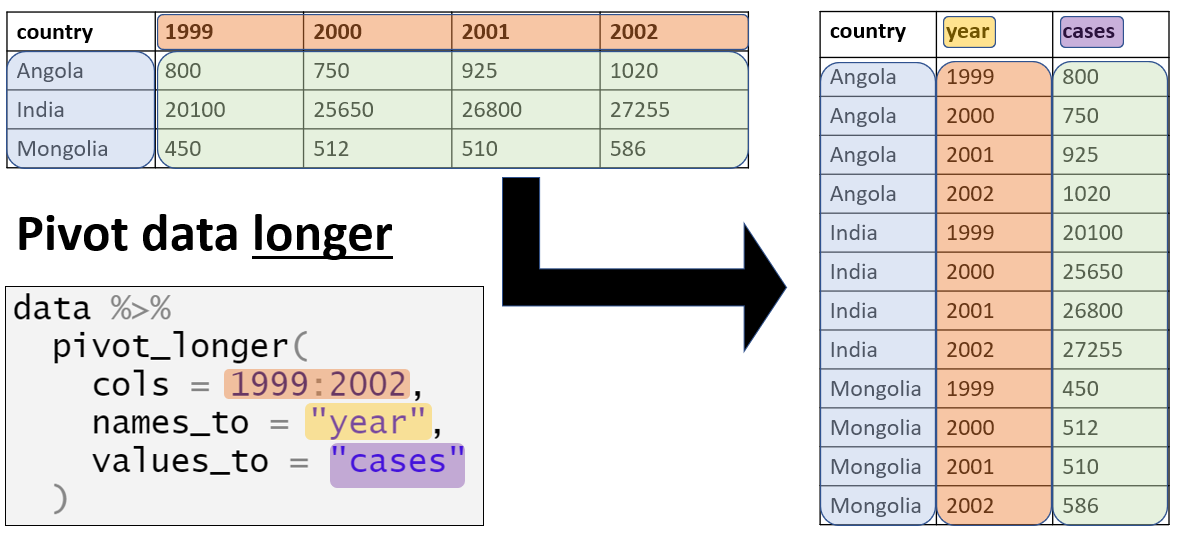
“Geniş” format
Veriler genellikle “geniş” bir biçimde girilir ve saklanır - burada bir gözlemin (kişinin) özellikleri veya yanıtları tek bir satırda depolanır. Bu sunum için faydalı olsa da, bazı analiz türleri için ideal değildir.
Örnek olarak yukarıdaki Hazırlık bölümünde içe aktarılan count_data veri setini ele alalım. Her satırın bir “tesis-gününü” temsil ettiğini görebilirsiniz. Gerçek vaka sayıları (en sağdaki sütunlar), belirli bir tesis günündeki farklı yaş grubuna ait bilgilerin ayrı sütunlar halinde ancak tek bir satırda saklanacağı şekilde “geniş” bir formatta bulunur.
Bu veri tabanındaki ScriptEach gözlemi, belirli bir tarihteki 65 tesisten birindeki sıtma sayımlarını gösterir; bu sayı, count_data$data_date %>% min() ile count_data$data_date %>% max() arasında değişir. Bu tesisler bir Province (il) (North) ve dört District (ilçe)de (Spring, Bolo, Dingo ve Barnard) bulunmaktadır. Veri seti, genel sıtma sayılarının yanı sıra üç yaş grubunun her birinde - <4 yaş, 5-14 yaş ve 15 yaş ve üzeri - yaşa özel sayıları da içermektedir.
Bunun gibi “geniş” veriler “düzenli veri” standartlarına uymaz, çünkü sütun başlıkları aslında “değişkenleri” temsil etmez - bunlar hipotetik bir “yaş grubu” değişkenini temsil eder.
Bu format, bilgileri bir tabloda sunmak veya vaka raporu formlarına veri girmek (örn. Excel’de) için faydalı olabilir. Ancak, analiz aşamasında, bu veriler tipik olarak “düzenli veri” standartlarıyla daha uyumlu “daha uzun” bir formata dönüştürülmelidir. Özellikle R paketi ggplot2, veriler “uzun” formatta olduğunda en iyi sonuçları verir.
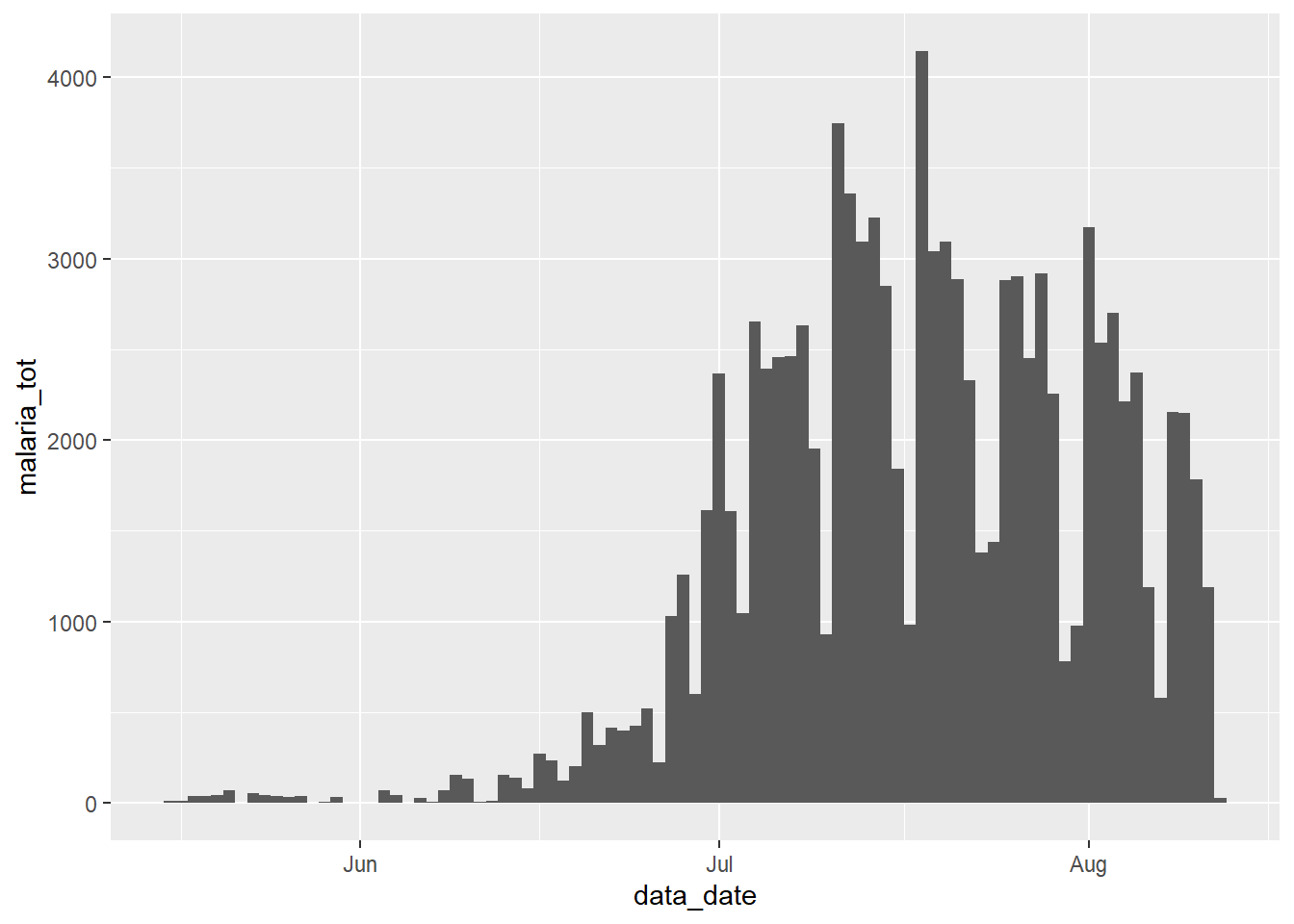
Toplam sıtma sayılarının zamana göre görselleştirilmesinde, mevcut formattaki verilerle herhangi bir zorluk yaşanmaz:
Ancak, her yaş grubunun bu toplam sayıya göreli katkılarını göstermek istersek ne olur? Bu durumda, ilgilenilen değişkenin (yaş grubu), veri kümesinde ggplot2’nin “haritalama estetiği” aes() argümanına aktarılabilecek tek bir sütunda bulunması gerekir.
pivot_longer()
tidyr fonksiyonu pivot_longer(), verileri “daha uzun” formata getirir. tidyr, R paketlerinin tidyverse kümesinin bir parçasıdır.
Bu fonksiyon, dönüştürülecek bir dizi sütunu kabul eder (cols = argümanı ile belirtilir). Bu nedenle, bir veri tabanının yalnızca bir kısmıyla çalışabilir. Bu fonksiyonla yalnızca vaka sayısı sütunlarını döndürmek istediğimizden, kullandığımız veritabanı için yararlıdı
Bu süreçte, biri kategorileri (eski sütun adları) ve diğeri de karşılık gelen değerleri (ör. vaka sayıları) içeren iki “yeni” sütun elde edeceksiniz. Bu yeni sütunlar için varsayılan adları kabul edebilir veya kendi adlarınızı sırasıyla names_to = ve values_to = argümanlarıyla belirtebilirsiniz.
Pivot_longer()’ı çalışırken görelim…
Standard pivoting
“Geniş” verileri “uzun” bir biçime dönüştürmek için tidyr’in pivot_longer() fonksiyonunu kullanmak istiyoruz. Spesifik olarak, sıtma sayılarına ilişkin verileri içeren dört sayısal sütunu iki yeni sütuna dönüştürmek istiyoruz. İki yeni sütundan biri yaş gruplarını ve diğeri yaş gruplarına karşılık gelen değerleri içerecektir.
df_long <- count_data %>%
pivot_longer(
cols = c(`malaria_rdt_0-4`, `malaria_rdt_5-14`, `malaria_rdt_15`, `malaria_tot`)
)
df_longYeni oluşturulan veri çerçevesinin (df_long) daha fazla satırı olduğuna dikkat edin (12.152’ye karşı 3.038); böylece veri çerçevesi daha uzun bir şekle sahip olur. Aslında, eski versiyonuna göre tam olarak dört kat daha uzundur, çünkü orijinal veri kümesindeki her satır şimdi df_long’da dört satırı temsil etmektedir (her bir sıtma sayımı gözlemi yaş grubu için bir tane (<4y, 5-14y, 15y+ ve toplam)).
Daha önce dört sütunda (malaria_ ön ekiyle başlayanlar) depolanan veriler şimdi iki sütunda depolandığından, daha uzun olmanın yanı sıra, yeni veri kümesi daha az sütuna (8’e karşı 10) sahiptir.
Bu dört sütunun adlarının tümü malaria_ önekiyle başladığından, aynı sonucu elde etmek için kullanışlı “tidyselect” start_with() işlevini kullanabilirdik (bu yardımcı işlevlerin daha fazlası için [Verileri temizleme ve temel işlevler] sayfasına bakın).
# tidyselect yardımcı işleviyle sütun sağlayın
count_data %>%
pivot_longer(
cols = starts_with("malaria_")
)## # A tibble: 12,152 × 8
## location_name data_date submitted_date Province District newid name value
## <chr> <date> <date> <chr> <chr> <int> <chr> <int>
## 1 Facility 1 2020-08-11 2020-08-12 North Spring 1 malaria_rdt_… 11
## 2 Facility 1 2020-08-11 2020-08-12 North Spring 1 malaria_rdt_… 12
## 3 Facility 1 2020-08-11 2020-08-12 North Spring 1 malaria_rdt_… 23
## 4 Facility 1 2020-08-11 2020-08-12 North Spring 1 malaria_tot 46
## 5 Facility 2 2020-08-11 2020-08-12 North Bolo 2 malaria_rdt_… 11
## 6 Facility 2 2020-08-11 2020-08-12 North Bolo 2 malaria_rdt_… 10
## 7 Facility 2 2020-08-11 2020-08-12 North Bolo 2 malaria_rdt_… 5
## 8 Facility 2 2020-08-11 2020-08-12 North Bolo 2 malaria_tot 26
## 9 Facility 3 2020-08-11 2020-08-12 North Dingo 3 malaria_rdt_… 8
## 10 Facility 3 2020-08-11 2020-08-12 North Dingo 3 malaria_rdt_… 5
## # … with 12,142 more rowsya da pozisyona göre:
# pozisyona göre sütun sağlamak
count_data %>%
pivot_longer(
cols = 6:9
)veya adlandırılmış aralığa göre:
# ardışık sütun aralıkları sağlama
count_data %>%
pivot_longer(
cols = `malaria_rdt_0-4`:malaria_tot
)Bu iki yeni sütuna varsayılan ad ve değerler verilir, ancak istediğimiz adları verebilmek için bu varsayılan adları geçersiz kılabiliriz; names_to ve values_to argümanlarını kullanarak yeni sütunlarda hangi bilgilerin depolandığını hatırlayabiliriz. Age_group adlarını ve sayılarını kullanalım:
df_long <-
count_data %>%
pivot_longer(
cols = starts_with("malaria_"),
names_to = "age_group",
values_to = "counts"
)
df_long## # A tibble: 12,152 × 8
## location_name data_date submitted_date Province District newid age_group counts
## <chr> <date> <date> <chr> <chr> <int> <chr> <int>
## 1 Facility 1 2020-08-11 2020-08-12 North Spring 1 malaria_rdt… 11
## 2 Facility 1 2020-08-11 2020-08-12 North Spring 1 malaria_rdt… 12
## 3 Facility 1 2020-08-11 2020-08-12 North Spring 1 malaria_rdt… 23
## 4 Facility 1 2020-08-11 2020-08-12 North Spring 1 malaria_tot 46
## 5 Facility 2 2020-08-11 2020-08-12 North Bolo 2 malaria_rdt… 11
## 6 Facility 2 2020-08-11 2020-08-12 North Bolo 2 malaria_rdt… 10
## 7 Facility 2 2020-08-11 2020-08-12 North Bolo 2 malaria_rdt… 5
## 8 Facility 2 2020-08-11 2020-08-12 North Bolo 2 malaria_tot 26
## 9 Facility 3 2020-08-11 2020-08-12 North Dingo 3 malaria_rdt… 8
## 10 Facility 3 2020-08-11 2020-08-12 North Dingo 3 malaria_rdt… 5
## # … with 12,142 more rowsŞimdi bu yeni veri kümesini ggplot2’ye aktarabiliriz ve yeni sütunlardaki count y eksenine ve yeni age_group sütununu fill = argümanına (sütun iç kısmının rengi) atayabiliriz. Bu komut, çubuk grafikte yaş grubuna göre sıtma sayılarını gösterir:
ggplot(data = df_long) +
geom_col(
mapping = aes(x = data_date, y = counts, fill = age_group),
width = 1
)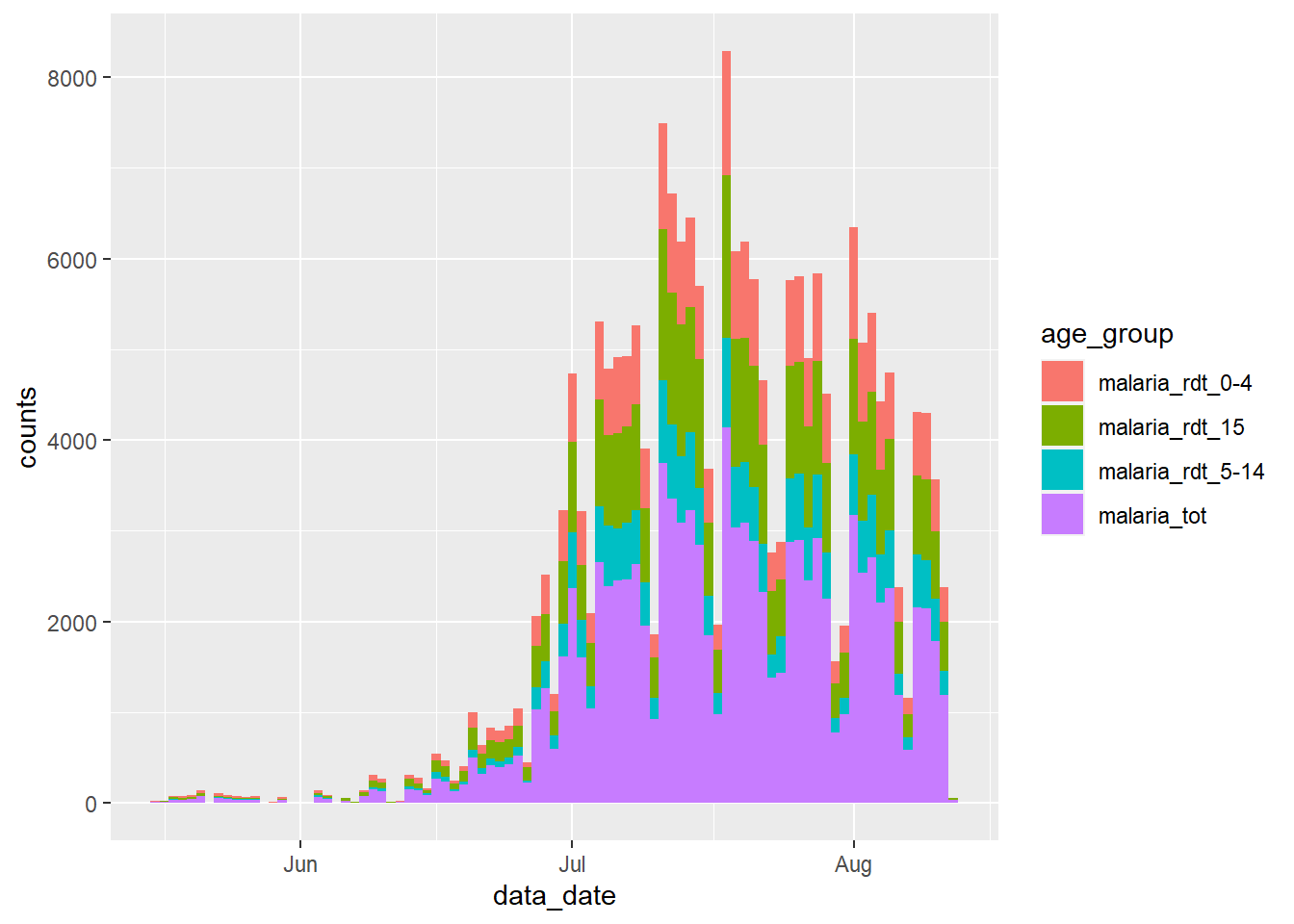
Bu yeni grafiği inceleyin ve daha önce oluşturduğumuz grafikle karşılaştırın – Sizce yanlış giden ne?
Sürveyans verilerini incelerken yaygın bir sorunla karşılaştık: Grafikteki her çubuğun boyu olması gerekenin iki katıdır, çünkü grafiğe malaria_tot sütunundaki toplam sayıları da dahil ettik.
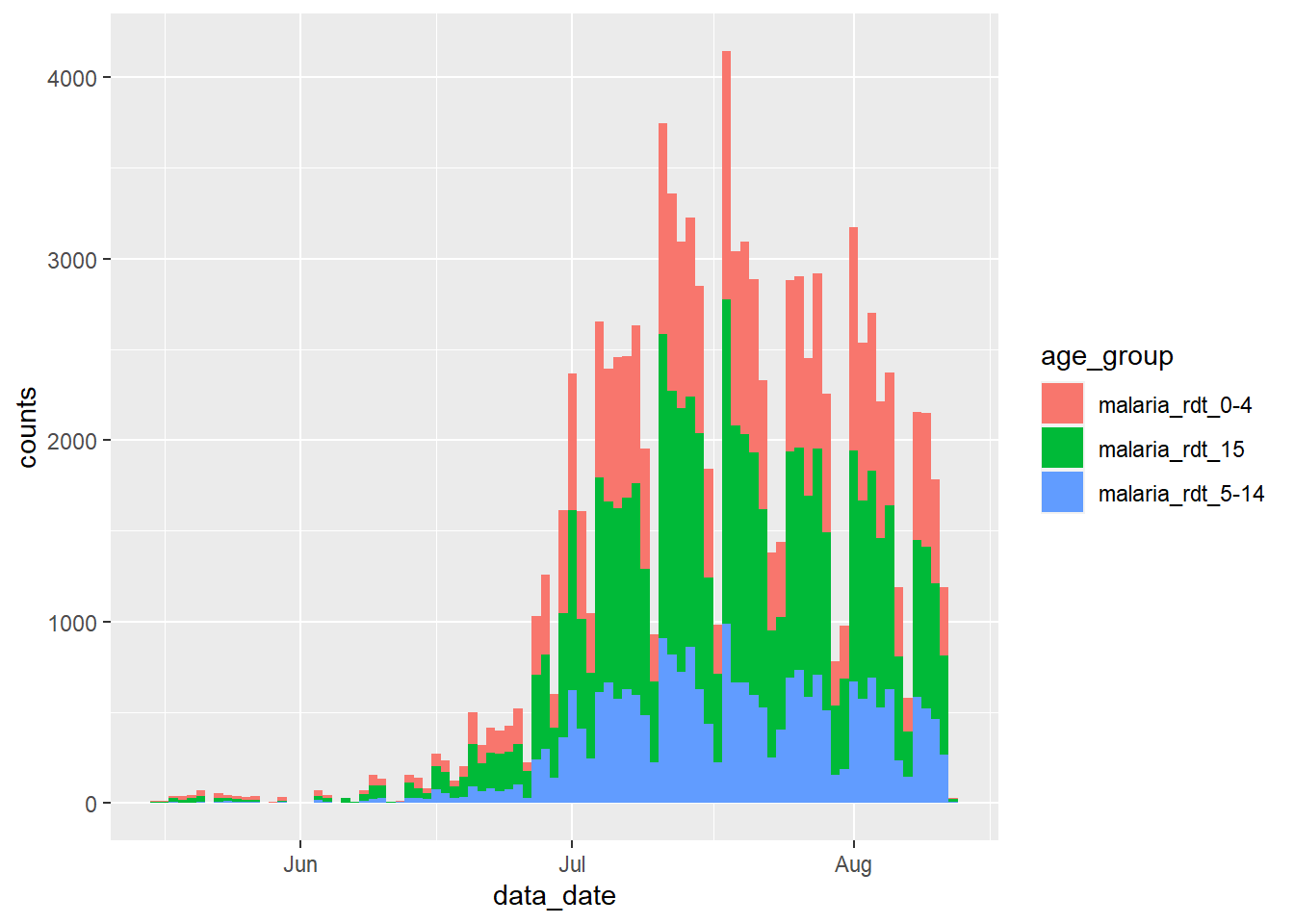
Bu sorunu birkaç şekilde halledebiliriz. ggplot()’a aktarmadan önce bu toplamları veri kümesinden filtreleyebiliriz:
df_long %>%
filter(age_group != "malaria_tot") %>%
ggplot() +
geom_col(
aes(x = data_date, y = counts, fill = age_group),
width = 1
)
Alternatif olarak, pivot_longer()’ı çalıştırdığımızda bu değişkeni hariç tutabilirdik, böylece sorun oluşturan bu değişkeni ayrı bir değişken olarak veri kümesinde tutabilirdik. Değerlerin yeni satırları doldurmak için nasıl “genişlediğini” inceleyin.
count_data %>%
pivot_longer(
cols = `malaria_rdt_0-4`:malaria_rdt_15, # does not include the totals column
names_to = "age_group",
values_to = "counts"
)## # A tibble: 9,114 × 9
## location_name data_date submitted_d…¹ Provi…² Distr…³ malar…⁴ newid age_g…⁵ counts
## <chr> <date> <date> <chr> <chr> <int> <int> <chr> <int>
## 1 Facility 1 2020-08-11 2020-08-12 North Spring 46 1 malari… 11
## 2 Facility 1 2020-08-11 2020-08-12 North Spring 46 1 malari… 12
## 3 Facility 1 2020-08-11 2020-08-12 North Spring 46 1 malari… 23
## 4 Facility 2 2020-08-11 2020-08-12 North Bolo 26 2 malari… 11
## 5 Facility 2 2020-08-11 2020-08-12 North Bolo 26 2 malari… 10
## 6 Facility 2 2020-08-11 2020-08-12 North Bolo 26 2 malari… 5
## 7 Facility 3 2020-08-11 2020-08-12 North Dingo 18 3 malari… 8
## 8 Facility 3 2020-08-11 2020-08-12 North Dingo 18 3 malari… 5
## 9 Facility 3 2020-08-11 2020-08-12 North Dingo 18 3 malari… 5
## 10 Facility 4 2020-08-11 2020-08-12 North Bolo 49 4 malari… 16
## # … with 9,104 more rows, and abbreviated variable names ¹submitted_date, ²Province,
## # ³District, ⁴malaria_tot, ⁵age_groupBirden çok sınıfa ait verinin pivotlanması
Yukarıdaki örnek, daha uzun şekilde pivotlamak istediğiniz tüm sütunların aynı sınıfta olduğu durumlarda (karakter, sayısal, mantıksal…) iyi çalışır.
Bununla birlikte, bir saha epidemiyoloğu olarak, bu konuda uzman olmayanlar tarafından hazırlanan ve standart dışı verilerle çalışacağınız birçok durum olacaktır - Hadley Wickham’ın Düzenli Veri ilkeleri üzerine ufuk açıcı makalesinde Tolstoy’a atıfta bulunarak belirttiği gibi “Düzenli veri kümelerinin hepsi birbirine benzer, ancak her dağınık veri kümesi kendi tarzında dağınıktır, aynı aileler gibi.”
Karşılaşacağınız özellikle yaygın bir sorun, farklı veri sınıfları içeren sütunları döndürme ihtiyacı olacaktır. Bu pivot işlemi, farklı veri türlerinin tek bir sütunda depolanmasına neden olur ve bu durum istenmeyen bir sonuçtur. Bunun yarattığı karışıklığı ortadan kaldırmak için çeşitli yaklaşımlar vardır. Ancak bu duruma düşmemek için pivot_longer() kullanımında uygulanabilecek önemli bir adım vardır.
A, B ve C öğelerinin her biri için farklı zamanlarda bir dizi gözlemin olduğu bir durumu ele alın. Bu öğeler örneğin farklı bireyler (örneğin 21 gün boyunca her gün bir Ebola vakasının temaslıları) veya hala çalışır durumda olduklarından emin olmak için yılda bir kez izlenen sağlık ocakları olabilir. Temaslı kişilerini izlemini ele alalım. Verilerin aşağıdaki gibi kaydedildiğini düşünelim:
Verinin durumu başlangıçta karmaşıktır. Her satır, bir öğe hakkında veri içerir, ancak zaman serilerinde zamandaki ilerleme ile sütunlarda sağa doğru ilerleme izlenmektedir. Ayrıca, sütun sınıfları sırayla tarih ve karakter değerleri almaktadır.
Bu araştırmacının karşılaştığı kötü bir örnek, 4 yıl boyunca her gün 8 yeni gözlem sütununun eklendiği kolera sürveyans verilerini içeriyordu. Bu verilerin depolandığı Excel dosyasını açmak, dizüstü bilgisayarda 10 dakikan fazla zaman almıştır.
Bu verilerle çalışmak için, veri çerçevesini uzun biçime dönüştürmemiz, ancak her öğeye ait tüm gözlemler için date ve character (durum) sütunu arasındaki ayrımı korumamız gerekir. Bunu yapmazsak, tek bir sütunda değişken türleri karışık şekilde bulunur (Veri yönetimi ve düzenli veriler söz konusu olduğunda gerçekten istenmeyen bir durumdur):
df %>%
pivot_longer(
cols = -id,
names_to = c("observation")
)## # A tibble: 18 × 3
## id observation value
## <chr> <chr> <chr>
## 1 A obs1_date 2021-04-23
## 2 A obs1_status Healthy
## 3 A obs2_date 2021-04-24
## 4 A obs2_status Healthy
## 5 A obs3_date 2021-04-25
## 6 A obs3_status Unwell
## 7 B obs1_date 2021-04-23
## 8 B obs1_status Healthy
## 9 B obs2_date 2021-04-24
## 10 B obs2_status Healthy
## 11 B obs3_date 2021-04-25
## 12 B obs3_status Healthy
## 13 C obs1_date 2021-04-23
## 14 C obs1_status Missing
## 15 C obs2_date 2021-04-24
## 16 C obs2_status Healthy
## 17 C obs3_date 2021-04-25
## 18 C obs3_status HealthyYukarıda, pivot komutu, tarihleri ve karakterleri tek bir değer sütununda birleştirdi. Bu durumda R, tüm sütunu sınıf karakterine dönüştür ve tarih özellikleri kaybolur.
Bu durumu önlemek için orijinal sütun adlarının sintaks yapısından yararlanabiliriz. Gözlem numarası, alt çizgi ve ardından “durum” veya “tarih” bilgisi içeren ortak bir adlandırma şeklini kullanmak faydalıdır. Pivot sonrası bu iki veri türünü ayrı sütunlarda tutmak için bu sintakstan yararlanabiliriz.
Bunu şu şekilde yapıyoruz:
- İkinci öğe (
".value") olmak üzere,names_to =argümanına bir karakter vektörü yazmalısınız. Bu özel terim, pivot sütunlarının adlarındaki bir karaktere göre nasıl bölüneceğini belirtir. - Ayrıca,
name_sep =argümanına “splitting-ayırma” karakterini de yazmalısınız. Bu durumda, ayırma karakteri alt çizgidir ( “_”)
Bu nedenle, yeni sütunların adlandırılması ve bölünmesi, mevcut değişken adlarındaki alt çizgiye göre gerçekleşir.
df_long <-
df %>%
pivot_longer(
cols = -id,
names_to = c("observation", ".value"),
names_sep = "_"
)
df_long## # A tibble: 9 × 4
## id observation date status
## <chr> <chr> <chr> <chr>
## 1 A obs1 2021-04-23 Healthy
## 2 A obs2 2021-04-24 Healthy
## 3 A obs3 2021-04-25 Unwell
## 4 B obs1 2021-04-23 Healthy
## 5 B obs2 2021-04-24 Healthy
## 6 B obs3 2021-04-25 Healthy
## 7 C obs1 2021-04-23 Missing
## 8 C obs2 2021-04-24 Healthy
## 9 C obs3 2021-04-25 HealthySon dokunuşlar:
Tarih sütununun şu anda character sınıfında olduğuna dikkat edin. Tarihlerle çalışma sayfasında açıklanan mutate() ve as_date() fonksiyonlarını kullanarak sınıfı kolayca uygun tarih sınıfına dönüştürebiliriz.
Ayrıca, “obs” ön ekini silerek observation sütununu sayısal bir biçime dönüştürmek isteyebiliriz. Bunu stringr paketinden str_remove_all() ile yapabiliriz (Karakterler ve dizeler sayfasına bakın).
df_long <-
df_long %>%
mutate(
date = date %>% lubridate::as_date(),
observation =
observation %>%
str_remove_all("obs") %>%
as.numeric()
)
df_long## # A tibble: 9 × 4
## id observation date status
## <chr> <dbl> <date> <chr>
## 1 A 1 2021-04-23 Healthy
## 2 A 2 2021-04-24 Healthy
## 3 A 3 2021-04-25 Unwell
## 4 B 1 2021-04-23 Healthy
## 5 B 2 2021-04-24 Healthy
## 6 B 3 2021-04-25 Healthy
## 7 C 1 2021-04-23 Missing
## 8 C 2 2021-04-24 Healthy
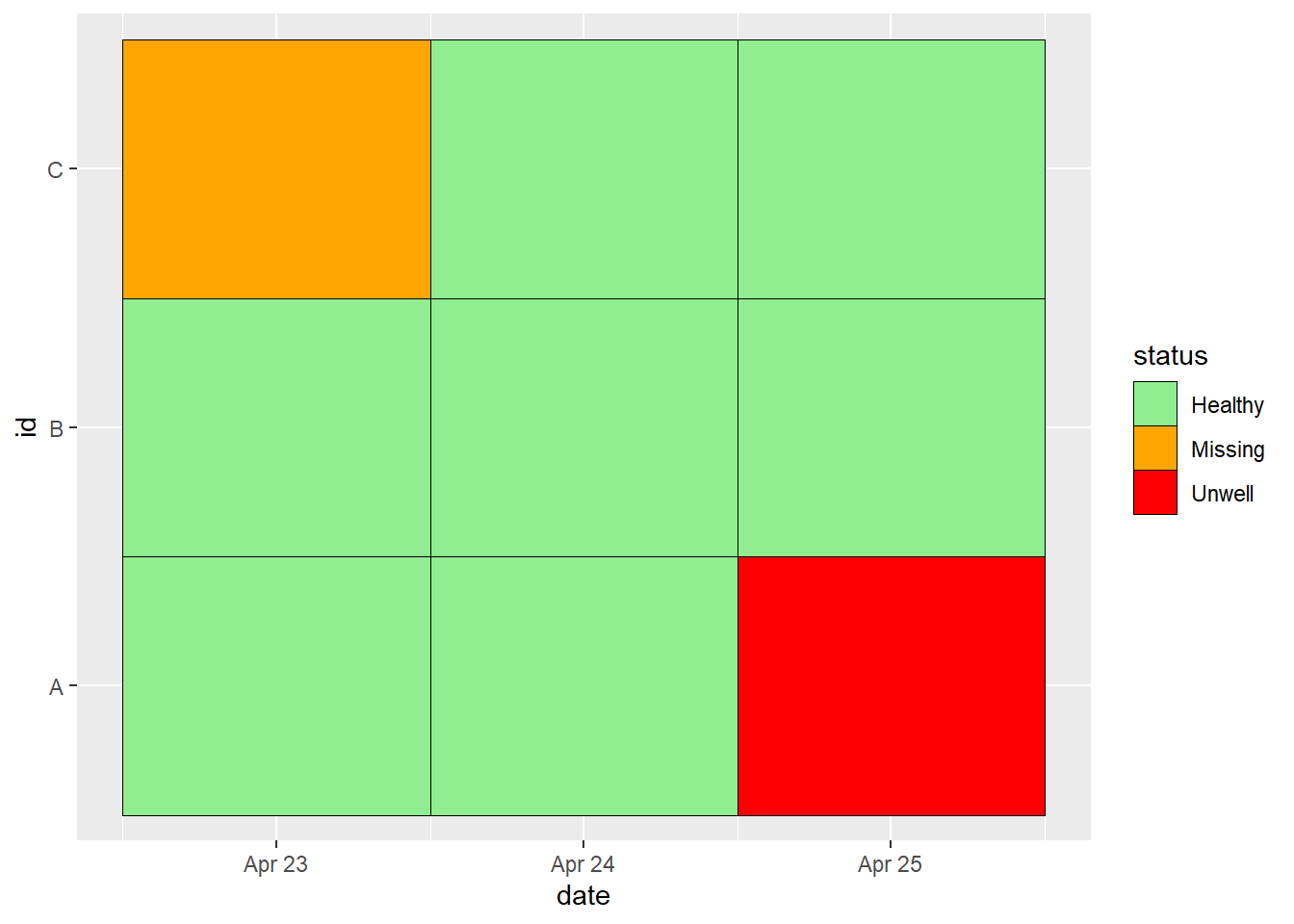
## 9 C 3 2021-04-25 HealthyVe şimdi bu formattaki verilerle çalışmaya başlayabiliriz, örn. açıklayıcı bir ısı döşemesi çizerek:
ggplot(data = df_long, mapping = aes(x = date, y = id, fill = status)) +
geom_tile(colour = "black") +
scale_fill_manual(
values =
c("Healthy" = "lightgreen",
"Unwell" = "red",
"Missing" = "orange")
)
12.3 Uzundan genişe
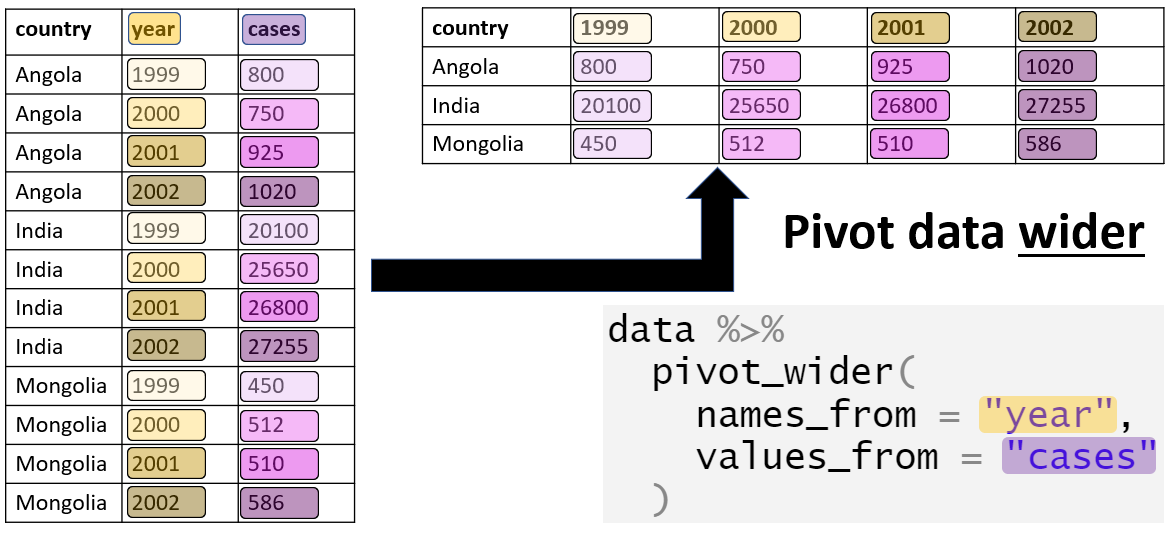
Bazı durumlarda, bir veri tabanını daha geniş bir formata dönüştürmek isteyebiliriz. Bunun için pivot_wider() fonksiyonunu kullanabiliriz.
Analiz sonuçlarını okuyucu için daha anlaşılır bir formata dönüştürmek istediğimiz zamandır (örneğin, [Sunum için bir tablo][Tables for presentation]), bu fonksiyonu. Genellikle bu işlem, bir gözlem için değerlerin birden çok satıra yayıldığı bir veri kümesinin, bu değerlerin tek bir satırda depolandığı bir formata dönüştürülmesini içerir.
Veri
Sayfanın bu bölümü için, vaka başına bir satır içeren satır listesini kullanacağız (Hazırlık bölümüne bakın).
İlk 50 satır:
Farklı yaş gruplarındaki bireylerin sayısını cinsiyete göre bilmek istediğimizi varsayalım:
## age_cat gender n
## 1 0-4 f 640
## 2 0-4 m 416
## 3 0-4 <NA> 39
## 4 5-9 f 641
## 5 5-9 m 412
## 6 5-9 <NA> 42
## 7 10-14 f 518
## 8 10-14 m 383
## 9 10-14 <NA> 40
## 10 15-19 f 359
## 11 15-19 m 364
## 12 15-19 <NA> 20
## 13 20-29 f 468
## 14 20-29 m 575
## 15 20-29 <NA> 30
## 16 30-49 f 179
## 17 30-49 m 557
## 18 30-49 <NA> 18
## 19 50-69 f 2
## 20 50-69 m 91
## 21 50-69 <NA> 2
## 22 70+ m 5
## 23 70+ <NA> 1
## 24 <NA> <NA> 86Bu bize, ggplot2’de görselleştirme için harika, ancak tabloda sunum için ideal olmayan uzun bir veri tabanı sağlar:
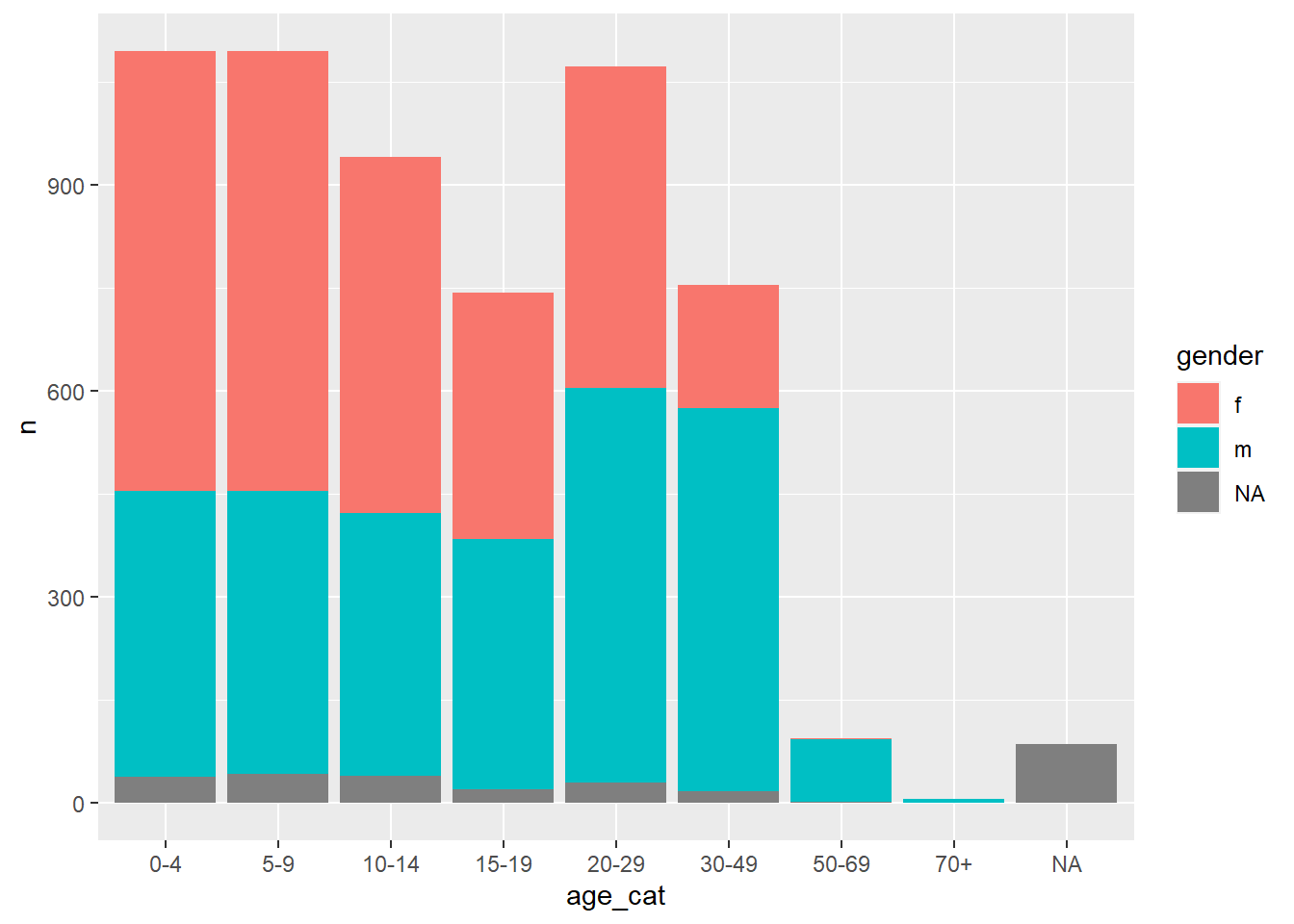
Daha geniş olarak pivotlama
Bu nedenle, verileri raporlara tablo olarak dahil etmek amaçlı olarak daha uygun biçime dönüştürmek için pivot_wider()’ı kullanabiliriz.
name_from argümanı, yeni sütun adlarının oluşturulacağı names sütunu belirtirken; values_from argümanı, hücreleri doldurmak için gereken değerlerin alınacağı sütunu belirtir. id_cols = argümanı isteğe bağlıdır, ancak döndürülmemesi gereken satırları içeren sütun adları vektörünü içerir ..
table_wide <-
df_wide %>%
pivot_wider(
id_cols = age_cat,
names_from = gender,
values_from = n
)
table_wide## # A tibble: 9 × 4
## age_cat f m `NA`
## <fct> <int> <int> <int>
## 1 0-4 640 416 39
## 2 5-9 641 412 42
## 3 10-14 518 383 40
## 4 15-19 359 364 20
## 5 20-29 468 575 30
## 6 30-49 179 557 18
## 7 50-69 2 91 2
## 8 70+ NA 5 1
## 9 <NA> NA NA 86Bu tablo daha okuyucu dostudur ve bu nedenle raporlarımıza dahil edilmesi daha uygundur. Flextable ve knitr dahil olmak üzere çeşitli paketlerle güzel bir tablolar oluşturulabilir. Bu süreç Sunum için tablolar sayfasında daha detaylı olarak anlatılmıştır.
table_wide %>%
janitor::adorn_totals(c("row", "col")) %>% # adds row and column totals
knitr::kable() %>%
kableExtra::row_spec(row = 10, bold = TRUE) %>%
kableExtra::column_spec(column = 5, bold = TRUE) | age_cat | f | m | NA | Total |
|---|---|---|---|---|
| 0-4 | 640 | 416 | 39 | 1095 |
| 5-9 | 641 | 412 | 42 | 1095 |
| 10-14 | 518 | 383 | 40 | 941 |
| 15-19 | 359 | 364 | 20 | 743 |
| 20-29 | 468 | 575 | 30 | 1073 |
| 30-49 | 179 | 557 | 18 | 754 |
| 50-69 | 2 | 91 | 2 | 95 |
| 70+ | NA | 5 | 1 | 6 |
| NA | NA | NA | 86 | 86 |
| Total | 2807 | 2803 | 278 | 5888 |
12.4 Doldurma
Bazı durumlarda pivot işlemi sonrası ve daha yaygın olarak bir birleştirme sonrası, bazı hücrelerde doldurmak istediğimiz boşluklar kalır.
Veri
Örneğin, ölçüm numarası, tesisin adı ve o andaki vaka sayısı için gözlemleri olan iki veri tabanını inceleyelim. Ancak, ikinci veri kümesinde ek olarak Year değişkeni vardır.
df1 <-
tibble::tribble(
~Measurement, ~Facility, ~Cases,
1, "Hosp 1", 66,
2, "Hosp 1", 26,
3, "Hosp 1", 8,
1, "Hosp 2", 71,
2, "Hosp 2", 62,
3, "Hosp 2", 70,
1, "Hosp 3", 47,
2, "Hosp 3", 70,
3, "Hosp 3", 38,
)
df1 ## # A tibble: 9 × 3
## Measurement Facility Cases
## <dbl> <chr> <dbl>
## 1 1 Hosp 1 66
## 2 2 Hosp 1 26
## 3 3 Hosp 1 8
## 4 1 Hosp 2 71
## 5 2 Hosp 2 62
## 6 3 Hosp 2 70
## 7 1 Hosp 3 47
## 8 2 Hosp 3 70
## 9 3 Hosp 3 38
df2 <-
tibble::tribble(
~Year, ~Measurement, ~Facility, ~Cases,
2000, 1, "Hosp 4", 82,
2001, 2, "Hosp 4", 87,
2002, 3, "Hosp 4", 46
)
df2## # A tibble: 3 × 4
## Year Measurement Facility Cases
## <dbl> <dbl> <chr> <dbl>
## 1 2000 1 Hosp 4 82
## 2 2001 2 Hosp 4 87
## 3 2002 3 Hosp 4 46İki veri kümesini birleştirmek için bir bind_rows() işlemi gerçekleştirdiğimizde, Year değişkeni, ön bilgi bulunmayan satırlar için (yani ilk veri kümesinde) NA ile doldurulur:
## # A tibble: 12 × 4
## Measurement Facility Cases Year
## <dbl> <chr> <dbl> <dbl>
## 1 1 Hosp 1 66 NA
## 2 1 Hosp 2 71 NA
## 3 1 Hosp 3 47 NA
## 4 1 Hosp 4 82 2000
## 5 2 Hosp 1 26 NA
## 6 2 Hosp 2 62 NA
## 7 2 Hosp 3 70 NA
## 8 2 Hosp 4 87 2001
## 9 3 Hosp 1 8 NA
## 10 3 Hosp 2 70 NA
## 11 3 Hosp 3 38 NA
## 12 3 Hosp 4 46 2002
fill()
Bu durumda, özellikle zaman içindeki eğilimleri keşfetmek istiyorsak, yıl dahil etmek için yararlı bir değişkendir. Bu nedenle, doldurulacak sütunu ve doldurma yönünü (bu durumda yukarı) belirterek boş hücreleri doldurmak için fill() komutunu kullanırız:
## # A tibble: 12 × 4
## Measurement Facility Cases Year
## <dbl> <chr> <dbl> <dbl>
## 1 1 Hosp 1 66 2000
## 2 1 Hosp 2 71 2000
## 3 1 Hosp 3 47 2000
## 4 1 Hosp 4 82 2000
## 5 2 Hosp 1 26 2001
## 6 2 Hosp 2 62 2001
## 7 2 Hosp 3 70 2001
## 8 2 Hosp 4 87 2001
## 9 3 Hosp 1 8 2002
## 10 3 Hosp 2 70 2002
## 11 3 Hosp 3 38 2002
## 12 3 Hosp 4 46 2002Alternatif olarak, verileri aşağı yönde dolduracak şekilde yeniden düzenleyebiliriz:
## # A tibble: 12 × 4
## Measurement Facility Cases Year
## <dbl> <chr> <dbl> <dbl>
## 1 1 Hosp 4 82 2000
## 2 1 Hosp 3 47 NA
## 3 1 Hosp 2 71 NA
## 4 1 Hosp 1 66 NA
## 5 2 Hosp 4 87 2001
## 6 2 Hosp 3 70 NA
## 7 2 Hosp 2 62 NA
## 8 2 Hosp 1 26 NA
## 9 3 Hosp 4 46 2002
## 10 3 Hosp 3 38 NA
## 11 3 Hosp 2 70 NA
## 12 3 Hosp 1 8 NA## # A tibble: 12 × 4
## Measurement Facility Cases Year
## <dbl> <chr> <dbl> <dbl>
## 1 1 Hosp 4 82 2000
## 2 1 Hosp 3 47 2000
## 3 1 Hosp 2 71 2000
## 4 1 Hosp 1 66 2000
## 5 2 Hosp 4 87 2001
## 6 2 Hosp 3 70 2001
## 7 2 Hosp 2 62 2001
## 8 2 Hosp 1 26 2001
## 9 3 Hosp 4 46 2002
## 10 3 Hosp 3 38 2002
## 11 3 Hosp 2 70 2002
## 12 3 Hosp 1 8 2002Artık görselleştirmek için kullanışlı bir veri setimiz var:
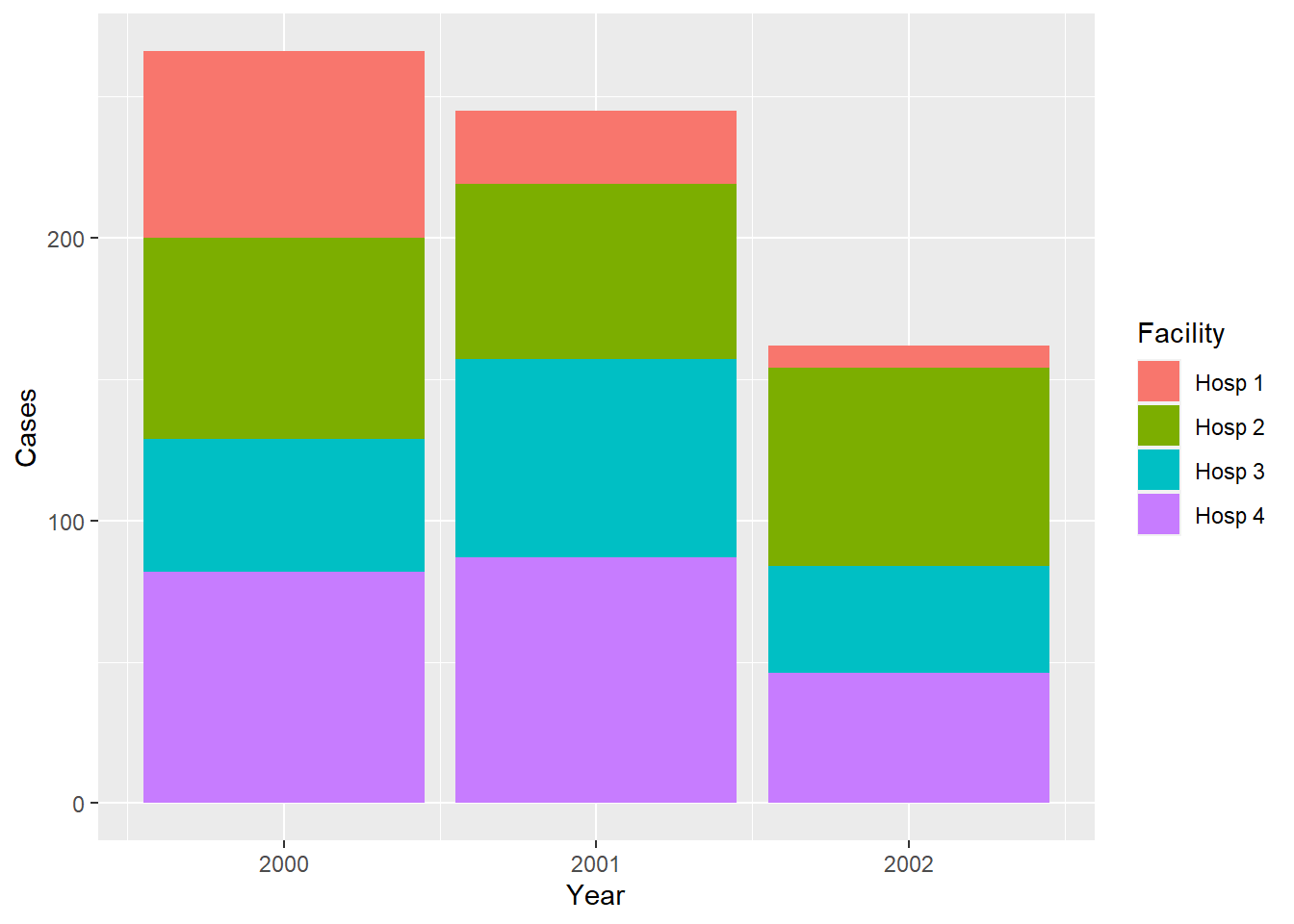
Ancak bu form tablo halinde sunmak için daha az kullanışlıdır, bu yüzden uzun, düzensiz veri çerçevesini daha geniş, düzenli bir veri çerçevesine dönüştürme alıştırması yapalım:
df_combined %>%
pivot_wider(
id_cols = c(Measurement, Facility),
names_from = "Year",
values_from = "Cases"
) %>%
arrange(Facility) %>%
janitor::adorn_totals(c("row", "col")) %>%
knitr::kable() %>%
kableExtra::row_spec(row = 5, bold = TRUE) %>%
kableExtra::column_spec(column = 5, bold = TRUE) | Measurement | Facility | 2000 | 2001 | 2002 | Total |
|---|---|---|---|---|---|
| 1 | Hosp 1 | 66 | NA | NA | 66 |
| 2 | Hosp 1 | NA | 26 | NA | 26 |
| 3 | Hosp 1 | NA | NA | 8 | 8 |
| 1 | Hosp 2 | 71 | NA | NA | 71 |
| 2 | Hosp 2 | NA | 62 | NA | 62 |
| 3 | Hosp 2 | NA | NA | 70 | 70 |
| 1 | Hosp 3 | 47 | NA | NA | 47 |
| 2 | Hosp 3 | NA | 70 | NA | 70 |
| 3 | Hosp 3 | NA | NA | 38 | 38 |
| 1 | Hosp 4 | 82 | NA | NA | 82 |
| 2 | Hosp 4 | NA | 87 | NA | 87 |
| 3 | Hosp 4 | NA | NA | 46 | 46 |
| Total |
|
266 | 245 | 162 | 673 |
Not: Bu durumda, ek değişken olan “ölçüm” tablonun oluşturulmasına engel olacağından, yalnızca Facility, Year ve Cases değişkenleri dahil edilmelidir:
df_combined %>%
pivot_wider(
names_from = "Year",
values_from = "Cases"
) %>%
knitr::kable()| Measurement | Facility | 2000 | 2001 | 2002 |
|---|---|---|---|---|
| 1 | Hosp 4 | 82 | NA | NA |
| 1 | Hosp 3 | 47 | NA | NA |
| 1 | Hosp 2 | 71 | NA | NA |
| 1 | Hosp 1 | 66 | NA | NA |
| 2 | Hosp 4 | NA | 87 | NA |
| 2 | Hosp 3 | NA | 70 | NA |
| 2 | Hosp 2 | NA | 62 | NA |
| 2 | Hosp 1 | NA | 26 | NA |
| 3 | Hosp 4 | NA | NA | 46 |
| 3 | Hosp 3 | NA | NA | 38 |
| 3 | Hosp 2 | NA | NA | 70 |
| 3 | Hosp 1 | NA | NA | 8 |
12.5 Kaynaklar
Yararlı bir eğitim