23 Zaman serileri ve salgınların tespit edilmesi
23.1 Genel bakış
Bu sekme, zaman serisi analizi için gereken birkaç paketin kullanımını göstermektedir. Bu tip analizler öncelikle tidyvers tidyverts ailesinden gelen paketlerle yapılır, ancak bulaşıcı hastalık epidemiyolojisi için daha uygun olan RECON trend paketini trending de kullanır.
Aşağıdaki örnekte, Almanya’da hazırlanmış Campylobacter bir veri setini kullandığımıza dikkat edin (ayrıntılar için el kitabının veri bölümüne bakabilirsiniz). Ancak, aynı kodu birden fazla ülke veya başka tabakalar içeren bir veritabanlarında çalıştırmak istiyorsanız, r4epis github deposunda örnek bir kod şablonu bulunmaktadır.r4epis github repo.
Kapsanan konular:
- Zaman serisi verileri
- Tanımlayıcı analiz
- Uyum (fitting) regresyonları
- İki zaman serisinin ilişkisi
- Salgın tespiti
- Kesintili zaman serisi
23.2 Hazırlık
Paketler
Bu kod kümesi, analizler için gereken paketlerin yüklenmesini gösterir. Bu el kitabında, gerekirse paketi kuran ve kullanım için yükleyen pacman’ın p_load() fonksiyonunu vurguluyoruz. Ayrıca temel R’dan library() ile paketleri yükleyebilirsiniz. R paketleri hakkında daha fazla bilgi için R temelleri sayfasına bakın. R temelleri
pacman::p_load(rio, # Dosyanın içe aktarılması
here, # Dosyanın yerinin bulunması
tidyverse, # veri yönetimi + ggplot2 grafikleri
tsibble, # zaman serisi veri tabanlarını işlemek
slider, # hareketli ortalamaları hesaplamak için
imputeTS, # eksik değerleri doldurmak için
feasts, # zaman serilerinde ayrıştırma ve otokorelasyon için
forecast, # sin ve cosin terimlerini verilere uydurun (not: feasts sonrası yüklenmelidir)
trending, # model oluştur ve değerlendir
tmaptools, # yer adlarına göre coğrafi koordinatları (boylam/enlem) almak için
ecmwfr, # copernicus uydu CDS API ile etkileşim için
stars, # .nc (iklim verileri) dosyalarını okumak için
units, # for defining units of measurement (climate data)
yardstick, # model doğruluğuna bakmak için
surveillance # sapma tespiti için
)Veri yükleme
Bu el kitabında kullanılan tüm verileri, [el kitabının ve verilerin indirilmesi] sayfasındaki talimatlar aracılığıyla indirebilirsiniz.
Bu bölümde kullanılan örnek veri seti, 2001 ve 2011 yılları arasında Almanya’da rapor edilen haftalık kampilobakter vaka sayılarıdır. Bu veri dosyasını (.xlsx) indirmek için buraya tıklayabilirsiniz.(.xlsx) olarak görülecektir.
Bu veri seti, surveillance paketinde bulunan veri setinin küçültülmüş bir versiyonudur. (detaylar için surveillance paketini yükleyin ve bkz. ?campyDE) surveillance
Bu verileri rio paketinden import() fonksiyonu ile içe aktarın (.xlsx, .csv, .rds gibi birçok dosya türünü işler - ayrıntılar için [İçe Aktarma ve Dışa Aktarma] sayfasına bakın).
# sayımları R'ye aktar
counts <- rio::import("campylobacter_germany.xlsx")Sayımların ilk 10 satırı aşağıda gösterilmiştir
Temiz veri
Aşağıdaki kod, tarih sütununun uygun biçimde olmasını sağlar. Bu sekme için tsibble paketini kullanacağız ve bu nedenle bir takvim haftası değişkeni oluşturmak için yearweek fonksiyonu kullanılacaktır. Bunu yapmanın başka yolları da vardır (ayrıntılar için Tarihlerle çalışma sayfasına bakın), ancak zaman serileri için en iyisi tarihleri tek bir çerçeve (tsibble) içinde tutmaktır.
İklim verisinin indirilmesi
Bu sayfanın iki zaman serisi bölümünde, kampilobakter vaka sayılarını iklim verileriyle karşılaştıracağız. Dünyanın herhangi bir yerindeki iklim verileri AB’nin Copernicus Uydusundan indirilebilir.
Bunlar kesin ölçümler değildir, ancak bir modele dayalıdır (interpolasyona benzer), tahminlerin yanı sıra küresel saatlik bilgi kapsamı elde edilebilir.
Bu iklim veri dosyalarının her birini [el kitabının ve verilerin indirilmesi] sayfasından indirebilirsiniz.
Burada gösterim amacıyla, verileri Copernicus iklim veri deposundan çekmek için ecmwfr paketini kullanmak için gereken R kodunu tanıtacağız. Bunun çalışması için ücretsiz bir hesap oluşturmanız gerekecektir. Paketin web sitesinde bunun nasıl yapılacağına dair yararlı bir yol gösterilmiştirwalkthrough.
Aşağıda, uygun API anahtarlarına sahip olduğunuzda, bunun nasıl yapılacağına ilişkin örnek kod verilmiştir. Aşağıdaki X’leri hesap kimliklerinizle değiştirmeniz gerekir. Her seferinde bir yıllık veri indirmeniz gerekir, aksi takdirde sunucu zaman aşımına uğrayacaktır.
Verilerini indirmek istediğiniz konumun koordinatlarından emin değilseniz, açık sokak haritalarından koordinatları almak için tmaptools paketini kullanabilirsiniz. Alternatif bir seçenek photon paketidir,photon ancak bu henüz CRAN’da yayınlanmamıştır; photon’un güzel yanı, aramanız için birkaç eşleşme olduğunda daha fazla bağlamsal veri sağlamasıdır.
## konum koordinatlarını al
coords <- geocode_OSM("Germany", geometry = "point")
## ERA-5 sorgulaması için boylamları/enlemleri bir araya getirin (sınırlayıcı kutu)
## (tek bir noktanın koordinatlarının tekrar edebileceği gibi)
request_coords <- str_glue_data(coords$coords, "{y}/{x}/{y}/{x}")
## Kopernik uydusundan modellenen verileri çekme (ERA-5 yeniden analizi)
## https://cds.climate.copernicus.eu/cdsapp#!/software/app-era5-explorer?tab=app
## https://github.com/bluegreen-labs/ecmwfr
## hava durumu verileri için anahtar ayarla
wf_set_key(user = "XXXXX",
key = "XXXXXXXXX-XXXX-XXXX-XXXX-XXXXXXXXXXX",
service = "cds")
## ilgilenilen her yıl için çalıştırın (aksi takdirde sunucu zaman aşımına uğrar)
for (i in 2002:2011) {
## sorguyu bir araya getir
## nasıl yapılacağı için buraya bakın: https://bluegreen-labs.github.io/ecmwfr/articles/cds_vignette.html#the-request-syntax
## yukarıdaki eklenti düğmesini kullanarak isteği bir listeye dönüştürün (listeye python)
## Hedef, çıktı dosyasının adıdır!
request <- request <- list(
product_type = "reanalysis",
format = "netcdf",
variable = c("2m_temperature", "total_precipitation"),
year = c(i),
month = c("01", "02", "03", "04", "05", "06", "07", "08", "09", "10", "11", "12"),
day = c("01", "02", "03", "04", "05", "06", "07", "08", "09", "10", "11", "12",
"13", "14", "15", "16", "17", "18", "19", "20", "21", "22", "23", "24",
"25", "26", "27", "28", "29", "30", "31"),
time = c("00:00", "01:00", "02:00", "03:00", "04:00", "05:00", "06:00", "07:00",
"08:00", "09:00", "10:00", "11:00", "12:00", "13:00", "14:00", "15:00",
"16:00", "17:00", "18:00", "19:00", "20:00", "21:00", "22:00", "23:00"),
area = request_coords,
dataset_short_name = "reanalysis-era5-single-levels",
target = paste0("germany_weather", i, ".nc")
)
# dosyayı indirin ve mevcut çalışma dizininde saklayın
file <- wf_request(user = "XXXXX", # kullanıcı ID (kimlik doğrulama için)
request = request, # istek
transfer = TRUE, # dosyayı indir
path = here::here("data", "Weather")) ## dosyayı kaydetmek için yolak
}İklim verisinin yüklenmesi
İster iklim verilerini el kitabımız üzerinden indirmiş olun, ister yukarıdaki kodu kullanmış olun, artık bilgisayarınızda aynı klasörde saklanan 10 yıllık “.nc” iklim veri dosyalarına sahip olmalısınız.
Bu dosyaları stars paketiyle R’a aktarmak için aşağıdaki kodu kullanın.
## hava durumu klasörüne giden yolu tanımlayın
file_paths <- list.files(
here::here("data", "time_series", "weather"), # replace with your own file path
full.names = TRUE)
## sadece şu anki ilgi alanına sahip olanları saklayın
file_paths <- file_paths[str_detect(file_paths, "germany")]
## tüm dosyaları stars nesnesi olarak oku
data <- stars::read_stars(file_paths)## t2m, tp,
## t2m, tp,
## t2m, tp,
## t2m, tp,
## t2m, tp,
## t2m, tp,
## t2m, tp,
## t2m, tp,
## t2m, tp,
## t2m, tp,Bu dosyalar nesne verisi olarak içe aktarıldıktan sonra onları bir veri çerçevesine dönüştüreceğiz.
## veri çerçevesine değiştir
temp_data <- as_tibble(data) %>%
## değişkenler ekleyin ve birimleri düzeltin
mutate(
## takvim haftası değişkeni oluşturun
epiweek = tsibble::yearweek(time),
## tarih değişkeni oluşturun (takvim haftasının başlangıcı)
date = as.Date(epiweek),
## sıcaklığı kelvin'den santigrat dereceye değiştir
t2m = set_units(t2m, celsius),
## yağışı metreden milimetreye değiştir
tp = set_units(tp, mm)) %>%
## haftaya göre gruplandır (tarihi de sakla)
group_by(epiweek, date) %>%
## haftalık ortalamanın özetlenmesi
summarise(t2m = as.numeric(mean(t2m)),
tp = as.numeric(mean(tp)))## `summarise()` has grouped output by 'epiweek'. You can override using the `.groups`
## argument.23.3 Zaman serisi verisi
Zaman serisi verilerini yapılandırmak ve işlemek için bir dizi farklı paket vardır. Bahsedildiği gibi, tidyverts paket ailesine odaklanacağız ve bu nedenle zaman serisi nesnemizi tanımlamak için tsibble paketini kullanacağız. Zaman serisi nesnesi olarak tanımlanan bir veri setine sahip olmak, analizimizi yapılandırmanın çok daha kolay olduğu anlamına gelir.
Bunu yapmak için tsibble() fonksiyonunu kullanırız ve “indeks”i, yani ilgilenilen zaman birimini belirten değişkeni belirtiriz. Bizim durumumuzda bu, epiweek değişkenidir.
Örneğin, il bazında haftalık sayıları olan bir veri setimiz olsaydı, key = değişkenini kullanarak gruplama değişkenini de belirleyebilirdik. Bu, her grup için analiz yapmamızı sağlar.
## zaman serisi nesnesini tanımla
counts <- tsibble(counts, index = epiweek)class(counts)’a baktığınızda, düzenli bir veri çerçevesi (“tbl_df”, “tbl”, “data.frame”) olmanın yanı sıra, bir zaman serisi veri çerçevesinin (“tbl_ts”) ek özelliklerine sahip olduğunu gösterir.
ggplot2 kullanarak verilerinize hızlıca göz atabilirsiniz. Olay örgüsünde net bir mevsimsel motif olduğunu ve eksik veri olmadığını görüyoruz. Ancak, her yılın başında raporlama ile ilgili bir sorun var gibi görünüyor; vakalar yılın son haftasında düşmekte ve sonraki yılın ilk haftasında artmaktadır.
## haftaya göre vakaların bir çizgi grafiğini çizin
ggplot(counts, aes(x = epiweek, y = case)) +
geom_line()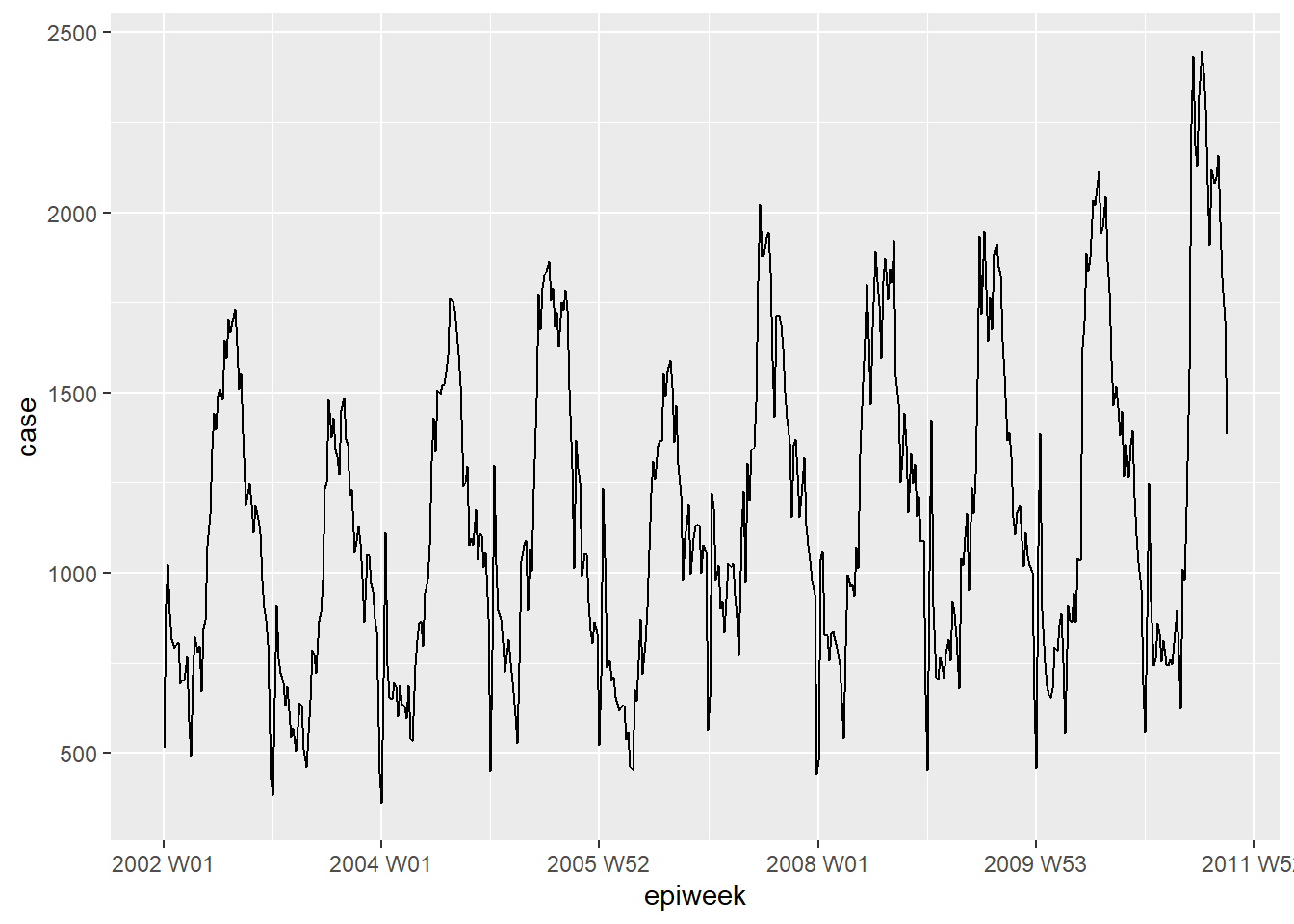
TEHLİKE: Çoğu veri kümesi bu örnek kadar temiz değildir. Aşağıdaki gibi duplikasyonları ve eksikleri kontrol etmeniz gerekecektir.
Duplikasyonlar
tsibble yinelenen gözlemlere izin vermez. Bu nedenle, her satırın benzersiz veya (anahtar değişkene göre) grup içinde benzersiz olması gerekir. Paket, kopyaları tanımlamaya yardımcı olan birkaç işleve sahiptir. Bunlar, satırın bir kopya olup olmadığına dair bir DOĞRU/YANLIŞ vektörü veren are_duplicated() ve size duplike satırların veri çerçevesini veren duplicates() fonksiyonlarını içerir.
İstediğiniz satırları nasıl seçeceğinizle ilgili daha fazla ayrıntı için Tekilleştirme (De-duplikasyon) sayfasına bakabilirsiniz. Tekilleştirme
## satırların kopya olup olmadığını DOĞRU/YANLIŞ vektörü olarak çıkarın
are_duplicated(counts, index = epiweek)
## yinelenen satırların veri çerçevesini çıkarın
duplicates(counts, index = epiweek) Eksikler
Yukarıdaki kısa incelememizde hiçbir eksik olmadığını gördük, ancak yeni yıl civarında raporlama gecikmesinde bir sorun olduğunu da gördük. Bu sorunu çözmenin bir yolu, bu değerleri eksik olarak ayarlamak ve ardından değerleri atfetmek olabilir. Zaman serisi atamasının en basit şekli, en son kayıp olmayan ve bir sonraki kayıp olmayan değer arasına düz bir çizgi çizmektir. Bunu yapmak için imputeTS paketinden na_interpolation() fonksiyonunu kullanacağız.
Tahmin için değerler atamada diğer seçenekler için Eksik veri sayfasına bakabilirsiniz.
Diğer bir alternatif, hareketli ortalama hesaplamak, belirgin raporlama sorunlarını denemek ve düzeltmek olabilir (bir sonraki bölüme ve Hareketli ortalamalar sayfasına bakın)Hareketli ortalamalar).
## raporlama sorunları olan haftalar yerine eksikleri olan bir değişken oluşturun
counts <- counts %>%
mutate(case_miss = if_else(
## epiweek 52, 53, 1 veya 2 içeriyorsa
str_detect(epiweek, "W51|W52|W53|W01|W02"),
## sonrasında eksik olarak ayarlayın
NA_real_,
## aksi takdirde değeri saklayın
case
))
## alternatif olarak eksikleri doğrusal trendle interpolasyon yapın
## en yakın iki bitişik nokta arasında
counts <- counts %>%
mutate(case_int = imputeTS::na_interpolation(case_miss)
)
## orijinaline kıyasla hangi değerlerin atfedildiğini (impute) kontrol etmek için
ggplot_na_imputations(counts$case_miss, counts$case_int) +
## geleneksel bir grafik oluşturun (siyah eksenli ve beyaz arka planlı)
theme_classic()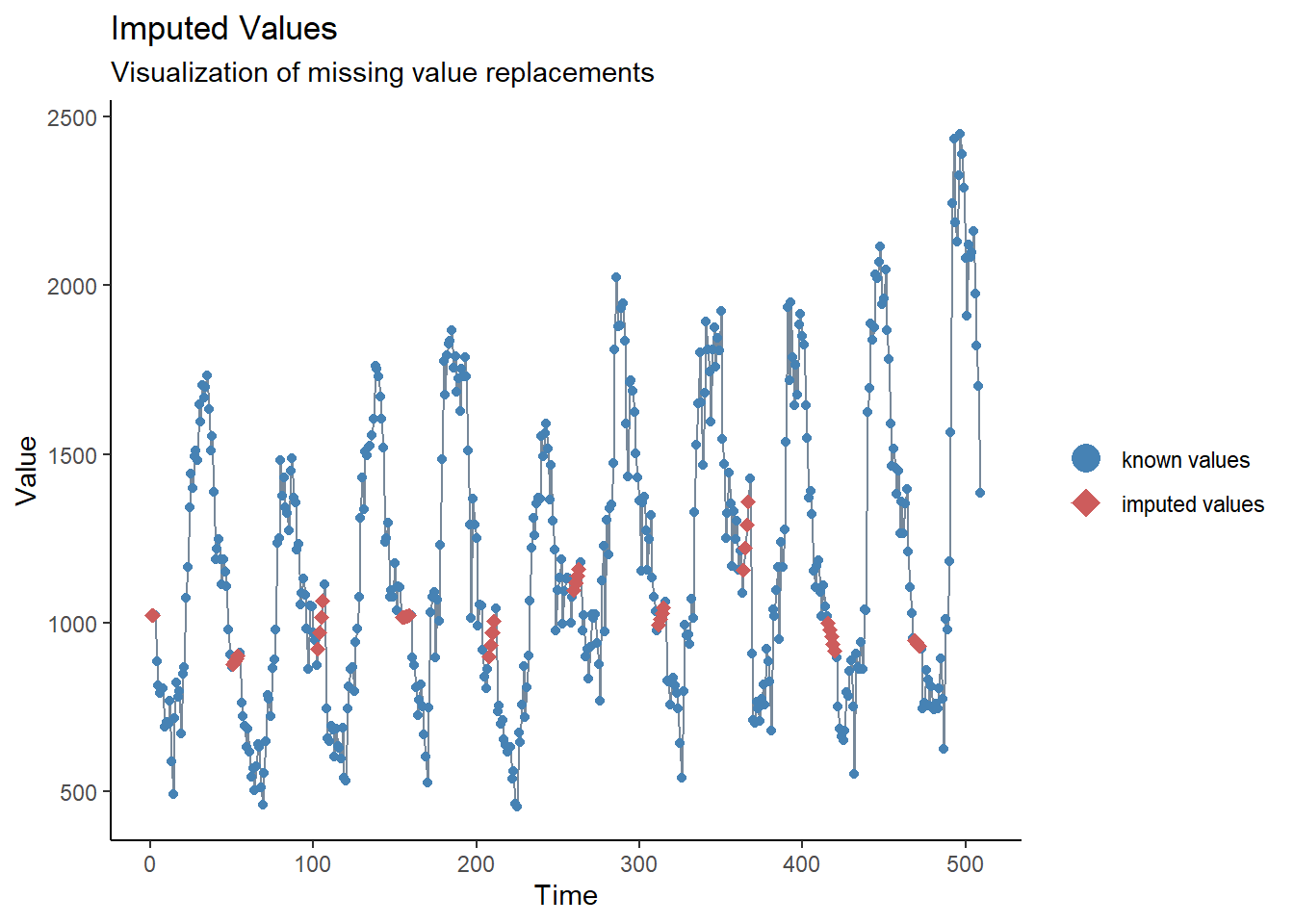
23.4 Tanımlayıcı analizler
Hareketli ortalamalar
Veriler çok gürültülüyse (yukarı ve aşağı atlamalar sayılır), hareketli bir ortalama hesaplamak yardımcı olabilir. Aşağıdaki örnekte, her hafta için önceki dört haftadaki ortalama vaka sayısını hesaplıyoruz. Bu hesap ile, verilerin daha yorumlanabilir hale gelmesi için düzeltme yapılır. Örneğimiz için bu düzeltmenin faydası kısıtlıdır, bu yüzden daha fazla analiz için interpolasyonlu verilere bağlı kalınacaktuır. Daha fazla ayrıntı için Hareketli ortalamalar sayfasına bakın. Hareketli ortalamalar.
## hareketli bir ortalama değişkeni oluşturun (eksiklerle ilgilenilecektir)
counts <- counts %>%
## ma_4w değişkenini oluştur
## case değişkeninin her satırının üzerine kaydırın
mutate(ma_4wk = slider::slide_dbl(case,
## her satır için ad hesapla
~ mean(.x, na.rm = TRUE),
## önceki dört haftayı kullan
.before = 4))
## farkını hızla görselleştirin
ggplot(counts, aes(x = epiweek)) +
geom_line(aes(y = case)) +
geom_line(aes(y = ma_4wk), colour = "red")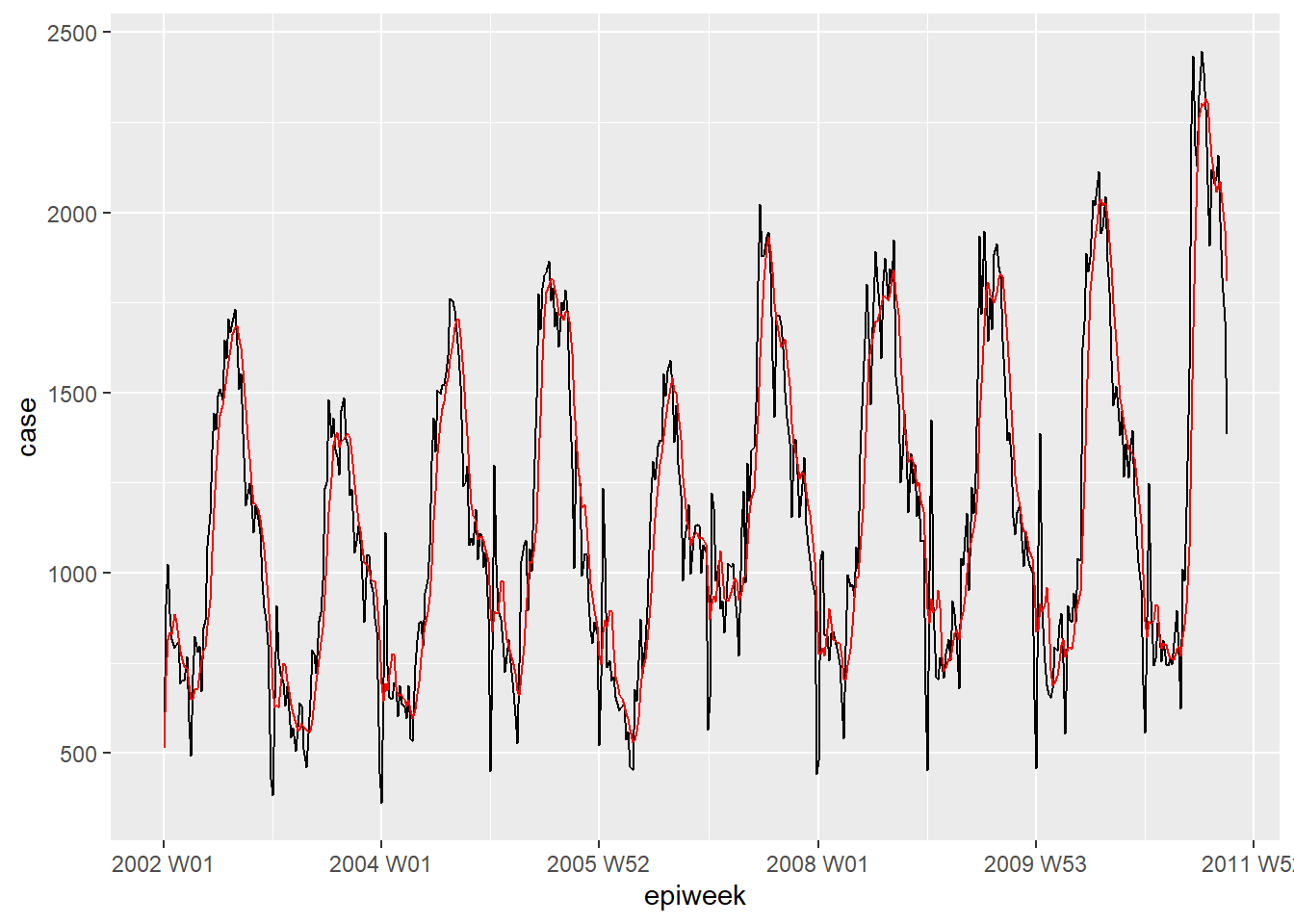
Periyodisite
Aşağıda bir periodogram oluşturmak için özel bir fonksiyon tanımlıyoruz. R’de fonksiyonların nasıl yazılacağı hakkında bilgi için Fonksiyon yazma sayfasına bakın.
İlk olarak fonksiyon tanımlanır. Fonksiyonun argümanları, sütun sayıları olan bir veri tabanını, veri tabanının ilk haftası olan başlangıç_haftası =, yılda kaç periyot olduğunu gösteren bir sayıyı (örneğin 52, 12) ve son olarak çıktı stilini içerir (aşağıdaki koddaki ayrıntılara bakın).
## Fonksiyonun argümanları
#####################
## x veritabanıdır
## x içindeki oranlar ya da sayımlar değişkendir
## start_week is the first week in your dataset
## periyot bir yıldaki birim sayısıdır
## çıktı, dönüş spektral periodogramı ya da peak görülen haftalardır.
## "periodogram" veya "weeks"
# Fonksiyonu tanımlamak
periodogram <- function(x,
counts,
start_week = c(2002, 1),
period = 52,
output = "weeks") {
## bir tsibble olmadığından emin olun, projeye filtre uygulayın ve yalnızca ilgilenilen sütunları tutun
prepare_data <- dplyr::as_tibble(x)
# veriyi hazırla <- prepare_data[prepare_data[[strata]] == j, ]
prepare_data <- dplyr::select(prepare_data, {{counts}})
## spec.pgram ile kullanılabilecek bir ara zaman serisi (“zoo”) oluşturun
zoo_cases <- zoo::zooreg(prepare_data,
start = start_week, frequency = period)
## hızlı fourier dönüşümü kullanmayan bir spektral periodogram elde edin
periodo <- spec.pgram(zoo_cases, fast = FALSE, plot = FALSE)
## pieak haftaları elde edin
periodo_weeks <- 1 / periodo$freq[order(-periodo$spec)] * period
if (output == "weeks") {
periodo_weeks
} else {
periodo
}
}
## en yüksek frekanslara sahip haftaları çıkarmak için spektral periodogram alın
## (mevsimsellik kontrolü)
periodo <- periodogram(counts,
case_int,
start_week = c(2002, 1),
output = "periodogram")
## görselleştirme için bir veri çerçevesine tam spektrum ve frekans yerleştirilir
periodo <- data.frame(periodo$freq, periodo$spec)
## en sık meydana gelen periyodikliği gösteren bir periodogram çizin
ggplot(data = periodo,
aes(x = 1/(periodo.freq/52), y = log(periodo.spec))) +
geom_line() +
labs(x = "Period (Weeks)", y = "Log(density)")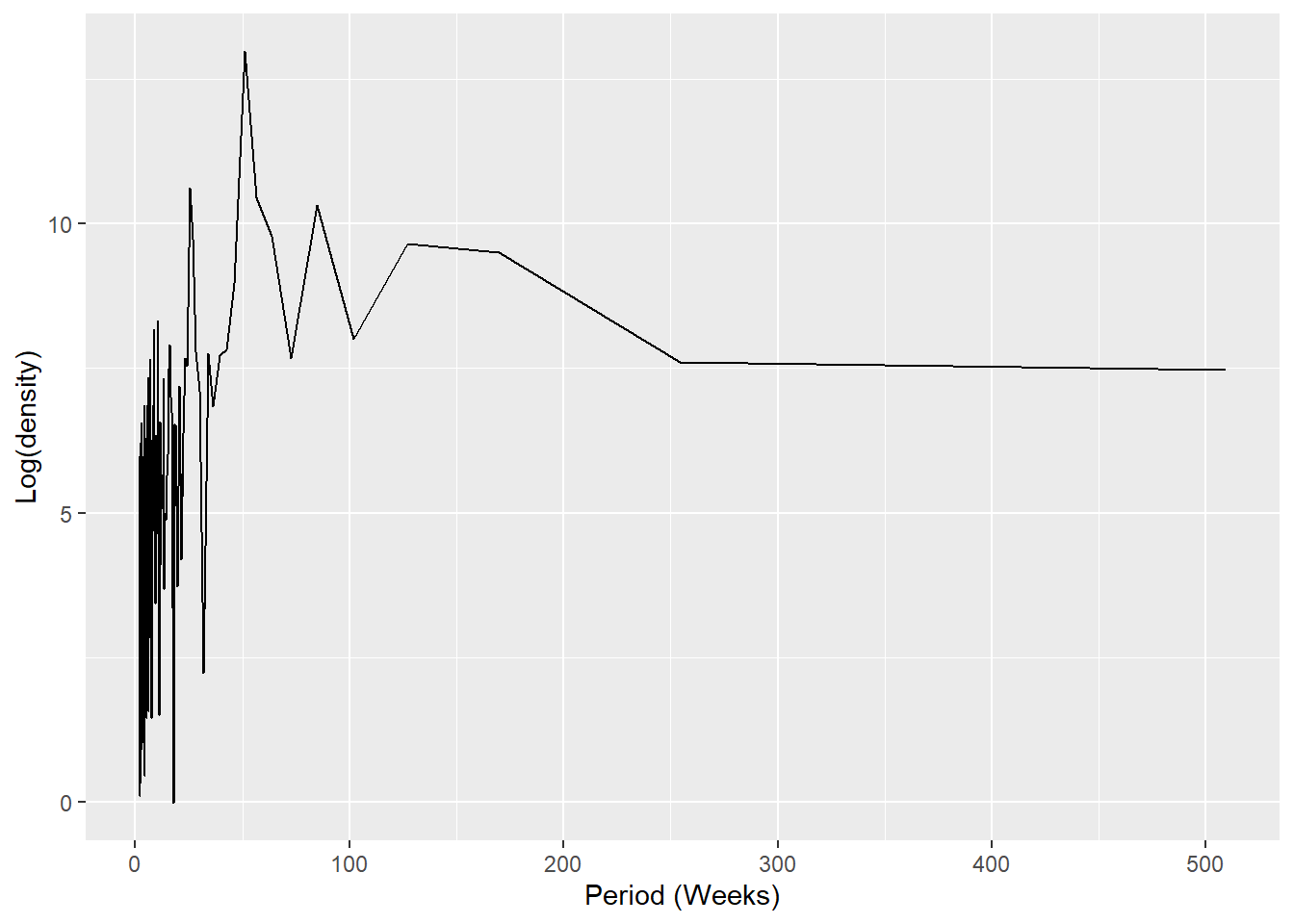
## haftaları içeren vektörü artan sırayla elde edin
peak_weeks <- periodogram(counts,
case_int,
start_week = c(2002, 1),
output = "weeks")NOT: Yukarıdaki haftaları sinüs ve kosinüs terimlerine eklemek mümkündür, ancak bu terimleri oluşturmak için bir fonksiyon kullanacağız (aşağıdaki regresyon bölümüne bakınız)
Ayrıştırma
Klasik ayrıştırma, bir zaman serisini parçalara ayırmak için kullanılır ve bu parçalar birlikte gördüğünüz modeli oluşturur. Bu farklı parçalar şunlardır:
• Trend döngüsü (verinin uzun vadeli seyri) • Mevsimsellik (tekrarlanan modeller) • Rastgele (trend ve sezon çıkarıldıktan sonra kalan)
## sayımlar veri tabanını ayrıştır
counts %>%
# additif klasik ayrıştırma modeli uygula
model(classical_decomposition(case_int, type = "additive")) %>%
## önemli bilgileri modelden çıkarın
components() %>%
## grafik oluştur
autoplot()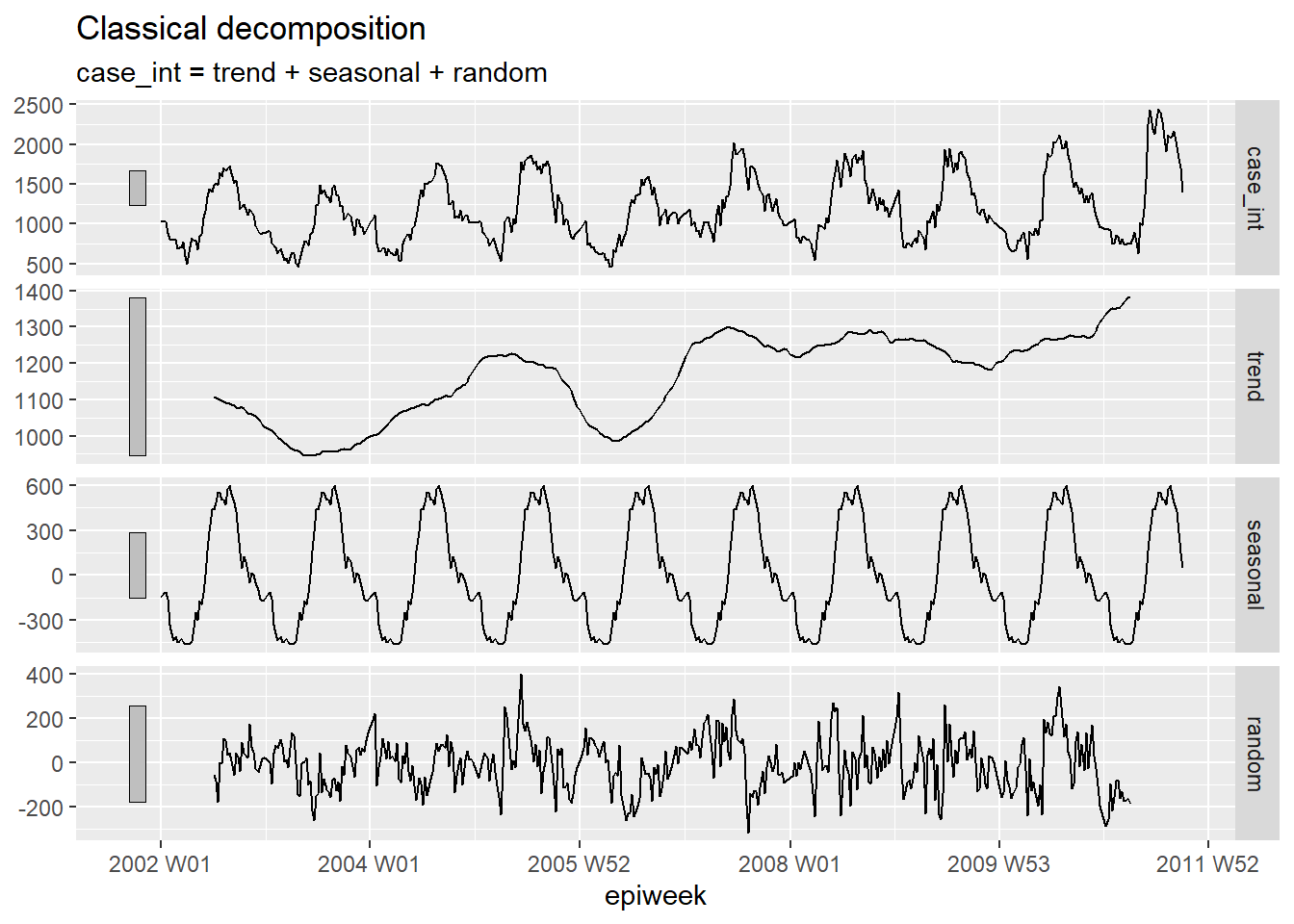
Otokorelasyon
Otokorelasyon size her haftadaki sayımlar ile önceki haftalardaki sayımlar (gecikme olarak adlandırılır) arasındaki ilişkiyi anlatır.
ACF() fonksiyonunu kullanarak, farklı gecikmelerdeki ilişkiyi bize bir dizi çizgi gösteren bir grafik oluşturabiliriz. Gecikmenin 0 (x = 0) olduğu yerde, çizgi gözlem ile kendisi arasındaki ilişkiyi gösterdiğinden (burada gösterilmemiştir) her zaman 1 olacaktır. Burada gösterilen ilk satır (x = 1), her gözlem ile bir önceki gözlem arasındaki ilişkiyi gösterir (1 gecikme), ikincisi gözlem ile iki önceki gözlem arasındaki ilişkiyi gösterir (2 gecikme) ve gecikme sayıları bu şekilde artarak devam eder (Her bir gözlem ile 1 yıllık (52 hafta öncesi) gözlem arasındaki ilişkiyi gösteren 52’ye kadar)
PACF() fonksiyonunun kullanılması (kısmi otokorelasyon için) aynı ilişkiyi gösterir, ancak aradaki diğer tüm haftalar için ayarlanmış bir ölçüttür. Kısmi otokorelasyon, periyodikliği belirlemek için daha az yararlıdır.
## sayım veri tabanını kullanma
counts %>%
## yıl içindeki gecikmeleri kullanarak otokorelasyonu hesaplayın
ACF(case_int, lag_max = 52) %>%
## grafik oluşturun
autoplot()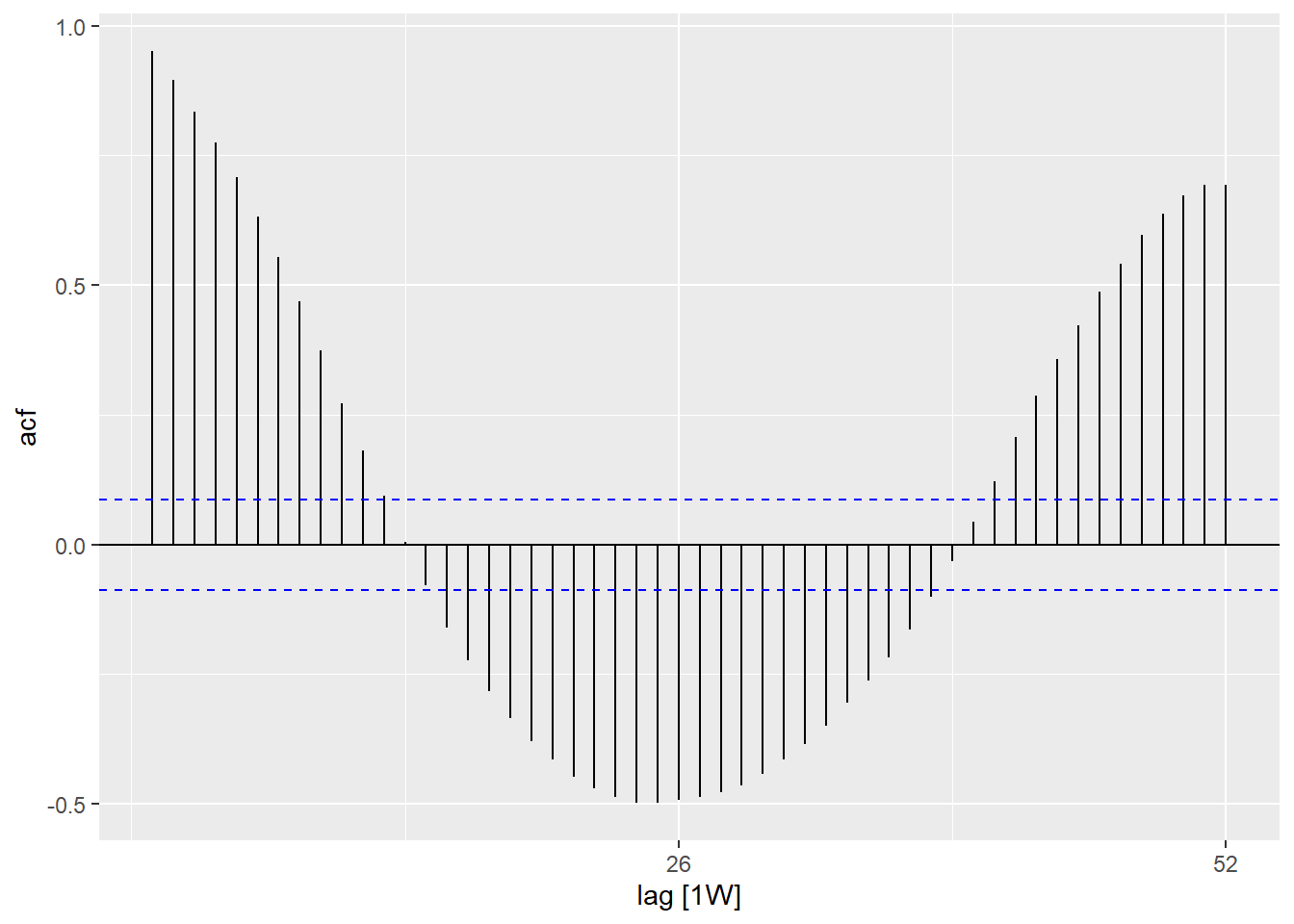
## sayım veri tabanını kullanma
counts %>%
## yıl içindeki gecikmeleri kullanarak kısmi otokorelasyonu hesaplayın
PACF(case_int, lag_max = 52) %>%
## grafik oluşturun
autoplot()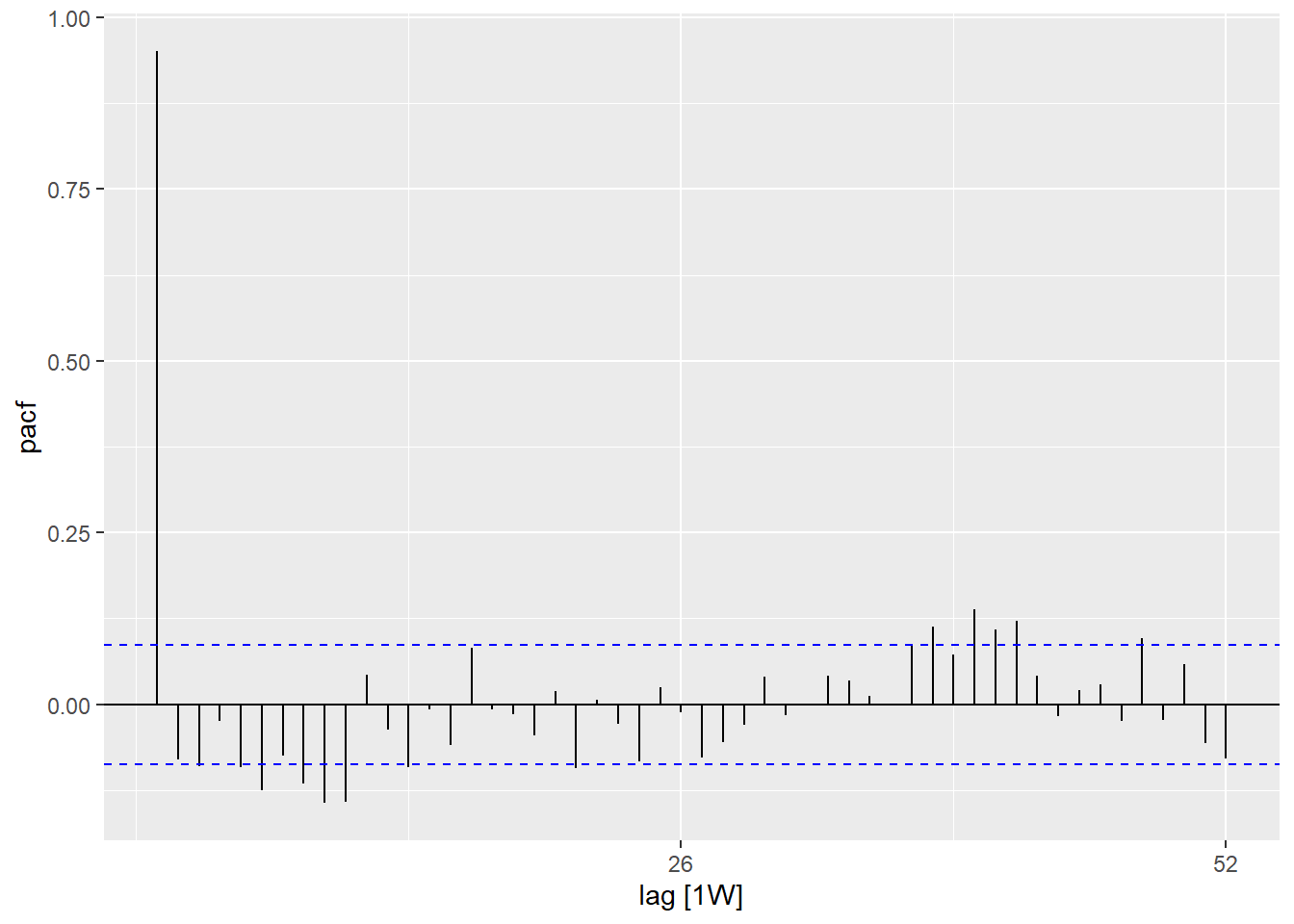
Ljung-Box testini (stats paketinde) kullanarak zaman serisinde null hipotezini (yani otokorelasyonlu olup olmadığı) test edebilirsiniz. Anlamlı bir p değeri, verilerde otokorelasyon olduğunu gösterir.
## bağımsızlık testi
Box.test(counts$case_int, type = "Ljung-Box")##
## Box-Ljung test
##
## data: counts$case_int
## X-squared = 462.65, df = 1, p-value < 2.2e-1623.5 Uyum regresyonları
Bir zaman serisine çok sayıda farklı regresyon sığdırmak mümkündür, ancak burada negatif iki terimli regresyonun nasıl sığdırılacağını göstereceğiz - çünkü bu genellikle bulaşıcı hastalıklardaki sayım verileri için en uygun olanıdır.
Fourier terimleri
Fourier terimleri, sinüs ve kosinüs eğrilerinin eş epi hafta ve case_int değişkenlerini kullanarak fourier terimleri ekleyindeğeridir. Aradaki fark, bu terimlerin verilerinizi açıklamak için en uygun eğri kombinasyonunu bulmaya dayalı bulunmasıdır.
Yalnızca bir fourier terimine kullanacak olsaydınız, periodogramınızda görülen en sık meydana gelen gecikmeniz için (bizim durumumuzda 52 hafta) bir sinüs ve bir kosinüs kullanmaya eşdeğer olacaktır. Forecast paketindeki fourier() fonksiyonunu kullanıyoruz.
Aşağıdaki kodda $ operatörünü kullanarak atadık, çünkü fourier() iki sütun oluşturur (biri sinüs, diğeri kosinüs için) ve bu nedenle bunlar veri tabanına “fourier” adı verilen bir liste olarak eklenir - ancak bu liste daha sonra regresyonda normal bir değişken olarak kullanılabilir:
Negatif binomial
Temel istatistik (base stats) veya MASS fonksiyonlarını (örn. lm(), glm() ve glm.nb()) kullanarak uyumlu regresyonlar oluşturmak mümkündür. Bununla birlikte, uygun güven ve tahmin aralıklarının (aksi takdirde mevcut olmayan) hesaplanmasına izin verdiği için, trending paketindeki fonksiyonları kullanacağız. Bu fonksiyonların sintaksı aynıdır ve bir çıktı değişkeni, ardından bir tilde (~) belirtir ve ardından artı (+) ile ayırarak ilgilendiğiniz çeşitli maruz kalma değişkenlerinizi eklersiniz.
Diğer fark, önce modeli tanımlamamız ve ardından onu verilere fit() ile uydurmamızdır. Bu, aynı sintaksa sahip birden çok farklı modeli karşılaştırmaya izin verdiği için yararlıdır.
İPUCU: Sayılar yerine oranları kullanmak istiyorsanız, offset(log(population) terimini ekleyerek popülasyon değişkenini logaritmik bir offset terimi olarak dahil edebilirsiniz. Bu durumda bir oran oluşturmak için predict() fonksiyonunu kullanmadan önce popülasyonu 1 olarak ayarlamanız gerekir.
İPUCU: ARIMA veya prophet gibi daha karmaşık modelleri uygulamak için fable paketine bakAbilirsiniz. fable.
## uydurmak istediğiniz modeli tanımlayın (negatif binom)
model <- glm_nb_model(
## ilgilenilen sonuç olarak vaka sayısını belirlemek
case_int ~
## trendi hesaba katmak için epiweek'i kullanın
epiweek +
## mevsimselliği hesaba katmak için fourier terimlerini kullanın
fourier)
## sayım veri tabanını kullanarak modelinize uydurun
fitted_model <- trending::fit(model, data.frame(counts))
## güven ve tahmin aralıklarını hesaplayın
observed <- predict(fitted_model, simulate_pi = FALSE)
## regresyonunuzu görselleştirin
ggplot(data = observed, aes(x = epiweek)) +
## model tahmini için bir satır ekleyin
geom_line(aes(y = estimate),
col = "Red") +
## tahmin aralıkları için bir bant ekleyin
geom_ribbon(aes(ymin = lower_pi,
ymax = upper_pi),
alpha = 0.25) +
## gözlemlenen vaka sayılarınız için bir satır ekleyin
geom_line(aes(y = case_int),
col = "black") +
## geleneksel bir grafik oluşturun (siyah eksenli ve beyaz arka planlı)
theme_classic()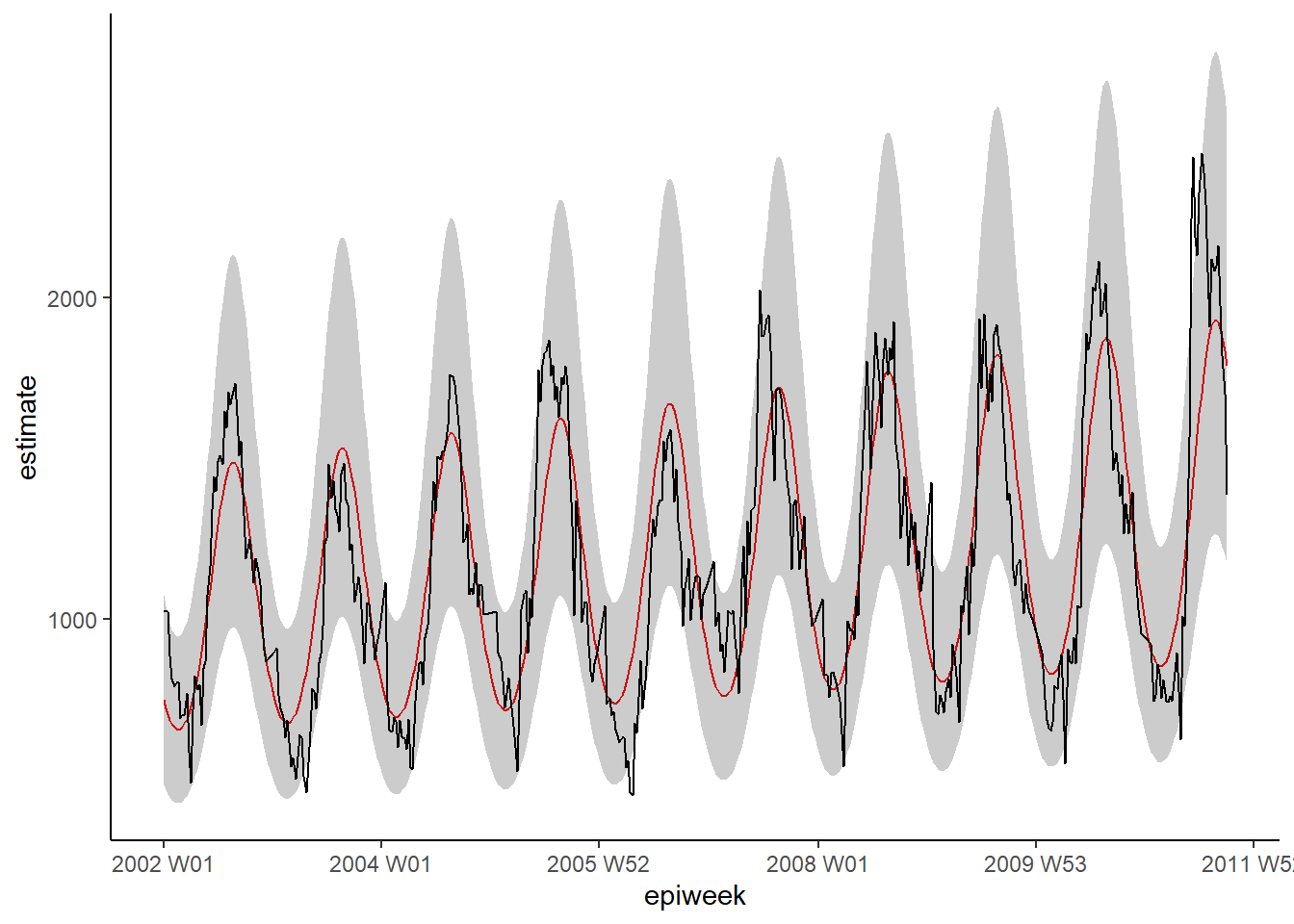
Artıklar (Residuals)
Modelimizin gözlemlenen verilere ne kadar uyum gösterdiğini görmek için artıklara bakmamız gerekir. Artıklar, gözlemlenen değerler ile modelden tahmin edilen değerler arasındaki farktır. Bunu basitçe case_int - tahmin formülünü kullanarak hesaplayabiliriz, ancak residuals() fonksiyonu bunu bizim için doğrudan regresyondan çıkarır.
Aşağıdan gördüğümüz gibi modelle tüm varyansı açıklamamaktayız. Daha fazla fourier terimi kullanmamız ve genliği ele almamız gerekebilir. Ancak bu örnek için olduğu gibi bırakacağız. Grafikler, modelimizin tepe ve diplerde (sayılar en yüksek ve en düşük olduğunda) daha kötü olduğunu ve gözlemlenen sayıları olduğundan daha az tahmin etme olasılığının (underestimate) yüksek olabileceğini gösteriyor.
## artıkların hesabı
observed <- observed %>%
mutate(resid = residuals(fitted_model$fitted_model, type = "response"))
## artıklar zaman içinde oldukça sabit mi (değilse: salgın? uygulamada değişiklik?)
observed %>%
ggplot(aes(x = epiweek, y = resid)) +
geom_line() +
geom_point() +
labs(x = "epiweek", y = "Residuals")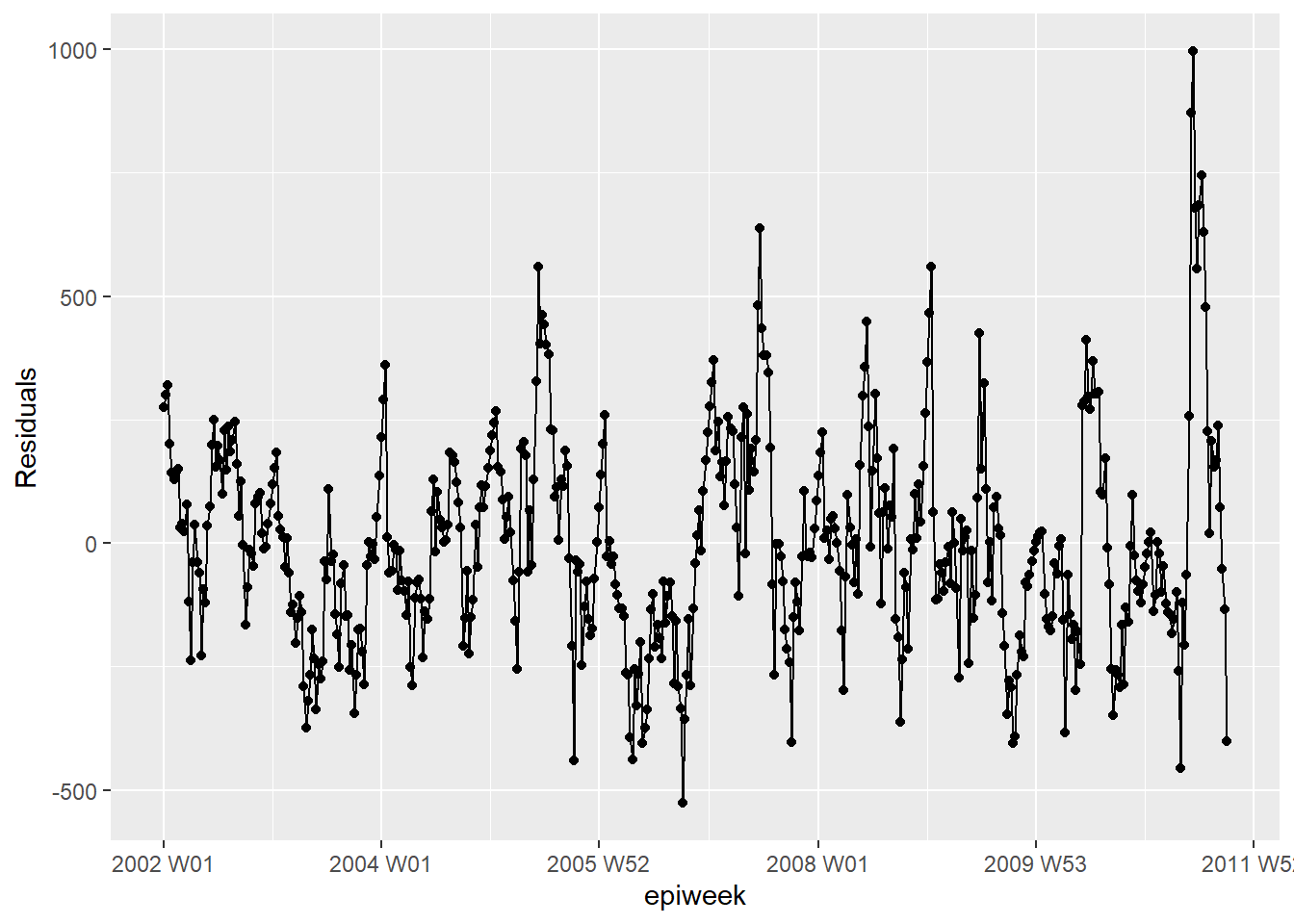
## artıklarda otokorelasyon var mı (hatanın bir motifi var mı?)
observed %>%
as_tsibble(index = epiweek) %>%
ACF(resid, lag_max = 52) %>%
autoplot()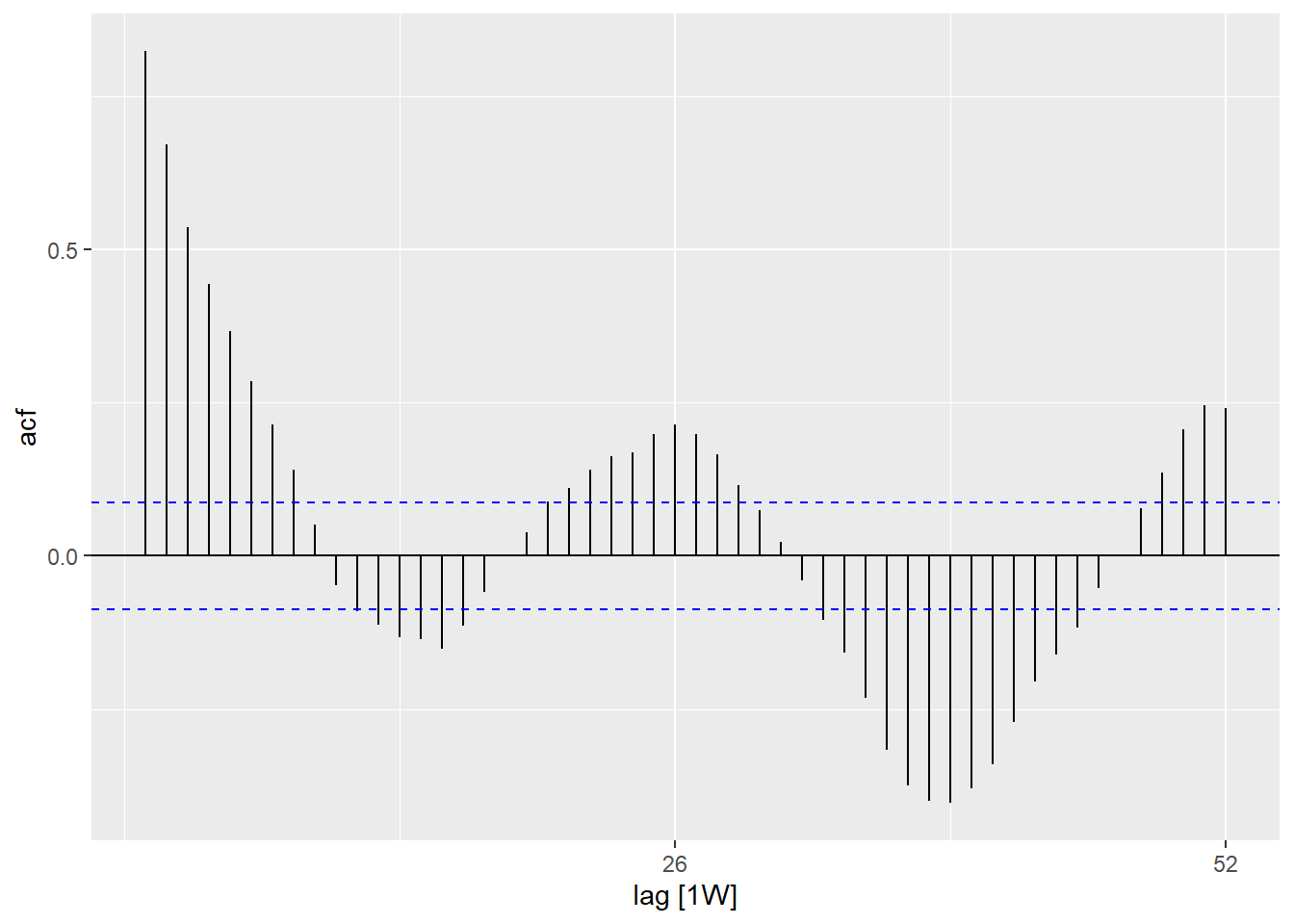
## artıklar normal olarak dağılmış mı (tahminin altında mı yoksa üzerinde mi?)
observed %>%
ggplot(aes(x = resid)) +
geom_histogram(binwidth = 100) +
geom_rug() +
labs(y = "count") 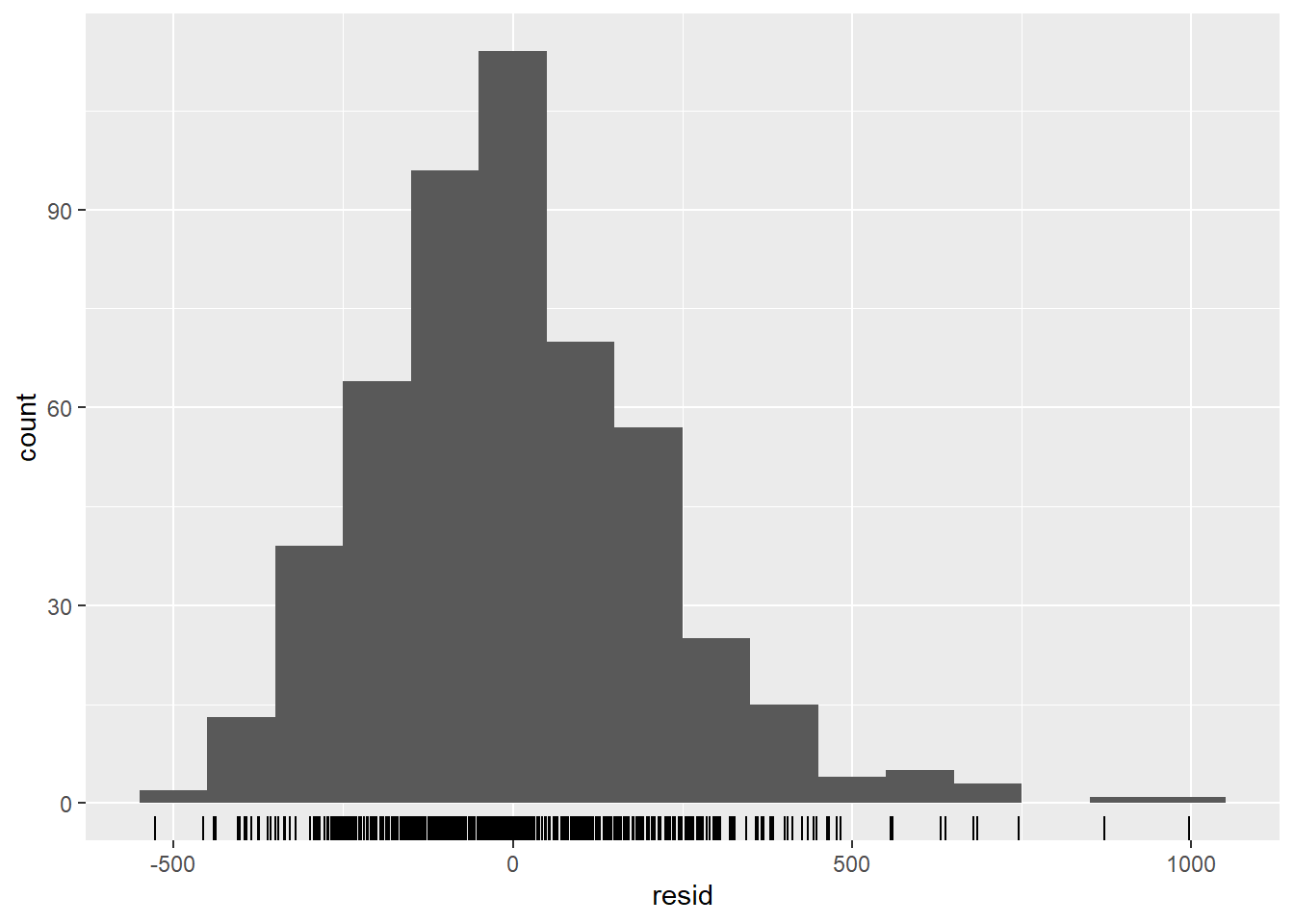
## gözlemlenen sayıları artıklarıyla karşılaştırın
## ayrıca motif görülmemeli
observed %>%
ggplot(aes(x = estimate, y = resid)) +
geom_point() +
labs(x = "Fitted", y = "Residuals")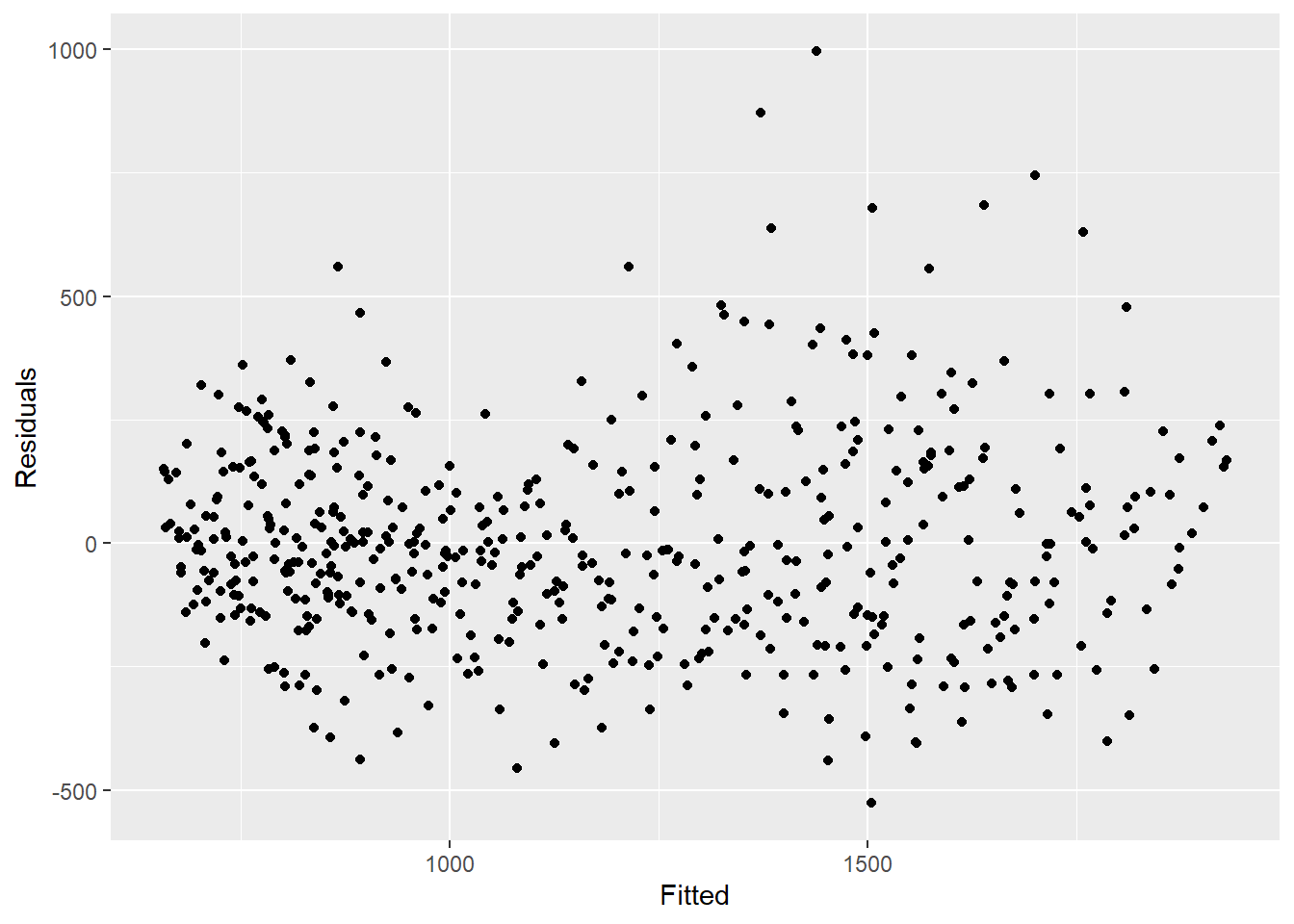
## artıkların otokorelasyonunu test edin
## H0, artıkların beyaz gürültü (yani rastgele) olmasıdır
## bağımsızlık testi
## p değeri anlamlıysa rastgele değil
Box.test(observed$resid, type = "Ljung-Box")##
## Box-Ljung test
##
## data: observed$resid
## X-squared = 346.64, df = 1, p-value < 2.2e-16
##
## Box-Ljung test
##
## data: observed$resid
## X-squared = 346.64, df = 1, p-value < 2.2e-1623.6 İki zaman serisinin ilişkisi
Burada, campylobacter vaka sayılarını açıklamak için hava durumu verilerini (özellikle sıcaklık) kullanmayı inceliyoruz.
Veritabanlarının birleştirilmesi
Hafta değişkenini kullanarak veri tabanlarımızı birleştirebiliriz. Birleştirme hakkında daha fazla bilgi için birleştirme ilgili el kitabı bölümüne bakın. joining.
Tanımlayıcı analizler
Görünür bir ilişki olup olmadığını görmek için önce verilerinizi çizin. Aşağıdaki grafik, iki değişkenin mevsimselliği yönünden görünür bir ilişki olduğunu ve sıcaklığın vaka sayısından birkaç hafta önce zirveye ulaştığını göstermektedir. Verilerin pivotlanması hakkında daha fazla bilgi için, verilerin pivotlanmasıyla ilgili el kitabı bölümüne bakın. pivoting data.
counts %>%
## sadece ilgilendiğimiz değişkenler veritabanında kalır
select(epiweek, case_int, t2m) %>%
## verilerinizi uzun formatta değiştirin
pivot_longer(
## anahtar olarak epiweek'i kullanın
!epiweek,
## sütun adlarını yeni "ölçü" sütununa taşıyın
names_to = "measure",
## hücre değerlerini yeni "değerler" sütununa taşı
values_to = "value") %>%
## yukarıdaki veritabanından bir grafik oluşturun
## epiweek'i x ekseninde ve değerler (sayılar/santigrat) y ekseninde grafik çizin
ggplot(aes(x = epiweek, y = value)) +
## sıcaklık ve vaka sayıları için ayrı bir grafik oluşturun
## kendi y eksenlerini ayarlamalarına izin verin
facet_grid(measure ~ ., scales = "free_y") +
## ikisini de bir çizgi grafiği olarak çizin
geom_line()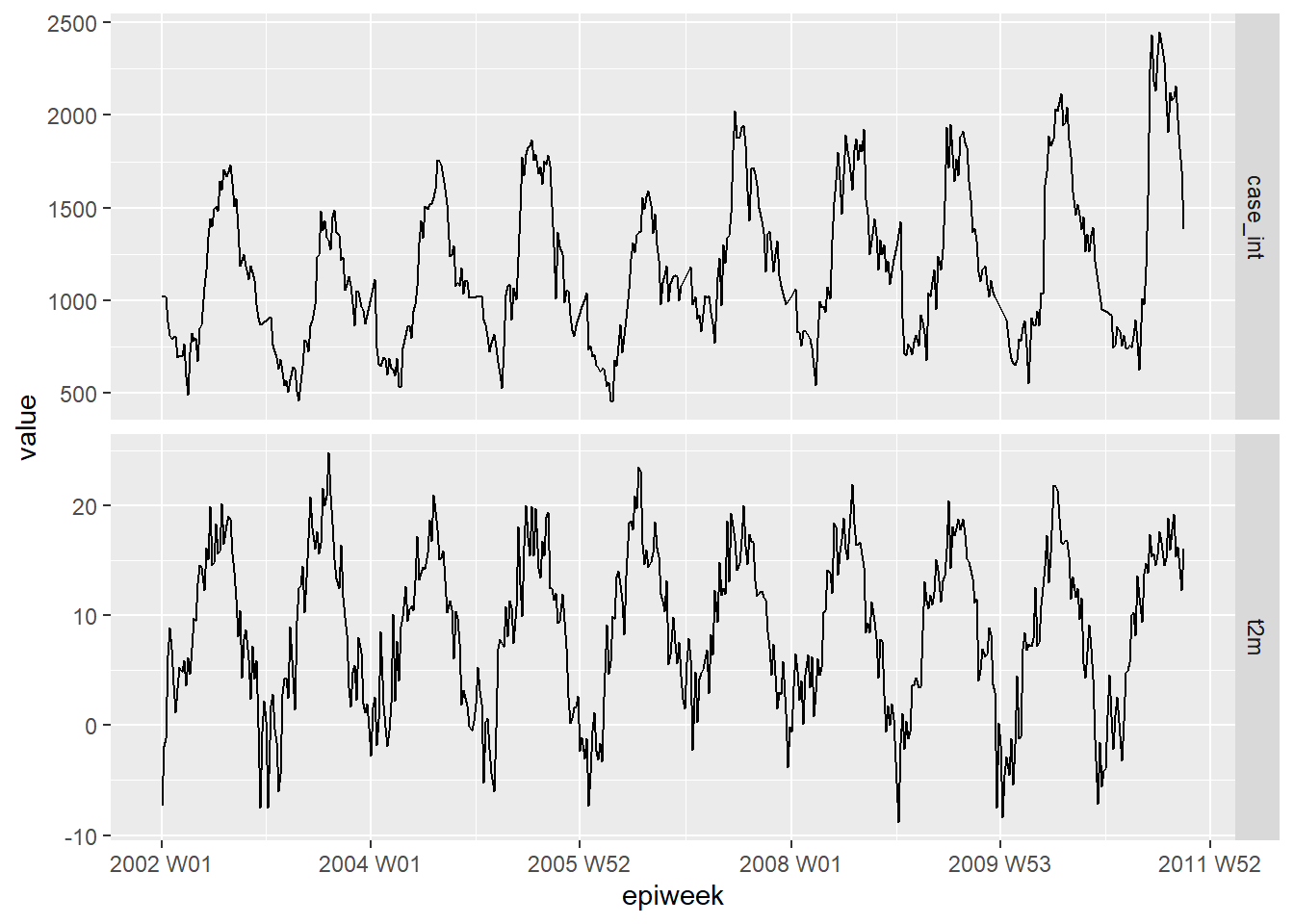
Gecikmeler ve çapraz korelasyonlar
Vakalar ve sıcaklık arasında hangi haftaların en çok ilişkili olduğunu test etmek çapraz korelasyon uygulanır. feasts paketinden çapraz korelasyon fonksiyonunu (CCF()) kullanabiliriz. Ayrıca autoplot() fonksiyonunu kullanarak (arrange kullanmak yerine) grafik oluşturabilirsiniz.
counts %>%
## interpolasyonlu sayımlar ve sıcaklık arasındaki çapraz korelasyonu hesaplayın
CCF(case_int, t2m,
## maksimum gecikmeyi 52 hafta olarak ayarlayın
lag_max = 52,
## korelasyon katsayısını hesaplayın
type = "correlation") %>%
## korelasyon katsayısını azalan biçimde dizin
## en yüksek ilişkili gecikmeleri göster
arrange(-ccf) %>%
## sadece ilk 10'u gösterin
slice_head(n = 10)## # A tsibble: 10 x 2 [1W]
## lag ccf
## <cf_lag> <dbl>
## 1 -4W 0.749
## 2 -5W 0.745
## 3 -3W 0.735
## 4 -6W 0.729
## 5 -2W 0.727
## 6 -7W 0.704
## 7 -1W 0.695
## 8 -8W 0.671
## 9 0W 0.649
## 10 47W 0.638Burada 4 haftalık bir gecikmenin en yüksek oranda ilişkili olduğunu görüyoruz, bu nedenle regresyonumuza dahil etmek için gecikmeli bir sıcaklık değişkeni oluşturuyoruz.
TEHLİKE: Gecikmeli sıcaklık değişkenindeki verilerin ilk dört haftasının eksik olduğunu (NA) unutmayın - çünkü veri almak için önceki dört hafta mevcut değildi. Bu veri tabanını trending predict() fonksiyonuyla kullanmak için, daha aşağıda predict() fonksiyonu içinde simulate_pi = FALSE argümanını kullanmamız gerekir. Simulate (simüle et) seçeneğini kullanmak istediysek, aşağıdaki kod parçasına drop_na(t2m_lag4) terimini ekleyerek eksikleri çıkararak yeni bir veri tabanı olarak kaydetmeliyiz.
İki değişkenli negatif binomial
Daha önce gösterildiği gibi negatif bir binom regresyon modeli geliştiriyoruz. Bu sefer modelimize dört hafta gecikmeli sıcaklık değişkenini ekliyoruz.
UYARI: predict() değişkeninde simulate_pi = FALSE kullanımına dikkat edin. Bunun nedeni, trending’in varsayılan davranışının bir tahmin aralığı oluşturmak için ciTools paketini kullanmasıdır. NA (eksik) sayılar varsa bu paket çalışmaz ve daha granüler aralıklar üretir. Ayrıntılar için ?trending::predict.trending_model_fit’e bakın.
## uydurmak istediğiniz modeli tanımlayın (negatif binom)
model <- glm_nb_model(
## ilgilenilen çıktı olarak vaka sayısını belirleyin
case_int ~
## trendi hesaba katmak için epiweek'i kullanın
epiweek +
## mevsimselliği hesaba katmak için fourier terimlerini kullanın
fourier +
## dört hafta gecikmeli sıcaklık değişkenini kullanın
t2m_lag4
)
## sayım veri tabanını kullanarak uyumlu modelinizi oluşturun
fitted_model <- trending::fit(model, data.frame(counts))
## güven aralıklarını ve tahmin aralıklarını hesaplayın
observed <- predict(fitted_model, simulate_pi = FALSE)Tek tek terimleri araştırmak için, get_model() fonksiyonunu kullanarak orijinal negatif binom regresyonunu trend formatından çıkarabilir sonrasında üstel tahminleri ve ilişkili güven aralıklarını elde etmek için broom paketi tidy() işlevine iletebiliriz. Bu bize, trend ve mevsimsellik için kontrol edildikten sonra gecikmeli sıcaklığın vaka sayılarına benzer olduğunu ve vaka sayılarıyla anlamlı olarak ilişkili (tahmini ~ 1) olduğunu gösterir. Bu sonuç , gecikmeli sıcaklık değişkeninin gelecekteki vaka sayılarını tahmin etmek için iyi bir değişken olabileceğini düşündürmektedir (iklim tahminleri zaten hazır olduğu için).
fitted_model %>%
## orijinal negatif binom regresyonunu elde edin
get_model() %>%
## sonuçların tidy veri çerçevesini elde edin
tidy(exponentiate = TRUE,
conf.int = TRUE)## # A tibble: 5 × 7
## term estimate std.error statistic p.value conf.low conf.high
## <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 (Intercept) 339. 0.108 53.8 0 274. 419.
## 2 epiweek 1.00 0.00000774 10.9 8.13e-28 1.00 1.00
## 3 fourierS1-52 0.752 0.0214 -13.3 1.84e-40 0.721 0.784
## 4 fourierC1-52 0.823 0.0200 -9.78 1.35e-22 0.791 0.855
## 5 t2m_lag4 1.01 0.00269 2.48 1.30e- 2 1.00 1.01Modelin hızlı görsel incelemesi, modelin gözlemlenen vaka sayılarını tahmin etmede daha iyi bir iş çıkarabileceğini gösterir.
## regresyonunuzu çiz
ggplot(data = observed, aes(x = epiweek)) +
## model tahmini için bir satır ekleyin
geom_line(aes(y = estimate),
col = "Red") +
## tahmin aralıkları için bir bant ekleyin
geom_ribbon(aes(ymin = lower_pi,
ymax = upper_pi),
alpha = 0.25) +
## gözlemlenen vaka sayılarınız için bir satır ekleyin
geom_line(aes(y = case_int),
col = "black") +
## geleneksel bir grafik oluşturun (siyah eksenli ve beyaz arka planlı)
theme_classic()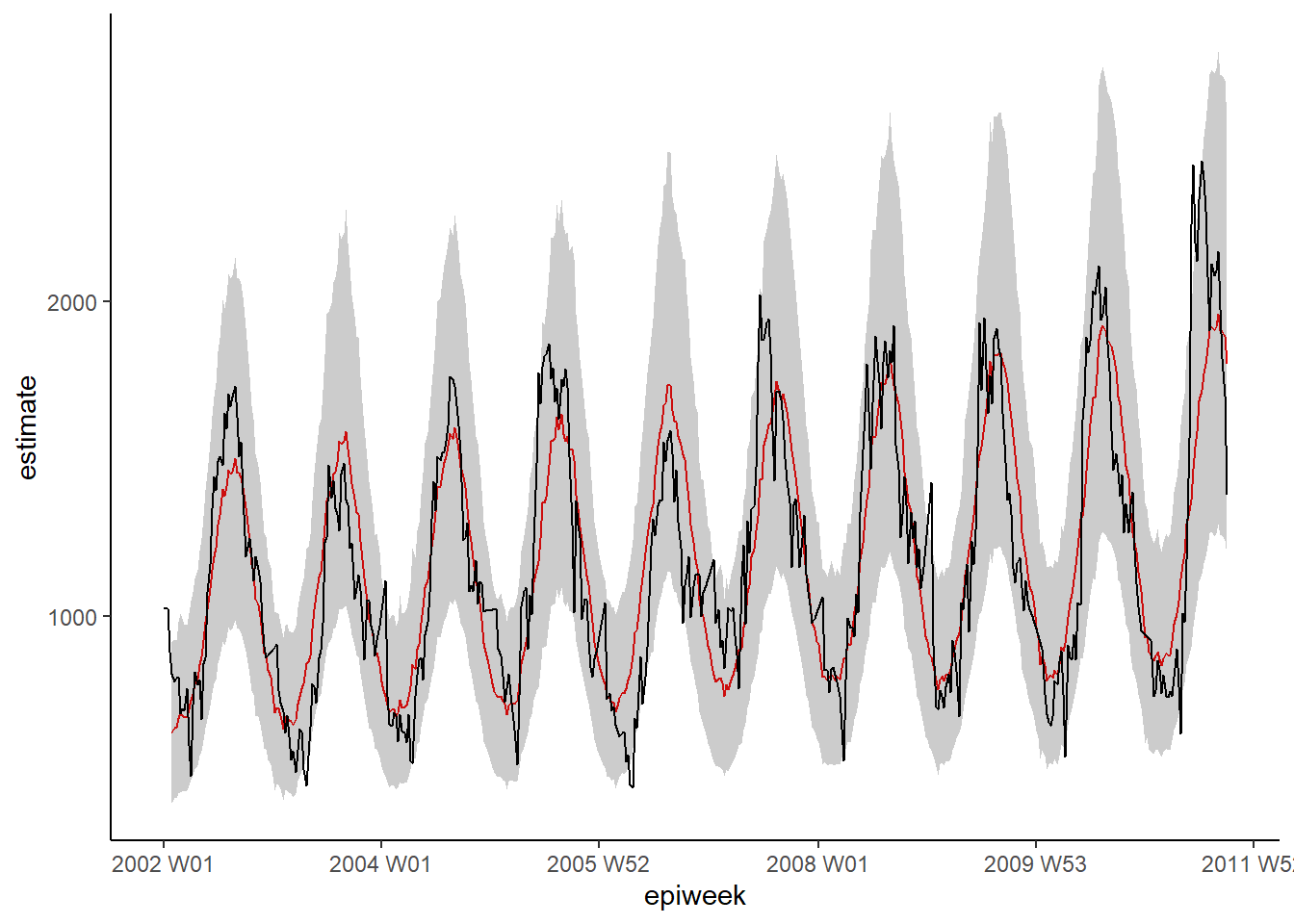
Rezidüeller (Artıklar)
Modelimizin gözlemlenen verilerle ne kadar uyumlu olduğunu görmek için artıkları tekrar araştırırız. Buradaki sonuçlar ve yorum önceki regresyona benzer, bu nedenle sıcaklık değişkenini içermeyen daha basit bir modele bağlı kalmak daha uygun olabilir.
## artıkları hesapla
observed <- observed %>%
mutate(resid = case_int - estimate)
## artıklar zaman içinde oldukça sabit mi (değilse: salgın? uygulamada değişiklik?)
observed %>%
ggplot(aes(x = epiweek, y = resid)) +
geom_line() +
geom_point() +
labs(x = "epiweek", y = "Residuals")## Warning: Removed 4 rows containing missing values (`geom_line()`).## Warning: Removed 4 rows containing missing values (`geom_point()`).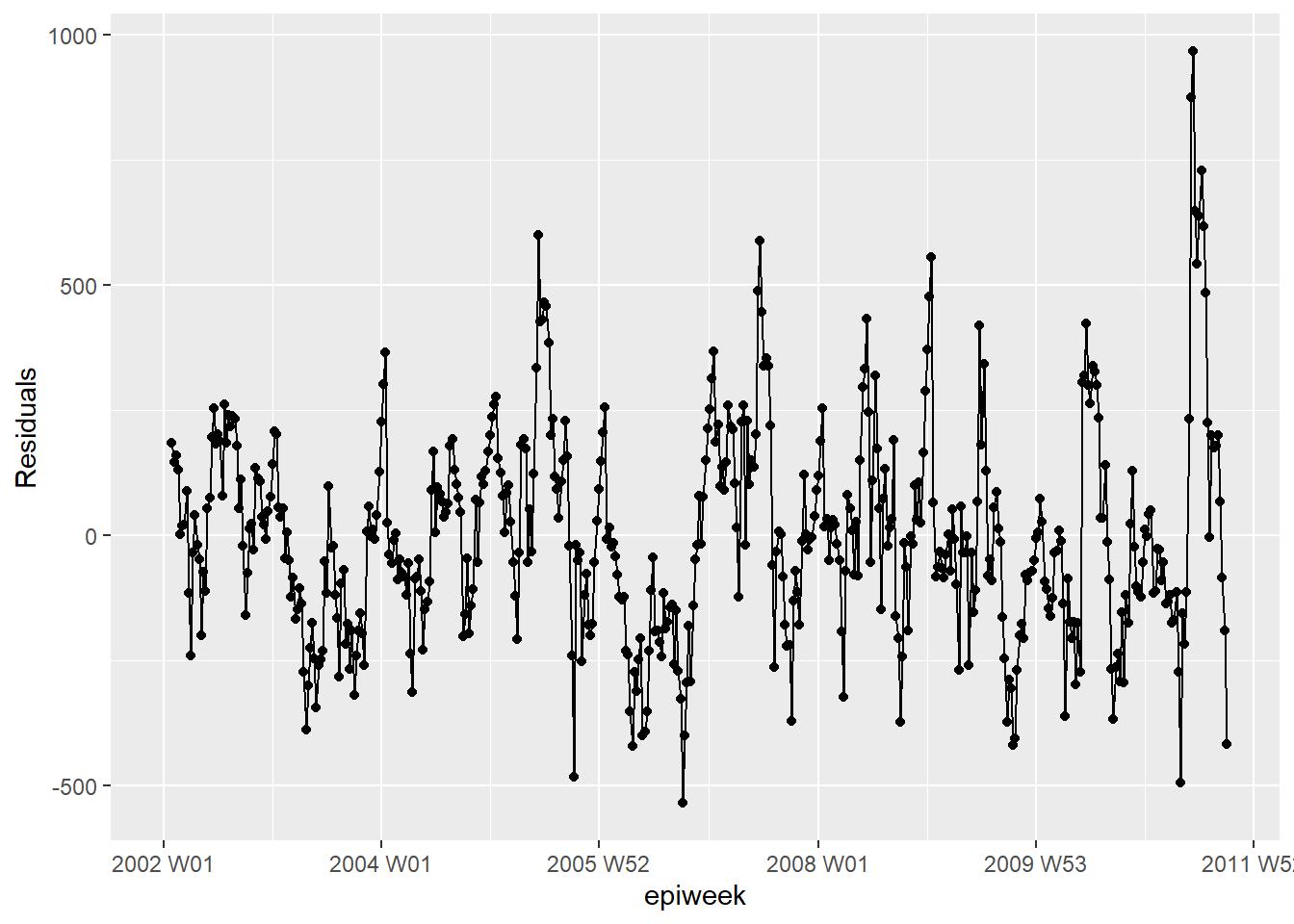
## Uyarı: Eksik değerler içeren 4 satır kaldırıldı (geom_path).
## artıklarda otokorelasyon var mı (hatanın bir motifi var mı?)
observed %>%
as_tsibble(index = epiweek) %>%
ACF(resid, lag_max = 52) %>%
autoplot()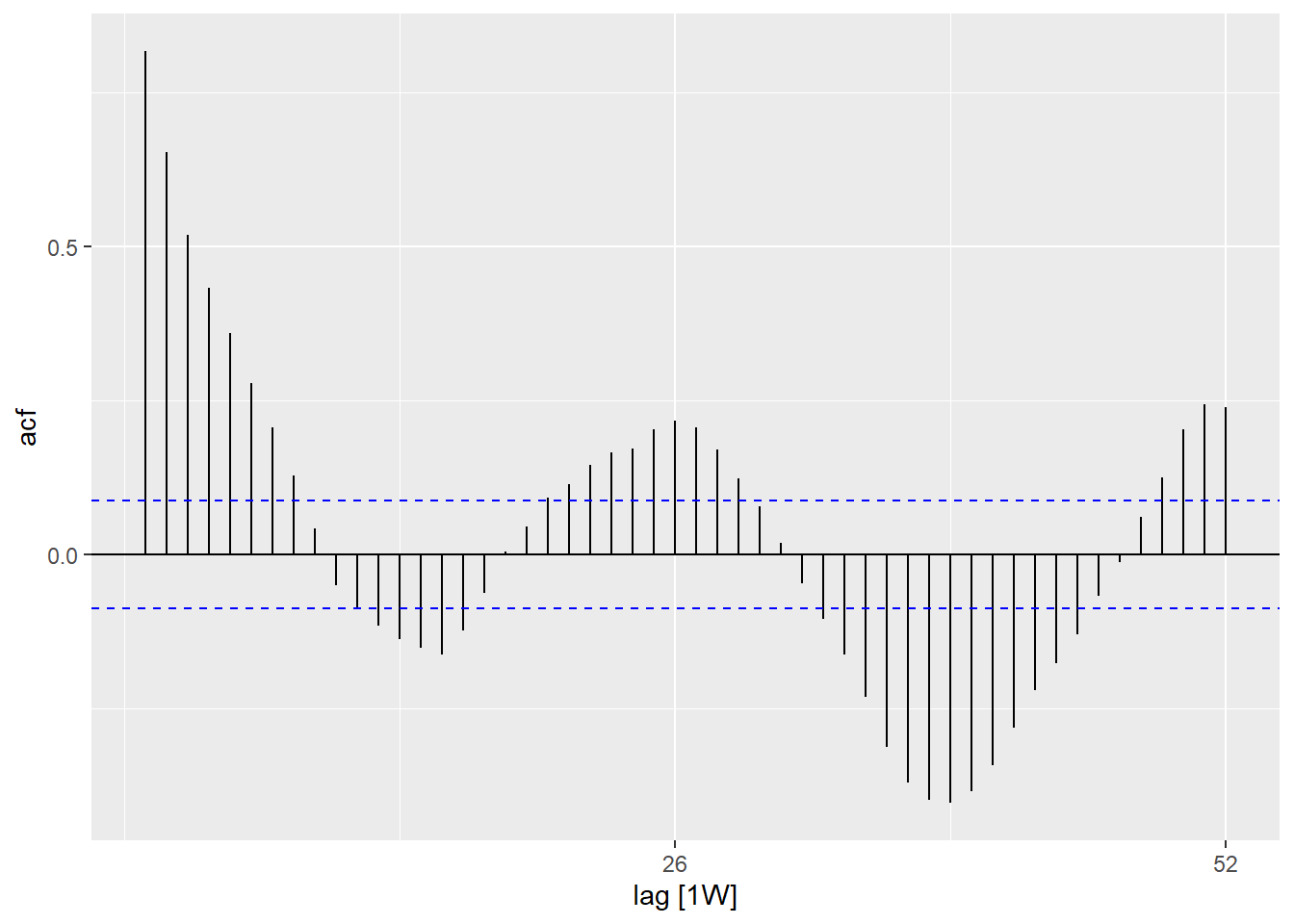
## artıklar normal dağılım gösteriyor mu (tahminin altında mı yoksa üzerinde mi?)
observed %>%
ggplot(aes(x = resid)) +
geom_histogram(binwidth = 100) +
geom_rug() +
labs(y = "count") ## Warning: Removed 4 rows containing non-finite values (`stat_bin()`).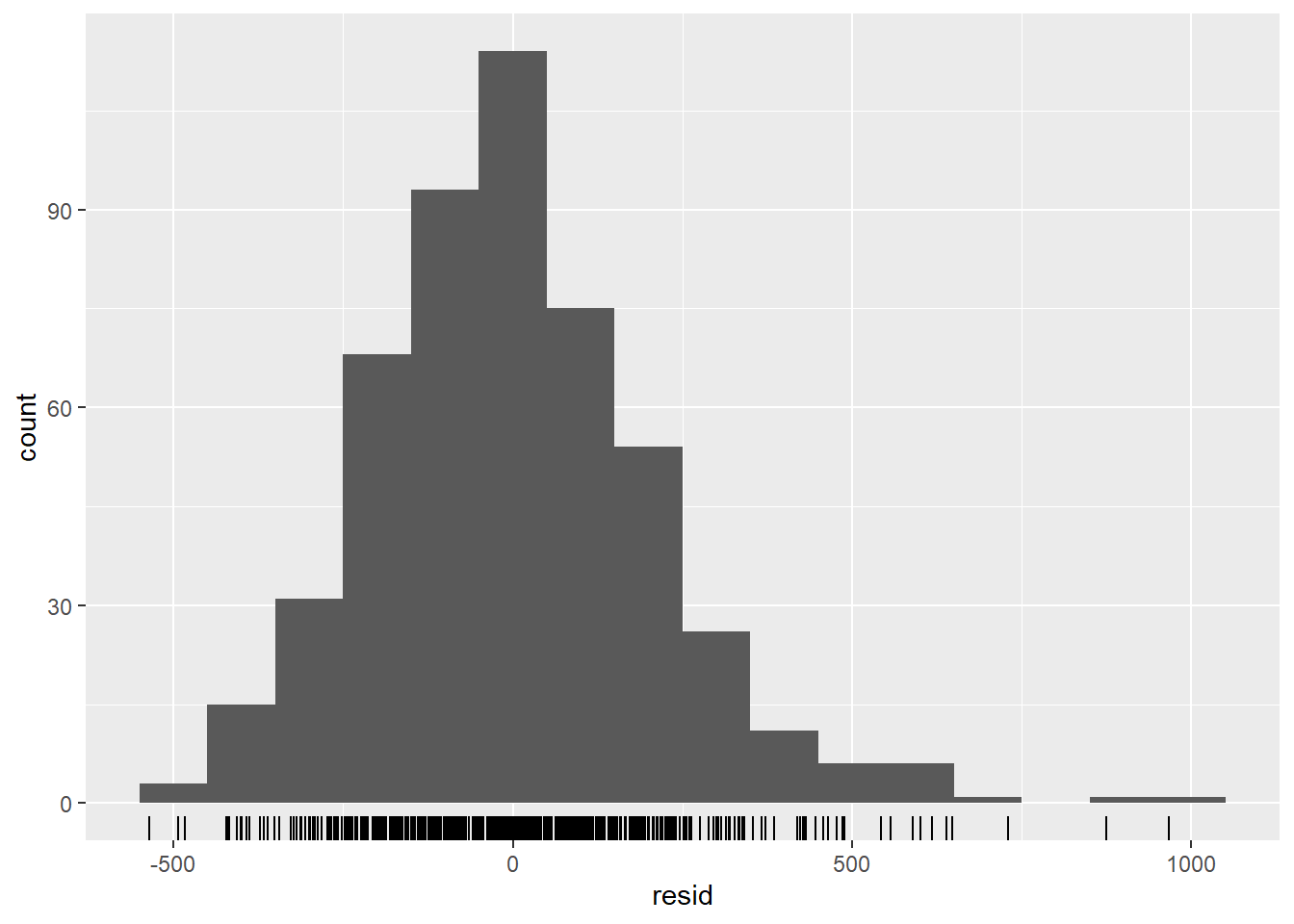
## Uyarı: Sonlu olmayan değerler içeren 4 satır kaldırıldı (stat_bin).
## gözlemlenen sayıları artıklarıyla karşılaştırın
## ayrıca tekrarlama olmamalı
observed %>%
ggplot(aes(x = estimate, y = resid)) +
geom_point() +
labs(x = "Fitted", y = "Residuals")## Warning: Removed 4 rows containing missing values (`geom_point()`).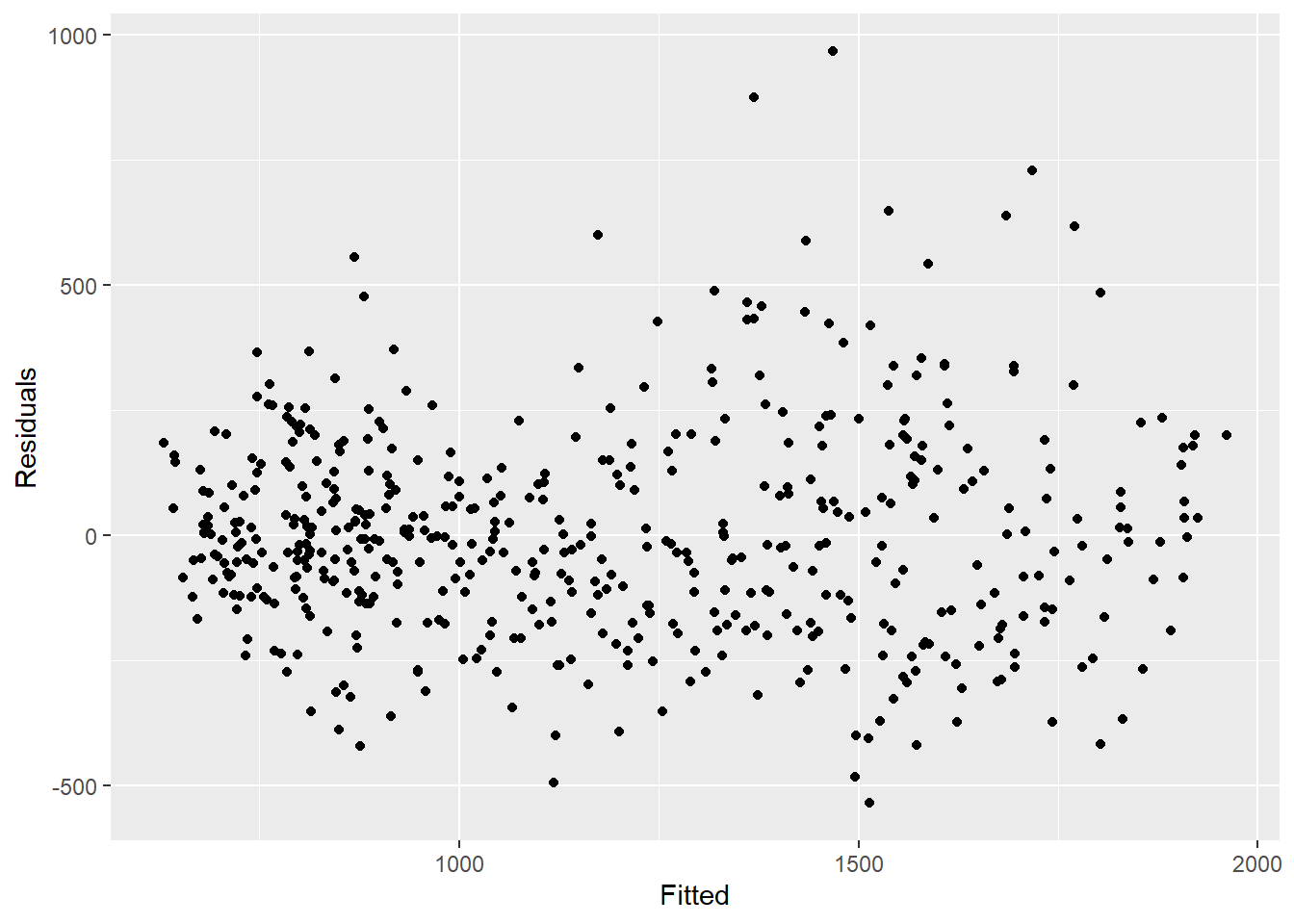
## Uyarı: Eksik değerler içeren 4 satır kaldırıldı (geom_point).
## artıkların otokorelasyonunu test edin
## H0, artıkların beyaz gürültü (yani rastgele) olmasıdır
## bağımsızlık testi
## p değeri anlamlıysa rastgele değildir
Box.test(observed$resid, type = "Ljung-Box")##
## Box-Ljung test
##
## data: observed$resid
## X-squared = 339.52, df = 1, p-value < 2.2e-1623.7 Salgınların tespit edilmesi
Burada salgınları tespit etmek için iki (benzer) yöntem göstereceğiz. İlki, yukarıdaki bölümler üzerine kuruludur. Regresyonları önceki yıllara uydurmak için trending paketini kullanıyoruz ve ardından gelecek yıl görmeyi umduğumuzu değerleri tahmin ediyoruz. Gözlenen sayımlar beklediğimizin üzerindeyse, bir salgın olduğunu gösterebilir. İkinci yöntem benzer ilkelere dayanmaktadır ancak aşırı değerlerin (aberration) tespiti için bir dizi farklı algoritmaya sahip surveillance paketini kullanır.
UYARI: Normalde, mevcut yılla ilgilenirsiniz (yalnızca mevcut haftaya kadar olan sayıları bildiğiniz için). Bu örnekte 2011’in 39. haftasındaymış gibi analiz yapıyoruz.
trending paketi
Bu yöntem için bir temel tanımlarız, genellikle yaklaşık 5 yıllık veri olmalıdır). Temel veriler için bir regresyon modeli oluştururuz ve ardından bunu gelecek yıl için tahmin elde etmek için kullanırız.
Kesim (eşik) tarihi
Tarihlerinizi tek bir yerde tanımlamak ve ardından bunları kodunuzun geri kalanında kullanmak daha kolaydır.
Burada bir başlangıç tarihi (gözlemlerimizin başladığı tarih) ve bir bitiş tarihi (temel dönemimizin sonu olan- ve tahmine ne zaman başlayacağımızın tarihi) tanımlarız. ~Ayrıca ilgilendiğimiz yılda (tahmin edeceğimiz yılda) kaç hafta olduğunu da tanımlarız ~. Ayrıca, temel kesim tarihimiz ile tahmin etmek istediğimiz bitiş tarihi arasında kaç hafta olduğunu da tanımlarız.
NOT: Bu örnekte, şu anda Eylül 2011’in sonundaymış gibi davranıyoruz (“2011 W39”).
## başlangıç tarihini tanımlayın (gözlemlerin başladığı zaman)
start_date <- min(counts$epiweek)
## bir bitiş haftası tanımlayın (temel verinin sonu, tahmin döneminin başlangıcı)
cut_off <- yearweek("2010-12-31")
## ilgilenilen son tarihi tanımlayın (yani tahminin sonu)
end_date <- yearweek("2011-12-31")
## ilgilenilen dönemde (yıl) kaç hafta bulun
num_weeks <- as.numeric(end_date - cut_off)Satır eklemek
Tidyverse formatında tahmin yapabilmek için, veri setimizde doğru sayıda satıra, yani yukarıda tanımlanan end_date tarihine kadar her hafta için bir satıra ihtiyacımız var. Aşağıdaki kod, bu satırları bir gruplama değişkenine göre eklemenize izin verir - örneğin, bir veri tabanında birden fazla ülkemiz varsa, ülkeye göre gruplayabilir ve ardından her biri için uygun satırlar ekleyebiliriz. tsibble’ın group_by_key() fonksiyonu , bu gruplandırmayı yapmamıza ve ardından gruplanmış verileri dplyr fonkisyonlarına, group_modify() ve add_row()’a geçirmemize izin verir. Ardından, verilerde mevcut olan maksimum hafta değerinden bir sonraki hafta ile bitiş haftası arasındaki hafta sırasını belirleriz.
Fourier terimleri
Fourier terimlerimizi yeniden tanımlamamız gerekiyor - çünkü bu terimleri temel tarihe uydurmak ve gelecek yıl için ekstrapole etmek istiyoruz. Bunu yapmak için fourier() fonkisyonundan iki çıktı listesini bir araya getirmemiz gerekiyor; ilki temel veriler içindir ve ikincisi ilgilenilen yılı (h argümanını tanımlayarak) tahmin eder.
Not: Satırları bağlamak için rbind() (tidyverse’teki bind_rows yerine) kullanmalıyız, çünkü fourier sütunları bir liste biçimindedir (yani ayrı ayrı adlandırılmaz).
## fourier terimlerini tanımlayın (sincos)
counts <- counts %>%
mutate(
## 2010 kesim tarihinden önceki ve sonraki haftalar için fourier terimlerini birleştirin
## (yani 2011 fourier terimleri tahmin edilmektedir)
fourier = rbind(
## önceki yıllar için fourier terimlerini getirin
fourier(
## sadece 2011'den önceki satırları tutun
filter(counts,
epiweek <= cut_off),
## bir dizi sin cos terimini dahil et
K = 1
),
## 2011 için fourier terimlerini tahmin edin (temel verileri kullanarak)
fourier(
## sadece 2011'den önceki satırları tutun
filter(counts,
epiweek <= cut_off),
## bir dizi sin cos terimini dahil et
K = 1,
## 52 hafta sonrasını tahmin et
h = num_weeks
)
)
)Verinin bölünmesi ve uyumlu regresyon
Şimdi veri setimizi temel periyoda (tahminin başladığı zamana kadarki veri) ve tahmin periyoduna bölmemiz gerekiyor. Bu, group_by()’den sonra dplyr group_split() fonkisyonu kullanılarak yapılır ve biri kesme işleminden önce diğeri sonra olmak üzere iki veri çerçevesi içeren bir liste oluşturur.
Daha sonra veri kümelerini listeden çıkarmak için purrr paketi pluck() fonksiyonunu kullanırız (bu fonksiyon köşeli parantez kullanmaya eşdeğer, örneğin dat[[1]] gibi ) ve daha sonra modelimizi temel verilere uydurabilir ve ardından kesintiden sonra ilgilendiğimiz veri için predict() fonksiyonunu kullanabiliriz.
Purrr hakkında daha fazla bilgi edinmek için Yineleme (iterasyon), döngüler ve listeler sayfasına bakın.
UYARI: predict() fonksiyonunda simulate_pi = FALSE argümanının kullanımına dikkat edin. Bunun nedeni, trend oluşturmanın varsayılan davranışının bir tahmin aralığını oluşturmak için ciTools paketinin kullanılmasıdır. NA (eksik) değerler varsa bu fonksiyon çalışmaz ve ayrıca daha granüler aralıklar üretir. Ayrıntılar için ?trending::predict.trending_model_fit’e bakın.
# uyum ve tahmin için bölünmüş veriler
dat <- counts %>%
group_by(epiweek <= cut_off) %>%
group_split()
## uydurmak istediğiniz modeli tanımlayın (negatif binom)
model <- glm_nb_model(
## set number of cases as outcome of interest
case_int ~
## use epiweek to account for the trend
epiweek +
## use the furier terms to account for seasonality
fourier
)
# Uyum ve tahmin için hangi verilerin kullanılacağını tanımlayın
fitting_data <- pluck(dat, 2)
pred_data <- pluck(dat, 1) %>%
select(case_int, epiweek, fourier)
# modeli uydurun
fitted_model <- trending::fit(model, data.frame(fitting_data))
# Uydurulan veriler için sınır ve tahmin değerleri elde edin
observed <- fitted_model %>%
predict(simulate_pi = FALSE)
# tahmin için kullanılacak veri ile tahmin
forecasts <- fitted_model %>%
predict(data.frame(pred_data), simulate_pi = FALSE)
## temel ve tahmin edilmiş veri tabanlarını birleştirin
observed <- bind_rows(observed, forecasts)Daha önce olduğu gibi modelimizi ggplot ile görselleştirebiliriz. %95 tahmin aralığının üzerindeki gözlemlenen değerleri dikkat çekmek amacıyla kırmızı noktalarla vurgularız. Bu sefer tahminin başladığı tarihi etiketlemek için dikey bir çizgi de ekliyoruz.
## regresyonunuzu görselleştirin
ggplot(data = observed, aes(x = epiweek)) +
## model tahmini için bir satır ekleyin
geom_line(aes(y = estimate),
col = "grey") +
## tahmin aralıkları için bir bant ekleyin
geom_ribbon(aes(ymin = lower_pi,
ymax = upper_pi),
alpha = 0.25) +
## gözlemlenen vaka sayılarınız için bir çizgi grafiği ekleyin
geom_line(aes(y = case_int),
col = "black") +
## beklenenin üzerinde gözlenen sayımlar için saçılım grafiği
geom_point(
data = filter(observed, case_int > upper_pi),
aes(y = case_int),
colour = "red",
size = 2) +
## tahminin nerede başladığını göstermek için dikey çizgi ve etiket ekleyin
geom_vline(
xintercept = as.Date(cut_off),
linetype = "dashed") +
annotate(geom = "text",
label = "Forecast",
x = cut_off,
y = max(observed$upper_pi) - 250,
angle = 90,
vjust = 1
) +
## geleneksel bir grafik oluşturun (siyah eksenli ve beyaz arka planlı)
theme_classic()## Warning: Removed 13 rows containing missing values (`geom_line()`).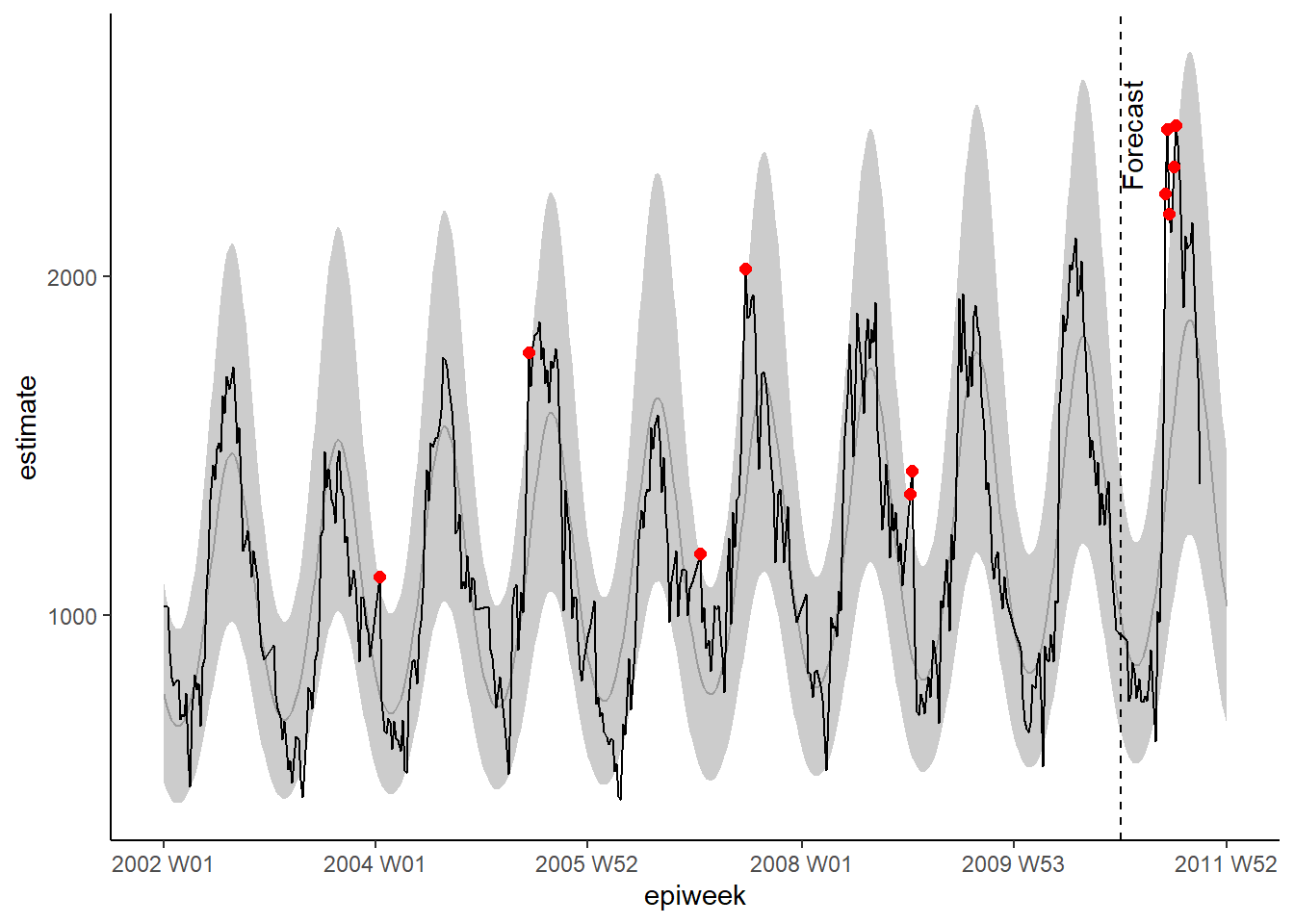
## Uyarı: Eksik değerler içeren 13 satır kaldırıldı(geom_path).Öngörünün doğrulanması (validasyonu)
Artıkları incelemenin ötesinde, modelinizin gelecekteki durumları tahmin etmede ne kadar iyi olduğunu araştırmak önemlidir. Bu size eşik uyarılarınızın ne kadar güvenilir olduğu konusunda fikir verir.
Doğrulamanın geleneksel yolu, önceki yılı ne kadar iyi tahmin edebileceğinizi görmektir (çünkü “şimdiki yıl” için sayıları henüz bilmiyorsunuz). Örneğin, veri setimizde 2010’u tahmin etmek için 2002’den 2009’a kadar olan verileri kullanıp sonra bu tahminlerin ne kadar doğru olduğunu ölçerdik. Ardından modeli 2010 verilerini içerecek şekilde uygulayıp 2011 sayılarını tahmin etmek için kullanın.
Aşağıdaki şekilde Hyndman ve arkadaşlarının “Tahmin ilkeleri ve uygulaması” bölümünde görülebileceği gibi. Hyndman et al “Forecasting principles and practice”.
 Görsel yazarlardan alınan izinle yeniden oluşturulmuştur
Görsel yazarlardan alınan izinle yeniden oluşturulmuştur
Bu yöntemin dezavantajı, elinizdeki tüm verileri kullanmamanız ve modelin tahmin için kullandığınız son model olmamasıdır.
Bir alternatif, çapraz doğrulama adı verilen bir yöntem kullanmaktır. Bu senaryoda, bir yıl sonrasını tahmin etmek için birden fazla modele uydurabilmek için mevcut tüm verileri kaydırırsınız. Aynı makaleden [Hyndman ve arkadaşlarına ait metninden]((https://otexts.com/fpp3/) alınan aşağıdaki şekilde görüldüğü gibi, her modelde giderek daha fazla veri kullanıyorsunuz.
Örneğin, ilk model 2003’ü tahmin etmek için 2002’yi kullanıyor , ikincisi 2004’ü tahmin etmek için 2002 ve 2003’ü kullanır, vb.
 Görsel yazarlardan alınan izinle yeniden oluşturulmuştur
Görsel yazarlardan alınan izinle yeniden oluşturulmuştur
Aşağıda, veri kümeleri üzerinde döngü işlemini uygulamak için purrr paketinden map() fonksiyonunu kullanıyoruz. Daha sonra, doğruluk ölçülerini hesaplama amacıyla yardstick paketini kullanmak için elde ettiğimiz tahminleri bir veri tabanına aktarır ve orijinal vaka sayılarıyla birleştiririz. Aşağıda belirtilen dört ölçüyü hesaplıyoruz: Kök Ortalama Kare Hatası (RMSE), Ortalama mutlak hata (MAE), Ortalama mutlak ölçekli hata (MASE), Ortalama mutlak yüzde hatası (MAPE).
UYARI: predict() fonksiyonunda simulate_pi = FALSE argümanının kullanımına dikkat edin. Bunun nedeni, trend oluşturmanın varsayılan davranışının bir tahmin aralığını oluşturmak için ciTools paketinin kullanılmasıdır. NA (eksik) değerler varsa bu fonksiyon çalışmaz ve ayrıca daha granüler aralıklar üretir. Ayrıntılar için ?trending::predict.trending_model_fit’e bakın.
## Çapraz doğrulama: Kayan pencereye dayalı olarak önümüzdeki haftaları tahmin etme
## 52 haftalık pencerelerde kaydırarak verilerinizi genişletin (öncesi + sonrası)
## 52 hafta sonrasını tahmin etmek
## (daha uzun gözlem zincirleri oluşturur- eski verileri tutar)
## kaydırmak istediğiniz pencereyi tanımlayın
roll_window <- 52
## tahmin etmek istediğiniz haftaları tanımlayın
weeks_ahead <- 52
## yinelenen, giderek daha uzun verilerden oluşan bir veri seti oluşturun
## her veri setini benzersiz bir kimlikle etiketleyin
## yalnızca ilgilenilen yıldan önceki vakaları kullanın (yani 2011)
case_roll <- counts %>%
filter(epiweek < cut_off) %>%
## sadece hafta ve vaka sayısı değişkenlerini veri tabanında tut
select(epiweek, case_int) %>%
## en son x sayıda gözlemi uzaklaştırın
## önümüzdeki kaç haftayı tahmin etmek istediğinize bağlı olarak
## (aksi takdirde "bilinmeyen" için tahmin yapılacaktır)
slice(1:(n() - weeks_ahead)) %>%
as_tsibble(index = epiweek) %>%
## gruplandırma kimliği oluşturmak amacıyla x’teki her hafta için kaydırın
## kaydırma pencerisinin özgüllüğüne bağlı
stretch_tsibble(.init = roll_window, .step = 1) %>%
## önceki vakası yokmuş gibi ilk çifti uzaklaştırın drop the first couple
filter(.id > roll_window)
## benzersiz veri setlerinin her biri için aşağıdaki kodu çalıştırın
forecasts <- purrr::map(unique(case_roll$.id),
function(i) {
## yalnızca mevcut katlantının uygun olmasını sağlayın
mini_data <- filter(case_roll, .id == i) %>%
as_tibble()
## tahmin için boş bir veri seti oluşturun
forecast_data <- tibble(
epiweek = seq(max(mini_data$epiweek) + 1,
max(mini_data$epiweek) + weeks_ahead,
by = 1),
case_int = rep.int(NA, weeks_ahead),
.id = rep.int(i, weeks_ahead)
)
## tahmin verilerini orijinale ekleyin
mini_data <- bind_rows(mini_data, forecast_data)
## en son eksik olmayan sayım verilerine dayalı olarak kesim tarhini tanımlayın
cv_cut_off <- mini_data %>%
## sadece eksik olmayan satırları tutun
drop_na(case_int) %>%
## en son haftayı elde edin
summarise(max(epiweek)) %>%
## bir veri çerçevesinde olmayacak şekilde dışa aktar
pull()
## mini_data’yı tsibble haline getirin
mini_data <- tsibble(mini_data, index = epiweek)
## fourier terimlerini tanımlayın (sincos)
mini_data <- mini_data %>%
mutate(
## fourier terimlerini kesim tarihinden önceki ve sonraki haftaları kombine etmek için kullanın
fourier = rbind(
## önceki yıllardan fourier terimelerini getirin
forecast::fourier(
## sadece kesim öncesi satırları tutun
filter(mini_data,
epiweek <= cv_cut_off),
## bir sin cos terim setini dahil edin
K = 1
),
## takiben eden yıl için fourier terimlerini tahmin edin (temel veriyi kullanarak)
fourier(
## sadece kesim öncesi satırları tutun
filter(mini_data,
epiweek <= cv_cut_off),
## bir sin cos terim setini dahil edin
K = 1,
## 52 hafta sonrasını tahmin edin
h = weeks_ahead
)
)
)
# uyum ve tahmin için bölünmüş veriler
dat <- mini_data %>%
group_by(epiweek <= cv_cut_off) %>%
group_split()
## uydurmak istediğiniz modeli tanımlayın (negatif binom)
model <- glm_nb_model(
## ilgilenilen çıktı olarak vaka sayısını ayarlayın
case_int ~
## trendi hesaba katmak için epiweek'i kullanın
epiweek +
## mevsimselliği hesaba katmak için fourier terimlerini kullanın
fourier
)
# Uyum ve tahmin için hangi verilerin kullanılacağını tanımlayın
fitting_data <- pluck(dat, 2)
pred_data <- pluck(dat, 1)
# modeli uydurun
fitted_model <- trending::fit(model, fitting_data)
# istenilen verilerle tahmin
forecasts <- fitted_model %>%
predict(data.frame(pred_data), simulate_pi = FALSE) %>%
## sadece hafta ve tahmin değerini tutun
select(epiweek, estimate)
}
)
## listeyi tüm tahminlerle birlikte bir veri çerçevesine dönüştürün
forecasts <- bind_rows(forecasts)
## tahmin ile gözlenen verileri birleştirin
forecasts <- left_join(forecasts,
select(counts, epiweek, case_int),
by = "epiweek")
## {yardstick} ile ölçümlerin hesabı
## RMSE: Kök Ortalama Kare Hatası
## MAE: Ortalama mutlak hata
## MASE: Ortalama mutlak ölçekli hata
## MAPE: Ortalama mutlak yüzde hatası
model_metrics <- bind_rows(
## tahmin edilen veri setinizde gözlemlenen ile tahmin edileni karşılaştırın
rmse(forecasts, case_int, estimate),
mae( forecasts, case_int, estimate),
mase(forecasts, case_int, estimate),
mape(forecasts, case_int, estimate),
) %>%
## yalnızca metrik türünü ve çıktısını tutun
select(Metric = .metric,
Measure = .estimate) %>%
## geniş formatta yapın, böylece satırları bağlayın
pivot_wider(names_from = Metric, values_from = Measure)
## metrik modeli getirin
model_metrics## # A tibble: 1 × 4
## rmse mae mase mape
## <dbl> <dbl> <dbl> <dbl>
## 1 252. 199. 1.96 17.3surveillance paketi
Bu bölümde, salgın tespit algoritmalarına dayalı uyarı eşikleri oluşturmak için surveillance paketini kullanıyoruz. Pakette birkaç farklı yöntem mevcuttur, ancak burada iki seçeneğe odaklanacağız. Ayrıntılar için, kullanılan algoritmaların uygulaması ve teorisi hakkındaki bu makalelere bakın. application ve theory.
İlk seçenek, iyileştirilmiş Farrington yöntemini kullanır. Bu yöntem, negatif bir binom glm’ye (trend dahil) uyum gösterir ve eşik değeri oluşturmak için geçmiş salgınları (aykırı değerler) düşük değer olarak kullanır.
İkinci seçenek glrnb yöntemini kullanır. Bu aynı zamanda negatif bir binom glm’ye (genelleştirilmiş doğrusal modele) de uyar, ancak trend ve fourier terimlerini içerir (burada tercih edilir). Regresyon, “kontrol ortalamasını” (~uyumlu değerleri) hesaplamak için kullanılır - daha sonra her hafta için ortalamada kayma olup olmadığını değerlendirmek için hesaplanmış bir genelleştirilmiş olabilirlik oranı (likelihood ratio) istatistiği kullanır. Her hafta için eşiğin önceki haftaları hesaba kattığını unutmayın, bu nedenle sürekli bir kayma varsa bir alarm tetiklenecektir. (Ayrıca her alarmdan sonra algoritmanın sıfırlandığını unutmayın)
surveillance paketiyle çalışmak için öncelikle çerçeveye sığacak bir “gözetim zaman serisi” nesnesi (sts() fonksiyonunu kullanarak) tanımlamamız gerekir.
## gözetim zaman serisi nesnesini tanımla
## not. popülasyon nesnesine bir payda ekleyebilirsiniz (bkz. ?sts)
counts_sts <- sts(observed = counts$case_int[!is.na(counts$case_int)],
start = c(
## yalnızca start_date tarihinden itibaren yılı tutmak için alt küme
as.numeric(str_sub(start_date, 1, 4)),
## yalnızca start_date tarihinden itibaren haftayı tutmak için alt küme
as.numeric(str_sub(start_date, 7, 8))),
## veri türünü tanımlayın (bu durumda haftalık)
freq = 52)
## dahil etmek istediğiniz hafta aralığını tanımlayın (ör. tahmin dönemi)
## not. sts nesnesi yalnızca kendilerine tanımlanmış hafta ya da yılları atamadan gözlemleri sayar
## bu nedenle uygun olanı tanımlamak için verilerimizi kullanırız
weekrange <- cut_off - start_dateDaha sonra Farrington yöntemi için parametrelerimizi bir liste halinde tanımlarız. Ardından farringtonFlexible() fonksiyonunu kullanarak algoritmayı çalıştırırız ve ardından bunu veri setimize dahil etmek için farringtonmethod@upperbound kullanarak bir uyarı eşiğini çıkarabiliriz. Farringtonmethod@alarm kullanarak her hafta için uyarı tetiklendiyse (eşiğin üzerindeyse) DOĞRU/YANLIŞ değerlerini çıkarmak da mümkündür.
## kontrolü tanımlayın
ctrl <- list(
## eşik için zaman periyodunu tanımlayın (yani 2011)
range = which(counts_sts@epoch > weekrange),
b = 9, ## temelden kaç yıl geriye
w = 2, ## haftalar halinde kaydırma penceresi
weightsThreshold = 2.58, ## geçmiş salgınların yeniden ağırlıklandırılması (geliştirilmiş noufaily yöntemi - orijinal öneriler 1)
## pastWeeksNotIncluded = 3, ## mevcut tüm haftaları kullanın (noufaily önerisi 26’yı çıkarın)
trend = TRUE,
pThresholdTrend = 1, ## 0.05 normally, however 1 is advised in the improved method (i.e. always keep)
thresholdMethod = "nbPlugin",
populationOffset = TRUE
)
## farrington’ın esnek yöntemini uygulayın
farringtonmethod <- farringtonFlexible(counts_sts, ctrl)
## orijinal veri setinde eşik adı verilen yeni bir değişken oluşturun
## farrington'dan gelen üst sınırı içerir
## not: bu yalnızca 2011'deki haftalar içindir (bu nedenle satırları alt kümelere ayırmanız gerekir)
counts[which(counts$epiweek >= cut_off &
!is.na(counts$case_int)),
"threshold"] <- farringtonmethod@upperboundDaha sonra sonuçları daha önce olduğu gibi ggplot’ta görselleştirebiliriz.
ggplot(counts, aes(x = epiweek)) +
## gözlemlenen vaka sayılarını bir çizgi olarak ekleyin
geom_line(aes(y = case_int, colour = "Observed")) +
## sapma algoritmasının üst sınırını ekleyin
geom_line(aes(y = threshold, colour = "Alert threshold"),
linetype = "dashed",
size = 1.5) +
## renkleri belirleyin
scale_colour_manual(values = c("Observed" = "black",
"Alert threshold" = "red")) +
## geleneksel bir grafik oluşturun (siyah eksenli ve beyaz arka planlı)
theme_classic() +
## lejantın (işaret tablosunun) başlığını kaldırın
theme(legend.title = element_blank())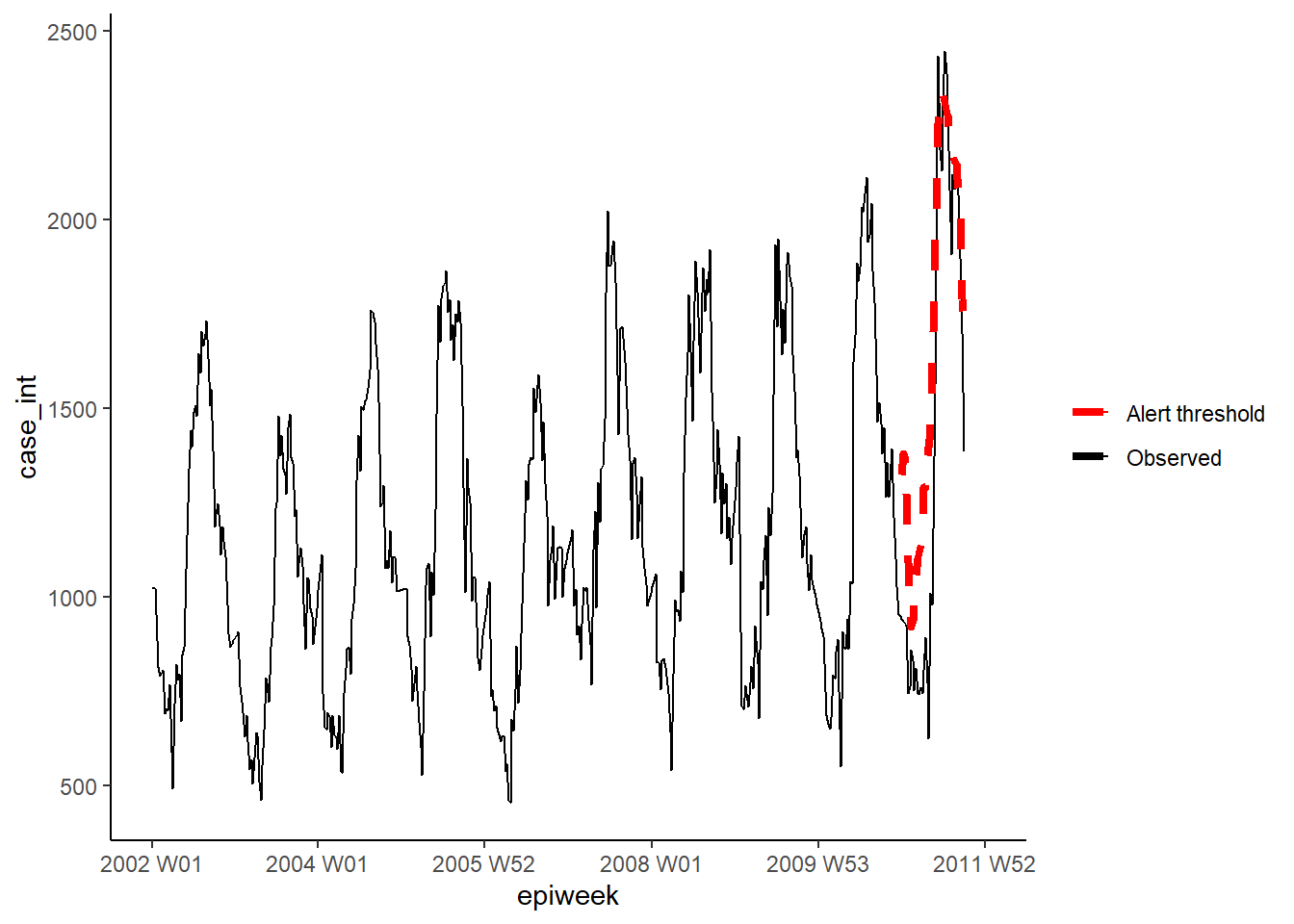
GLRNB yöntemi
Benzer şekilde GLRNB yöntemi için her bir parametremizi bir liste halinde tanımlıyoruz, ardından algoritmayı uygun hale getiriyoruz ve üst sınırları çıkarıyoruz.
UYARI: Bu yöntem, eşikleri hesaplamak için “kaba kuvvet” (önyüklemeye [bootstrapping’e] benzer) kullanır, bu nedenle uzun zaman alabilir!
Ayrıntılar için GLRNB gösterimi bakabilirsiniz.
## kontrol ayarlarını belirleyin
ctrl <- list(
## hangi zaman dilimi için eşik değer isteneceğini tanımlayın (yani 2011)
range = which(counts_sts@epoch > weekrange),
mu0 = list(S = 1, ## dahil edilecek fourier terimlerinin (harmonik) sayısı
trend = TRUE, ## trendin dahil edilip edilmeyeceği
refit = FALSE), ## her alarmdan sonra modelin yeniden uydurulup uydurulmayacağı
## cARL = GLR istatistiği için eşik (keyfi)
## 3 ~ yanlış pozitifleri en aza indirmek için orta yol
## 1, glm.nb'nin %99 tahmin aralığına uyar - zirvelerden sonraki değişikliklerle (uyarı için eşik düşürüldü)
c.ARL = 2,
# theta = log(1.5), ## salgın vakalarında %50 artışa eşittir
ret = "cases" ## vaka sayısı olarak üst sınıra dönüş eşiği
)
## glrnb yöntemini uygula
glrnbmethod <- glrnb(counts_sts, control = ctrl, verbose = FALSE)
## orijinal veri setinde eşik adı verilen yeni bir değişken oluşturun
## glrnb'den üst sınırı içeren
## not. bu yalnızca 2011'deki haftalar içindir (bu nedenle satırları alt kümelere ayırmanız gerekir)
counts[which(counts$epiweek >= cut_off &
!is.na(counts$case_int)),
"threshold_glrnb"] <- glrnbmethod@upperboundÇıktıları daha önce olduğu gibi görselleştirin.
ggplot(counts, aes(x = epiweek)) +
## gözlemlenen vaka sayılarını bir çizgi olarak ekleyin
geom_line(aes(y = case_int, colour = "Observed")) +
## sapma algoritmasının üst sınırına ekleyin
geom_line(aes(y = threshold_glrnb, colour = "Alert threshold"),
linetype = "dashed",
size = 1.5) +
## renkleri belirleyin
scale_colour_manual(values = c("Observed" = "black",
"Alert threshold" = "red")) +
## geleneksel bir grafik oluşturun (siyah eksenli ve beyaz arka planlı)
theme_classic() +
## lejantın (işaret tablosunun) başlığını kaldırın
theme(legend.title = element_blank())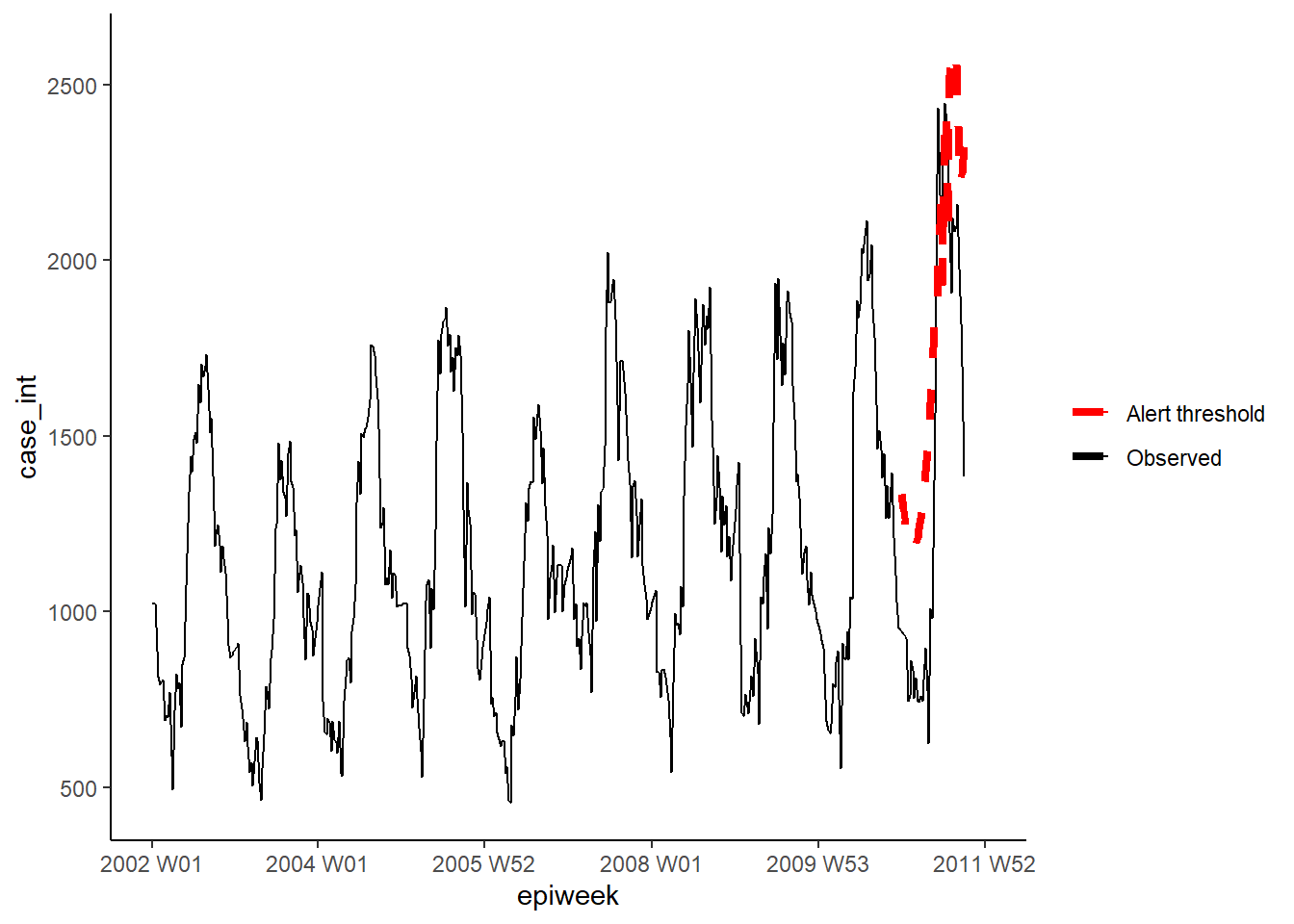
23.8 Kesintili zaman serileri
Kesintili zaman serileri (ayrıca bölümlere ayrılmış regresyon veya müdahale analizi olarak da adlandırılır), genellikle aşıların hastalık insidansı üzerindeki etkisini değerlendirmede kullanılır. Ancak çok çeşitli müdahalelerin etkisini değerlendirmek için kullanılabilir. Örneğin, hastane prosedürlerinde değişiklikler veya bir popülasyona yeni bir hastalık türünün görülmesi. Bu örnekte, 2008’in sonunda Almanya’ya yeni bir Campylobacter türünün geldiğini farz edeceğiz ve bunun vaka sayısını etkileyip etkilemediğini göreceğiz. Negatif binom regresyonunu tekrar kullanacağız. Bu seferki regresyon, müdahaleden önce (veya burada yeni suşun gelişi) ve diğeri (ön/pre- ve son/post dönemler) olmak üzere iki kısma ayrılacaktır. Bu, iki zaman periyodunu karşılaştıran bir insidans oranı hesaplamamızı sağlar. Denklemi açıklamak anlatımı daha net hale getirebilir (eğer değilse, görmezden gelin!).
Negatif binom regresyon aşağıdaki gibi tanımlanabilir:
\[\log(Y_t)= β_0 + β_1 \times t+ β_2 \times δ(t-t_0) + β_3\times(t-t_0 )^+ + log(pop_t) + e_t\]
Neresi: \(Y_t\), \(t\) anında gözlemlenen vaka sayısıdır \(pop_t\), \(t\) anındaki 100.000’lerdeki nüfus büyüklüğüdür (burada kullanılmaz) \(t_0\) ön dönemin son yılıdır (varsa geçiş süresi dahil) \(δ(x\) gösterge işlevidir (x≤0 ise 0 ve x>0 ise 1’dir) \((x)^+\) kesme operatörüdür (x>0 ise x, aksi takdirde 0’dır) \(e_t\) kalıntıyı belirtir Gerektiğinde ek terimler trend ve sezon eklenebilir.
\(β_2 \times δ(t-t_0) + β_3\times(t-t_0 )^+\) genelleştirilmiş doğrusaldır sonrası dönemin bir parçasıdır ve ön dönemde sıfırdır. Bu, \(β_2\) ve \(β_3\) tahminlerinin müdahalenin etkileri olduğu anlamına gelir.
Elimizdeki tüm verileri (yani geriye dönük olarak) kullanacağımızdan, burada tahmin yapmadan fourier terimlerini yeniden hesaplamamız gerekiyor. Ek olarak, regresyon için gereken ekstra terimleri hesaplamamız gerekiyor.
## epi hafta ve case_int değişkenlerini kullanarak fourier terimleri ekleyin
counts$fourier <- select(counts, epiweek, case_int) %>%
as_tsibble(index = epiweek) %>%
fourier(K = 1)
## müdahale haftasını tanımla
intervention_week <- yearweek("2008-12-31")
## regresyon için değişkenleri tanımlayın
counts <- counts %>%
mutate(
## formüldeki t'ye karşılık gelir
## hafta sayısı (muhtemelen düz epiweeks değişkenini de kullanabilir)
# linear = row_number(epiweek),
## formüldeki delta(t-t0)'a karşılık gelir
## müdahale öncesi veya sonrası dönem
intervention = as.numeric(epiweek >= intervention_week),
## formüldeki (t-t0)^+'a karşılık gelir
## müdahale sonrası hafta sayısı
## (0 ile hesaplanan değer arasındaki sayılardan daha büyük sayıyı seçin)
time_post = pmax(0, epiweek - intervention_week + 1))Daha sonra bu terimleri negatif bir binom regresyonuna uydurmak için kullanırız ve çıktısı yüzde değişimi olan bir tablo elde ederiz. Bu örnekte, önemli bir değişiklik görülmemiştir.
DİKKAT: Npredict() değişkeninde simulate_pi = FALSE kullanımına dikkat edin. Bunun nedeni, trending’in varsayılan davranışının bir tahmin aralığı oluşturmak için ciTools paketini kullanmasıdır. NA (eksik) sayılar varsa bu paket çalışmaz ve daha granüler aralıklar üretir. Ayrıntılar için ?trending::predict.trending_model_fit’e bakın.
## uydurmak istediğiniz modeli tanımlayın (negatif binom)
model <- glm_nb_model(
## ilgilenilen çıktı olarak vaka sayısını belirleyeni
case_int ~
## trendi hesaba katmak için epiweek'i kullanın
epiweek +
## mevsimselliği hesaba katmak için fourier terimlerini kullanın
fourier +
## öncesi (pre-) veya sonrası (post-) dönemde ekleyin
intervention +
## müdahale sonrası zamanı ekleyin
time_post
)
## sayım veri setini kullanarak modelinizi uydurun
fitted_model <- trending::fit(model, counts)
## güven ve tahmin aralıklarını hesaplayın
observed <- predict(fitted_model, simulate_pi = FALSE)
## tabloda tahminleri ve yüzde değişimini gösterin
fitted_model %>%
## orijinal negatif binom regresyonunu çıkarın
get_model() %>%
## sonuçların düzenli (tidy) veri çerçevesini elde edin
tidy(exponentiate = TRUE,
conf.int = TRUE) %>%
## sadece müdahale değerini koruyun
filter(term == "intervention") %>%
## tahmin ve güven aralıkları için IRR'yi yüzde değişikliğine değiştirin
mutate(
## ilgilenilen sütunların her biri için - yeni bir sütun oluşturun
across(
all_of(c("estimate", "conf.low", "conf.high")),
## yüzde değişimini hesaplamak için formülü uygulayın
.f = function(i) 100 * (i - 1),
## yeni sütun adlarına bir sonek "_perc" ekleyin
.names = "{.col}_perc")
) %>%
## yalnızca belirli sütunları tutun (ve yeniden adlandırın)
select("IRR" = estimate,
"95%CI low" = conf.low,
"95%CI high" = conf.high,
"Percentage change" = estimate_perc,
"95%CI low (perc)" = conf.low_perc,
"95%CI high (perc)" = conf.high_perc,
"p-value" = p.value)## # A tibble: 1 × 7
## IRR `95%CI low` `95%CI high` `Percentage change` 95%CI low (perc…¹ 95%CI…² p-val…³
## <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 0.936 0.874 1.00 -6.40 -12.6 0.306 0.0645
## # … with abbreviated variable names ¹`95%CI low (perc)`, ²`95%CI high (perc)`,
## # ³`p-value`Daha önce olduğu gibi, regresyonun çıktılarını görselleştirebiliriz.
ggplot(observed, aes(x = epiweek)) +
## gözlemlenen vaka sayılarını bir çizgi olarak ekle
geom_line(aes(y = case_int, colour = "Observed")) +
## model tahmini için bir satır ekleyin
geom_line(aes(y = estimate, col = "Estimate")) +
## tahmin aralıkları için bir bant ekleyin
geom_ribbon(aes(ymin = lower_pi,
ymax = upper_pi),
alpha = 0.25) +
## tahminin nerede başladığını göstermek için dikey çizgi ve etiket ekleyin
geom_vline(
xintercept = as.Date(intervention_week),
linetype = "dashed") +
annotate(geom = "text",
label = "Intervention",
x = intervention_week,
y = max(observed$upper_pi),
angle = 90,
vjust = 1
) +
## renkleri tanımlayın
scale_colour_manual(values = c("Observed" = "black",
"Estimate" = "red")) +
## geleneksel bir grafik oluşturun (siyah eksenli ve beyaz arka planlı)
theme_classic()## Warning: Removed 13 rows containing missing values (`geom_line()`).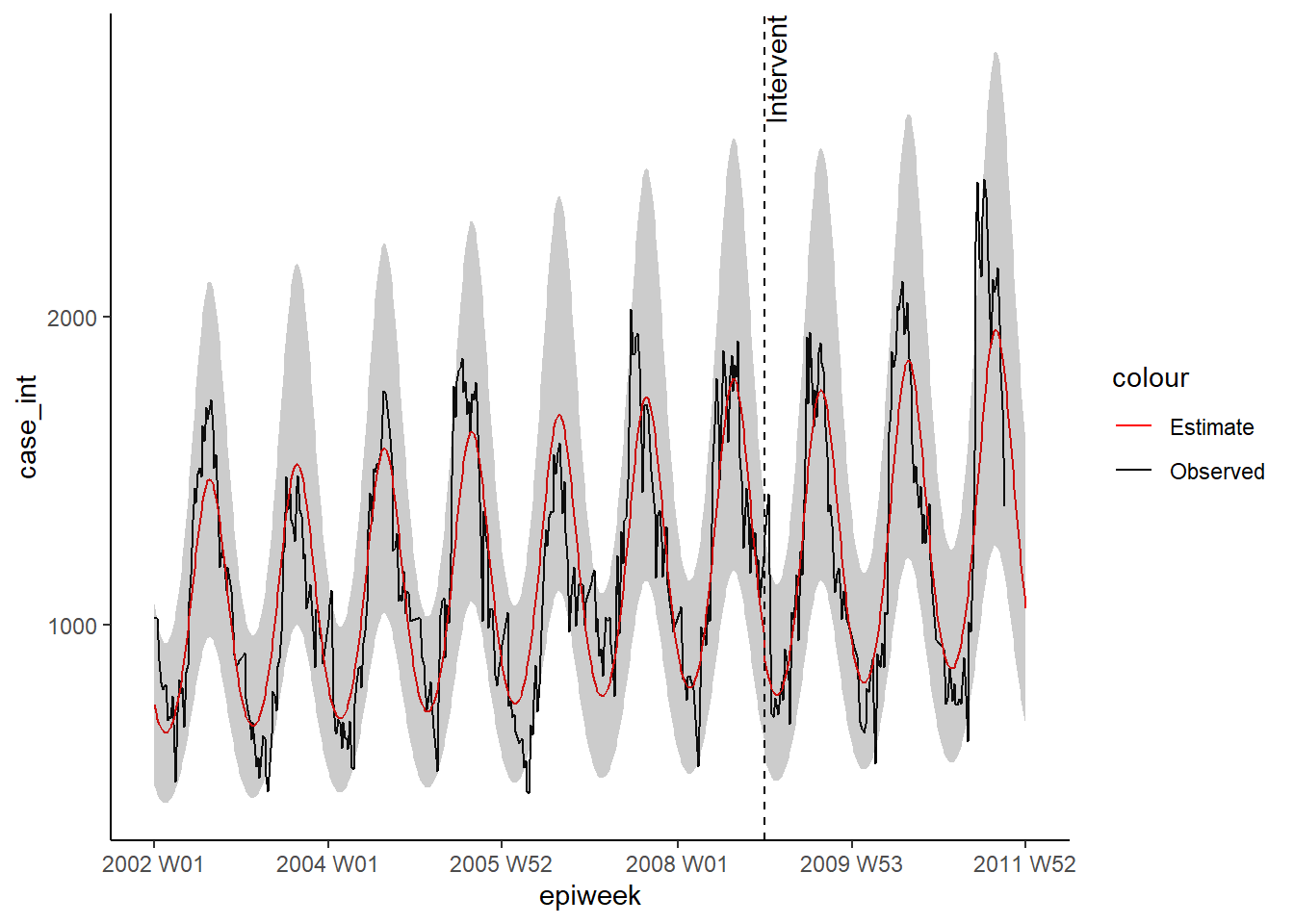
## Uyarı: Eksik değerler içeren 13 satır kaldırıldı (geom_path).