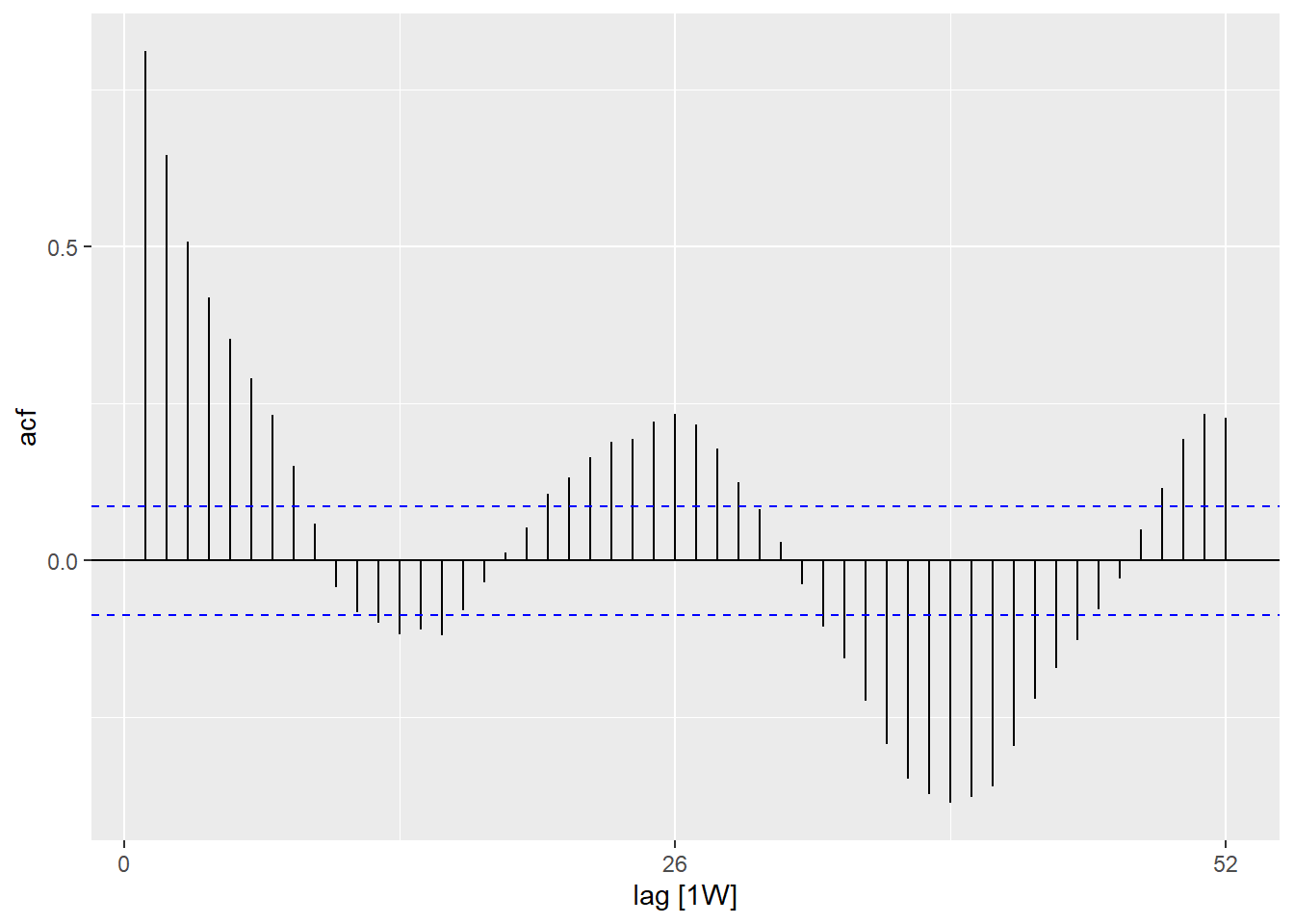pacman::p_load(rio, # импорт файлов
here, # путь к файлам
tidyverse, # управление данными + графика ggplot2
tsibble, # работа с наборами данных с временными рядами
slider, # для расчета скользящих средних значений
imputeTS, # для заполненеия отсутствующих значений
feasts, # для разложения временных рядов и автокорреляции
forecast, # построение sin и cosin по данным (примечание: необходимо загружать после feasts)
trending, # построение и оценка моделей
tmaptools, # для получения геокоординат (долготоа/широта) на основе названий локаций
ecmwfr, # для взаимодействия с CDS API спутника copernicus
stars, # для прочтения файлов .nc (климатические данные)
units, # для определения единиц измерения (климатические данные)
yardstick, # для рассмотрения точности модели
surveillance # для выявления аберраций
)23 Временные ряды и обнаружение вспышек
23.1 Обзор
На этой странице демонстрируется использование нескольких пакетов для анализа временных рядов. Мы в основном полагаемся на пакеты из семейства tidyverts, но также будем использовать пакет RECON trending, чтобы строить модели, которые больше подходят для эпидемиологии инфекционных заболеваний.
Обратите внимание, что в примере ниже мы используем набор данных из пакета surveillance по кампилобактеру в Германии (см. главу по данным руководства для получения дополнительной информации). Однако, если бы вы хотели выполнить тот же код для набора данных с несколькими странами или другими стратами, тогда вот есть пример шаблона кода для этих целей в репозитории r4epis github.
Рассматриваемые темы включают:
- Данные временных рядов
- Описательный анализ
- Построение регрессий
- Отношения между двумя временными рядами
- Обнаружение вспышек
- Прерванные временные ряды
23.2 Подготовка
Пакеты
Данный фрагмент кода показывает загрузку пакетов, необходимых для анализа. В данном руководстве мы фокусируемся на использовании p_load() из пакета pacman, которая устанавливает пакет, если необходимо, и загружает его для использования. Вы можете также загрузить установленные пакеты с помощью library() из базового R. См. страницу Основы R для получения дополнительной информации о пакетах R.
Загрузка данных
Вы можете скачать все данные, используемые в данном руководстве, следуя инструкциям на странице [Скачивание руководства и данных].
Пример набора данных, используемый в данном разделе, отражает еженедельное количество случаев кампилобактера, зарегистрированных в Германии в период с 2001 по 2011. Кликните сюда для скачивания этого файла с данными (.xlsx).
Этот набор данных - сокращенная версия набора данных, доступного в пакете surveillance. (для дополнительной информации загрузите пакет surveillance и посмотрите ?campyDE)
Импортируйте эти данные с помощью функции import() из пакета rio (он работает со многими типами файлов, такими как .xlsx, .csv, .rds - см. детали на страниец Импорт и экспорт).
# импорт количества случаев в R
counts <- rio::import("campylobacter_germany.xlsx")Первые 10 строк количества случаев отображены ниже.
Вычистка данных
Код ниже проверяет, что столбец даты находится в правильном формате. Для этой вкладки мы будем использовать пакет tsibble, так что для создания переменной календарной недели будет использоваться функция yearweek. Существует ряд других способов это сделать (см. страницу Работа с датами для получения дополнительной информации), однако для временных рядов лучше придерживаться одной системы (tsibble).
## проверяем правильность формата столбца даты
counts$date <- as.Date(counts$date)
## создаем переменную календарной недели
## соответствующую определению недель ИСПО с началом недели в понедельник
counts <- counts %>%
mutate(epiweek = yearweek(date, week_start = 1))Скачивание климатических данных
В разделе отношения между двумя временными рядами этой страницы мы будем сравнивать количество случаев кампилобактер с климатическими данными.
Климатические данные для любой точки мира можно скачать со спутника ЕС Copernicus. Эти измерения не являются абсолютно точными, а основаны на модели (похоже на интерполяцию), однако преимуществом является глобальный ежечасный охват и прогнозы.
Вы можете скачать эти файлы климатических данных со страницы Скачивание руководства и данных.
В демонстрационных целях мы покажем код R для использования пакета ecmwfr, чтобы получить эти данные из Хранилища климатических данных Copernicus. Вам нужно будет создать для этого бесплатный аккаунт. На веб-сайте пакета есть полезная пошаговая инструкция о том, как это сделать. Ниже приведен пример кода, который вы можете использовать, как только у вас будут необходимые ключи API. Вы должны заменить X ниже на свое ID аккаунта. Вам нужно скачивать по году данных за раз, иначе истекает время ожидания на сервере.
Если вы не уверены в координатах нужной вам локации, для которой вы хотите скачать данные, вы можете использовать пакет tmaptools, чтобы взять координаты с open street maps. В качестве альтернативы есть пакет photon, однако он еще не выпущен на CRAN; плюс пакета photon в том, что он дает больше контекстуальных данных, когда есть несколько совпадений по вашему поиску.
## получение координат места
coords <- geocode_OSM("Germany", geometry = "point")
## свести воедино долготы/широты в формате для запросов к ERA-5 (ограничительная рамка)
## (поскольку нужна только одна точка, можно повторить координаты)
request_coords <- str_glue_data(coords$coords, "{y}/{x}/{y}/{x}")
## Получения данных, смоделированных спутником copernicus(ERA-5 реанализ)
## https://cds.climate.copernicus.eu/cdsapp#!/software/app-era5-explorer?tab=app
## https://github.com/bluegreen-labs/ecmwfr
## создание ключа для погодных данных
wf_set_key(user = "XXXXX",
key = "XXXXXXXXX-XXXX-XXXX-XXXX-XXXXXXXXXXX",
service = "cds")
## выполнение для каждого интересующего года (otherwise server times out)
for (i in 2002:2011) {
## формирование запроса
## см. тут как это сделать: https://bluegreen-labs.github.io/ecmwfr/articles/cds_vignette.html#the-request-syntax
## изменяем запрос на список, используя кнопку добавления выше (python в list)
## Target (цель) - имя файла на выходе!!
request <- request <- list(
product_type = "reanalysis",
format = "netcdf",
variable = c("2m_temperature", "total_precipitation"),
year = c(i),
month = c("01", "02", "03", "04", "05", "06", "07", "08", "09", "10", "11", "12"),
day = c("01", "02", "03", "04", "05", "06", "07", "08", "09", "10", "11", "12",
"13", "14", "15", "16", "17", "18", "19", "20", "21", "22", "23", "24",
"25", "26", "27", "28", "29", "30", "31"),
time = c("00:00", "01:00", "02:00", "03:00", "04:00", "05:00", "06:00", "07:00",
"08:00", "09:00", "10:00", "11:00", "12:00", "13:00", "14:00", "15:00",
"16:00", "17:00", "18:00", "19:00", "20:00", "21:00", "22:00", "23:00"),
area = request_coords,
dataset_short_name = "reanalysis-era5-single-levels",
target = paste0("germany_weather", i, ".nc")
)
## скачиваем файл и сохраняем в текущей рабочей директории
file <- wf_request(user = "XXXXX", # ID пользователя (для авторизации)
request = request, # запрос
transfer = TRUE, # скачиваем файл
path = here::here("data", "Weather")) ## путь для сохранения данных
}Загрузка климатических данных
Вне зависимости от того, скачали ли вы данные по климату с помощью руководства или кода выше, у вас в одной папке на компьютере теперь должны быть в наличии файлы “.nc” с климатическими данными за 10 лет.
Используйте код ниже, чтобы импортировать эти файлы в R с помощью пакета stars.
## определяем путь к папке с погодными данными (weather)
file_paths <- list.files(
here::here("data", "time_series", "weather"), # замените на собственный путь к файлу
full.names = TRUE)
## сохраняем только те, которые имеют интересующее нас в данный момент имя
file_paths <- file_paths[str_detect(file_paths, "germany")]
## прочитываем все файлы как объект stars
data <- stars::read_stars(file_paths)t2m, tp,
t2m, tp,
t2m, tp,
t2m, tp,
t2m, tp,
t2m, tp,
t2m, tp,
t2m, tp,
t2m, tp,
t2m, tp, Как только эти файлы были импортированы как объект data, мы их конвертируем в датафрейм.
## изменяем на датафрейм
temp_data <- as_tibble(data) %>%
## добавляем переменные и правильные единицы измерения
mutate(
## создаем переменную календарная неделя
epiweek = tsibble::yearweek(time),
## создаем переменную дата (начало календарной недели)
date = as.Date(epiweek),
## меняем температуру с градусов по Кельвину на градусы по Цельсию
t2m = set_units(t2m, celsius),
## меняем осадки из метров в миллиметры
tp = set_units(tp, mm)) %>%
## группируем по неделе (но сохраняем дату)
group_by(epiweek, date) %>%
## получаем среднее за неделю
summarise(t2m = as.numeric(mean(t2m)),
tp = as.numeric(mean(tp)))`summarise()` has grouped output by 'epiweek'. You can override using the
`.groups` argument.23.3 Данные временных рядов
Существует ряд различных пакетов для структурирования и рабботы с данными временных рядов. Как мы говорили, мы будем фокусироваться на семействе пакетов tidyverts, поэтому мы используем пакет tsibble для определения нашего объекта временного ряда. Определение набора данных как объекта временного ряда означает, что нам будет легче структурировать анализ.
Чтобы это сделать, используем функцию tsibble() и уточним “index” (индекс), т.е. переменную, указывающую интересующую нас единицу времени. В данном случае это переменная эпиднеделя (epiweek).
Если бы у нас был набор данных с еженедельным количеством случаев по провинции, например, мы бы также могли определить переменную группирования, используя аргумент key =. Это позволило бы нам проводить анализ для каждой группы.
## определяем объект временного ряда
counts <- tsibble(counts, index = epiweek)Если рассмотреть class(counts), вы увидите, что это не просто аккуратный датафрейм (“tbl_df”, “tbl”, “data.frame”), но у него еще есть дополнительные свойства датафрейма временного ряда (“tbl_ts”).
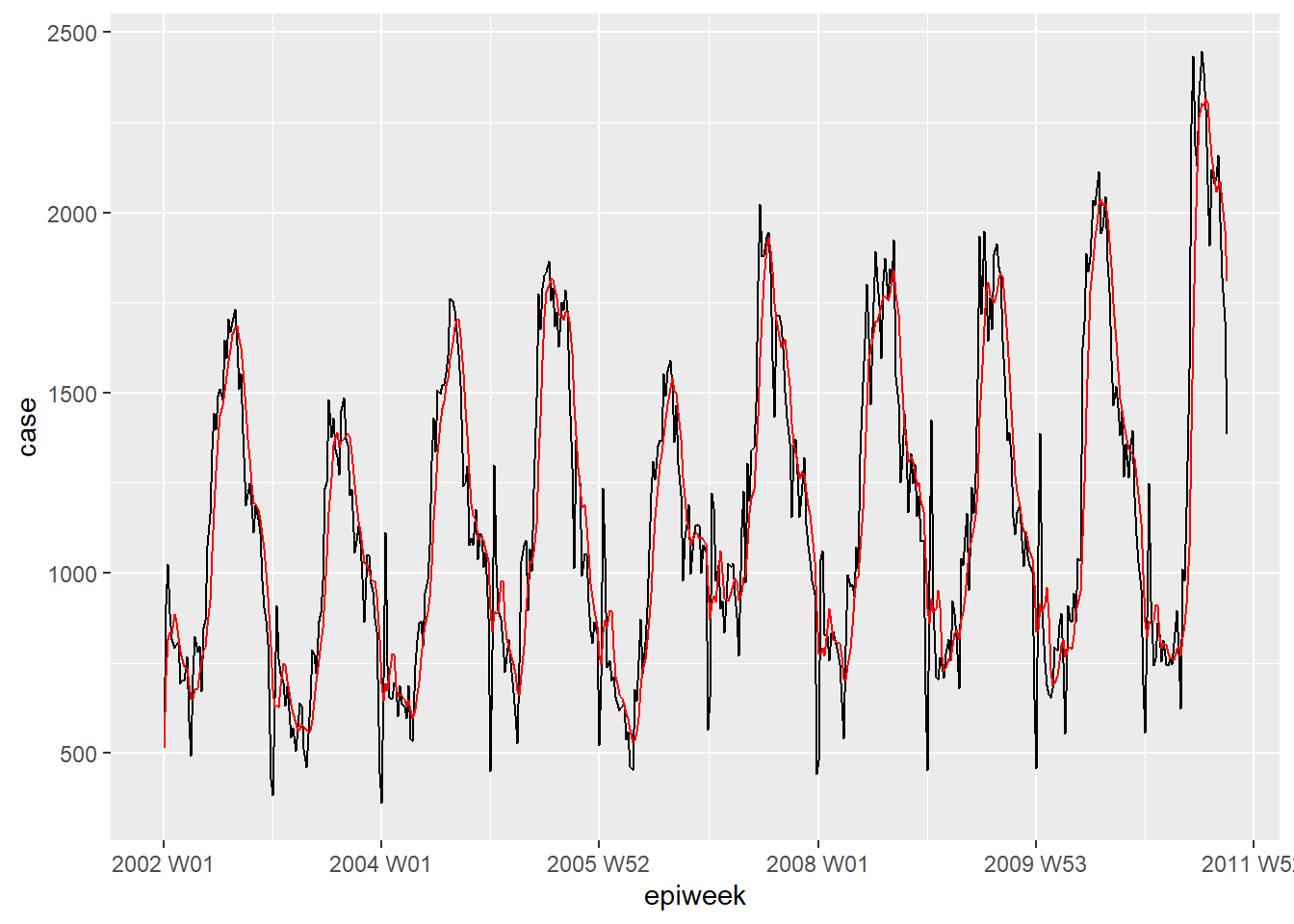
Вы можете быстро просмотреть свои данные, используя ggplot2. Из графика мы видим, что есть четкая сезонность, а также что нет отсутствующих данных. Однако кажется есть проблема с отчетностью в начале каждого года; количество случаев падает в последнюю неделю года, а затем увеличивается в первую неделю следующего года.
## строим линейный график количества случаев по неделям
ggplot(counts, aes(x = epiweek, y = case)) +
geom_line()ВНИМАНИЕ: Большинство наборов данных не такие чистые, как этот пример. Вам нужно будет проверить дубликаты и отсутствующие данные, как показано ниже.
Дубликаты
tsibble не допускает дублирующиеся наблюдения. Поэтому каждая строка должна быть уникальной, либо уникальной внутри группы (переменная key). В пакете есть функции, которые помогают выявить дубликаты. Они включают are_duplicated(), которая даст вам вектор TRUE/FALSE (ИСТИНА/ЛОЖЬ), показывающий является ли строка дубликатом, и duplicates(), которая даст вам датафрейм дублирующихся строк.
См. деталио том, как выбирать нужные вам строки на странице Дедупликация.
## получаем вектор TRUE/FALSE (ИСТИНА/ЛОЖЬ) того, являются ли строки дубликатами
are_duplicated(counts, index = epiweek)
## получаем датафрейм любых дублирующихся строк
duplicates(counts, index = epiweek) Отсутствующие данные
Из краткого рассмотрения данных выше мы видели, что нет отсутствующих данных, но при этом мы заметили проблему с задержкой в отчетности в районе нового года. Один из способов решить эту проблему - задать эти значения как отсутствующие и затем вменить значения. Самая простая форма вменения временных рядов - прочертить прямую линию между последним неотсутствующим и следующим неотсутствующим значением. Чтобы это сделать, мы используем функцию na_interpolation() из пакета imputeTS.
См. страницу Отсутствующие данные для рассмотрения других вариантов вменения.
Еще одна альтернатива - рассчитать скользящее среднее, чтобы сгладить эти проблемы с отчетностью (см. следующий раздел и страницу Скользящие средние значения).
## создаем переменную с отсутствующими значениями вместо недель с проблемами с отчетностью
counts <- counts %>%
mutate(case_miss = if_else(
## если эпиднеделя содержит 52, 53, 1 или 2
str_detect(epiweek, "W51|W52|W53|W01|W02"),
## тогда установить на отсутствующее
NA_real_,
## в остальных случаях - сохранить значение case
case
))
## альтернативно интерполяция отсутствующих значений по линейному тренду
## между двумя ближайшими соседними точками
counts <- counts %>%
mutate(case_int = imputeTS::na_interpolation(case_miss)
)
## чтобы проверить, какие значения были вменены по сравнению с оригинальными
ggplot_na_imputations(counts$case_miss, counts$case_int) +
## создаем традиционную диаграмму (с черными осями и белым фоном)
theme_classic()23.4 Описательный анализ
Скользящие средние значения
Если в данных много шума (количество подскакивает и падает), тогда может быть полезно рассчитать скользящие средние значения. В примере ниже мы рассчитываем для каждой недели среднее количество случаев за четыре предыдущих недели. Это выравнивает данные, чтобы сделать их более легко интерпретируемыми. В нашем случае это не слишком много меняет, поэтому мы будем использовать интерполированные данные для дальнейшего анализа. См. страницу Скользящие средние значения для получения дополнительной информации.
## создаем переменную скользящего среднего (работает с отсутствующими значениями)
counts <- counts %>%
## создаем переменную ma_4w
## скользим по каждой строке переменной case (случаи)
mutate(ma_4wk = slider::slide_dbl(case,
## для каждой строки рассчитываем имя
~ mean(.x, na.rm = TRUE),
## используем 4 предыдущих недели
.before = 4))
## создаем визуализацию различий
ggplot(counts, aes(x = epiweek)) +
geom_line(aes(y = case)) +
geom_line(aes(y = ma_4wk), colour = "red")Периодичность
Ниже мы определяем пользовательскую функцию, чтобы создать периодограмму. См. страницу [Написание функций] для получения информации о том, как писать функции в R.
Сначала определяется функция. Ее аргументы включают набор данных со столбцом counts, start_week =, которая является первой неделей набора данных, число, которое указывает количество периодов в году (например, 52, 12), и наконец, стиль выходных данных (см. детали в коде ниже).
## Аргументы функции
#####################
## x - набор данных
## counts - переменная с данными о количестве или коэффициентами в x
## start_week - первая неделя вашего набора данных
## period - период, сколько единиц в году
## output - выходные данные - вы хотите получить спектральную периодограмму или пиковые недели
## "periodogram" или "weeks"
# Определяем функцию
periodogram <- function(x,
counts,
start_week = c(2002, 1),
period = 52,
output = "weeks") {
## убедитесь, что это не tsibble, фильтр, чтобы защитить и сохранить только интересующие столбцы
prepare_data <- dplyr::as_tibble(x)
# готовим данные: prepare_data <- prepare_data[prepare_data[[strata]] == j, ]
prepare_data <- dplyr::select(prepare_data, {{counts}})
## создаем промеждуточный временной ряд "zoo", чтобы использовать для spec.pgram
zoo_cases <- zoo::zooreg(prepare_data,
start = start_week, frequency = period)
## получаем спектральную периодограмму, не используя быстрые трансформации фурье
periodo <- spec.pgram(zoo_cases, fast = FALSE, plot = FALSE)
## на выходе получаем пиковые недели
periodo_weeks <- 1 / periodo$freq[order(-periodo$spec)] * period
if (output == "weeks") {
periodo_weeks
} else {
periodo
}
}
## получаем спектральную периодограмму для извлечения недель с саамыми высокими частотами
## (проверка сезонности)
periodo <- periodogram(counts,
case_int,
start_week = c(2002, 1),
output = "periodogram")
## переносим спектр и частоту в датафрейм для построения графика
periodo <- data.frame(periodo$freq, periodo$spec)
## строим периодограмму, показывающую наиболее часто возникающую периодичность
ggplot(data = periodo,
aes(x = 1/(periodo.freq/52), y = log(periodo.spec))) +
geom_line() +
labs(x = "Period (Weeks)", y = "Log(density)")## получаем вектор недель в возрастающем порядке
peak_weeks <- periodogram(counts,
case_int,
start_week = c(2002, 1),
output = "weeks")ПРИМЕЧАНИЕ: Можно использовать указанные выше недели, чтобы добавить их в условия sin и cosine, однако мы будем использовать функцию для генерации этих условий (см. раздел регрессия ниже)
Разложение
Классическое разложение используется для разбивки временного ряда на несколько частей, которые при рассмотрении вместе создают закономерности, которые вы видите. Эти различные части включают:
- Цикл тренда (долгосрочное направление данных)
- Сезонность (повторяющиеся закономерности)
- Случайность (то, что осталось после удаления тренда и сезонности)
Автокорреляция
Автокорреляция показывает вам отношения между количеством каждой недели и недель до нее (называемых лагами).
Используя функцию ACF(), мы можем создать график, который покажет нам количество линий для отношения при разных лагах. То, где лаг составляет 0 (x = 0), эта линия всегда будет 1, так как она показывает отношение между наблюдением и самим собой (здесь не показано). Первая линия, показанная здесь (x = 1), отражает отношениие между каждым наблюдением и наблюдением перед ним (лаг 1), вторая - показывает отношение между каждым наблюдением и предпоследним наблюдением до него (лаг 2) и так далее до лага 52, который отражает отношение между каждым наблюдением и наблюдением за 1 год до этого (52 недели ранее).
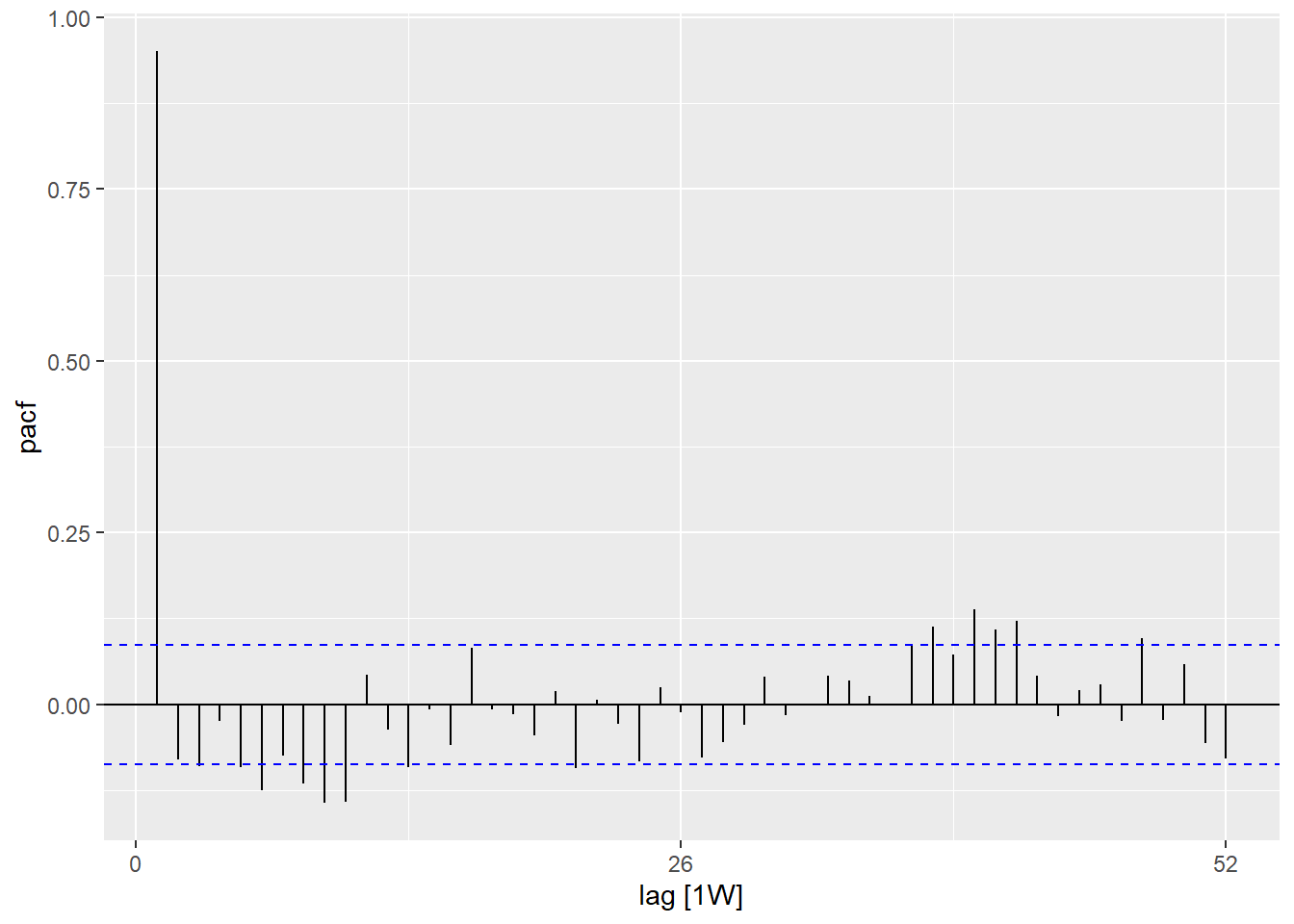
При использовании функции PACF() (для частичной автокорреляции) мы увидим тот же тип отношений, но скорректированный для всех промежуточных недель. Это менее информативно для определения периодичности.
## используем набор данных количества случаев counts
counts %>%
## рассчитываем автокорреляцию, используя лаги на 1 год
ACF(case_int, lag_max = 52) %>%
## показываем график
autoplot()## используем набор данных количества случаев counts
counts %>%
## рассчитываем частичную автокорреляцию, используя лаги за полный год
PACF(case_int, lag_max = 52) %>%
## показываем график
autoplot()Вы можете формально протестировать нулевую гипотезу независимости во временном ряде (т.е. что нет автокорреляции), используя тест Льюнг-Бокса (в пакете stats). Значительное p-значение указывает на то, что существует автокорреляция в данных.
## тест на независимость
Box.test(counts$case_int, type = "Ljung-Box")
Box-Ljung test
data: counts$case_int
X-squared = 462.65, df = 1, p-value < 2.2e-1623.5 Построение регрессий
Для временного ряда можно построить большое количество разных регрессий, однако здесь мы продемонстрируем, как построить отрицательную биномиальную регрессию - так как часто она является наиболее подходящей для данных об абсолютном количестве для инфекционных заболеваний.
Условия Фурье
Условия Фурье - эквивалент кривых синуса и косинуса. Разница состоит в том, что они строятся на основе поиска наиболее подходящей комбинации кривых для объяснения ваших данных.
Построение только одного условия фурье будет эквивалентно построению синусоиды и косинусоиды для наиболее часто встречающегося лага в периодограмме (в нашем случае 52 недели). Мы используем функцию fourier() из пакета forecast.
В коде ниже мы проводим присваивание с помощью $, поскольку fourier() выдает два столбца (один для синуса и один для косинуса), они добавляются в набор данных в виде списка под названием “fourier” - но этот список затем можно использовать как обычную переменную в регрессии.
## добавляем условия фурье, используя переменные epiweek и case_int
counts$fourier <- select(counts, epiweek, case_int) %>%
fourier(K = 1)Отрицательная биномиальная
Можно построить регрессии, используя базовые функции stats или MASS (например, lm(), glm() и glm.nb()). Однако мы будем использовать функции из пакета trending, так как они позволяют рассчитывать соответствующие доверительные интервалы и интервалы прогнозирования (которые иначе недоступны). Синтаксис тот же самый, и вы задаете переменную результата, затем тильда (~), а затем добавляете различные интересующие переменные воздействия, отделяемые знаками плюс (+).
Еще отличие заключается в том, что мы сначала определяем модель, а затем строим ее с помощью fit() с использованием данных. Это полезно, поскольку это позволяет сравнивать несколько разных моделей
с одинаковым синтаксисом.
СОВЕТ: Если вы хотите использовать коэффициенты, а не абсолютное количество, вытможете включить популяционную переменную в качестве логарифмического условия смещения, добавив offset(log(population). Затем нужно задать популяцию как 1, прежде чем использовать predict(), чтобы получить коэффициент.
СОВЕТ: Чтобы построить более сложные модели, такие как ARIMA или prophet, см. пакет fable.
## определяем модель, которую хотим построить (отрицательная биномиальная)
model <- glm_nb_model(
## задаем количество случаев как интересующий результат
case_int ~
## используем эпиднедели (epiweek), чтобы учесть тренд
epiweek +
## используем условия фурье, чтобы учесть сезонность
fourier)
## строим модель, используя набор данных counts
fitted_model <- trending::fit(model, data.frame(counts))
## рассчитываем доверительные интервалы и интервалы прогнозирования
observed <- predict(fitted_model, simulate_pi = FALSE)
estimate_res <- data.frame(observed$result)
## строим график регрессии
ggplot(data = estimate_res, aes(x = epiweek)) +
## добавляем линию с оценкой модели
geom_line(aes(y = estimate),
col = "Red") +
## добавляем полосу интервалов прогнозирования
geom_ribbon(aes(ymin = lower_pi,
ymax = upper_pi),
alpha = 0.25) +
## добавляем линию для наблюдаемого количества случаев
geom_line(aes(y = case_int),
col = "black") +
## создаем традиционный график (с черными осями и белым фоном)
theme_classic()Остатки
Чтобы посмотреть, насколько хорошо наша модель соответствует наблюдаемым данным, необходимо посмотреть на остатки. Остатки - разница между наблюдаемым количеством и количеством, оцененным моделью Мы можем их рассчитать просто с помощью case_int - estimate, но функция residuals() извлекает их для нас напрямую из регрессии.
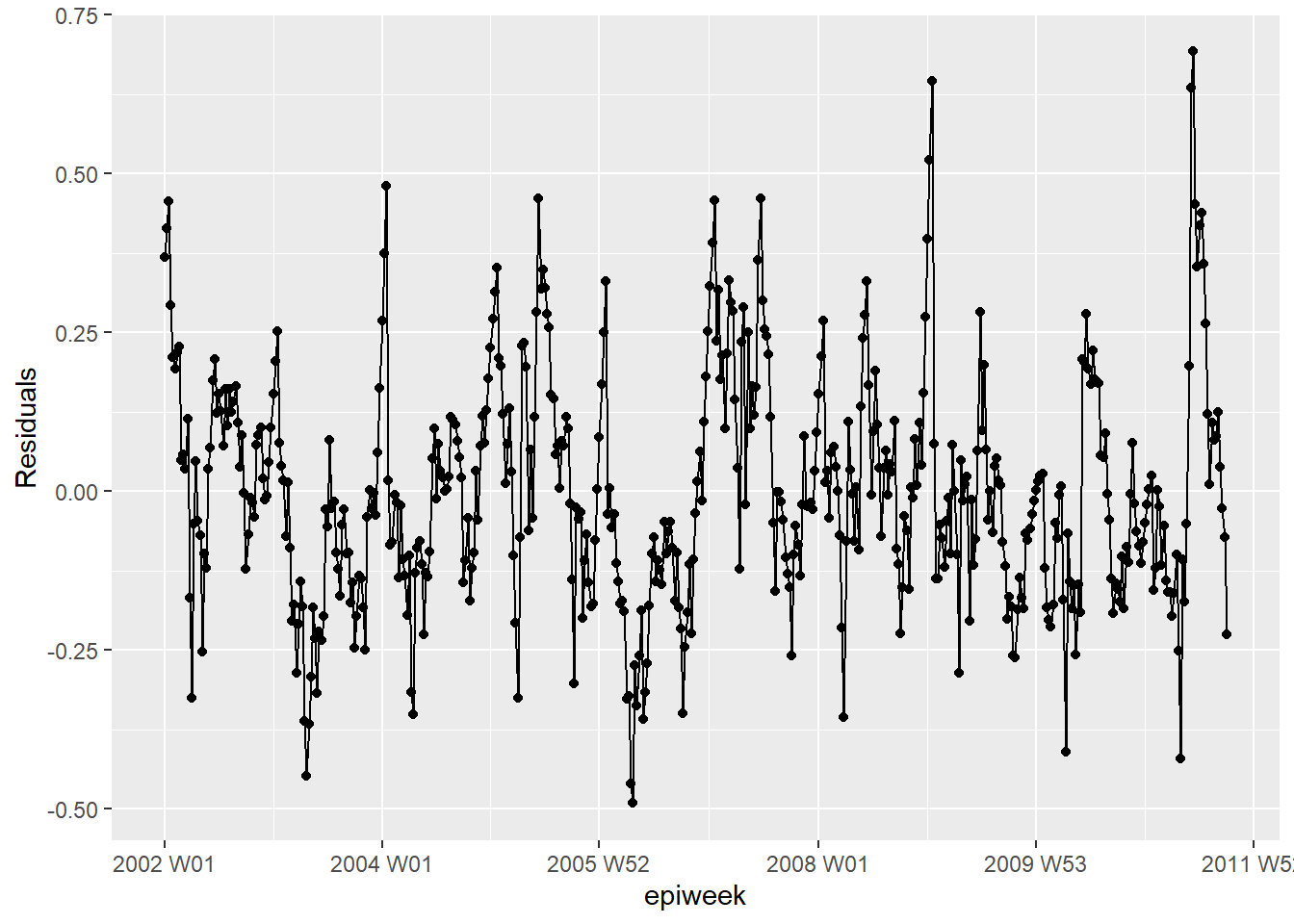
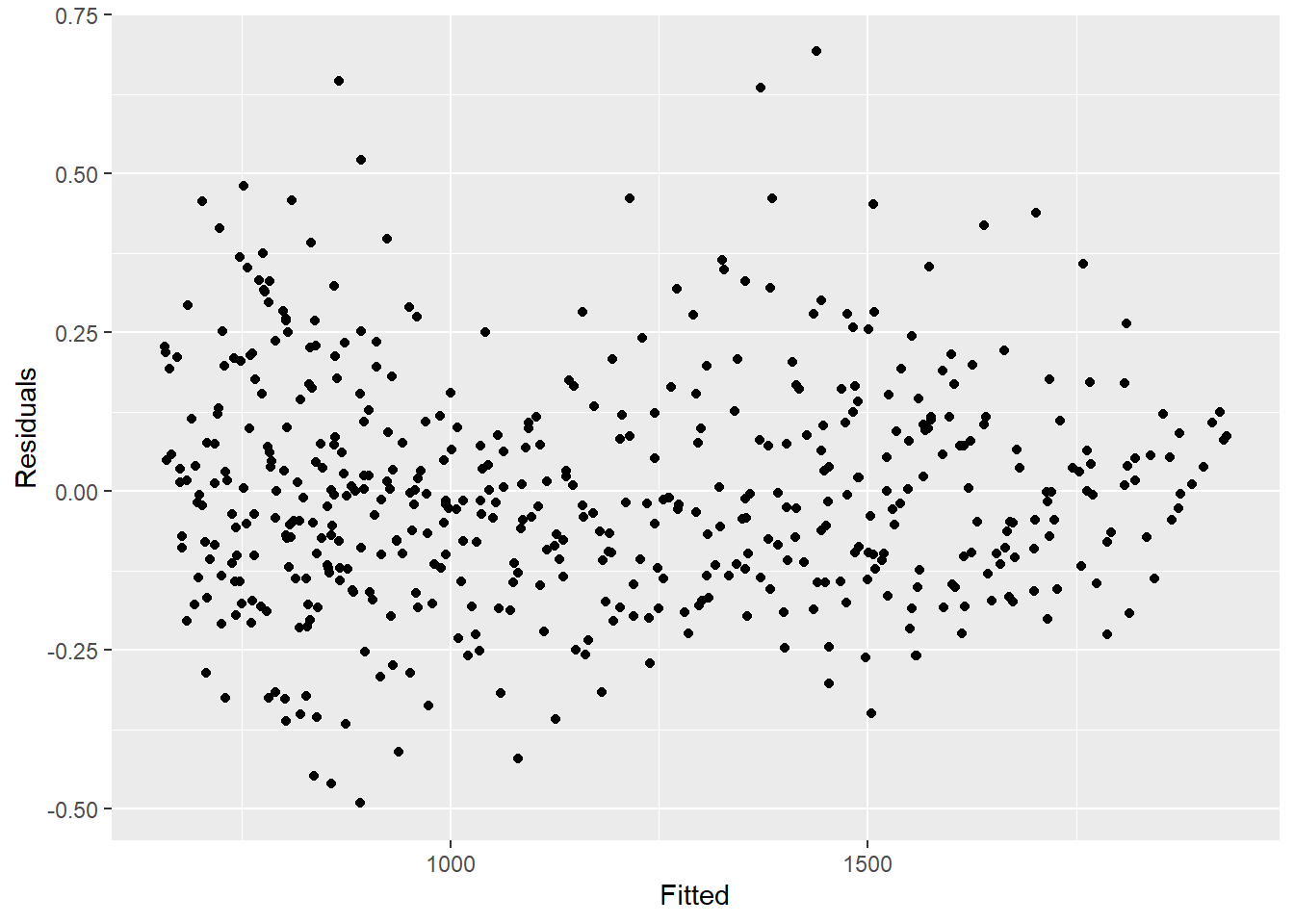
Что мы видим ниже - мы не объяснили все что мы не объясняем всю вариацию которые мы могли бы объяснить с помощью этой модели. Возможно, нам следует подобрать больше условий Фурье, и обратить внимание на амплитуду. Однако для данного примера мы оставим все как есть. Графики показывают нам, что модель дает более плохие результаты в периоды пиков и минимумов (когда количество случаев самое большое или самое низкое) и что модель с большей вероятностью недооценивает наблюдаемое количество.
## рассчитываем остатки
estimate_res <- estimate_res %>%
mutate(resid = fitted_model$result[[1]]$residuals)
## достаточно ли постоянны остатки в разные периоды времени (если нет: вспышки? изменение практики?)
estimate_res %>%
ggplot(aes(x = epiweek, y = resid)) +
geom_line() +
geom_point() +
labs(x = "epiweek", y = "Residuals")## есть ли автокорреляция в остатках (есть ли закономерности в ошибке?)
estimate_res %>%
as_tsibble(index = epiweek) %>%
ACF(resid, lag_max = 52) %>%
autoplot()## имеют ли остатки нормальное распределение (недооценивание или переоценивание?)
estimate_res %>%
ggplot(aes(x = resid)) +
geom_histogram(binwidth = 100) +
geom_rug() +
labs(y = "count") ## сравниваем наблюдаемое количество с остатками
## также не должно быть закономерностей
estimate_res %>%
ggplot(aes(x = estimate, y = resid)) +
geom_point() +
labs(x = "Fitted", y = "Residuals")## формально тестируем автокорреляцию остатков
## H0 - остатки представляют собой ряд с белым шумом (т.е. случайные)
## тестируем на независимость
## если значение p значимое, тогда не случайность
Box.test(estimate_res$resid, type = "Ljung-Box")
Box-Ljung test
data: estimate_res$resid
X-squared = 336.25, df = 1, p-value < 2.2e-1623.6 Отношения двух временных рядов
Здесь мы рассмотрим использование погодных данных (в частности, температуры) для объяснения количество случаев кампилобактера.
Объединение наборов данных
Мы можем объединить наши наборы данных, используя переменную недели. Дополнительную информацию об объединении см. в разделе руководства Соединение данных.
## используем левое соединение, чтобы у нас были только те строки, которые уже имеются в counts
## удаляем переменную даты из temp_data (иначе она будет дублироваться)
counts <- left_join(counts,
select(temp_data, -date),
by = "epiweek")Описательный анализ
Сначала постройте график ваших данных, чтобы увидеть, есть ли какие-то очевидные связи. График ниже показывает, что есть четкая связь с сезонностью двух переменных, и что пик температуры, возможно, достигается за несколько недель до пика количества случаев. Для получения дополнительной информации о повороте данных, см. раздел руководства Поворот данных.
counts %>%
## сохраняем интересующие нас переменные
select(epiweek, case_int, t2m) %>%
## меняем данные в длинный формат
pivot_longer(
## используем epiweek в качестве ключа
!epiweek,
## перемещаем имена столбцов в новый столбец "measure"
names_to = "measure",
## перемещаем значения ячеек в новый столбец "values"
values_to = "value") %>%
## создаем график с набором данных, указанным выше
## откладываем эпиднедели (epiweek) на оси x, а значения (values) (количество/градусы по Цельсию) на оси y
ggplot(aes(x = epiweek, y = value)) +
## создаем отдельный график для температуры и количества случаев
## разрешаем задать собственные оси y
facet_grid(measure ~ ., scales = "free_y") +
## составляем линейный график обоих
geom_line()Лаги и взаимнокорреляционная функция (кросс-корреляция)
Чтобы формально протестировать, для каких недель самая большая связь между случаями и температурой. Мы можем использовать функцию кросс-корреляции (CCF()) из пакета feasts. Вы также можете визуализировать (вместо использования arrange) с помощью функции autoplot().
counts %>%
## рассчитываем кросс-корреляцию между интерполированными подсчетами и температурой
CCF(case_int, t2m,
## устанавливаем максимальный лаг на 52 недели
lag_max = 52,
## выдаем коэффициент корреляции
type = "correlation") %>%
## сортируем в порядке уменьшения коэффициента корреляции
## показываем наиболее связанные лаги
arrange(-ccf) %>%
## показываем только топ 10
slice_head(n = 10)# A tsibble: 10 x 2 [1W]
lag ccf
<cf_lag> <dbl>
1 -4W 0.749
2 -5W 0.745
3 -3W 0.735
4 -6W 0.729
5 -2W 0.727
6 -7W 0.704
7 -1W 0.695
8 -8W 0.671
9 0W 0.649
10 47W 0.638Тз этого мы видим, что лаг 4 недель имеет наиболее высокую корреляцию, поэтому мы создаем переменную температуры с лагом, которую включим в нашу регрессию.
ВНИМАНИЕ: Обратите внимание, что первые 4 недели данных в переменной температуры с лагом отсутствуют (NA) - так как не существует четырех недель до, из которых можно было бы получить эти данные. Чтобы использовать этот набор данных в функции trending predict(), нам нужно использовать аргумент simulate_pi = FALSE внутри predict() ниже. Если мы хотим использовать опцию симуляции, тогда нам нужно отбросить эти отсутствующие значения и сохранить как новый набор данных, добавив drop_na(t2m_lag4) во фрагмент кода ниже.
counts <- counts %>%
## создаем новую переменную температуры с лагом 4 недели
mutate(t2m_lag4 = lag(t2m, n = 4))Отрицательная биномиальная с двумя переменными
Мы строим отрицательную биномиальную регрессию, как это делалось ранее. В этот раз добавим переменную температуры с лагом 4 недели.
ВНИМАНИЕ: Обратите внимание на использование simulate_pi = FALSE внутри аргумента predict(). Это необходимо потому, что поведение trending по умолчанию состоит в использовании пакета ciTools для оценки интервала прогнозирования. Это не работает, если есть количество NA, и также создает более детальные интервалы. См. дополнительную информацию в ?trending::predict.trending_model_fit.
## определяем модель, по которой мы хотим построить регрессию (отрицательную биномиальную)
model <- glm_nb_model(
## задаем количество случаев как интересующий результат
case_int ~
## используем эпиднедели (epiweek), чтобы учесть тренд
epiweek +
## используем условие фурье, чтобы учесть сезонность
fourier +
## используем температуру с лагом 4 недели
t2m_lag4
)
## строим модель с набором данных counts
fitted_model <- trending::fit(model, data.frame(counts))
## рассчитываем доверительные интервалы и интервалы прогнозирования
observed <- predict(fitted_model, simulate_pi = FALSE)Чтобы изучить индивидуальные условия, мы можем извлечь оригинальную отрицательную биномиальную регрессию из формата trending, используя get_model() и передать ее в функцию tidy() из пакета broom, чтобы получить возведенные в степень оценки и соответствующие доверительные интервалы.
Это нам показывает, что температура с лагом, после учета тренда и сезонности, схожа с количеством случаев (оценка ~ 1) и имеет значительную связь. Это указывает на то, что это может быть хорошей переменной для прогнозирования будущего количества случаев (так как климатические прогнозы легко доступны).
fitted_model %>%
## извлекаем оригинальную отрицательную биномиальную регрессию
get_fitted_model() #%>% [[1]]
Call: glm.nb(formula = case_int ~ epiweek + fourier + t2m_lag4, data = data.frame(counts),
init.theta = 32.80689607, link = log)
Coefficients:
(Intercept) epiweek fourierS1-52 fourierC1-52 t2m_lag4
5.825e+00 8.464e-05 -2.850e-01 -1.954e-01 6.672e-03
Degrees of Freedom: 504 Total (i.e. Null); 500 Residual
(4 observations deleted due to missingness)
Null Deviance: 2015
Residual Deviance: 508.2 AIC: 6784 ## получаем аккуратный датафрейм результатов
#tidy(exponentiate = TRUE,
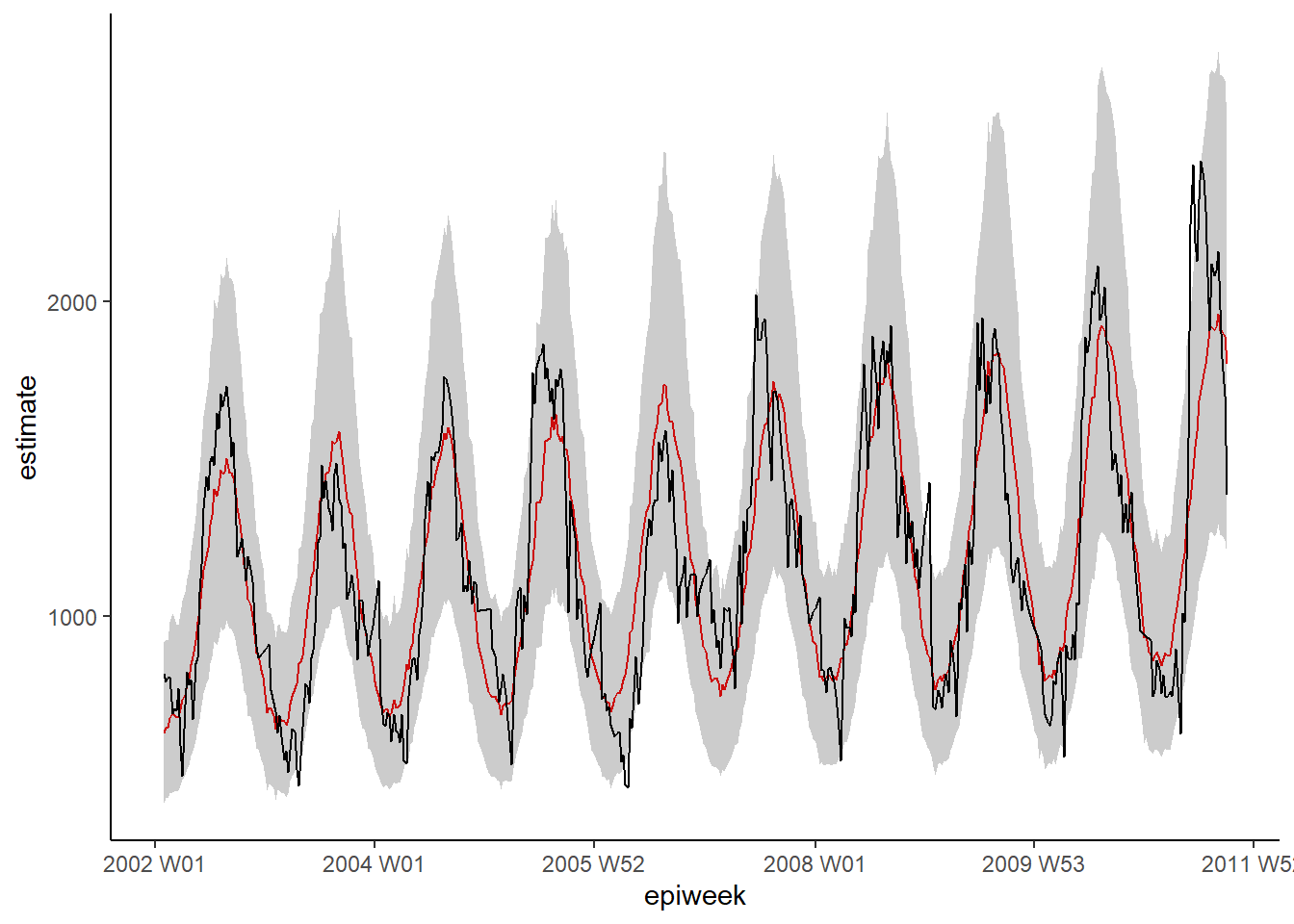
# conf.int = TRUE)Быстрый визуальный осмотр модели показывает, что она может лучше справиться с оценкой наблюдаемого количества случаев.
estimate_res <- data.frame(observed$result)
## строим график регрессии
ggplot(data = estimate_res, aes(x = epiweek)) +
## добавляем линию для оценки модели
geom_line(aes(y = estimate),
col = "Red") +
## добавляем диапазон интервалов прогнозирования
geom_ribbon(aes(ymin = lower_pi,
ymax = upper_pi),
alpha = 0.25) +
## добавляем линию наблюдаемого количества случаев
geom_line(aes(y = case_int),
col = "black") +
## создаем классический график (с черными осями и белым фоном)
theme_classic()Остатки
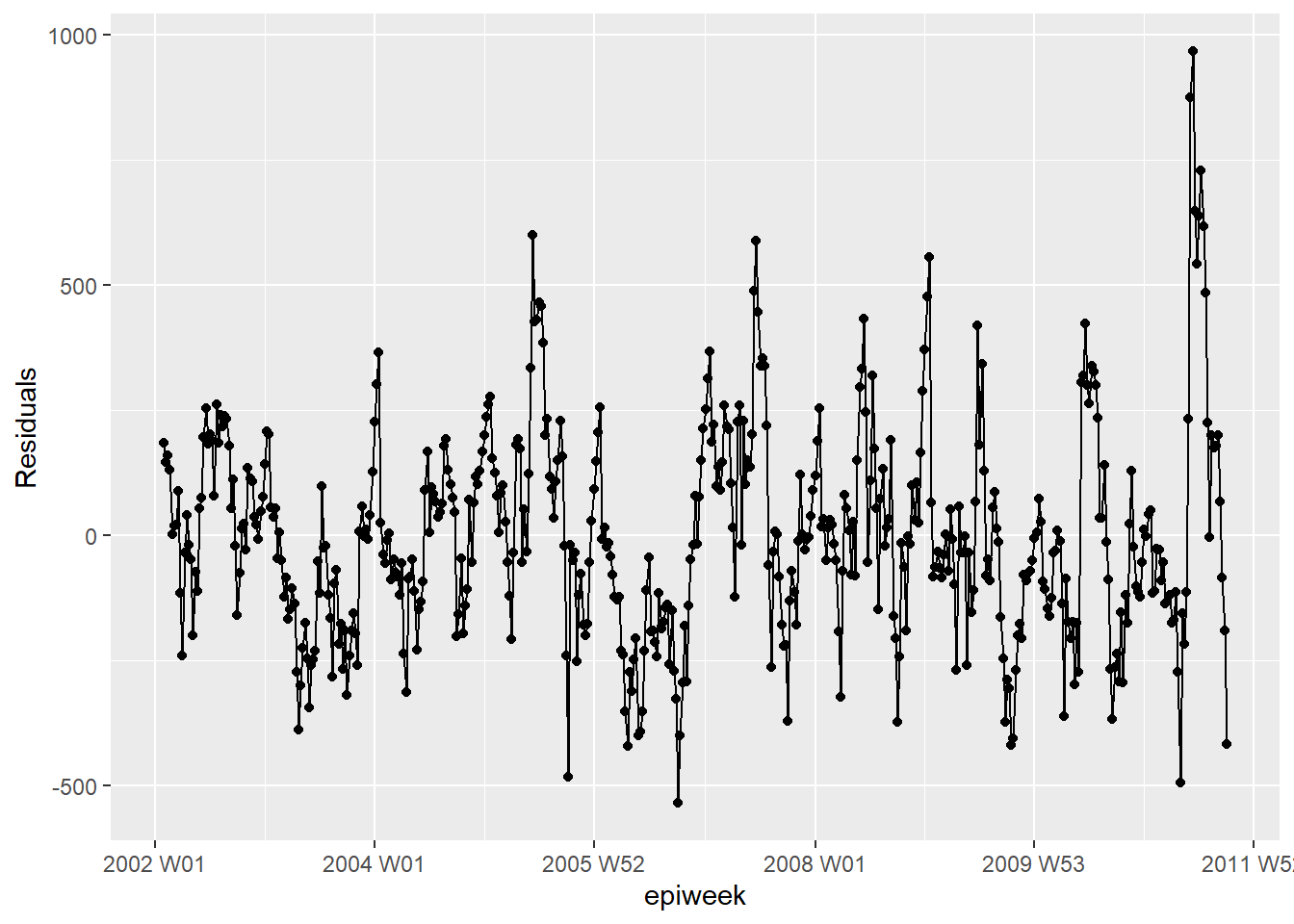
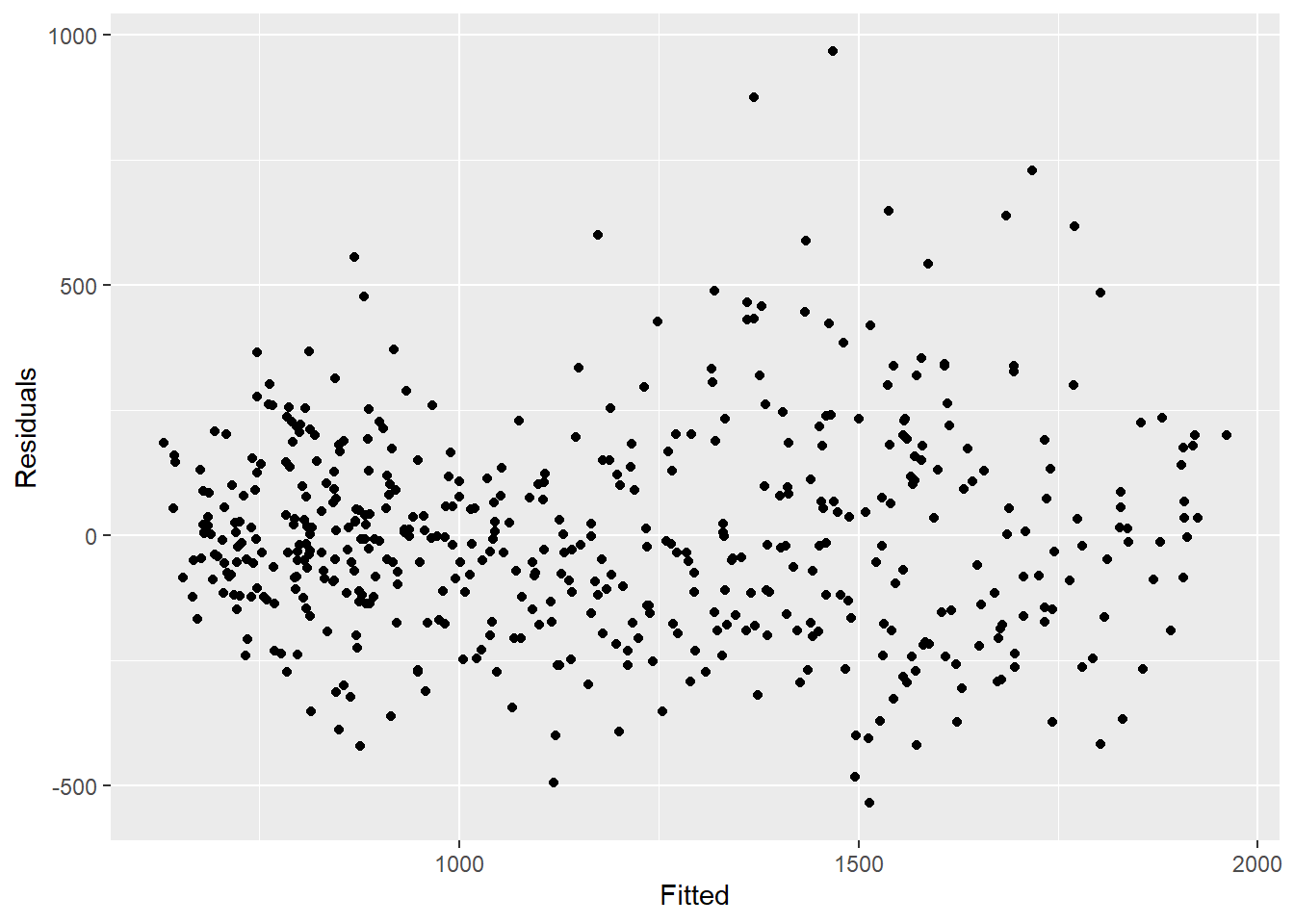
Мы снова исследуем остатки, чтобы проверить, насколько хорошо наша модель согласуется с наблюдаемыми данными. Результаты и интерпретация здесь аналогичны результатам предыдущей регрессии, поэтому, возможно, целесообразнее придерживаться более простой модели без температуры.
## рассчитываем остатки
estimate_res <- estimate_res %>%
mutate(resid = case_int - estimate)
## достаточно ли постоянны остатки в разные периоды времени (если нет: вспышки? изменение практики?)
estimate_res %>%
ggplot(aes(x = epiweek, y = resid)) +
geom_line() +
geom_point() +
labs(x = "epiweek", y = "Residuals")## есть ли автокорреляция в остатках (есть ли закономерности в ошибке?)
estimate_res %>%
as_tsibble(index = epiweek) %>%
ACF(resid, lag_max = 52) %>%
autoplot()## имеют ли остатки нормальное распределение (недооценивание или переоценивание?)
estimate_res %>%
ggplot(aes(x = resid)) +
geom_histogram(binwidth = 100) +
geom_rug() +
labs(y = "count") ## сравниваем наблюдаемое количество с остатками
## также не должно быть закономерностей
estimate_res %>%
ggplot(aes(x = estimate, y = resid)) +
geom_point() +
labs(x = "Fitted", y = "Residuals")## формально тестируем автокорреляцию остатков
## H0 - остатки представляют собой ряд с белым шумом (т.е. случайные)
## тестируем на независимость
## если значение p значимое, тогда не случайность
Box.test(estimate_res$resid, type = "Ljung-Box")
Box-Ljung test
data: estimate_res$resid
X-squared = 339.52, df = 1, p-value < 2.2e-1623.7 Обнаружение вспышки
Здесь мы продемонстрируем два (похожих) метода обнаружения вспышки. Первый метод основан на описанных выше разделах. Мы используем пакет trending, чтобы построить регрессии по предыдущим годам, а потом прогнозируем, что мы ожидаем увидеть в следующем году. Если наблюдаемое количество больше, чем мы ожидали, тогда это может указывать на вспышку. Второй метод основан на похожих принципах, но использует пакет surveillance, в котором есть ряд различных алгоритмов для выявления аберраций.
ВНИМАНИЕ: Как правило, вас интересует текущий год (где вы знаете количество только до текущей недели). Поэтому в этом примере мы представим, что мы находимся на 39 неделе 2011.
Пакет trending
Для этого метода мы определим базовый уровень (как правило, для этого нужно около 5 лет данных). Мы строим регрессию по базовым данным, а затем используем для прогнозирования оценок на следующий год.
Дата окончания сбора данных
Легче всего определить ваши даты в одном месте и затем использовать их в остальном коде.
Здесь мы задаем дату начала (когда начались наши наблюдения) и дату окончания сбора данных (конец базового периода - и начало периода прогнозирования). ~Мы также определяем, сколько недель в интересующем нас году (том, который мы прогнозируем)~. Мы также задаем, сколько недель между нашей последней датой базового периода и последней датой для прогнозирования.
ПРИМЕЧАНИЕ: В этом примере мы представим, что мы в настоящее время находимся в конце сентября 2011 (“2011 W39”).
## определяем дату начала (когда начались наблюдения)
start_date <- min(counts$epiweek)
## определяем неделю окончания сбора данных (окончание базового периода, начало периода прогнозирования)
cut_off <- yearweek("2010-12-31")
## определяем последнюю интересующую дату (т.е. окончание прогнозирования)
end_date <- yearweek("2011-12-31")
## считаем количество недель в интересующем периоде (году)
num_weeks <- as.numeric(end_date - cut_off)Добавление строк
Чтобы мы могли прогнозировать в формате tidyverse, нам нужно иметь правильное количество строк в нашем наборе данных, т.е. по одной строке для каждой недели до end_date, определенной ниже. Код ниже позволяет вам добавить эти строки по переменной группирования - например, если у нас в одном наборе данных несколько стран, мы можем сгруппировать по странам, а затем добавить строки для каждой. Функция group_by_key() из tsibble позволяет нам выполнить это группирование и затем передать
сгруппированные данные в функции dplyr, group_modify() и add_row(). Затем мы задаем последовательность недель между одной после максимальной недели имеющейся в данных, и конечной неделей.
## добавляем отсутствующие недели до конца года
counts <- counts %>%
## группируем по региону
group_by_key() %>%
## для каждой группы добавляем строки от самой последней эпиднедели до конца года
group_modify(~add_row(.,
epiweek = seq(max(.$epiweek) + 1,
end_date,
by = 1)))Условия Фурье
Нам необходимо переопределить наши условия Фурье - поскольку мы хотим подогнать их только к базовой дате, а затем предсказать (экстраполировать) эти условия на следующий год. Чтобы это сделать, нам нужно объединить два полученных на выходе списка из функции fourier(); первый список для базовых данных, а второй - прогнозирует интересующий год (с помощью определения аргумента h).
Примечание чтобы связать строки нам нужно использовать rbind() (а не bind_rows из tidyverse), поскольку столбцы фурье являются списком (не именованы индивидуально).
## определяем условия фурье (sincos)
counts <- counts %>%
mutate(
## объединяем условия фурье для недель до и после даты окончания в 2010
## (примечание: условия фурье на 2011 прогнозируются)
fourier = rbind(
## получаем условия фурье по предыдущим годам
fourier(
## сохраняем только строки до 2011
filter(counts,
epiweek <= cut_off),
## включаем один набор условий sin cos
K = 1
),
## прогнозируем условия фурье на 2011 (используя базовые данные)
fourier(
## сохраняем только строки до 2011
filter(counts,
epiweek <= cut_off),
## включаем один набор условий sin cos
K = 1,
## прогнозируем на 52 недели вперед
h = num_weeks
)
)
)Разделение данных и построение регрессии
Теперь нам надо разделить наш набор данных на базовый период и период прогнозирования. Это делается с помощью функции group_split() из dplyr после group_by(), что создаст список с двумя датафреймами - один до даты окончания сбора данных и один после.
Затем мы используем функцию pluck() из пакета purrr, чтобы извлечь наборы данных из списка (эквивалентно использованию квадратных скобок, например, dat[[1]]), и затем можем построить модель по базовым данным, а затем используем функцию predict() для интересующих нас данных после даты окончания сбора данных.
См. страницу [Итерация, циклы и списки], чтобы получить дополнительную информацию о purrr.
ВНИМАНИЕ: Обратите внимание на использование simulate_pi = FALSE внутри аргумента predict(). Это происходит из-за того, что поведение по умолчанию для trending - использовать пакет ciTools, чтобы оценить интервал прогнозирования. Это не работает, если есть количество NA, а также создает более детальные интервалы. см. детали в ?trending::predict.trending_model_fit.
# разделяем данные для построения модели и прогнозирования
dat <- counts %>%
group_by(epiweek <= cut_off) %>%
group_split()
## определяем модель, которую мы хотим построить (отрицательная биномиальная)
model <- glm_nb_model(
## задаем число случаев как интересующий результат
case_int ~
## используем epiweek, чтобы учесть тренд
epiweek +
## используем условия фурье, чтобы учесть сезонность
fourier
)
# определяем, какие данные использовать для построения, а какие - для прогнозирования
fitting_data <- pluck(dat, 2)
pred_data <- pluck(dat, 1) %>%
select(case_int, epiweek, fourier)
# строим модель
fitted_model <- trending::fit(model, data.frame(fitting_data))
# получаем доверительный интервал и оценки по построенным данным
observed <- fitted_model %>%
predict(simulate_pi = FALSE)
# прогнозируем с данными, с которыми нужен прогноз
forecasts <- fitted_model %>%
predict(data.frame(pred_data), simulate_pi = FALSE)
## объединяем базовый и прогнозный набор данных
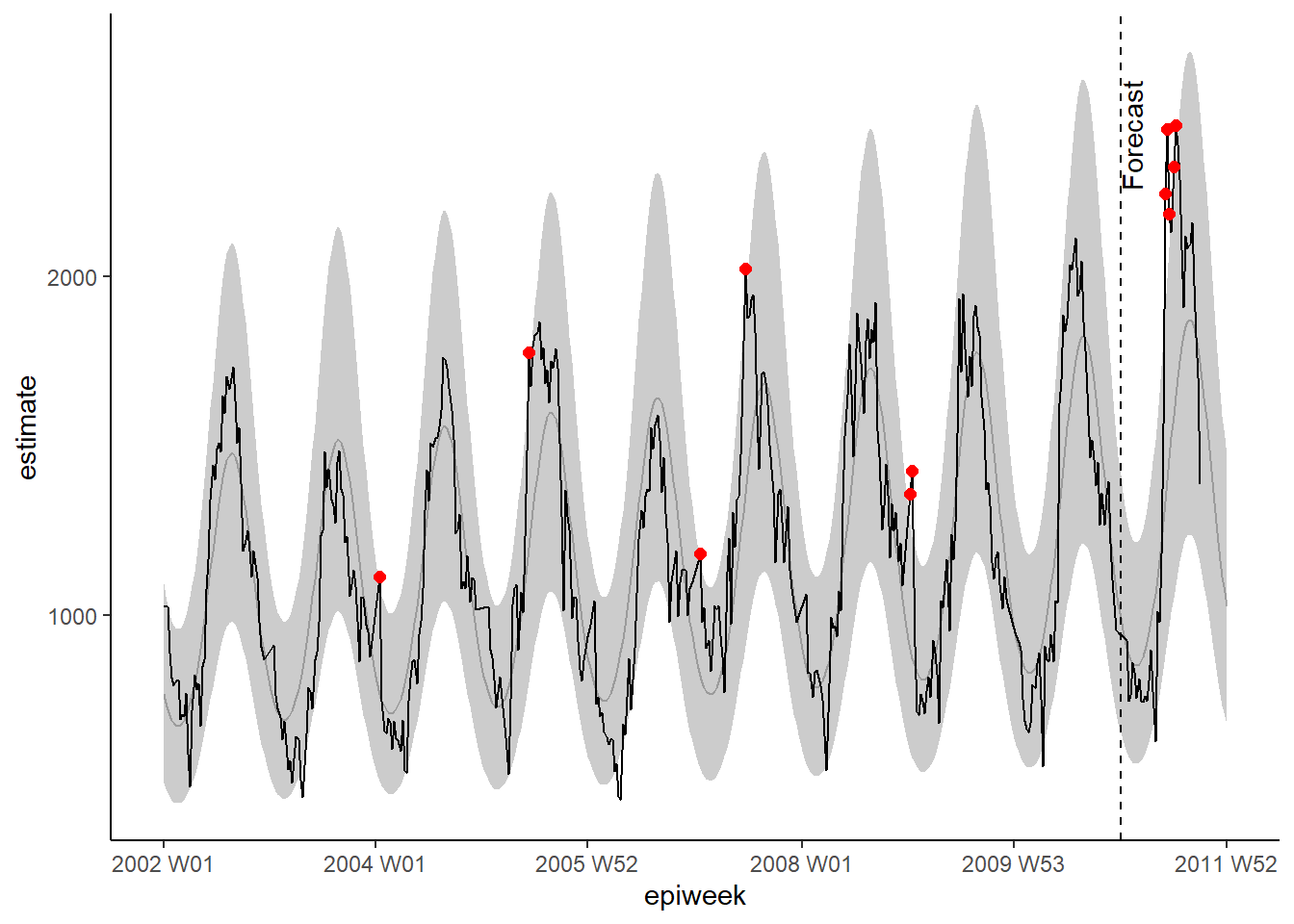
observed <- bind_rows(observed$result, forecasts$result)Как и ранее, мы можем визуализировать нашу модель с помощью ggplot. Мы выделяем предупреждения красными точками для наблюдаемого количества, превышающего 95% интервал прогнозирования. В этот раз мы также добавляем вертикальную линию к подписи, чтобы показать, где начинается прогнозирование.
## строим график регрессии
ggplot(data = observed, aes(x = epiweek)) +
## добавляем линию для оценки модели
geom_line(aes(y = estimate),
col = "grey") +
## добавляем полосу для интервалов прогнозирования
geom_ribbon(aes(ymin = lower_pi,
ymax = upper_pi),
alpha = 0.25) +
## добавляем линию для наблюдаемого количества случаев
geom_line(aes(y = case_int),
col = "black") +
## откладываем на графике точки для наблюдаемого количества, превышающего ожидаемое
geom_point(
data = filter(observed, case_int > upper_pi),
aes(y = case_int),
colour = "red",
size = 2) +
## добавляем вертикальную линию и подпись, чтобы показать, где начинается прогнозирование
geom_vline(
xintercept = as.Date(cut_off),
linetype = "dashed") +
annotate(geom = "text",
label = "Forecast",
x = cut_off,
y = max(observed$upper_pi) - 250,
angle = 90,
vjust = 1
) +
## создаем традиционный график (с черными осями и белым фоном)
theme_classic()Warning: Removed 13 rows containing missing values or values outside the scale range
(`geom_line()`).Валидация прогноза
Помимо проверки остатков, важно изучить, насколько хорошо ваша модель при прогнозировании случаев в будущем. Это дает представление о том, насколько надежны ваши пороговые предупреждения.
Традиционный способ валидации заключается в том, чтобы проверить, насколько хорошо вы можете предсказать последний год, предшествующий текущему (поскольку вы еще не знаете подсчетов для “текущего года”). Например, в нашем наборе данных мы используем данные с 2002 по 2009 год, чтобы предсказать 2010 год, а затем посмотрим, насколько точны эти прогнозы. Затем мы перестраиваем модель, добавляя в нее данные за 2010 год и используем их для прогнозирования количествав 2011 году.
Как можно увидеть на рисунке ниже, подготовленном Hyndman и соавторами в “Принципах и практике прогнозирования”.
 рисунок приведен с разрешения авторов
рисунок приведен с разрешения авторов
Недостатком является то, что вы не используете все доступные для вас данные, и это не финальная модель, которую вы используете для прогноза.
Альтернативным вариантом является использование метода, называемого кросс-валидацией. В этом сценарии перебираются все имеющиеся данные, чтобы построить несколько моделей для прогнозирования на один год вперед. В каждой модели используется все больше и больше данных, как показано на рисунке ниже из той же [работы Хиндмана и соавторов]((https://otexts.com/fpp3/). Например, первая модель использует 2002, чтобы спрогнозировать 2003, вторая использует 2002 и 2003 для прогнозирования 2004, и так далее.  рисунок приведен с разрешения авторов
рисунок приведен с разрешения авторов
Ниже мы используем функцию map() из пакета purrr, чтобы пройти цикл по каждому набору данных. Мы затем размещаем оценки в один набор данных и объединяем его с оригинальным количеством случаев, чтобы использовать пакет yardstick для расчета мер точности. Мы рассчитываем четыре меры, включая: средняя квадратическая ошибка (RMSE), средняя абсолютная ошибка
(MAE), Средняя абсолютная масштабированная ошибка (MASE), Средняя абсолютная процентная ошибка (MAPE).
ВНИМАНИЕ: Обратите внимание на использование simulate_pi = FALSE внутри аргумента predict(). Это происходит, поскольку поведение trending по умолчанию состоит в использовании пакета ciTools для оценки интервала прогнозирования. Это не работает, если есть количество NA, а также создает более детальные интервалы. См. детали в ?trending::predict.trending_model_fit.
## Перекрестная валидация: прогнозирование на неделю(и) вперед на основе скользящего окна
## расширим данные путем скольжения по окнам в 52 недели (до + после)
## чтобы спрогнозировать на 52 вперед
## (создает все более длинные и длинные цепочки наблюдений - сохраняет более старые данные)
## определяем окно, по которому мы хотим скользить
roll_window <- 52
## определяем недели вперед, на которые хотим спрогнозировать
weeks_ahead <- 52
## создаем набор данных повторяющихся все более длинных данных
## подписываем каждый набор данных уникальным id
## используем только случаи до интересующего года (т.е 2011)
case_roll <- counts %>%
filter(epiweek < cut_off) %>%
## сохраняем только переменные недели и количества случаев
select(epiweek, case_int) %>%
## отбрасываем последние x наблюдений
## в зависимости от того, на сколько недель вперед прогнозируем
## (иначе прогноз будет до "неизвестно")
slice(1:(n() - weeks_ahead)) %>%
as_tsibble(index = epiweek) %>%
## скользим по каждой недели в x по окнам, чтобы создать ID группы
## в зависимости от того, что задают скользящие окна
stretch_tsibble(.init = roll_window, .step = 1) %>%
## отбрасываем первые несколько - поскольку нет случаев "до"
filter(.id > roll_window)
## для каждого уникального набора данных выполняем код ниже
forecasts <- purrr::map(unique(case_roll$.id),
function(i) {
## сохраняем только текущий раз построения
mini_data <- filter(case_roll, .id == i) %>%
as_tibble()
## создаем пустой набор данных для прогнозирования
forecast_data <- tibble(
epiweek = seq(max(mini_data$epiweek) + 1,
max(mini_data$epiweek) + weeks_ahead,
by = 1),
case_int = rep.int(NA, weeks_ahead),
.id = rep.int(i, weeks_ahead)
)
## добавляем данные прогноза к оригинальным
mini_data <- bind_rows(mini_data, forecast_data)
## определяем последнюю дату на основе последних неотсутствующих данных о количестве
cv_cut_off <- mini_data %>%
## сохраняем только неотсутствующие строки
drop_na(case_int) %>%
## получаем последнюю неделю
summarise(max(epiweek)) %>%
## извлекаем, чтобы она не была в датафрейме
pull()
## создаем mini_data в виде tsibble
mini_data <- tsibble(mini_data, index = epiweek)
## определяем условия фурье (sincos)
mini_data <- mini_data %>%
mutate(
## объединяем условия фурье для недель до и после даты окончания сбора данных
fourier = rbind(
## получаем условия фурье по предыдущим годам
forecast::fourier(
## сохраняем только строки до даты окончания сбора данных
filter(mini_data,
epiweek <= cv_cut_off),
## включаем один набор условий sin cos
K = 1
),
## прогнозируем условия фурье для следующего года (используя базовые данные)
fourier(
## сохраняем только строки до даты окончания сбора данных
filter(mini_data,
epiweek <= cv_cut_off),
## включаем один набор условий sin cos
K = 1,
## прогнозируем на 52 недели вперед
h = weeks_ahead
)
)
)
# разделяем данные для построения регрессии и прогнозирования
dat <- mini_data %>%
group_by(epiweek <= cv_cut_off) %>%
group_split()
## определяем модель, которую мы хотим построить (отрицательная биномиальная)
model <- glm_nb_model(
## задаем число случаев в качестве интересующего результата
case_int ~
## используем эпиднеделю (epiweek), чтобы учесть тренд
epiweek +
## используем условия фурье, чтобы учесть сезонность
fourier
)
# определяем, какие данные использовать для построения регрессии и какие - для прогноза
fitting_data <- pluck(dat, 2)
pred_data <- pluck(dat, 1)
# строим модель
fitted_model <- trending::fit(model, fitting_data)
# прогнозируем с данными, с которыми мы хотим выполнить прогноз
forecasts <- fitted_model %>%
predict(data.frame(pred_data), simulate_pi = FALSE)
forecasts <- data.frame(forecasts$result[[1]]) %>%
## сохраняем только неделю и прогнозную оценку
select(epiweek, estimate)
}
)
## превращаем список в датафрейм со всеми прогнозами
forecasts <- bind_rows(forecasts)
## соединяем прогнозы с наблюдаемыми данными
forecasts <- left_join(forecasts,
select(counts, epiweek, case_int),
by = "epiweek")
## используя {yardstick} рассчитываем метрики
## RMSE: средняя квадратическая ошибка
## MAE: средняя абсолютная ошибка
## MASE: средняя абсолютная масштабированная ошибка
## MAPE: средняя абсолютная процентная ошибка
model_metrics <- bind_rows(
## в вашем спрогнозированном наборе данных сравниваем наблюдаемые данные с прогнозом
rmse(forecasts, case_int, estimate),
mae( forecasts, case_int, estimate),
mase(forecasts, case_int, estimate),
mape(forecasts, case_int, estimate),
) %>%
## сохраняем только тип метрики и его выходной результат
select(Metric = .metric,
Measure = .estimate) %>%
## превращаем в широкий формат, чтобы можно было после этого связать строки
pivot_wider(names_from = Metric, values_from = Measure)
## выдаем метрики модели
model_metrics# A tibble: 1 × 4
rmse mae mase mape
<dbl> <dbl> <dbl> <dbl>
1 252. 199. 1.96 17.3пакет surveillance
В этом разделе мы используем пакет surveillance, чтобы создать пороги предупреждения, основанные на алгоритмах обнаружения вспышек. Существуют ряд разных методов, доступных в этом пакете, однако мы сфокусируемся на двух опциях. Для получения детальной информации см. работы по применению и теории использованных алгоритмов.
Первая опция использует улучшенный метод Фаррингтона. Он строит отрицательную биномиальную обобщенную линейную модель (glm) (включая тренд) и снижает веса прошлых вспышек (аномальных значений) для создания порогового уровня.
Второй вариант - использовать метод glrnb. Он также строит отрицательную биномиальную обобщенную линейную модель (glm), но включает тренд и условия фурье (поэтому мы его считаем предпочтительным). Регрессия используется для расчета “контрольного среднего” (~построенные значения) - затем используется рассчитанная статистика обобщенного отношения правдоподобия, чтобы оценить, есть ли смещение среднего для каждой недели. Обратите внимание, что порог для каждой недели принимает во внимание предыдущие недели, так что есть существует устойчивый сдвиг, сработает предупреждение. (Также обратите вниимание, что после каждого срабатывания предупреждения, алгоритм перезапускается)
Чтобы работать с пакетом surveillance, нам нужно сначала определить объект “временной ряд эпиднадзора (surveillance time series)” (используя функцию sts() ), чтобы он соответствовал рамкам.
## определяем объект временной ряд эпиднадзора
## примечание: вы можете включить знаменатель с популяционным объектом (см. ?sts)
counts_sts <- sts(observed = counts$case_int[!is.na(counts$case_int)],
start = c(
## подмножество, чтобы сохранить только год с момента start_date
as.numeric(str_sub(start_date, 1, 4)),
## подмножество, чтобы сохранить только неделю с момента start_date
as.numeric(str_sub(start_date, 7, 8))),
## определяем тип данных (в данном случае случаев в неделю)
freq = 52)
## определяем диапазон недель, которые мы хотим включить (т.е. период прогнозирования)
## примечание: объект sts только считает наблюдения без присваивания им идентификатора
## недели или года - поэтому мы используем наши данные, чтобы определить соответствующие наблюдения
weekrange <- cut_off - start_dateМетод Фаррингтона
Мы определяем каждый из наших параметров для метода Фаррингтона в списке list. Затем мы выполняем алгоритм, используя farringtonFlexible() и затем мы можем извлечь порог предупреждения, используя farringtonmethod@upperbound, чтобы включить его в наш набор данных. Можно также извлечь ИСТИНУ/ЛОЖЬ (TRUE/FALSE) для каждой недели, чтобы узнать, сработало ли предупреждение (превышен ли порог), используя farringtonmethod@alarm.
## определяем контроль
ctrl <- list(
## определяем для какого временного периода нам нужен порог (т.е. 2011)
range = which(counts_sts@epoch > weekrange),
b = 9, ## на сколько лет назад базовый уровень
w = 2, ## размер скользящего окна в неделях
weightsThreshold = 2.58, ## изменение весов прошлых вспышек (улучшенный метод нуфайли (noufaily) - оригинальный предлагает 1)
## pastWeeksNotIncluded = 3, ## используем все доступные недели (noufaily предлагает отбросить 26)
trend = TRUE,
pThresholdTrend = 1, ## 0.05 как правило, однако в улучшенном методе рекомендуется 1 (т.е. всегда сохранять)
thresholdMethod = "nbPlugin",
populationOffset = TRUE
)
## применяем гибкий метод фаррингтона
farringtonmethod <- farringtonFlexible(counts_sts, ctrl)
## создаем новую переменную в оригинальном наборе данных, называемую threshold (порог)
## содержашую верхнюю границу из фаррингтона
## примечание: это только для недель в 2011 (так что нужно подмножество строк)
counts[which(counts$epiweek >= cut_off &
!is.na(counts$case_int)),
"threshold"] <- farringtonmethod@upperboundМы можем визуализировать результаты на ggplot, как это делали ранее.
ggplot(counts, aes(x = epiweek)) +
## добавляем наблюдаемое количество случаев как линию
geom_line(aes(y = case_int, colour = "Observed")) +
## добавляем верхнюю границу алгоритма аберрации
geom_line(aes(y = threshold, colour = "Alert threshold"),
linetype = "dashed",
size = 1.5) +
## определяем цвета
scale_colour_manual(values = c("Observed" = "black",
"Alert threshold" = "red")) +
## создаем традиционный график (с черными осями и белым фоном)
theme_classic() +
## удаляем заголовок легенды
theme(legend.title = element_blank())Метод GLRNB
Аналогично, для метода GLRNB (процедура на основе обобщенного отношения правдоподобия), мы определяем каждый из наших параметров в списке list, затем строим алгоритм и извлекаем верхние границы.
ВНИМАНИЕ: Этот метод использует “грубую силу” (похож на бутстреппинг) для расчета порогов, поэтому может занять много времени!
См. виньетку GLRNB для получения дополнительной информации.
## определяем опции контроля
ctrl <- list(
## определяем, для какого периода нам нужен порог (т.е. 2011)
range = which(counts_sts@epoch > weekrange),
mu0 = list(S = 1, ## количество условий фурье (гармоник), которые нужно включить
trend = TRUE, ## включать ли тренд
refit = FALSE), ## строить ли заново модель после каждого сигнала предупреждения
## cARL = порог для статистики обобщенной линейной модели (GLR) (спорный)
## 3 ~ средняя позиция для минимизации ложных срабатываний
## 1 строит до 99% интервала прогнозирования glm. примечание - с изменениями после пиков (снижается порог предупреждения)
c.ARL = 2,
# theta = log(1.5), ## равняется 50% увеличению случаев во вспышке
ret = "cases" ## выдает верхнюю границу порога как количество случаев
)
## применяем метод glrnb
glrnbmethod <- glrnb(counts_sts, control = ctrl, verbose = FALSE)
## создаем новую переменную в оригинальном наборе данных под именем threshold (порог)
## содержащую верхнюю границу glrnb
## примечание: это только для недель в 2011 (так что нужно подмножество строк)
counts[which(counts$epiweek >= cut_off &
!is.na(counts$case_int)),
"threshold_glrnb"] <- glrnbmethod@upperboundВизуализируем выходные результаты как ранее.
ggplot(counts, aes(x = epiweek)) +
## добавляем наблюдаемое количество случаев как линию
geom_line(aes(y = case_int, colour = "Observed")) +
## добавляем верхнюю границу алгоритма аберрации
geom_line(aes(y = threshold_glrnb, colour = "Alert threshold"),
linetype = "dashed",
size = 1.5) +
## определяем цвета
scale_colour_manual(values = c("Observed" = "black",
"Alert threshold" = "red")) +
## создаем традиционный график (с черными осями и белым фоном)
theme_classic() +
## удаляем заголовок легенды
theme(legend.title = element_blank())23.8 Прерванные временные ряды
Прерванные временные ряды (также называемые сегментированная регрессия или анализ вмешательств), часто используются при оценке воздействия вакцин на заболеваемость. Но они могут использоваться для оценки широкого спекта вмешательств или заражений. Например, изменений в больничных процедурах или появление нового штамма заболевания в популяции. В данном примере представим, что появился новый штамм Кампилобактера в Германии в конце 2008, и посмотрим, повлияло ли это на количество случаев. Мы снова используем отрицательную биномиальную регрессию. В этот раз регрессия будет поделена на две части, одна до вмешательства (или в данном случае появления нового штамма) и одна после (периоды до и после). Это позволит нам рассчитать коэффициент частоты заболеваемости, сравнивающий два периода. Объяснение уравнения может прояснить ситуацию (если нет - просто игнорируйте его!).
Отрицательную биномиальную регрессию можно определить следующим образом:
\[\log(Y_t)= β_0 + β_1 \times t+ β_2 \times δ(t-t_0) + β_3\times(t-t_0 )^+ + log(pop_t) + e_t\]
Где: \(Y_t\) - количество случаев, наблюдаемых во время \(t\)
\(pop_t\) - размер популяции в 100,000 во время \(t\) (здесь не используется)
\(t_0\) - последний год периода до (включая время перехода, если есть)
\(δ(x\) - индикаторная функция (она будет it0, если x≤0 и 1 при x>0)
\((x)^+\) оператор отсечки (он равен x если x>0 и 0 в других случаях)
\(e_t\) означает остаток При необходимости можно добавить дополнительные условия тренда и сезона.
\(β_2 \times δ(t-t_0) + β_3\times(t-t_0 )^+\) - обобщенная линейная часть периода после и равна нулю в период до. Это означает, что оценки \(β_2\) и \(β_3\) являются эффектами вмешательства.
Нам нужно пересчитать условия фурье без прогнозирования, поскольку мы используем все доступные нам данные (т.е. ретроспективно). Кроме того, нам нужно рассчитать дополнительные условия, необходимые для регрессии.
## добавляем условия фурье, используя переменные epiweek и case_int
counts$fourier <- select(counts, epiweek, case_int) %>%
as_tsibble(index = epiweek) %>%
fourier(K = 1)
## определяем неделю вмешательства
intervention_week <- yearweek("2008-12-31")
## определяем переменные для регрессии
counts <- counts %>%
mutate(
## соответствует t в формуле
## количество недель (можно просто использовать напрямую переменную epiweeks)
# linear = row_number(epiweek),
## соответствует дельта(t-t0) в формуле
## период до и после вмешательства
intervention = as.numeric(epiweek >= intervention_week),
## соответствует (t-t0)^+ в формуле
## количество недель после вмешательства
## (выбираем более большое число между 0 и тем, что будет получено в расчетах)
time_post = pmax(0, epiweek - intervention_week + 1))Затем мы используем эти условия для построения отрицательной биномиальной регрессии и создаем таблицу с процентными изменениями. Этот пример показывает, что не было существенного изменения.
ВНИМАНИЕ: Обратите внимание на использование simulate_pi = FALSE внутри аргумента predict(). Это происходит потому, что поведение по умолчанию trending состоит в том, чтобы использовать пакет ciTools для оценки интервала прогнозирования. Это не работает, если существует количество NA, и также создает более детальные интервалы. См. детали в ?trending::predict.trending_model_fit.
## определяем модель, которую надо построить (отрицательная биномиальная)
model <- glm_nb_model(
## задаем количество случаев как интересующий результат
case_int ~
## используем эпиднеделю (epiweek), чтобы учесть тренд
epiweek +
## используем условия фурье, чтобы учесть сезонность
fourier +
## добавляем в периоде до или после
intervention +
## добавляем время после вмешательства
time_post
)
## строим модель, используя набор данных counts
fitted_model <- trending::fit(model, counts)
## рассчитываем доверительные интервалы и интервалы прогнозирования
observed <- predict(fitted_model, simulate_pi = FALSE)## показываем оценки и процентное изменение в таблице
fitted_model %>%
## извлекаем оригинальную отрицательную биномиальную регрессию
get_model() %>%
## получаем аккуратный датафрейм результатов
tidy(exponentiate = TRUE,
conf.int = TRUE) %>%
## сохраняем только значение вмешательства
filter(term == "intervention") %>%
## изменяем коэффициент частоты заболеваемости на процентное изменение для оценки и ДИ
mutate(
## для каждого из интересующих столбцов создаем новый столбец
across(
all_of(c("estimate", "conf.low", "conf.high")),
## применяем формулу, чтобы рассчитать процентные изменения
.f = function(i) 100 * (i - 1),
## добавляем суффикс к именам новых столбцов "_perc"
.names = "{.col}_perc")
) %>%
## сохраняем (и переименовываем) некоторые столбцы
select("IRR" = estimate,
"95%CI low" = conf.low,
"95%CI high" = conf.high,
"Percentage change" = estimate_perc,
"95%CI low (perc)" = conf.low_perc,
"95%CI high (perc)" = conf.high_perc,
"p-value" = p.value)Как и ранее, мы можем визуализировать выходные результаты регрессии.
estimate_res <- data.frame(observed$result)
ggplot(estimate_res, aes(x = epiweek)) +
## добавляем наблюдаемое количество случаев как линию
geom_line(aes(y = case_int, colour = "Observed")) +
## добавляем линию для оценки модели
geom_line(aes(y = estimate, col = "Estimate")) +
## добавляем полосу для интервалов прогнозирования
geom_ribbon(aes(ymin = lower_pi,
ymax = upper_pi),
alpha = 0.25) +
## добавляем вертикальную линию и подпись, чтобы показать, где началось прогнозирование
geom_vline(
xintercept = as.Date(intervention_week),
linetype = "dashed") +
annotate(geom = "text",
label = "Intervention",
x = intervention_week,
y = max(observed$upper_pi),
angle = 90,
vjust = 1
) +
## определяем цвета
scale_colour_manual(values = c("Observed" = "black",
"Estimate" = "red")) +
## создаем традиционный график (с черными осями и белым фоном)
theme_classic()Warning: Unknown or uninitialised column: `upper_pi`.Warning in max(observed$upper_pi): no non-missing arguments to max; returning
-Inf23.9 Ресурсы
Учебник Прогнозирование: принципы и практики
Кейсы EPIET по анализу временных рядов
Курс Penn State Рукопись по пакету Surveillance