pacman::p_load(
rio, # импорт файлов
here, # путь к файлам
tidyverse, # управление данными + графика ggplot2,
stringr, # манипуляции с текстовыми последовательностями
purrr, # циклы над объектами по принципам аккуратных данных
gtsummary, # сводная статистика и тесты
broom, # приведение результатов регрессий в аккуратный вид
lmtest, # тесты на отношение правдоподобия
parameters, # альтернатива для приведения результатов регрессий в аккуратный вид
see # альтернатива для визуализации форест-графиков
)19 Однофакторная и многофакторная регрессия
На этой странице демонстрируется использование функций регрессии из базового R, таких как glm(), и пакета gtsummary для рассмотрения связей между переменными (например, отношения шансов, отношения рисков и отношения моментных рисков). Для вычистки выходных данных регрессии также используются такие функции, как tidy() из пакета broom.
- Однофакторная: таблицы два-на-два
- Стратифицированная: Оценка Мантеля-Хензеля
- Многофакторная: выбор переменной, выбор модели, итоговая таблица
- Форест-графики
Для информации о Пропорциональной регрессии рисков Кокса см. страницу Анализ выживаемости.
ПРИМЕЧАНИЕ: Мы используем понятие многофакторная для регрессии с несколькими независимыми переменными. В этом смысле моделью с множественным откликом будет регрессия с несколькими результатами - см. эту редакционную статью для получения детальной информации
19.1 Подготовка
Загрузка пакетов
Данный фрагмент кода показывает загрузку пакетов, необходимых для анализа. В данном руководстве мы фокусируемся на использовании p_load() из пакета pacman, которая устанавливает пакет, если необходимо, и загружает его для использования. Вы можете также загрузить установленные пакеты с помощью library() из базового R. См. страницу Основы R для получения дополнительной информации о пакетах R.
Импорт данных
Мы импортируем набор данных о случаях имитированной эпидемии Эболы. Если вы хотите выполнять действия параллельно, кликните, чтобы скачать “чистый” построчный список (как файл .rds). Импортируйте данный с помощью функции import() из пакета rio (он работает с многими типами файлов, такими как .xlsx, .csv, .rds - см. детали на странице Импорт и экспорт).
# импорт построчного списка
linelist <- import("linelist_cleaned.rds")Первые 50 строк построчного списка отображены ниже.
Вычистка данных
Сохранение независимых переменных
Мы храним имена столбцов независимых переменных как текстовый вектор. На него мы будем ссылаться позже.
## определяем интересующие переменные
explanatory_vars <- c("gender", "fever", "chills", "cough", "aches", "vomit")Конвертация в 1 и 0
Ниже мы конвертируем столбцы независимых переменных с “yes”/“no”, “m”/“f”, и “dead”/“alive” на 1 / 0, чтобы соответствовать ожиданиям моделей логистической регрессии. Чтобы это сделать эффективным образом, используем across() из dplyr, чтобы преобразовать сразу несколько столбцов. Функция, которую мы применяем к каждому столбцу, называется case_when() (также из dplyr), она применяет логику для конвертации указанных значений в 1 и 0. См. разделы по across() и case_when() на странице Вычистка данных и ключевые функции).
Примечание: “.” ниже представляет столбец, который обрабатывается в этот момент функцией across().
## конвертируем двоичные переменные в 0/1
linelist <- linelist %>%
mutate(across(
.cols = all_of(c(explanatory_vars, "outcome")), ## для каждого указанного столбца и "outcome"
.fns = ~case_when(
. %in% c("m", "yes", "Death") ~ 1, ## перекодируем, мужской пол, да и смерть на 1
. %in% c("f", "no", "Recover") ~ 0, ## женский пол, нет и выздоровление на 0
TRUE ~ NA_real_) ## в остальных случаях - отсутствующее значение
)
)Удаление строк с отсутствующими значениями
Чтобы удалить строки с отсутствующими значениям, мы можем использовать функцию drop_na() из tidyr. Однако мы хотим это сделать только для строк, у которых отсутствуют значения в интересующих нас столбцах.
Первое, что нам нужно сделать, это убедиться, что наш вектор независимых переменных explanatory_vars включает столбец возраста age (age выдал бы ошибку в предыдущей операции case_when(), которая была только для двоичных переменных). Затем мы передаем linelist в команду drop_na(), чтобы удалить строки с отсутствующими значениями в столбце outcome или любом из столбцов независимых переменных explanatory_vars.
До выполнения кода количество строк в linelist составляет nrow(linelist).
## добавляем age_category к независимым переменным
explanatory_vars <- c(explanatory_vars, "age_cat")
## удаляем строки с отсутствующей информацией по интересующим переменным
linelist <- linelist %>%
drop_na(any_of(c("outcome", explanatory_vars)))Количество строк, остающихся в linelist, составляет nrow(linelist).
19.2 Однофакторный
Как и в материалах на странице Описательные таблицы, конкретный способ применения определит, какой пакет R вам использовать. Мы представим две опции для проведения однофакторного анализа:
- Используйте функции, доступные в базовом R, чтобы быстро напечатать результаты в консоли. Используйте пакет broom, чтобы привести выходные данные в аккуратный вид.
- Используйте пакет gtsummary, чтобы моделировать и получать готовые к публикации выходные данные.
базовый R
Линейная регрессия
Функция базового R lm() создает линейную регрессию, оценивая отношения между числовыми независимыми и зависимыми переменными, между которыми предполагается линейное отношение.
Задайте уровнение в виде формулы, разделив названия столбцов независимой и зависимой переменной тильдой ~. Также уточните набор данных в аргументе data =. Определите результаты модели как объект R, чтобы их можно было использовать позже.
lm_results <- lm(ht_cm ~ age, data = linelist)Затем вы можете выполнить summary() для результатов модели, чтобы увидеть коэффициенты (возможные значения), P-значение, остатки и другие меры.
summary(lm_results)
Call:
lm(formula = ht_cm ~ age, data = linelist)
Residuals:
Min 1Q Median 3Q Max
-128.579 -15.854 1.177 15.887 175.483
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 69.9051 0.5979 116.9 <2e-16 ***
age 3.4354 0.0293 117.2 <2e-16 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 23.75 on 4165 degrees of freedom
Multiple R-squared: 0.7675, Adjusted R-squared: 0.7674
F-statistic: 1.375e+04 on 1 and 4165 DF, p-value: < 2.2e-16Альтернативно вы можете использовать функцию tidy() из пакета broom, чтобы извлечь результаты в таблицу. Результаты нам говорят, что для каждого года увеличения возраста рост увеличивается на 3.5 см. и это является статистически значимым.
tidy(lm_results)# A tibble: 2 × 5
term estimate std.error statistic p.value
<chr> <dbl> <dbl> <dbl> <dbl>
1 (Intercept) 69.9 0.598 117. 0
2 age 3.44 0.0293 117. 0Затем вы можете также использовать эту регрессию, чтобы добавить ее к ggplot, для этого мы сначала переносим точки из наблюдаемых данных и построенной линии в один датафрейм, используя функцию augment() из broom.
## сводим точки регрессии и наблюдаемые данные в один набор данных
points <- augment(lm_results)
## строим график данных, используя возраст в качестве оси x
ggplot(points, aes(x = age)) +
## добавляем точки для роста
geom_point(aes(y = ht_cm)) +
## добавляем линию регрессии
geom_line(aes(y = .fitted), colour = "red")Также можно добавить простую линейную регрессию сразу в ggplot, используя функцию geom_smooth().
## добавляем данные к графику
ggplot(linelist, aes(x = age, y = ht_cm)) +
## показываем точки
geom_point() +
## добавляем линейную регрессию
geom_smooth(method = "lm", se = FALSE)`geom_smooth()` using formula = 'y ~ x'См. раздел Ресурсы в конце главы, в котором приведены более подробные руководства.
Логистическая регрессия
Функция glm() из пакета stats (часть базового R) используется, чтобы строить Обобщенные линейные модели (GLM).
glm() может использоваться для однофакторной и многофакторной логистической регрессии (например, чтобы получить отношения шансов). Вот ключевые части:
# аргументы для glm()
glm(formula, family, data, weights, subset, ...)formula =Модель задакется вglm()в виде уравнения, с зависимыми переменными слева, а независимыми переменными справа от тильды~.
family =Определяет тип модели, который надо выполнить. Для логистической регрессии используйтеfamily = "binomial", для пуассоновской - используйтеfamily = "poisson". Другие примеры приведены в таблице ниже.
data =Уточните ваш датафрейм
Если необходимо, вы можете также уточнить функцию связку через синтаксис family = familytype(link = "linkfunction")). Вы можете более подробно ознакомиться с документацией о других семействах и опциональных аргументах, таких как weights = и subset = (?glm).
| Семейство | Функция связки по умолчанию |
|---|---|
"биномиальное" |
(link = "logit") |
"гауссово" |
(link = "identity") |
"гамма" |
(link = "inverse") |
"обратное гауссово" |
(link = "1/mu^2") |
"пуассона" |
(link = "log") |
"квази" |
(link = "identity", variance = "constant") |
"квазибиномиальное" |
(link = "logit") |
"квазипуассона" |
(link = "log") |
При выполнении glm() чаще всего результаты сохраняются как именованный объект R. Затем вы можете напечатать результаты в консоли, используя summary(), как показано ниже, либо провести другие операции с результатами (например, возвести в степень).
Если вам нужно выполнить отрицательную биномиальную регрессию, вы можете использовать пакет MASS; glm.nb() использует тот же синтаксис, что glm(). Для описания работы разных видов регрессии, см. страницу статистики UCLA.
Однофакторная glm()
В данном примере мы оцениваем связь между разными возрастными категориями и исходом смерти (закодирована как 1 в разделе Подготовка). Ниже представлена однофакторная модель исхода outcome по возрастной категории age_cat. Мы сохраняем выходные данные модели как model, затем печатаем их с помощью summary() в консоли. Обратите внимание, что представленные оценки являются логарифмами отношения шансов, и что базовый уровень является первым уровнем фактора в возрастной категории age_cat (“0-4”).
model <- glm(outcome ~ age_cat, family = "binomial", data = linelist)
summary(model)
Call:
glm(formula = outcome ~ age_cat, family = "binomial", data = linelist)
Coefficients:
Estimate Std. Error z value Pr(>|z|)
(Intercept) 0.233738 0.072805 3.210 0.00133 **
age_cat5-9 -0.062898 0.101733 -0.618 0.53640
age_cat10-14 0.138204 0.107186 1.289 0.19726
age_cat15-19 -0.005565 0.113343 -0.049 0.96084
age_cat20-29 0.027511 0.102133 0.269 0.78765
age_cat30-49 0.063764 0.113771 0.560 0.57517
age_cat50-69 -0.387889 0.259240 -1.496 0.13459
age_cat70+ -0.639203 0.915770 -0.698 0.48518
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
(Dispersion parameter for binomial family taken to be 1)
Null deviance: 5712.4 on 4166 degrees of freedom
Residual deviance: 5705.1 on 4159 degrees of freedom
AIC: 5721.1
Number of Fisher Scoring iterations: 4Чтобы изменить базовый уровень определенной переменной, убедитесь, что столбец относится к классу фактор и передвиньте нужный уровень на первую позицию с помощью fct_relevel() (см. страницу Факторы). Например, ниже мы берем столбец age_cat и устанавливаем “20-29” в качестве базового уровня до передачи измененного датафрейма в glm().
linelist %>%
mutate(age_cat = fct_relevel(age_cat, "20-29", after = 0)) %>%
glm(formula = outcome ~ age_cat, family = "binomial") %>%
summary()
Call:
glm(formula = outcome ~ age_cat, family = "binomial", data = .)
Coefficients:
Estimate Std. Error z value Pr(>|z|)
(Intercept) 0.26125 0.07163 3.647 0.000265 ***
age_cat0-4 -0.02751 0.10213 -0.269 0.787652
age_cat5-9 -0.09041 0.10090 -0.896 0.370220
age_cat10-14 0.11069 0.10639 1.040 0.298133
age_cat15-19 -0.03308 0.11259 -0.294 0.768934
age_cat30-49 0.03625 0.11302 0.321 0.748390
age_cat50-69 -0.41540 0.25891 -1.604 0.108625
age_cat70+ -0.66671 0.91568 -0.728 0.466546
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
(Dispersion parameter for binomial family taken to be 1)
Null deviance: 5712.4 on 4166 degrees of freedom
Residual deviance: 5705.1 on 4159 degrees of freedom
AIC: 5721.1
Number of Fisher Scoring iterations: 4Печать результатов
Для большинства случаев использования необходимо внести некоторые изменения в указанные выше выходные данные. Функция tidy() из пакета broom является удобной, чтобы сделать результаты модели презентабельными.
Здесь мы демонстрируем, как комбинировать выходные данные модели с таблицей подсчетов.
- Получите возведенные в степень оценочные логарифмы отношения шансов и доверительных интервалов, передав модель в функцию
tidy()и установивexponentiate = TRUEиconf.int = TRUE.
model <- glm(outcome ~ age_cat, family = "binomial", data = linelist) %>%
tidy(exponentiate = TRUE, conf.int = TRUE) %>% # возводим в степень и создаем ДИ
mutate(across(where(is.numeric), round, digits = 2)) # округляем все числовые столбцыWarning: There was 1 warning in `mutate()`.
ℹ In argument: `across(where(is.numeric), round, digits = 2)`.
Caused by warning:
! The `...` argument of `across()` is deprecated as of dplyr 1.1.0.
Supply arguments directly to `.fns` through an anonymous function instead.
# Previously
across(a:b, mean, na.rm = TRUE)
# Now
across(a:b, \(x) mean(x, na.rm = TRUE))Ниже представлена полученная на выходе таблица tibble model:
- Объединяем эти результаты модели с таблицей подсчета. Ниже мы создаем перекрестную таблицу подсчета с помощью функции
tabyl()из пакета janitor, как объяснялось на странице Описательные таблицы.
counts_table <- linelist %>%
janitor::tabyl(age_cat, outcome)Вот как выглядит этот датафрейм counts_table:
Теперь мы можем связать counts_table и результаты модели model вместе горизонтально с помощью bind_cols() (dplyr). Помните, что при использовании bind_cols() строки в двух датафреймах должны идеально соответствовать. В данном коде поскольку мы связываем внутри цепочки канала, мы используем ., чтобы представить поставленный в канал объект counts_table, когда мы его привязываем к модели model. Чтобы завершить процесс, мы используем select(), чтобы выбрать необходимые столбцы и их порядок, и наконец применяем функцию из базового R round() ко всем числовым столбцам, чтобы уточнить два знака после запятой.
combined <- counts_table %>% # начинаем с таблицы подсчета
bind_cols(., model) %>% # объединяем с выходными данными регрессии
select(term, 2:3, estimate, # выбираем и изменяем порядок столбцов
conf.low, conf.high, p.value) %>%
mutate(across(where(is.numeric), round, digits = 2)) ## округляем до 2 знаков после запятойВот так выглядит объединенный датафрейм, красиво напечатанный в виде изображения с помощью функции из flextable. На странице Таблицы для презентации объясняется, как адаптировать такие таблицы с помощью flextable, либо вы можете использовать другие пакеты, такие как knitr или GT.
combined <- combined %>%
flextable::qflextable()Создание цикла для нескольких однофакторных моделей
Ниже мы представим метод с использованием glm() и tidy() для более простого подхода, см. раздел gtsummary.
Чтобы выполнить модели на нескольких переменных воздействия, чтобы получить однофакторные отношения шансов (т.е. без контроля по отношению друг к другу), вы можете использовать подход, описанный ниже. В нем используется str_c() из пакета stringr для создания однофакторных формул (см. Текст и последовательности), выполняется регрессия glm() для каждой формулы, каждый результат регрессии glm() передается в tidy() и наконец объединяются все результаты моделей с помощью bind_rows() из tidyr. Этот подход использует для итераций map() из пакета purrr - см. страницу Итерации, циклы и списки для более подробной информации об этом инструменте.
Создаем вектор имен столбцов независимых переменных. У нас уже есть
explanatory_varsиз раздела Подготовка на этой странице.Используем
str_c(), чтобы создать несколько формул последовательности, при этомoutcomeбудет в левой части, а название столбца изexplanatory_vars- в правой. Точка.заменяет название столбца изexplanatory_vars.
explanatory_vars %>% str_c("outcome ~ ", .)[1] "outcome ~ gender" "outcome ~ fever" "outcome ~ chills"
[4] "outcome ~ cough" "outcome ~ aches" "outcome ~ vomit"
[7] "outcome ~ age_cat"Передайте эти формулы-последовательности в
map()и установите~glm()в качестве функции, которая будет применяться к каждому входному параметру. Внутриglm()установите формулу регрессию какas.formula(.x), где.xбудет заменяться на формулу-последовательность, определенную шагом выше.map()выполнит цикл для каждой из формул-последовательностей, выполнив для каждой регрессию.Выходные данные этой первой функции
map()передаются во вторую командуmap(), которая применяетtidy()к результатам регрессии.Наконец, выходные данные второй
map()(список аккуратных датафреймов) сжимается с помощьюbind_rows(), что приводит к созданию одного датафрейма со всеми однофакторными результатами.
models <- explanatory_vars %>% # начинаем с интересующей переменной
str_c("outcome ~ ", .) %>% # объединяем каждую переменную в формулу ("исход (outcome) ~ интересующая переменная")
# проводим итерацию через каждую однофакторную формулу
map(
.f = ~glm( # по одной передаем формулы в glm()
formula = as.formula(.x), # внутри glm() формула-последовательность - это .x
family = "binomial", # уточняем тип glm (логистическая)
data = linelist)) %>% # набор данных
# приводим результаты регрессий glm, сделанных выше, в аккуратный формат
map(
.f = ~tidy(
.x,
exponentiate = TRUE, # возводим в степень
conf.int = TRUE)) %>% # получаем доверительные интервалы
# сжимаем список выходных данных регрессии в один датафрейм
bind_rows() %>%
# округляем все числовые столбцы
mutate(across(where(is.numeric), round, digits = 2))В этот раз конечный объект models длиннее, поскольку теперь представляет объединенные результаты нескольких однофакторных регрессий. Пролистайте все строки model.
Как и ранее, мы создаем таблицу подсчета из linelist для каждой независимой переменной, связываем ее с models и создаем прекрасную таблицу. Мы начинаем с переменных, проводим по ним итерацию с помощью map(). Мы проводим итерацию по разным определенным пользователям функциям, что требует создания таблицы подсчета с помощью функций dplyr. Затем результаты объединяются и связываются с результатами модели models.
## для каждой независимой переменной
univ_tab_base <- explanatory_vars %>%
map(.f =
~{linelist %>% ## начинаем с построчного списка
group_by(outcome) %>% ## группируем набор данных по исходу
count(.data[[.x]]) %>% ## создаем подсчет по интересующей переменной
pivot_wider( ## разворачиваем на широкий формат (как в кросс-табуляции)
names_from = outcome,
values_from = n) %>%
drop_na(.data[[.x]]) %>% ## удаляем строки с отсутствующими значениями
rename("variable" = .x) %>% ## меняем столбец интересующей переменной на "variable"
mutate(variable = as.character(variable))} ## конвертируем в текст, иначе недвоичные (категориальные) переменные выйдут, как фактор, и их нельзя будет объединить
) %>%
## сжимаем список выходных данных подсчетов в один датафрейм
bind_rows() %>%
## объединяем с выходными данными регрессии
bind_cols(., models) %>%
## сохраняем только интересующие нас столбцы
select(term, 2:3, estimate, conf.low, conf.high, p.value) %>%
## округляем знаки после запятой
mutate(across(where(is.numeric), round, digits = 2))Ниже представлен датафрейм. См. страницу Таблицы для презентации, чтобы ознакомиться с идеями о том, как еще конвертировать эту таблицу в красивую таблицу в HTML (например, с помощью flextable).
Пакет gtsummary
Ниже мы представляем использование tbl_uvregression() из пакета gtsummary. Как и на странице Описательные таблицы, функции gtsummary хорошо справляются с расчетом статистики и подготовкой профессионально выглядящих результатов. Эта функция создает таблицу результатов однофакторной регрессии.
Мы выбираем только необходимые столбцы из linelist (независимые переменные и переменная исхода) и подставляем их в tbl_uvregression(). Мы выполним однофакторную регрессию для каждого из столбцов, который мы определили как explanatory_vars на этапе Подготовки данных (пол, жар, озноб, кашель, боли, рвота, и возрастные категории age_cat).
Внутри самой функции мы задаем method = как glm (без кавычек), y = столбец исхода (outcome), уточняем в method.args =, что мы хотим выполнить логистическую регрессию с помощью family = binomial, а также говорим, что нужно возвести результаты в степень
Выходным результатом будет HTML, который содержит подсчеты
univ_tab <- linelist %>%
dplyr::select(explanatory_vars, outcome) %>% ## выбираем интересующие переменные
tbl_uvregression( ## создаем однофакторную таблицу
method = glm, ## задаем, какую регрессию выполнить (обобщенная линейная модель)
y = outcome, ## задаем переменную исхода
method.args = list(family = binomial), ## dопределяем, какой тип glm надо выполнить (логистическая)
exponentiate = TRUE ## возводим в степень, чтобы получить отношения шансов (а не логарифм отношения шансов)
)
## просматриваем таблицу однофакторных результатов
univ_tab| Characteristic | N | OR1 | 95% CI1 | p-value |
|---|---|---|---|---|
| gender | 4,167 | 1.00 | 0.88, 1.13 | >0.9 |
| fever | 4,167 | 1.00 | 0.85, 1.17 | >0.9 |
| chills | 4,167 | 1.03 | 0.89, 1.21 | 0.7 |
| cough | 4,167 | 1.15 | 0.97, 1.37 | 0.11 |
| aches | 4,167 | 0.93 | 0.76, 1.14 | 0.5 |
| vomit | 4,167 | 1.09 | 0.96, 1.23 | 0.2 |
| age_cat | 4,167 | |||
| 0-4 | — | — | ||
| 5-9 | 0.94 | 0.77, 1.15 | 0.5 | |
| 10-14 | 1.15 | 0.93, 1.42 | 0.2 | |
| 15-19 | 0.99 | 0.80, 1.24 | >0.9 | |
| 20-29 | 1.03 | 0.84, 1.26 | 0.8 | |
| 30-49 | 1.07 | 0.85, 1.33 | 0.6 | |
| 50-69 | 0.68 | 0.41, 1.13 | 0.13 | |
| 70+ | 0.53 | 0.07, 3.20 | 0.5 | |
| 1 OR = Odds Ratio, CI = Confidence Interval | ||||
С этими выходными данными вы можете сделать множество модификаций, например, адаптировать текстовые подписи, выделить строки жирным в зависимости от их p-значений и т.п. См. самоучители тут и в других онлайн источниках.
19.3 Стратифицированный
Стратифицированный анализ в настоящее время разрабатывается для gtsummary, эта страница будет обновляться.
19.4 Многофакторный
Для многофакторного анализа мы также представим два подхода:
glm()иtidy()
- пакет gtsummary
Поток работ будет похож для них, отличаться будет только последний шаг свода в итоговую таблицу.
Проведение многофакторного анализа
Здесь мы используем glm(), но добавляем больше переменных для анализа в правой стороне уравнения, отделяя символом плюс (+).
Чтобы выполнить модель со всеми независимыми переменными, мы выполним:
mv_reg <- glm(outcome ~ gender + fever + chills + cough + aches + vomit + age_cat, family = "binomial", data = linelist)
summary(mv_reg)
Call:
glm(formula = outcome ~ gender + fever + chills + cough + aches +
vomit + age_cat, family = "binomial", data = linelist)
Coefficients:
Estimate Std. Error z value Pr(>|z|)
(Intercept) 0.069054 0.131726 0.524 0.600
gender 0.002448 0.065133 0.038 0.970
fever 0.004309 0.080522 0.054 0.957
chills 0.034112 0.078924 0.432 0.666
cough 0.138584 0.089909 1.541 0.123
aches -0.070705 0.104078 -0.679 0.497
vomit 0.086098 0.062618 1.375 0.169
age_cat5-9 -0.063562 0.101851 -0.624 0.533
age_cat10-14 0.136372 0.107275 1.271 0.204
age_cat15-19 -0.011074 0.113640 -0.097 0.922
age_cat20-29 0.026552 0.102780 0.258 0.796
age_cat30-49 0.059569 0.116402 0.512 0.609
age_cat50-69 -0.388964 0.262384 -1.482 0.138
age_cat70+ -0.647443 0.917375 -0.706 0.480
(Dispersion parameter for binomial family taken to be 1)
Null deviance: 5712.4 on 4166 degrees of freedom
Residual deviance: 5700.2 on 4153 degrees of freedom
AIC: 5728.2
Number of Fisher Scoring iterations: 4Если вы хотите включить две переменные и взаимодействие между ними, вы можете отделить их звездочкой * вместо +. Отделяйте их двоеточием :, если вы только уточняете взаимодействие. Например:
glm(outcome ~ gender + age_cat * fever, family = "binomial", data = linelist)Опционально вы можете использовать этот код, чтобы воспользоваться заранее определенным вектором имен столбцов и воссоздать использованную выше команду с помощью str_c(). Это может быть полезным, если у вас меняются имена независимых переменных или если вы не хотите их все снова печатать.
## выполняем регрессию со всеми интересующими переменными
mv_reg <- explanatory_vars %>% ## начинаем с вектора имен столбцов независимых переменных
str_c(collapse = "+") %>% ## объединяем все имена интересующих переменных, которые отделяются плюсом
str_c("outcome ~ ", .) %>% ## объединяем имена интересующих переменных с исходом в стиле формулы
glm(family = "binomial", ## задаем тип glm как логистическую регрессию,
data = linelist) ## определяем набор данныхПостроение модели
Вы можете построить свою модель шаг за шагом, сохраняя разные модели, которые включают определенные независимые переменные. Вы можете сравнить эти модели с помощью тестов на отношение правдоподобия, используя lrtest() из пакета lmtest, как показано ниже:
ПРИМЕЧАНИЕ: Использование базовой функции anova(model1, model2, test = "Chisq) даст те же результаты
model1 <- glm(outcome ~ age_cat, family = "binomial", data = linelist)
model2 <- glm(outcome ~ age_cat + gender, family = "binomial", data = linelist)
lmtest::lrtest(model1, model2)Likelihood ratio test
Model 1: outcome ~ age_cat
Model 2: outcome ~ age_cat + gender
#Df LogLik Df Chisq Pr(>Chisq)
1 8 -2852.6
2 9 -2852.6 1 2e-04 0.9883Еще один вариант - взять объект модели и применить функцию step() из пакета stats. При построении этой модели уточните, какое направление выбора переменной вы хотите использовать.
## выберите модель, используя выбор вперед на основе критерия AIC
## вы также можете сделать выбор "назад" ("backward") или в обе стороны ("both"), исправив направление
final_mv_reg <- mv_reg %>%
step(direction = "forward", trace = FALSE)Вы также можете отключить экспоненциальный формат записи в вашей сессии R для ясности:
options(scipen=999)Как описывается в разделе по однофакторнуму анализу, передайте выходные данные модели в tidy(), чтобы возвести в степень логарифмы отношения шансов и ДИ. Наконец, мы округляем все числовые столбцы до двух знаков после запятой. Просмотрите все строки.
mv_tab_base <- final_mv_reg %>%
broom::tidy(exponentiate = TRUE, conf.int = TRUE) %>% ## получаем аккуратный датафрейм с оценками
mutate(across(where(is.numeric), round, digits = 2)) ## округляем Вот как выглядит получившийся датафрейм:
Объединение однофакторного и многофакторного анализа
Объединяем с помощью gtsummary
В пакете gtsummary есть функция tbl_regression(), которая берет выходные результаты регрессии (в данном случае glm()) и создает хорошую суммарную таблицу.
## показываем таблицу результатов итоговой регрессии
mv_tab <- tbl_regression(final_mv_reg, exponentiate = TRUE)Давайте посмотрим таблицу:
mv_tab| Characteristic | OR1 | 95% CI1 | p-value |
|---|---|---|---|
| gender | 1.00 | 0.88, 1.14 | >0.9 |
| fever | 1.00 | 0.86, 1.18 | >0.9 |
| chills | 1.03 | 0.89, 1.21 | 0.7 |
| cough | 1.15 | 0.96, 1.37 | 0.12 |
| aches | 0.93 | 0.76, 1.14 | 0.5 |
| vomit | 1.09 | 0.96, 1.23 | 0.2 |
| age_cat | |||
| 0-4 | — | — | |
| 5-9 | 0.94 | 0.77, 1.15 | 0.5 |
| 10-14 | 1.15 | 0.93, 1.41 | 0.2 |
| 15-19 | 0.99 | 0.79, 1.24 | >0.9 |
| 20-29 | 1.03 | 0.84, 1.26 | 0.8 |
| 30-49 | 1.06 | 0.85, 1.33 | 0.6 |
| 50-69 | 0.68 | 0.40, 1.13 | 0.14 |
| 70+ | 0.52 | 0.07, 3.19 | 0.5 |
| 1 OR = Odds Ratio, CI = Confidence Interval | |||
Вы также можете объединять несколько разных таблиц выходных результатов, созданных gtsummary с помощью функции tbl_merge(). Сейчас мы объединим результаты многофакторного анализа с помощью gtsummary с результатам однофакторного анализа, который мы создали выше:
## объединяем с результатами однофакторного анализа
tbl_merge(
tbls = list(univ_tab, mv_tab), # объединяем
tab_spanner = c("**Univariate**", "**Multivariable**")) # задаем имена заголовков| Characteristic | Univariate | Multivariable | |||||
|---|---|---|---|---|---|---|---|
| N | OR1 | 95% CI1 | p-value | OR1 | 95% CI1 | p-value | |
| gender | 4,167 | 1.00 | 0.88, 1.13 | >0.9 | 1.00 | 0.88, 1.14 | >0.9 |
| fever | 4,167 | 1.00 | 0.85, 1.17 | >0.9 | 1.00 | 0.86, 1.18 | >0.9 |
| chills | 4,167 | 1.03 | 0.89, 1.21 | 0.7 | 1.03 | 0.89, 1.21 | 0.7 |
| cough | 4,167 | 1.15 | 0.97, 1.37 | 0.11 | 1.15 | 0.96, 1.37 | 0.12 |
| aches | 4,167 | 0.93 | 0.76, 1.14 | 0.5 | 0.93 | 0.76, 1.14 | 0.5 |
| vomit | 4,167 | 1.09 | 0.96, 1.23 | 0.2 | 1.09 | 0.96, 1.23 | 0.2 |
| age_cat | 4,167 | ||||||
| 0-4 | — | — | — | — | |||
| 5-9 | 0.94 | 0.77, 1.15 | 0.5 | 0.94 | 0.77, 1.15 | 0.5 | |
| 10-14 | 1.15 | 0.93, 1.42 | 0.2 | 1.15 | 0.93, 1.41 | 0.2 | |
| 15-19 | 0.99 | 0.80, 1.24 | >0.9 | 0.99 | 0.79, 1.24 | >0.9 | |
| 20-29 | 1.03 | 0.84, 1.26 | 0.8 | 1.03 | 0.84, 1.26 | 0.8 | |
| 30-49 | 1.07 | 0.85, 1.33 | 0.6 | 1.06 | 0.85, 1.33 | 0.6 | |
| 50-69 | 0.68 | 0.41, 1.13 | 0.13 | 0.68 | 0.40, 1.13 | 0.14 | |
| 70+ | 0.53 | 0.07, 3.20 | 0.5 | 0.52 | 0.07, 3.19 | 0.5 | |
| 1 OR = Odds Ratio, CI = Confidence Interval | |||||||
Объединение с помощью dplyr
Альтернативным способом объединения результатов однофакторного и многофакторного анализа glm()/tidy() будет использование функций соединения из dplyr.
- Объединяем полученные ранее результаты однофакторного анализа (
univ_tab_base, где содержится подсчет) с аккуратными результатами многофакторного анализаmv_tab_base
- Используем
select(), чтобы сохранить только те столбцы, которые нам нужны, уточнить их порядок и переименовать их
- Используем
round(), чтобы задать два знака после запятой всем столбцам в классе Double (число двойной точности)
## объединяем однофакторную и многофакторную таблицу
left_join(univ_tab_base, mv_tab_base, by = "term") %>%
## выбираем столбцы и переименовываем
select( # новое имя = старое имя
"characteristic" = term,
"recovered" = "0",
"dead" = "1",
"univ_or" = estimate.x,
"univ_ci_low" = conf.low.x,
"univ_ci_high" = conf.high.x,
"univ_pval" = p.value.x,
"mv_or" = estimate.y,
"mvv_ci_low" = conf.low.y,
"mv_ci_high" = conf.high.y,
"mv_pval" = p.value.y
) %>%
mutate(across(where(is.double), round, 2)) # A tibble: 20 × 11
characteristic recovered dead univ_or univ_ci_low univ_ci_high univ_pval
<chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 (Intercept) 909 1168 1.28 1.18 1.4 0
2 gender 916 1174 1 0.88 1.13 0.97
3 (Intercept) 340 436 1.28 1.11 1.48 0
4 fever 1485 1906 1 0.85 1.17 0.99
5 (Intercept) 1472 1877 1.28 1.19 1.37 0
6 chills 353 465 1.03 0.89 1.21 0.68
7 (Intercept) 272 309 1.14 0.97 1.34 0.13
8 cough 1553 2033 1.15 0.97 1.37 0.11
9 (Intercept) 1636 2114 1.29 1.21 1.38 0
10 aches 189 228 0.93 0.76 1.14 0.51
11 (Intercept) 931 1144 1.23 1.13 1.34 0
12 vomit 894 1198 1.09 0.96 1.23 0.17
13 (Intercept) 338 427 1.26 1.1 1.46 0
14 age_cat5-9 365 433 0.94 0.77 1.15 0.54
15 age_cat10-14 273 396 1.15 0.93 1.42 0.2
16 age_cat15-19 238 299 0.99 0.8 1.24 0.96
17 age_cat20-29 345 448 1.03 0.84 1.26 0.79
18 age_cat30-49 228 307 1.07 0.85 1.33 0.58
19 age_cat50-69 35 30 0.68 0.41 1.13 0.13
20 age_cat70+ 3 2 0.53 0.07 3.2 0.49
# ℹ 4 more variables: mv_or <dbl>, mvv_ci_low <dbl>, mv_ci_high <dbl>,
# mv_pval <dbl>19.5 Форест-график
Этот раздел показывает, как создать график с результатами вашей регрессии. Есть две опции, вы можете построить график самостоятельно с помощью ggplot2 или использовать мета-пакет под названием easystats (пакет, который включает много пакетов).
См. страницу Основы ggplot, если вы не знакомы с пакетом построения графиков ggplot2.
Пакет ggplot2
Вы можете построить форест-график с помощью ggplot(), построив график с результатами многофакторной регрессии. Добавляйте слои на графики с помощью “геомов”:
- оценочные данные с помощью
geom_point()
- доверительные интервалы с помощью
geom_errorbar()
- вертикальная линия на ОШ = 1 с
geom_vline()
Прежде чем построить график, возможно нужно использовать fct_relevel() из пакета forcats, чтобы задать порядок переменных/уровни на оси y. ggplot() может отобразить их в буквенно-числовом порядке, который не очень хорошо работает для значений возрастных категорий (“30” будет раньше, чем “5”). См. дополнительную информацию на странице Факторы.
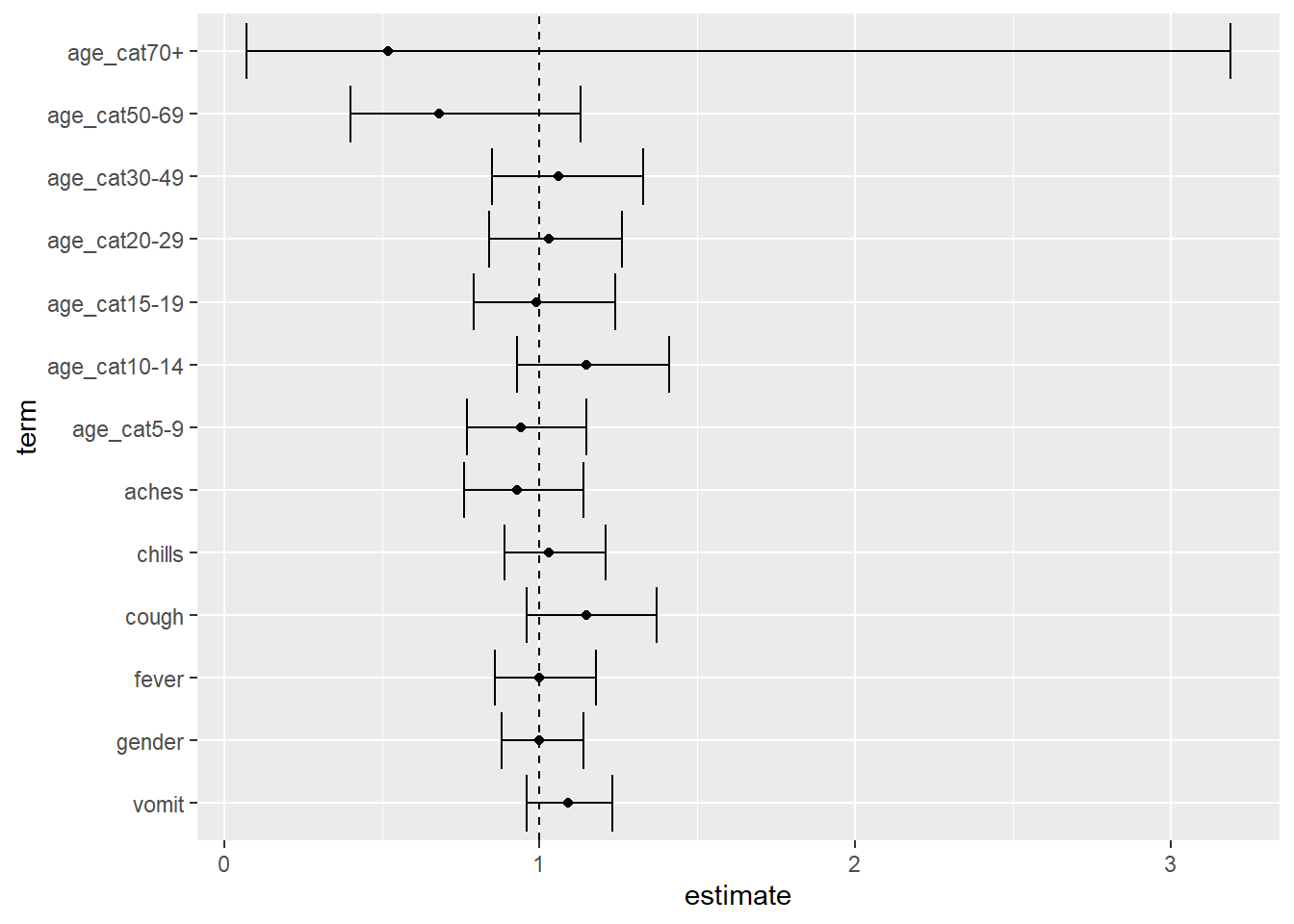
## удаляем свободный член из результатов многофакторного анализа
mv_tab_base %>%
#задаем порядок уровней по оси y
mutate(term = fct_relevel(
term,
"vomit", "gender", "fever", "cough", "chills", "aches",
"age_cat5-9", "age_cat10-14", "age_cat15-19", "age_cat20-29",
"age_cat30-49", "age_cat50-69", "age_cat70+")) %>%
# удаляем строку "intercept" из графика
filter(term != "(Intercept)") %>%
## строим график с переменной по оси y и оценочным показателем (ОШ) по оси x
ggplot(aes(x = estimate, y = term)) +
## показываем оценку как точку
geom_point() +
## добавляем столбец ошибки для доверительных интервалов
geom_errorbar(aes(xmin = conf.low, xmax = conf.high)) +
## показываем, где ОШ = 1 в виде пунктирной линии для справки
geom_vline(xintercept = 1, linetype = "dashed")Пакеты easystats
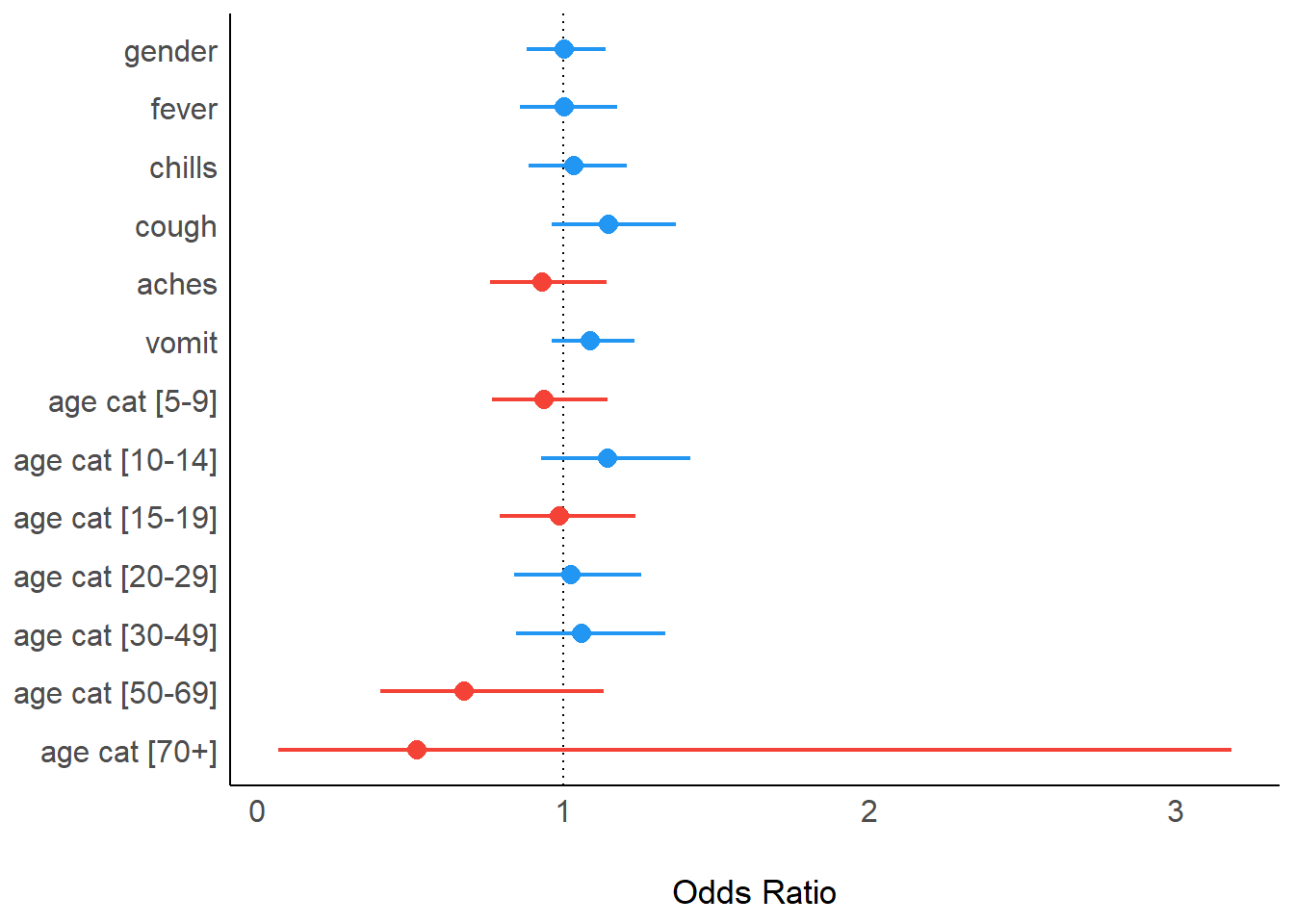
В качестве альтернативы, если вам не нужен такой детальный уровень контроля, как дает ggplot2, можно использовать комбинацию пакетов easystats.
Функция model_parameters() из parameters выполняет действия, эквивалентные функции tidy() из пакета broom. Пакет see затем принимает эти выходные данные и создает форест-график по умолчанию в виде объекта ggplot().
19.6 Ресурсы
Информация для содержания этой страницы была взята из следующих онлайн ресурсов и виньеток: