pacman::p_load(rio, # Importation du fichier
here, # Localisation de fichiers
tidyverse, # gestion des données + graphiques ggplot2
tsibble, # gère les ensembles de données de séries temporelles
slider, # pour calculer les moyennes mobiles
imputeTS, # pour remplir les valeurs manquantes
feasts, # pour la décomposition des séries temporelles et l'autocorrélation
forecast, # ajustement des termes sin et cosin aux données (note : doit être chargé après feasts)
trending, # ajustement et évaluation des modèles
tmaptools, # pour obtenir des géocoordonnées (lon/lat) à partir de noms de lieux
ecmwfr, # pour interagir avec l'API CDS de copernicus sateliate
stars, # pour lire les fichiers .nc (données climatiques)
units, # pour définir les unités de mesure (données climatiques)
yardstick, # pour l'examen de la précision du modèle
surveillance # pour la détection des aberrations
)23 Série temporelle et détection des épidémies
23.1 Aperçu
Cet onglet démontre l’utilisation de plusieurs paquets pour l’analyse des séries temporelles. Il s’appuie principalement sur les paquets de la famille tidyverts mais utilise également le paquet RECON trending pour ajuster des modèles qui sont plus appropriés à l’épidémiologie des maladies infectieuses.
Notez que dans l’exemple ci-dessous, nous utilisons un ensemble de données provenant du paquet surveillance. sur Campylobacter en Allemagne (voir le chapitre sur les données, du manuel pour plus de détails). Cependant, si vous vouliez exécuter le même code sur un ensemble de données avec plusieurs pays ou d’autres strates, il y a un exemple de code pour cela dans le fichier r4epis github repo.
Les sujets abordés sont les suivants :
- Données de séries temporelles
- Analyse descriptive
- Régressions ajustées
- Relation de deux séries temporelles
- Détection de l’épidémie
- Séries chronologiques interrompues
23.2 Préparation
Paquets
Ce morceau de code montre le chargement des paquets nécessaires aux analyses. Dans ce manuel, nous mettons l’accent sur p_load() de pacman, qui installe le paquet si nécessaire et le charge pour l’utiliser. Vous pouvez également charger des paquets avec library() depuis base R. Voir la page sur bases de R pour plus d’informations sur les paquets R.
Chargement des données
Vous pouvez télécharger toutes les données utilisées dans ce manuel en suivant les instructions de la page Télécharger le manuel et les données.
L’ensemble de données d’exemple utilisé dans cette section est le décompte hebdomadaire des cas de campylobacter signalés en Allemagne entre 2001 et 2011. Vous pouvez cliquer ici pour télécharger ce fichier de données (.xlsx)..
Cet ensemble de données est une version réduite de l’ensemble de données disponible dans le paquet surveillance (pour plus de détails, chargez le paquet surveillance et voyez ?campyDE)
Importez ces données avec la fonction import() du paquet rio (elle gère de nombreux types de fichiers comme .xlsx, .csv, .rds - voir la page Importation et exportation pour plus de détails).
# Importez les comptes dans R
counts <- rio::import("campylobacter_germany.xlsx")Les 10 premières lignes des comptages sont affichées ci-dessous.
Nettoyer les données
Le code ci-dessous s’assure que la colonne de date est dans le format approprié. Pour cet onglet, nous utiliserons le package tsibble et donc la fonction yearweek sera utilisée pour créer une variable de semaine calendaire. Il existe plusieurs autres façons de le faire (voir la page Manipuler les dates pour plus de détails), mais pour les séries temporelles, il est préférable de rester dans un seul cadre (tsibble).
## s'assurer que la colonne date est dans le format approprié
counts$date <- as.Date(counts$date)
## créer une variable de semaine calendaire
## adapter les définitions ISO des semaines commençant un lundi
counts <- counts %>%
mutate(epiweek = yearweek(date, week_start = 1))Télécharger les données climatiques
Dans la partie relation de deux séries temporelles de cette page, nous allons comparer le nombre de cas de campylobacter aux données climatiques.
Les données climatiques de n’importe quel endroit du monde peuvent être téléchargées à partir du satellite Copernicus de l’UE. Il ne s’agit pas de mesures exactes, mais de données basées sur un modèle (similaire à l’interpolation), mais l’avantage est une couverture horaire globale ainsi que des prévisions.
Vous pouvez télécharger chacun de ces fichiers de données climatiques à partir de la page Télécharger le manuel et les données.
Pour les besoins de la démonstration, nous allons présenter le code R permettant d’utiliser le paquet ecmwfr pour extraire ces données du magasin de données climatiques Copernicus Climate Data Store. Vous devrez créer un compte gratuit pour que cela fonctionne. Le site Web du paquet contient un demo utile sur la manière de procéder. Vous trouverez ci-dessous un exemple de code expliquant comment procéder, une fois que vous les clés API appropriées. Vous devez remplacer les X ci-dessous par les identifiants de votre compte. Vous devrez télécharger une année de données à la fois, sinon le serveur s’arrête.
Si vous n’êtes pas sûr des coordonnées d’un lieu pour lequel vous voulez télécharger des données, vous pouvez utiliser le paquet tmaptools pour extraire les coordonnées des cartes routières ouvertes. Une autre option est le paquet photon mais il n’a pas encore été publié sur CRAN. photon fournit plus de données contextuelles lorsqu’il y a plusieurs correspondances pour votre recherche.
## récupérer les coordonnées de l'emplacement
coords <- geocode_OSM("Germany", geometry = "point")
## rassembler les long/lats dans un format pour les requêtes ERA-5 (bounding box)
## (comme on ne veut qu'un seul point, on peut répéter les coordonnées)
request_coords <- str_glue_data(coords$coords, "{y}/{x}/{y}/{x}")
## Extraction des données modélisées à partir du satellite copernicus (réanalyse ERA-5)
## https://cds.climate.copernicus.eu/cdsapp#!/software/app-era5-explorer?tab=app
## https://github.com/bluegreen-labs/ecmwfr
## Configurer la clé pour les données météo
wf_set_key(user = "XXXXX",
key = "XXXXXXXXX-XXXX-XXXX-XXXX-XXXXXX-XXXXXXXXX",
service = "cds")
## Exécution pour chaque année d'intérêt (sinon le serveur s'arrête)
for (i in 2002:2011) {
## construire une requête
## voir ici pour savoir comment faire : https://bluegreen-labs.github.io/ecmwfr/articles/cds_vignette.html#the-request-syntax
## changer la requête en une liste en utilisant le bouton addin ci-dessus (python to list)
## La cible est le nom du fichier de sortie ! !!
request <- request <- list(
product_type = "reanalysis",
format = "netcdf",
variable = c("2m_temperature", "total_precipitation"),
year = c(i),
month = c("01", "02", "03", "04", "05", "06", "07", "08", "09", "10", "11", "12"),
day = c("01", "02", "03", "04", "05", "06", "07", "08", "09", "10", "11", "12",
"13", "14", "15", "16", "17", "18", "19", "20", "21", "22", "23", "24",
"25", "26", "27", "28", "29", "30", "31"),
time = c("00:00", "01:00", "02:00", "03:00", "04:00", "05:00", "06:00", "07:00",
"08:00", "09:00", "10:00", "11:00", "12:00", "13:00", "14:00", "15:00",
"16:00", "17:00", "18:00", "19:00", "20:00", "21:00", "22:00", "23:00"),
area = request_coords,
dataset_short_name = "reanalysis-era5-single-levels",
target = paste0("germany_weather", i, ".nc")
)
### Télécharger le fichier et le stocker dans le répertoire de travail actuel.
file <- wf_request(user = "XXXXX", # ID utilisateur (pour l'authentification)
request = request, # la requête
transfer = TRUE, # télécharger le fichier
path = here::here("data", "Weather")) ## chemin pour sauvegarder les données
}Charger les données climatiques
Que vous ayez téléchargé les données climatiques via notre manuel ou que vous ayez utilisé le code ci-dessus, vous devriez maintenant avoir 10 ans de fichiers de données climatiques “.nc” stockés dans le même dossier sur votre ordinateur.
Utilisez le code ci-dessous pour importer ces fichiers dans R avec le paquet stars.
## définir le chemin vers le dossier météo
file_paths <- list.files(
here::here("data", "time_series", "weather"), # remplacer par votre propre chemin de fichier
full.names = TRUE)
## ne garder que ceux qui ont le nom courant d'intérêt
file_paths <- file_paths[str_detect(file_paths, "germany")]
## lire tous les fichiers en tant qu'objet stars
data <- stars::read_stars(file_paths)t2m, tp,
t2m, tp,
t2m, tp,
t2m, tp,
t2m, tp,
t2m, tp,
t2m, tp,
t2m, tp,
t2m, tp,
t2m, tp, Une fois que ces fichiers ont été importés en tant qu’objet data, nous allons les convertir en un cadre de données.
## conversion en cadre de données
temp_data <- as_tibble(data) %>%
## ajouter des variables et corriger les unités
mutate(
## créer une variable de semaine calendaire
epiweek = tsibble::yearweek(time),
## créer une variable de date (début de la semaine calendaire)
date = as.Date(epiweek),
## changer la température de kelvin en celsius
t2m = set_units(t2m, celsius),
## changer les précipitations de mètres en millimètres
tp = set_units(tp, mm)) %>%
## regrouper par semaine (en gardant la date aussi)
group_by(epiweek, date) %>%
## obtenir la moyenne par semaine
summarise(t2m = as.numeric(mean(t2m)),
tp = as.numeric(mean(tp)))23.3 Données de séries temporelles
Il existe un certain nombre de paquets différents pour structurer et traiter les données de données de séries temporelles. Comme nous l’avons dit, nous nous concentrerons sur la famille de paquets tidyverts et nous utiliserons donc le paquet tsibble pour définir notre objet série temporelle. Avoir un ensemble de données défini comme un objet de série temporelle, il est beaucoup plus facile de structurer notre analyse.
Pour ce faire, nous utilisons la fonction tsibble() et spécifions l’“index”, c’est-à-dire la variable spécifiant l’unité de temps qui nous intéresse. Dans notre cas, il s’agit de la variable epiweek.
Si nous avions un ensemble de données avec des comptages hebdomadaires par province, par exemple, nous pourrions également spécifier la variable de regroupement en utilisant l’argument key =. Cela nous permettrait d’effectuer une analyse pour chaque groupe.
## Définir un objet de série temporelle
counts <- tsibble(counts, index = epiweek)En regardant class(counts), on constate qu’en plus d’être un cadre de données ordonné (“tbl_df”, “tbl”, “data.frame”), il possède les propriétés supplémentaires d’un cadre de données de série temporelle (“tbl_ts”).
Vous pouvez jeter un coup d’oil rapide à vos données en utilisant ggplot2. Nous voyons sur le graphique que qu’il existe un modèle saisonnier clair, et qu’il n’y a pas de manques. Cependant, il semble y avoir un problème avec la déclaration au début de chaque année, dans la dernière semaine de l’année, puis augmentent pour la première semaine de l’année suivante.
## tracer un graphique linéaire des cas par semaine
ggplot(counts, aes(x = epiweek, y = case)) +
geom_line()ATTENTION: La plupart des ensembles de données ne sont pas aussi propres que cet exemple. Vous devrez vérifier les doublons et les valuers qui manques comme ci-dessous.
Duplicates
tsibble n’autorise pas les observations en double. Ainsi, chaque ligne devra être unique, ou unique au sein du groupe (variable key). Le paquet a quelques fonctions qui aident à identifier les doublons. Celles-ci incluent are_duplicated() qui vous donne un vecteur VRAI/FAUX indiquant si la ligne est un dupliqué, et duplicates() qui vous donne un cadre de données des lignes dupliquées.
Voir la page sur De-duplication pour plus de détails sur la façon de sélectionner les lignes que vous voulez.
## obtient un vecteur de VRAI/FAUX si les lignes sont des doublons
are_duplicated(counts, index = epiweek)
## Obtenez un cadre de données pour les lignes dupliquées.
duplicates(counts, index = epiweek) Manques
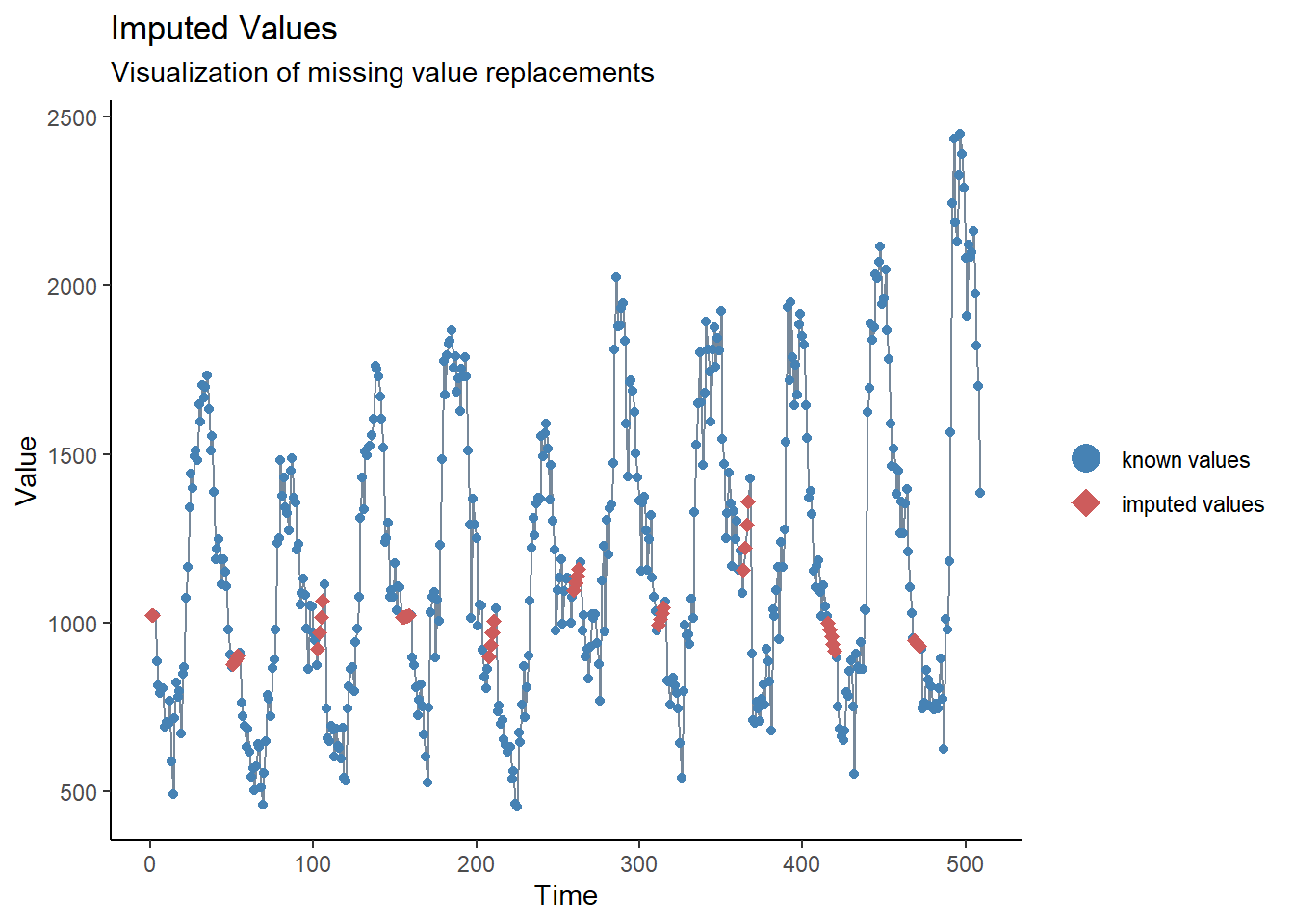
Nous avons vu lors de notre brève inspection ci-dessus qu’il n’y a pas de manques, mais nous avons aussi vu qu’il semble y avoir un problème de retard de déclaration autour du nouvel an. Une façon de résoudre ce problème pourrait être de définir ces valeurs comme manquantes, puis d’imputer les valeurs. La forme la plus simple d’imputation de séries chronologiques consiste à tracer une ligne droite entre les dernières valeurs non manquantes et les valeurs manquantes. Pour ce faire, nous utiliserons la fonction na_interpolation() du package imputeTS.
Consultez la page Données manquantes pour connaître les autres options d’imputation.
Une autre solution consisterait à calculer une moyenne mobile, pour essayer de pour tenter d’aplanir ces problèmes de déclaration apparents (voir la section suivante et la page sur les Moyennes mobiles).
## créez une variable avec les manques au lieu des semaines avec des problèmes de déclaration.
counts <- counts %>%
mutate(case_miss = if_else(
## si epiweek contient 52, 53, 1 ou 2
str_detect(epiweek, "W51|W52|W53|W01|W02"),
## alors définie comme manquante
NA_real_,
## sinon, conservez la valeur dans le cas
case
))
## alternativement interpoler les manquants par une tendance linéaire
## entre deux points adjacents les plus proches
counts <- counts %>%
mutate(case_int = imputeTS::na_interpolation(case_miss)
)
## pour vérifier quelles valeurs ont été imputées par rapport à l'original.
ggplot_na_imputations(counts$case_miss, counts$case_int) +
## faire un graphique traditionnel (avec des axes noirs et un fond blanc)
theme_classic()23.4 Analyse descriptive
Moyennes mobiles
Si les données sont très bruyantes (les comptes sautent en haut et en bas), alors il peut être utile de calculer une moyenne mobile. Dans l’exemple ci-dessous, pour chaque semaine, nous calculons le nombre moyen de cas des quatre semaines précédentes. Cela permet de lisser les données, pour les rendre plus interprétables. Dans notre cas, cela n’apporte pas grand-chose. Nous nous en tiendrons aux données interpolées pour la suite de l’analyse. Voir la page Moyennes mobiles pour plus de détails.
## créer une variable de moyenne mobile (traite les manques)
counts <- counts %>%
## créer la variable ma_4w
## glisser sur chaque ligne de la variable case
mutate(ma_4wk = slider::slide_dbl(case,
## pour chaque ligne, calculez le nom
~ mean(.x, na.rm = TRUE),
## utiliser les quatre semaines précédentes
.before = 4))
### faire une visualisation rapide de la différence
ggplot(counts, aes(x = epiweek)) +
geom_line(aes(y = case)) +
geom_line(aes(y = ma_4wk), colour = "red")Périodicité
Nous définissons ci-dessous une fonction personnalisée pour créer un périodogramme. Voir la page ecrire les fonctions pour des informations sur la façon d’écrire des fonctions dans R.
Tout d’abord, la fonction est définie. Ses arguments incluent un jeu de données avec une colonne counts, start_week = qui est la première semaine du jeu de données, un nombre pour indiquer combien de périodes par an (par exemple 52, 12), et enfin le style de sortie (voir les détails dans le code ci-dessous).
## Arguments de la fonction
#####################
## x est un ensemble de données
## counts est une variable avec des données de comptage ou des taux dans x
## start_week est la première semaine de votre série de données
## period est le nombre d'unités dans une année.
## output indique si vous souhaitez renvoyer le périodogramme spectral ou les semaines de pointe.
## "périodogramme" ou "semaines".
# Définissez la fonction
periodogram <- function(x,
counts,
start_week = c(2002, 1),
period = 52,
output = "weeks") {
## s'assurer que ce n'est pas un tsibble, filtrer sur le projet et ne garder que les colonnes d'intérêt.
prepare_data <- dplyr::as_tibble(x)
# prepare_data <- prepare_data[prepare_data[[strata]] == j, ]
prepare_data <- dplyr::select(prepare_data, {{counts}})
## créer une série temporelle "zoo" intermédiaire pour pouvoir l'utiliser avec spec.pgram
zoo_cases <- zoo::zooreg(prepare_data,
start = start_week, frequency = period)
## obtenir un périodogramme spectral n'utilisant pas la transformée de fourier rapide.
periodo <- spec.pgram(zoo_cases, fast = FALSE, plot = FALSE)
## retourner les semaines de pointe
periodo_weeks <- 1 / periodo$freq[order(-periodo$spec)] * period
if (output == "weeks") {
periodo_weeks
} else {
periodo
}
}
## obtenir le périodogramme spectral pour extraire les semaines avec les fréquences les plus élevées
## (vérification de la saisonnalité)
periodo <- periodogram(counts,
case_int,
start_week = c(2002, 1),
output = "periodogram")
## Tirez le spectre et la fréquence dans un cadre de données pour le tracé.
periodo <- data.frame(periodo$freq, periodo$spec)
## Tracez un périodogramme montrant la périodicité la plus fréquente.
ggplot(data = periodo,
aes(x = 1/(periodo.freq/52), y = log(periodo.spec))) +
geom_line() +
labs(x = "Period (weeks)", y = "Log(density)")## obtenir un vecteur semaines dans l'ordre croissant
peak_weeks <- periodogram(counts,
case_int,
start_week = c(2002, 1),
output = "weeks")ATTENTION: Il est possible d’utiliser les semaines ci-dessus pour les ajouter aux termes sin et cosinus, cependant nous utiliserons une fonction pour générer ces termes (voir la section régression ci-dessous) .
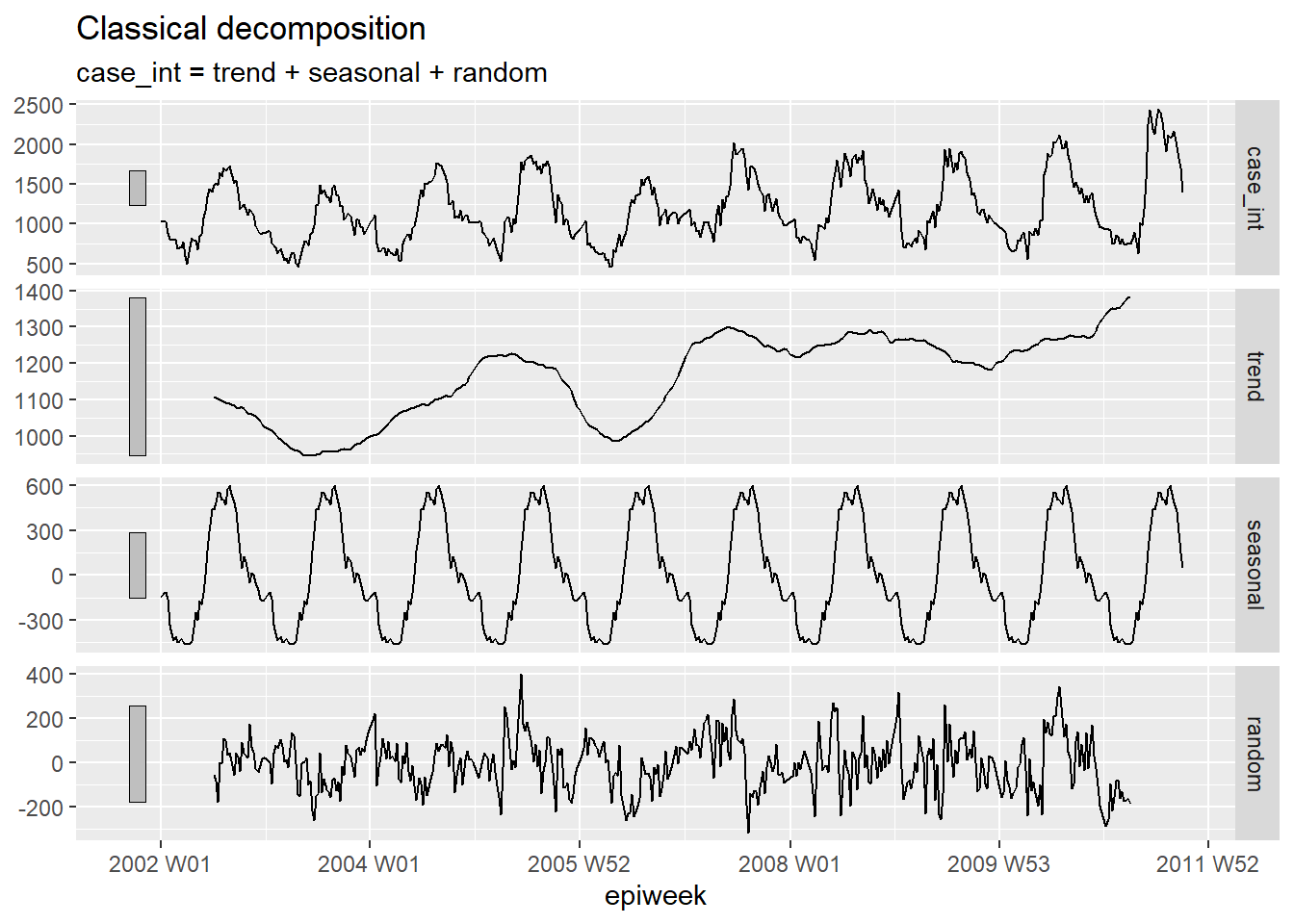
Décomposition
La décomposition classique est utilisée pour décomposer une série temporelle en plusieurs parties, qui lorsqu’elles sont prises ensemble constituent le modèle que vous voyez.
Ces différentes parties sont :
- La tendance-cycle (la direction à long terme des données)
- La saisonnalité (motifs répétitifs)
- L’aléatoire (ce qui reste après avoir retiré la tendance et la saison).
Autocorrélation
L’autocorrélation vous renseigne sur la relation entre les comptes de chaque semaine et les semaines qui la précèdent (appelées décalages).
En utilisant la fonction ACF(), nous pouvons produire un graphique qui nous montre un certain nombre de lignes pour la relation à différents décalages. Si le décalage est de 0 (x = 0), cette ligne sera toujours égale à 1, car elle montre la relation entre les deux. La première ligne illustrée ici (x = 1) montre la relation entre chaque observation et l’observation qui la précède (décalage de 1), la seconde montre la relation entre chaque observation et l’avant-dernière (décalage de 2) et ainsi de suite jusqu’à un décalage de 52 qui montre la relation entre chaque observation et l’observation d’un an (52 semaines avant).
L’utilisation de la fonction PACF() (pour l’autocorrélation partielle) montre le même type de relation, mais ajustée pour toutes les autres semaines intermédiaires. Ceci est moins informatif pour déterminer la périodicité.
## en utilisant l'ensemble de données counts
counts %>%
## calculer l'autocorrélation en utilisant une année complète de lags
ACF(case_int, lag_max = 52) %>%
## afficher un graphique
autoplot()## en utilisant l'ensemble de données counts
counts %>%
## calculer l'autocorrélation partielle en utilisant une année complète de décalages
PACF(case_int, lag_max = 52) %>%
## Afficher un graphique
autoplot()Vous pouvez tester formellement l’hypothèse nulle d’indépendance d’une série temporelle (c’est à dire qu’elle n’est pas autocorrélée) en utilisant le test de Ljung-Box (dans le paquet stats). Une valeur p significative suggère qu’il existe une autocorrélation dans les données.
## Test d'indépendance
Box.test(counts$case_int, type = "Ljung-Box")
Box-Ljung test
data: counts$case_int
X-squared = 462.65, df = 1, p-value < 2.2e-1623.5 Ajustement des régressions
Il est possible d’ajuster un grand nombre de régressions différentes à une série temporelle, Cependant, nous allons montrer ici comment ajuster une régression binomiale négative, c’est souvent la plus appropriée pour les données de comptage dans les maladies infectieuses.
Termes de Fourier
Les termes de Fourier sont l’équivalent des courbes sin et cosin. La différence est que ceux-ci sont ajustés en fonction de la recherche de la combinaison de courbes la plus appropriée pour expliquervos données.
Si vous n’ajustez qu’un seul terme de Fourier, cela équivaudrait à ajuster une courbe sin et un cosin pour le décalage le plus fréquent de votre périodogramme (dans notre cas 52 semaines). Nous utilisons la fonction fourier() du paquet forecast.
Dans le code ci-dessous, nous assignons en utilisant le $, car fourier() renvoie deux colonnes (une pour le sin et une pour le cosin) et celles-ci sont ajoutées à l’ensemble de données sous forme de liste, appelée “fourier”. Mais cette liste peut ensuite être utilisée comme une variable normale dans une régression.
## ajout des termes de fourier en utilisant les variables epiweek et case_int
counts$fourier <- select(counts, epiweek, case_int) %>%
fourier(K = 1)Binomiale négative
Il est possible d’ajuster des régressions en utilisant les fonctions stats ou MASS de base (par exemple, lm(), glm() et glm.nb()). Cependant, nous utiliserons celles du paquet trending, car cela permet de calculer les intervalles de confiance et de prédictionconfiance et les intervalles de prédiction appropriés (qui ne sont pas disponibles autrement). La syntaxe est la même, et vous spécifiez une variable de résultat puis un tilde (~) puis vous ajoutez vos diverses variables d’exposition d’intérêt séparées par un plus (+).
L’autre différence est que nous définissons d’abord le modèle et ensuite fit() aux données. Ceci est utile car cela permet de comparer plusieurs modèles différents avec la même syntaxe.
ATTENTION: Si vous vouliez utiliser des taux, plutôt que des comptes, vous pourriez inclure la variable population comme terme de décalage logarithmique, en ajoutant offset(log(population). Vous auriez alors besoin de définir la population à 1, avant d’utiliser predict() afin de produire un taux.
ATTENTION: Pour l’ajustement de modèles plus complexes tels que les modèles comme ARIMA ou prophète, voir le paquet fable.
## définissez le modèle que vous voulez ajuster (binomiale négative).
model <- glm_nb_model(
## définir le nombre de cas comme résultat d'intérêt
case_int ~
## utiliser epiweek pour tenir compte de la tendance
epiweek +
## utiliser les termes de fourier pour tenir compte de la saisonnalité
fourier)
## ajustez votre modèle en utilisant le jeu de données de comptage
fitted_model <- trending::fit(model, data.frame(counts))
### calculer les intervalles de confiance et les intervalles de prédiction
observed <- predict(fitted_model, simulate_pi = FALSE)
estimate_res <- data.frame(observed$result)
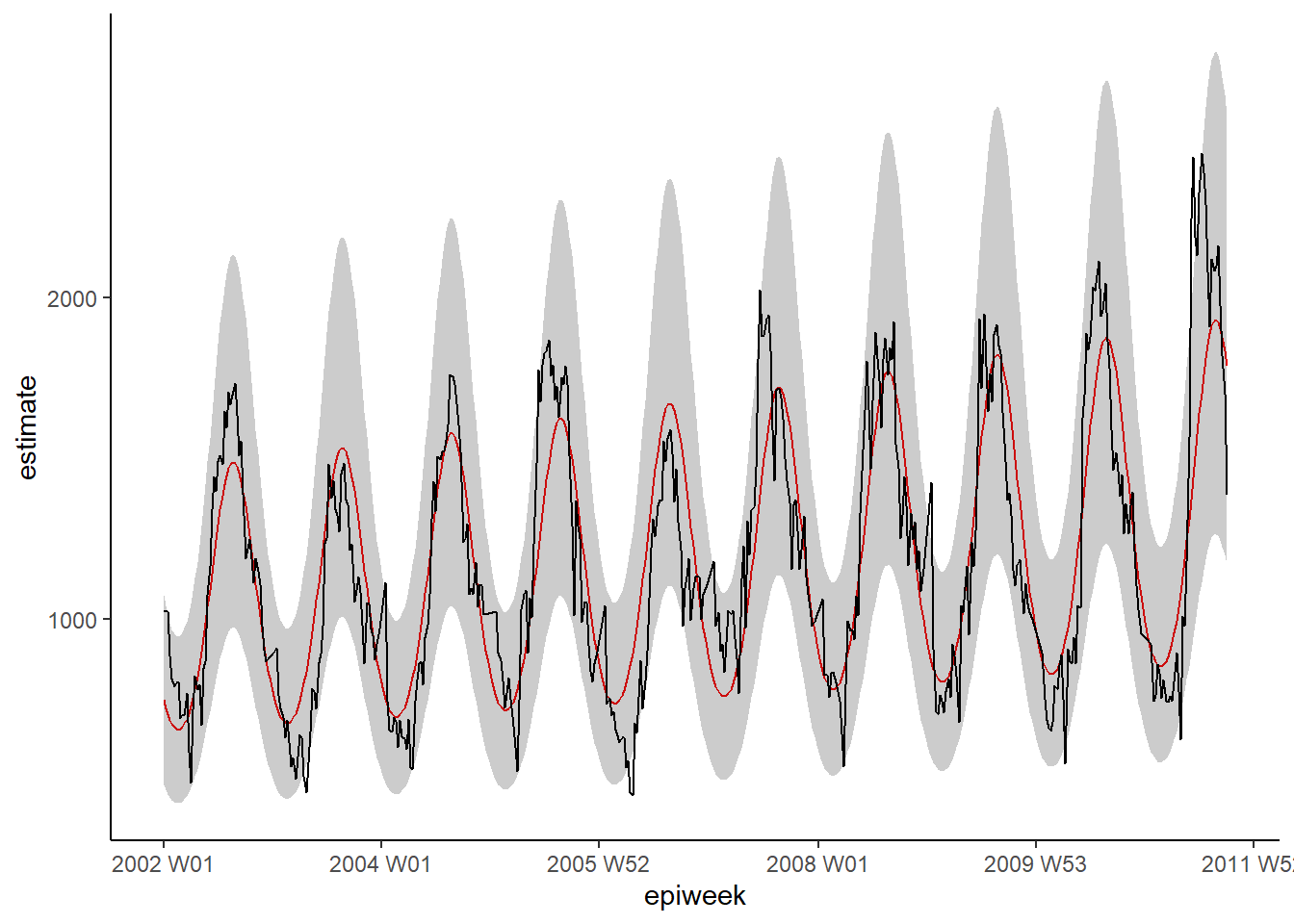
## Tracez votre régression
ggplot(data = estimate_res, aes(x = epiweek)) +
## ajouter une ligne pour l'estimation du modèle
geom_line(aes(y = estimate),
col = "red") +
## ajouter une bande pour les intervalles de prédiction
geom_ribbon(aes(ymin = lower_pi,
ymax = upper_pi),
alpha = 0.25) +
## ajouter une ligne pour le nombre de cas observés
geom_line(aes(y = case_int),
col = "black") +
## faire un graphique traditionnel (avec des axes noirs et un fond blanc)
theme_classic()Résidus
Pour voir si notre modèle s’adapte bien aux données observées, nous devons examiner les résidus. Les résidus sont la différence entre les comptes observés et les comptes estimés à partir du modèle. Nous pourrions calculer cela simplement en utilisant case_int - estimate, mais la fonction residuals() l’extrait directement de la régression pour nous.
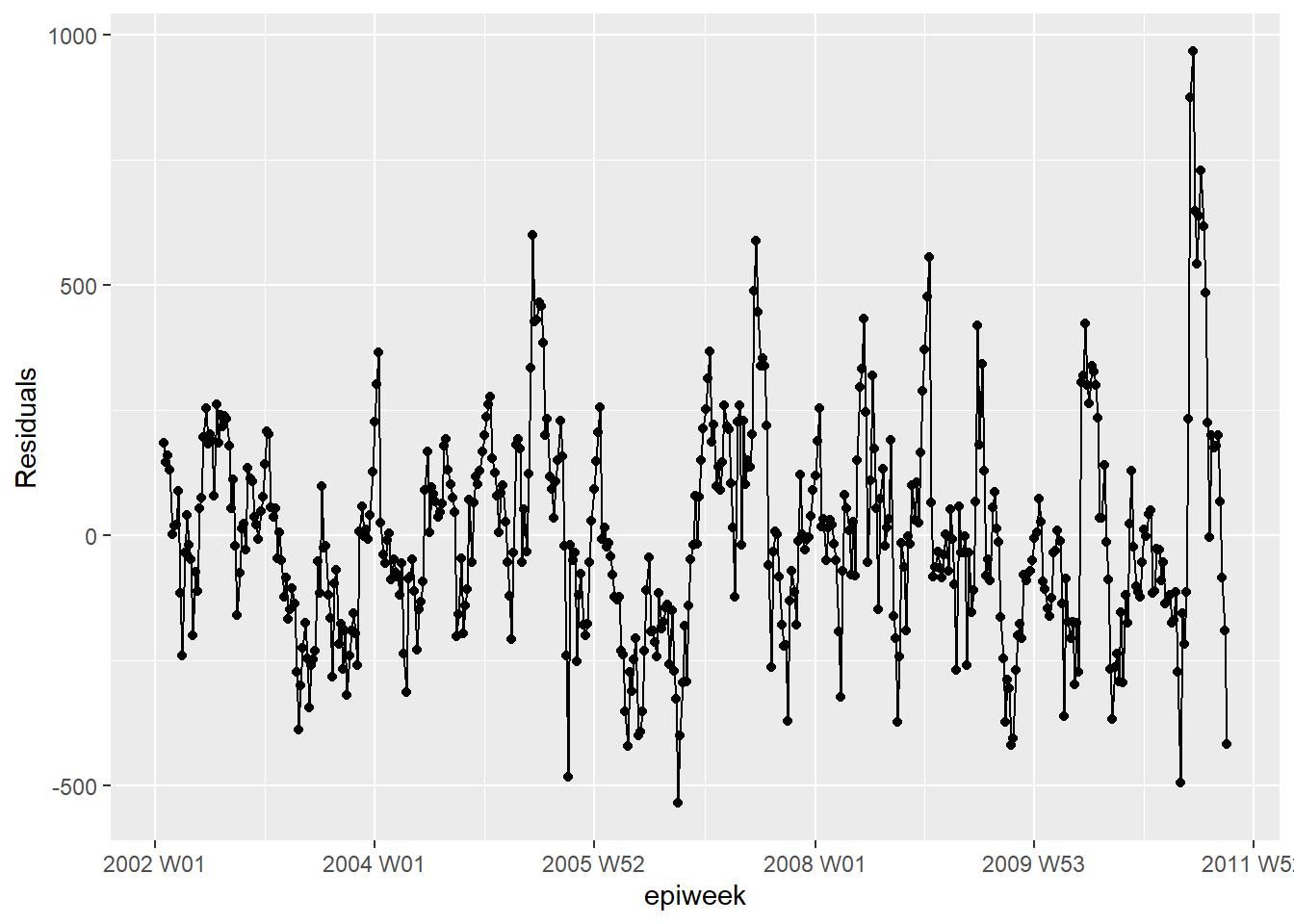
Ce que nous voyons ci-dessous, c’est que nous n’expliquons pas toutes les variations que nous pourrions expliquer avec le modèle. Il se peut que nous devions ajuster plus de termes de Fourier, et s’attaquer à l’amplitude. Cependant, pour cet exemple, nous allons laisser les choses telles quelles. Les graphiques montrent que notre modèle est moins bon dans les pics et les creux (lorsque les comptages sont les plus élevés et les plus bas) et qu’il est plus susceptible de sous-estimer les comptagees observés.
## calculate the residuals
estimate_res <- estimate_res %>%
mutate(resid = fitted_model$result[[1]]$residuals)
## are the residuals fairly constant over time (if not: outbreaks? change in practice?)
estimate_res %>%
ggplot(aes(x = epiweek, y = resid)) +
geom_line() +
geom_point() +
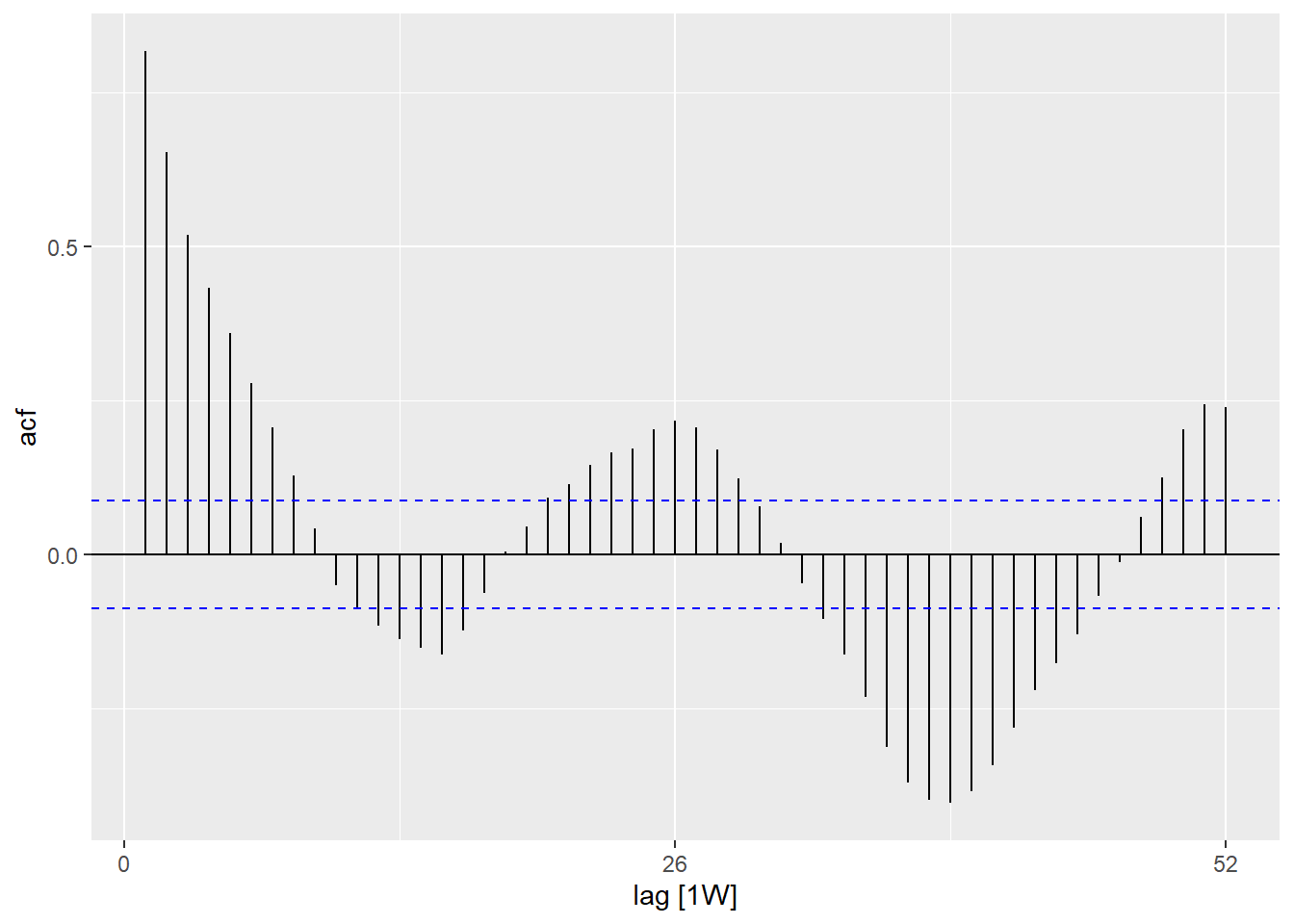
labs(x = "epiweek", y = "Residuals")## is there autocorelation in the residuals (is there a pattern to the error?)
estimate_res %>%
as_tsibble(index = epiweek) %>%
ACF(resid, lag_max = 52) %>%
autoplot()## are residuals normally distributed (are under or over estimating?)
estimate_res %>%
ggplot(aes(x = resid)) +
geom_histogram(binwidth = 0.01) +
geom_rug() +
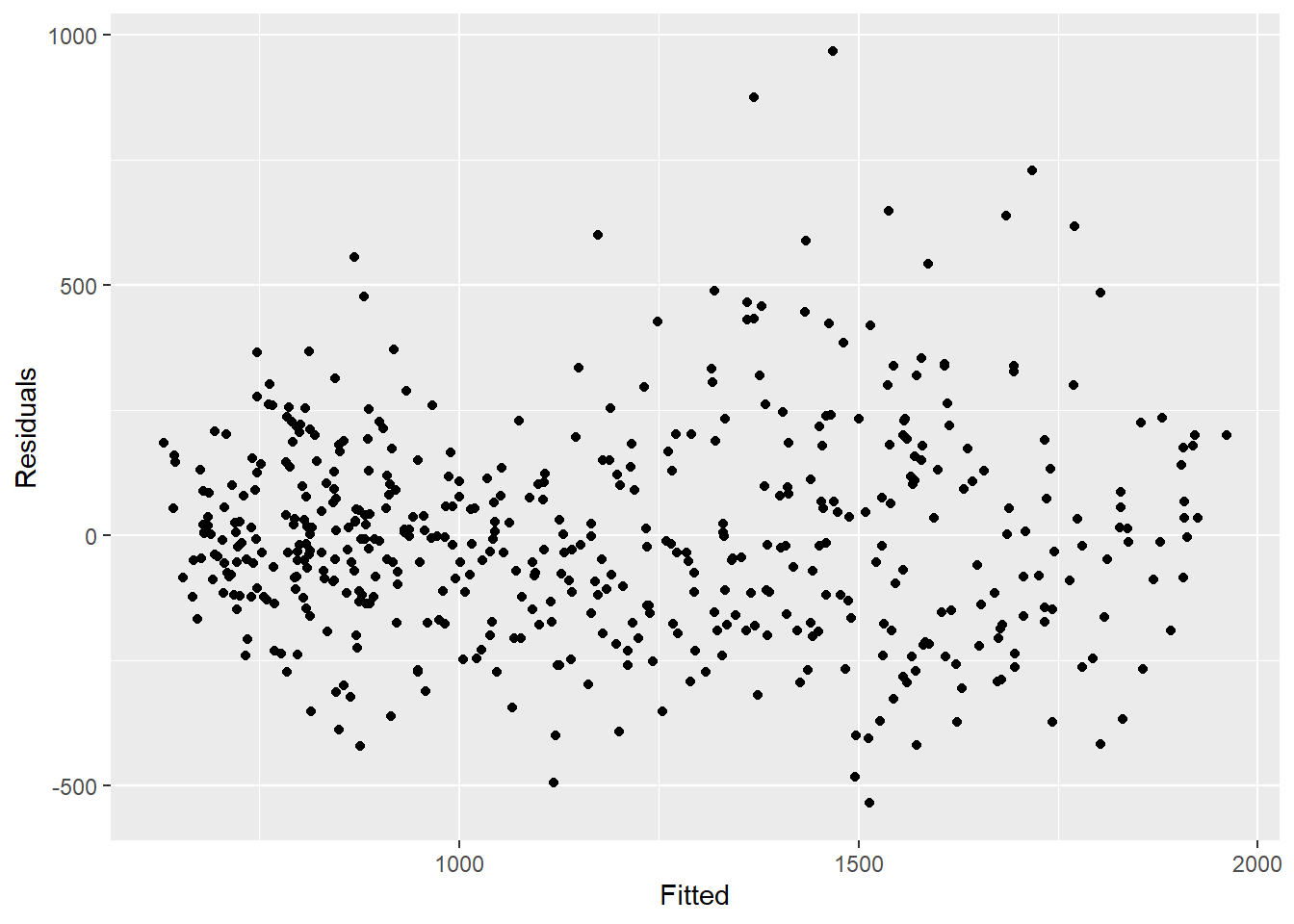
labs(y = "count") ## compare observed counts to their residuals
## should also be no pattern
estimate_res %>%
ggplot(aes(x = estimate, y = resid)) +
geom_point() +
labs(x = "Fitted", y = "Residuals")## formally test autocorrelation of the residuals
## H0 is that residuals are from a white-noise series (i.e. random)
## test for independence
## if p value significant then non-random
Box.test(estimate_res$resid, type = "Ljung-Box")
Box-Ljung test
data: estimate_res$resid
X-squared = 336.25, df = 1, p-value < 2.2e-1623.6 Relation entre deux séries temporelles
Nous examinons ici l’utilisation de données météorologiques (en particulier la température) pour expliquer le nombre de cas de campylobacter.
Fusionner des ensembles de données
Nous pouvons fusionner nos ensembles de données à l’aide de la variable week. Pour plus d’informations sur la fusion, voir la section du manuel sur les joindres.
## left join so that we only have the rows already existing in counts
## drop the date variable from temp_data (otherwise is duplicated)
counts <- left_join(counts,
select(temp_data, -date),
by = "epiweek")counts %>%
## garder les variables qui nous intéressent
select(epiweek, case_int, t2m) %>%
## Changez vos données en format long
pivot_longer(
## utiliser epiweek comme clé
!epiweek,
## déplacez les noms des colonnes vers la nouvelle colonne "measure".
names_to = "measure",
## déplacez les valeurs des cellules vers la nouvelle colonne "values".
values_to = "value") %>%
## créer un graphique avec l'ensemble de données ci-dessus
## Tracez l'epiweek sur l'axe des x et les valeurs (nombres/celsius) sur l'axe des y.
ggplot(aes(x = epiweek, y = value)) +
## créez un graphique séparé pour les comptages tempérés et les comptages de cas.
## laissez-les définir leurs propres axes y
facet_grid(measure ~ ., scales = "free_y") +
## Tracez les deux comme une ligne
geom_line()Lags et corrélation croisée
Pour tester formellement quelles semaines sont les plus fortement liées entre les cas et la température. Nous pouvons utiliser la fonction de corrélation croisée (CCF()) du paquet feasts. Vous pouvez également visualiser (plutôt que d’utiliser arrange) en utilisant la fonction autoplot().
counts %>%
## calculer la corrélation croisée entre les comptages interpolés et la température
CCF(case_int, t2m,
## fixer le délai maximum à 52 semaines
lag_max = 52,
## retourne le coefficient de corrélation
type = "correlation") %>%
## arange dans l'ordre décroissant du coefficient de corrélation
## montre les décalages les plus associés
arrange(-ccf) %>%
## montrer seulement les dix premiers
slice_head(n = 10)# A tsibble: 10 x 2 [1W]
lag ccf
<cf_lag> <dbl>
1 -4W 0.749
2 -5W 0.745
3 -3W 0.735
4 -6W 0.729
5 -2W 0.727
6 -7W 0.704
7 -1W 0.695
8 -8W 0.671
9 0W 0.649
10 47W 0.638Nous voyons qu’un décalage de 4 semaines est le plus fortement corrélé, donc nous créons une variable de température décalée à inclure dans notre régression.
ATTENTION: Notez que les quatre premières semaines de nos données dans la variable de température décalée sont manquantes (NA) - car il n’y a pas quatre semaines précédentes pour obtenir des données. Afin d’utiliser cet ensemble de données avec la fonction predict() du paquet trending, nous devons utiliser l’argument simulate_pi = FALSE dans la fonction predict() plus bas. Si nous voulions utiliser l’option simulate, alors nous devons supprimer ces manques et les stocker comme un nouvel ensemble de données en ajoutant drop_na(t2m_lag4). au morceau de code ci-dessous.
counts <- counts %>%
## créer une nouvelle variable pour la température décalée de quatre semaines.
mutate(t2m_lag4 = lag(t2m, n = 4))Binomiale négative avec deux variables
Nous ajustons une régression binomiale négative comme nous l’avons fait précédemment. Cette fois, nous ajoutons la variable de température retardée de quatre semaines.
ATTENTION: Notez l’utilisation de simulate_pi = FALSE dans l’argument predict(). Ceci est dû au fait que le comportement par défaut de trending est d’utiliser le paquet ciTools pour estimer un intervalle de prédiction. Cela ne fonctionne pas s’il y a des comptes NA, et produit également des intervalles plus granulaires. Voir ?trending::predict.trending_model_fit pour plus de détails.
## définissez le modèle que vous voulez ajuster (binomial négatif).
model <- glm_nb_model(
## définir le nombre de cas comme résultat d'intérêt
case_int ~
## utiliser epiweek pour tenir compte de la tendance
epiweek +
## utiliser les termes de fourier pour tenir compte de la saisonnalité
fourier +
## utiliser la température retardée de quatre semaines
t2m_lag4
)
## ajustez votre modèle en utilisant l'ensemble de données de comptage
fitted_model <- trending::fit(model, data.frame(counts))
### calculer les intervalles de confiance et les intervalles de prédiction
observed <- predict(fitted_model, simulate_pi = FALSE)Pour étudier les termes individuels, nous pouvons extraire la régression binomiale négative originale du paquet trending en utilisant get_fitted_model() et la passer au fonction tidy() du paquetage broom pour récupérer les estimations exponentielles et lesintervalles de confiance associés.
Ce que cela nous montre est que la température décalée, après avoir contrôlé la tendance et la saisonnalité, est similaire au nombre de cas (estimation ~ 1) et significativement associée. Cela suggère qu’il pourrait s’agir d’une bonne variable à utiliser pour prédire le nombre de cas futurs (les prévisions climatiques étant facilement accessibles).
fitted_model %>%
## extraire la régression binomiale négative originale
get_fitted_model() #%>% [[1]]
Call: glm.nb(formula = case_int ~ epiweek + fourier + t2m_lag4, data = data.frame(counts),
init.theta = 32.80689607, link = log)
Coefficients:
(Intercept) epiweek fourierS1-52 fourierC1-52 t2m_lag4
5.825e+00 8.464e-05 -2.850e-01 -1.954e-01 6.672e-03
Degrees of Freedom: 504 Total (i.e. Null); 500 Residual
(4 observations deleted due to missingness)
Null Deviance: 2015
Residual Deviance: 508.2 AIC: 6784 ## obtenir un cadre de données ordonné des résultats
#broom::tidy(exponentiate = TRUE,
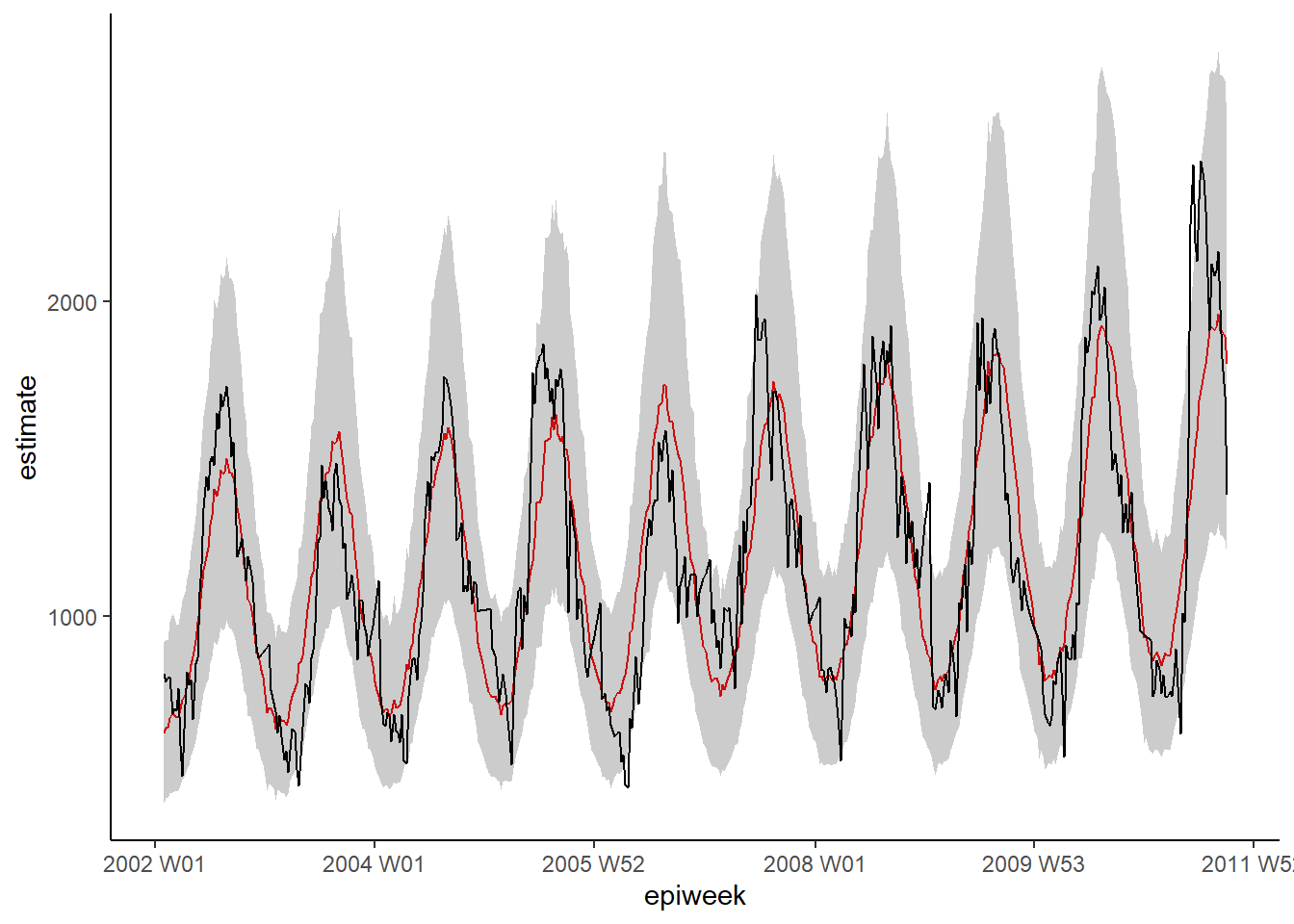
# conf.int = TRUE)Une rapide inspection visuelle du modèle montre qu’il pourrait faire un meilleur travail pour d’estimer le nombre de cas observés.
estimate_res <- data.frame(observed$result)
## Tracez votre régression
ggplot(data = estimate_res, aes(x = epiweek)) +
## ajouter une ligne pour l'estimation du modèle
geom_line(aes(y = estimate),
col = "red") +
## ajouter une bande pour les intervalles de prédiction
geom_ribbon(aes(ymin = lower_pi,
ymax = upper_pi),
alpha = 0.25) +
## ajouter une ligne pour le nombre de cas observés
geom_line(aes(y = case_int),
col = "black") +
## faire un graphique traditionnel (avec des axes noirs et un fond blanc)
theme_classic()Résidus
Nous examinons à nouveau les résidus pour voir si notre modèle s’adapte bien aux données observées. Les résultats et l’interprétation sont ici similaires à ceux de la régression précédente, donc il est peut-être plus judicieux de s’en tenir au modèle plus simple sans température.
## calculer les résidus
estimate_res <- estimate_res %>%
mutate(resid = case_int - estimate)
## les résidus sont-ils assez constants dans le temps (si non : épidémies ? changement de pratique ?)
estimate_res %>%
ggplot(aes(x = epiweek, y = resid)) +
geom_line() +
geom_point() +
labs(x = "epiweek", y = "Residuals")## Y a-t-il une autocorrélation dans les résidus (y a-t-il un modèle d'erreur ?) ?
estimate_res %>%
as_tsibble(index = epiweek) %>%
ACF(resid, lag_max = 52) %>%
autoplot()## les résidus sont-ils normalement distribués (y a-t-il sous-estimation ou surestimation ?)
estimate_res %>%
ggplot(aes(x = resid)) +
geom_histogram(binwidth = 100) +
geom_rug() +
labs(y = "count") ## comparer les comptages observés à leurs résidus
## il ne devrait pas y avoir de modèle
estimate_res %>%
ggplot(aes(x = estimate, y = resid)) +
geom_point() +
labs(x = "Fitted", y = "Residuals")## tester formellement l'autocorrélation des résidus
## H0 est que les résidus proviennent d'une série à bruit blanc (c'est-à-dire aléatoire)
## Test d'indépendance
### si la valeur p est significative alors non aléatoire
Box.test(estimate_res$resid, type = "Ljung-Box")
Box-Ljung test
data: estimate_res$resid
X-squared = 339.52, df = 1, p-value < 2.2e-1623.7 Détection des épidémies
Nous allons démontrer ici deux méthodes (similaires) de détection des épidémies. La première s’appuie sur les sections précédentes. Nous utilisons le paquet trending pour ajuster les régressions aux années précédentes, et puisprédire ce que nous nous attendons à voir l’année suivante. Si les comptages observés sont supérieursce que nous attendons, cela pourrait suggérer qu’il y a une épidémie. La deuxième méthode est basée sur des principes similaires mais utilise le paquet surveillance,qui possède un certain nombre d’algorithmes différents pour la détection des aberrations.
ATTENTION: Normalement, vous vous intéressez à l’année en cours (où vous ne connaissez que les comptages jusqu’à la semaine actuelle). Donc, dans cet exemple, nous prétendons être dans la semaine 39 de 2011.
tendance paquet
Pour cette méthode, nous définissons une ligne de base (qui devrait généralement être d’environ 5 ans de données). Nous ajustons une régression aux données de base, puis nous l’utilisons pour prédire les estimations pour l’année suivante.
Date limite
Il est plus facile de définir vos dates à un endroit, puis de les utiliser dans le reste de votre code.
Ici nous définissons une date de début (quand nos observations ont commencé) et une date limite (la fin de notre période de référence - et le début de la période pour laquelle nous voulons prédire).Nous définissons également le nombre de semaines entre la date limite de la période de référence et la date de fin de la période pour laquelle nous sommes intéressés à prédire.
ATTENTION: Dans cet exemple, nous prétendons être actuellement à la fin du mois de septembre 2011 (“2011 W39”).
## définir la date de début (quand les observations ont commencé)
start_date <- min(counts$epiweek)
## définir une semaine de coupure (fin de la ligne de base, début de la période de prédiction)
cut_off <- yearweek("2010-12-31")
## définir la dernière date qui nous intéresse (c'est-à-dire la fin de la prédiction)
end_date <- yearweek("2011-12-31")
## trouver combien de semaines dans la période (année) d'intérêt.
num_weeks <- as.numeric(end_date - cut_off)23.7.0.1 Ajoutez des lignes {.unnumbered}.
Pour pouvoir faire des prévisions dans un format tidyverse, nous devons avoir le bon nombre de lignes dans notre jeu de données, c’est-à-dire une ligne pour chaque semaine jusqu’à la date de fin définie ci-dessus. Le code ci-dessous vous permet d’ajouter ces lignes par une variable de regroupement - par exemple, si nous avons plusieurs pays dans un même ensemble de données, nous pouvons ajouter des lignes pour chacun d’entre eux. La fonction group_by_key() de tsibble nous permet d’effectuer ce regroupement, et ensuite de passer les données groupées aux fonctions dplyr, group_modify() et add_row(). Ensuite, nous spécifions la séquence des semaines entre une après la semaine maximale actuellement disponible dans les données et la semaine de fin.
## ajouter les semaines manquantes jusqu'à la fin de l'année
counts <- counts %>%
## regrouper par région
group_by_key() %>%
## pour chaque groupe, ajoutez les lignes à partir de la semaine d'épi la plus élevée jusqu'à la fin de l'année
group_modify(~add_row(.,
epiweek = seq(max(.$epiweek) + 1,
end_date,
by = 1)))Termes de Fourier
Nous devons redéfinir nos termes de Fourier, car nous voulons les adapter à la date de base uniquement, puis prédire (extrapoler) ces termes pour l’annee suivante. Pour ce faire, nous devons combiner deux listes de sortie de la fonction fourier() ensemble ; la première est pour les données de base, et la seconde prédit pour l’année qui nous intéresse (en définissant le paramètre fourier()).
N.b. pour lier les lignes, nous devons utiliser rbind() (plutôt que tidyverse bind_rows) carles colonnes de fourier sont une liste (et ne sont donc pas nommées individuellement).
## définir les termes de fourier (sincos)
counts <- counts %>%
mutate(
## combiner les termes de fourier pour les semaines avant et après la date limite de 2010
## (nb. les termes de fourier de 2011 sont prédits)
fourier = rbind(
## obtenir les termes de fourier pour les années précédentes
fourier(
## garder uniquement les lignes avant 2011
filter(counts,
epiweek <= cut_off),
## inclure un ensemble de termes sin cos
K = 1
),
## prédire les termes de fourier pour 2011 (en utilisant les données de base)
fourier(
## garder uniquement les lignes avant 2011
filter(counts,
epiweek <= cut_off),
## inclure un ensemble de termes sin cos
K = 1,
## prédire 52 semaines à l'avance
h = num_weeks
)
)
)Diviser les données et ajuster la régression
Nous devons maintenant diviser notre ensemble de données en deux périodes : la période de base et la période de prédiction. Ceci est fait en utilisant la fonction dplyr group_split() après group_by(), et créera une liste avec deux cadres de données, un pour la période avant la coupure et un pour la période après la coupure.
Nous utilisons ensuite la fonction pluck() du paquet purrr pour extraire les ensembles de données de la liste (ce qui équivaut à utiliser des crochets, par exemple dat[[1]]), et nous pouvons alors ajuster notre modèle aux données de base, et ensuite utiliser la fonction predict() pour nos données d’intérêt après la coupure.
Consultez la page sur l’itération, les boucles et les listes pour en savoir plus sur purrr.
ATTENTION: Notez l’utilisation de simulate_pi = FALSE dans l’argument predict(). Ceci est dû au fait que le comportement par défaut de trending est d’utiliser le paquet ciTools pour estimer un intervalle de prédiction. Cela ne fonctionne pas s’il y a des comptes NA, et produit également des intervalles plus granulaires. Voir ?trending::predict.trending_model_fit pour plus de détails.
# diviser les données pour l'ajustement et la prédiction
dat <- counts %>%
group_by(epiweek <= cut_off) %>%
group_split()
## définir le modèle que vous voulez ajuster (binomial négatif)
model <- glm_nb_model(
## définir le nombre de cas comme résultat d'intérêt
case_int ~
## utiliser epiweek pour tenir compte de la tendance
epiweek +
## utiliser les termes de fourier pour tenir compte de la saisonnalité
fourier
)
# définir les données à utiliser pour l'ajustement et celles pour la prédiction.
fitting_data <- pluck(dat, 2)
pred_data <- pluck(dat, 1) %>%
select(case_int, epiweek, fourier)
# ajustement du modèle
fitted_model <- trending::fit(model, data.frame(fitting_data))
# obtenir le confint et les estimations pour les données ajustées
observed <- fitted_model %>%
predict(simulate_pi = FALSE)
# prévoir avec les données que l'on veut prévoir avec
forecast <- fitted_model %>%
predict(data.frame(pred_data), simulate_pi = FALSE)
## combiner les ensembles de données de base et prédits
observed <- bind_rows(observed$result, forecast$result)Comme précédemment, nous pouvons visualiser notre modèle avec ggplot. Nous mettons en évidence les alertes avecpoints rouges pour les comptes observés au-dessus de l’intervalle de prédiction de 95 %. Cette fois, nous ajoutons également une ligne verticale pour indiquer quand la prévision commence.
## Tracez votre régression
ggplot(data = observed, aes(x = epiweek)) +
## ajoutez une ligne pour l'estimation du modèle
geom_line(aes(y = estimate),
col = "grey") +
## ajouter une bande pour les intervalles de prédiction
geom_ribbon(aes(ymin = lower_pi,
ymax = upper_pi),
alpha = 0.25) +
## ajouter une ligne pour le nombre de cas observés
geom_line(aes(y = case_int),
col = "black") +
## Tracez des points pour les nombres observés supérieurs aux prévisions.
geom_point(
data = filter(observed, case_int > upper_pi),
aes(y = case_int),
color = "red",
size = 2) +
## ajouter une ligne verticale et une étiquette pour montrer où la prévision a commencé
geom_vline(
xintercept = as.Date(cut_off),
linetype = "dashed") +
annotate(geom = "text",
label = "Forecast",
x = cut_off,
y = max(observed$upper_pi) - 250,
angle = 90,
vjust = 1
) +
## faire un graphique traditionnel (avec des axes noirs et un fond blanc)
theme_classic()Warning: Removed 13 rows containing missing values or values outside the scale range
(`geom_line()`).Validation de la prédiction
Au-delà de l’inspection des résidus, il est important d’étudier la capacité de votre modèle à prédire les cas dans le futur. Cela vous donne une idée de la fiabilité de vos seuils d’alerte.
La méthode traditionnelle de validation consiste à voir dans quelle mesure vous pouvez prédire l’année la plus récente avant l’année en cours (parce que vous ne pouvez pas prédire l’année en cours).Par exemple, dans notre ensemble de données, nous utiliserions les données de 2002 à 2009 pour prédire 2010, et ensuite voir si ces prédictions sont exactes. Ensuite, nous réajustons le modèle pour incluredonnées de 2010 et les utiliser pour prédire les comptages de 2011.
Comme on peut le voir dans la figure ci-dessous, réalisée par Hyndman et al dans “Forecasting principles and practice”.
 figure reproduite avec l’autorisation des auteurs.
figure reproduite avec l’autorisation des auteurs.
L’inconvénient de cette méthode est que vous n’utilisez pas toutes les données dont vous disposez et que vous n’obtenez pas le modèle final que vous utilisez pour la prédiction.
Une alternative consiste à utiliser une méthode appelée validation croisée. Dans ce scénario, vous passez en revue toutes les données disponibles pour ajuster plusieurs modèles afin de prédire un an à l’avance. Vous utilisez de plus en plus de données dans chaque modèle, comme le montre la figure ci-dessous tirée de la même [Hyndman et al texte]((https://otexts.com/fpp3/). Par exemple, le premier modèle utilise 2002 pour prédire 2003, le second utilise 2002 et 2003 pour prédire 2004, et ainsi de suite.  figure reproduite avec l’autorisation des auteurs.
figure reproduite avec l’autorisation des auteurs.
Dans la figure ci-dessous, nous utilisons la fonction map() du paquet purrr pour boucler sur chaque ensemble de données. Nous mettons ensuite les estimations dans un seul ensemble de données et les fusionnons avec le nombre de cas original, pour utiliser le paquet yardstick pour calculer les mesures de précision. Nous calculons quatre mesures, notamment Erreur quadratique moyenne (RMSE), Erreur absolue moyenne (MAE) l’erreur absolue moyenne mise à l’échelle (MASE), l’erreur absolue moyenne en pourcentage (MAPE).
CAUTION: Notez l’utilisation de simulate_pi = FALSE dans l’argument predict(). Ceci est dû au fait que le comportement par défaut de trending est d’utiliser le paquet ciTools pour estimer un intervalle de prédiction. Cela ne fonctionne pas s’il y a des comptes NA, et produit également des intervalles plus granulaires. Voir ?trending::predict.trending_model_fit pour plus de détails.
## Validation croisée : prédire la ou les semaines à venir en fonction de la fenêtre glissante.
## élargissez vos données en les faisant glisser dans des fenêtres de 52 semaines (avant + après).
## pour prédire les 52 semaines à venir
## (crée des chaînes d'observations de plus en plus longues - conserve les données plus anciennes)
## définir la fenêtre que l'on veut faire glisser
roll_window <- 52
## Définir les semaines à venir à prévoir
weeks_ahead <- 52
## créer un ensemble de données répétitives, de plus en plus longues.
## étiqueter chaque ensemble de données avec un identifiant unique.
## utiliser seulement les cas avant l'année d'intérêt (i.e. 2011)
case_roll <- counts %>%
filter(epiweek < cut_off) %>%
## Garder uniquement les variables de la semaine et du nombre de cas.
select(epiweek, case_int) %>%
## laisser tomber les x dernières observations
## en fonction du nombre de semaines d'anticipation de la prévision.
## (sinon ce sera une prévision réelle à "inconnu")
slice(1 :(n() - weeks_ahead)) %>%
as_tsibble(index = epiweek) %>%
## reconduire chaque semaine dans x après les fenêtres pour créer l'ID de regroupement
## en fonction de la fenêtre de roulement spécifiée
stretch_tsibble(.init = roll_window, .step = 1) %>%
## laisser tomber les deux premiers - car il n'y a pas de cas "avant".
filter(.id > roll_window)
## pour chacun des ensembles de données uniques, exécutez le code ci-dessous
forecasts <- purrr::map(unique(case_roll$.id),
function(i) {
## garder uniquement le pli courant en cours d'ajustement
mini_data <- filter(case_roll, .id == i) %>%
as_tibble()
## créer un ensemble de données vides pour la prévision sur
forecast_data <- tibble(
epiweek = seq(max(mini_data$epiweek) + 1,
max(mini_data$epiweek) + weeks_ahead,
by = 1),
case_int = rep.int(NA, weeks_ahead),
.id = rep.int(i, weeks_ahead)
)
## ajouter les données de prévision à l'original
mini_data <- bind_rows(mini_data, forecast_data)
## définir le cut off basé sur les dernières données de comptage non manquantes.
cv_cut_off <- mini_data %>%
## ne garder que les lignes non manquantes
drop_na(case_int) %>%
## obtenir la dernière semaine
summarise(max(epiweek)) %>%
## extraire ce qui n'est pas dans un dataframe
pull()
## Remettre mini_data dans un tsibble
mini_data <- tsibble(mini_data, index = epiweek)
## définir les termes de fourier (sincos)
mini_data <- mini_data %>%
mutate(
## Combinez les termes de Fourier pour les semaines avant et après la date limite.
fourier = rbind(
## obtenir les termes de fourier pour les années précédentes
forecast::fourier(
### ne conserve que les lignes avant la date butoir
filter(mini_data,
epiweek <= cv_cut_off),
## inclure un ensemble de termes sin cos
K = 1
),
## prédire les termes de fourier pour l'année suivante (en utilisant les données de base)
fourier(
## conserver uniquement les lignes avant la coupure
filter(mini_data,
epiweek <= cv_cut_off),
## inclure un ensemble de termes sin cos
K = 1,
## prédire 52 semaines à l'avance
h = weeks_ahead
)
)
)
# diviser les données pour l'ajustement et la prédiction
dat <- mini_data %>%
group_by(epiweek <= cv_cut_off) %>%
group_split()
## définir le modèle que vous voulez ajuster (binomiale négative)
model <- glm_nb_model(
## définir le nombre de cas comme résultat d'intérêt
case_int ~
## utiliser epiweek pour tenir compte de la tendance
epiweek +
## utiliser les termes de fourier pour tenir compte de la saisonnalité
fourier
)
# définir les données à utiliser pour l'ajustement et celles pour la prédiction.
fitting_data <- pluck(dat, 2)
pred_data <- pluck(dat, 1)
# ajuster le modèle
fitted_model <- trending::fit(model, data.frame(fitting_data))
# prévoir avec les données que l'on veut prédire avec
forecasts <- fitted_model %>%
predict(data.frame(pred_data), simulate_pi = FALSE)
forecasts <- data.frame(forecasts$result[[1]]) %>%
## garder seulement la semaine et l'estimation de la prévision
select(epiweek, estimate)
}
)
## Transformer la liste en un cadre de données avec toutes les prévisions.
forecasts <- bind_rows(forecasts)
## joindre les prévisions aux données observées
forecasts <- left_join(forecasts,
select(counts, epiweek, case_int),
by = "epiweek")
## en utilisant {yardstick} calculer les métriques
## RMSE : Root mean squared error (erreur quadratique moyenne)
## MAE : Erreur absolue moyenne
## MASE : Mean absolute scaled error (erreur absolue moyenne mise à l'échelle)
## MAPE : Erreur absolue moyenne en pourcentage
model_metrics <- bind_rows(
## dans votre ensemble de données forcées, comparez les données observées aux données prédites.
rmse(forecasts, case_int, estimate),
mae( forecasts, case_int, estimate),
mase(forecasts, case_int, estimate),
mape(forecasts, case_int, estimate),
) %>%
### ne conserve que le type de métrique et sa sortie
select(Metric = .metric,
Measure = .estimate) %>%
## faire en sorte que le format soit large pour pouvoir lier les lignes ensuite
pivot_wider(names_from = Metric, values_from = Measure)
## Retourner les métriques du modèle
model_metrics# A tibble: 1 × 4
rmse mae mase mape
<dbl> <dbl> <dbl> <dbl>
1 252. 199. 1.96 17.3surveillance paquet
Dans cette section, nous utilisons le paquet surveillance pour créer des seuils d’alerte basés sur des algorithmes de détection d’épidémies. Il existe plusieurs méthodes différentes disponibles dans le paquet, mais nous nous concentrerons ici sur deux options. Pour plus de détails, voir ces articles sur l’application et théorie des alogirths utilisés.
La première option utilise la méthode améliorée de Farrington. Celle-ci ajuste un glm binomial négatif (y compris la tendance) et pondère à la baisse les épidémies passées (valeurs aberrantes) pour créer un niveau de seuil.
La deuxième option utilise la méthode glrnb. Celle-ci ajuste également un glm binomial négatif binomiale négative, mais inclut la tendance et les termes de Fourier (elle est donc privilégiée ici). La régression est utilisée pour calculer la “moyenne de contrôle” (~valeurs ajustées) - elle utilise ensuite une statistique de rapport de vrai semblance généralisé calculé pour évaluer s’il y a un changement de la moyenne pour chaque semaine. Notez que le seuil pour chaque semaine prend en compte les semaines précédentes, de sorte que s’il y a un changement soutenu, une alarme sera déclenchée. (Notez également qu’après chaque alarme, l’algorithme est réinitialisé).
Afin de travailler avec le paquet surveillance, nous devons d’abord définir un objet “série temporelle de surveillance” (en utilisant la fonction sts()) pour s’intégrer dans le cadre.
## Définir un objet de série temporelle de surveillance
## nb. vous pouvez inclure un dénominateur avec l'objet population (voir ?sts)
counts_sts <- sts(observed = counts$case_int[!is.na(counts$case_int)],
start = c(
## sous-ensemble pour ne garder que l'année de start_date.
as.numeric(str_sub(start_date, 1, 4)),
## sous-ensemble pour ne conserver que la semaine à partir de la date de départ
as.numeric(str_sub(start_date, 7, 8))),
## définir le type de données (dans ce cas, hebdomadaire)
freq = 52)
## définir la plage de semaines que vous voulez inclure (c'est-à-dire la période de prédiction)
## nb. l'objet sts ne compte que les observations sans leur attribuer un identifiant de semaine ou d'année.
## d'année - nous utilisons donc nos données pour définir les observations appropriées.
weekrange <- cut_off - start_dateMéthode Farrington
Nous définissons ensuite chacun de nos paramètres pour la méthode de Farrington dans une liste. Ensuite, nous exécutons l’algorithme en utilisant farringtonFlexible() et ensuite nous pouvons extraire le seuil d’une alerte en utilisant farringtonmethod@upperbound pour l’inclure dans notre données. Il est également possible d’extraire un VRAI/FAUX pour chaque semaine si elle a déclenché une alerte (au-dessus du seuil) en utilisant farringtonmethod@alarm.
## définir le contrôle
ctrl <- list(
## définissez la période pour laquelle vous voulez un seuil (i.e. 2011)
range = which(counts_sts@epoch > weekrange),
b = 9, ## nombre d'années en arrière pour la ligne de base
w = 2, ## taille de la fenêtre de roulement en semaines
weightsThreshold = 2.58, ## repondération des épidémies passées (méthode noufaily améliorée - l'originale suggère 1)
## pastWeeksNotIncluded = 3, ## utilisation de toutes les semaines disponibles (noufaily suggère d'en éliminer 26)
trend = TRUE,
pThresholdTrend = 1, ## 0.05 normalement, mais 1 est conseillé dans la méthode améliorée (c'est-à-dire toujours garder)
thresholdMethod = "nbPlugin",
populationOffset = TRUE
)
## appliquer la méthode flexible de Farrington
farringtonmethod <- farringtonFlexible(counts_sts, ctrl)
## créer une nouvelle variable dans le jeu de données original appelée threshold.
## contenant la limite supérieure de Farrington.
## nb. ceci est seulement pour les semaines de 2011 (donc besoin de sous-ensembler les lignes)
counts[which(counts$epiweek >= cut_off &
!is.na(counts$case_int)),
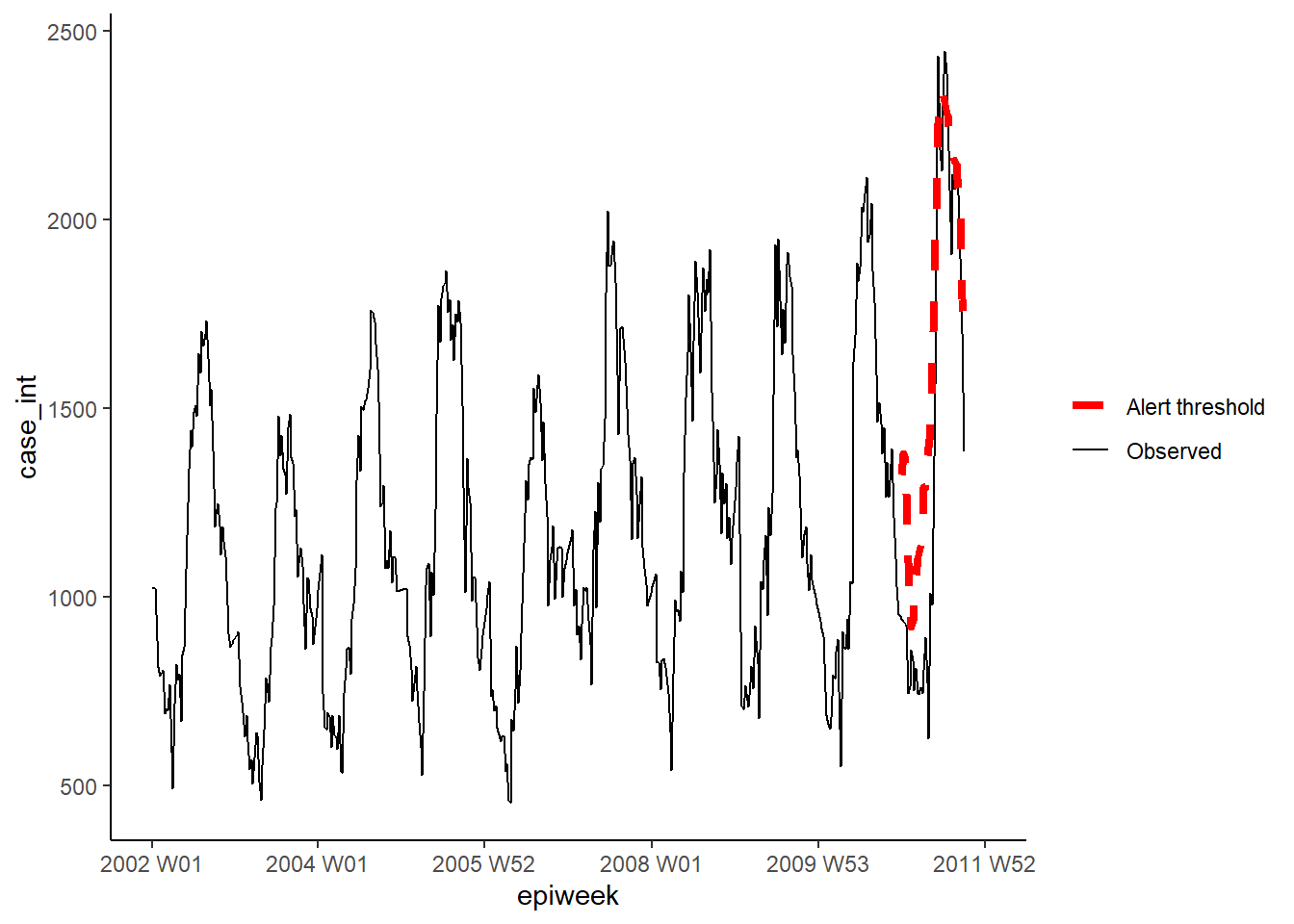
"threshold"] <- farringtonmethod@upperboundNous pouvons ensuite visualiser les résultats dans ggplot comme nous l’avons fait précédemment.
ggplot(counts, aes(x = epiweek)) +
## ajouter le nombre de cas observés sous forme de ligne
geom_line(aes(y = case_int, colour = "Observed")) +
## ajout de la limite supérieure de l'algorithme d'aberration
geom_line(aes(y = threshold, colour = "Alert threshold"),
linetype = "dashed",
size = 1.5) +
## définir les couleurs
scale_colour_manual(values = c("Observed" = "black",
"Alert threshold" = "red")) +
## faire un graphique traditionnel (avec des axes noirs et un fond blanc)
theme_classic() +
## supprimer le titre de la légende
theme(legend.title = element_blank())Méthode GLRNB
De même pour la méthode GLRNB, nous définissons chacun de nos paramètres pour le dans une liste, puis nous ajustons l’algorithme et extrayons les limites supérieures.
ATTENTION: Cette méthode utilise la “force brute” (similaire au bootstrapping) pour calculer les seuils, donc peut prendre beaucoup de temps!.
Voir la vignette GLRNB pour plus de détails.
## définir les options de contrôle
ctrl <- list(
## définir la période pour laquelle on veut un seuil (i.e. 2011)
range = which(counts_sts@epoch > weekrange),
mu0 = list(S = 1, ## nombre de termes de fourier (harmoniques) à inclure
trend = TRUE, ## inclusion ou non de la tendance
refit = FALSE), ## si l'on refit le modèle après chaque alarme
## cARL = seuil pour la statistique GLR (arbitraire)
## 3 ~ seuil intermédiaire pour minimiser les faux positifs
## 1 s'ajuste aux 99%PI de glm.nb - avec des changements après les pics (seuil abaissé pour l'alerte)
c.ARL = 2,
# thêta = log(1.5), ## équivaut à une augmentation de 50% des cas dans une épidémie
ret = "cases" ## retourne la limite supérieure du seuil sous forme de nombre de cas
)
## appliquer la méthode glrnb
glrnbmethod <- glrnb(counts_sts, control = ctrl, verbose = FALSE)
## créer une nouvelle variable dans l'ensemble de données original appelée threshold
## contenant la limite supérieure de glrnb.
## nb. ceci est seulement pour les semaines de 2011 (donc besoin de sous-ensembler les lignes)
counts[which(counts$epiweek >= cut_off &
!is.na(counts$case_int)),
"threshold_glrnb"] <- glrnbmethod@upperboundVisualisez les sorties comme précédemment.
ggplot(counts, aes(x = epiweek)) +
## ajouter le nombre de cas observés sous forme de ligne
geom_line(aes(y = case_int, colour = "Observed")) +
## ajout de la limite supérieure de l'algorithme d'aberration
geom_line(aes(y = threshold_glrnb, color = "Alert threshold"),
linetype = "dashed",
size = 1.5) +
## définir les couleurs
scale_colour_manual(values = c("Observed" = "black",
"Alert threshold" = "red")) +
## faire un graphique traditionnel (avec des axes noirs et un fond blanc)
theme_classic() +
## supprimer le titre de la légende
theme(legend.title = element_blank())23.8 Série chronologique interrompue
Les séries chronologiques interrompues (également appelées régression segmentée ou analyse d’intervention), est souvent utilisée pour évaluer l’impact des vaccins sur l’incidence des maladies. Mais elle peut être utilisée pour évaluer l’impact d’un large éventail d’interventions ou d’introductions. Par exemple, des changements dans les procédures hospitalières ou l’introduction d’une nouvelle souche de maladie dans une population. Dans cet exemple, nous supposerons qu’une nouvelle souche de Campylobacter a été introduite en Allemagne fin 2008, et nous verrons si cela affecte le nombre de cas. Nous utiliserons à nouveau la régression binomiale négative. Cette fois-ci, la régression sera divisée en deux parties, l’une avant l’intervention (ou l’introduction de la nouvelle souche ici) et une autre après (les périodes pré et post). Cela nous permet de calculer un ratio de taux d’incidence comparant les deux périodes. Une explication de l’équation pourrait rendre les choses plus claires (sinon, ignorez-la !).
La régression binomiale négative peut être définie comme suit :
\[\log(Y_t)= β_0 + β_1 \times t+ β_2 \times δ(t-t_0) + β_3\times(t-t_0 )^+ + log(pop_t) + e_t\]
Où : \(Y_t\) est le nombre de cas observés au temps \(t\).
\(pop_t\) est la taille de la population en 100 000 au moment \(t\) (non utilisé ici)
\(t_0\) est la dernière année de la pré-période (y compris la période de transition, le cas échéant).
\(Î'(x\) est la fonction indicatrice (elle vaut 0 si xâ¤0 et 1 si x>0)
\((x)^+\) est l’opérateur de coupure (il vaut x si x>0 et 0 sinon)
\(e_t\) désigne le résidu Des termes supplémentaires, tendance et saison, peuvent être ajoutés si nécessaire.
\(β_2 \times Î'(t-t_0) + β_3\times(t-t_0 )^+\) est la partie linéaire généralisée de la post-période et est nulle dans la pré-période. Cela signifie que les estimations de \(β_2\) et \(β_3\) sont les effets de l’intervention.
Nous devons recalculer les termes de fourier sans faire de prévision ici, car nous utiliserons toutes les données dont nous disposons (c’est-à-dire rétrospectivement). De plus, nous devons calculerles termes supplémentaires nécessaires à la régression.
## ajouter les termes de fourier en utilisant les variabless epiweek et case_int
counts$fourier <- select(counts, epiweek, case_int) %>%
as_tsibble(index = epiweek) %>%
fourier(K = 1)
## définir la semaine d'intervention
intervention_week <- yearweek("2008-12-31")
## définir les variables pour la régression
counts <- counts %>%
mutate(
## correspond à t dans la formule
## nombre de semaines (on pourrait probablement aussi utiliser la variable epiweeks)
# linear = row_number(epiweek),
## correspond au delta(t-t0) dans la formule
## période de pré ou post intervention
intervention = as.numeric(epiweek >= intervention_week),
## correspond à (t-t0)^+ dans la formule
## nombre de semaines après l'intervention
## (choisir le plus grand nombre entre 0 et ce qui ressort du calcul)
time_post = pmax(0, epiweek - intervention_week + 1))Nous utilisons ensuite ces termes pour ajuster une régression binomiale négative, et produisons un tableau avec le pourcentage de changement. Cet exemple montre qu’il n’y a pas eu de changement significatif.
ATTENTION: Notez l’utilisation de simulate_pi = FALSE dans l’argument predict(). Ceci est dû au fait que le comportement par défaut de trending est d’utiliser le paquet ciTools pour estimer un intervalle de prédiction. Cela ne fonctionne pas s’il y a des comptes NA, et produit également des intervalles plus granulaires. Voir ?trending::predict.trending_model_fit pour plus de détails.
## définissez le modèle que vous voulez ajuster (binomial négatif).
model <- glm_nb_model(
## définir le nombre de cas comme résultat d'intérêt
case_int ~
## utiliser epiweek pour tenir compte de la tendance
epiweek +
## utiliser les termes fourier pour tenir compte de la saisonnalité
fourier +
## ajouter si dans la pré ou post-période
intervention +
## ajouter le temps après l'intervention
time_post
)
## ajustez votre modèle en utilisant l'ensemble de données de comptage
fitted_model <- trending::fit(model, data.frame(counts))
### calculer les intervalles de confiance et les intervalles de prédiction
observed <- predict(fitted_model, simulate_pi = FALSE)## Afficher les estimations et le pourcentage de changement dans un tableau
fitted_model %>%
## extraire la régression binomiale négative originale
get_fitted_model() %>%
## obtenir un cadre de données ordonné des résultats
tidy(exponentiate = TRUE,
conf.int = TRUE) %>% ### ne conserve que les valeurs d'intervention
### ne conserve que la valeur d'intervention
filter(term == "intervention") %>%
## changer le IRR en pourcentage de changement pour l'estimation et les ICs
mutate(
## pour chacune des colonnes d'intérêt - créer une nouvelle colonne
across(
all_of(c("estimate", "conf.low", "conf.high")),
## appliquer la formule pour calculer le pourcentage de changement
.f = function(i) 100 * (i - 1),
## ajouter un suffixe aux nouveaux noms de colonnes avec "_perc".
.names = "{.col}_perc")
) %>%
## ne garder (et renommer) que certaines colonnes
select( "IRR" = estimate,
"95%CI low" = conf.low,
"95%CI high" = conf.high,
"Variation in percentag" = estimate_perc,
"95%CI low (perc)" = conf.low_perc,
"95%CI high (perc)" = conf.high_perc,
"p-value" = p.value)Comme précédemment, nous pouvons visualiser les résultats de la régression.
estimate_res <- data.frame(observed$result)
ggplot(estimate_res, aes(x = epiweek)) +
## ajouter le nombre de cas observés sous forme de ligne
geom_line(aes(y = case_int, colour = "Observed")) +
## ajout d'une ligne pour l'estimation du modèle
geom_line(aes(y = estimate, col = "Estimate")) +
## ajouter une bande pour les intervalles de prédiction
geom_ribbon(aes(ymin = lower_pi,
ymax = upper_pi),
alpha = 0.25) +
## ajouter une ligne verticale et une étiquette pour montrer où la prévision a commencé
geom_vline(
xintercept = as.Date(intervention_week),
linetype = "dashed") +
annotate(geom = "text",
label = "Intervention",
x = intervention_week,
y = max(estimate_res$upper_pi),
angle = 90,
vjust = 1
) +
## définir les couleurs
scale_colour_manual(values = c("Observed" = "black",
"Estimate" = "red")) +
## faire un graphique traditionnel (avec des axes noirs et un fond blanc)
theme_classic()23.9 Ressources
Prévision : principes et pratique - manuel
Études de cas d’analyse de séries temporelles EPIET
Cours de Penn State Manuscrit du paquet de surveillance