7 Importer et exporter des données
Sur cette page, nous décrivons les moyens de localiser, d’importer et d’exporter des fichiers :
Utilisation du package rio pour
import()etexport()de manière flexible de nombreux types de fichiersUtilisation du package here pour localiser des fichiers relatifs à la racine d’un projet R - pour prévenir des complications liées aux chemins d’accès de fichiers qui sont spécifiques à un ordinateur
Des scénarios d’importation spécifiques, tels que :
Feuilles d’Excel spécifiques
En-têtes désordonnés et lignes sautées
Feulles de calcul Google
À partir des données publiées sur les sites web
Avec des APIs
Importer le plus récent fichier
Saisie manuelle des données
Type de fichier spécifique à R tels que RDS et RData
Exportation/sauvegarde des fichiers et des graphiques
7.1 Aperçu
Lorsque vous importez un “jeu de données” dans R, vous créez généralement un nouvel objet appelé data frame dans votre environnement R et vous le définissez comme un fichier importé (par exemple Excel, CSV, TSV, RDS) qui se trouve dans votre répertoire de dossiers à un certain chemin d’accès au fichier.
Vous pouvez importer/exporter de nombreux types de fichiers, y compris ceux créés par d’autres programmes statistiques (SAS, STATA, SPSS). Vous pouvez également vous connecter à des bases de données relationnelles.
R a même ses propres formats de données :
- Un fichier RDS (.rds) stocke un seul objet R tel qu’un dataframe. Ils sont utiles pour stocker des données nettoyées, car ils conservent les classes de colonnes R. Pour en savoir plus, consultez cette section.
- Un fichier RData (.Rdata) peut être utilisé pour stocker plusieurs objets, voire un espace de travail R complet. Pour en savoir plus, consultez cette section.
7.2 Le package rio
Le paquet R que nous recommandons est : rio. Le nom “rio” est une abréviation de “R I/O” (input/output).
Ses fonctions import() et export() peuvent traiter de nombreux types de fichiers différents (par exemple .xlsx, .csv, .rds, .tsv). Lorsque vous fournissez un chemin d’accès au fichier à l’une de ces fonctions (y compris l’extension de fichier comme “.csv”), rio lira l’extension et utilisera le bon outil pour importer ou exporter le fichier.
L’alternative à l’utilisation de rio est d’utiliser les fonctions de nombreux autres packages, chacun d’entre eux étant spécifique à un type de fichier. Par exemple, read.csv() (base R), read.xlsx() (openxlsx package), et write_csv() (readr pacakge), etc. Ces alternatives peuvent être difficiles à mémoriser, alors qu’utiliser import() et export() de rio est facile.
Les fonctions import() et export() de rio utilisent le package et la fonction appropriés pour un fichier donné, en se basant sur son extension. Consultez la fin de cette page pour un tableau complet des paquets/fonctions que rio utilise en arrière-plan. Il peut également être utilisé pour importer des fichiers STATA, SAS, et SPSS, parmi des dizaines d’autres types de fichiers.
L’importation/exportation de shapefile nécessite d’autres packages, tel que détaillé dans la page sur Introduction aux SIG.
7.3 Le package here
Le package here et sa fonction here() permettent d’indiquer facilement à R où trouver et enregistrer vos fichiers - en fait, il construit les chemins d’accès aux fichiers.
Utilisé en conjonction avec un projet R, here vous permet de décrire l’emplacement des fichiers dans votre projet R par rapport au répertoire racine du projet R (le dossier de niveau supérieur). Cela est utile lorsque le projet R peut être partagé ou accessible par plusieurs personnes/ordinateurs. Cela évite les complications dues aux chemins d’accès aux fichiers uniques sur différents ordinateurs (par exemple "C:/Users/Laura/Documents..." en “commençant” le chemin d’accès à un endroit commun pour tous les utilisateurs (la racine du projet R).
Voici comment here() fonctionne dans un projet R :
- Lorsque le package here est chargé pour la première fois dans le projet R, il place un petit fichier appelé “.here” dans le dossier racine de votre projet R comme “repère” ou “ancre”.
- Dans vos scripts, pour référencer un fichier dans les sous-dossiers du projet R, vous utilisez la fonction
here()pour construire le chemin d’accès au fichier en relation avec cette ancre - Pour construire le chemin d’accès au fichier, écrivez les noms des dossiers au-delà de la racine, entre guillemets, séparés par des virgules, et terminez par le nom du fichier et son extension, comme indiqué ci-dessous.
- Les chemins d’accès
here()peuvent être utilisés à la fois pour l’importation et l’exportation.
Par exemple, ci-dessous, la fonction import() reçoit un chemin d’accès construit avec here().
linelist <- import(here("data", "linelists", "ebola_linelist.xlsx"))La commande here("data", "linelists", "ebola_linelist.xlsx") fournit en fait le chemin d’accès complet au fichier qui est unique à l’ordinateur de l’utilisateur :
"C:/Users/Laura/Documents/my_R_project/data/linelists/ebola_linelist.xlsx"L’avantage est que la commande R utilisant here() peut être exécutée avec succès sur n’importe quel ordinateur accédant au projet R.
TIP: Si vous n’êtes pas certaine de l’emplacement de la racine “here”, exécutez la fonction here() avec des parenthèses vides.
Plus d’informations sur le package ici en cliquant sur ce lien.
7.4 Chemins d’accès aux fichiers
Lorsque vous importez ou exportez des données, vous devez fournir un chemin d’accès au fichier. Vous pouvez le faire de trois manières différentes:
- Recommandé: fournir un chemin d’accès “relatif” avec le package here
- Fournir le chemin d’accès “complet” / “absolu”
- Sélection manuelle des fichiers
Chemins d’accès “relatifs”
Dans R, les chemins d’accès “relatifs” consistent en un chemin d’accès relatif à la racine d’un projet R. Ils permettent d’obtenir des chemins d’accès plus simples qui peuvent fonctionner sur différents ordinateurs (par exemple, si le projet R se trouve sur un disque partagé ou est envoyé par courrier électronique). Comme décrit ci-dessus, les chemins de fichiers relatifs sont facilités par l’utilisation du package here.
Voici un exemple de chemin d’accès relatif construit avec here(). Nous supposons que le travail se trouve dans un projet R qui contient un sous-dossier “data” et dans celui-ci un sous-dossier “linelists”, dans lequel se trouve le fichier .xlsx qui nous intéresse.
linelist <- import(here("data", "linelists", "ebola_linelist.xlsx"))Chemins d’accès “absolus”
Les chemins d’accès absolus ou “complets” peuvent être fournis à des fonctions comme import() mais ils sont “fragiles” car ils sont uniques à l’ordinateur spécifique de l’utilisateur et donc non recommandés.
Vous trouverez ci-dessous un exemple de chemin d’accès absolu à un fichier. Dans l’ordinateur de Laura, il existe un dossier “analysis”, un sous-dossier “data” et un sous-dossier “linelists”, dans lequel se trouve le fichier .xlsx en question.
linelist <- import("C:/Users/Laura/Documents/analysis/data/linelists/ebola_linelist.xlsx")Quelques points à noter concernant les chemins d’accès absolus aux fichiers:
- Évitez les chemins d’accès absolus parce qu’ils vont interrompre le script si ils sont utilisés sur un autre ordinateur différent
- Utilisez des barres obliques (/), comme dans l’exemple ci-dessus (remarque : ce n’est PAS l’option par défaut pour les chemins d’accès aux fichiers de Windows).
- Les chemins de fichiers qui commencent par des doubles barres obliques (par exemple, “//…”) ne seront probablement pas reconnus par R et produiront une erreur. Pensez à déplacer votre travail sur un lecteur “nommé” ou “lettré” qui commence par une lettre (par exemple “J :” ou “C :”). Voir la page sur les Interactions avec les répertoires pour plus de détails sur cette question.
Un scénario dans lequel les chemins de fichier absolus peuvent être appropriés est celui où vous voulez importer un fichier d’un lecteur partagé qui a le même chemin d’accès complet pour tous les utilisateurs.
TIP: Pour convertir rapidement tous les \ à /, surlignez le code d’intérêt, utilisez Ctrl+f (avec Windows), sélectionnez l’option “Dans la sélection”, et ensuite utilisez la fonction remplacement pour les convertir.
Sélectionner les fichier manuellement
Vous pouvez importer des données de façon manuelle via une de ces méthodes:
- Dans le volet Environnement de RStudio, cliquez sur “Import Dataset”, et sélectionnez le type de données
- Cliquez sur File / Import Dataset / (sélectionnez le type de données)
- Pour une sélection manuelle complète, utilisez la commande
file.choose()de la base R (en laissant les parenthèses vides) pour d’éclencher l’apparition d’une fenêtre pop-up qui permet à l’utilisateur de sélectionner manuellement le fichier sur son ordinateur. Par exemple :
# Sélection manuelle d'un fichier. Lorsque cette commande est exécutée, une fenêtre POP-UP apparaîtra.
# Le chemin du fichier sélectionné sera fourni à la commande import().
my_data <- import(file.choose())TIP: Il est possible que la fenêtre pop-up apparaisse DERRIÈRE votre fenêtre de RStudio.
7.5 Importer des données
Il est assez simple d’utiliser import() pour importer un ensemble des données. Il suffit de fournir le chemin d’accès au fichier (y compris le nom et l’extension du fichier) entre guillemets. Si vous utilisez here() pour construire le chemin d’accès au fichier, suivez les insTIPtions ci-dessus. Voici quelques exemples:
Importation d’un fichier csv situé dans votre “répertoire de travail” ou dans le dossier racine du projet R:
linelist <- import("linelist_cleaned.csv")Importation de la première feuille d’un Excel qui se trouve dans les sous-dossiers “data” et “linelists” du projet R (le chemin du fichier construit à l’aide de here()):
linelist <- import(here("data", "linelists", "linelist_cleaned.xlsx"))Importing d’un data frame (un fichier .rds) à l’aide d’un chemin d’accès absolu:
linelist <- import("C:/Users/Laura/Documents/tuberculosis/data/linelists/linelist_cleaned.rds")Feuilles spécifiques d’Excel
Par défaut, si vous fournissez un fichier Excel (.xlsx) à import(), la première feuille du fichier sera importée. Si vous voulez importer une feuille spécifique, incluez le nom de la feuille dans l’argument which =. Par exemple:
my_data <- import("my_excel_file.xlsx", which = "Sheetname")Si vous utilisez la méthode here() pour fournir un chemin d’accès relatif à import(), vous pouvez toujours indiquer une feuille spécifique en ajoutant l’argument which = après les parenthèses de fermeture de la fonction here().
# Demonstration: importing a specific Excel sheet when using relative pathways with the 'here' package
linelist_raw <- import(here("data", "linelist.xlsx"), which = "Sheet1")` Pour exporter un data frame à partir de R vers une feuille Excel spécifique tout en gardant inchangé le reste du fichier Excel, vous devrez importer, modifier et exporter avec un package alternatif spécifique pour cet objectif tel que openxlsx. Pour davantage d’information consultez la page Interactions avec les répertoires ou cette page github.
Si votre fichier Excel est .xlsb (fichier Excel en format binaire) il est possible que vous ne puissiez pas l’importer en utilisant rio. Envisagez de le réenregistrer en format .xlsx, ou bien en utilisant un package tel que readxlsb qui fut conçu à cet effet.
Données manquantes
Vous pouvez désigner la ou les valeurs de votre ensemble de données qui doivent être considérées comme manquantes. Comme expliqué dans la page Données manquantes, la valeur dans R pour les données manquantes est NA, mais peut-être que l’ensemble de données que vous voulez importer utilise 99, “Manquant”, ou juste un espace vide “” à la place.
Utilisez l’argument na = avec import() et fournissez la ou les valeurs entre guillemets (même si ce sont des nombres). Vous pouvez spécifier plusieurs valeurs en les incluant dans un vecteur, en utilisant c() comme indiqué ci-dessous.
Ici, la valeur “99” dans le jeu de données est considéré comme manquant et converti à NA dans R.
linelist <- import(here("data", "my_linelist.xlsx"), na = "99")Ici, toutes les valeurs “Missing”, “” (cellule vide), ou ” ” (espace unique) dans l’ensemble de données importées sont converties en NA dans R.
linelist <- import(here("data", "my_linelist.csv"), na = c("Missing", "", " "))Sauter des lignes
Parfois, vous pouvez vouloir éviter d’importer une ligne de données. Vous pouvez le faire avec l’argument skip = si vous utilisez import() de rio sur un fichier .xlsx ou .csv. Indiquez le nombre de lignes que vous souhaitez ignorer.
linelist_raw <- import("linelist_raw.xlsx", skip = 1) # n'importe pas la ligne d'en-têteMalheureusement, skip = n’accepte qu’une seule valeur entière, pas une plage (par exemple, “2:10” ne fonctionne pas). Pour sauter l’importation de lignes spécifiques qui ne sont pas consécutives à partir du haut, pensez à importer plusieurs fois et à utiliser bind_rows() de dplyr. Voir l’exemple ci-dessous pour sauter seulement la ligne 2.
Gérer une deuxième ligne d’en-tête
Parfois, vos données peuvent avoir une deuxième ligne, par exemple s’il s’agit d’une ligne de “dictionnaire de données” comme indiqué ci-dessous. Cette situation peut être problématique car elle peut entraîner l’importation de toutes les colonnes en tant que classe “caractère”.
Voici un exemple de ce type d’ensemble de données (la première ligne étant le dictionnaire de données).
Supprimez la deuxième ligne d’en-tête
Pour supprimer la deuxième ligne d’en-tête, vous devrez probablement importer les données deux fois.
- Importez les données afin de stocker les bons noms des colonnes.
- Importez à nouveau les données, en sautant les deux premières lignes (en-tête et deuxième ligne).
- Reliez les noms corrects sur le cadre de données réduit.
L’argument exact utilisé pour relier les bons noms de colonnes dépend du type de fichier de données (.csv, .tsv, .xlsx, etc.). Ceci est dû au fait que rio utilise une fonction différente pour les différents types de fichiers (voir le tableau ci-dessus).
Pour les documents Excel: (col_names =)
# importer pour la première fois; stocker les noms des colonnes
linelist_raw_names <- import("linelist_raw.xlsx") %>% names() # stocker les bons noms des colonnes
# importer pour la deuxième fois; sauter la deuxième ligne, et assigner les noms des colonnes à l'argument col_names =
linelist_raw <- import("linelist_raw.xlsx",
skip = 2,
col_names = linelist_raw_names
) Pour les fichiers CSV: (col.names =)
# importer pour la première fois; stocker les noms des colonnes
linelist_raw_names <- import("linelist_raw.csv") %>% names() # save true column names
# notez que l'argument pour les fichiers CSV est 'col.names = '
linelist_raw <- import("linelist_raw.csv",
skip = 2,
col.names = linelist_raw_names
) Option de secours - changer les noms des colonnes à l’aide d’une commande additionnelle
# attribuer/supprimer des en-têtes en utilisant la fonction de base 'colnames()'
colnames(linelist_raw) <- linelist_raw_namesCréer un dictionnaire de données
Bonus! Si vous avez une deuxième ligne qui est un dictionnaire de données, vous pouvez facilement créer un dictionnaire de données approprié à partir de celle-ci. Cette astuce est adaptée de cet article.
dict <- linelist_2headers %>% # début: liste des case avec le dictionnaire en première ligne
head(1) %>% # garder seulement les noms des colonnes et la premièere ligne étant le dictionnaire
pivot_longer(cols = everything(), # pivoter toutes les colonnes au format long
names_to = "Column", # définir de nouveaux noms pour les colonnes
values_to = "Description")Combiner les deux lignes d’en-tête
Dans certains cas, lorsque votre ensemble de données brutes comporte deux lignes d’en-tête (ou plus précisément, la deuxième ligne de données est un en-tête secondaire), vous pouvez souhaiter les “combiner” ou ajouter les valeurs de la deuxième ligne d’en-tête à la première ligne d’en-tête.
La commande ci-dessous définit les noms des colonnes du cadre de données comme la combinaison (collage) des premiers en-têtes (vrais) avec la valeur située immédiatement en dessous (dans la première ligne).
names(my_data) <- paste(names(my_data), my_data[1, ], sep = "_")Feuille de calcul Google
Vous pouvez importer des données à partir d’une feuille de calcul Google en ligne avec le paquet googlesheet4 et en authentifiant votre accès à la feuille de calcul.
pacman::p_load("googlesheets4")Ci-dessous, une feuille de calcul Google de démonstration est importée et sauvegardée. Cette commande peut vous demander de confirmer l’authentification de votre compte Google. Suivez les insTIPtions et les fenêtres contextuelles de votre navigateur Internet pour accorder aux packages API Tidyverse les autorisations de modifier, créer et supprimer vos feuilles de calcul dans Google Drive.
La feuille ci-dessous est “consultable par toute personne ayant le lien” et vous pouvez essayer de l’importer.
Gsheets_demo <- read_sheet("https://docs.google.com/spreadsheets/d/1scgtzkVLLHAe5a6_eFQEwkZcc14yFUx1KgOMZ4AKUfY/edit#gid=0")La feuille ci-dessous est “consultable par toute personne ayant le lien” et vous pouvez essayer de l’importer.
Gsheets_demo <- read_sheet("1scgtzkVLLHAe5a6_eFQEwkZcc14yFUx1KgOMZ4AKUfY")Un autre package, googledrive, propose des fonctions utiles pour écrire, modifier et supprimer des feuilles Google. Par exemple, en utilisant les fonctions gs4_create() et sheet_write() trouvées dans ce package.
Voici d’autres tutoriels en ligne utiles:
Tutoriel de base sur l’importation de feuilles de calcul Google sheets
tutoriel plus détaillé
intéraction entre googlesheets4 et tidyverse
7.6 Fichiers multiples - importation, exportation, fractionnement, combinaison
Consultez la page Itération, boucles et listes pour obtenir des exemples sur la manière d’importer et de combiner plusieurs fichiers, ou plusieurs fichiers de classeur Excel. Cette page contient également des exemples sur la façon de diviser un cadre de données en plusieurs parties et d’exporter chacune d’entre elles séparément, ou en tant que feuilles nommées dans un classeur Excel.
7.7 Importer de Github
L’importation de données directement de Github dans R peut être très facile ou nécessiter quelques étapes, selon le type de fichier. Voici quelques approches:
Fichiers CSV
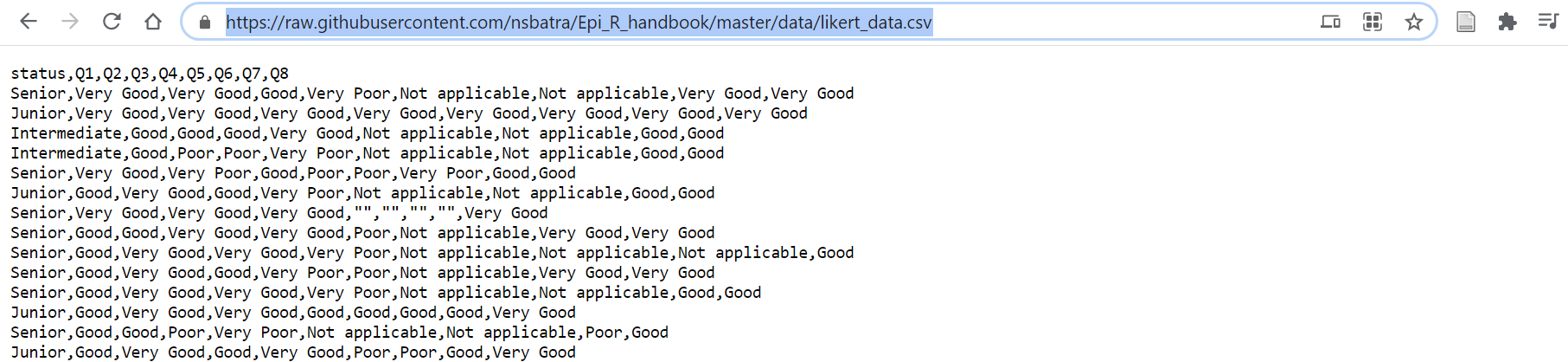
Il peut être facile d’importer un fichier .csv directement de Github dans R avec une commande R.
- Allez sur le repo Github, localisez le fichier qui vous intéresse et cliquez dessus.
- Cliquez sur le bouton “Raw” (vous verrez alors les données csv “brutes”, comme indiqué ci-dessous)
- Copiez l’adresse URL
- Placez l’URL entre guillemets dans la commande R
import()
Fichiers XLSX
Il se peut que vous ne puissiez pas visualiser les données “brutes” pour certains fichiers (e.x. .xlsx, .rds, .nwk, .shp)
- Allez sur le repo Github, localisez le fichier qui vous intéresse et cliquez dessus.
- Cliquez sur le bouton “Download”, comme indiqué ci-dessous
- Sauvegardez le fichier sur votre ordinateur, et importez-le dans R
Shapefiles
Les fichiers Shapefiles comportent de nombreux sous-fichiers, chacun avec une extension de fichier différente. Un fichier aura l’extension “.shp”, mais d’autres peuvent avoir “.dbf”, “.prj”, etc. Pour télécharger un shapefile à partir de Github, vous devrez télécharger chacun des fichiers de sous-composants individuellement, et les enregistrer dans le même dossier sur votre ordinateur. Dans Github, cliquez sur chaque fichier individuellement et téléchargez-les en cliquant sur le bouton ” Download “.
Une fois enregistré sur votre ordinateur, vous pouvez importer le shapefile comme indiqué sur la page Bases de SIG st_read() du paquet sf. Il vous suffit de fournir le chemin d’accès et le nom du fichier ” .shp “, à condition que les fichiers associés se trouvent dans le même dossier sur votre ordinateur.
Ci-dessous, vous pouvez voir comment le shapefile “sle_adm3” se compose de plusieurs fichiers, chacun devant être téléchargé depuis Github.
7.8 Entrée de données de façon manuelle
Entrée par lignes
Utilisez la fonction tribble du paquet tibble du tidyverse, ici un référence tibble en ligne
Notez que les en-têtes de colonne commencent par une tilde (~). Notez également que chaque colonne ne doit contenir qu’une seule classe de données (caractère, numérique, etc.). Vous pouvez utiliser des tabulations, des espaces et de nouvelles lignes pour rendre la saisie des données plus intuitive et plus lisible. Les espaces ne comptent pas entre les valeurs, mais chaque ligne est représentée par une nouvelle ligne de code. Par exemple:
# create the dataset manually by row
manual_entry_rows <- tibble::tribble(
~colA, ~colB,
"a", 1,
"b", 2,
"c", 3
)Et maintenant nous affichons le nouveau jeu de données:
Entrée par colonnes
Étant donné qu’un cadre de données est constitué de vecteurs (colonnes verticales), l’approche de base de la création manuelle de cadres de données dans R prévoit que vous définissiez chaque colonne, puis que vous les reliiez entre elles. Cela peut être contre-intuitif en épidémiologie, car nous pensons généralement à nos données en lignes (comme ci-dessus).
# define each vector (vertical column) separately, each with its own name
PatientID <- c(235, 452, 778, 111)
Treatment <- c("Yes", "No", "Yes", "Yes")
Death <- c(1, 0, 1, 0)ATTENTION: Tous les vecteurs doivent avoir la même longueur (même nombre de valeurs).
Les vecteurs peuvent ensuite être liés entre eux à l’aide de la fonction data.frame():
# combiner les colonnes dans un cadre de données, en référençant les noms de vecteurs
manual_entry_cols <- data.frame(PatientID, Treatment, Death)Et maintenant nous affichons le nouveau jeu de données:
Collage à partir du presse-papiers
Si vous copiez des données d’un autre endroit et que vous les avez dans votre presse-papiers, vous pouvez essayer l’une des deux méthodes ci-dessous :
A partir du package clipr, vous pouvez utiliser read_clip_tbl() pour importer comme un cadre de données, ou simplement read_clip() pour importer comme un vecteur de caractères. Dans les deux cas, laissez les parenthèses vides.
linelist <- clipr::read_clip_tbl() # importe le presse-papiers actuel comme cadre de données
linelist <- clipr::read_clip() # importations en tant que vecteur de caractèresVous pouvez aussi facilement exporter vers le presse-papiers de votre système avec clipr. Voir la section ci-dessous sur l’Exportation.
Alternativement, vous pouvez utiliser la fonction read.table() de base R avec file = "clipboard") pour importer comme un cadre de données:
df_from_clipboard <- read.table(
file = "clipboard", # specify this as "clipboard"
sep = "t", # separator could be tab, or commas, etc.
header=TRUE) # if there is a header row7.9 Importer le fichier le plus récent
Il arrive souvent que vous receviez des mises à jour quotidiennes de vos ensembles de données. Dans ce cas, vous voudrez écrire un code qui importe le fichier le plus récent. Nous présentons ci-dessous deux façons d’aborder cette question:
- Sélection du fichier en fonction de la date figurant dans le nom du fichier
- Sélection du fichier sur la base des métadonnées du fichier (dernière modification)
Dates dans le nom du fichier
Cette approche repose sur trois prémisses:
- Vous faites confiance aux dates dans les noms de fichiers
- Les dates sont numériques et apparaissent généralement dans le même format (par exemple, année, mois, jour)
- Il n’y a pas d’autres chiffres dans le nom du fichier
Nous vous expliquerons chaque étape, puis nous vous les montrerons combinées à la fin.
Tout d’abord, utilisez dir() de base R pour extraire uniquement les noms de fichiers pour chaque fichier dans le dossier qui vous intéresse. Pour plus de détails sur dir(), consultez la page sur Interactions avec les répertoires. Dans cet exemple, le dossier concerné est le dossier ” linelists ” situé dans le dossier ” example ” situé dans ” data ” au sein du projet R.
linelist_filenames <- dir(here("data", "example", "linelists")) # get file names from folder
linelist_filenames # print[1] "20201007linelist.csv" "case_linelist_2020-10-02.csv"
[3] "case_linelist_2020-10-03.csv" "case_linelist_2020-10-04.csv"
[5] "case_linelist_2020-10-05.csv" "case_linelist_2020-10-08.xlsx"
[7] "case_linelist20201006.csv" Une fois que vous avez ce vecteur de noms, vous pouvez en extraire les dates en appliquant str_extract() de stringr en utilisant cette expression régulière. Elle extrait tous les nombres dans le nom de fichier (y compris tout autre caractère au milieu comme les tirets ou les barres obliques). Vous pouvez en savoir plus sur stringr à la page Caractères et chaînes de caractères.
linelist_dates_raw <- stringr::str_extract(linelist_filenames, "[0-9].*[0-9]") # extract numbers and any characters in between
linelist_dates_raw # print[1] "20201007" "2020-10-02" "2020-10-03" "2020-10-04" "2020-10-05"
[6] "2020-10-08" "20201006" En supposant que les dates sont généralement écrites dans le même format (par exemple, Année puis Mois puis Jour) et que les années ont 4 chiffres, vous pouvez utiliser les fonctions de conversion flexibles de lubridate (ymd(), dmy(), ou mdy()) pour les convertir en dates. Pour ces fonctions, les tirets, les espaces ou les barres obliques n’ont pas d’importance, seul l’ordre des chiffres compte. Pour en savoir plus, consultez la page Manipuler les dates.
linelist_dates_clean <- lubridate::ymd(linelist_dates_raw)
linelist_dates_clean[1] "2020-10-07" "2020-10-02" "2020-10-03" "2020-10-04" "2020-10-05"
[6] "2020-10-08" "2020-10-06"La fonction R base which.max() peut alors être utilisée pour retourner la position de l’index (par exemple, 1ère, 2ème, 3ème, …) de la valeur maximale de la date. Le dernier fichier est correctement identifié comme étant le 6ème fichier - “case_linelist_2020-10-08.xlsx”.
index_latest_file <- which.max(linelist_dates_clean)
index_latest_file[1] 6Si nous condensons toutes ces commandes, le code complet pourrait ressembler à ce qui suit. Notez que le . de la dernière ligne est un caractère de remplacement pour l’objet pipé à ce point de la séquence de pipelines. A ce stade, la valeur est simplement le nombre 6. Celui-ci est placé entre doubles crochets pour extraire le 6ème élément du vecteur de noms de fichiers produit par dir().
# Charger les packages
pacman::p_load(
tidyverse, # data management
stringr, # work with strings/characters
lubridate, # work with dates
rio, # import / export
here, # relative file paths
fs) # directory interactions
# extraire le nom de fichier du dernier fichier
latest_file <- dir(here("data", "example", "linelists")) %>% # noms du fichier du sous-dossier "linelists"
str_extract("[0-9].*[0-9]") %>% # extraire les dates (nombres)
ymd() %>% # convertir les nombres en dates (assumant le format année-mois-jour)
which.max() %>% # obtenir l'index de la date maximale (dernier fichier)
dir(here("data", "example", "linelists"))[[.]] # retourne le nom de fichier de la dernière liste de diffusion
latest_file # imprimer le nom du dernier fichier[1] "case_linelist_2020-10-08.xlsx"Vous pouvez maintenant utiliser ce nom pour terminer le chemin de fichier relatif, avec here():
here("data", "example", "linelists", latest_file) Et vous pouvez maintenant importer le dernier fichier:
# import
import(here("data", "example", "linelists", latest_file)) # import Utiliser l’information du fichier
Si vos fichiers n’ont pas de date dans leur nom (ou si vous ne faites pas confiance à ces dates), vous pouvez essayer d’extraire la date de dernière modification à partir des métadonnées du fichier. Utilisez les fonctions du paquet fs pour examiner les informations des métadonnées de chaque fichier, qui comprennent l’heure de dernière modification et le chemin d’accès au fichier.
Ci-dessous, nous fournissons le dossier d’intérêt à la fonction dir_info() de fs. Dans ce cas, le dossier d’intérêt se trouve dans le projet R dans le dossier “data”, le sous-dossier “example”, et son sous-dossier “linelists”. Le résultat est un cadre de données avec une ligne par fichier et des colonnes pour modification_time, path, etc. Vous pouvez voir un exemple visuel de ceci dans la page sur Interactions avec les répertoires.
Nous pouvons trier ce cadre de données de fichiers par la colonne modification_time, et ensuite ne garder que la ligne (fichier) la plus haute/la plus récente avec la fonction base de R head(). Ensuite, nous pouvons extraire le chemin d’accès de ce dernier fichier uniquement avec la fonction dplyr pull() sur la colonne path. Enfin, nous pouvons passer ce chemin de fichier à import(). Le fichier importé est enregistré sous le nom de latest_file.
latest_file <- dir_info(here("data", "example", "linelists")) %>% # collecte des informations sur tous les fichiers du répertoire
arrange(desc(modification_time)) %>% # trier par temps de modification
head(1) %>% # ne conserver que le fichier le plus récent
pull(path) %>% # extraire uniquement le chemin du fichier
import() # importer le fichier7.10 APIs
Une “interface de programmation automatisée” (API) peut être utilisée pour demander directement des données à un site web. Les API sont un ensemble de règles qui permettent à une application logicielle d’interagir avec une autre. Le client (vous) envoie une “requête” et reçoit une “réponse” contenant du contenu. Les packages R httr et jsonlite peuvent faciliter ce processus.
Chaque site Web compatible avec l’API possède sa propre documentation et ses propres spécificités avec lesquelles il faut se familiariser. Certains sites sont accessibles au public et peuvent être consultés par n’importe qui. D’autres, comme les plates-formes avec des identifiants et des références d’utilisateur, nécessitent une authentification pour accéder à leurs données.
Il va sans dire qu’il est nécessaire de disposer d’une connexion Internet pour importer des données via l’API. Nous donnerons brièvement des exemples d’utilisation des API pour importer des données, et nous vous renverrons à d’autres ressources.
Note : rappelons que les données peuvent être affichées* sur un site web sans API, ce qui peut être plus facile à récupérer. Par exemple, un fichier CSV affiché peut être accessible simplement en fournissant l’URL du site à import() comme décrit dans la section sur importation de Github.*
Requête HTTP
L’échange d’API se fait le plus souvent par le biais d’une requête HTTP. HTTP, qui signifie Hypertext Transfer Protocol, est le format sous-jacent d’une demande/réponse entre un client et un serveur. Les données d’entrée et de sortie exactes peuvent varier en fonction du type d’API, mais le processus est le même : une “demande” (souvent une requête HTTP) de la part de l’utilisateur, contenant souvent une requête, suivie d’une “réponse”, contenant des informations d’état sur la demande et éventuellement le contenu demandé.
Voici quelques éléments d’une requête HTTP:
- L’URL du point de terminaison de l’API
- La “Méthode” (ou “Verbe”)
- En-tête
- Corps
La “méthode” de la requête HTTP est l’action que vous voulez effectuer. Les deux méthodes HTTP les plus courantes sont GET et POST mais d’autres peuvent inclure PUT, DELETE, PATCH, etc. Lorsque vous importez des données dans R, il est très probable que vous utilisiez la méthode GET.
Après votre requête, votre ordinateur recevra une “réponse” dans un format similaire à celui que vous avez envoyé, comprenant l’URL, l’état HTTP (l’état 200 est ce que vous voulez !), le type de fichier, la taille et le contenu souhaité. Vous devrez ensuite analyser cette réponse et la transformer en un cadre de données exploitable dans votre environnement R.
Packages
Le package httr fonctionne bien pour traiter les requêtes HTTP dans R. Il nécessite peu de connaissances préalables sur les API Web et peut être utilisé par des personnes moins familières avec la terminologie du développement logiciel. En outre, si la réponse HTTP est un fichier .json, vous pouvez utiliser jsonlite pour analyser la réponse.
# load packages
pacman::p_load(httr, jsonlite, tidyverse)Données disponibles au public
Voici un exemple de requête HTTP, emprunté à un tutoriel du site the Trafford Data Lab. Ce site propose plusieurs autres ressources pour apprendre et des exercices API.
Scénario : Nous souhaitons importer une liste des établissements de restauration rapide de la ville de Trafford, au Royaume-Uni. Les données sont accessibles à partir de l’API de la Food Standards Agency, qui fournit des données sur l’évaluation de l’hygiène alimentaire au Royaume-Uni.
Voici les paramètres de notre requête :
- Verbe HTTP: GET
- URL du point d’accès à l’API: http://api.ratings.food.gov.uk/Establishments
- Paramètres sélectionnés: name, address, longitude, latitude, businessTypeId, ratingKey, localAuthorityId
- En-tête: “x-api-version”, 2
- Format(s) des données: JSON, XML
- Documentation: http://api.ratings.food.gov.uk/help
Le code R serait le suivant:
# préparer la requête
path <- "http://api.ratings.food.gov.uk/Establishments"
request <- GET(url = path,
query = list(
localAuthorityId = 188,
BusinessTypeId = 7844,
pageNumber = 1,
pageSize = 5000),
add_headers("x-api-version" = "2"))
# Vérifier s'il y a une erreur de serveur ("200" est bon !)
request$status_code
# soumettre la requête, analyser la réponse et la convertir en un cadre de données
response <- content(request, as = "text", encoding = "UTF-8") %>%
fromJSON(flatten = TRUE) %>%
pluck("establishments") %>%
as_tibble()Vous pouvez maintenant nettoyer et utiliser le cadre de données response, qui contient une ligne par établissement de restauration rapide.
Authentification requise
Certaines API nécessitent une authentification, c’est-à-dire que vous devez prouver votre identité pour pouvoir accéder à des données restreintes. Pour importer ces données, vous devrez peut-être d’abord utiliser une méthode POST pour fournir un nom d’utilisateur, un mot de passe ou un code. Vous obtiendrez alors un jeton d’accès, qui pourra être utilisé pour les demandes ultérieures par la méthode GET afin de récupérer les données souhaitées.
Vous trouverez ci-dessous un exemple d’interrogation de données à partir de Go.Data, qui est un outil d’investigation des épidémies. Go.Data utilise une API pour toutes les interactions entre le frontal web et les applications pour smartphones utilisées pour la collecte des données. Go.Data est utilisé dans le monde entier. Comme les données sur les épidémies sont sensibles et que vous ne devez pouvoir accéder qu’aux données concernant votre épidémie, une authentification est requise.
Vous trouverez ci-dessous un exemple de code R utilisant httr et jsonlite pour se connecter à l’API Go.Data afin d’importer des données sur le suivi des contacts de votre épidémie.
# définir les informations d'identification pour l'autorisation
url <- "https://godatasampleURL.int/" # url d'instance valide de Go.Data
username <- "username" # nom d'utilisateur valide Go.Data
password <- "password" # mot de passe Go,Data valide
outbreak_id <- "xxxxxx-xxxx-xxxx-xxxx-xxxxxxx" # identifiant d'épidémie Go.Data valide
# obtenir le jeton d'accès
url_request <- paste0(url,"api/oauth/token?access_token=123") # define base URL request
# préparer la requête
response <- POST(
url = url_request,
body = list(
username = username, # utiliser le nom d'utilisateur et le mot de passe enregistrés ci-dessus pour l'autorisation
password = password),
encode = "json")
# exécuter la demande et analyser la réponse
content <-
content(response, as = "text") %>%
fromJSON(flatten = TRUE) %>% # aplatir les JSON imbriqués
glimpse()
# Sauvegarder le jeton d'accès de la réponse
access_token <- content$access_token # sauvegarder le jeton d'accès pour permettre les appels API suivants
# importer les contacts de l'épidémie
# Utiliser le jeton d'accès
response_contacts <- GET(
paste0(url,"api/outbreaks/",outbreak_id,"/contacts"), # obtenir (GET) la requête
add_headers(
Authorization = paste("Bearer", access_token, sep = " ")))
json_contacts <- content(response_contacts, as = "text") # convertir en texte JSON
contacts <- as_tibble(fromJSON(json_contacts, flatten = TRUE)) # aplatir JSON en tibbleATTENTION: Si vous importez de grandes quantités de données à partir d’une API nécessitant une authentification, il se peut que le délai d’attente soit dépassé. Pour éviter cela, récupérez à nouveau l’access_token avant chaque requête GET de l’API et essayez d’utiliser des filtres ou des limites dans la requête.
Pour plus de détails, consultez la documentation sur LoopBack Explorer, la page Suivi des contacts ou les astuces API sur Go.Data Github repository
Vous pouvez en savoir plus sur le package httr here
Cette section a aussi été informée par ce tutoriel et ce tutoriel.
7.11 Exporter
Avec le package rio
Avec rio, vous pouvez utiliser la fonction export() de manière très similaire à import(). Donnez d’abord le nom de l’objet R que vous voulez sauvegarder (par exemple linelist) et ensuite entre guillemets mettez le chemin du fichier où vous voulez sauvegarder le fichier, en incluant le nom de fichier désiré et l’extension de fichier. Par exemple :
Cette opération permet de sauvegarder le cadre de données linelist comme un classeur Excel dans le répertoire de travail/ dossier racine du projet :
export(linelist, "my_linelist.xlsx") # will save to working directoryVous pouvez enregistrer le même cadre de données comme un fichier csv en changeant l’extension. Par exemple, nous l’enregistrons également dans un chemin de fichier construit avec here() :
export(linelist, here("data", "clean", "my_linelist.csv"))Vers le presse-papiers
Pour exporter un cadre de données vers le “presse-papiers” de votre ordinateur (pour ensuite le coller dans un autre logiciel comme Excel, Google Spreadsheets, etc.) vous pouvez utiliser write_clip() du paquet clipr.
# exporter le cadre de données de la liste de cas vers le presse-papiers de votre système
clipr::write_clip(linelist)7.12 Documents RDS
Outre les fichiers .csv, .xlsx, etc., vous pouvez également exporter/enregistrer des cadres de données R sous forme de fichiers .rds. Il s’agit d’un format de fichier spécifique à R, très utile si vous savez que vous allez retravailler les données exportées dans R.
Les classes de colonnes sont stockées, de sorte que vous n’avez pas à refaire le nettoyage lors de l’importation (avec un fichier Excel ou même un fichier CSV, cela peut être un casse-tête !) C’est aussi un fichier plus petit, ce qui est utile pour l’exportation et l’importation si votre ensemble de données est grand.
Par exemple, si vous travaillez dans une équipe d’épidémiologie et que vous devez envoyer des fichiers à une équipe SIG pour la cartographie, et qu’ils utilisent également R, envoyez-leur simplement le fichier .rds ! Toutes les classes de colonnes sont alors conservées et ils ont moins de travail à faire.
export(linelist, here("data", "clean", "my_linelist.rds"))7.13 Fichiers Rdata et listes
Les fichiers .Rdata peuvent stocker plusieurs objets R - par exemple plusieurs cadres de données, des résultats de modèles, des listes, etc. Cela peut être très utile pour consolider ou partager un grand nombre de vos données pour un projet donné.
Dans l’exemple ci-dessous, plusieurs objets R sont stockés dans le fichier exporté “my_objects.Rdata”:
rio::export(my_list, my_dataframe, my_vector, "my_objects.Rdata")Note : si vous essayez d’importer une liste, utilisez import_list() de rio pour l’importer avec la sTIPture et le contenu originaux complets.
rio::import_list("my_list.Rdata")7.14 Sauvegarde des graphiques
Les insTIPtions sur la façon de sauvegarder les tracés, tels que ceux créés par ggplot(), sont discutées en profondeur dans la page bases de ggplot.
En bref, lancez ggsave("my_plot_filepath_and_name.png") après avoir imprimé votre tracé. Vous pouvez soit fournir un objet de tracé sauvegardé à l’argument plot =, ou seulement spécifier le chemin du fichier de destination (avec l’extension du fichier) pour sauvegarder le tracé le plus récemment affiché. Vous pouvez aussi contrôler le width =, height =, units =, et dpi =.
L’enregistrement d’un graphe de réseau, tel qu’un arbre de transmission, est abordé dans la page Chaînes de transmission.
7.15 Ressources
Le Manuel pour Importer/Exporter
Chapitre sur l’importaiton de données de R 4 Data Science
Documation pour ggsave()
Voici un tableau tiré de la vignette en ligne de rio. Pour chaque type de données, il indique : l’extension de fichier attendue, le package que rio utilise pour importer ou exporter les données, et si cette fonctionnalité est incluse dans la version installée par défaut de rio.
| Format | Extension Typique | Package d’Importation | Package d’Exportation | Installé par Defaut |
|---|---|---|---|---|
| Comma-separated data | .csv | data.table fread() |
data.table | Oui |
| Pipe-separated data | .psv | data.table fread() |
data.table | Oui |
| Tab-separated data | .tsv | data.table fread() |
data.table | Oui |
| SAS | .sas7bdat | haven | haven | Oui |
| SPSS | .sav | haven | haven | Oui |
| Stata | .dta | haven | haven | Oui |
| SAS | XPORT | .xpt | haven | Oui |
| SPSS Portable | .por | haven | Oui | |
| Excel | .xls | readxl | Oui | |
| Excel | .xlsx | readxl | openxlsx | Oui |
| R syntax | .R | base | base | Oui |
| Saved R objects | .RData, .rda | base | base | Oui |
| Serialized R objects | .rds | base | base | Oui |
| Epiinfo | .rec | foreign | Oui | |
| Minitab | .mtp | foreign | Oui | |
| Systat | .syd | foreign | Oui | |
| “XBASE” | database files | .dbf | foreign | Oui |
| Weka Attribute-Relation File Format | .arff | foreign | foreign | Oui |
| Data Interchange Format | .dif | utils | Oui | |
| Fortran data | pas d’extension reconnue | utils | Oui | |
| Fixed-width format data | .fwf | utils | utils | Oui |
| gzip comma-separated data | .csv.gz | utils | utils | Oui |
| CSVY (CSV + YAML metadata header) | .csvy | csvy | csvy | Non |
| EViews | .wf1 | hexView | Non | |
| Feather R/Python interchange format | .feather | feather | feather | Non |
| Fast Storage | .fst | fst | fst | Non |
| JSON | .json | jsonlite | jsonlite | Non |
| Matlab | .mat | rmatio | rmatio | Non |
| OpenDocument Spreadsheet | .ods | readODS | readODS | Non |
| HTML Tables | .html | xml2 | xml2 | Non |
| Shallow XML documents | .xml | xml2 | xml2 | Non |
| YAML | .yml | yaml | yaml | Non |
| Clipboard default is tsv | clipr | clipr | Non |