12 Restructurer les données
Dans le contexte de la gestion des données, le pivot des données fait référence à l’un des deux processus suivants :
La création de tableaux croisés, qui sont des tableaux de statistiques résumant les données d’un tableau plus étendu.
La restructuration d’un tableau du format long au format large, ou vice versa.
Dans ce chapitre, nous allons nous focaliser sur le second processus. Résumer ses données dans des tableau est une étape cruciale de l’analyse des données et est traitée dans les chapitres sur le regroupement des données et les tableaux descriptifs.
Ce chapitre traite des formats de données. Il est utile de connaître l’idée de “données bien rangées / ordonnées”, dans laquelle chaque variable a sa propre colonne, chaque observation a sa propre ligne et chaque valeur a sa propre cellule. Vous trouverez plus d’informations sur ce sujet dans ce chapitre en ligne de R for Data Science(en Anglais).
12.1 Étapes préliminaires
Importation des paquets
Ces lignes de code chargent les paquets nécessaires aux analyses. Dans ce guide, nous mettons l’accent sur p_load() de pacman, qui installe le paquet si nécessaire puis l’importe pour l’utiliser. Vous pouvez également charger les paquets installés avec library() de base R. Voir la page sur bases de R pour plus d’informations sur les paquets R.
pacman::p_load(
rio, # import des fichiers
here, # gestion des chemins d'accès
kableExtra,
tidyverse) # gestion des données + graphiques (ggplot2)Importation des données
Cas de Malaria
Dans ce chapitre, nous utiliserons un jeu de données fictif de cas quotidiens de paludisme, par établissement et par groupe d’âge. Pour reproduire les étapes, cliquez ici pour télécharger les données (en tant que fichier .rds). Ou importez des données avec la fonction import() du paquet rio (elle gère de nombreux types de fichiers comme .xlsx, .csv, .rds - voir la page Importation et exportation des données pour plus de détails).
# Importation des données
count_data <- import("malaria_facility_count_data.rds")Les premières cinquantes lignes sont affichées ci-dessous.
Linelist des cas
A la fin de ce chapitre, nous utiliserons également une liste des cas d’une épidémie d’Ebola simulée. Pour reproduire les étapes, cliquez pour télécharger la linelist “propre” (en tant que fichier .rds). Importez vos données avec la fonction import() du paquet rio (elle accepte de nombreux types de fichiers comme .xlsx, .rds, .csv - voir la page Importation et exportation des données pour plus de détails).
# Importer la linelist
linelist <- import("linelist_cleaned.xlsx")12.2 Transformation du format large vers long
Le format “large”
Les données sont souvent saisies et stockées dans un format “large” (ou “étendu”), où les caractéristiques ou les réponses d’un sujet/d’un item sont entrées dans une même ligne. Cette structure de données est utile pour la saisie et la présentation des données, mais elle n’est pas appropriée pour de nombreuses analyses.
Par exemple, dans le jeu de données count_data importé auparavant, chaque ligne représente un établissement à une date donnée. Les nombres de cas sont contenus dans les colonnes les plus à droites, avec une colonne par classe d’age, et une colonne pour le nombre total de cas ce jour là dans cet établissement. L’information “nombre de cas” est donc contenues sur plusieurs colonnes, au lieu d’une seule, d’où la structure dite “large”.
Plus précisément, chaque ligne dans ce tableau contient le nombre de cas de paludisme dans l’un des 65 établissements à une date donnée, dans la période allant de count_data$data_date %>% min() à count_data$data_date %>% max(). Ces établissements sont situés dans une province (Nord) et quatre districts (Spring, Bolo, Dingo, et Barnard). Le dataframe contient les totaux des cas de paludisme, globaux et pour chaque classe d’age (<4 ans, 5-14 ans, et 15 ans et plus).
Les données sous format “large” comme celle-ci ne respectent pas les normes de “données rangées”, car les en-têtes de colonne ne représentent pas réellement des “variables”: ils contiennent les valeurs d’une hypothétique variable “groupe d’âge”.
Ce format est utile pour présenter les informations dans un tableau, ou pour saisir des données (dans Excel par exemple) à partir de formulaires de notification des cas. Cependant, au stade de l’analyse, ces données doivent généralement être restructurées et rangées en un format plus long. Le paquet de visualisations ggplot2, fonctionne également mieux lorsque les données sont dans un format “long”.
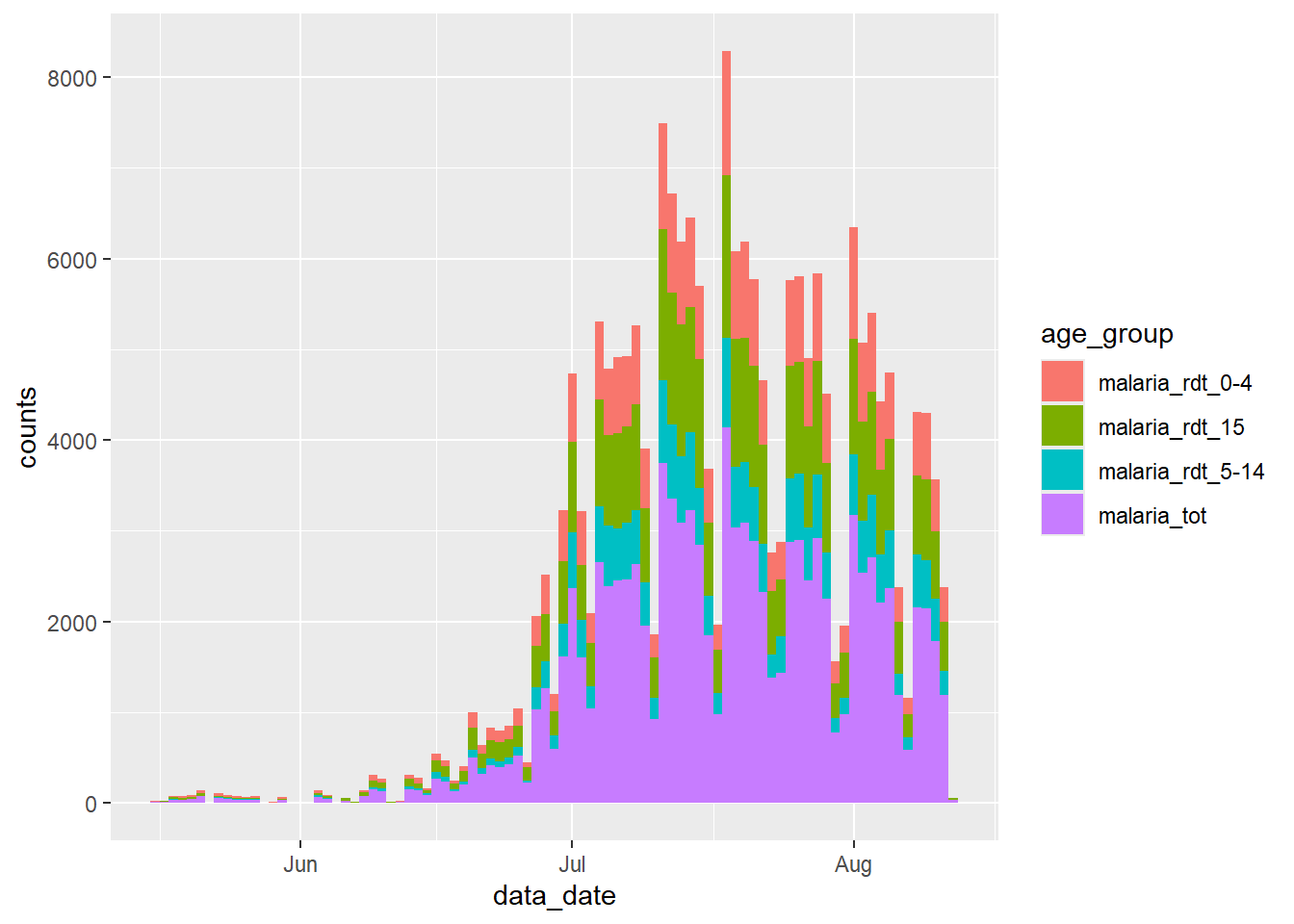
La visualisation du nombre total de cas de paludisme dans le temps ne pose aucun problème avec les données dans leur format actuel :
Cependant, les choses se compliquent si l’on veut visualiser les contributions relatives de chaque groupe d’âge au total des cas ? Nous devons alors nous assurer que la variable d’intérêt (le groupe d’âge) ait sa propre colonne dans le dataframe, colonne qui peut être passée à l’argument “mapping aesthetics” aes() de {ggplot2}.
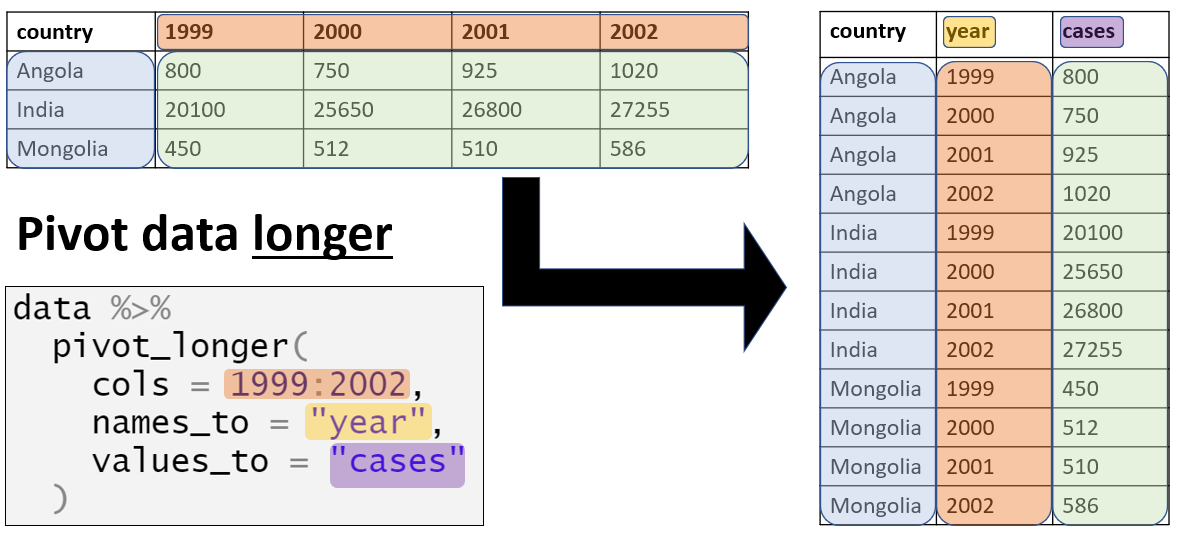
pivot_longer()
La fonction pivot_longer() de tidyr transforme un jeu de données au format “large” en un jeu de données “plus long”. tidyr fait partie du méta-paquet tidyverse.
Elle accepte une ou plusieurs colonnes à transformer (argument cols =), ce qui donne un contrôle fin sur les colonnes à restructurer. Par exemple, pour les données sur le paludisme, nous ne voulons faire pivoter que les colonnes contenant des nombre de cas.
Suite à ce processus, nous obtenons deux “nouvelles” colonnes: l’une contenant les catégories (anciennement sotckées dans les noms de colonnes), et l’autre avec les valeurs correspondantes (ici, le nombre de cas). Nous pouvons accepter les noms par défaut pour ces nouvelles colonnes, ou spécifier de nouveaux noms dans names_to = et values_to = respectivement.
Voyons comment utiliser pivot_longer()…
Transformation simple
Nous utilisons la fonction pivot_longer() de tidyr pour convertir les données d’un format “large” à un format “long”. Plus précisément, il s’agit de convertir les quatre colonnes numériques contenant des nombre de cas de paludisme en deux nouvelles colonnes : une qui contient les groupes d’âge et une qui contient les valeurs correspondantes.
df_long <- count_data %>%
pivot_longer(
cols = c(`malaria_rdt_0-4`, `malaria_rdt_5-14`,
`malaria_rdt_15`, `malaria_tot`)
)
df_longNotons que le dataframe nouvellement crée (df_long) a plus de lignes (12 152 contre 3 038) : il est devenu plus long. Pour être précis, il est quatre fois plus long, car chaque ligne du tableau d’origine a donné quatre lignes dans `df_long``, une pour chacun des colonnes restructurées (<4 ans, 5-14 ans, 15 ans et plus, et total).
Le nouveau tableau a également moins de colonnes (8 contre 10), car les données précédemment stockées dans quatre colonnes (celles qui commencent par le préfixe malaria_) sont maintenant stockées dans deux colonnes.
Note : puisque les noms de quatre colonnes transformées commencent tous par le préfixe malaria_, nous aurions pu sélectionner les colonnes à transformer en utilisant la fonction starts_with() pour obtenir le même résultat (voir la page sur le nettoyage des données et les fonctions de base pour plus de ces fonctions d’aide de type “tidyselect”).
# choisir les colonnes avec l'aide d'une fonction "tidyselect"
count_data %>%
pivot_longer(
cols = starts_with("malaria_")
)# A tibble: 12,152 × 8
location_name data_date submitted_date Province District newid name value
<chr> <date> <date> <chr> <chr> <int> <chr> <int>
1 Facility 1 2020-08-11 2020-08-12 North Spring 1 malari… 11
2 Facility 1 2020-08-11 2020-08-12 North Spring 1 malari… 12
3 Facility 1 2020-08-11 2020-08-12 North Spring 1 malari… 23
4 Facility 1 2020-08-11 2020-08-12 North Spring 1 malari… 46
5 Facility 2 2020-08-11 2020-08-12 North Bolo 2 malari… 11
6 Facility 2 2020-08-11 2020-08-12 North Bolo 2 malari… 10
7 Facility 2 2020-08-11 2020-08-12 North Bolo 2 malari… 5
8 Facility 2 2020-08-11 2020-08-12 North Bolo 2 malari… 26
9 Facility 3 2020-08-11 2020-08-12 North Dingo 3 malari… 8
10 Facility 3 2020-08-11 2020-08-12 North Dingo 3 malari… 5
# ℹ 12,142 more rowsou par position :
# Choisir les colonnes à partir de leur position dans le tableau
count_data %>%
pivot_longer(
cols = 6:9
)ou dans le cas de colonnes consécutives, avec la première et la dernière colonne :
# Choisir les colonnes avec un "intervalle"
count_data %>%
pivot_longer(
cols = `malaria_rdt_0-4`:malaria_tot
)Les deux nouvelles colonnes crées lors de la restructuration reçoivent les noms par défaut de name et value, mais nous pouvons remplacer ces valeurs par défaut par des noms qui décrivent mieux le contenu des colonnes en utilisant les arguments names_to et values_to. Par exemple, si nous voulons renommer les colonnes age_group et counts :
df_long <- count_data %>%
pivot_longer(
cols = starts_with("malaria_"),
names_to = "age_group",
values_to = "counts"
)
df_long# A tibble: 12,152 × 8
location_name data_date submitted_date Province District newid age_group
<chr> <date> <date> <chr> <chr> <int> <chr>
1 Facility 1 2020-08-11 2020-08-12 North Spring 1 malaria_rdt_…
2 Facility 1 2020-08-11 2020-08-12 North Spring 1 malaria_rdt_…
3 Facility 1 2020-08-11 2020-08-12 North Spring 1 malaria_rdt_…
4 Facility 1 2020-08-11 2020-08-12 North Spring 1 malaria_tot
5 Facility 2 2020-08-11 2020-08-12 North Bolo 2 malaria_rdt_…
6 Facility 2 2020-08-11 2020-08-12 North Bolo 2 malaria_rdt_…
7 Facility 2 2020-08-11 2020-08-12 North Bolo 2 malaria_rdt_…
8 Facility 2 2020-08-11 2020-08-12 North Bolo 2 malaria_tot
9 Facility 3 2020-08-11 2020-08-12 North Dingo 3 malaria_rdt_…
10 Facility 3 2020-08-11 2020-08-12 North Dingo 3 malaria_rdt_…
# ℹ 12,142 more rows
# ℹ 1 more variable: counts <int>Nous pouvons maintenant passer ce nouveau jeu de données à {ggplot2}, et placer la nouvelle colonne count dans l’axe des y et colorer les barres en fonction des valeurs de la colonne age_group grâce à l’argument fill =. Nous obtenons alors un diagramme en bâtons des cas de paludisme par groupe d’âge :
ggplot(data = df_long) +
geom_col(
mapping = aes(x = data_date,
y = counts,
fill = age_group),
width = 1
)Examinez ce nouveau tracé et comparez-le avec le tracé que nous avons créé précédemment : qu’est-ce qui cloche ?
Nous fait une erreur classique du traitement des données de surveillance et inclus le nombre de cas totaux de la colonne malaria_tot. La conséquence est que chaque barre du graphique est deux fois plus élevée qu’elle ne devrait l’être.
Nous pouvons résoudre ce problème de plusieurs façons. Tout d’abord nous pouvons simplement filtrer les données avant de les passer à ggplot() :
df_long %>%
filter(age_group != "malaria_tot") %>%
ggplot() +
geom_col(
aes(x = data_date,
y = counts,
fill = age_group),
width = 1
)Autrement, nous aurions pu exclure cette variable lors du pivot_longer(), la conservant comme une variable séparée dans le tableau :
count_data %>%
pivot_longer(
cols = `malaria_rdt_0-4`:malaria_rdt_15, # does not include the totals column
names_to = "age_group",
values_to = "counts"
)# A tibble: 9,114 × 9
location_name data_date submitted_date Province District malaria_tot newid
<chr> <date> <date> <chr> <chr> <int> <int>
1 Facility 1 2020-08-11 2020-08-12 North Spring 46 1
2 Facility 1 2020-08-11 2020-08-12 North Spring 46 1
3 Facility 1 2020-08-11 2020-08-12 North Spring 46 1
4 Facility 2 2020-08-11 2020-08-12 North Bolo 26 2
5 Facility 2 2020-08-11 2020-08-12 North Bolo 26 2
6 Facility 2 2020-08-11 2020-08-12 North Bolo 26 2
7 Facility 3 2020-08-11 2020-08-12 North Dingo 18 3
8 Facility 3 2020-08-11 2020-08-12 North Dingo 18 3
9 Facility 3 2020-08-11 2020-08-12 North Dingo 18 3
10 Facility 4 2020-08-11 2020-08-12 North Bolo 49 4
# ℹ 9,104 more rows
# ℹ 2 more variables: age_group <chr>, counts <int>Les valeurs sont alors répétées dans les lignes des groupes d’age.
Transformer les données de plusieurs classes
L’exemple ci-dessus fonctionne bien dans les situations où toutes les colonnes que l’on veut faire pivoter sont de la même classe (chaîne de caractère, numérique, logique…).
Cependant, en tant qu’épidémiologiste de terrain, vous serez amené à travailler avec des données qui ont été préparées par des non-spécialistes, appliquant leur propre logique. Cela aboutit parfois à des jeux de données non-standard, voire totalement désorganisés. Comme Hadley Wickham l’a noté (en faisant référence à Tolstoï) dans son article séminal sur les principes des données rangées, organisées (tidy data) :
Comme les familles, les tableaux de données rangés et organisés se ressemblent tous, mais chaque fichier de données désorganisé / mal rangé l’est à sa manière.
Un problème particulièrement courant est la nécessité de restructurer des colonnes qui contiennent différentes types de données. Cette transformation aurait pour conséquence de stocker différents types de données dans une seule colonne, ce qui est déconseillé. Il y a plusieurs manière de gérer les problèmes associés à ce type de données mais la restructuration avec pivot_longer() est une étape importante.
Imaginons cette situation : une série d’observations a été effectuée à différents pas de temps pour chacun des trois éléments A, B et C. Il peut s’agir d’individus (par exemple, les contacts d’un cas d’Ebola sont suivis chaque jour pendant 21 jours) ou de postes de santé de villages éloignés qui sont contrôlés une fois par an pour s’assurer qu’ils sont toujours fonctionnels. Reprenons l’exemple de la recherche des contacts. Imaginons que les données soient stockées comme suit :
Le format de ces données est un peu plus compliqué que dans l’exemple précédent : chaque ligne stocke des informations sur un élément, et des paires de colonnes contiennent des séries d’observations à différentes dates. Le fichier s’allonge avec de nouvelles colonnes à droite au fur et à mesure des observations. Les classes de colonnes alternent entre dates et chaînes de caractères.
Pour la petite histoire, un des pires exemples de ce type de données qu’il m’ait été donné de rencontrer concernait des données de surveillance du choléra, dans lesquelles 8 nouvelles colonnes d’observations étaient ajoutées chaque jour, pendant 4 ans. L’ouverture du fichier Excel sur mon ordinateur portable a pris plus de dix minutes…
Pour travailler avec ces données, nous devons les transformer en format “long” tout en gardant la séparation entre une colonne date et une colonne caractère (statut), pour chaque observation pour chaque élément. Ceci afin d’éviter de nous retrouver avec un mélange de types de variables dans une seule colonne, une situation que vous devrez chercher à éviter à tout prix dans votre gestion de données, en particulier avec des données ordonnées.
C’est malheureusement ce qui se produit si l’on effectue une transformation simple :
df %>%
pivot_longer(
cols = -id,
names_to = c("observation")
)# A tibble: 18 × 3
id observation value
<chr> <chr> <chr>
1 A obs1_date 2021-04-23
2 A obs1_status Healthy
3 A obs2_date 2021-04-24
4 A obs2_status Healthy
5 A obs3_date 2021-04-25
6 A obs3_status Unwell
7 B obs1_date 2021-04-23
8 B obs1_status Healthy
9 B obs2_date 2021-04-24
10 B obs2_status Healthy
11 B obs3_date 2021-04-25
12 B obs3_status Healthy
13 C obs1_date 2021-04-23
14 C obs1_status Missing
15 C obs2_date 2021-04-24
16 C obs2_status Healthy
17 C obs3_date 2021-04-25
18 C obs3_status Healthy Ci-dessus, notre restructuration a fusionné dates et caractères en une seule colonne valeur. Face à deux colonnes de classes différentes, la fonction convertit par défaut la colonne entière en chaîne de caractères.
Pour éviter cette situation, nous pouvons tirer parti de la structure des noms de colonnes dans le tableau original, qui respectent le même format : le numéro de l’observation, un “_” puis soit “statut” soit “date”.
Pour cela, il nous faut :
fournir un vecteur de chaîne de caractères un peu spécial à l’argument
names_to =. Dans ce vecteur, le second élément est".value", ce terme spécial indiquant que les colonnes restructurées seront divisées sur la base d’un mot dans le nom des colonnes.fournir le caractère utilisé comme séparateur à l’argument
names_sep =. Dans le cas présent, il s’agit du tiret-bas “_“.
Ainsi, le nommage et la division des nouvelles colonnes sont basés sur le “_” dans les noms des variables existantes.
df_long <-
df %>%
pivot_longer(
cols = -id,
names_to = c("observation", ".value"),
names_sep = "_"
)
df_long# A tibble: 9 × 4
id observation date status
<chr> <chr> <chr> <chr>
1 A obs1 2021-04-23 Healthy
2 A obs2 2021-04-24 Healthy
3 A obs3 2021-04-25 Unwell
4 B obs1 2021-04-23 Healthy
5 B obs2 2021-04-24 Healthy
6 B obs3 2021-04-25 Healthy
7 C obs1 2021-04-23 Missing
8 C obs2 2021-04-24 Healthy
9 C obs3 2021-04-25 HealthyDerniers détails :
La colonne date est actuellement sous la forme d’une chaîne de caractères mais nous pouvons facilement la convertir en classe date en utilisant les fonctions mutate() et as_date() décrites dans la page travailler avec des dates.
Nous pouvons aussi améliorer la colonne observation en supprimant le préfixe “obs” et en la convertissant en format numérique. Nous pouvons le faire avec str_remove_all() du paquet stringr (voir la page sur les chaînes de caractères).
df_long <-
df_long %>%
mutate(
date = date %>% lubridate::as_date(),
observation =
observation %>%
str_remove_all("obs") %>%
as.numeric()
)
df_long# A tibble: 9 × 4
id observation date status
<chr> <dbl> <date> <chr>
1 A 1 2021-04-23 Healthy
2 A 2 2021-04-24 Healthy
3 A 3 2021-04-25 Unwell
4 B 1 2021-04-23 Healthy
5 B 2 2021-04-24 Healthy
6 B 3 2021-04-25 Healthy
7 C 1 2021-04-23 Missing
8 C 2 2021-04-24 Healthy
9 C 3 2021-04-25 HealthyNous pouvons maintenant travailler avec les données dans ce format allongé. par exemple, en créant une carte de chaleur :
12.3 Transformation du format long en large
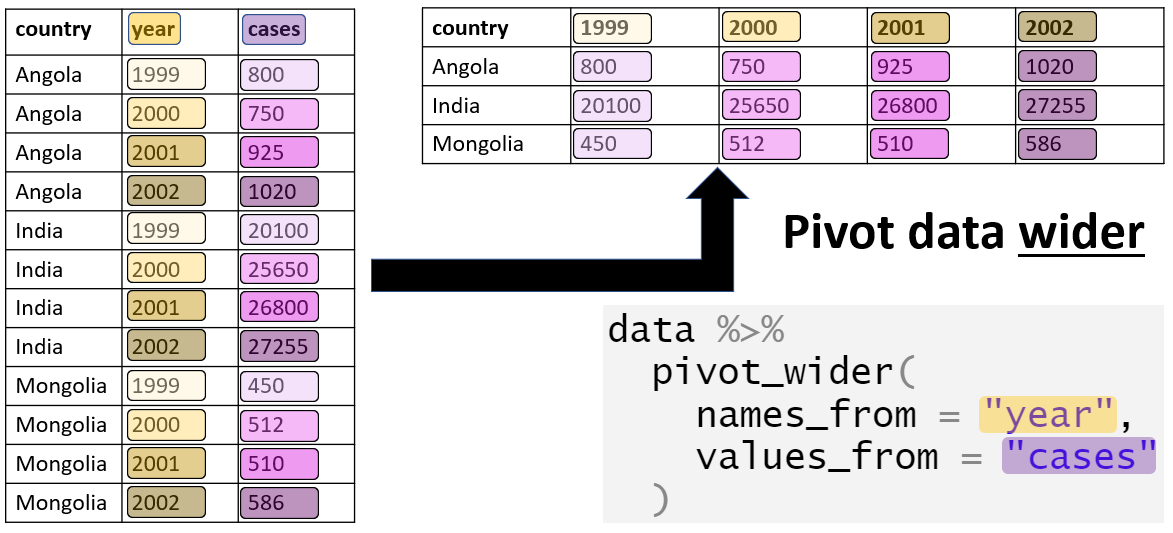
Il peut être utile de transformer un jeu de données d’un format “long” en un format plus large à l’aide de la fonction pivot_wider().
Un cas d’utilisation typique est lorsque qu’il faut transformer les résultats d’une analyse dans un format plus digeste pour le lecteur, tel qu’un tableau résumé. En général, il s’agit de transformer un dataframe dans lequel les informations relatives à un sujet sont réparties sur plusieurs lignes en un format dans lequel ces informations sont stockées sur une seule ligne.
Données utilisées
Pour cette section nous utiliserons la liste des cas (voir la section Etapes préliminaires), qui contient une ligne par cas.
Voici les 50 premières lignes :
Imaginons que nous voulions voir les nombres d’individus dans les différentes classes d’age, par genre :
df_wide <-
linelist %>%
count(age_cat, gender)
df_wide age_cat gender n
1 0-4 f 640
2 0-4 m 416
3 0-4 <NA> 39
4 5-9 f 641
5 5-9 m 412
6 5-9 <NA> 42
7 10-14 f 518
8 10-14 m 383
9 10-14 <NA> 40
10 15-19 f 359
11 15-19 m 364
12 15-19 <NA> 20
13 20-29 f 468
14 20-29 m 575
15 20-29 <NA> 30
16 30-49 f 179
17 30-49 m 557
18 30-49 <NA> 18
19 50-69 f 2
20 50-69 m 91
21 50-69 <NA> 2
22 70+ m 5
23 70+ <NA> 1
24 <NA> <NA> 86Cette commande renvoi un dataframe en format long, qui est très adapté à la création de graphiques avec ggplot2, mais moins idéal pour un tableau résumé dans un rapport :
Nous pouvons utiliser la fonction pivot_wider() pour restructurer les données dans un format plus lisible par un lecteur humain.
pivot_wider()
L’argument names_from spécifie la colonne depuis laquelle générer les nouveaux noms de colonne, tandis que l’argument values_from spécifie la colonne depuis laquelle prendre les valeurs pour remplir les cellules. L’argument id_cols = est facultatif, mais peut servir à fournir un vecteur de noms de colonnes qui ne doivent pas être pivotées, et qui serviront d’identifiant pour chaque ligne.
table_wide <-
df_wide %>%
pivot_wider(
id_cols = age_cat,
names_from = gender,
values_from = n
)
table_wide# A tibble: 9 × 4
age_cat f m `NA`
<fct> <int> <int> <int>
1 0-4 640 416 39
2 5-9 641 412 42
3 10-14 518 383 40
4 15-19 359 364 20
5 20-29 468 575 30
6 30-49 179 557 18
7 50-69 2 91 2
8 70+ NA 5 1
9 <NA> NA NA 86Ce tableau est beaucoup plus facile à lire, et est une base pour créer des tableaux résumés dans des rapports et articles. Nous pouvons améliorer son apparence à l’aide de paquets tels que flextable et knitr (voir la page Tableaux pour la présentation.
table_wide %>%
janitor::adorn_totals(c("row", "col")) %>% # adds row and column totals
knitr::kable() %>%
kableExtra::row_spec(row = 10, bold = TRUE) %>%
kableExtra::column_spec(column = 5, bold = TRUE) | age_cat | f | m | NA | Total |
|---|---|---|---|---|
| 0-4 | 640 | 416 | 39 | 1095 |
| 5-9 | 641 | 412 | 42 | 1095 |
| 10-14 | 518 | 383 | 40 | 941 |
| 15-19 | 359 | 364 | 20 | 743 |
| 20-29 | 468 | 575 | 30 | 1073 |
| 30-49 | 179 | 557 | 18 | 754 |
| 50-69 | 2 | 91 | 2 | 95 |
| 70+ | NA | 5 | 1 | 6 |
| NA | NA | NA | 86 | 86 |
| Total | 2807 | 2803 | 278 | 5888 |
12.4 Remplissage des colonnes
Parfois, après un pivot, ou plus fréquemment après un bind, nous nous retrouvons avec des vides dans certaines cellules que nous aimerions remplir.
Données
Par exemple, prenons deux jeux de données qui contiennent tout deux des observations pour le numéro de mesure, le nom de l’établissement et le nombre de cas à ce moment-là. En plus de cela, le deuxième jeu de données a également une variable Year.
df1 <-
tibble::tribble(
~Measurement, ~Facility, ~Cases,
1, "Hosp 1", 66,
2, "Hosp 1", 26,
3, "Hosp 1", 8,
1, "Hosp 2", 71,
2, "Hosp 2", 62,
3, "Hosp 2", 70,
1, "Hosp 3", 47,
2, "Hosp 3", 70,
3, "Hosp 3", 38,
)
df1 # A tibble: 9 × 3
Measurement Facility Cases
<dbl> <chr> <dbl>
1 1 Hosp 1 66
2 2 Hosp 1 26
3 3 Hosp 1 8
4 1 Hosp 2 71
5 2 Hosp 2 62
6 3 Hosp 2 70
7 1 Hosp 3 47
8 2 Hosp 3 70
9 3 Hosp 3 38df2 <-
tibble::tribble(
~Year, ~Measurement, ~Facility, ~Cases,
2000, 1, "Hosp 4", 82,
2001, 2, "Hosp 4", 87,
2002, 3, "Hosp 4", 46
)
df2# A tibble: 3 × 4
Year Measurement Facility Cases
<dbl> <dbl> <chr> <dbl>
1 2000 1 Hosp 4 82
2 2001 2 Hosp 4 87
3 2002 3 Hosp 4 46Lorsque nous effectuons une liaison avec bind_rows() pour joindre les deux ensembles de données ensemble, la variable Year est remplie avec de NA pour les lignes où il n’y avait pas d’information préalable (c’est-à-dire le premier ensemble de données) :
df_combined <-
bind_rows(df1, df2) %>%
arrange(Measurement, Facility)
df_combined# A tibble: 12 × 4
Measurement Facility Cases Year
<dbl> <chr> <dbl> <dbl>
1 1 Hosp 1 66 NA
2 1 Hosp 2 71 NA
3 1 Hosp 3 47 NA
4 1 Hosp 4 82 2000
5 2 Hosp 1 26 NA
6 2 Hosp 2 62 NA
7 2 Hosp 3 70 NA
8 2 Hosp 4 87 2001
9 3 Hosp 1 8 NA
10 3 Hosp 2 70 NA
11 3 Hosp 3 38 NA
12 3 Hosp 4 46 2002fill()
Dans ce cas, Year est une variable utile à inclure si nous souhaitons explorer les tendances temporelle. Nous pouvons utiliser fill() pour remplir les cellules vides, en spécifiant la colonne à remplir et la direction (dans ce cas up) :
df_combined %>%
fill(Year, .direction = "up")# A tibble: 12 × 4
Measurement Facility Cases Year
<dbl> <chr> <dbl> <dbl>
1 1 Hosp 1 66 2000
2 1 Hosp 2 71 2000
3 1 Hosp 3 47 2000
4 1 Hosp 4 82 2000
5 2 Hosp 1 26 2001
6 2 Hosp 2 62 2001
7 2 Hosp 3 70 2001
8 2 Hosp 4 87 2001
9 3 Hosp 1 8 2002
10 3 Hosp 2 70 2002
11 3 Hosp 3 38 2002
12 3 Hosp 4 46 2002Alternativement, nous pouvons réordonner les données pour remplir vers le bas :
df_combined <-
df_combined %>%
arrange(Measurement, desc(Facility))
df_combined# A tibble: 12 × 4
Measurement Facility Cases Year
<dbl> <chr> <dbl> <dbl>
1 1 Hosp 4 82 2000
2 1 Hosp 3 47 NA
3 1 Hosp 2 71 NA
4 1 Hosp 1 66 NA
5 2 Hosp 4 87 2001
6 2 Hosp 3 70 NA
7 2 Hosp 2 62 NA
8 2 Hosp 1 26 NA
9 3 Hosp 4 46 2002
10 3 Hosp 3 38 NA
11 3 Hosp 2 70 NA
12 3 Hosp 1 8 NAdf_combined <-
df_combined %>%
fill(Year, .direction = "down")
df_combined# A tibble: 12 × 4
Measurement Facility Cases Year
<dbl> <chr> <dbl> <dbl>
1 1 Hosp 4 82 2000
2 1 Hosp 3 47 2000
3 1 Hosp 2 71 2000
4 1 Hosp 1 66 2000
5 2 Hosp 4 87 2001
6 2 Hosp 3 70 2001
7 2 Hosp 2 62 2001
8 2 Hosp 1 26 2001
9 3 Hosp 4 46 2002
10 3 Hosp 3 38 2002
11 3 Hosp 2 70 2002
12 3 Hosp 1 8 2002Nous avons à présent un jeu de données facilement visualisable à l’aide de ggplot2 :
Mais ce jeu de données est peu lisible si présenté tel quel dans un rapport. Nous pouvons appliquer pivot_larger() pour le transformer en un format plus large :
df_combined %>%
pivot_wider(
id_cols = c(Measurement, Facility),
names_from = "Year",
values_from = "Cases"
) %>%
arrange(Facility) %>%
janitor::adorn_totals(c("row", "col")) %>%
knitr::kable() %>%
kableExtra::row_spec(row = 5, bold = TRUE) %>%
kableExtra::column_spec(column = 5, bold = TRUE) | Measurement | Facility | 2000 | 2001 | 2002 | Total |
|---|---|---|---|---|---|
| 1 | Hosp 1 | 66 | NA | NA | 66 |
| 2 | Hosp 1 | NA | 26 | NA | 26 |
| 3 | Hosp 1 | NA | NA | 8 | 8 |
| 1 | Hosp 2 | 71 | NA | NA | 71 |
| 2 | Hosp 2 | NA | 62 | NA | 62 |
| 3 | Hosp 2 | NA | NA | 70 | 70 |
| 1 | Hosp 3 | 47 | NA | NA | 47 |
| 2 | Hosp 3 | NA | 70 | NA | 70 |
| 3 | Hosp 3 | NA | NA | 38 | 38 |
| 1 | Hosp 4 | 82 | NA | NA | 82 |
| 2 | Hosp 4 | NA | 87 | NA | 87 |
| 3 | Hosp 4 | NA | NA | 46 | 46 |
| Total | - | 266 | 245 | 162 | 673 |
Note : dans ce cas, nous avons dû spécifier de n’inclure que les trois variables Facility, Year, et Cases car la variable supplémentaire Measurement interférait avec la création de la table :
df_combined %>%
pivot_wider(
names_from = "Year",
values_from = "Cases"
) %>%
knitr::kable()| Measurement | Facility | 2000 | 2001 | 2002 |
|---|---|---|---|---|
| 1 | Hosp 4 | 82 | NA | NA |
| 1 | Hosp 3 | 47 | NA | NA |
| 1 | Hosp 2 | 71 | NA | NA |
| 1 | Hosp 1 | 66 | NA | NA |
| 2 | Hosp 4 | NA | 87 | NA |
| 2 | Hosp 3 | NA | 70 | NA |
| 2 | Hosp 2 | NA | 62 | NA |
| 2 | Hosp 1 | NA | 26 | NA |
| 3 | Hosp 4 | NA | NA | 46 |
| 3 | Hosp 3 | NA | NA | 38 |
| 3 | Hosp 2 | NA | NA | 70 |
| 3 | Hosp 1 | NA | NA | 8 |
12.5 Resources
Voici un tutoriel utilel