35 Diagramas y gráficos
Esta página cubre el código para producir:
- Diagramas de flujo utilizando DiagrammeR y el lenguaje
DOT - Diagramas aluviales/Diagramas de Sankey
- Calendario de eventos
35.1 Preparación
Cargar paquetes
Este trozo de código muestra la carga de los paquetes necesarios para los análisis. En este manual destacamos p_load() de pacman, que instala el paquete si es necesario y lo carga para su uso. También es posible cargar los paquetes instalados con library() de R base. Consulta la página sobre fundamentos de R para obtener más información sobre los paquetes de R.
pacman::p_load(
DiagrammeR, # para diagramas de flujo
networkD3, # para diagramas aluviales/Sankey
tidyverse) # gestión y visualización de datosImportar datos
La mayor parte del contenido de esta página no requiere unos datos. Sin embargo, en la sección del diagrama de Sankey, utilizaremos la lista de casos de una epidemia de ébola simulada. Si quieres seguir esta parte, clica para descargar linelist “limpio” (como archivo .rds). Importa los datos con la función import() del paquete rio (maneja muchos tipos de archivos como .xlsx, .csv, .rds - consulta la página importación y exportación para más detalles).
# importa linelist
linelist <- import("linelist_cleaned.rds")A continuación se muestran las primeras 50 filas del listado.
35.2 Diagramas de flujo
Se puede utilizar el paquete R DiagrammeR para crear gráficos/gráficos de flujo. Pueden ser estáticos o ajustarse de forma dinámica en función de los cambios en unos datos.
Herramientas
La función grViz() se utiliza para crear un diagrama “Graphviz”. Esta función acepta una cadena de caracteres de entrada que contiene las instrucciones para hacer el diagrama. Dentro de esa cadena, las instrucciones están escritas en un lenguaje diferente, llamado DOT - es bastante fácil aprender lo básico.
Estructura básica
- Abre las instrucciones
grViz(" - Especifica la dirección y el nombre del gráfico, y abre los paréntesis, por ejemplo,
digraph my_flow_chart { - Define los elementos del gráfico (layout, rank direction)
- Establece los nodos (create nodes)
- Establece las conexiones entre nodos
- Cierra las instrucciones
}")
Ejemplos sencillos
A continuación, dos sencillos ejemplos
Un ejemplo mínimo:
# Un gráfico mínimo
DiagrammeR::grViz("digraph {
graph[layout = dot, rankdir = LR]
a
b
c
a -> b -> c
}")Un ejemplo con un contexto de salud pública quizás más aplicado:
grViz(" # Todas las instrucciones están dentro de una cadena de caracteres grandes
digraph surveillance_diagram { # 'digraph' significa 'gráfico direccional', luego el nombre del gráfico
# enunciado del gráfico
#######################
graph [layout = dot,
rankdir = TB,
overlap = true,
fontsize = 10]
# nodos
#######
node [shape = circle, # forma = círculo
fixedsize = true
width = 1.3] # anchura de los círculos
Primary # nombres de los nodos
Secondary
Tertiary
# Bordes
#######
Primary -> Secondary [label = ' case transfer']
Secondary -> Tertiary [label = ' case transfer']
}
")Sintaxis
Sintaxis básica
Los nombres de los nodos, o las etiquetas de las conexiones (edges), pueden separarse con espacios, punto y coma o nuevas líneas.
Dirección del rango
Se puede reorientar un gráfico para que se mueva de izquierda a derecha ajustando el argumento rankdir dentro de la sentencia del gráfico. El valor predeterminado es TB (top-bottom, de arriba a abajo), pero puede ser LR (Left-Right, de izquierda a derecha), RL o BT.
Nombres de los nodos
Los nombres de los nodos pueden ser palabras sueltas, como en el sencillo ejemplo anterior. Para utilizar nombres con varias palabras o caracteres especiales (por ejemplo, paréntesis, guiones), pon el nombre del nodo entre comillas simples (’ ’). Puede ser más fácil tener un nombre de nodo corto, y asignar una etiqueta como se muestra a continuación entre corchetes [ ]. Si quieres tener una nueva línea dentro del nombre del nodo, debes hacerlo a través de una etiqueta - utiliza \n en la etiqueta del nodo entre comillas simples, como se muestra a continuación.
Subgrupos
Al definir las conexiones (aristas), se pueden crear subgrupos a ambos lados de la arista con corchetes ({ }). La arista se aplica entonces a todos los nodos en el corchete - es una forma abreviada.
Diseños
- dot (establecer
rankdirentre TB, LR, RL, BT, ) - neato
- twopi
- circo
Nodos - atributos editables
label(texto, entre comillas simples si es de varias palabras)
fillcolor(muchos colores posibles)
fontcolor(color de la fuente)alpha(transparencia 0-1)
shape(ellipse, oval, diamond, egg, plaintext, point, square, triangle)
style(estilo)sides(lados)peripheries(periferia)fixedsize(h x w) (tamaño fijo (alto x ancho))height(alto)width(ancho)distortion(dstorsión)penwidth(ancho del borde de la forma)
x(left/right) (desplazamiento a la izquierda/derecha)y(up/down) (desplazamiento arriba/abajo)fontname(nombre de la fuente)fontsize(tamaño de letra)icon
Conexioness - atributos editables
arrowsize(tamaño de la flecha)arrowhead(normal, box, crow, curve, diamond, dot, inv, none, tee, vee)
arrowtail(cola de flecha)dir(dirección, )
style(guiones, …)
color
alpha
headport(texto delante de la punta de la flecha)
tailport(texto detrás de la cola de flecha)fontname(nombre de la fuente)fontsize(tamaño de letra)fontcolor(color de la fuente)penwidth(anchura de la flecha)
minlen(longitud mínima)
Nombres de los colores: valores hexadecimales o nombres de colores “X11”, véase aquí para los detalles de X11
Ejemplos complejos
El siguiente ejemplo amplía el surveillance_diagram, añadiendo nombres de nodos complejos, conexiones agrupadas, colores y estilos
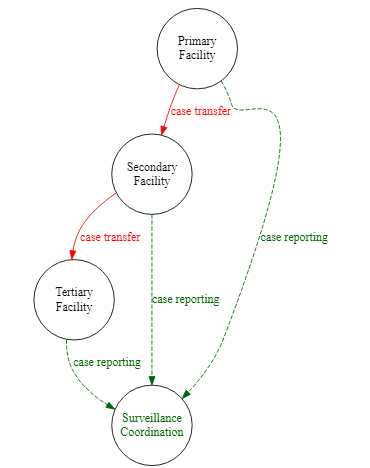
DiagrammeR::grViz(" # Todas las instrucciones están dentro de una cadena de caracteres grandes
digraph surveillance_diagram { # 'digraph' significa 'gráfico direccional', luego el nombre del gráfico
# enunciado del gráfico
#######################
graph [layout = dot,
rankdir = TB, # disposición de arriba abajo
fontsize = 10]
# nodes (circles)
#################
node [shape = circle, # forma = círculo
fixedsize = true
width = 1.3]
Primary [label = 'Primary\nFacility']
Secondary [label = 'Secondary\nFacility']
Tertiary [label = 'Tertiary\nFacility']
SC [label = 'Surveillance\nCoordination',
fontcolor = darkgreen]
# Bordes
#######
Primary -> Secondary [label = ' case transfer',
fontcolor = red,
color = red]
Secondary -> Tertiary [label = ' case transfer',
fontcolor = red,
color = red]
# Bordes agrupados
{Primary Secondary Tertiary} -> SC [label = 'case reporting',
fontcolor = darkgreen,
color = darkgreen,
style = dashed]
}
")Agrupaciones de subgráficos
Para agrupar los nodos en clústeres de cajas, ponlos dentro del mismo subgrafo (subgraph name {}). Para que cada subgrafo se identifique dentro de una caja delimitadora, comienza el nombre del subgrafo con “cluster”, como se muestra con las 4 cajas de abajo.
DiagrammeR::grViz(" # Todas las instrucciones están dentro de una cadena de caracteres grandes
digraph surveillance_diagram { # 'digraph' significa 'gráfico direccional', luego el nombre del gráfico
# enunciado del gráfico
#######################
graph [layout = dot,
rankdir = TB,
overlap = true,
fontsize = 10]
# nodos (círculos)
###################
node [shape = circle, # forma = círculo
fixedsize = true
width = 1.3] # anchura de los círculos
subgraph cluster_passive {
Primary [label = 'Primary\nFacility']
Secondary [label = 'Secondary\nFacility']
Tertiary [label = 'Tertiary\nFacility']
SC [label = 'Surveillance\nCoordination',
fontcolor = darkgreen]
}
# nodos (cajas)
###############
node [shape = box, # forma del nodo
fontname = Helvetica] # fuente de texto del nodo
subgraph cluster_active {
Active [label = 'Active\nSurveillance']
HCF_active [label = 'HCF\nActive Search']
}
subgraph cluster_EBD {
EBS [label = 'Event-Based\nSurveillance (EBS)']
'Social Media'
Radio
}
subgraph cluster_CBS {
CBS [label = 'Community-Based\nSurveillance (CBS)']
RECOs
}
# Bordes
#######
{Primary Secondary Tertiary} -> SC [label = 'case reporting']
Primary -> Secondary [label = 'case transfer',
fontcolor = red]
Secondary -> Tertiary [label = 'case transfer',
fontcolor = red]
HCF_active -> Active
{'Social Media' Radio} -> EBS
RECOs -> CBS
}
")
Formas de los nodos
El siguiente ejemplo, tomado de este tutorial, muestra las formas de los nodos aplicados y una abreviatura de las conexiones de los bordes en serie
DiagrammeR::grViz("digraph {
graph [layout = dot, rankdir = LR]
# define los estilos globales de los nodos. Podemos anular estos en la caja si lo deseamos
node [shape = rectangle, style = filled, fillcolor = Linen]
data1 [label = 'Dataset 1', shape = folder, fillcolor = Beige]
data2 [label = 'Dataset 2', shape = folder, fillcolor = Beige]
process [label = 'Process \n Data']
statistical [label = 'Statistical \n Analysis']
results [label= 'Results']
# definiciones de las bordes con los ID de los nodos
{data1 data2} -> process -> statistical -> results
}")Salidas
Cómo manejar y guardar las salidas
- Las salidas aparecerán en el panel del Visor de RStudio, por defecto en la parte inferior derecha junto a Files, Plots, Packages, y Help.
- Para exportarlos puedes “Save as image” o “Copy to clipboard” desde el Visor. El gráfico se ajustará al tamaño especificado.
Figuras parametrizadas
Esta es una cita a este tutorial: https://mikeyharper.uk/flowcharts-in-r-using-diagrammer/
“Figuras parametrizadas”: Una gran ventaja de diseñar figuras dentro de R es que podemos conectar las figuras directamente con nuestro análisis leyendo los valores de R directamente en nuestros diagramas de flujo. Por ejemplo, imagina que has creado un proceso de filtrado que elimina valores después de cada etapa de un proceso, puedes hacer que una figura muestre el número de valores que quedan en el conjunto de datos después de cada etapa de su proceso. Para hacer esto, puedes utilizar el símbolo @@X directamente dentro de la figura, y luego hacer referencia a esto en el pie de página del gráfico utilizando [X]:, donde X es el índice numérico único”.
Te animamos a revisar este tutorial si te interesa la parametrización.
35.3 Diagramas Aluviales/Sankey
Cargar paquetes
Este trozo de código muestra la carga de los paquetes necesarios para los análisis. En este manual destacamos p_load() de pacman, que instala el paquete si es necesario y lo carga para su uso. También puedes cargar los paquetes instalados con library() de R base. Consulta la página sobre fundamentos de R para obtener más información sobre los paquetes de R.
Cargamos el paquete networkD3 para producir el diagrama, y también tidyverse para los pasos de preparación de datos.
pacman::p_load(
networkD3,
tidyverse)Trazado desde los datos
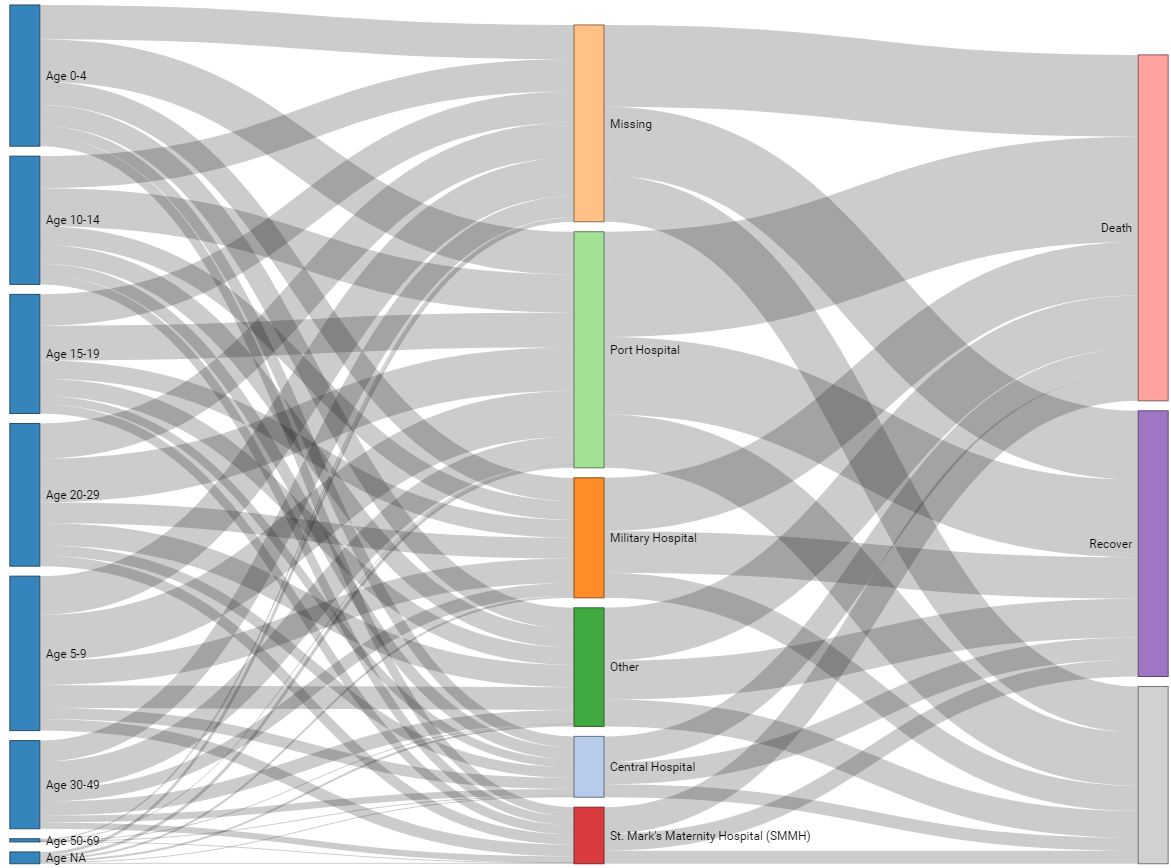
Trazado de las conexiones en unos datos. A continuación mostramos el uso de este paquete con linelist Aquí hay un tutorial en línea.
Comenzamos obteniendo los recuentos de casos para cada combinación única de categoría de edad y hospital. Hemos eliminado los valores con categoría de edad ausente para mayor claridad. También reetiquetamos las columnas hospital y age_cat como source y target respectivamente. Estos serán los dos lados del diagrama aluvial.
# recuentos por hospital y categoría de edad
links <- linelist %>%
drop_na(age_cat) %>%
select(hospital, age_cat) %>%
count(hospital, age_cat) %>%
rename(source = hospital,
target = age_cat)El conjunto de datos tiene ahora este aspecto:
Ahora creamos un dataframe de todos los nodos del diagrama, bajo la columna name. Esto consiste en todos los valores de hospital y age_cat. Observa que nos aseguramos de que todos son de tipo carácter antes de combinarlos. Ajustamos las columnas ID para que sean números en lugar de etiquetas:
# Nombres únicos de los nodos
nodes <- data.frame(
name=c(as.character(links$source), as.character(links$target)) %>%
unique()
)
nodes # imprime name
1 Central Hospital
2 Military Hospital
3 Missing
4 Other
5 Port Hospital
6 St. Mark's Maternity Hospital (SMMH)
7 0-4
8 5-9
9 10-14
10 15-19
11 20-29
12 30-49
13 50-69
14 70+A continuación editamos el dataframe links, que hemos creado anteriormente con count(). Añadimos dos columnas numéricas IDsource e IDtarget que reflejarán/crearán los enlaces entre los nodos. Estas columnas contendrán los números de ruta (posición) de los nodos de origen y destino. Se resta 1 para que estos números de posición comiencen en 0 (no en 1).
# coincide con números, no con nombres
links$IDsource <- match(links$source, nodes$name)-1
links$IDtarget <- match(links$target, nodes$name)-1El conjunto de datos links tiene ahora este aspecto:
Ahora traza el diagrama Sankey con sankeyNetwork(). Puedes leer más sobre cada argumento ejecutando ?sankeyNetwork en la consola. Ten en cuenta que a menos que establezcas iterations = 0 el orden de los nodos puede no ser el esperado.
# gráfico
#########
p <- sankeyNetwork(
Links = links,
Nodes = nodes,
Source = "IDsource",
Target = "IDtarget",
Value = "n",
NodeID = "name",
units = "TWh",
fontSize = 12,
nodeWidth = 30,
iterations = 0) # asegura que el orden de los nodos es como en los datos
pEste es un ejemplo en el que también se incluye el resultado del paciente. Obsérva que en el paso de preparación de los datos tenemos que calcular los recuentos de casos entre la edad y el hospital, y por separado entre el hospital y el resultado - y luego unir todos estos recuentos con bind_rows().
# recuentos por hospital y categoría de edad
age_hosp_links <- linelist %>%
drop_na(age_cat) %>%
select(hospital, age_cat) %>%
count(hospital, age_cat) %>%
rename(source = age_cat, # renombra
target = hospital)
hosp_out_links <- linelist %>%
drop_na(age_cat) %>%
select(hospital, outcome) %>%
count(hospital, outcome) %>%
rename(source = hospital, # renombra
target = outcome)
# combina las conexiones
links <- bind_rows(age_hosp_links, hosp_out_links)
# Nombres únicos de los nodos
nodes <- data.frame(
name=c(as.character(links$source), as.character(links$target)) %>%
unique()
)
# Crear números de identificación
links$IDsource <- match(links$source, nodes$name)-1
links$IDtarget <- match(links$target, nodes$name)-1
# gráfico
#########
p <- sankeyNetwork(Links = links,
Nodes = nodes,
Source = "IDsource",
Target = "IDtarget",
Value = "n",
NodeID = "name",
units = "TWh",
fontSize = 12,
nodeWidth = 30,
iterations = 0)
phttps://www.displayr.com/sankey-diagrams-r/
35.4 Calendario de eventos
Para hacer una línea de tiempo que muestre eventos específicos, puedes utilizar el paquete vistime.
Mira esta viñeta
# cargar paquetes
pacman::p_load(vistime, # hacer la línea de tiempo
plotly # para visualización interactiva
)Este es el conjunto de datos de eventos con el que comenzamos:
p <- vistime(data) # aplica vistime
library(plotly)
# paso 1: transformar en una lista
pp <- plotly_build(p)
# paso 2: tamaño del marcador
for(i in 1:length(pp$x$data)){
if(pp$x$data[[i]]$mode == "markers") pp$x$data[[i]]$marker$size <- 10
}
# paso 3: tamaño del texto
for(i in 1:length(pp$x$data)){
if(pp$x$data[[i]]$mode == "text") pp$x$data[[i]]$textfont$size <- 10
}
# paso 4: posición del texto
for(i in 1:length(pp$x$data)){
if(pp$x$data[[i]]$mode == "text") pp$x$data[[i]]$textposition <- "right"
}
#imprimir
pp35.5 DAGs
Puedes construir un DAG manualmente utilizando el paquete DiagammeR y el lenguaje DOT como se ha descrito anteriormente.
Como alternativa, existen paquetes como ggdag y daggity
35.6 Recursos
Gran parte de lo anterior sobre el lenguaje DOT está adaptado del tutorial de este sitio
Otro tutorial más detallado sobre DiagammeR
Esta página sobre los diagramas de Sankey