41 Organização de relatórios de rotina
Esta página cobre o pacote reportfactory, que é um acompanhamento do uso do R Markdown para relatórios.
Em cenários onde você executa relatórios rotineiramente (diariamente, semanalmente, etc.), isso facilita a compilação de vários arquivos R Markdown e a organização de seus resultados. Em essência, ele fornece uma “fábrica” a partir da qual você pode executar os relatórios R Markdown, obter pastas com carimbo de data e hora automaticamente para as saídas e ter controle de versão “leve”.
reportfactory é um dos pacotes desenvolvidos pelo RECON (R Epidemics Consortium). Aqui está o site e Github.
41.1 Preparação
Carregar pacotes
De dentro do RStudio, instale a versão mais recente do pacote reportfactory do Github.
Você pode fazer isso através do pacote pacman com p_load_current_gh () que forçará a instalação da última versão do Github. Forneça a cadeia de caracteres “reconverse/reportfactory”, que especifica a organização Github (reconverse) e o repositório (reportfactory). Você também pode usar install_github () do pacote remotes, como alternativa.
# Instale e carregue a última versão do pacote do Github
pacman::p_load_current_gh("reconverse/reportfactory")
#remotes::install_github("reconverse/reportfactory") # alternative41.2 Nova fábrica
Para criar uma nova fábrica, execute a função new_factory(). Isso criará uma nova pasta de projeto R independente. Por padrão:
- A fábrica será adicionada ao seu diretório de trabalho
- O nome do projeto de fábrica R será denominado “new_factory.Rproj”
- Sua sessão RStudio irá “mover-se” para este projeto R
# Isso criará a fábrica no diretório de trabalho
new_factory()Olhando dentro da fábrica, você pode ver que as subpastas e alguns arquivos foram criados automaticamente.
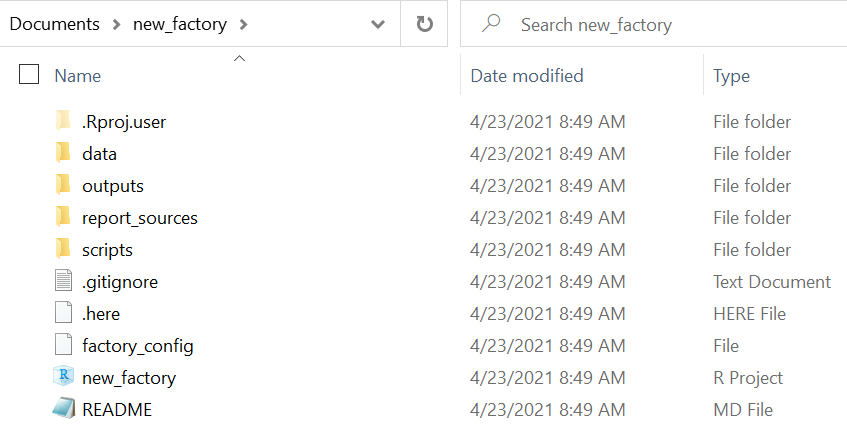
- A pasta report_sources manterá seus scripts R Markdown, que geram seus relatórios
- A pasta * outputs * conterá os resultados do relatório (por exemplo, HTML, Word, PDF, etc.)
- A pasta scripts pode ser usada para armazenar outros scripts R (por exemplo, que são fornecidos por seus scripts Rmd)
- A pasta data pode ser usada para armazenar seus dados (subpastas “brutas” e “limpas” estão incluídas)
- Um arquivo .here, para que você possa usar o pacote here para chamar arquivos em subpastas por sua relação com esta pasta raiz (consulte a página Projetos R para obter detalhes)
- Um arquivo gitignore foi criado no caso de você vincular este projeto R a um repositório Github (consulte Controle de versão e colaboração com Github)
- Um arquivo README vazio, se você usar um repositório Github
**_ CUIDADO: _** dependendo da configuração do seu computador, arquivos como “.here” podem existir, mas são invisíveis.
Das configurações padrão, abaixo estão várias que você pode querer ajustar dentro do comando new_factory ():
-
factory =- Fornece um nome para a pasta de fábrica (o padrão é “new_factory”)
-
path =- Designa um caminho de arquivo para a nova fábrica (o padrão é o diretório de trabalho)
-
report_sources =Fornece um nome alternativo para a subpasta que contém os scripts R Markdown (o padrão é “report_sources”)
-
outputs =Fornece um nome alternativo para a pasta que contém os resultados do relatório (o padrão é “outputs”)
Veja ?New_factory para uma lista completa dos argumentos.
Quando você cria a nova fábrica, sua sessão R é transferida para o novo projeto R, então você deve carregar novamente o pacote reportfactory.
pacman::p_load(reportfactory)Agora você pode executar o comando factory_overview() para ver a estrutura interna (todas as pastas e arquivos) na fábrica.
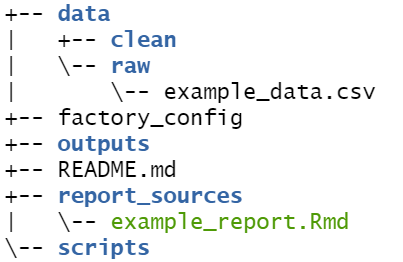
factory_overview() # imprime visão geral da fábrica para o consoleA seguinte “árvore” das pastas e arquivos da fábrica é impressa no console R. Observe que na pasta “dados” existem subpastas para dados “brutos” e “limpos” e dados CSV de exemplo. Também existe “example_report.Rmd” na pasta “report_sources”.
41.3 Crie um relatório
De dentro do projeto R de fábrica, crie um relatório R Markdown como faria normalmente e salve-o na pasta “report_sources”. Consulte a página R Markdown para obter instruções. Para fins de exemplo, adicionamos o seguinte à fábrica:
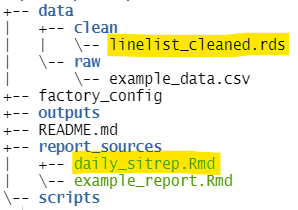
- Um novo script de markdown R intitulado “daily_sitrep.Rmd”, salvo na pasta “report_sources”
- Dados para o relatório (“linelist_cleaned.rds”), salvos na subpasta “clean” dentro da pasta “data”
Podemos ver usando factory_overview() nosso R Markdown na pasta “report_sources” e o arquivo de dados na pasta de dados “clean” (destacado):
Abaixo está uma captura de tela do início do R Markdown “daily_sitrep.Rmd”. Você pode ver que o formato de saída é definido como HTML, por meio do cabeçalho YAML output: html_document.
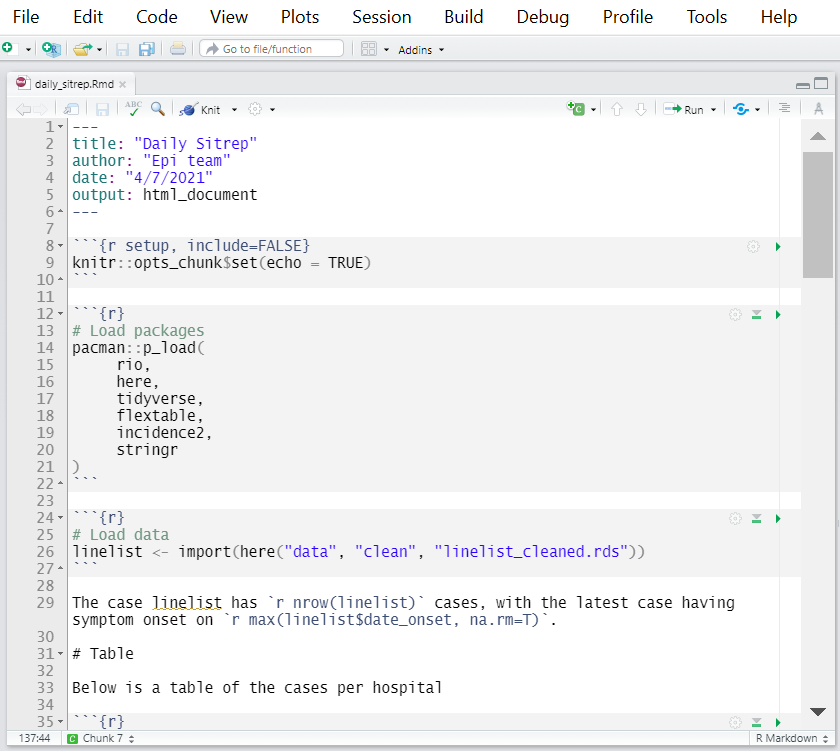
Neste script simples, existem comandos para:
- Carregar os pacotes necessários
- Importar os dados de lista de linha usando um caminho de arquivo do pacote here (leia mais na página em Importar e exportar)
- Imprima uma tabela de resumo de casos e exporte-a com
export()como um arquivo .csv
- Imprima uma epicurva e exporte-a com
ggsave()como um arquivo .png
Você pode revisar apenas a lista de relatórios R Markdown na pasta “report_sources” com este comando:
list_reports()41.4 Compilar
Em uma fábrica de relatórios, “compilar” um relatório R Markdown significa que o script .Rmd será executado e a saída será produzida (conforme especificado no script YAML, por exemplo, como HTML, Word, PDF, etc).
A fábrica criará automaticamente uma pasta com carimbo de data e hora para as saídas na pasta “saídas”.
O próprio relatório e todos os arquivos exportados produzidos pelo script (por exemplo, csv, png, xlsx) serão salvos nesta pasta. Além disso, o próprio script Rmd será salvo nesta pasta, então você tem um registro dessa versão do script.
Isso contrasta com o comportamento normal de um R Markdown “knitado” ou “tricotado”, que salva as saídas no local do script Rmd. Esse comportamento padrão pode resultar em pastas lotadas e confusas. A fábrica visa melhorar a organização quando é necessário executar relatórios com frequência.
Compilar por nome
Você pode compilar um relatório específico executando compile_reports() e fornecendo o nome do script Rmd (sem extensão .Rmd) para reports =. Para simplificar, você pode pular o reports = e apenas escrever o nome R Markdown entre aspas, como abaixo.

Este comando compilaria apenas o relatório “daily_sitrep.Rmd”, salvando o relatório HTML e as exportações da tabela .csv e epicurva .png para uma subpasta com carimbo de data e hora específica para o relatório, dentro da pasta “outputs”.
Observe que se você optar por fornecer a extensão .Rmd, deverá digitar corretamente a extensão conforme ela é salva no nome do arquivo (.rmd vs. .Rmd).
Observe também que, ao compilar, você pode ver vários arquivos aparecerem temporariamente na pasta “report_sources” - mas eles irão desaparecer em breve, pois são transferidos para a pasta “outputs” correta.
Compilar por número
Você também pode especificar o script Rmd para compilar, fornecendo um número ou vetor de números para relatórios =. Os números devem estar alinhados com a ordem em que os relatórios aparecem quando você executa list_reports().
# Compile o segundo e o quarto Rmds na pasta "report_sources"
compile_reports(reports = c(2, 4))Compilar todos
Você pode compilar todos os relatórios R Markdown na pasta “report_sources” definindo o argumento reports = para TRUE.

Compilar da subpasta
Você pode adicionar subpastas à pasta “report_sources”. Para executar um relatório R Markdown de uma subpasta, simplesmente forneça o nome da pasta para subpasta =. Abaixo está um exemplo de código para compilar um relatório Rmd que reside em uma subpasta de “report_sources”.
compile_reports(
reports = "summary_for_partners.Rmd",
subfolder = "for_partners")Você pode compilar todos os relatórios Rmd dentro de uma subpasta fornecendo o nome da subpasta para reports =, com uma barra no final, como abaixo.
compile_reports(reports = "for_partners/")Parametrização
Conforme observado na página em Relatórios com R Markdown, você pode executar relatórios com parâmetros especificados. Você pode passar esses parâmetros como uma lista para compile_reports() através do argumento params =. Por exemplo, neste relatório fictício, há três parâmetros fornecidos para os relatórios R Markdown.
compile_reports(
reports = "daily_sitrep.Rmd",
params = list(most_recent_data = TRUE,
region = "NORTHERN",
rates_denominator = 10000),
subfolder = "regional"
)Usando um “arquivo de execução”
Se você tiver vários relatórios para executar, considere a criação de um script R que contenha todos os comandos de compile_reports (). Um usuário pode simplesmente executar todos os comandos neste script R e todos os relatórios serão compilados. Você pode salvar este “arquivo de execução” na pasta “scripts”.
41.5 Saídas
Depois de termos compilado os relatórios algumas vezes, a pasta “outputs” pode ter a seguinte aparência (destaques adicionados para maior clareza):
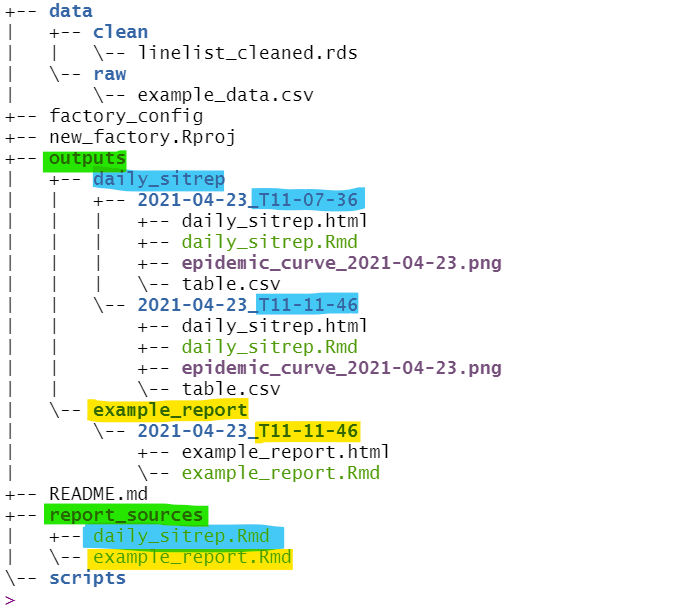
- Dentro de “saídas”, subpastas foram criadas para cada relatório Rmd
- Dentro delas, outras subpastas foram criadas para cada compilação única
- Estes são marcados com data e hora (“2021-04-23_T11-07-36” significa 23 de abril de 2021 às 11:07:36)
- Você pode editar o formato do carimbo de data / hora. Veja
?Compile_reports
- Estes são marcados com data e hora (“2021-04-23_T11-07-36” significa 23 de abril de 2021 às 11:07:36)
- Dentro de cada pasta compilada de data / hora, a saída do relatório é armazenada (por exemplo, HTML, PDF, Word) junto com o script Rmd (controle de versão!) e quaisquer outros arquivos exportados (por exemplo, table.csv, epidemic_curve.png)
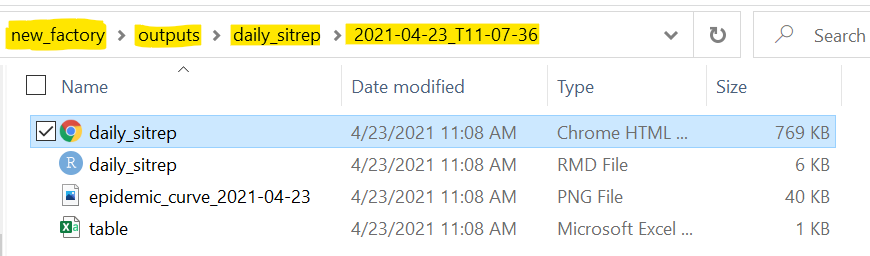
Aqui está uma visão dentro de uma das pastas com carimbo de data / hora, para o relatório “daily_sitrep”. O caminho do arquivo é destacado em amarelo para ênfase.
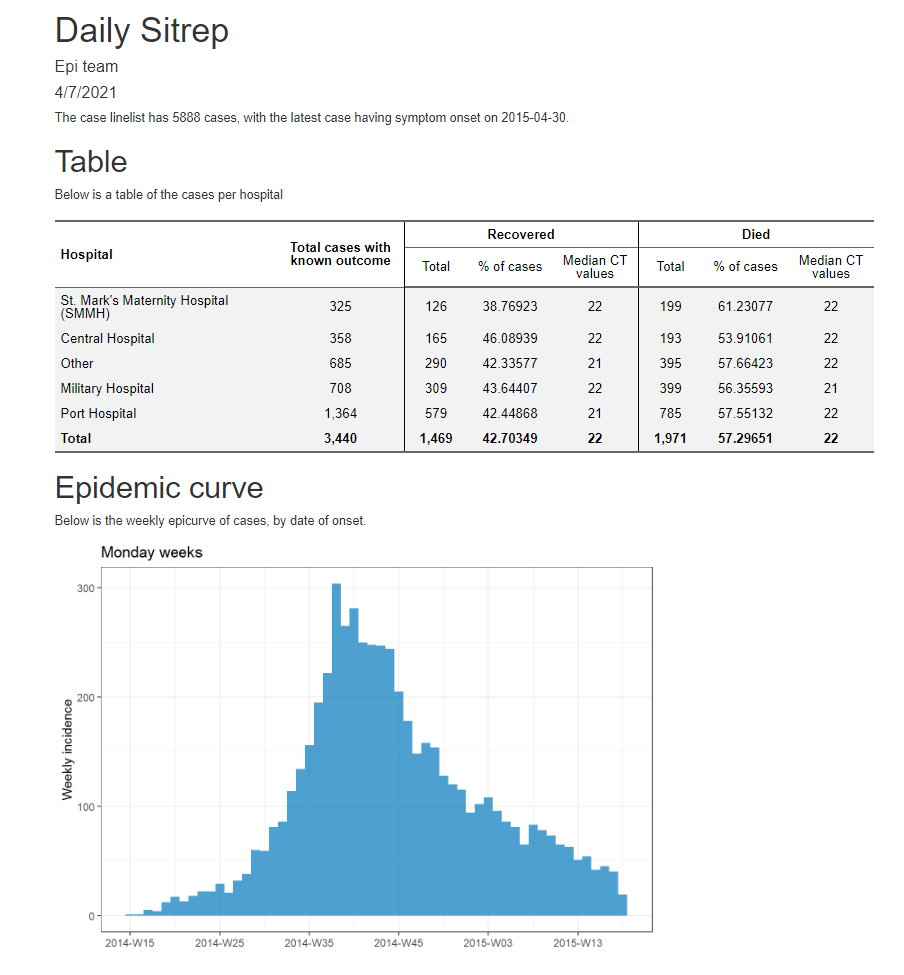
Finalmente, abaixo está uma captura de tela da saída do relatório HTML.
Você pode usar list_outputs() para revisar uma lista das saídas.
41.6 Diversos
41.6.1 Knit
Você ainda pode “tricotar” um de seus relatórios R Markdown pressionando o botão “Knit”, se desejar. Se você fizer isso, como padrão, as saídas aparecerão na pasta onde o Rmd foi salvo - a pasta “report_sources”. Nas versões anteriores do reportfactory, ter qualquer arquivo não-Rmd em “report_sources” impedia a compilação, mas não é mais o caso. Você pode executar compile_reports() e nenhum erro ocorrerá.
Scripts
Incentivamos você a utilizar a pasta “scripts” para armazenar “arquivos de execução” ou scripts .R originados de seus scripts .Rmd. Consulte a página em R Markdown para dicas sobre como estruturar seu código em vários arquivos.
Extras
Com reportfactory, você pode usar a função
list_deps()para listar todos os pacotes necessários em todos os relatórios em toda a fábrica.-
Há um pacote de acompanhamento em desenvolvimento chamado rfextras que oferece mais funções auxiliares para auxiliá-lo na construção de relatórios, como:
-
load_scripts()- origina / carrega todos os scripts .R em uma determinada pasta (a pasta “scripts” por padrão)
-
find_latest()- encontra a versão mais recente de um arquivo (por exemplo, o conjunto de dados mais recente)
-