# 最新版の Epi R Handbook パッケージをインストールする
pacman::p_install_gh("appliedepi/epirhandbook")2 ハンドブックとデータのダウンロード
2.1 オフライン版のダウンロード
このハンドブックのオフライン版を HTML ファイルとしてダウンロードすることで、インターネットに接続できない場合でも、Web ブラウザでファイルを閲覧することができます。オフラインでの利用を検討されている方は、以下の点にご注意ください。
- ファイルを開いた際、画像と目次の読み込みに 1 ~ 2 分かかる場合があります。
- オフラインのハンドブックは、1 ページが非常に長く、左側に目次があるなど、レイアウトが少し異なります。特定の用語を検索する際は、Ctrl + f (Cmd - f) を使用してください。
- インターネットに接続できなくなる前に、適切な R パッケージをインストールするため、推奨するパッケージ の章をご参照ください。
- すべてのサンプルデータを含む R パッケージ epirhandbook をインストールしてください(インストール方法は以下の通りです)。
ハンドブックをダウンロードする方法は2つあります。
ダウンロードリンクの利用
こちらの リンク を右クリックし、「リンク先を別名で保存(Save link as)」を選択すると、素早くダウンロードすることができます。
Mac をお使いの場合は、Cmd を押しながらクリックしてください。モバイルの場合は、リンクを長押しし、「リンクを保存(Save link)」を選択すると、ハンドブックがお使いの端末にダウンロードされます。HTML コードが表示された場合は、上記の手順が正しく行われているか確認する、または次の方法をお試しください。
R パッケージの利用
Epirhandbook という R パッケージがあります。このパッケージには、Github リポジトリからハンドブックファイルをあなたのパソコンにダウンロードするための関数 download_book() が含まれています。
また、このパッケージには、すべてのサンプルデータをダウンロードするための関数 get_data() も含まれています。
次のコードを実行し、Github repository appliedepi から R パッケージ epirhandbook パッケージをインストールします。このパッケージは CRAN にないので、Github からインストールする場合は特別な関数 p_install_gh() を使ってください。
ここで、今開いている R セッションで(これらのデータを)使用するために、パッケージを読み込みます。
# 使用するパッケージを読み込む
pacman::p_load(epirhandbook)次に、epirhandbook パッケージに含まれている関数 download_book() (括弧内は空白)を実行して、ハンドブックをパソコンにダウンロードします。RStudio を使用している場合、保存場所を選択するウィンドウが表示されます。
# オフラインハンドブックをパソコンにダウンロードする
download_book()2.2 サンプルデータのダウンロード
ハンドブックの内容を「実際に体験しながら」学習したい場合、サンプルデータと出力結果をダウンロードすることができます。
R パッケージの利用
最も簡単にすべてのデータをダウンロードする方法は、前述した epirhandbook パッケージをインストールすることです。このパッケージには、すべてのサンプルデータをパソコンの任意のフォルダに保存する関数 get_data() が含まれています。
epirhandbook パッケージをインストールするには、次のコードを実行してください。このパッケージは CRAN にないため、インストールする際には p_install_gh() という関数を使用し、このハンドブックの Github Organization(“appliedepi”)と epirhandbook パッケージを指定してください。
# 最新版の Epi R Handbook パッケージをインストールする
pacman::p_install_gh("appliedepi/epirhandbook")ダウンロード後、今開いている R セッションで、インストールしたパッケージを読み込みます。
# 使用するパッケージを読み込む
pacman::p_load(epirhandbook)次に、epirhandbook パッケージに含まれている関数 get_data() を使って、サンプルデータをパソコンにダウンロードします。get_data("all") を実行するとすべてのサンプルデータがダウンロードできます。特定のファイルのみをダウンロードしたい場合は、引用符の中でファイル名と拡張子を指定してください。
データはすでにパッケージと一緒にダウンロードされているため、あとはパソコンのフォルダに移動させるだけです。ポップアップウィンドウが表示されますので、保存先のフォルダを選択してください。約 30 のファイル(サンプルデータ、サンプルデータの出力結果を含む)ありますので、新規に「data」フォルダを作成することをおすすめします。
# すべてのサンプルデータをパソコンのフォルダにダウンロードする
get_data("all")
# ラインリストのサンプルデータのみをパソコンのフォルダにダウンロードする
get_data(file = "linelist_cleaned.rds")# ある特定のファイルをパソコンのフォルダにダウンロードする
get_data("linelist_cleaned.rds")get_data() を使ってファイルをパソコンに保存した後、それを R にインポートする必要があります。詳細は、データのインポート・エクスポート の章をご参照ください。
また、ご希望の場合は、このハンドブックで使用したすべてのデータを、Github リポジトリの 「data」フォルダ で確認することができます。
1 つずつダウンロードする
このオプションは、Github リポジトリからファイルごとにデータをダウンロードするもので、リンクまたはファイル固有の R コマンドのいずれかを使用します。ファイルの種類によっては、ダウンロードボタンでダウンロードできるものもあれば、R コマンドでダウンロードできるものもあります。
症例ラインリスト
これは、outbreaks パッケージの練習用データセット ebola_sim から本ハンドブックチームによって拡張された架空のエボラ出血熱の発生状況のデータです。
「加工前の」ラインリストのダウンロードはこちら(.xlsx)。「加工前の」症例ラインリストは、雑多なデータを含む Excel のスプレッドシートです。データクリーニングと主要関数 の章を学ぶ際にご利用ください。
「前処理された」ラインリストのダウンロードはこちら(.rds)。このファイルは、このハンドブック内でラインリストを使用するすべての章で使用します。.rds ファイルは、列のデータ型を保持する R 特有のファイル形式です。これにより、R にデータをインポートした後、最小限のクリーニングを行うだけでよくなります。
その他の関連ファイル
データクリーニングの章の一部では、「クリーニングディクショナリ(cleaning dictionary)」(.csv ファイル)を使用しています。次のコマンドを実行すると、R に直接読み込むことができます。
pacman::p_load(rio) # rio パッケージのインストールと読み込み
# Github から直接ファイルをインポートする
cleaning_dict <- import("https://github.com/appliedepi/epiRhandbook_eng/raw/master/data/case_linelists/cleaning_dict.csv")マラリア症例数のデータ
これらのデータは、年齢層別、施設別、日別のマラリア症例数の架空のデータです。.rds ファイルは、列のデータ型を保持する R 特有のファイル形式です。これにより、R にデータをインポートした後、最小限のクリーニングを行うだけでよくなります。
リッカート尺度のデータ
これは、人口ピラミッドとリッカート尺度 の章で使用する、リッカート尺度による架空の調査データです。次のコマンドを実行すると、R に直接読み込むことができます。
pacman::p_load(rio) # rio パッケージのインストールと読み込み
# Github から直接ファイルをインポートする
likert_data <- import("https://raw.githubusercontent.com/nsbatra/Epi_R_handbook/master/data/likert_data.csv")柔軟なダッシュボード
以下は、R Markdownで作るダッシュボード の章に関連するファイルへのリンクです。
接触者の追跡
接触者の追跡 の章では、Go.Data のデータを例に、接触者追跡に関するデータ解析について紹介しました。この章で使用したデータは、次のリンクをクリックすると、.rds ファイルとしてダウンロードすることができます。
感染者調査データのダウンロードはこちら(.rds file)
接触者登録データのダウンロードはこちら(.rds file)
接触者フォローアップのデータのダウンロードはこちら(.rds file)
注釈:他のソフトウェア(KoBo、DHIS2 Tracker、CommCare など)の構造化された接触者の追跡データは、見た目が異なる場合があります。この章の代替サンプルデータやコンテンツをご提供いただける場合は こちら へご連絡ください。
ヒント:Go.Data を展開していて、API に接続したい場合は、データのインポート・エクスポートの章 (API セクション) と Go.Data Community of Practiceをご参照ください。
GIS
シェイプファイル(Shapefiles)には多くのサブファイルがあり、それぞれ異なるファイル拡張子を持っています。あるファイルの拡張子は「.shp」ですが、他のファイルは「.dbf」、「.prj」などの拡張子があります。
GIS の基礎 の章には、Humanitarian Data Exchange ウェブサイトへのリンクがあり、zip ファイルとしてシェイプファイルを直接ダウンロードすることができます。
例えば、医療施設ポイントデータは、こちら からダウンロードできます。「hotosm_sierra_leone_health_facilities_points_shp.zip」をダウンロードし、パソコンに保存したら、フォルダーを「解凍」してください。拡張子が異なる複数のファイル(例:「.shp」、「.prj」、「.shx」)が表示されますので、これらすべてのファイルをパソコンの同じフォルダ内に保存してください。そして、R にインポートするには、sf パッケージの st_read() に 「.shp」 ファイルのパスとファイル名を指定します(GIS の基礎 の章に記載されています)。
先述したオプション 1 の方法に従って epirhandbook パッケージを利用してすべてのサンプルデータをダウンロードする場合、すべてのシェイプファイルが含まれます。
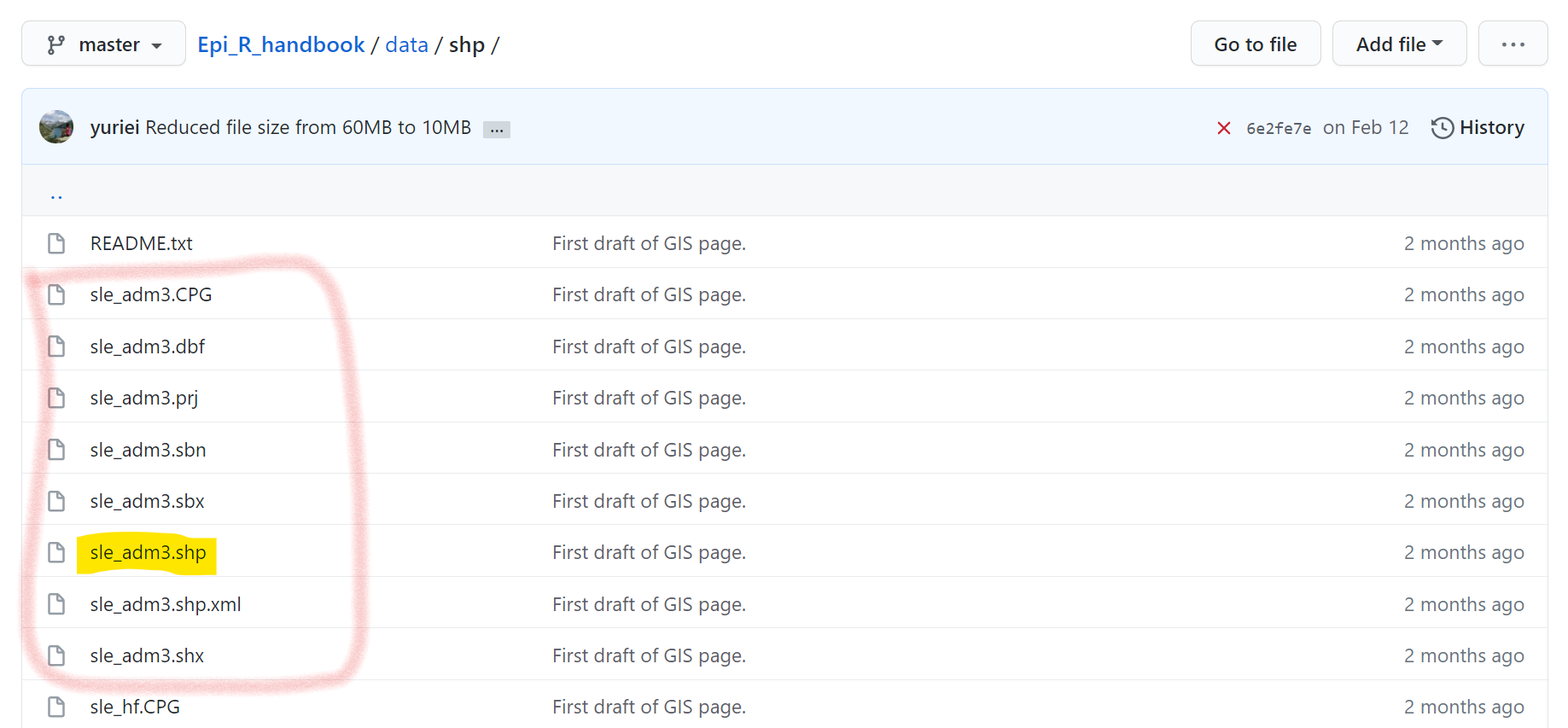
または、R Handbook Github の「data」フォルダ(「gis」 サブフォルダを参照)からシェイプファイルをダウンロードすることも可能です。ただし、各サブファイルを個別にパソコンにダウンロードする必要があることに注意してください。Github で、各ファイルを個別にクリックし、「Download」ボタンをクリックしてダウンロードできます。下の図は、シェイプファイル「sle_adm3」が多くのファイルから構成されており、それぞれを Github からダウンロードする必要があることを示しています。
系統樹
系統樹 の章をご参照ください。299 の Shigella sonnei サンプルの全ゲノムシークエンスから構築した系統樹の Newick ファイルと対応するサンプルデータ(テキストファイルへ変換)です。ベルギーのサンプルとそのデータは、ECDC EUPHEM フェローのプロジェクトの一環として、サルモネラ菌と赤痢菌についてベルギー NRC から提供されたもので、原稿も掲載される予定です。国際的なデータは、公共データベース(ncbi)で公開されており、過去に発表されています。
- 系統樹ファイル「Shigella_tree.txt」をダウンロードするには、この リンク を右クリック(Mac の場合は Cmd を押しながらクリック)し、「リンク先を別名で保存(Save link as)」を選択してください。
- 各サンプルの追加情報を含む「sample_data_Shigella_tree.csv」をダウンロードするには、このリンクを右クリック(Mac の場合は Cmd を押しながらクリック)し、「リンク先を別名で保存(Save link as)」を選択してください。
- 新しく作成されたサブセット・ツリーを見るには、このリンクを右クリック(Mac の場合は Cmd を押しながらクリック)し、「リンク先を別名で保存(Save link as)」を選択してください。.txt ファイルがあなたのパソコンにダウンロードされます。
インストール後、系統樹の章で説明されているとおり、ape パッケージの read.tree() で .txt ファイルを取り込むことができます。
ape::read.tree("Shigella_tree.txt")標準化
標準化率 の章をご参照ください。次のコマンドで、インターネット上の Github リポジトリから直接 R セッションにデータを読み込むことができます。
# rio パッケージのインストールと読み込み
pacman::p_load(rio)
##############
# Country A
##############
# A 国の人口統計データを Github から直接インポートする
A_demo <- import("https://github.com/appliedepi/epiRhandbook_eng/raw/master/data/standardization/country_demographics.csv")
# A 国の死因を Github から直接インポートする
A_deaths <- import("https://github.com/appliedepi/epiRhandbook_eng/raw/master/data/standardization/deaths_countryA.csv")
##############
# Country B
##############
# B 国の人口統計データを Github から直接インポートする
B_demo <- import("https://github.com/appliedepi/epiRhandbook_eng/raw/master/data/standardization/country_demographics_2.csv")
# B 国の死因を Github から直接インポートする
B_deaths <- import("https://github.com/appliedepi/epiRhandbook_eng/raw/master/data/standardization/deaths_countryB.csv")
###############
# Reference Pop
###############
# B 国の人口統計データをGithubから直接インポートする
standard_pop_data <- import("https://github.com/appliedepi/epiRhandbook_eng/raw/master/data/standardization/world_standard_population_by_sex.csv")時系列分析とアウトブレイクの検出
時系列分析とアウトブレイクの検出 の章をご参照ください。surveillance パッケージに含まれている、2002 年から 2011 年までにドイツで報告されたカンピロバクター症例を使用しています(注:このデータセットは、学習のために 2011 年末から 3 ヶ月分のデータを削除し、元のデータから改編したものです)。
ドイツのカンピロバクターのデータのダウンロードはこちら(.xlsx)
また、時系列分析とアウトブレイクの検出の章では、ドイツの 2002 年から 2011 年の気候データ(気温:摂氏、降水量:ミリメートル)も使用します。これらのデータは ecmwfr パッケージを使用して EU Copernicus 衛星再解析データセットからダウンロードしたものです。時系列分析の章で説明されているように、これらのデータをすべてダウンロードし、stars::read_stars() でインポートする必要があります。
ドイツの 2002 年の天気予報のダウンロードはこちら(.nc file)
ドイツの 2003 年の天気予報のダウンロードはこちら(.nc file)
ドイツの 2004 年の天気予報のダウンロードはこちら(.nc file)
ドイツの 2005 年の天気予報のダウンロードはこちら(.nc file)ドイツの 2006 年の天気予報のダウンロードはこちら(.nc file)
ドイツの 2007 年の天気予報のダウンロードはこちら(.nc file)
ドイツの 2008 年の天気予報のダウンロードはこちら(.nc file)
ドイツの 2009 年の天気予報のダウンロードはこちら(.nc file)
標本調査データ分析
標本調査データ分析 の章では、MSF OCA 調査テンプレートをもとにした架空の死亡率調査データを使用しています。この架空のデータは、「R4Epis」プロジェクト の一環として作成されたものです。
Shiny
Shiny で作るダッシュボード の章では、マラリアのデータを表示する簡単なアプリの作成方法を紹介しています。
Shiny アプリを作成する R ファイルのダウンロードは以下の通りです。
Shiny アプリの UI とサーバーのコードを含む app.R ファイルのダウンロードはこちら
Shiny アプリ用のマラリアデータを含む facility_count_data.rds ファイルのダウンロードはこちら。なお、here() のファイルパスが正しく動作するように、「data」フォルダ内に保存しなければならない場合があります。
章内で説明されている、アプリを開く前に実行すべき global.R ファイルのダウンロードはこちら
global.R がもととなっている plot_epicurve.R ファイルのダウンロードはこちら なお、here() のファイルパスが正しく動作するために、「funcs」フォルダ内に保存しなければならない場合があります。